扩展功能
文章信息
- 牛世全, 龙洋, 李海云, 达文燕, 胡山, 李渭娟, 朱学泰, 孔维宝
- NIU Shi-Quan, LONG Yang, LI Hai-Yun, DA Wen-Yan, HU Shan, LI Wei-Juan, ZHU Xue-Tai, KONG Wei-Bao
- 应用Illumina MiSeq高通量测序技术分析河西走廊地区盐碱土壤微生物多样性
- Microbial diversity in saline alkali soil from Hexi Corridor analyzed by Illumina MiSeq high-throughput sequencing system
- 微生物学通报, 2017, 44(9): 2067-2078
- Microbiology China, 2017, 44(9): 2067-2078
- DOI: 10.13344/j.microbiol.china.160824
-
文章历史
- 收稿日期: 2016-11-14
- 接受日期: 2017-06-06
- 优先数字出版日期(www.cnki.net): 2017-06-26
2. 甘肃农业大学草业学院 甘肃 兰州 730070
2. Pratacultural College, Gansu Agricultural University, Lanzhou, Gansu 730070, China
土壤作为地球生态系统的重要组成部分,是人类赖以生存的基础。土壤中微生物数量大、种类多,但大量微生物的不可培养性成为传统微生物生态研究中最大的障碍。近年来,使用不同方法对土壤微生物多样性的研究越来越多。盐碱土壤作为一种极端环境,其中的微生物具有不同于一般微生物的遗传特性、生理功能和特殊结构,并且这些微生物的生命活动决定了土壤的理化性质,会影响土壤结构和肥力[1]。土壤微生物多样性研究在评价生态系统、维护生态平衡中发挥着巨大的作用[2]。因此探索盐碱化生态环境土壤微生物多样性对开发和利用盐碱土壤微生物资源具有重要意义,同时也为极端盐碱环境下微生物的分离和研究提供了理论依据。
河西走廊是甘肃省主要的粮食作物产区,也是丝绸之路的重要通道。它分为3个独立的内流盆地,盐碱化土壤普遍存在于这些地区,严重影响了区域内农业的发展。目前,对于河西走廊盐碱土壤微生物多样性的研究主要包括对河西走廊盐碱土细菌种群结构多样性的研究[3],探究了河西走廊春季不同盐碱土壤中微生物数量、酶活性与理化因子的关系[4];对河西走廊酒泉地区盐碱土未培养放线菌多样性的研究[5];对河西走廊石羊河下游地区盐碱土中放线菌多样性的研究[6];对河西走廊酒泉地区盐碱土可培养放线菌多样性的研究[7]。这些研究主要利用传统的培养方法或16S rRNA基因文库构建的分子生态学方法研究了盐碱地区细菌及放线菌群落结构的多样性。
高通量测序技术是目前应用最普遍的新一代测序技术。它在分析微生物的群落结构时有着独特的优势,能够通过从环境样本中直接获取的总DNA进行文库构建并测序[8],关键在于能够产生测序覆盖深度非常高的16S rRNA基因的测序数据,用来估计微生物群落的物种构成,能更加真实地揭示原位环境中微生物群落的复杂性和多样性[9]。对于任何一种复杂的生态系统,对其微生物多样性的研究是对其环境描述的基础[10]。赵婉雨等[11]采用高通量测序手段与传统培养技术相结合的方式对柴达木盆地达布逊盐湖微生物多样性的研究发现,该环境细菌多样性和丰度远高于古菌,并且通过纯培养方法得到的细菌主要是厚壁菌门,而通过高通量测序结果显示有53%为放线菌门;李新等[12]应用变性梯度凝胶电泳技术对内蒙古河套灌区三类不同盐碱程度的土壤细菌群落多样性进行研究发现,盐碱土壤的主要类群是变形菌门。国际上对盐碱环境中微生物群落结构的研究也很关注,Yousuf等[13]研究印度沿海盐碱地细菌多样性和群落结构,发现了细菌群落在3个样地中存在很大的差异,可能是与土壤中盐度、碳含量和硫含量有关,在农田土中化能无机自养型的微生物占优势,而在盐碱生态环境中绿弯菌门占优势;Canfora等[14]研究半干旱地中海地区极端高盐度土壤微生物群落结构和生物多样性,发现古细菌群落的丰度和多样性受土壤盐度的影响,而细菌群落随盐度梯度的增加而呈下降趋势。河西走廊地区由于其特殊的地理位置以及与周围地理环境的特定关系,农牧发展尤为令人注目,但其生态环境十分脆弱,由于不合理的生产和生活方式,导致该地区生态结构发生了不利于人类生存和发展的变化。笔者课题组之前的研究初步表明该地区微生物多样性丰富,但对于该地区盐碱土壤微生物多样性的研究还不够充分,了解土壤微生物群落构成,对生态环境的保护有一定的帮助。基于对前期研究的总结,本研究试图通过高通量测序手段来反映该地区微生物群落结构的多样性,以期完整了解河西走廊盐碱土壤微生物群落结构的多样性。
1 材料与方法 1.1 研究区域概况河西走廊位于甘肃省的西北部,东起乌鞘岭,西至古玉门关,南北介于南山(祁连山和阿尔金山)和北山(马鬃山、合黎山和龙首山)间,东经93°23′-104°12′,北纬37°17′-42°48′,长约900 km,宽数千米至近百千米,为西北-东南走向的狭长平地。年降水量29 mm-490 mm,年蒸发量1 800 mm-3 000 mm。全年日照时数3 000-3 400 h,年平均气温5-10。河西走廊分为3个独立的内流盆地,即疏勒河流域、黑河流域和石羊河流域。
1.2 样品采集2012年12月,土壤样品采集于甘肃省河西走廊地区(疏勒河流域、黑河流域和石羊河流域)分布的盐碱土。采用五点取样法采集土样,去除地表板结的部分,取0-20 cm的土壤样品保存于灭菌的密封袋中,置于冰袋中冷藏迅速带回实验室,土样在-80下保存,供土壤微生物种群结构多样性的分析。土壤样品基本信息见表 1。
| 土壤类型 Soil type |
编号 Soil No. |
采样点 Location |
经纬度 Longitude and latitude |
海拔 Altitude (m) |
主要植被 Main vegetation |
| 原生盐碱土Primary saline-alkali soil | S1 | 玉门市清泉镇 | E97.677 579°, N39.986 469° | 1 647.7 | 冰草、芦苇 |
| S2 | 高台县黑泉乡 | E99.638 251°, N39.506 657° | 1 272.8 | 红柳、芦苇 | |
| S3 | 民勤县昌宁乡 | E102.594 085°, N38.572 124° | 1 314.4 | 芦苇 | |
| 次生盐碱土 Secondary saline-alkali soil |
S4 | 瓜州县广至乡 | E95.575 876°, N40.438 953° | 1 071.3 | 白刺、芦苇 |
| S5 | 临泽县新华镇 | E99.933 506°, N39.262 469° | 1 322.2 | 芦苇 | |
| S6 | 民勤县薛百乡 | E102.979 748°, N38.570 892° | 1 330.1 | 沙棘、芦苇 | |
| 农田土 Farmland soil |
S7 | 瓜州县广至乡 | E95.575 981°, N40.439 062° | 1 071.8 | 棉花 |
| S8 | 高台县罗城乡 | E99.678 663°, N39.554 537° | 1 252.4 | 玉米 | |
| S9 | 民勤县三滩乡 | E103.236 211°, N38.756 648° | 1 293.2 | 棉花 |
Omega土壤DNA提取试剂盒、DNA胶回收纯化试剂盒购于北京全式金生物技术有限公司。
H-1650R台式高速冷冻离心机,湘仪离心机仪器有限公司;721分光光度计、K9840自动凯氏定氮仪,上海仪电分析仪器有限公司;WFX-210原子吸收分光光度计,北京瑞利公司;凝胶成像系统,Bio-Rad公司;电泳仪,北京市六一仪器厂;奥豪斯测试笔,兰州鑫泽华科生物科技有限公司。
1.4 土壤理化性质的测定风干及过筛后的土壤样品进行以下理化性质分析:pH、电导率、有机质、总氮、速效钾、有效磷。pH和电导率使用奥豪斯测试笔测定;速效钾采用醋酸铵浸提法[15];有效磷采用钼锑抗比色法[16];全氮采用凯氏定氮法[17];有机碳采用重铬酸钾容量法-水合热法测定[18]。
1.5 土壤总DNA的提取及16S rRNA基因的PCR扩增土壤总DNA基因组的提取采用DNA提取试剂盒进行。采用引物520F (5′-AYTGGGYDTAAAGN G-3′)和802R (5′-TACNVGGGTATCTAATCC-3′)扩增土壤微生物16S rRNA基因V4区。25 μL PCR反应体系:Q5 reaction buffer (5×) 5.0 μL,Q5 GC high enhancer (5×) 5.0 μL,dNTPs (2.5 mmol/L) 2.00 μL,模板(0.2 ng/μL) 1.0 μL,引物F (10 μmol/L) 1.0 μL,引物R (10 μmol/L) 1.0 μL,Q5 Polymerase (5 U/μL) 0.25 μL,灭菌超纯水9.75 μL。PCR反应条件:98;98,50,72,25个循环;72 min。扩增后的PCR产物进行2%琼脂糖凝胶电泳,经纯化后的样品用BioTek酶标仪进行定量检测,每个样品将重复的3个PCR产物混合后进行测定。
1.6 测序及分析经纯化检测后的样品送至上海派森诺生物科技有限公司利用Illumina Miseq高通量测序技术进行测序。
测序所得结果运用QIIME[19]进行序列过滤。运用Mothur[20]软件中UCHIME[21]的方法去除嵌合体序列。数据过滤的标准:(1) 去除5′端引物错配碱基数 > 1的序列;(2) 去除含有模糊碱基(Ambiguous)或长度≤150 bp的序列;(3) 去除嵌合体序列。
OTU (Operational taxonomic unit)聚类分析通过在QIIME中调用Uclust[22]方法对优质序列按相似度0.97进行聚类,选取每个类最长的序列为代表序列。OTU注释通过在QIIME中采用blast[23]的方法对序列数据库进行比对,获得每个OTU分类学信息。OTU维恩图利用Mothur软件或者R软件生成样本间(或组间) OTU的维恩图。稀释曲线图使用OTU表并利用QIIME软件制成。根据OTU列表中的各样品物种丰度情况,应用软件Mothur中的Summary.single命令,计算3种常用的生物多样性指数。将优势菌门与土壤理化因子结合制成RDA图。将属水平上的分类信息进行聚类,应用软件R (Pheatmap)绘制Heatmap图。
2 结果与分析 2.1 土壤理化性质土壤电导率和pH值反映出土壤的盐碱化程度,土壤有机质含量和土壤有效磷含量分别与土壤肥力和土壤中磷循环是密切相关的,土壤中总氮含量和速效钾含量可以反映出土壤氮素和速效钾的供应状况。本研究的9个样品理化性质如表 2所示,土壤电导率在0.14-13.66 ms之间,pH在8.3-9.0之间。有机质介于0.21-1.38 g/kg之间,总氮介于0.67-0.96 g/kg之间,速效钾介于134.77-300.79 μg/L之间,有效磷介于2.99-43.71 mg/kg之间。本研究土壤有机质和有效磷含量在不同盐碱化程度土壤中的分布与牛世全等[4]对河西走廊春季不同盐碱土壤中理化因子的研究结果相似,均为农田土有机质含量高于其他两类土壤,有效磷含量次生盐碱土低于其他两类土壤。与崔嵘等[24]对小兴安岭红树林土壤理化性质的结果比较,发现本研究中土壤pH明显高于红树林土壤pH,而速效钾、有效磷和有机质含量明显低于红树林土壤中的含量。本研究的土壤肥力均较低,不利于生物的生长。
| 样品 Sample |
pH | 电导率 Conductivity (ms/cm) |
总氮 Total nitrogen (g/kg) |
速效钾 Quick-acting potassium (μg/L) |
有效磷 Available phosphorous (mg/kg) |
有机质 Corg (g/kg) |
| S1 | 9.0 | 13.66 | 0.87 | 156.96 | 8.03 | 0.44 |
| S2 | 9.0 | 8.64 | 0.89 | 300.79 | 19.75 | 1.38 |
| S3 | 8.7 | 6.55 | 0.94 | 216.82 | 13.48 | 0.69 |
| S4 | 8.5 | 1.43 | 0.85 | 134.77 | 3.31 | 0.47 |
| S5 | 8.8 | 7.24 | 0.77 | 149.56 | 6.75 | 1.17 |
| S6 | 8.6 | 0.18 | 0.83 | 140.11 | 15.36 | 0.21 |
| S7 | 8.4 | 1.38 | 0.74 | 168.88 | 2.99 | 1.34 |
| S8 | 8.3 | 1.14 | 0.67 | 267.24 | 10.23 | 0.96 |
| S9 | 8.5 | 0.14 | 0.96 | 147.23 | 43.71 | 1.01 |
将9个样品分为3组,group1组土样均为原生盐碱土,所含样品为S1、S2、S3,Group2组土样均为次生盐碱土,所含样品为S4、S5、S6,Group3组土样均为农田土,所含样品为S7、S8、S9。
如图 1所示,其中Group1组共得到3 317个OTU,Group2组共得到5 448个OTU,Group3组共得到5 055个OTU。分别将这三组两两比较发现,Group1与Group2共有的OTU为2 145个,Group1与Group3共有的OTU为1 944个,Group2与Group3共有的OTU为4 007个。Group1中特有的OTU为868个,Group2中特有的OTU为936个,Group3中特有的OTU为744个。这三组的OTU数一共为7 364个,而它们共同包含的OTU数为1 640个。
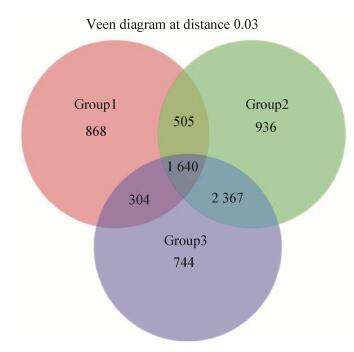
|
| 图 1 盐碱土壤样品微生物OTU数量的韦恩图 Figure 1 Venn diagram of OTU number based on 16S rRNA gene sequence in saline alkali soil samples |
|
|
结果显示,不同类型的土壤中所含OTU数Group2 > Group3 > Group1,说明了次生盐碱土的微生物类群最丰富,原生盐碱土的微生物类群最少;Group2次生盐碱土与Group3农田土微生物类群的一致性最高,差异较小,Group1原生盐碱土与Group3农田土、Group1原生盐碱土与Group2次生盐碱土微生物类群一致性较低,差异非常明显。图 1表明这三种土壤类型有部分一致的微生物类群,但从整体来看差异还是比较明显。
2.3 微生物多样性指数分析Shannon指数和Simpson指数可以用来估算样品中微生物的多样性,Shannon指数值越大,说明群落多样性越高;Simpson指数越大,说明群落多样性越低[25]。表 3的结果显示S6号Shannon指数最大,Simpson指数最小;S1号Shannon指数最小,Simpson指数最大,说明S1号土样的群落多样性最低,S6号土样的群落多样性最高。各样点的覆盖率分别为94.9%、94.5%、91.7%、93.4%、95.8%、93.9%、93.1%、94.7%和97.0%,说明各样地的微生物物种信息基本得到了充分的体现。
| 样品 Samples |
辛普森指数 Simpson |
香农指数 Shannon |
覆盖率 Coverage (%) |
| S1 | 0.033 | 4.788 | 94.9 |
| S2 | 0.006 | 6.600 | 94.5 |
| S3 | 0.009 | 6.366 | 91.7 |
| S4 | 0.003 | 7.174 | 93.4 |
| S5 | 0.011 | 5.665 | 95.8 |
| S6 | 0.002 | 7.176 | 93.9 |
| S7 | 0.002 | 7.166 | 93.1 |
| S8 | 0.002 | 7.053 | 94.7 |
| S9 | 0.007 | 6.635 | 97.0 |
从图 2中可以看出9个样品的稀释曲线均基本上趋于平缓,说明测序趋于饱和,取样基本合理,能够比较真实地反应出这9个土壤样品中的微生物群落,结合各样品覆盖率,说明测序结果中包含了大多数微生物类群,基本可以反映该区域土壤中微生物群落结构组成,但可能仍有少量微生物种类未被发现。

|
| 图 2 9个样品的稀释曲线 Figure 2 Rarefaction curves of nine soil samples at cutoff level of 3% |
|
|
9个土壤样品高通量测序后共获得23个门和未确定类群。如图 3所示,其中23个门分别属于古菌域的泉古菌门(Crenarchaeota)、广古菌门(Euryarchaeota)和细菌域的热袍菌门(Thermotogae)、支原体门(Mollicutes)、互养菌门(Synergistetes)、螺旋体门(Spirochaetes)、变形菌门(Proteobacteria)、浮霉菌门(Planctomycetes)、硝化螺菌门(Nitrospira)、芽单胞菌门(Gemmatimonadetes)、梭杆菌门(Fusobacteriales)、厚壁菌门(Firmicutes)、纤维杆菌门(Fibrobacteria)、酸杆菌门(Acidobacteria)、迷踪菌门(Elusimicrobia)、蓝藻门(Cyanobacteria)、绿弯菌门(Chloroflexi)、疣微菌门(Verrucomicrobia)、衣原体门(Chlamydiales)、绿菌门(Chlorobi)、拟杆菌门(Bacteroidetes)、装甲菌门(Armatimonadetes)、放线菌门(Actinobacteria)。将这9个样品依据微生物丰度分布特征,从属水平上进行聚类,图 4中红绿色代表着菌属的丰度,越偏向红色丰度越高,而越偏向绿色丰度越低。从图 4中可以看出,S4号与S5号微生物类群相似度最高,与S6号聚为一类,S8号与S7号微生物类群相似度最高,与S9号聚为一类,这6个样聚为一大类;S1号与S3号微生物类群相似度最高,与S2号聚为一类,与上述的6个样聚成的一大类为同一水平。

|
| 图 3 不同盐碱土壤样品中微生物菌门组成 Figure 3 Relative abundances of microbial phyla in different saline alkali soil samples |
|
|

|
| 图 4 基于属水平上的聚类分析 Figure 4 Based on the level of genus cluster analysis |
|
|
经分析,S1号样品有14个门,S2号样品有18个门,S3号样品有15个门,S4号样品有16个门,S5号样品有12个门,S6号样品有15个门,S7号样品有16个门,S8号样品有16个门,S9号样品有17个门。在这些盐碱土壤中占优势的菌门有:变形菌门(Proteobacteria) 28.56%、放线菌门(Actinobacteria) 17.2%、拟杆菌门(Bacteroidetes) 12.3%。还有一些菌门占有较低的百分比,依次是酸杆菌门(Acidobacteria) 6.08%、浮霉菌门(Planctomycetes) 6.02%、绿弯菌门(Chloroflexi) 5.78%、芽单胞菌门(Gemmatimonadetes) 5.43%、厚壁菌门(Firmicutes) 4.90%、疣微菌门(Verrucomicrobia) 4.46%;剩余的一些门占有的比例非常低:装甲菌门(Armatimonadetes)、绿菌门(Chlorobi)、蓝藻门(Cyanobacteria)、硝化螺菌门(Nitrospira)、广古菌门(Euryarchaeota)、泉古菌门(Crenarchaeota)、热袍菌门(Thermotogae)、支原体门(Mollicutes)、互养菌门(Synergistaceae)、螺旋体门(Spirochaetes)、梭杆菌门(Fusobacteriales)、纤维杆菌门(Fibrobacteria)、迷踪菌门(Elusimicrobia)、衣原体门(Chlamydiales)。原生盐碱土和农田土中占优势的菌门是变形菌门(Proteobacteria),次生盐碱土中占优势的菌门是放线菌门(Actinobacteria);原生盐碱土中的厚壁菌门(Firmicutes)所占的比例明显高于农田土和次生盐碱土,放线菌门(Actinobacteria)所占比例明显低于农田土和次生盐碱土。三类土壤中共有的菌门有广古菌门(Euryarchaeota)、酸杆菌门(Acidobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、绿菌门(Chlorobi)、绿弯菌门(Chloroflexi)、蓝藻门(Cyanobacteria)、厚壁菌门(Firmicutes)、芽单胞菌门(Gemmatimonadetes)、硝化螺菌门(Nitrospira)、浮霉菌门(Planctomycetes)、变形菌门(Proteobacteria)和疣微菌门(Verrucomicrobia)。
变形菌门(Proteobacteria)中分布的微生物属种类最多,其中α-变形菌纲(Alphaproteobacteria)在次生盐碱土中占优势,β-变形菌纲(Betaproteobacteria)在原生盐碱土和农田土中均占有优势,δ-变形菌纲(Deltaproteobacteria)和γ-变形菌纲(Gammaproteobacteria)在次生盐碱土中占优势。原生盐碱土的优势菌属有假单胞菌属(Pseudomonas) (9.8%)、KSA1 (5.1%)、乳球菌属(Lactococcus) (4.9%)、芽孢杆菌属(Bacillus) (3.1%)、嗜冷杆菌属(Psychrobacter) (2.2%)、不动杆菌属(Acinetobacter) (1.7%)、尤泽比氏菌属(Euzebya) (1.5%)、黄杆菌属(Flavobacterium) (1.3%)和气味黄杆菌属(Myroides) (1%);次生盐碱土的优势菌属有B-42 (3.1%)、Chitinophaga (2.2%)、小双孢菌属(Microbispora) (1.5%)和吉莱氏菌属(Gillisia) (0.9%);农田土的优势菌属有浮霉状菌属(Planctomyces) (2.0%)、Skermanella (1.5%)和鞘氨醇杆菌属(Sphingobacterium) (1.0%)。
2.6 土壤理化性质对微生物群落的影响RDA分析是为了检测微生物群落组成与环境变量之间的关系。轴一和轴二的解释量分别是50.11%和23.51%。环境因子包括pH、总氮、有机碳和土壤电导率对微生物群落组成影响较大,而土壤速效钾和有效磷对微生物群落组成影响不显著。由图 5可以看出同一类型盐碱土壤样品的点距离较近,说明它们的微生物群落构成相似。但次生盐碱土中3个样地之间的距离都较远,样品之间的微生物群落构成的差异较其它两类土壤更为明显,可能与次生盐碱化程度的差异有关。拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)与pH和电导率呈正相关,;拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、浮霉菌门(Planctomycetes)和厚壁菌门(Firmicutes)与总氮含量呈正相关;绿弯菌门(Chloroflexi)、芽单胞菌门(Gemmatimonadetes)、浮霉菌门(Planctomycetes)、酸杆菌门(Acidobacteria)、疣微菌门(Verrucomicrobia)与速效钾的含量成正相关;绿弯菌门(Chloroflexi)、浮霉菌门(Planctomycetes)、芽单胞菌门(Gemmatimonadetes)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)与有效磷含量呈正相关;有机质含量与拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、浮霉菌门(Planctomycetes)和芽单胞菌门(Gemmatimonadetes)呈正相关。

|
| 图 5 土壤样品微生物群落与土壤理化因子的冗余分析 Figure 5 Redundancy analysis biplot between the microbial communities and soil chemical properties |
|
|
土壤中含有巨大数量的微生物类群,这也体现出土壤微生物群落结构的复杂性。研究表明[3, 5-7],河西走廊酒泉地区盐碱土放线菌多样性为原生盐碱土 > 次生盐碱土 > 农田土,原生盐碱土中放线菌优势类群为棒状杆菌亚目和假诺卡氏菌亚目,次生盐碱土和农田土优势类群为微球菌亚目;石羊河下游地区盐碱土放线菌多样性为次生盐碱土 > 原生盐碱土 > 农田土,三类盐碱土的优势类群均为微球菌亚目;河西走廊盐碱土细菌种群结构多样性为次生盐碱土 > 原生盐碱土 > 农田土,原生盐碱土和次生盐碱土中优势类群为厚壁菌门和γ-变形菌纲,农田土中厚壁菌门为优势类群。本研究应用Illumina Miseq高通量测序技术对甘肃省河西走廊三大流域的9个土壤样品的微生物多样性进行16S rRNA基因测序分析,由于原核生物基因组内的16S rRNA基因是多重拷贝的,所以可能会导致本研究最后得出的微生物多样性和丰度结果比环境中实际存在的要高,Sun等[26]的研究表明在使用以16S rRNA基因为基础的研究方法中会过高地估计环境中微生物的多样性,在细菌中以16S rRNA基因为基础的分析理想目标区域为V4和V5区。本实验的扩增区域为16S rRNA基因的V4区,以尽可能地减少对于该环境中微生物多样性和丰度的高估。结果表明原生盐碱土与次生盐碱土、原生盐碱土与农田土微生物群落构成差异较大,次生盐碱土与农田土微生物群落构成差异较小,所得OTU最后分别归入23个门和未确定类群,其中占优势的菌门是变形菌门,其次是放线菌门,第三是拟杆菌门。有研究表明影响土壤微生物群落结构组成和多样性的因素有自然因素和人为因素[2]。虽然同为河西走廊地区的盐碱土壤,但由于不同的采样地点可能会受该地区人为干扰的影响而导致微生物群落组成发生变化,本研究采集样品的pH和电导率普遍高于之前的研究,同时本研究的RDA分析又表明了pH对于微生物群落的影响最显著,所以这有可能是引起同一地区不同采样点微生物群落组成具有较大差异的原因。在自然情况下形成的原生盐碱土中存在适应该环境特有的微生物群落[27],而由于人口压力增加,在大力发展生产的同时疏忽了排灌工程的配套、用水的管理和调节,这些人为原因使农田土盐碱化形成次生盐碱土,在微生物群落构成上会与农田土具有一定的相似性。应用高通量测序技术可以更为充分地反映出该地区盐碱土壤微生物群落的多样性和分布情况。
Canfora等[28]在对意大利西西里岛不同盐浓度的盐碱土对细菌多样性的影响研究中共得到52 623条序列,被归为15个门,占优势的菌门包括变形菌门、放线菌门、酸杆菌门、疣微菌门和厚壁菌门,其中变形菌门是最大的种群,占95.5%,其次是放线菌门,占83.39%。赵婉雨等[11]对达布逊盐湖的研究发现,8 378条序列分别属于广古菌门、放线菌门、拟杆菌门、变形菌门和疣微菌门。这些微生物门类同样存在于本研究中,说明在相似的盐碱环境中微生物类群的分布具有一定的相似性。其他研究也发现,在盐碱土中变形菌门是最常见的微生物类群[29-30],这与本研究的结果一致。目前发现大约50个属的细菌中含有嗜盐和耐盐菌株,它们分别属于变形菌门、厚壁菌门、拟杆菌门和放线菌门[31]。本研究发现的大部分微生物都属于这些细菌类群。张广志等[32]在盐碱土壤中分离鉴定耐盐细菌时发现了黄杆菌属,并且发现在盐浓度达到25%时黄杆菌属也能生存。黄杆菌属在本研究中为原生盐碱土的优势菌属,说明黄杆菌属的耐盐碱能力较强。有研究表明黄杆菌可以合成视紫红质[33],与一些嗜盐古菌合成的视紫红质一样,是一种通过质子泵捕捉光能的特殊产能方式[34]。这种特殊的代谢方式可能与黄杆菌能够在盐碱环境下生存有潜在的联系。有研究表明假单胞菌属也是一类耐盐碱的种群[14]。郑贺云等[35]对新疆阿克苏地区盐碱地细菌类群多样性分析中发现了21个优势菌属,Amorphus、黏球菌属、不动杆菌属、苍白杆菌属、微小杆菌属、假单胞菌属、芽孢杆菌属、草螺菌属、微杆菌属、葡萄球菌属、盐单胞菌属、Solibacillus、Planobacterium、红育菌属、Paenisporosarcina、需盐杆菌属、海杆菌属、链霉菌属、盐杆菌属、Salimicrobium、丙酸杆菌属,其中不动杆菌属、假单胞菌属和芽孢杆菌属在本研究中也都属于原生盐碱土的优势菌属,其余的18个属在本研究的三类土壤中都没有发现。虽然同为盐碱土壤,但所处地区生态环境不同,也可能导致微生物生态分布的不同。
本研究利用高通量测序方法对9个土样的微生物多样性进行了探究,共发现23个门174个纲322个目527个科898个属,变形菌门为原生盐碱土和农田土中的优势菌门,放线菌门是次生盐碱土中的优势菌门。研究发现原生盐碱土中微生物多样性最低,但占优势的菌属较次生盐碱土和农田土多;次生盐碱土多样性最高。表明河西走廊盐碱土微生物多样性非常丰富,尤其是在次生盐碱土中,分布的细菌多为嗜盐碱细菌,它们的生态分布具有明显的地区特异性。
4 结论(1) 对河西走廊三大流域的三类土壤利用高通量测序手段对微生物多样性进行研究。通过对9个土壤样品的OTU聚类分析、物种分布规律和多样性指数分析发现次生盐碱土的微生物类群最丰富,与农田土微生物类群组成差异较小,而原生盐碱土与农田土、原生盐碱土与次生盐碱土微生物类群组成差异非常显著。
(2) RDA分析、物种门分类水平上的丰度分析和属水平上的聚类分析表明,发现的23个细菌门类和未确定类群,其中变形菌门、放线菌门和拟杆菌门占优势。假单胞菌属、KSA1、乳球菌属、芽孢杆菌属、嗜冷杆菌属、不动杆菌属、尤泽比氏菌属、黄杆菌属和气味黄杆菌是原生盐碱土的主要类群;B-42、Chitinophaga、小双孢菌属和吉莱氏菌是次生盐碱土的主要类群;浮霉状菌属、Skermanella和鞘氨醇杆菌属是农田土的主要类群。发现土壤理化指标中速效钾和有效磷对于微生物群落组成的影响不显著,而土壤pH的影响尤为显著。
| [1] |
Ma Y, Zhao H, Xu CY, et al. Study on bacterial diversity in the eastern side of Ningxia Helan mountain[J]. Northern Horticulture, 2015(17): 157-160. (in Chinese) 马悦, 赵辉, 徐春燕, 等. 宁夏贺兰山东麓贫营养土壤中细菌多样性[J]. 北方园艺, 2015(17): 157-160. |
| [2] |
Cao HJ, Ni HW. Research advances in soil microbial diversity and its impact factors[J]. Territory & Natural Resources Study, 2015(3): 85-88. (in Chinese) 曹宏杰, 倪红伟. 土壤微生物多样性及其影响因素研究进展[J]. 国土与自然资源研究, 2015(3): 85-88. |
| [3] |
Niu SQ, Jing CH, Liao SQ, et al. Bacterial population structure diversity in saline-alkali soil in Hexi Corridor[J]. Journal of Northwest Normal University:Natural Science, 2013, 49(2): 90-95. (in Chinese) 牛世全, 景彩虹, 廖世齐, 等. 河西走廊盐碱土细菌种群结构多样性的研究[J]. 西北师范大学学报:自然科学版, 2013, 49(2): 90-95. |
| [4] |
Niu SQ, Yang JW, Hu L, et al. Relationship with soil microbial quantity, soil enzyme activity and physicochemical factor between different saline-alkali soil in Hexi Corridor in spring[J]. Microbiology China, 2012, 39(3): 416-427. (in Chinese) 牛世全, 杨建文, 胡磊, 等. 河西走廊春季不同盐碱土壤中微生物数量、酶活性与理化因子的关系[J]. 微生物学通报, 2012, 39(3): 416-427. |
| [5] |
Li HY, Niu SQ, Kong WB, et al. Diversity of uncultured actinomycetes in saline-alkali soil from Jiuquan area of Hexi Corridor[J]. Chinese Journal of Applied Ecology, 2015, 26(9): 2817-2823. (in Chinese) 李海云, 牛世全, 孔维宝, 等. 河西走廊酒泉地区盐碱土未培养放线菌多样性[J]. 应用生态学报, 2015, 26(9): 2817-2823. |
| [6] |
Li HY, Niu SQ, Kong WB, et al. Diversity of actinomycetes at the saline-alkali soils in downstream area of Shiyang River of Hexi Corridor-illustrated by the case of Minqin Country[J]. China Environmental Science, 2015, 35(6): 1805-1813. (in Chinese) 李海云, 牛世全, 孔维宝, 等. 河西走廊石羊河下游地区盐碱土中放线菌多样性——以民勤县为例[J]. 中国环境科学, 2015, 35(6): 1805-1813. |
| [7] |
Li HY, Hu L, Niu SQ, et al. Diversity of culturable actinomycetes in saline-alkali soil in Jiuquan region of Hexi Corridor[J]. Chinese Journal of Ecology, 2015, 34(2): 367-372. (in Chinese) 李海云, 胡磊, 牛世全, 等. 河西走廊酒泉地区盐碱土壤中可培养放线菌多样性[J]. 生态学杂志, 2015, 34(2): 367-372. |
| [8] |
Qin N, Li DF, Yang RF. Next-generation sequencing technologies and the application in microbiology-a review[J]. Acta Microbiologica Sinica, 2011, 51(4): 445-457. (in Chinese) 秦楠, 栗东芳, 杨瑞馥. 高通量测序技术及其在微生物学研究中的应用[J]. 微生物学报, 2011, 51(4): 445-457. |
| [9] |
Li QG, Tao L. High-throughput sequencing technology and its application in life science[J]. Animal Husbandry and Feed Science, 2012, 33(2): 25-28. (in Chinese) 李庆岗, 陶立. 高通量测序技术及其在生命科学中的应用[J]. 畜牧与饲料科学, 2012, 33(2): 25-28. |
| [10] |
Gans J, Wolinsky M, Dunbar J. Computational improvements reveal great bacterial diversity and high metal toxicity in soil[J]. Science, 2005, 309(5739): 1387-1390. DOI:10.1126/science.1112665 |
| [11] |
Zhao WY, Yang J, Dong HL, et al. Microbial diversity in the hypersaline Dabuxun lake in Qaidam basin, China[J]. Earth and Environment, 2013, 41(4): 398-405. (in Chinese) 赵婉雨, 杨渐, 董海良, 等. 柴达木盆地达布逊盐湖微生物多样性研究[J]. 地球与环境, 2013, 41(4): 398-405. |
| [12] |
Li X, Jiao Y, Dai G, et al. Soil bacterial community diversity under different degrees of saline-alkaline in the Hetao area of Inner Mongolia[J]. China Environmental Science, 2016, 36(1): 249-260. (in Chinese) 李新, 焦燕, 代钢, 等. 内蒙古河套灌区不同盐碱程度的土壤细菌群落多样性[J]. 中国环境科学, 2016, 36(1): 249-260. |
| [13] |
Yousuf B, Sanadhya P, Keshri J, et al. Comparative molecular analysis of chemolithoautotrophic bacterial diversity and community structure from coastal saline soils, Gujarat, India[J]. BMC Microbiology, 2012, 12(1): 1. DOI:10.1186/1471-2180-12-1 |
| [14] |
Canfora L, Lo Papa G, Antisari LV, et al. Spatial microbial community structure and biodiversity analysis in "extreme" hypersaline soils of a semiarid Mediterranean area[J]. Applied Soil Ecology, 2015, 93: 120-129. DOI:10.1016/j.apsoil.2015.04.014 |
| [15] |
Zhang NF. The determination of soil available potassium-the method of ammonium acetate extraction and flame photometer[J]. Soils and Fertilizers, 1974(1): 33-35. (in Chinese) 张乃凤. 土壤速效钾的测定——醋酸铵浸提、火焰光度计测定法[J]. 土壤肥料, 1974(1): 33-35. |
| [16] |
Ding L, Li JH, Zhao SF, et al. Effects of application of biological organic fertilizer and antagonistic bacteria on soil available nutrients and soil enzyme activities[J]. Xinjiang Agricultural Sciences, 2011, 48(3): 504-510. (in Chinese) 丁雷, 李俊华, 赵思峰, 等. 生物有机肥和拮抗菌对土壤有效养分和土壤酶活性的影响[J]. 新疆农业科学, 2011, 48(3): 504-510. DOI:10.6048/j.issn.1001-4330.2011.03.018 |
| [17] |
Shen W, Chen JZ, Dai CJ. Determination of protein content in milk by microwave digestion-kjeldahl[J]. Food Research and Development, 2009, 30(5): 96-97. (in Chinese) 沈文, 陈均志, 代春吉. 微波消解——凯式定氮法测定牛奶中蛋白质含量[J]. 食品研究与开发, 2009, 30(5): 96-97. |
| [18] |
Yu R, Xu MG, Wang BR. Study on methods for determining labile orange matter of soils[J]. Soils and Fertilizers, 2005(2): 49-52. (in Chinese) 于荣, 徐明岗, 王伯仁. 土壤活性有机质测定方法的比较[J]. 中国土壤肥料, 2005(2): 49-52. |
| [19] |
Caporaso JG, Kuczynski J, Stombaugh J. QⅡME allows analysis of high-throughput community sequencing data[J]. NatureMethods, 2010, 7(5): 335-336. |
| [20] |
Schloss PD, Westcott SL, Ryabin T, et al. Introducing mothur:open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541. DOI:10.1128/AEM.01541-09 |
| [21] |
Edgar RC, Haas BJ, Clemente JC, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-2200. DOI:10.1093/bioinformatics/btr381 |
| [22] |
Edgar RC. Search and clustering orders of magnitude faster than blast[J]. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [23] |
Altschul SF, Gish W, Miller W, et al. Basic local alignment search tool[J]. Journal of Molecular Biology, 1990, 215(3): 403-410. DOI:10.1016/S0022-2836(05)80360-2 |
| [24] |
Cui R, Zou L, Yu Y, et al. Variations of soil enzyme activities and soil physical and chemical properties in Korean Pine forest of Xiaoxing'an mountains[J]. Journal of Northeast Forestry University, 2016, 44(8): 49-54. (in Chinese) 崔嵘, 邹丽, 于洋, 等. 小兴安岭红松林土壤酶活性与土壤理化性质的时空变化[J]. 东北林业大学学报, 2016, 44(8): 49-54. |
| [25] |
He Z, Zhao TT, Xing ZL, et al. Analysis of bacterial community composition in landfill cover soil[J]. China Environmental Science, 2015, 35(12): 3744-3753. (in Chinese) 何芝, 赵天涛, 邢志林, 等. 典型生活垃圾填埋场覆盖土微生物群落分析[J]. 中国环境科学, 2015, 35(12): 3744-3753. DOI:10.3969/j.issn.1000-6923.2015.12.028 |
| [26] |
Sun DL, Jiang X, Wu QL, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19): 5962-5969. DOI:10.1128/AEM.01282-13 |
| [27] |
Zahran HH. Diversity, adaptation and activity of the bacterial flora in saline environments[J]. Biology and Fertility of Soils, 1997, 25(3): 211-223. DOI:10.1007/s003740050306 |
| [28] |
Canfora L, Bacci G, Pinzari F, et al. Salinity and bacterial diversity:to what extent does the concentration of salt affect the bacterial community in a saline soil?[J]. PLoS One, 2014, 9(9): e106662. DOI:10.1371/journal.pone.0106662 |
| [29] |
Ma B, Gong J. A meta-analysis of the publicly available bacterial and archaeal sequence diversity in saline soils[J]. World Journal of Microbiology and Biotechnology, 2013, 29(12): 2325-2334. DOI:10.1007/s11274-013-1399-9 |
| [30] |
Valenzuela-Encinas C, Neria-González I, Alcántara-Hernández RJ, et al. Changes in the bacterial populations of the highly alkaline saline soil of the former lake Texcoco (Mexico) following flooding[J]. Extremophiles, 2009, 13(4): 609-621. DOI:10.1007/s00792-009-0244-4 |
| [31] |
Hou MF, He SL, Li D, et al. Bacterial diversity in Lianyungang marine sediment and Qinghai lake sediment[J]. Environmental Science, 2011, 32(9): 2681-2688. (in Chinese) 侯梅锋, 何士龙, 李栋, 等. 连云港海底底泥及青海湖底泥细菌多样性研究[J]. 环境科学, 2011, 32(9): 2681-2688. |
| [32] |
Zhang GZ, Zhou HZ, Yang HT, et al. Isolation and identification of halotolerant bacteria from salt-soda soil[J]. Shandong Agricultural Sciences, 2008(9): 49-50. (in Chinese) 张广志, 周红姿, 杨合同, 等. 盐碱土壤中耐盐细菌的分离与鉴定[J]. 山东农业科学, 2008(9): 49-50. |
| [33] |
Gómez-Consarnau L, González JM, Coll-Lladó M, et al. Light stimulates growth of proteorhodopsin-containing marine Flavobacteria[J]. Nature, 2007, 445(7124): 210-213. DOI:10.1038/nature05381 |
| [34] |
Jiao NZ, Feng FY, Wei B. Proteorhodopsin-a new path for biological utilization of light energy in the sea[J]. Chinese Science Bulletin, 2006, 51(8): 889-896. DOI:10.1007/s11434-008-0889-x |
| [35] |
Zheng HY, Li ZK, Li C, et al. Analysis of the bacterial diversity and dominant population in Akesu saline-alkali in Xinjiang[J]. Microbiology China, 2012, 39(7): 1031-1043. (in Chinese) 郑贺云, 黎志坤, 李超, 等. 新疆阿克苏地区盐碱地细菌类群多样性及优势菌群分析[J]. 微生物学通报, 2012, 39(7): 1031-1043. |
 2017, Vol. 44
2017, Vol. 44




