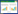扩展功能
文章信息
- 徐怀英, 黄迪海, 张伟, 秦春芝, 刘星丽, 王友令, 欧阳文军, 秦卓明
- XU Huai-Ying, HUANG Di-Hai, ZHANG Wei, QIN Chun-Zhi, LIU Xing-Li, WANG You-Ling, OUYANG Wen-Jun, QIN Zhuo-Ming
- 中国禽源新城疫病毒(NDV)流行株F和HN基因的遗传演化和变异频率
- Genetic evolution and substitutions frequency of F and HN gene of Newcastle disease virus isolates from poultry in China
- 微生物学通报, 2017, 44(12): 2933-2941
- Microbiology China, 2017, 44(12): 2933-2941
- DOI: 10.13344/j.microbiol.china.170422
-
文章历史
- 收稿日期: 2017-06-13
- 接受日期: 2017-08-28
- 优先数字出版日期(www.cnki.net): 2017-09-15
2. 山东省健牧生物药业有限公司 山东 济南 250100;
3. 山东师范大学 山东 济南 250014
2. Shandong Jianmu Biological Pharmaceutical Co., Ltd., Jinan, Shandong 250100, China;
3. Shandong Normal University, Jinan, Shandong 250014, China
新城疫(Newcastle disease,ND)主要是由新城疫病毒(Newcastle disease virus,NDV)引起的家禽、野鸟等感染的一种烈性传染病,是危害中国乃至全世界养禽业最重要的疫病之一[1-2]。自1926年首次在英国发现以来,该病已在世界范围内流行近90年,给全球家禽和野生鸟类造成了严重危害,带来了巨大的经济损失。
NDV是一种具有囊膜的单股、负链、不分节段的RNA病毒。依据其基因组大小,初步分为Class Ⅰ和Class Ⅱ两大类。Class Ⅰ主要源自水禽,由15 198个核苷酸组成,主要为弱毒。Class Ⅱ由15 186和15 192两类核苷酸组成,除基因Ⅰ型和部分基因Ⅱ型为弱毒外,大部分为强毒。NDV基因组包含6个基因,分别编码3′NP-P-M-F-HN-L5′六种结构蛋白和两种非结构蛋白[3]。NDV的囊膜由F和HN两个糖蛋白组成,F糖蛋白能够融合宿主细胞膜,使病毒侵入宿主细胞内;HN糖蛋白具有血凝素和神经氨酸酶活性,在病毒侵染过程中识别细胞受体,介导病毒吸附细胞膜,是极其重要的保护性抗原,这两种蛋白决定着NDV的抗原性(包括毒力等)和免疫原性[4-5]。
为探讨近20年来中国NDV分子遗传变异的趋势,课题组在总结前期研究的基础上,结合已在GenBank登录的国内外364株NDV序列,并以最早在国内发生的Ⅶ型Go/GD/QY/1997 (简写“QY97”)为参考毒株,从F和HN基因的核苷酸和氨基酸水平上分析比较其替代频率,旨在进一步探讨NDV的遗传变异规律,为NDV的合理防控提供依据。
1 材料与方法 1.1 NDV基因序列来源本课题组1997-2015年期间分离、鉴定并测序的89株NDV流行毒株的基因序列,以及从GenBank下载的364株具有代表性的国内外禽源NDV流行毒株和15株国内外经典毒株序列。
1.2 方法(1) 利用Lasergene 7.1和MEGA 5.1软件对NDV毒株的F和HN基因核苷酸(nt)和氨基酸(aa)序列进行比对,利用F和HN基因nt序列全长绘制系统发育树,参照Diel等方法进行基因分型[6]。
(2) 参照文献[7-8]的方法,利用MEGA 5.1软件,比较NDV流行株不同基因型的F和HN基因的核苷酸(氨基酸)替代频率。同时,以国内最早发生的Ⅶ型QY97为参考(GenBank登录号为KJ782375),重点评价了1997-2015年在我国广泛流行的Ⅶ型NDV流行株的替代频率,并对其关键氨基酸位点进行分析,进而探讨其分子变异机制。
2 结果与分析 2.1 系统发育树的构建利用NDV F和HN基因全长核苷酸(nt)序列绘制系统进化树,二者的绘制结果基本一致。图 1显示的是依据F基因nt全长绘制的系统发育树。依据Diel等[6]的标准,初步将NDV毒株分为15个基因型(图 1)。本实验室分离的89株NDV流行株主要分布于基因Ⅱ型(8株)、Ⅵ型(6株)、Ⅶ型(71株)和Ⅸ型(4株)。其中,基因Ⅶ型进一步演化为不同的亚型(图 1),而Ⅶ d型多达67株(占Ⅶ型的94.4%),这表明Ⅶ d亚型不仅是Ⅶ型中的优势株,也是我国NDV流行株中的优势株。
|
| 图 1 中国1997-2015年新城疫流行株F基因系统发育树 Figure 1 F gene phylogenetic tree of NDV strains from 1997 to 2015 in China 注:GenBank号列于NDV毒株名称之前;◀:表示含2个以上毒株;括号中的数字表示毒株数量;标尺刻度代表序列差异. Note: GenBank accession number were listed before the names of NDV. ◀: More than two strains; The numbers in brackets indicate NDV isolates; The scale bar means sequence difference. |
|
|
国外NDV流行株主要分布于Ⅹ型、Ⅺ型和ⅩⅢ型,与中国NDV流行株明显不同,具体分布如下:基因Ⅹ型(3株,源自北美洲的美国)、Ⅺ型(3株,源自非洲的马达加斯加)和ⅩⅢ型(4株,源自南亚的巴基斯坦)。
比较发现:NDV流行株基因型的分布与地域、时间密切相关。不同时间、不同国家和区域,其基因型不同,具有地域性特点,显示出NDV遗传演化的多样性和复杂性。
2.2 相似性比较NDV F和HN基因相似性比较显示:同一基因型内核苷酸(nt)相似性较高(表 1)。以HN基因为例:基因Ⅶ型毒株间nt相似性为92.1%-99.9%;基因Ⅱ型毒株间为95.6%-100%;基因Ⅰ型毒株间为93.1%-100%;基因Ⅲ型毒株间为99.3%-99.9%;基因Ⅸ毒株间为99.4%-99.9%;基因Ⅵ型毒株间为91.4%-100%。相反,不同基因型间差异较大,如La Sota与基因Ⅶ型毒株之间nt/aa相似性为78.1%-80.1%;与基因Ⅸ型相似性为86.8%-87.4%;与基因Ⅵ型相似性为79.9%-81.2%。F和HN基因nt相似性结果一致。
| 基因型 Genotype |
年代 year |
F基因 F gene |
HN基因 HN gene |
|||||
| 毒株数量 Number of strains |
基因型内的同源性 Homology in the same genotype (%) |
平均替代率 Average substitution rate (×10-3) |
毒株数量 Number of strains |
基因型内的同源性 Homology in the same genotype (%) |
平均替代率 Average substitution rate (×10-3) |
|||
| Ⅰ | 1967-2014 | 23 | 95.5-99.9 | 1.01 | 23 | 93.1-100 | 0.97 | |
| Ⅱ | 1946-2015 | 23 | 96.3-99.6 | 0.07 | 17 | 95.6-100 | 0.08 | |
| Ⅲ | 1950-2009 | 8 | 96.0-100 | 0.02 | 6 | 99.3-99.9 | 0.07 | |
| Ⅸ | 1948-2014 | 25 | 98.9-99.8 | 0.02 | 12 | 99.4-99.9 | 0.06 | |
| Ⅵ | 1972-2015 | 62 | 92.7-100 | 2.22 | 47 | 91.4-100 | 1.82 | |
| Ⅶ | 1997-2015 | 327 | 93.3-99.9 | 2.26 | 274 | 92.1-99.9 | 3.23 | |
|
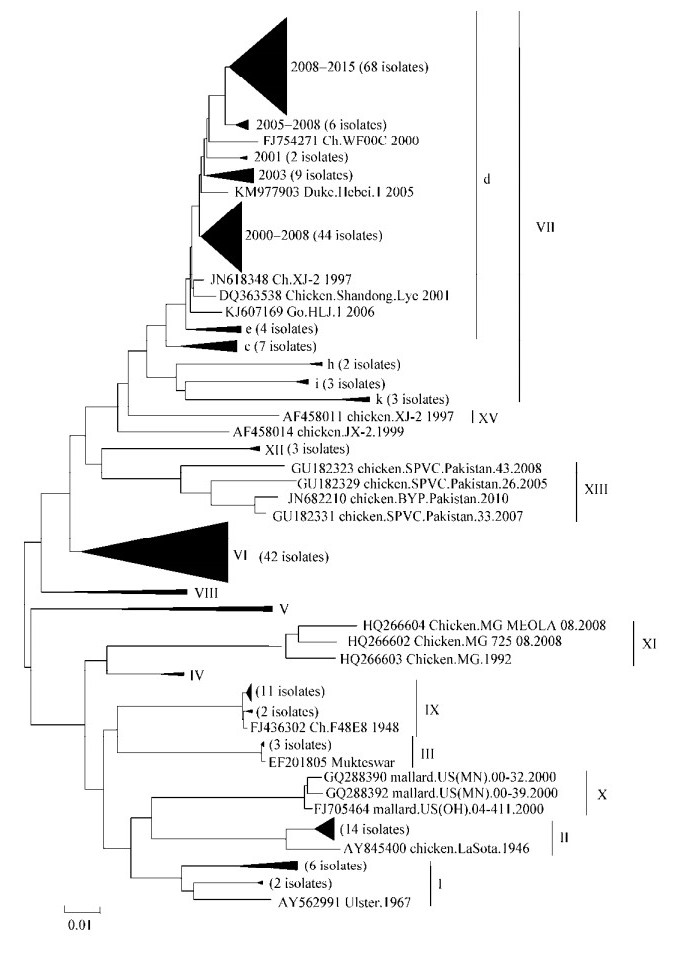
| 图 2 NDV流行株与La Sota株F基因的核苷酸差异及其与年代的相关性 Figure 2 The relationship between F gene nt homologies of NDV epidemic strains against La Sota in different years |
|
|
| 阶段 Period |
年代 Year |
毒株数量 Number of strains |
基因长度 Length (nt) |
变异数量 Number of variation |
变异率 Variation rate (%) |
转换数 P |
颠换数 Q |
替代率 substitution rate (%) |
平均替代率 Average substitution rate (×10-3) |
| 未使用基因Ⅶ型疫苗 Unused genotype Ⅶ vaccine |
1997 | 1 | 1 662 | 0 | 0 | 0 | 0 | 0 | 4.72* |
| 1998 | 1 | 1 662 | 40.0 | 2.41 | 38.0 | 2.0 | 2.53 | ||
| 1999 | 2 | 1 662 | 75.0 | 4.51 | 69.5 | 5.5 | 4.90 | ||
| 2000 | 5 | 1 662 | 39.4 | 2.37 | 35.6 | 3.8 | 2.54 | ||
| 2001 | 6 | 1 662 | 36.5 | 2.20 | 32.8 | 3.7 | 2.36 | ||
| 使用基因Ⅶ型疫苗 Using genotype Ⅶ vaccine |
2002 | 6 | 1 662 | 34.3 | 2.06 | 29.5 | 3.8 | 2.16 | 1.60 |
| 2003 | 23 | 1 662 | 46.2 | 2.78 | 41.5 | 4.7 | 3.00 | ||
| 2004 | 6 | 1 662 | 39.5 | 2.38 | 34.5 | 5.0 | 2.59 | ||
| 2005 | 16 | 1 662 | 42.5 | 2.56 | 38.0 | 4.5 | 2.76 | ||
| 2006 | 33 | 1 662 | 47.5 | 2.86 | 42.4 | 5.1 | 3.10 | ||
| 2007 | 42 | 1 662 | 51.9 | 3.12 | 46.2 | 5.7 | 3.40 | ||
| 2008 | 47 | 1 662 | 53.5 | 3.22 | 48.0 | 5.5 | 3.50 | ||
| 2009 | 20 | 1 662 | 56.8 | 3.42 | 51.6 | 5.2 | 3.70 | ||
| 2010 | 20 | 1 662 | 62.8 | 3.78 | 56.6 | 6.2 | 4.11 | ||
| 2011 | 45 | 1 662 | 63.7 | 3.83 | 57.6 | 6.1 | 4.17 | ||
| 2012 | 17 | 1 662 | 67.2 | 4.04 | 61.1 | 6.1 | 4.40 | ||
| 2013 | 12 | 1 662 | 64.4 | 3.87 | 58.2 | 6.2 | 4.22 | ||
| 2014 | 4 | 1 662 | 59.0 | 3.55 | 53.0 | 6.0 | 3.86 | ||
| 2015 | 5 | 1 662 | 67.1 | 4.04 | 60.8 | 6.3 | 4.40 | ||
| 注:*:与2002-2015年NDV分离株差异显著(p < 0.01). Note: *: highly significiant difference comparing NDV isolates in 2002-2015 with separate periods (p < 0.01). |
|||||||||
| 阶段 Period |
年代 Year |
毒株数量 Number of strains |
基因长度 Length (nt) |
变异数量 Number of variation |
变异率 Variation rate (%) |
转换数 P |
颠换数 Q |
替代率 Substitution rate (%) |
平均替代率 Average substitution rate (×10-3) |
| 未使用基因Ⅶ型疫苗 Unused genotype Ⅶ vaccine |
1997 | 1 | 1 716 | 0.0 | 0.00 | 0.0 | 0.0 | 0.00 | 8.20* |
| 1998 | 1 | 1 716 | 8.0 | 3.08 | 7.0 | 1.0 | 0.50 | ||
| 1999 | 3 | 1 716 | 52.7 | 3.00 | 48.0 | 4.7 | 3.30 | ||
| 2000 | 9 | 1 716 | 51.4 | 3.78 | 45.2 | 6.2 | 3.26 | ||
| 2001 | 8 | 1 716 | 64.7 | 3.53 | 58.6 | 6.1 | 4.13 | ||
| 使用基因Ⅶ型疫苗 Using genotype Ⅶ vaccine |
2002 | 9 | 1 716 | 60.5 | 4.01 | 53.5 | 7.0 | 3.83 | 1.84 |
| 2003 | 22 | 1 716 | 68.7 | 4.37 | 61.1 | 7.6 | 4.38 | ||
| 2004 | 11 | 1 716 | 69.5 | 4.38 | 62.7 | 6.8 | 4.44 | ||
| 2005 | 14 | 1 716 | 74.9 | 4.67 | 66.5 | 8.4 | 4.85 | ||
| 2006 | 20 | 1 716 | 75.0 | 4.43 | 66.5 | 8.5 | 4.79 | ||
| 2007 | 24 | 1 716 | 80.0 | 4.65 | 71.2 | 8.8 | 5.14 | ||
| 2008 | 37 | 1 716 | 75.9 | 4.43 | 67.7 | 8.2 | 4.89 | ||
| 2009 | 11 | 1 716 | 79.6 | 4.65 | 71.0 | 8.6 | 5.14 | ||
| 2010 | 9 | 1 716 | 83.0 | 4.45 | 74.0 | 9.0 | 5.35 | ||
| 2011 | 29 | 1 716 | 90.5 | 5.29 | 79.6 | 10.9 | 5.92 | ||
| 2012 | 19 | 1 716 | 92.6 | 5.41 | 83.0 | 9.6 | 5.99 | ||
| 2013 | 11 | 1 716 | 96.6 | 5.64 | 86.2 | 10.4 | 6.26 | ||
| 2014 | 2 | 1 716 | 75.5 | 4.41 | 65.5 | 10.0 | 4.87 | ||
| 2015 | 2 | 1 716 | 98.5 | 5.57 | 88.0 | 10.5 | 6.41 | ||
| 注:*:与2002-2015年NDV分离株差异显著(p < 0.01). Note: *: highly significant difference comparing NDV isolates in 2002-2015 with separate periods (p < 0.01). |
|||||||||
由表 3可见:1997-2015年间,NDV流行株HN基因的年平均核苷酸替代率为3.37×10-3。其中,1997-2001年(未使用基因Ⅶ型疫苗) HN基因的年平均核苷酸替代率为8.28×10-3;2002-2015年(疫苗使用后)为1.84×10-3。
(2) 氨基酸替代率比较:参照上述方法,对近20年的NDV基因Ⅶ流行株氨基酸替代率进行比较。结果发现F基因在1997-2015年间的年平均氨基酸替代率为1.14×10-3。其中,1997-2001年间的替代率为3.86×10-3,而在2002-2015年间的替代率仅为0.31×10-3。HN基因在1997-2015年间的年平均氨基酸替代率为2.35×10-3。其中,1997-2001年间的替代率为5.86×10-3,而2002-2015年间的替代率仅为1.49×10-3。
综合比较显示:HN基因的年平均替代率略高于F,显示出二者具有相同的进化规律。
2.4 NDV基因Ⅶ型的关键氨基酸位点分析通过对71株国内NDV基因Ⅶ型的氨基酸基序和系统发育比较发现,NDV基因Ⅶ型流行毒病还可以进一步分为不同的基因亚型,该结果已在多篇文献中描述[9-10]。本研究证实,基因Ⅶ d亚型不仅为优势流行株,还具有其它亚型所不具备的氨基酸位点,如71R、474I和500A等;同时,F蛋白出现了较多的氨基酸替代,即78K→R、145N→K、279Q→H、387K→R、480K→R、513V→I和520G→A,其中除4个氨基酸为保守性替换外,其它3个氨基酸的改变涉及到极性所带电荷的变化,而且具有氨基酸变异的累积。
HN基因与F基因相似,也出现了多个特异性的氨基酸替代位点,如45V→A、48T→M、49T→E、102T→V、118A→E、120N→S、129D→G、310D→G、362G→A、347E→K、443T→M、479H→Y和540V→A等。
3 讨论 3.1 NDV基因型的多样性和复杂性中国是一个养禽大国,免疫接种是防控ND的基本国策,通过疫苗的推广应用,我国已经把ND控制到比较低的水平。尽管如此,中国依然是世界上NDV流行较为严重的国家。近期的第四次NDV世界大流行无论在疫病流行的规模、速度、宿主的种类、危害程度及持续时间上,均超过了前三次[11-13]。目前,中国NDV流行株主要包含基因Ⅱ、Ⅵ、Ⅶ、Ⅸ、Ⅻ和ⅩⅤ型[6-13],是世界上NDV基因型存在较多的国家之一,这无疑对我国的NDV防控提出了新的挑战。
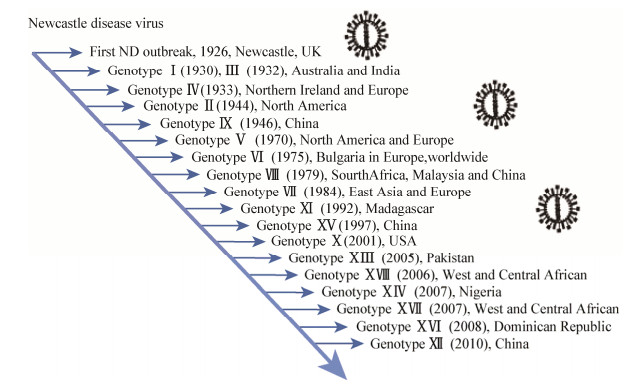
基因分型是在NDV分类中应用最广泛、最便捷和最准确的方法,因为它建立在对病原分子序列分析的基础上,不同基因型之间的进化距离和系统发育存在着更为精准的相关性。研究者们在参照国际公认的禽流感基因型划分标准的同时,对NDV提出了更为合理的分型依据,初步确定了NDV不同基因型间的平均进化距离(10%),并把其作为鉴定新基因型的标准[6]。依据新的分类方法,Class Ⅰ病毒由单一基因型组成,而Class Ⅱ则由18个基因型组成(本实验有15个基因型),包括先前确定的10个(Ⅰ-Ⅸ和Ⅺ)基因型和8个新基因型(Ⅹ、Ⅻ-ⅩⅧ)[14-16],新基因型大都来自北美洲、非洲和欧洲,但Ⅻ和ⅩⅤ源自中国。图 3显示了不同时间的NDV进化和新基因型出现的时间,显示出在一定的免疫压力下,点突变、不同宿主种间传播、气候、地域等因素共同促进了NDV的演化。
|
| 图 3 新城疫病毒不同年代基因型的演化历史 Figure 3 Evolutionary history of different genotypes of Newcastle disease virus in different years |
|
|
遗传和变异是病毒适应外界环境并维持生存的一种重要方式,也是病毒逃避和抵抗宿主疫苗免疫的重要手段。NDV属于RNA病毒,单股、负链、不分阶段,其复制酶缺乏校正机制,因此,NDV的变异频率相对于DNA病毒较高[17]。NDV不同基因型之间,其遗传变异速率不一,以基因Ⅵ型和Ⅶ型变异频率最大,而其它基因型则相对较小。该F基因变异频率结果与Lee等的研究相近(年核苷酸变异频率为2.89×10-3)[18]。进一步的研究表明,1997-2001年NDV基因Ⅶ型最早出现时,其HN基因核苷酸年平均进化率明显比F基因高,这暗示着对NDV抗原性影响较大的HN基因则必然会影响疫苗的免疫效果。部分养殖场为提高疫苗的保护效果,陆续接种了基因Ⅶ灭活疫苗,因此在2002-2015年间,HN基因核苷酸年平均替代率仅为1.84×10-3。这表明,当流行株与疫苗株完全匹配时,可大大降低免疫鸡群的排毒量和载毒量,减少NDV变异的频率[19]。该结果与低致病性禽流感H9N2的变异十分相似。但对于多片段的禽流感病毒而言,H9N2流行株HA基因的年均替代率明显高于NDV的HN变异频率[8]。
3.3 NDV变异对于疫苗防控的启示尽管NDV只有一个血清型,但其编码的基因型已进化到18个。这表明,NDV在长期的免疫选择压力下正处于持续的进化之中。前期的研究表明,NDV的HN基因和F基因同源性均与HI交叉抑制试验具有相关性,但主要与HN基因有关,这也在一定程度上证实了长期的点突变积累造成了NDV抗原性的变化[20]。
HN蛋白是NDV囊膜上最大的纤突糖蛋白,在免疫反应中具有决定作用,是重要的保护性抗原。HN蛋白结构呈β螺旋折叠,内有4股反向平行的6个β折叠区。相对应的氨基酸区域位点为:175-228 (β1:抗原对应位点23)、237-288 (β2:抗原对应位点3)、316-396 (β3:抗原对应位点3、1、4和14)、401-403 (β4)、472-515 (β5:抗原对应位点2)和521-577C末端(β6:抗原对应位点2)[21-22]。生物信息学分析发现,NDV流行株的HN基因在上述氨基酸关键位点已经发生了改变,如347位为“E→K”、362位“G→A”等,上述位点的改变均会对NDV的抗原性产生影响[23]。
新城疫在全球已流行近90年,疫苗预防在控制ND流行方面发挥了重要作用。但再完美的疫苗也不能阻挡NDV的遗传演变和替代,对病原的杀灭净化和生物安全措施应成为下一步疫病防控的重点。
| [1] |
Ma BC, He YF, Xu HY, et al. Isolation, identification and molecular characterization of a velogenic NDV isolate from a broiler flock in high antibody level against NDV[J]. Microbiology China, 2007, 34(1): 95-100. 马保臣, 何叶峰, 徐怀英, 等. 高抗体水平种鸡NDV的分离鉴定和分子特性[J]. 微生物学通报, 2007, 34(1): 95-100. |
| [2] |
Wang YK, Tian HF, Zhou JH, et al. A primary study on the Geese's paramyxovirus infection[J]. China Poultry, 1998, 20(4): 3-5. 王永坤, 田慧芳, 周继宏, 等. 鹅副粘病毒病的研究[J]. 中国家禽, 1998, 20(4): 3-5. |
| [3] |
Kattenbelt JA, Stevens MP, Gould AR. Sequence variation in the Newcastle disease virus genome[J]. Virus Research, 2006, 116(1/2): 168-184. |
| [4] |
de Leeuw OS, Koch G, Hartog L, et al. Virulence of Newcastle disease virus is determined by the cleavage site of the fusion protein and by both the stem region and globular head of the haemagglutinin-neuraminidase protein[J]. Journal of General Virology, 2005, 86(6): 1759-1769. DOI:10.1099/vir.0.80822-0 |
| [5] |
Jin JH, Zhao J, Ren YC, et al. Contribution of HN protein length diversity to Newcastle disease virus virulence, replication and biological activities[J]. Scientific Reports, 2016, 6: 36890. DOI:10.1038/srep36890 |
| [6] |
Diel DG, da Silva LH, Liu HL, et al. Genetic diversity of avian paramyxovirus type 1: proposal for a unified nomenclature and classification system of Newcastle disease virus genotypes[J]. Infection, Genetics and Evolution, 2012, 12(8): 1770-1779. DOI:10.1016/j.meegid.2012.07.012 |
| [7] |
Du NX, Pan QQ, Zhang SF. The wrong idea of genetic distance and the exploration of the trace of virus evolution[J]. Virologica Sinica, 2005, 20(3): 335-339. 杜念兴, 潘芹芹, 张素芳. 遗传距离的错误观点和病毒进化踪迹的探索[J]. 中国病毒学, 2005, 20(3): 335-339. |
| [8] |
Meng F, Xu HY, Zhang W, et al. Genetic evolution and substitution frequency of avian influenza virus HA gene in chicken H9N2 subtype in China in the last 20 years[J]. Acta Microbiologica Sinica, 2016, 56(1): 35-43. 孟芳, 徐怀英, 张伟, 等. 近20年中国部分地区鸡源H9N2亚型禽流感病毒HA基因遗传演化及其变异频率[J]. 微生物学报, 2016, 56(1): 35-43. |
| [9] |
Xu HY. Antigenicity and genetic variation of Newcastle disease virus isolated from Shandong Province[D]. Beijing: Doctoral Dissertation of China Agricultural University, 2014 (in Chinese) 徐怀英. 新城疫病毒山东流行株分子演化及抗原性变异分析[D]. 北京: 中国农业大学博士学位论文, 2014 http://cdmd.cnki.com.cn/Article/CDMD-10019-1014336309.htm |
| [10] |
Qin ZM, Tan LT, Xu HY, et al. Pathotypical characterization and molecular epidemiology of Newcastle disease virus isolates from different hosts in China from 1996 to 2005[J]. Journal of Clinical Microbiology, 2008, 46(2): 601-611. DOI:10.1128/JCM.01356-07 |
| [11] |
Hu ZL, Hu J, Hu SL, et al. High levels of virus replication and an intense inflammatory response contribute to the severe pathology in lymphoid tissues caused by Newcastle disease virus genotype Ⅶd[J]. Archives of Virology, 2015, 160(3): 639-648. DOI:10.1007/s00705-014-2301-2 |
| [12] |
Liu XF, Hu SL. Molecular epidemiology of Newcastle disease virus and new vaccine development in China[J]. China Poultry, 2010, 32(21): 1-4. 刘秀梵, 胡顺林. 我国新城疫病毒的分子流行病学及新疫苗研制[J]. 中国家禽, 2010, 32(21): 1-4. |
| [13] |
Qin ZM, Xu HY, Liu YS, et al. Prevention and dynamic evolution of antigenic and core genes of Newcastle disease virus[J]. China Poultry, 2011, 33(14): 1-6. 秦卓明, 徐怀英, 刘玉山, 等. 新城疫病毒抗原性和核心基因的动态演化及防控思考[J]. 中国家禽, 2011, 33(14): 1-6. |
| [14] |
Khorajiya JH, Pandey S, Ghodasara PD, et al. Patho-epidemiological study on Genotype-XⅢ Newcastle disease virus infection in commercial vaccinated layer farms[J]. Veterinary World, 2015, 8(3): 372-381. DOI:10.14202/vetworld. |
| [15] |
Samuel A, Nayak B, Paldurai A, et al. Phylogenetic and pathotypic characterization of newcastle disease viruses circulating in west Africa and efficacy of a current vaccine[J]. Journal of Clinical Microbiology, 2013, 51(3): 771-781. DOI:10.1128/JCM.02750-12 |
| [16] |
Snoeck CJ, Owoade AA, Couacy-Hymann E, et al. High genetic diversity of Newcastle disease virus in poultry in West and Central Africa: cocirculation of genotype ⅩⅣ and newly defined genotypes ⅩⅦ and ⅩⅧ[J]. Journal of Clinical Microbiology, 2013, 51(7): 2250-2260. DOI:10.1128/JCM.00684-13 |
| [17] |
Dimitrov KM, Lee DH, Williams-Coplin D, et al. Newcastle disease viruses causing recent outbreaks worldwide show unexpectedly high genetic similarity to historical virulent isolates from the 1940s[J]. Journal of Clinical Microbiology, 2016, 54(5): 1228-1235. DOI:10.1128/JCM.03044-15 |
| [18] |
Lee EK, Jeon WJ, Kwon JH, et al. Molecular epidemiological investigation of Newcastle disease virus from domestic ducks in Korea[J]. Veterinary Microbiology, 2009, 134(3/4): 241-248. |
| [19] |
Hu SL, Sun Q, Wu YT, et al. Attenuation of a genotype Ⅶd Newcastle disease virus ZJI strain of goose origin by reverse genetics[J]. Acta Microbiologica Sinica, 2007, 47(2): 197-200. 胡顺林, 孙庆, 吴艳涛, 等. 用反向遗传技术致弱基因Ⅶd型鹅源新城疫病毒ZJI株[J]. 微生物学报, 2007, 47(2): 197-200. |
| [20] |
Qin ZM. Heterogeneities of pathogenicity and antigenicity in relation to the genetic variation of F and HN genes in prevailed Newcastle disease viruses[D]. Tai'an: Doctoral Dissertation of Shandong Agricultural University, 2006 (in Chinese) 秦卓明. 新城疫病毒流行株致病性和抗原性及其与F和HN基因变异的相关性[D]. 泰安: 山东农业大学博士学位论文, 2006 http://cdmd.cnki.com.cn/Article/CDMD-10434-2006100732.htm |
| [21] |
Connaris H, Takimoto T, Russell R, et al. Probing the sialic acid binding site of the hemagglutinin-neuraminidase of newcastle disease virus: identification of key amino acids involved in cell binding, catalysis, and fusion[J]. Journal of Virology, 2002, 76(4): 1816-1824. DOI:10.1128/JVI.76.4.1816-1824.2002 |
| [22] |
McGinnes LW, Morrison TG. Inhibition of receptor binding stabilizes Newcastle disease virus HN and F protein-containing complexes[J]. Journal of Virology, 2006, 80(6): 2894-2903. DOI:10.1128/JVI.80.6.2894-2903.2006 |
| [23] |
Zhu J. Molecular epidemiology of Newcastle disease viruses isolated in Eastern China during 2008-2013 and the effect of E347K mutation in HN gene on antigenicity[D]. Yangzhou: Doctoral Dissertation of Yangzhou University, 2015 (in Chinese) 朱杰. 2008-2013年华东地区新城疫病毒分子流行病学及HN基因E347K变异对病毒抗原性的影响[D]. 扬州: 扬州大学博士学位论文, 2015 http://cdmd.cnki.com.cn/Article/CDMD-11117-1015657840.htm |
 2017, Vol. 44
2017, Vol. 44