扩展功能
文章信息
- 王永远, 叶剑, 郑志永, 高敏杰, 李佳佳, 詹晓北
- WANG Yong-Yuan, YE Jian, ZHENG Zhi-Yong, GAO Min-Jie, LI Jia-Jia, ZHAN Xiao-Bei
- 土壤杆菌氧调控基因fnrN突变株发酵性能及基于RNA-Seq的基因表达
- Fermentation characteristics of Agrobacterium sp. with oxygen regulation gene fnrN mutation and genes expression analysis based on RNA-Seq
- 微生物学通报, 2017, 44(10): 2261-2268
- Microbiology China, 2017, 44(10): 2261-2268
- DOI: 10.13344/j.microbiol.china.160939
-
文章历史
- 收稿日期: 2016-12-20
- 接受日期: 2017-02-13
- 优先数字出版日期(www.cnki.net): 2017-02-24
热凝胶是一种水不溶性的线性β-(1, 3)葡聚糖[1],具有良好的凝胶特性,有利于保持速冻食品中的纹理和水分[2],因此被广泛地应用于冻肉、面条、饺子和面团等食品中。热凝胶于1996年被FDA批准可用作食品添加剂,此外,热凝胶还具有抗菌、抗病毒、抗过敏和调节免疫力等功能和潜力[3-4]。
土壤杆菌(Agrobacterium sp.) ATCC 31749是热凝胶合成的工业生产菌株,属于革兰氏阴性菌,其合成热凝胶过程可以分为两个阶段:菌体生长期和产胶期。土壤杆菌ATCC 31749在菌体生长期不合成热凝胶;氮源耗尽后菌体停止生长,开始合成热凝胶并分泌到胞外[5]。随着热凝胶在菌体表面或周围的积累,溶氧的传递就变得愈加困难。Zhang等[6]研究发现在微氧和缺氧的情况下,热凝胶的合成受到严重限制,不同溶氧水平下与热凝胶合成相关基因的转录水平分析表明,所考察基因的转录水平随溶氧的增加而增强,溶氧在50%时所考察基因的转录水平最高,细胞内cyoA、catD、fixN、icd、sdhB、mdh、glmM和galU基因的转录水平是低溶氧条件下(5%)的3−6倍;戴小萌等[7]用蛋白质二维电泳技术研究了土壤杆菌在5%、25%、50%和75%溶氧水平下产胶期胞内总蛋白的表达差异,在4个溶氧水平下鉴定出15个显著差异蛋白,其中葡萄糖磷酸变位酶(Pgm)和乳清苷5-磷酸脱羧酶(PyrF)直接参与调控热凝胶的合成;Ruffing等[8]完成了土壤杆菌ATCC 31749基因组的测序工作,并对测序结果进行基因预测和注释,通过基因组学分析发现fnrN作为全局调节因子参与土壤杆菌ATCC 31749热凝胶合成的氧调控过程。在其他微生物中,关于fnrN的研究也有一些报道:Tsoy等[9]通过比较基因组学分析认为,在变形菌纲中,溶氧调节系统包括单组分系统FnrN/FixK和双组分系统FixLJ,其中FnrN作为Crp-Fnr家族转录调节因子,通过铁硫蛋白直接感知胞内溶氧水平,而FixLJ系统响应的是胞外溶氧的变化;在布鲁氏菌中,低氧条件可以诱导fnrN的表达,regA的突变可以使fnrN转录水平在微氧和厌氧条件下分别降低4倍和22倍[10];而根癌土壤杆菌C58中,FnrN通过调控nnrR的表达进而调节亚硝酸盐还原酶和氮氧化物还原酶的表达[11],Ramey等[12]也发现在溶氧限制条件下,根癌土壤杆菌C58中FNR型转录调节因子sinR的表达需要FnrN的参与。
然而,fnrN基因对热凝胶合成的影响及其调控机理尚不清楚。在土壤杆菌ATCC 31749中,fnrN基因GenBank登录号为333790673,编码产物为Crp家族转录调节因子,Blast比对分析发现,土壤杆菌ATCC 31749和根癌土壤杆菌C58中fnrN的一致性达到98%。本研究利用三亲本接合法,把含有fnrN同源臂的自杀式质粒pJQ-fnrN-kan导入土壤杆菌ATCC 31749中,构建ΔfnrN菌,进而考察ΔfnrN菌的生长和发酵特性,最后基于RNA-Seq技术分析土壤杆菌ATCC 31749野生菌和ΔfnrN菌在产胶期相关基因表达转录水平的差异,以此探究fnrN对热凝胶合成的调控机理。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒: 实验采用的菌株和质粒见表 1。| 名称Name | 特性Characteristic | 来源Source |
| strains | ||
| Agrobacterium sp. ATCC 31749 | StrR | Laboratory stock[13] |
| E. coli JM109 | Cloning vector | Laboratory stock |
| E. coli DH5α λ pir | λ pir, cloning vector | Laboratory stock |
| Plasmids | ||
| PRK2013 | KnR, helper plasmid | Laboratory stock |
| pCAMBIA3300 | KnR, with kan gene | Laboratory stock |
| pJQ200KS | GmR, SacB, suicide plasmid | Laboratory stock |
| pMD-fnrN | AmpR, with fnrN gene | This work |
| pMD-kan | KnR, with kan gene | This work |
| pMD-fnrN-kan | AmpR, KnR | This work |
| pJQ-fnrN-kan | KnR, GmR, SacB | This work |
| Primers | Sequences (5′→3′) | Sizes (bp) |
| fnrN-F | CGGGATCCTACACGGCAATCGGCTCGT | 27 |
| fnrN-R | GCTCTAGATCATGAGCAAGGGGCTGCA | 27 |
| kan-F | GTCGACCACACAGGAAACAGACCATGATTGAACAAGATGGATTGC | 45 |
| kan-R | CAAGCTTAGTCCCGCTCAGAAGAAC | 25 |
| fnrN double exchange-F | CATCAGGCTGACCATGTCATCAGG | 24 |
| fnrN double exchange-R | TTTCGGAAGCCGAATACAAACGC | 23 |
| 注:引物序列中带有下划线的大写字母表示酶切位点. Note: Capital letters underlined represent restriction enzyme cutting sites in the sequences of primers. |
||
从土壤杆菌ATCC 31749野生菌基因组中PCR扩增得到fnrN片段,连接克隆载体pMD19-T Simple得到pMD-fnrN,测序显示fnrN片段大小为1 565 bp,将测序结果在NCBI中进行Blast比对,测序结果与Agrobacterium sp. ATCC 31749染色体基因组上fnrN基因序列一致性达到100%,确认克隆得到的基因片段是正确的。
2.2 fnrN突变菌株的筛选通过三亲接合的方法,将自杀式质粒pJQ-fnrN-kan导入到野生菌中,质粒中fnrN-kan两端序列与基因组上fnrN两端发生同源重组,fnrN-kan片段重组到原fnrN的位置,通过抗生素筛选、菌落PCR和测序验证获得ΔfnrN突变株。抗性筛选结果表明突变株能在50 mg/L Kan平板上生长,而野生株不能。PCR显示从fnrN突变株中只扩增到预期大小的fnrN-kan片段,而从野生菌株中只扩增到fnrN片段(图 1)。将扩增得到的fnrN-kan片段进行测序分析,分析显示扩增的条带是fnrN-kan片段,以上三方面结果均证明自杀式质粒整合到基因组DNA上,fnrN基因结构被破坏。

|
| 图 1 PCR产物的凝胶电泳 Figure 1 Gel electrophoresis of the PCR products 注:1:野生菌的fnrN PCR产物;2:ΔfnrN菌的fnrN PCR产物. Note: 1: fnrN PCR product in wild strain; 2: fnrN PCR product in ΔfnrN strain. |
|
|
通过摇瓶发酵实验,比较分析了ΔfnrN突变株与野生菌株在硫酸铵消耗速率和生长期生物量之间的差异,土壤杆菌进入产胶期后,由于氮源耗尽,生物量不再增加,所以只测定了生长期的生物量。如图 2所示,菌体生长期结束时,ΔfnrN突变株生物量(4.76 g/L)仅比野生菌株(4.88 g/L)降低了2.46%,其变化并不明显,属于误差范围;野生菌和ΔfnrN突变菌对硫酸铵的利用速率也基本一致。这说明fnrN的突变并不影响菌体对氮源的利用和菌体生物量的积累。
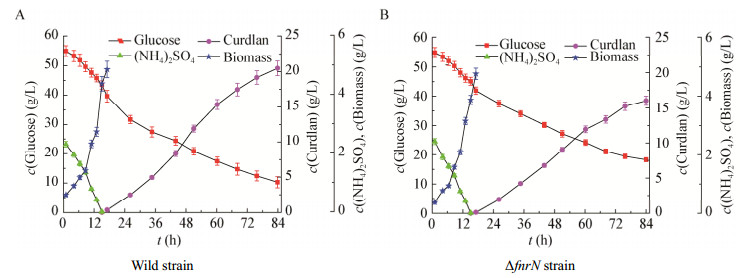
|
| 图 2 土壤杆菌野生菌株(A)和ΔfnrN菌株(B)发酵过程曲线 Figure 2 Fermentation process curves of Agrobacterium sp. wild strain (A) and ΔfnrN (B) |
|
|
UDP-Glucose (UDPG)是热凝胶合成的前体物质,热凝胶的合成需要不断地消耗葡萄糖[16]。实验研究分析了ΔfnrN突变株和野生菌株在葡萄糖利用和热凝胶合成方面的差异。如图 2所示,氮源耗尽后,ΔfnrN和野生菌株均进入产胶期,开始合成热凝胶。在产胶期,ΔfnrN对葡萄糖的消耗速率一直低于野生菌,同时其合成热凝胶的速率也低于野生菌株,特别是在发酵60 h后,ΔfnrN突变株合成热凝胶的速率明显低于野生菌株。
如表 3所示,发酵结束时野生菌的热凝胶产量达到20.5 g/L,突变株只能达到16.0 g/L,同比降低22.0%。土壤杆菌合成热凝胶的理论转化率为0.74[17],在发酵84 h后,突变菌株产物对碳源的转化率比野生菌株降低13.8%。在土壤杆菌ATCC 31749中,碳源的消耗主要用于热凝胶的合成、菌体生长繁殖和有机酸合成或CO2的释放,fnrN基因的突变使土壤杆菌在产胶期对葡萄糖的消耗量由野生菌的30.7 g/L减少为27.8 g/L,降低9.45%,同时产物的生产强度也由301 mg/(L·h)减少为250 mg/(L·h),降低16.9%。野生菌和ΔfnrN菌的实验条件相同,仅仅是fnrN基因发生突变,因此认为fnrN可能参与热凝胶的合成调控。
| 特性 Characteristics |
野生菌株 Wild strain |
ΔfnrN突变株 ΔfnrN strain |
| Glucose consumption in curdlan-producing phase (g/L) | 30.7 | 27.8 |
| Product concentration (g/L) | 20.5 | 16.0 |
| Conversion rate of carbon substrate into product (g/g) | 0.668 | 0.576 |
| Product productivity (mg/(L·h)) | 301 | 250 |
利用荧光定量RT-PCR技术,分析产胶期土壤杆菌野生菌目的基因在50%和5%溶氧条件下的转录水平差异,如图 3所示,与50%溶氧相比,5%溶氧条件下,cydAB和fixK的转录水平提高了2−3倍,fixG和fixH的转录水平提高了8倍,而fixN、fixQ的转录水平提高了23倍,编码生物膜合成调节因子的sinR转录水平提高了17倍。在土壤杆菌ATCC 31749中,fixGHIS共用一个启动子,而fixNOPQ共用一个启动子,因此可以推测,在微氧条件下fixGHIS和fixNOPQ的转录表达水平会显著提高。
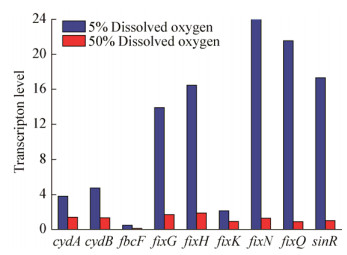
|
| 图 3 RT-PCR结果 Figure 3 Results of RT-PCR |
|
|

|
| 图 4 野生菌与ΔfnrN菌差异表达基因分布 Figure 4 Distribution of differentially expressed genes in wild strain and ΔfnrN strain |
|
|
| 类别 Categories |
基因 Gene |
变化倍数 Fold change |
描述 Description |
| Curdlan biosynthesis | crdA | −1.9 | Hypothetical protein |
| crdS | −1.7 | β-1, 3-glucan synthase catalytic subunit | |
| crdC | −1.3 | Hypothetical protein | |
| ecfR | −2.0 | ECF family RNA polymerase sigma factor | |
| sinR | −69.0 | Fnr family regulator of biofilm formation | |
| C-di-GMP biosynthesis | AGRO_2215 | 1.6 | GGDEF family protein |
| AGRO_1294 | 1.5 | GGDEF family protein | |
| AGRO_1185 | 1.5 | GGDEF family protein | |
| AGRO_4075 | 1.5 | GGDEF family protein | |
| Oxygen signaling associated | cydA | −2.1 | Cytochrome D oxidase |
| cydB | −1.9 | Cytochrome D oxidase | |
| cy2 | −2.4 | Cytochrome C 2 | |
| fixQ | −4.0 | Cytochrome C oxidase | |
| fixP | −3.6 | Cytochrome C oxidase | |
| fixO | −12.4 | Cytochrome C oxidase | |
| fixN | −13.6 | Cytochrome C oxidase | |
| AGRO_4698 | 136.0 | Phosphopantetheinyl transferase |
sinR编码产物是一种生物膜合成调节因子,fnrN突变会使其表达量下调69倍。在与土壤杆菌亲缘关系较近的根癌土壤杆菌中有研究报道,sinR可由微氧条件或fnrN诱导表达,并且sinR的过表达可以加速生物膜的合成[12]。荧光定量PCR分析土壤杆菌野生菌在不同溶氧条件下产胶期样本基因表达差异发现,在5%溶氧条件下sinR基因的表达量是50%溶氧的17倍(图 3)。戴小萌研究发现,在5%溶氧条件下土壤杆菌生产热凝胶的能力会大大降低[7]。fnrN的突变显著影响sinR的表达,而胞外多糖的合成与生物膜有着密不可分的关系,fnrN可能通过调节sinR的表达来调控热凝胶的合成。
核苷酸第二信使(c-di-GMP)是一种全局调节因子,能调控纤维质、褐藻胶等胞外多糖的合成,而合成c-di-GMP的蛋白通常含有GGDEF特征区域。在土壤杆菌基因组中有21个基因编码产物可能含有GGDEF区域,其中只有AGRO_2215、AGRO_1294、AGRO_1185和AGRO_4075在ΔfnrN菌中上调1.5倍以上,但这4个基因是否参与调控热凝胶的合成还需进一步实验的验证。
2.5.3 fnrN突变对氧调控相关基因的影响: cydAB、cy2、fixNOPQ等这些与细胞色素有关的基因在ΔfnrN菌中均显著下调,可能是由于fnrN的突变,氧气的传递过程受阻,细胞中氧信号通路被迫发生改变。在根瘤菌中,fnrNd能在微氧的情况下上调fixNOPQd启动子的表达[19];Schlüter等也发现无论是在独立生存的条件还是在根瘤共生区,在微氧的情况下fixN都会被诱导表达,并且fnrN突变株和fixL突变株中,fixN的表达量受到严重影响[20]。本研究发现,fnrN的突变会使编码细胞色素氧化酶的fixO、fixN的表达量均下调10倍以上,我们认为在土壤杆菌中,fnrN通过调控fixNOPQ的表达参与溶氧调控。在ΔfnrN菌中,AGRO_4698表达上调136倍,其编码产物为磷酸泛酰巯基乙胺基转移酶,与载体蛋白的激活有关。GO (Gene ontology)注释分析表明AGRO_4698与肠菌素的合成、酰基载体蛋白合成酶活性或者镁离子接合有关。微生物系统中,肠菌素是一种亲和力较高的含铁载体,主要结合三价铁离子。而细菌中的铁硫蛋白是氧化应激的传感器[21],还有学者认为细菌主要依靠含有铁硫集群或亚铁血红素的感应蛋白来调节基因的表达以应对外界环境中溶氧的变化[22],fnrN的突变使氧调控的一条途径受阻,但fnrN并不影响细胞生长繁殖,说明AGRO_4698可能参与氧调控的某一支路。
3 结论与讨论土壤杆菌ATCC 31749合成热凝胶是一个非常复杂的过程,其中氮信号级联、以核苷酸为基础的第二信使c-di-GMP、溶氧等都会影响热凝胶的合成。本研究构建了ΔfnrN菌株,发现该基因突变并不影响菌体生长,但突变株合成热凝胶的能力严重减弱,RNA-Seq分析结果发现ΔfnrN菌株中热凝胶合成关键基因的表达量均有所下降,fnrN通过调控ecfR和sinR的表达进而影响热凝胶的合成,并且通过下调fixNOPQ的表达来参与氧信号调控。本研究也发现,fnrN基因突变株在低氧条件下培养一段时间后,其菌液会逐渐变为砖红色,这可能与编码细胞色素相关基因的转录下调有关。
在大肠杆菌中,fnr是Crp-Fnr家族的调节因子,fnr通过铁硫蛋白簇接受氧信号,并调节许多基因的转录来适应外界溶氧的变化[23],对大肠杆菌fnr和土壤杆菌fnrN编码的蛋白质结构域进行分析发现,两者均含有CAP_ED结构域和HTH_CRP (螺旋-转角-螺旋)结构域,这说明fnrN很可能是类似于fnr的一个溶氧调节的全局调节因子。已有文献报道证明氧通过信号转导途径调控微生物多糖的合成,在Agrobacterium tumefaciens中fnrN既与氧信号转导有关,也与琥珀酰葡聚糖合成有关[12]。
本研究通过构建ΔfnrN突变株,证明了fnrN与热凝胶合成的相关性,利用RNA-Seq分析出ΔfnrN菌中的差异表达基因,并对fnrN调控热凝胶合成的可能途径进行分析,发现编码σ因子的ecfR和编码生物膜合成调节因子sinR是主要的中间调节因子。此外,本研究发现了cydAB和fixGHISNOPQ的表达与溶氧有关,并且fnrN通过调控cydAB和fixNOPQ的表达参与氧信号调控。本研究将有利于全面掌握热凝胶合成过程中的氧信号转导途径。
| [1] |
Hua JL, Um HJ, Yin CJ, et al. Proteomic analysis of curdlan-producing Agrobacterium sp. in response to pH downshift[J]. Journal of Biotechnology, 2008, 138(3/4): 80-87. |
| [2] |
Shin HD, Liu L, Kim MK, et al. Metabolic engineering of Agrobacterium sp. ATCC 31749 for curdlan production from cellobiose[J]. Journal of Industrial Microbiology & Biotechnology, 2016, 43(9): 1323-1331. |
| [3] |
Zhu FM, Du B, Xu BJ. A critical review on production and industrial applications of beta-glucans[J]. Food Hydrocolloids, 2016, 52: 275-288. DOI:10.1016/j.foodhyd.2015.07.003 |
| [4] |
Kawashima S, Hirose K, Iwata A, et al. β-glucan curdlan induces IL-10-producing CD4+ T cells and inhibits allergic airway inflammation[J]. The Journal of Immunology, 2012, 189(12): 5713-5721. DOI:10.4049/jimmunol.1201521 |
| [5] |
Gummadi SN, Kumar K. Production of extracellular water insoluble β-1, 3-glucan (Curdlan) from Bacillus sp. SNC07[J]. Biotechnology and Bioprocess Engineering, 2005, 10(6): 546-551. DOI:10.1007/BF02932292 |
| [6] |
Zhang HT, Zhan XB, Zheng ZY, et al. Sequence and transcriptional analysis of the genes responsible for curdlan biosynthesis in Agrobacterium sp. ATCC 31749 under simulated dissolved oxygen gradients conditions[J]. Applied Microbiology and Biotechnology, 2011, 91(1): 163-175. DOI:10.1007/s00253-011-3243-1 |
| [7] |
Dai XM, Yang LB, Zheng ZY, et al. Proteomic analysis of curdlan-producing Agrobacterium sp. ATCC 31749 in response to dissolved oxygen[J]. Acta Microbiologica Sinica,, 2015, 55(8): 1018-1025. 戴小萌, 杨利博, 郑志永, 等. 溶氧影响土壤杆菌ATCC 31749发酵生产热凝胶的蛋白质组学[J]. 微生物学报, 2015, 55(8): 1018-1025. |
| [8] |
Ruffing AM. Metabolic engineering and omics analysis of Agrobacterium sp. ATCC 31749 for oligosaccharide synthesis[D]. Atlanta: Doctoral Dissertation of Georgia Institute of Technology, 2010
|
| [9] |
Tsoy OV, Ravcheev DA, Čuklina J, et al. Nitrogen fixation and molecular oxygen: comparative genomic reconstruction of transcription regulation in Alphaproteobacteria[J]. Frontiers in Microbiology, 2016, 7: 1343. |
| [10] |
Abdou E, Deredjian A, de Bagüés MPJ, et al. RegA, the regulator of the two-component system RegB/RegA of Brucella suis, is a controller of both oxidative respiration and denitrification required for chronic infection in mice[J]. Infection and Immunity, 2013, 81(6): 2053-2061. DOI:10.1128/IAI.00063-13 |
| [11] |
Baek SH, Hartsock A, Shapleigh JP. Agrobacterium tumefaciens C58 uses ActR and FnrN to control nirK and nor expression[J]. Journal of Bacteriology, 2008, 190(1): 78-86. DOI:10.1128/JB.00792-07 |
| [12] |
Ramey BE, Matthysse AG, Fuqua C. The FNR-type transcriptional regulator SinR controls maturation of Agrobacterium tumefaciens biofilms[J]. Molecular Microbiology, 2004, 52(5): 1495-1511. DOI:10.1111/j.1365-2958.2004.04079.x |
| [13] |
Yu LJ, Wu JR, Liu J, et al. Enhanced curdlan production in Agrobacterium sp. ATCC 31749 by addition of low-polyphosphates[J]. Biotechnology and Bioprocess Engineering, 2011, 16(1): 34-41. DOI:10.1007/s12257-010-0145-5 |
| [14] |
Yu LJ, Lu J, Wu JR, et al. Influence of low-polyphosphates on curdlan production by Alcaligenes faecalis var. myxogene[J]. Microbiology China, 2010, 37(5): 664-670. 于丽珺, 路敬, 吴剑荣, 等. 低聚磷酸盐对粪产碱杆菌合成热凝胶的影响[J]. 微生物学通报, 2010, 37(5): 664-670. |
| [15] |
Zhou X, Wang SY, Song LX, et al. Developing complex probiotics used in treating resoluble nitrogen wastewater from seawater culture[J]. Microbiology China, 2010, 37(4): 543-546. 周鑫, 王素英, 宋霖霞. 海水养殖含氮废水处理的复合微生态制剂研制[J]. 微生物学通报, 2010, 37(4): 543-546. |
| [16] |
Zhan XB, Lin CC, Zhang HT. Recent advances in curdlan biosynthesis, biotechnological production, and applications[J]. Applied Microbiology and Biotechnology, 2012, 93(2): 525-531. DOI:10.1007/s00253-011-3740-2 |
| [17] |
Zheng ZY, Lee JW, Zhan XB, et al. Effect of metabolic structures and energy requirements on curdlan production by Alcaligenes faecalis[J]. Biotechnology and Bioprocess Engineering, 2007, 12(4): 359-365. DOI:10.1007/BF02931057 |
| [18] |
Mortazavi A, Williams BA, McCue K, et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq[J]. Nature Methods, 2008, 5(7): 621-628. DOI:10.1038/nmeth.1226 |
| [19] |
Lopez O, Morera C, Miranda-Rios J, et al. Regulation of gene expression in response to oxygen in Rhizobium etli: role of FnrN in fixNOQP expression and in symbiotic nitrogen fixation[J]. Journal of Bacteriology, 2001, 183(24): 6999-7006. DOI:10.1128/JB.183.24.6999-7006.2001 |
| [20] |
Schlüter A, Patschkowski T, Quandt J, et al. Functional and regulatory analysis of the two copies of the fixNOQP operon of Rhizobium leguminosarum strain VF39[J]. Molecular Plant-Microbe Interactions, 1997, 10(5): 605-616. DOI:10.1094/MPMI.1997.10.5.605 |
| [21] |
Kobayashi K, Fujikawa M, Kozawa T. Oxidative stress sensing by the iron-sulfur cluster in the transcription factor, SoxR[J]. Journal of Inorganic Biochemistry, 2014, 133: 87-91. DOI:10.1016/j.jinorgbio.2013.11.008 |
| [22] |
Taabazuing CY, Hangasky JA, Knapp MJ. Oxygen sensing strategies in mammals and bacteria[J]. Journal of Inorganic Biochemistry, 2014, 133: 63-72. DOI:10.1016/j.jinorgbio.2013.12.010 |
| [23] |
Kiley PJ, Beinert H. Oxygen sensing by the global regulator, FNR: the role of the iron-sulfur cluster[J]. FEMS Microbiology Reviews, 1998, 22(5): 341-352. DOI:10.1111/j.1574-6976.1998.tb00375.x |
 2017, Vol. 44
2017, Vol. 44




