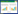扩展功能
文章信息
- 廖恒毅, 王旭昊, 崔逸儒, 李晓翠, 王红岩, 侯贺磊, 王晶晶, 李秀颖, 杨毅
- LIAO Hengyi, WANG Xuhao, CUI Yiru, LI Xiaocui, WANG Hongyan, HOU Helei, WANG Jingjing, LI Xiuying, YANG Yi
- 还原性脱卤酶的生物信息学分析
- Bioinformatics analysis of reductive dehalogenases
- 微生物学通报, 2024, 51(12): 5105-5120
- Microbiology China, 2024, 51(12): 5105-5120
- DOI: 10.13344/j.microbiol.china.240533
-
文章历史
- 收稿日期: 2024-06-30
- 接受日期: 2024-12-03
- 网络首发日期: 2024-12-10
2. 中国科学院沈阳应用生态研究所 森林生态与保育重点实验室, 辽宁 沈阳 110016;
3. 中国科学院大学, 北京 100049;
4. 沈阳药科大学, 辽宁 沈阳 110016
2. Key Laboratory of Forest Ecology and Conservation, Institute of Applied Ecology, Chinese Academy of Sciences, Shenyang 110016, Liaoning, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China;
4. Shenyang Pharmaceutical University, Shenyang 110016, Liaoning, China
有机卤化物(halogenated organic compound, HOC)是指含有氟、氯、溴或碘等卤素元素的有机化合物[1]。原始自然环境中,有机卤化物可通过生物体(例如细菌、真菌和植物)、生物量燃烧、火山活动及其他地热活动形成,是卤素生物地球化学循环不可或缺的一部分[2-3]。目前,由于其优异的理化特性,有机卤化物被广泛应用于工农业等众多领域[4-5]。然而,由于其不合理的生产及不当的释放,加之有机卤化物通常具有化学稳定性强、难降解、全球转移及生物富集等特点,导致环境中这些化合物的累积量越来越多[6-9]。这些有机卤化物随后可通过食物网的生物积累和生物放大作用,对人类健康和生态系统功能产生不同程度的潜在风险和危害[10-12]。
有机卤呼吸细菌(organohalide-respiring bacteria, OHRB)与还原性脱卤酶(reductive dehalogenases, RDases)是有机卤化物生物修复的关键工具[13]。OHRB能以有机卤化物作为电子受体,以氢气或甲酸、乙酸等小分子有机酸作为电子供体,基于电子传递过程裂解有机卤化物中的碳-卤素键[14-17]。有机卤取代基以无机卤的形式被释放,有机卤化物进而可被转化为无毒或低毒的终产物,或是生物降解性更高的中间产物[18]。而RDases作为有机卤呼吸的末端电子受体还原酶,是该过程的核心[15, 19-20]。
RDases由催化亚基(RdhA)和膜锚定蛋白(RdhB)两部分组成[21]。其中,RdhA是催化亚基,具有钴胺素辅因子和2个Fe-S簇[13]。该亚基可通过膜锚定亚基RdhB锚定到细胞质膜[21]。OHRB的基因组中存在许多编码RDases的基因,其功能与结构的多样性促使不同OHRB参与到不同的脱卤过程中。然而,大多数RDases基因尚未进行功能鉴定。迄今为止,RDases生化研究仍因其纯化与表征方面的困难而受阻,其活性位点、电子传递的组成部分及序列结构特征等相关信息也知之甚少,这从根源上制约了OHRB在有机卤污染场地原位修复中的实际应用及相关修复技术的发展。
本研究通过生物信息学分析方法对44个RDases蛋白的基本理化性质、跨膜结构、信号肽、磷酸化位点、系统发育、多序列比对的同源性矩阵、保守基序、保守结构域、二级结构、三级结构与无序区域进行预测分析,以期为发现更多新型RDases并研究其功能奠定基础,也为基于RDases的OHRB应用于实际场地修复提供科学依据与理论参考。
1 材料与方法 1.1 还原性脱卤酶序列的收集本研究以44个RDases蛋白氨基酸编码序列作为分析对象(蛋白名称与登录号见表 1)。其中,39个RDases蛋白氨基酸编码序列来源于还原性脱卤酶数据库(http://RDaseDB.biozone.utoronto.ca)[22]。5个RDases蛋白氨基酸编码序列[PceA-Lab、DcaA-Lab、TcaA-Lab、DcpA(1)-Lab与DcpA(2)-Lab]来自本实验室。
| 编号 No. |
名称 Name |
登录号 Accession number |
编号 No. |
名称 Name |
登录号 Accession number |
|
| 1 | 3-CBA RDase | AFM24124 | 23 | PceA-4 | AAO60101 | |
| 2 | 3-C-4-H-P RDase | WP_015942994 | 24 | PceA-5 | WP_011460641 | |
| 3 | BvcA | AAT64888 | 25 | PceA-6 | AAW40342 | |
| 4 | CbrA | CAI82345 | 26 | PceA-7 | AHF10727 | |
| 5 | CerA | PMP99143 | 27 | PceA-8 | ABV38373 | |
| 6 | CfrA | AFV05253 | 28 | PceA-9 | AHJ12791 | |
| 7 | CprA-1 | BAE45337 | 29 | PceA-DCE | AGW23613 | |
| 8 | CprA-2 | AAG49544 | 30 | PceA-TCE | AGW23615 | |
| 9 | CprA-3 | AAG46187 | 31 | PdrA | BAE45338 | |
| 10 | CprA-4 | AAQ54585 | 32 | TcbA | WP_068882928 | |
| 11 | CprA-5 | AAL84925 | 33 | TceA-1 | AIZ97109 | |
| 12 | CrdA | AAK95329 | 34 | TceA-2 | AAW39060 | |
| 13 | CtrA | AGO27983 | 35 | TdrA | AKG53095 | |
| 14 | DcaA | CAJ75430 | 36 | TmrA | WP_034377773 | |
| 15 | DcpA | WP_013218938 | 37 | VcrA-1 | AQY73737 | |
| 16 | DcrA | AFV02209 | 38 | VcrA-2 | AOV99943 | |
| 17 | DebcprA | AGC09147 | 39 | VcrA-3 | WP_012882535 | |
| 18 | MbrA | ADF96893 | 40 | PceA-Lab | WP_278545147 | |
| 19 | NpRdhA | EKF18105 | 41 | DcaA-Lab | MDV2988852 | |
| 20 | PceA-1 | AAC60788 | 42 | TcaA-Lab | MDJ0306437 | |
| 21 | PceA-2 | AAG49543 | 43 | DcpA(1)-Lab | WZD84542 | |
| 22 | PceA-3 | CAD28792 | 44 | DcpA(2)-Lab | WZD84543 | |
| 3-C-4-H-P RDase: 3-chloro-4-hydroxy-phenylacetate reductive dehalogenase. | ||||||
44个RDases蛋白氨基酸编码序列采用各种生物信息学分析工具对其序列与结构特征进行分析预测,具体生物信息学分析工具见表 2。
| 工具名称 Tool name |
网址 Website |
功能 Function |
| ProtParam | https://web.expasy.org/protparam/ | 蛋白的分子量、理论等电点(pI)、氨基酸组成、不稳定指数、总平均亲水指数和脂肪族指数预测 Prediction of proteinʼs molecular weight, theoretical isoelectric point (pI), amino acid composition, instability index, grand average of hydropathicity, and aliphatic index |
| ProtScale | https://web.expasy.org/protscale/ | 蛋白的亲水性与疏水性预测 Prediction of protein hydrophilicity and hydrophobicity |
| DeepTMHMM 1.0 | https://services.healthtech.dtu.dk/services/DeepTMHMM-1.0/ | 蛋白跨膜结构预测 Prediction of protein transmembrane structure |
| SignalP 6.0 | https://services.healthtech.dtu.dk/services/SignalP-6.0/ | 蛋白信号肽预测 Prediction of protein signal peptide |
| NetPhosBac 1.0 | https://services.healthtech.dtu.dk/services/NetPhosBac-1.0/ | 蛋白的丝氨酸、苏氨酸、酪氨酸3种磷酸化位点预测 Prediction of phosphorylation sites on protein serine, threonine, and tyrosine |
| MEME | https://meme-suite.org/meme/ | 蛋白保守基序预测 Prediction of protein conserved motif |
| Pfam | http://pfam-legacy.xfam.org/ | 蛋白保守结构域预测 Prediction of protein conserved domain |
| SOPMA | https://npsa.lyon.inserm.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html | 蛋白二级结构预测 Prediction of protein secondary structure |
| AlphaFold 3 | https://alphafoldserver.com/ | 蛋白三级结构预测 Prediction of protein tertiary structure |
| PyMOL | – | 蛋白三级结构的可视化 Visualization of protein tertiary structure |
| DISOPRED3 | http://bioinf.cs.ucl.ac.uk/psipred/ | 蛋白无序区域预测 Prediction of protein disordered region |
| DNAMAN; MEGA 11 | – | 蛋白多序列比对与进化树构建 Protein multiple sequence alignments and phylogenetic tree construction |
| TBtools | – | 蛋白保守基序与保守结构域的可视化 Visualization of protein conserved motif and domain |
| RStudio | – | 部分分析结果的可视化 Visualization of partial analysis results |
| Adobe Illustrator | – | 图片的组装与美化 Assembly and enhancement of figures |
| –:非在线工具 –: Non-online tools. |
||
如图 1A所示,不同RDases蛋白的分子量存在一定差异。其中,NpRdhA蛋白的分子量最高,为76 503.1 Da;而CrdA和DebcprA蛋白的分子量较低,分别为35 456.4 Da和36 088.7 Da。其余RDases蛋白分子量均在49−65 kDa之间。由图 1B可知,不同RDases蛋白的理论等电点也有所不同。其中,DebcprA、3-CBA RDase、DcaA与CprA-5蛋白的理论等电点较高,分别为9.15、8.42、8.28与8.02。其余RDases蛋白的理论等电点均在5.5−8.0之间。各个RDases蛋白的脂肪族指数也有所差异(图 1C)。其中,DebcprA蛋白的脂肪族指数较高,为93.24。其余RDases蛋白的脂肪族指数均在56−78之间。蛋白的亲水性是维持其稳定性和功能的基础。由图 1D可知,不同RDases蛋白的总平均亲水指数在–0.509−–0.044之间,均为亲水性蛋白。由图 1E可知,除TcaA-Lab、CtrA、TmrA与CfrA这4个酶蛋白外,其余RDases蛋白的不稳定系数均小于40,属于稳定蛋白。
|
| 图 1 44个还原性脱卤酶的基本理化性质 Figure 1 The basic physicochemical properties of 44 reductive dehalogenases. A: The molecular weights of 44 reductive dehalogenases. B: Theoretical isoelectric points (pI) of 44 reductive dehalogenases. C: The aliphatic indices of 44 reductive dehalogenases. D: The grand average of hydropathicity for 44 reductive dehalogenases. E: The instability index of 44 reductive dehalogenases. F: The number of amino acids in 44 reductive dehalogenases. A:44个还原性脱卤酶的分子量. B:44个还原性脱卤酶的理论等电点. C:44个还原性脱卤酶的脂肪族指数. D:44个还原性脱卤酶的总平均亲水指数. E:44个还原性脱卤酶的不稳定指数. F:44个还原性脱卤酶的氨基酸数量 |
|
|
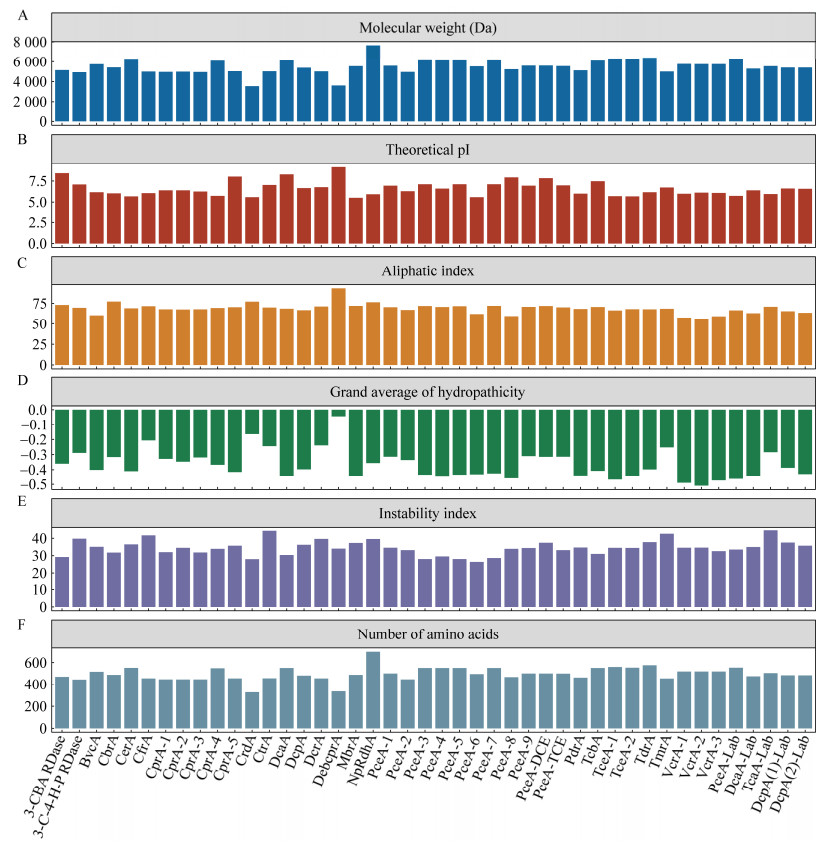
由图 1F可知,不同RDases蛋白的氨基酸数量也存在一定差异。在44个RDases蛋白中,NpRdhA蛋白的氨基酸数量最多,共含698个氨基酸。而CrdA与DebcprA蛋白的氨基酸数量较少,分别具有327个和336个氨基酸。其余RDases蛋白的氨基酸数量均在445−576之间。如图 2所示,44个RDases蛋白的氨基酸组成具有一定的相似性。在不同RDases蛋白中,丙氨酸(Ala)、甘氨酸(Gly)与赖氨酸(Lys)均为含量占比较高的氨基酸。
|
| 图 2 44个还原性脱卤酶的氨基酸组成 Figure 2 Amino acid compositions of 44 reductive dehalogenases. Ala: Alanine; Cys: Cysteine; His: Histidine; Met: Methionine; Thr: Threonine; Arg: Arginine; Gln: Glutamine; Ile: Isoleucine; Phe: Phenylalanine; Trp: Tryptophan; Asn: Asparagine; Glu: Glutamic acid; Leu: Leucine; Pro: Proline; Tyr: Tyrosine; Asp: Aspartic acid; Gly: Glycine; Lys: Lysine; Ser: Serine; Val: Valine. Ala:丙氨酸;Cys:半胱氨酸;His:组氨酸;Met:甲硫氨酸;Thr:苏氨酸;Arg:精氨酸;Gln:谷氨酰胺;Ile:异亮氨酸;Phe:苯丙氨酸;Trp:色氨酸;Asn:天冬酰胺;Glu:谷氨酸;Leu:亮氨酸;Pro:脯氨酸;Tyr:酪氨酸;Asp:天冬氨酸;Gly:甘氨酸;Lys:赖氨酸;Ser:丝氨酸;Val:缬氨酸 |
|
|
通过对44个RDases蛋白的跨膜结构进行预测分析发现,除TcaA-Lab蛋白外,其余43个RDases蛋白均无跨膜结构。这意味着绝大多数RDases蛋白并非膜定位蛋白,在有机卤呼吸细菌的生命过程中并不能发挥跨膜蛋白功能作用(如信号传递、物质转运和细胞通信等)。
44个RDases蛋白的信号肽预测分析结果如表 3所示。NpRdhA、DcaA-Lab与TcaA-Lab的蛋白序列并无明显信号肽,为非经典型分泌蛋白。其余RDases蛋白均存在信号肽,为分泌蛋白。其中,仅CrdA存在Sec转运蛋白转运并被信号肽酶Ⅰ切割的“标准”分泌信号肽(Sec/SPI),其切割位点位于24−25位氨基酸之间;而其余RDases蛋白均只存在由Tat转运蛋白转运并被信号肽酶Ⅰ切割的Tat信号肽(Tat/SPI),其切割位点大多数在40−50位氨基酸之间。
| 蛋白名称 Protein name |
信号肽类型 Signal peptide type |
切割位点 Cleavage site |
蛋白名称 Protein name |
信号肽类型 Signal peptide type |
切割位点 Cleavage site |
|
| 3-CBA RDase | Tat/SPI | 39–40 | PceA-4 | Tat/SPI | 39–40 | |
| 3-C-4-H-P RDase | Tat/SPI | 42–43 | PceA-5 | Tat/SPI | 39–40 | |
| BvcA | Tat/SPI | 43–44 | PceA-6 | Tat/SPI | 42–43 | |
| CbrA | Tat/SPI | 35–36 | PceA-7 | Tat/SPI | 39–40 | |
| CerA | Tat/SPI | 31–32 | PceA-8 | Tat/SPI | 47–48 | |
| CfrA | Tat/SPI | 53–54 | PceA-9 | Tat/SPI | 37–38 | |
| CprA-1 | Tat/SPI | 42–43 | PceA-DCE | Tat/SPI | 37–38 | |
| CprA-2 | Tat/SPI | 42–43 | PceA-TCE | Tat/SPI | 37–38 | |
| CprA-3 | Tat/SPI | 42–43 | PdrA | Tat/SPI | 49–50 | |
| CprA-4 | Tat/SPI | 27–28 | TcbA | Tat/SPI | 39–40 | |
| CprA-5 | Tat/SPI | 51–52 | TceA-1 | Tat/SPI | 40–41 | |
| CrdA | Sec/SPI | 24–25 | TceA-2 | Tat/SPI | 34–35 | |
| CtrA | Tat/SPI | 53–54 | TdrA | Tat/SPI | 55–56 | |
| DcaA | Tat/SPI | 39–40 | TmrA | Tat/SPI | 53–54 | |
| DcpA | Tat/SPI | 45–46 | VcrA-1 | Tat/SPI | 35–36. | |
| DcrA | Tat/SPI | 53–54 | VcrA-2 | Tat/SPI | 29–30 | |
| DebcprA | Tat/SPI | 39–40 | VcrA-3 | Tat/SPI | 35–36 | |
| MbrA | Tat/SPI | 28–29 | PceA-Lab | Tat/SPI | 34–35 | |
| NpRdhA | – | – | DcaA-Lab | – | – | |
| PceA-1 | Tat/SPI | 37–38 | TcaA-Lab | – | – | |
| PceA-2 | Tat/SPI | 42–43 | DcpA(1)-Lab | Tat/SPI | 40–41 | |
| PceA-3 | Tat/SPI | 39–40 | DcpA(2)-Lab | Tat/SPI | 45–46 | |
| 数字代表氨基酸位点;–:不含信号肽 Numbers represent amino acid sites; –: No signal peptide. |
||||||
磷酸化修饰对于细胞生长、分化、凋亡和细胞信号转导等多种细胞过程的调节均具有重要作用,是目前研究最为广泛的蛋白质翻译后修饰(post-translational modification, PTM)之一[23-24]。44个RDases蛋白的磷酸化位点预测结果如表 4所示。结果表明,磷酸化位点在RDases蛋白序列中普遍存在,且主要以丝氨酸(serine)为主,而苏氨酸(threonine)较少。磷酸化位点的普遍存在表明RDases蛋白很可能是通过自身磷酸化来调节有机卤呼吸相关的生物学过程。
| 蛋白名称 Protein name |
丝氨酸 Serine |
苏氨酸 Threonine |
蛋白名称 Protein name |
丝氨酸 Serine |
苏氨酸 Threonine |
蛋白名称 Protein name |
丝氨酸 Serine |
苏氨酸 Threonine |
||
| 3-CBA RDase | 11 | 0 | DcrA | 6 | 0 | PdrA | 10 | 1 | ||
| 3-C-4-H-P RDase | 8 | 1 | DebcprA | 5 | 1 | TcbA | 5 | 0 | ||
| BvcA | 9 | 0 | MbrA | 7 | 0 | TceA-1 | 12 | 2 | ||
| CbrA | 13 | 2 | NpRdhA | 7 | 1 | TceA-2 | 10 | 1 | ||
| CerA | 10 | 1 | PceA-1 | 5 | 2 | TdrA | 13 | 2 | ||
| CfrA | 6 | 0 | PceA-2 | 4 | 1 | TmrA | 7 | 0 | ||
| CprA-1 | 4 | 1 | PceA-3 | 5 | 0 | VcrA-1 | 6 | 2 | ||
| CprA-2 | 4 | 0 | PceA-4 | 7 | 0 | VcrA-2 | 6 | 2 | ||
| CprA-3 | 4 | 1 | PceA-5 | 5 | 0 | VcrA-3 | 6 | 2 | ||
| CprA-4 | 10 | 1 | PceA-6 | 7 | 2 | PceA-Lab | 13 | 1 | ||
| CprA-5 | 8 | 0 | PceA-7 | 5 | 0 | DcaA-Lab | 12 | 2 | ||
| CrdA | 1 | 1 | PceA-8 | 8 | 0 | TcaA-Lab | 8 | 0 | ||
| CtrA | 6 | 0 | PceA-9 | 5 | 2 | DcpA(1)-Lab | 12 | 2 | ||
| DcaA | 7 | 0 | PceA-DCE | 6 | 1 | DcpA(2)-Lab | 13 | 3 | ||
| DcpA | 11 | 2 | PceA-TCE | 7 | 1 |
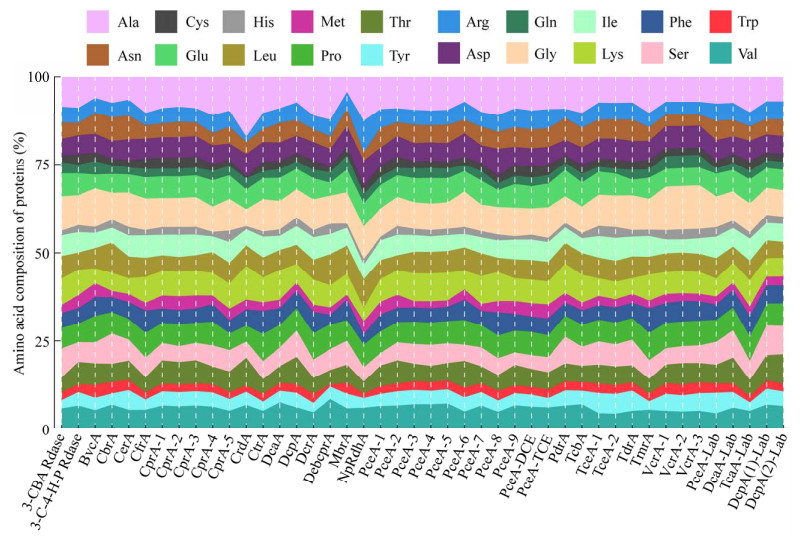
为分析44个RDases蛋白的亲缘关系,本研究基于邻近(neighbor-joining, NJ)法构建了44个RDases蛋白的系统发育树,检验方法为步长检验方法(bootstrap method),检验次数1 000次。由图 3A可知,多数RDases蛋白间的进化关系较近。CrdA单个蛋白独立成分支,这或许是由于其蛋白序列与其它序列差异较大所致。44个RDases蛋白多序列比对的同源性矩阵结果如图 3B所示。结果表明,具有相似或相同底物的RDases蛋白序列具有更高的相似性,而其他RDases蛋白序列之间的相似性则相对较低。此外,多序列比对结果还显示,44个RDases蛋白间的保守氨基酸所占的比例也不高,并且分散在整个序列长度范围内。
|
| 图 3 44个还原性脱卤酶的系统发育分析(A)与同源性矩阵(B) Figure 3 Phylogenetic analysis (A) and homology matrix (B) of 44 reductive dehalogenases. |
|
|
应用在线工具MEME对44个RDases蛋白的保守基序进行了预测分析,共获得了10个保守基序,分别命名为Motif 1−Motif 10 (保守基序的序列信息见表 5)。由图 4A可知,在44个RDases蛋白的保守基序中,Motif 1−Motif 7的分布位置与顺序高度相似且保守性较高。
| 保守基序名 Conserved motif name |
氨基酸序列 Amino acid sequence |
| Motif 1 | TDLPLAPDKPIDFGVREFCKTCKKCADAC |
| Motif 2 | GSSCGNCVAVCPFNK |
| Motif 3 | EENSQMVKKAARFFGADLVGI |
| Motif 4 | VPFAVQAGLGEASRTG |
| Motif 5 | HSTISRRBFLKVLGLAGAGAA |
| Motif 6 | VSTTPVFNSFFKKMDDAFGYG |
| Motif 7 | SYSQMGEVAYKLAQFJRGLGYNAVPS |
| Motif 8 | KPKSVIVMMIEQDYETMKTSPSYLGSAAA |
| Motif 9 | EFPYKVDAKYQRFNSLKNFFEKAFDPEPNKTPJKFHFDDVS |
| Motif 10 | NSGTLRWYVDSNKCLEFWATN |
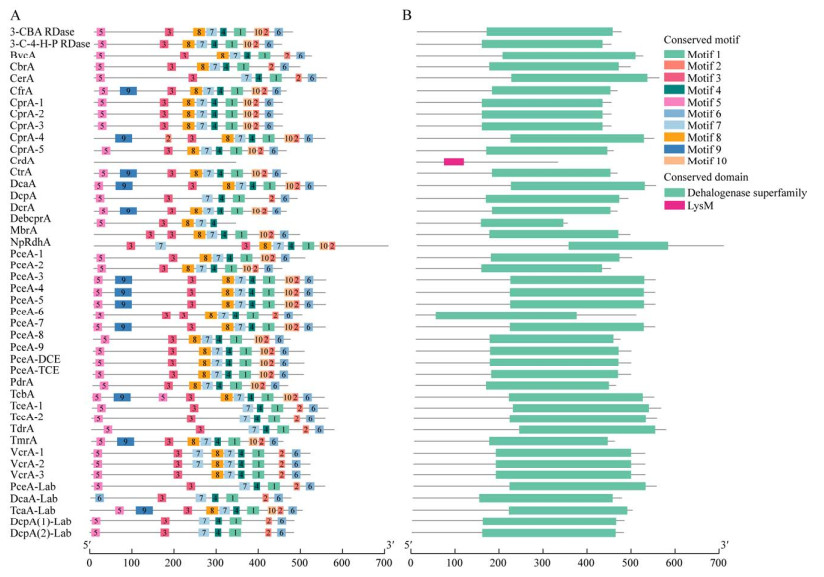
|
| 图 4 44个还原性脱卤酶的保守基序(A)与保守结构域(B)分析 Figure 4 Conserved motifs (A) and conserved domains (B) analysis of 44 reductive dehalogenases. |
|
|
应用在线工具Pfam对44个RDases蛋白的保守结构域进行预测分析,结果如图 4B所示。除CrdA蛋白外,其余RDases蛋白均包含dehalogenase super family结构域。LysM结构域特定存在于CrdA蛋白中,可能与其特定的还原脱卤功能或独特的作用方式有关。
2.5 蛋白质的二级结构与三级结构预测44个RDases蛋白的二级结构预测结果如图 5所示。结果表明,不同RDases蛋白的二级结构具有明显的相似性。在各个RDases蛋白中,无规卷曲(random coil)的比例均占据了绝对优势,其次为α螺旋(α-helix)。而延伸链(extended)与β-转角(β-turn)的占比较小。由此可见,RDases蛋白二级结构的相似性大于其蛋白序列的相似性。然而,尽管二级结构较为保守,部分RDases蛋白的底物范围差异仍然显著。这表明,某些氨基酸(尤其是在结构域和活性位点上的残基)的差异可能是决定功能特征的关键因素。
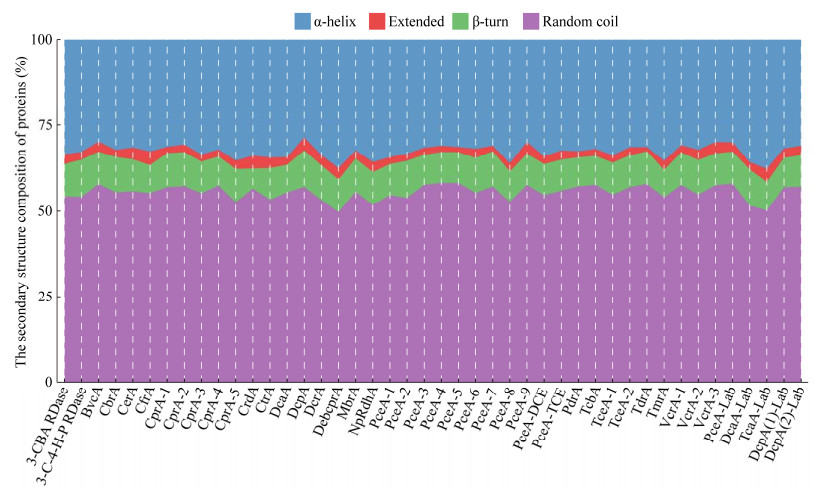
|
| 图 5 44个还原性脱卤酶的二级结构预测结果 Figure 5 The predicted secondary structure results of 44 reductive dehalogenases. |
|
|
应用AlphaFold 3和PyMOL对44个RDases蛋白进行预测建模,结果如图 6所示。分析表明,RDases蛋白三级结构与其对应的脱卤底物类别似乎有着密切联系。底物相似性较高的RDases蛋白在三级结构上表现出较高的保守性,这可能反映了它们在底物识别和催化机制上的相似性。然而,尽管部分RDases蛋白在整体的三级结构上表现出较高的保守性,其拓扑结构与聚合角度等仍存在一定差异。这些微小但关键的结构差异可能是决定其底物范围、催化效率及催化产物类型差异的重要因素。

|
| 图 6 44个还原性脱卤酶的三级结构预测结果 Figure 6 The predicted tertiary structure results of 44 reductive dehalogenases. |
|
|
长期以来形成了“序列-结构-功能”的研究范式,即蛋白质氨基酸序列决定其三维结构,进而决定其生物学功能[25-26]。伴随着研究的深入,不断有研究证实某些在生理条件下缺乏有序稳定的高级结构的蛋白质/蛋白区域也能够参与多种生物学过程,行使特定的生物学功能,这类具有生物活性的蛋白/蛋白区域被称为固有无序蛋白(intrinsically disordered protein, IDP)/固有无序区域(intrinsically disordered region, IDR)[25, 27]。使用DISOPRED3在线工具对44个RDases蛋白进行无序区域的预测,发现这些蛋白质均存在无序区域。除DcpA、MbrA、NpRdhA、DcaA-Lab、TcaA-Lab、DcpA(1)-Lab与DcpA(2)-Lab外,其余37个RDases蛋白均存在由连续30个以上的氨基酸组成的无序区域,属于部分无序的固有无序蛋白[28]。此外,预测结果中RDases蛋白的无序区域又根据是否具有蛋白质的结合位点分为“disordered”与“disordered, protein binding”两种类型。预测结果显示(图 7A),44个RDases蛋白的无序区域中均存在蛋白质结合位点,提示这些无序区域可能在RDases蛋白与底物结合的调控网络中发挥重要作用。尽管这些蛋白的氨基酸组成与二级结构在不同酶之间具有较高的保守性,然而不同底物的RDases蛋白的“disordered, protein binding”占比以及氨基酸组成等无序区域特征(图 7A–7C)表现出显著差异。这一现象进一步暗示了无序区域在决定脱卤酶底物特异性方面可能发挥着关键作用。另外,即使是对应相同底物的RDases蛋白,其无序区域特征依然存在一定的差异,这可能是导致某些具有相同底物的RDases蛋白在进化树中出现在不同分支,并且在催化效率和催化产物类型上有所不同的原因之一。
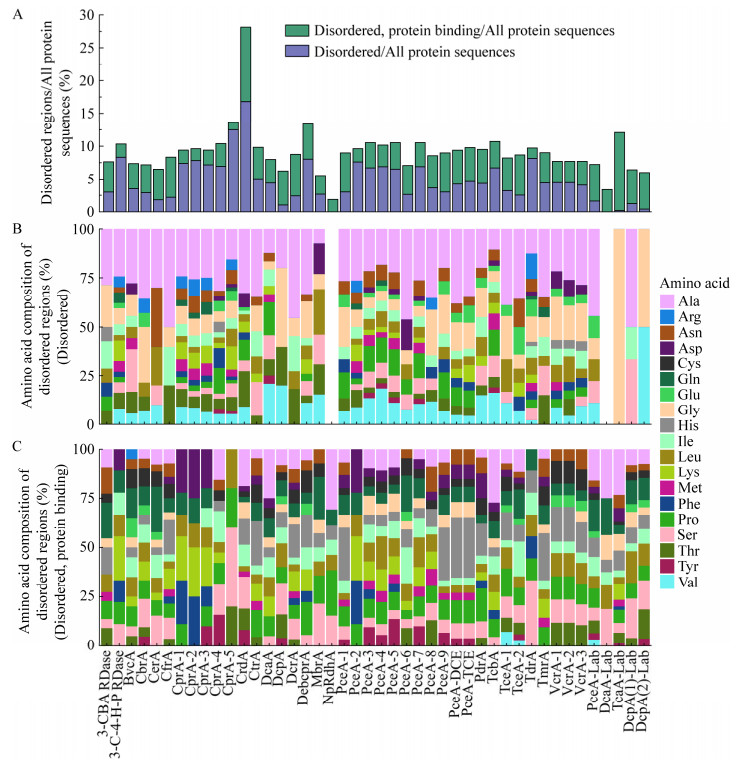
|
| 图 7 44个还原性脱卤酶的无序区域特征 Figure 7 Characteristics of disordered regions of 44 reductive dehalogenases. A: Composition of disordered regions of 44 reductive dehalogenases. B: Amino acid composition of disordered regions (disordered). C: Amino acid composition of disordered regions (Disordered, protein binding). Ala: Alanine; Arg: Arginine; Asn: Asparagine; Asp: Aspartic acid; Cys: Cysteine; Gln: Glutamine; Glu: Glutamic acid; Gly: Glycine; His: Histidine; Ile: Isoleucine; Leu: Leucine; Lys: Lysine; Met: Methionine; Phe: Phenylalanine; Pro: Proline; Ser: Serine; Thr: Threonine; Tyr: Tyrosine; Val: Valine. A:44个还原性脱卤酶的无序区域组成. B:无序区域(disordered)的氨基酸组成. C:无序区域(disordered, protein binding)的氨基酸组成. Ala:丙氨酸;Arg:精氨酸;Asn:天冬酰胺;Asp:天冬氨酸;Cys:半胱氨酸;Gln:谷氨酰胺;Glu:谷氨酸;Gly:甘氨酸;His:组氨酸;Ile:异亮氨酸;Leu:亮氨酸;Lys:赖氨酸;Met:甲硫氨酸;Phe:苯丙氨酸;Pro:脯氨酸;Ser:丝氨酸;Thr:苏氨酸;Tyr:酪氨酸;Val:缬氨酸 |
|
|
目前,关于有机卤呼吸细菌还原脱卤内电子转移反应过程中底物攻击位点与反应机理尚未达成共识。在PceA催化四氯乙烯脱氯反应的研究中,Zhang等[29]提出了质子耦合电子转移(proton-coupled electron transfer, PCET)反应机理;其中,质子来源于高度保守的Arg305,而非普遍认为的Try246,电子则来源于[4Fe-4S]1+簇。然而,其他RDases是否以Arg305或其他残基作为质子来源,仍需进一步验证。此外,既往研究表明还原脱卤发生在OHRB细胞外膜电子传递链末端脱卤酶催化活性空间[30-31]。然而,信号肽预测结果显示,NpRdhA、DcaA-Lab与TcaA-Lab的蛋白序列中均未发现明显的信号肽,表明它们不属于分泌蛋白。其具体的还原脱卤反应机制仍需进一步挖掘。
IDR/IDP在分子与细胞功能中发挥着重要作用,挑战了传统的序列-结构-功能范式,对其深入研究有助于阐明一些目前被忽视的生物学机理。无序区域的预测结果显示,44个RDases蛋白均存在具有蛋白结合位点的无序区域,并且多数RDases蛋白属于部分无序的IDP。不同底物,甚至相同底物的RDases蛋白在无序区域特征上的差异,揭示了无序区域在决定RDase功能和底物范围方面的潜在作用。然而,由于IDR/IDP缺乏稳定的三维结构,像AlphaFold等常见的结构预测方法难以捕捉其可能的三维构象特性,这使得实验表征IDR/IDP的结构特性极具挑战性,也可能是依赖晶体结构的RDases蛋白结构与功能机制等相关研究受限的原因之一。此外,研究表明IDR对溶剂的物理化学性质高度敏感,pH、温度、氧化还原状态和高渗压等条件均会导致某些IDR的构象变化,这意味着借助IDR环境敏感性来调节蛋白活性方面存在潜力[32-33]。未来借助更先进的技术研究RDases蛋白IDR的环境敏感性及特殊功能域等,不仅可为深入理解RDases蛋白的结构与功能关系提供新视角,也可为未来原位修复中OHRB或RDases的环境适应性问题提供新策略。
4 结论本研究利用多种生物信息学分析工具对44个RDases蛋白的结构与序列特征进行了预测分析。结果表明,不同RDases蛋白的理化性质存在差异,但呈现一定保守性。不同RDases蛋白的磷酸化位点、保守结构域分布、保守基序分布,以及二级结构的保守性较高。底物类别相似或相同的RDases在蛋白序列和三级结构上更为相似。大多数还原性脱卤酶蛋白的亲缘较近。RDases蛋白主要为分泌型蛋白、非膜定位蛋白和固有无序蛋白。因此,序列与结构的保守性支持了不同RDases蛋白在有机卤化物降解过程中可能执行相似的生物学功能。本研究结果旨在为未来RDases、有机卤呼吸细菌、有机卤化物污染修复及其他相关基础课题的研究设计提供理论支持。未来通过生物信息学对RDases理化特性与作用机制的不断揭示,并结合人工突变筛选高效有机卤呼吸细菌,将推动有机卤呼吸细菌在有机卤污染场地原位修复的应用。
| [1] |
LIU YH, WANG L, LIU RL, FANG JS. Biogeochemical cycling of halogenated organic compounds in the ocean: current progress and future directions[J]. Deep Sea Research Part Ⅰ: Oceanographic Research Papers, 2024, 205: 104237. DOI:10.1016/j.dsr.2024.104237 |
| [2] |
AGARWAL V, MILES ZD, WINTER JM, EUSTÁQUIO AS, EL GAMAL AA, MOORE BS. Enzymatic halogenation and dehalogenation reactions: pervasive and mechanistically diverse[J]. Chemical Reviews, 2017, 117(8): 5619-5674. DOI:10.1021/acs.chemrev.6b00571 |
| [3] |
ATASHGAHI S, HÄGGBLOM MM, SMIDT H. Organohalide respiration in pristine environments: implications for the natural halogen cycle[J]. Environmental Microbiology, 2018, 20(3): 934-948. DOI:10.1111/1462-2920.14016 |
| [4] |
ZHANG M, SHI Q, SONG XZ, WANG H, BIAN ZY. Recent electrochemical methods in electrochemical degradation of halogenated organics: a review[J]. Environmental Science and Pollution Research International, 2019, 26(11): 10457-10486. DOI:10.1007/s11356-019-04533-3 |
| [5] |
LOOS R, LOCORO G, COMERO S, CONTINI S, SCHWESIG D, WERRES F, BALSAA P, GANS O, WEISS S, BLAHA L, BOLCHI M, GAWLIK BM. Pan-European survey on the occurrence of selected polar organic persistent pollutants in ground water[J]. Water Research, 2010, 44(14): 4115-4126. DOI:10.1016/j.watres.2010.05.032 |
| [6] |
JOHNSON-RESTREPO B, KANNAN K, ADDINK R, ADAMS DH. Polybrominated diphenyl ethers and polychlorinated biphenyls in a marine foodweb of coastal Florida[J]. Environmental Science & Technology, 2005, 39(21): 8243-8250. |
| [7] |
XU Y, YU RMK, ZHANG XW, MURPHY MB, GIESY JP, LAM MHW, LAM PKS, WU RSS, YU HX. Effects of PCBs and MeSO2–PCBs on adrenocortical steroidogenesis in H295R human adrenocortical carcinoma cells[J]. Chemosphere, 2006, 63(5): 772-784. DOI:10.1016/j.chemosphere.2005.08.013 |
| [8] |
LU QH, QIU L, YU L, ZHANG SW, de TOLEDO RA, SHIM H, WANG SQ. Microbial transformation of chiral organohalides: distribution, microorganisms and mechanisms[J]. Journal of Hazardous Materials, 2019, 368: 849-861. DOI:10.1016/j.jhazmat.2019.01.103 |
| [9] |
WU RF, WANG SQ. Integration of microbial reductive dehalogenation with persulfate activation and oxidation (Bio-RD-PAO) for complete attenuation of organohalides[J]. Frontiers of Environmental Science & Engineering, 2021, 16(2): 22. |
| [10] |
FALANDYSZ J, ROSE M, FERNANDES AR. Mixed poly-brominated/chlorinated biphenyls (PXBs): widespread food and environmental contaminants[J]. Environment International, 2012, 44: 118-127. DOI:10.1016/j.envint.2012.03.006 |
| [11] |
ZHANG ZW, PEI NC, SUN YX, LI JL, LI XP, YU S, XU XR, HU YX, MAI BX. Halogenated organic pollutants in sediments and organisms from mangrove wetlands of the Jiulong River Estuary, South China[J]. Environmental Research, 2019, 171: 145-152. DOI:10.1016/j.envres.2019.01.028 |
| [12] |
RAI AR, SINGH RP, SRIVASTAVA AK, DUBEY RC. Structure prediction and evolution of a halo-acid dehalogenase of Burkholderia mallei[J]. Bioinformation, 2012, 8(22): 1111-1113. DOI:10.6026/97320630081111 |
| [13] |
JUGDER BE, ERTAN H, BOHL S, LEE M, MARQUIS CP, MANEFIELD M. Organohalide respiring bacteria and reductive dehalogenases: key tools in organohalide bioremediation[J]. Frontiers in Microbiology, 2016, 7: 249. |
| [14] |
SUFLITA JM, HOROWITZ A, SHELTON DR, TIEDJE JM. Dehalogenation: a novel pathway for the anaerobic biodegradation of haloaromatic compounds[J]. Science, 1982, 218(4577): 1115-1117. DOI:10.1126/science.218.4577.1115 |
| [15] |
LEYS D, ADRIAN L, SMIDT H. Organohalide respiration: microbes breathing chlorinated molecules[J]. Philosophical Transactions of the Royal Society of London Series B, Biological Sciences, 2013, 368(1616): 20120316. DOI:10.1098/rstb.2012.0316 |
| [16] |
RICHARDSON RE. Genomic insights into organohalide respiration[J]. Current Opinion in Biotechnology, 2013, 24(3): 498-505. DOI:10.1016/j.copbio.2013.02.014 |
| [17] |
BOMMER M, KUNZE C, FESSELER J, SCHUBERT T, DIEKERT G, DOBBEK H. Structural basis for organohalide respiration[J]. Science, 2014, 346(6208): 455-458. DOI:10.1126/science.1258118 |
| [18] |
HUG LA, MAPHOSA F, LEYS D, LÖFFLER FE, SMIDT H, EDWARDS EA, ADRIAN L. Overview of organohalide-respiring bacteria and a proposal for a classification system for reductive dehalogenases[J]. Philosophical Transactions of the Royal Society of London Series B, Biological Sciences, 2013, 368(1616): 20120322. DOI:10.1098/rstb.2012.0322 |
| [19] |
SCHUBERT T, DIEKERT G. Comparative biochemistry of organohalide respiration[M]// Organohalide-Respiring Bacteria. Berlin, Heidelberg: Springer Berlin Heidelberg, 2016: 397-427.
|
| [20] |
SCHUBERT T, ADRIAN L, SAWERS RG, DIEKERT G. Organohalide respiratory chains: composition, topology and key enzymes[J]. FEMS Microbiology Ecology, 2018, 94(4): fiy035. |
| [21] |
NAKAMURA R, OBATA T, NOJIMA R, HASHIMOTO Y, NOGUCHI K, OGAWA T, YOHDA M. Functional expression and characterization of tetrachloroethene dehalogenase from Geobacter sp.[J]. Frontiers in Microbiology, 2018, 9: 1774. DOI:10.3389/fmicb.2018.01774 |
| [22] |
MOLENDA O, PUENTES JÁCOME LA, CAO X, NESBØ CL, TANG SQ, MORSON N, PATRON J, LOMHEIM L, WISHART DS, EDWARDS EA. Insights into origins and function of the unexplored majority of the reductive dehalogenase gene family as a result of genome assembly and ortholog group classification[J]. Environmental Science Processes & Impacts, 2020, 22(3): 663-678. |
| [23] |
JAMAL S, ALI W, NAGPAL P, GROVER A, GROVER S. Predicting phosphorylation sites using machine learning by integrating the sequence, structure, and functional information of proteins[J]. Journal of Translational Medicine, 2021, 19(1): 218. |
| [24] |
ARDITO F, GIULIANI M, PERRONE D, TROIANO G, MUZIO LL. The crucial role of protein phosphorylation in cell signaling and its use as targeted therapy (Review)[J]. International Journal of Molecular Medicine, 2017, 40(2): 271-280. DOI:10.3892/ijmm.2017.3036 |
| [25] |
DUNKER AK, LAWSON JD, BROWN CJ, WILLIAMS RM, ROMERO P, OH JS, OLDFIELD CJ, CAMPEN AM, RATLIFF CM, HIPPS KW, AUSIO J, NISSEN MS, REEVES R, KANG C, KISSINGER CR, BAILEY RW, GRISWOLD MD, CHIU W, GARNER EC, OBRADOVIC Z. Intrinsically disordered protein[J]. Journal of Molecular Graphics and Modelling, 2001, 19(1): 26-59. DOI:10.1016/S1093-3263(00)00138-8 |
| [26] |
ZHU YF, CHEN YR, CAO ZX, WANG JH. Study on intrinsically disordered proteins (IDPs) by prediction and molecular dynamics simulation[J]. Journal of Dezhou University, 2014, 30(6): 6-13. (in Chinese) 朱玉风, 陈艳如, 曹赞霞, 王吉华. 固有无序蛋白质(IDPs)的预测和分子动力学模拟研究[J]. 德州学院学报, 2014, 30(6): 6-13. |
| [27] |
UVERSKY VN. Introduction to intrinsically disordered proteins (IDPs)[J]. Chemical Reviews, 2014, 114(13): 6557-6560. DOI:10.1021/cr500288y |
| [28] |
FUKUCHI S, HOSODA K, HOMMA K, GOJOBORI T, NISHIKAWA K. Binary classification of protein molecules into intrinsically disordered and ordered segments[J]. BMC Structural Biology, 2011, 11: 29. DOI:10.1186/1472-6807-11-29 |
| [29] |
ZHANG X, WANG ZK, LI Z, SHAIK S, WANG BJ. [4Fe–4S]-mediated proton-coupled electron transfer enables the efficient degradation of chloroalkenes by reductive dehalogenases[J]. ACS Catalysis, 2023, 13(2): 1173-1185. |
| [30] |
ZHANG SW, WEN W, XIA XH, OUYANG W, MAI BX, ADRIAN L, SCHÜÜRMANN G. Insight into the mechanism underlying Dehalococcoides mccartyi strain CBDB1-mediated B12-dependent aromatic reductive dehalogenation[J]. Environmental Science & Technology, 2023, 57(29): 10773-10781. |
| [31] |
JUGDER BE, ERTAN H, LEE M, MANEFIELD M, MARQUIS CP. Reductive dehalogenases come of age in biological destruction of organohalides[J]. Trends in Biotechnology, 2015, 33(10): 595-610. |
| [32] |
THEILLET FX, BINOLFI A, FREMBGEN-KESNER T, HINGORANI K, SARKAR M, KYNE C, LI CG, CROWLEY PB, GIERASCH L, PIELAK GJ, ELCOCK AH, GERSHENSON A, SELENKO P. Physicochemical properties of cells and their effects on intrinsically disordered proteins (IDPs)[J]. Chemical Reviews, 2014, 114(13): 6661-6714. |
| [33] |
CUEVAS-VELAZQUEZ CL, VELLOSILLO T, GUADALUPE K, SCHMIDT HB, YU F, MOSES D, BROPHY JAN, COSIO-ACOSTA D, DAS A, WANG LX, JONES AM, COVARRUBIAS AA, SUKENIK S, DINNENY JR. Intrinsically disordered protein biosensor tracks the physical-chemical effects of osmotic stress on cells[J]. Nature Communications, 2021, 12(1): 5438. |
 2024, Vol. 51
2024, Vol. 51