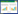扩展功能
文章信息
- 苟丽霞, 蔡楠, 刘孟欣, 汪志军, 韩铁生
- GOU Lixia, CAI Nan, LIU Mengxin, WANG Zhijun, HAN Tiesheng
- 卡西霉素生物合成基因簇中调控基因calR1的功能
- Functional analysis of the regulatory gene calR1 in calcimycin biosynthetic gene cluster
- 微生物学通报, 2023, 50(3): 1195-1204
- Microbiology China, 2023, 50(3): 1195-1204
- DOI: 10.13344/j.microbiol.china.220555
-
文章历史
- 收稿日期: 2022-06-09
- 接受日期: 2022-08-18
- 网络首发日期: 2022-10-08
2. 上海交通大学生命科学技术学院 微生物代谢国家重点实验室, 上海 200240;
3. 华北理工大学公共卫生学院 河北省煤矿卫生与安全重点实验室, 河北 唐山 063210
2. State Key Laboratory of Microbial Metabolism, School of Life Sciences and Biotechnology, Shanghai Jiao Tong University, Shanghai 200240, China;
3. Hebei Provincial Key Laboratory of Occupational Health and Safety for Coal Industry, School of Public Health, North China University of Science and Technology, Tangshan 063210, Hebei, China
卡西霉素(calcimycin,A23187,图 1)是1972年Reed等在教酒链霉菌NRRL3882中发现的聚醚类化合物,其结构由α-酮基吡咯、螺缩酮及苯并噁唑3部分组成[1-2]。卡西霉素是重要的离子载体抗生素,目前已有17 000多篇文献对其报道,其能与生理上重要的阳离子如钙离子、镁离子等结合,形成脂溶性复合物通过生物膜,进而发挥药理作用[3]。近年来,研究者发现卡西霉素通过将细胞外的钙离子逆浓度梯度泵入卵母细胞,从而有效激活卵母细胞,因此广泛应用于辅助生殖的临床实践中[4-5]。基于卡西霉素独特的结构特征和广泛的生理活性[6-8],其生物合成途径的研究引起科学家的广泛关注。
对卡西霉素生物合成途径的研究始于1980年[9-10]。2011年,Wu等[11]从教酒链霉菌NRRL3882中克隆到负责合成卡西霉素的基因簇。根据生物信息学预测,calA1-A5是5个聚酮合成酶(polyketide synthase, PKS)基因,负责螺缩酮的合成;calN1-N3负责α-酮基吡咯的合成;calT为抗性基因;calR1-R3为3个调控基因;calU1-U5为未知功能基因。2013年,我们通过基因敲除及喂养实验证实基因簇上的calB1-B4基因负责3-羟基邻氨基苯甲酸(3-HAA,苯并噁唑环的前体物)的生物合成[12],并通过体外催化实验证实CalM负责卡西霉素中苯并噁唑结构的氮甲基化[13]。2018年,Wu等通过体内外实验证实了CalC是一种ATP依赖的cezomycin-CoA连接酶[14],而硫酯酶CalG催化cezomycin-CoA复合物的解离[15]。
卡西霉素生物合成基因簇上共有3个可能的转录调控基因calR1、calR2和calR3,分别属于TylR、LuxR和TetR家族。前期工作中,我们通过基因敲除、回补及体外催化实验证实calR3是卡西霉素生物合成中的负调控基因,calR3基因缺失突变株的卡西霉素产量提高了7倍[16]。通过构建calR2基因缺失突变菌株并对其代谢产物及突变株基因转录水平分析,证实calR2在卡西霉素的生物合成中起正调控作用[1]。
本研究以另一个潜在的调控基因calR1为研究对象,生物信息学比对分析发现,CalR1与泰乐菌素(tylosin)生物合成基因簇中的调控因子TylR及碳霉素(carbomycin)生物合成基因簇的调控因子AcyB2均具有较高的相似性。本研究通过PCR-targeting的方法,构建calR1基因中断的突变株、利用整合型回补质粒pJTU2170载体构建∆calR1: calR1的回补菌株,对∆calR1突变菌株及回补菌株的发酵产物进行HPLC分析,并比较分析在野生型及突变型菌株中卡西霉素生物合成关键基因的转录差异,以期初步阐明calR1基因对卡西霉素产量的影响,为卡西霉素生物合成调控网络的研究提供有力依据,并为后续利用分子生物学手段提高卡西霉素产量奠定基础。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒Escherichia coli ET12567/pUZ8002用于接合转移,BW25113/pIJ790用于PCR-targeting敲除宿主目的基因,整合型质粒pJTU2170用于突变株的回补,以上菌株及质粒均来自上海交通大学分子微生物学实验室,具体信息见表 1。
| Strain or plasmid | Characteristics | Resource |
| Strain | ||
| E. coli | ||
| BW25113/pIJ790 | RepA101(ts), araBp-gam-be-exo, araC, Cml | [17] |
| ET12567/pUZ8002 | Kan, CmL, dcmdam hsdS Tra+ | [17] |
| DH10B | lacZ ΔM15F-recA | Invitrogen |
| Streptomyces chartreusis | ||
| NRRL3882 | Wild type | NRRL |
| GLX24 | calR1 disruption mutant | This study |
| GLX27 | GLX24 complemented with calR1 | This study |
| Plasmid | ||
| p16F9 | CmL, repE ori2 oriV | [11] |
| pJTU3789 | p16F9 derived plasmid carrying a defective calR1, Apr | This study |
| pJTU2170 | aac(3)IV, lacz, reppuc, attФC31, oriT | [11] |
| pJTU2170-R1 | pJTU2170 containing calR1 | This study |
LA及LB培养基[13]用于大肠杆菌培养;TSBY培养基[13]用于链霉菌总DNA及RNA的提取;SFM固体培养基[13]及SFM液体培养基分别用于链霉菌的培养及发酵。
1.1.3 主要试剂和仪器KOD plus和rTaq,TOYOBO公司;限制性内切酶,New England Biolabs公司;RNA提取试剂盒、DNA胶回收试剂盒、FastQuant RT Kit和SuperReal qPCR PreMix (SYBR Green)试剂盒,天根生化科技有限公司;卡西霉素标准品,Sigma-Aldrich公司。抗生素、如氨苄霉素、卡那霉素、阿泊拉霉素、氯霉素的浓度及HPLC、LC/MS条件见文献[1]。
恒温摇床ZWF-200、恒温培养箱ZXRD-B2050,上海智城分析仪器制造有限公司;用于DNA定量的ScanDrop200,耶拿分析仪器股份公司;用于RT-qPCR的ABI7900HT,应用生物系统公司;HPLC (Agilent 1200)、LC/MS (Agilent 6410)及ZORBAX SB-C18色谱柱(2.1 mm×150 mm, 3.5 μm),安捷伦科技有限公司。
1.2 方法 1.2.1 CalR1蛋白的序列比对分析CalR1 (GenBank登录号为AEH42475.1)、TylR (GenBank登录号为AAF29380.1)、AcyB2 (GenBank登录号为ALK28498.1)的氨基酸序列采用Clustal Omega工具进行序列比对[18],采用Needleman-Wunsch BLAST方法进行一致性和相似性的两两比较。
1.2.2 calR1基因敲除突变菌株的构建本研究中,calR1基因敲除采用的是PCR-targeting方法。设计引物R1-F1和R1-F2 (表 2),PCR扩增用于敲除calR1基因的替换片段。通过Escherichia coli BW25113/pIJ790的λ-RED重组系统将含有阿泊拉霉素抗性基因及oriT的cassette与柯斯质粒p16F9上的calR1基因进行双交换同源重组,获得重组质粒pJTU3789。pJTU3789的接合转移及接合子筛选条件同文献[1]。挑取接合子扩大培养,提取其基因组DNA,采用引物R1-F3和R1-F4 (表 2)进行PCR扩增验证。链霉菌基因组DNA的提取方法及PCR反应体系、条件见文献[1]。
| Primer | Sequence (5′→3′) | Size (bp) | Usage |
| RTC-F1 | CGGCTGGGGTCATTGTT | 17 | calC RT-qPCR |
| RTC-F2 | AAGGCGTTCGACTTGTA | 17 | |
| R1-F1 | TTGCCCCGCAGCGACCAGCGCGGGCGCGGCCTCGACTACATTCCGGGGATCCGTCGACC | 59 | Disruption of calR1 |
| R1-F2 | CCGGCGCACCGGGTCGCCCAGCCAGACACCCGACAGCAGTGTAGGCTGGAGCTGCTTC | 58 | |
| R1-F3 | CCGTGCGACCGATCGAAC | 18 | GLX24 and GLX27 validation |
| R1-F4 | GCACCGCACACATCTCCA | 18 | |
| R1-F5 | GGAGTTCCATATGATCACCGGACACGACCTCGAAG | 35 | |
| R1-F6 | CCGGAATTCGAGAACGACATCCTGCTGACCCT | 32 |
设计引物R1-F5和R1-F6 (表 2),以教酒链霉菌NRRL3882的基因组DNA为模板,通过高保真的KOD-plus DNA聚合酶扩增完整的calR1基因。PCR产物通过限制性内切酶Nde I及EcoR I酶切后连接到pJTU2170上,构建回补质粒pJTU2170-R1。在辅助质粒pUZ8002的协助下,通过大肠杆菌和链霉菌之间的两亲本接合转移,将pJTU2170-R1导入到ΔcalR1突变株中,采用卡那霉素抗性筛选,并使用PCR扩增回补菌株的总DNA以验证回补是否成功。
1.2.4 菌株发酵及发酵产物分析将野生菌株NRRL3882、突变菌株GLX24及回补菌株GLX27 (ΔcalR1: calR1)的孢子接种培养。种子液及发酵的培养条件、发酵液的收集、萃取、浓缩,以及HPLC或LC/MS检测分析条件见文献[1],其中色谱检测程序:0−5 min,70%甲醇等梯度洗脱;6−20 min,70%−90%甲醇洗脱;21−40 min,90%−100%甲醇洗脱;41−45 min,100%甲醇洗脱。
1.2.5 链霉菌总RNA的提取和反转录将野生NRRL3882菌株和突变菌株GLX24的孢子接种于TSBY培养基中,30 ℃、220 r/min培养5 d,吸取少量菌液于12 000 r/min离心1 min,弃上清。然后按照Total RNA Isolation Kit操作说明提取RNA,之后将去除DNA的RNA反转录为cDNA,根据FastQuant RT Kit的操作说明书进行,详见文献[1]。
1.2.6 卡西霉素生物合成基因转录水平检测采用荧光定量PCR分析野生菌株及calR1中断的突变株的卡西霉素生物合成相关基因的转录水平。以cDNA为模板,16S rRNA基因为内参,根据ΔΔCt的计算公式[1],通过比较Ct值计算目标基因在突变菌株和野生菌株的转录水平差异。根据卡西霉素生物合成相关基因的功能及其在基因簇上的分布筛选了14个目标基因,其中calC的RT-qPCR引物序列见本文表 2,其他基因的引物见文献[1]的表 2。根据SuperReal qPCR PreMix试剂盒配制20 μL Real-time PCR反应体系:10 μL Super Real SYBR Green PreMix,上、下游引物(300 nmol/L)各3 μL;模板cDNA (4 ng/μL) 4 μL。PCR反应条件见文献[1]。
2 结果与分析 2.1 CalR1的生物信息学分析如图 2的氨基酸序列比对显示,CalR1与兽药抗生素泰乐菌素生物合成基因簇中的调控因子TylR[19-21],以及源自耐热链霉菌(Streptomyces thermotolerans)的碳霉素生物合成基因簇的调控因子AcyB2[22-25]均具有较高的相似性。其中CalR1与TylR的一致性和相似性分别为38%和49%,与AcyB2的一致性和相似性分别为37%和49%。这提示我们,CalR1可能为卡西霉素生物合成途径的调控因子。

|
| 图 2 CalR1蛋白和同源蛋白氨基酸序列比对 Figure 2 Amino acid sequence alignment of CalR1 with its homologous proteins. CalR1 (GenBank登录号为AEH42475.1)来源于Streptomyces chartreusis NRRL 3882;TylR (GenBank登录号为AAF29380.1)来源于Streptomyces fradiae;AcyB2 (GenBank登录号为ALK28498.1)来源于Streptomyces thermotolerans CalR1 (GenBank accession No.: AEH42475.1) from Streptomyces chartreusis NRRL 3882; TylR (GenBank accession No.: AAF29380.1) from Streptomyces fradiae; AcyB2 (GenBank accession No.: ALK28498.1) from Streptomyces thermotolerans. |
|
|
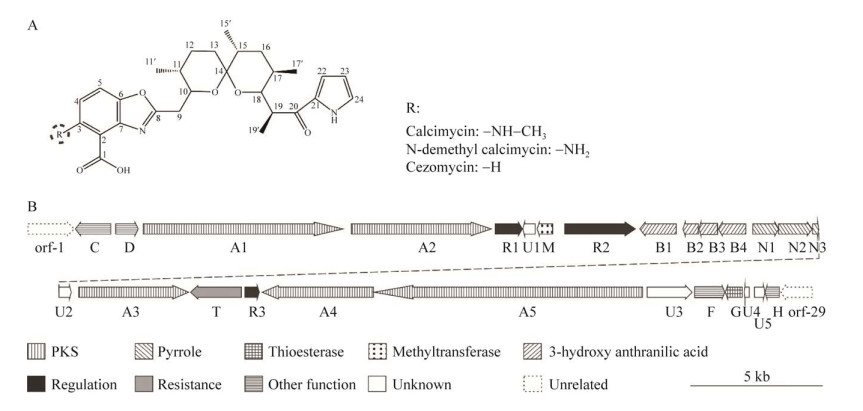
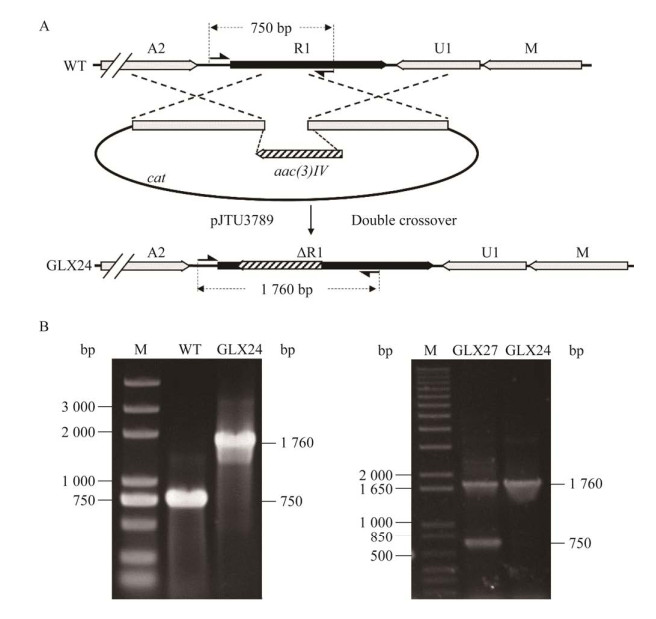
为了确认calR1基因的功能,通过PCR-targeting技术,对教酒链霉菌NRRL3882基因组上的calR1基因进行敲除及回补实验,通过双交换同源重组构建了用于敲除calR1基因的基因敲除质粒pJTU3789,见图 3A。将pJTU3789转入大肠杆菌ET12567/pUZ8002,接着与野生菌株接合转移,筛选含阿泊拉霉素抗性的接合子。通过PCR验证接合子的基因型,其中双交换突变株GLX24的PCR扩增片段为1 760 bp,而野生菌株的扩增片段为750 bp,结果见图 3B,与预期相吻合。
|
| 图 3 突变菌株GLX24的构建及GLX24与回补菌株GLX27的PCR验证 Figure 3 Construction of the GLX24 mutant, and PCR verification of the mutant GLX24 and the complementation strain GLX27. A:通过基因置换用aac(3)IV抗性基因中断calR1基因得到突变菌株GLX24. B:突变株GLX24与回补菌株GLX27的PCR验证. M:DNA molecular weight marker;WT:以野生菌株的总DNA为模板的扩增产物;GLX24:以突变株GLX24的总DNA为模板的扩增产物;GLX27:以回补菌株GLX27的总DNA为模板的扩增产物 A: A schematic diagram elucidating the generation of GLX24. B: Validation of the mutant strain GLX24 and complementation strain GLX27 by PCR analysis. M: Marker; WT: PCR product using total DNA of wild-type strain as template; GLX24: PCR product using total DNA of the mutant GLX24 as template; GLX27: PCR product using total DNA of the complementation strainGLX27 as template. |
|
|
将本研究构建的calR1基因回补质粒pJTU2170-R1导入到突变菌株GLX24 (ΔcalR1)中获得回补菌株GLX27(ΔcalR1: calR1),提取回补菌株GLX27的DNA,PCR扩增验证。因为pJTU2170为整合型质粒,所以回补菌株含有750 bp的野生型条带及1 760 bp的重组条带(图 3C)。
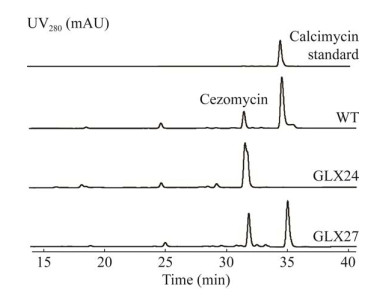
2.3 菌株代谢产物分析将S. chartreusis NRRL3882、GLX24及GLX27分别进行液体发酵,30 ℃、220 r/min培养7 d,采用HPLC分析发酵产物,以calcimycin标准品作为阳性对照。HPLC结果见图 4,野生菌株产生卡西霉素及cezomycin[12],突变菌株GLX24失去产生卡西霉素的能力,但有cezomycin的积累,结合文中2.1的生物信息学预测结果,说明calR1基因可能在卡西霉素的生物合成中起调控功能,可能负责从cezomycin至卡西霉素之间的转化。calR1基因对突变株GLX24进行回补后,回补菌株GLX27恢复产生卡西霉素的能力,表明calR1基因中断对卡西霉素产量的影响并不是由极性效应产生,进一步说明calR1基因在卡西霉素生物合成中的调控作用。
|
| 图 4 野生菌株(WT)、GLX24 (突变株)与GLX27 (回补菌株)发酵产物的HPLC检测 Figure 4 HPLC analysis of the metabolites of wild-type, calR1 knock-out mutant strain GLX24 and complementary strain GLX27. 卡西霉素标准品作参照 Calcimycin standard as control. |
|
|
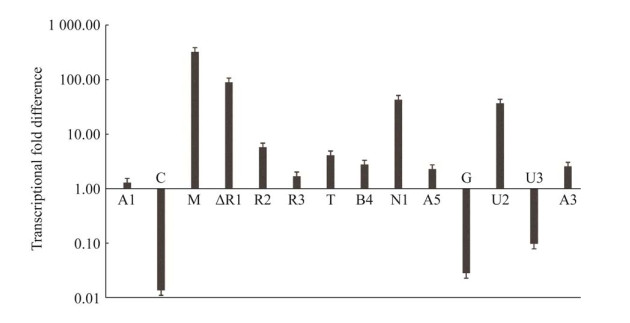
通过RT-qPCR分析calR1基因对卡西霉素合成相关基因的转录调控,提取培养96 h (平台期)[16]的S. chartreusis NRRL3882与GLX24的总RNA,分析卡西霉素生物合成基因簇上14个不同功能基因(图 5)的转录水平,测定这些基因在突变菌株与野生菌株的转录差异,各基因的分布及预测的功能见图 1B。
|
| 图 5 GLX24与野生型菌株的目标基因转录水平差异分析 Figure 5 Detection of the difference of transcription level between the wild-type strain and GLX24 mutant. 纵轴为各基因在GLX24中相较于野生型菌株中的转录水平差异倍数 The vertical axis is the fold difference in the transcription level of each gene in GLX24 relative to the wild-type strain. |
|
|
结果如图 5所示,与野生型相比,突变株中calC、calG和calU3的转录水平显著下降,而其他11个基因的转录水平均有明显上升。由图 4可知,calR1基因的敲除导致中间产物cezomycin大量积累,几乎不产生终产物calcimycin。因此,在野生型菌株中,calR1可能通过正调控calC、calG及calU3的转录水平,促进cezomycin向calcimycin的转化。
3 讨论与结论calR1在生物信息学预测中属于TylR家族基因,但该家族基因的实验研究较少,目前只有泰乐菌素生物合成基因簇中的tylR及碳霉素生物合成基因簇的acyB2有研究报道。本研究通过将calR1基因进行敲除及回补,通过代谢产物比较分析,证实其负责调控从中间产物cezomycin向终产物卡西霉素的转化。卡西霉素在3位上比cezomycin多一个甲氨基(图 1A),因此calR1可能负责调控cezomycin的3位氨基化。
通过对基因簇中14个代表性基因进行转录水平差异检测,发现敲除calR1后,calC、calG、calU3的转录水平均显著下降(分别下降约0.013、0.028、0.01倍)。最近的研究报道显示,CalC与cezomycin在3位的活化有关[14],而CalG催化CalC的逆反应[15]。Cezomycin经CalC活化后,可能经过多步反应在CalU3等蛋白作用下3位氨基化(CalU3功能研究待发表),然后由CalM进一步甲基化[13],形成终产物卡西霉素。因此calC转录水平的显著下调将阻碍cezomycin的进一步反应,这与本研究观察到的敲除calR1前后基因转录水平差异结果相一致。
敲除calR1后,calM等11个基因的转录水平均有不同程度的上升,显示calR1是一个识别位点较宽泛的转录调控因子,该性质与tylR相似。在Streptomyces fradiae中敲除tylR后,泰乐菌素生物合成基因簇中有至少19个基因转录水平下调或不表达,同时至少有13个基因转录水平上调[19]。这种生物合成途径内的“全局性”调控现象,可能与CalR1或TylR的DNA结合结构域的识别特异性有关。
敲除calR1后,calR2、calR3的转录水平均有不同程度上升(图 5),显示3个调控基因之间可能具有相互作用。已有的研究显示,敲除calR2后,calR1、calR3的转录水平基本无变化,而calA1、calA3、calA5、calM、calB4、calT、calU2等基因均显著下调,导致完全不产卡西霉素及cezomycin[1]。敲除calR3后,calR2转录水平约上升3倍,calR1基本不变,除了calA1略有下降,其余关键基因的转录水平均有不同程度上升,其中calT的转录水平大约上升170倍,导致卡西霉素的产量上升了约8倍[16]。综合以上基因转录差异数据,可推测calR1负调控calR3及calR2,calR3也负调控calR2,但该推测还需要进一步实验验证,因其可能并非直接调控关系,可能涉及中间产物产量变化等导致的间接调控。关于高产菌株改造,calR1及calR2与卡西霉素的高产关系还不明确,需要进一步研究,而敲除calR3的突变株产量提升明显,有望改造成工业发酵高产菌株。综上所述,本研究通过基因敲除回补、代谢产物比较分析、基因转录差异检测等鉴定了calR1为卡西霉素生物合成途径中的调控因子,其可能通过调控calC、calG及calU3的表达,催化中间产物cezomycin转化为卡西霉素。本研究将为揭示卡西霉素的3位甲氨基化修饰机制打下良好基础。
| [1] |
GE JX, HAN TS, LIU WX, WU C, WANG ZJ, GOU LX. Function of the regulatory gene calR2 involved in calcimycin biosynthesis[J]. Microbiology China, 2018, 45(7): 1518-1526. (in Chinese) 盖婧璇, 韩铁生, 刘文秀, 武策, 汪志军, 苟丽霞. 卡西霉素生物合成调控基因calR2的功能[J]. 微生物学通报, 2018, 45(7): 1518-1526. |
| [2] |
REED PW, LARDY HA. A23187: a divalent cation ionophore[J]. The Journal of Biological Chemistry, 1972, 247(21): 6970-6977. DOI:10.1016/S0021-9258(19)44681-4 |
| [3] |
REED PW. Effects of divalent cation ionophore A23187 on potassium permeability of rat erythrocytes[J]. The Journal of Biological Chemistry, 1976, 251(11): 3489-3494. DOI:10.1016/S0021-9258(17)33464-6 |
| [4] |
XU ZW, YAO GD, NIU WB, FAN HY, MA XS, SHI SL, JIN HX, SONG WY, SUN YP. Calcium ionophore (A23187) rescues the activation of unfertilized oocytes after intracytoplasmic sperm injection and chromosome analysis of blastocyst after activation[J]. Frontiers in Endocrinology, 2021, 12: 692082. DOI:10.3389/fendo.2021.692082 |
| [5] |
SAMPAIO B, ORTIZ I, RESENDE H, FELIX M, VARNER D, HINRICHS K. Factors affecting intracellular calcium influx in response to calcium ionophore A23187 in equine sperm[J]. Andrology, 2021, 9(5): 1631-1651. DOI:10.1111/andr.13036 |
| [6] |
HOLLAND DR, STEINBERG MI, ARMSTRONG WM. A23187: a calcium ionophore that directly increases cardiac contractility (38704)[J]. Proceedings of the Society for Experimental Biology and Medicine Society for Experimental Biology and Medicine (New York, N. Y.), 1975, 148(4): 1141-1145. DOI:10.3181/00379727-148-38704 |
| [7] |
MATSUSHIMA K, OPPENHEIM JJ. Calcium ionophore (A23187) increases interleukin 1 (IL-1) production by human peripheral blood monocytes and interacts synergistically with IL-1 to augment concanavalin A stimulated thymocyte proliferation[J]. Cellular Immunology, 1985, 90(1): 226-233. DOI:10.1016/0008-8749(85)90184-4 |
| [8] |
KAN KS, COLEMAN R. The calcium ionophore A23187 increases the tight-junctional permeability in rat liver[J]. The Biochemical Journal, 1988, 256(3): 1039-1041. DOI:10.1042/bj2561039 |
| [9] |
ZMIJEWSKI MJ Jr. Biosynthesis of antibiotic A23187. incorporation of precursors into A23187[J]. The Journal of Antibiotics, 1980, 33(4): 447-450. DOI:10.7164/antibiotics.33.447 |
| [10] |
DAVID L, EMADZADEH S. Biosynthesis of the ionophorous antibiotic A23187[J]. The Journal of Antibiotics, 1982, 35(11): 1616-1617. DOI:10.7164/antibiotics.35.1616 |
| [11] |
WU QL, LIANG JD, LIN SJ, ZHOU XF, BAI LQ, DENG ZX, WANG ZJ. Characterization of the biosynthesis gene cluster for the pyrrole polyether antibiotic calcimycin (A23187) in Streptomyces chartreusis NRRL 3882[J]. Antimicrobial Agents and Chemotherapy, 2011, 55(3): 974-982. DOI:10.1128/AAC.01130-10 |
| [12] |
GOU LX, WU QL, LIN SJ, LI XM, LIANG JD, ZHOU XF, AN DR, DENG ZX, WANG ZJ. Mutasynthesis of pyrrole spiroketal compound using calcimycin 3-hydroxy anthranilic acid biosynthetic mutant[J]. Applied Microbiology and Biotechnology, 2013, 97(18): 8183-8191. DOI:10.1007/s00253-013-4882-1 |
| [13] |
WU QL, GOU LX, LIN SJ, LIANG JD, YIN J, ZHOU XF, BAI LQ, AN DR, DENG ZX, WANG ZJ. Characterization of the N-methyltransferase CalM involved in calcimycin biosynthesis by Streptomyces chartreusis NRRL 3882[J]. Biochimie, 2013, 95(7): 1487-1493. DOI:10.1016/j.biochi.2013.03.014 |
| [14] |
WU H, LIANG JD, WANG JL, LIANG WJ, GOU LX, WU QL, ZHOU XF, BRUCE IJ, DENG ZX, WANG ZJ. Cezomycin is activated by CalC to its ester form for further biosynthesis steps in the production of calcimycin in Streptomyces chartreusis NRRL 3882[J]. Applied and Environmental Microbiology, 2018, 84(12): e00586-e00518. |
| [15] |
WU H, LIANG JD, GOU LX, WU QL, LIANG WJ, ZHOU XF, BRUCE IJ, DENG ZX, WANG ZJ. Recycling of overactivated acyls by a type II thioesterase during calcimycin biosynthesis in Streptomyces chartreusis NRRL 3882[J]. Applied and Environmental Microbiology, 2018, 84(12): e00587-e00518. |
| [16] |
GOU LX, HAN TS, WANG XX, GE JX, LIU WX, HU F, WANG ZJ. A novel TetR family transcriptional regulator, CalR3, negatively controls calcimycin biosynthesis in Streptomyces chartreusis NRRL 3882[J]. Frontiers in Microbiology, 2017, 8: 2371. DOI:10.3389/fmicb.2017.02371 |
| [17] |
KIESER T, BIBB MJ, BUTTNER MJ, CHATER KF, HOPWOOD DA. Practical Streptomyces Genetics[M]. John Innes Foundation, Norwich, 2000.
|
| [18] |
MADEIRA F, PARK YM, LEE J, BUSO N, GUR T, MADHUSOODANAN N, BASUTKAR P, TIVEY ARN, POTTER SC, FINN RD, LOPEZ R. The EMBL-EBI search and sequence analysis tools APIs in 2019[J]. Nucleic Acids Research, 2019, 47(W1): W636-W641. DOI:10.1093/nar/gkz268 |
| [19] |
STRATIGOPOULOS G, BATE N, CUNDLIFFE E. Positive control of tylosin biosynthesis: pivotal role of TylR[J]. Molecular Microbiology, 2004, 54(5): 1326-1334. DOI:10.1111/j.1365-2958.2004.04347.x |
| [20] |
BATE N, BIGNELL DRD, CUNDLIFFE E. Regulation of tylosin biosynthesis involving 'SARP-helper' activity[J]. Molecular Microbiology, 2006, 62(1): 148-156. DOI:10.1111/j.1365-2958.2006.05338.x |
| [21] |
CUNDLIFFE E. Control of tylosin biosynthesis in Streptomyces fradiae[J]. Journal of Microbiology and Biotechnology, 2008, 18(9): 1485-1491. |
| [22] |
ARISAWA A, KAWAMURA N, TSUNEKAWA H, OKAMURA K, TONE H, OKAMOTO R. Cloning and nucleotide sequences of two genes involved in the 4″-O-acylation of macrolide antibiotics from Streptomyces thermotolerans[J]. Bioscience, Biotechnology, and Biochemistry, 1993, 57(12): 2020-2025. |
| [23] |
ZHONG JJ, LU ZL, DAI JL, HE WQ. Identification of two regulatory genes involved in carbomycin biosynthesis in Streptomyces thermotolerans[J]. Archives of Microbiology, 2017, 199(7): 1023-1033. |
| [24] |
LIU JJ, ZHANG Y, HE WQ. Construction of a novel carrimycin-producing strain by using CRISPR-Cas9 and ribosome engineering techniques[J]. Chinese Journal of Biotechnology, 2021, 37(6): 2116-2126. (in Chinese) 刘娟娟, 张妍, 赫卫清. 利用CRISPR-Cas9系统与核糖体工程获得新型可利霉素产生菌[J]. 生物工程学报, 2021, 37(6): 2116-2126. |
| [25] |
DAI JL, WANG YG, LIU JJ, HE WQ. The regulatory genes involved in spiramycin and bitespiramycin biosynthesis[J]. Microbiological Research, 2020, 240: 126532. DOI:10.1016/j.micres.2020.126532 |
 2023, Vol. 50
2023, Vol. 50