扩展功能
文章信息
- 肖瑀轩, 赵舒然, 文秋, 徐畅, 封雨晴, 胡永飞
- XIAO Yuxuan, ZHAO Shuran, WEN Qiu, XU Chang, FENG Yuqing, HU Yongfei
- 鸡源暹罗芽孢杆菌CML548的益生特性及全基因组分析
- Probiotic characteristics and whole genome analysis of Bacillus siamensis CML548 from chicken
- 微生物学通报, 2023, 50(2): 584-599
- Microbiology China, 2023, 50(2): 584-599
- DOI: 10.13344/j.microbiol.china.220475
-
文章历史
- 收稿日期: 2022-05-11
- 接受日期: 2022-07-15
- 网络首发日期: 2022-08-18
家禽养殖业是我国畜牧业的重要支柱产业之一[1]。长期以来,抗生素作为饲料添加剂在动物疫病防治、提高饲料转化率、促进畜禽生长方面发挥了重要作用[2]。但其滥用所造成的耐药基因和耐药细菌的传播对人类健康造成了潜在威胁。我国自2021年7月1日开始,已经全面禁止促生长类药物饲料添加剂的使用,给畜牧业带来了新的挑战和机遇[3]。为了实现绿色健康养殖,酶制剂、微生态制剂、酸化剂和噬菌体等饲料添加剂正成为研究热点。其中,芽孢杆菌由于具有产生多种消化酶、生长迅速、耐酸、耐胆盐、耐高温和抑制病原微生物生长等特性,被认为是益生菌的重要来源。多种芽孢杆菌已被《饲料添加剂目录》所收录[3]。
暹罗芽孢杆菌(Bacillus siamensis)于2010年被鉴定并正式命名[4]。研究表明,暹罗芽孢杆菌对革兰氏阳性菌和革兰氏阴性菌具有广谱的抑菌活性[5],还具有分泌α-淀粉酶、蛋白酶、纤维素酶的能力,有望作为饲料发酵剂和添加剂应用于畜禽生产当中[6]。近年来,研究者从不同来源分离获得多株暹罗芽孢杆菌菌株并开展了分离菌株的生物学特性研究。Xu等[7]在带鱼胃肠道中分离得到一株暹罗芽孢杆菌JF15,其对多种耐药水生细菌病原体具有拮抗作用。Gorai等[8]在鹰嘴豆的根瘤中分离得到一株对真菌具有广谱抑菌活性且具有良好宿主定殖潜力的B. siamensis CEN6。Gao等[9]从一株分离自海洋的暹罗芽孢杆菌的次级代谢产物中发现了5种新的大环内酰亚胺(macrolactin),其中3种对甘蔗黑穗病真菌具有显著的抑制作用。一株分离自中国传统发酵食物的B. siamensis CU-XJ-9能够分泌伊枯草菌素(iturin),对禾谷镰刀菌的生长具有抑制作用[10]。分离自泰国泡菜的B. siamensis B44v能够产生一种类似细菌素的物质,对革兰氏阳性菌和革兰氏阴性菌具有广谱抗菌活性,尤其是对鱼类病原体嗜水气单胞菌(Aeromonas hydrophila)和无乳链球菌(Streptococcus agalactiae)具有抑制作用;并且该菌能够在模拟肠道条件下存活并产生蛋白酶和纤维素酶,作为饲料添加剂促进鲶鱼生长[5]。这些研究结果表明,不同环境来源暹罗芽孢杆菌的益生特性具有一定差别,筛选具有优良益生性状的分离株并开展其益生机制尤其是抑菌机制的研究,将为暹罗芽孢杆菌的开发利用奠定重要基础。
本研究对B. siamensis CML548体外益生特性进行评价,包括抑制病原菌能力、耐酸能力、耐胆盐能力及产酶能力;同时对其进行高通量测序和基因组学分析,进一步探究其抑菌及潜在的益生机制,以期为B. siamensis CML548作为家禽益生菌的开发利用提供参考。
1 材料与方法 1.1 菌株菌株CML548分离于广东省云浮市矮项鸡的盲肠食糜,由本实验室保存。禽致病性大肠杆菌(Escherichia coli) CAU 0757、鼠伤寒沙门氏菌(Salmonella Typhimurium) CMCC 50115和产气荚膜梭菌(Clostridium perfringens) ATCC 13124为本实验室保存菌种。
1.2 培养基、主要试剂和仪器LB肉汤培养基,北京奥博星生物技术有限责任公司;FTG培养基,青岛高科技工业园海博生物技术有限公司;TSC培养基,青岛高科技工业园海博生物技术有限公司。QIAamp DNA Mini试剂盒,凯杰企业管理(上海)有限公司。Illumina HiSeq 2500测序平台,Illumina公司。
1.3 体外抑菌能力检测将沙门氏菌、大肠杆菌分别接种于LB肉汤培养基中,37 ℃、200 r/min培养12 h;将产气荚膜梭菌接种于FTG培养基中,37 ℃厌氧箱内培养12 h。挑取鸡源暹罗芽孢杆菌CML548单菌落接种于5 mL无菌LB肉汤中,37 ℃、200 r/min培养12 h后8 000 r/min离心10 min。
将OD600为0.7的沙门氏菌、大肠杆菌菌液以0.5%的体积含量加入含0.7%琼脂的LB培养基中并充分混匀;将OD600为0.7的产气荚膜梭菌菌液以0.5%的体积含量加入含0.7%琼脂的FTG培养基中并充分混匀。适时将含病原菌的软琼脂倾倒至放有牛津杯的LB固体培养基上,制作双层板。待上层培养基冷却后,移除牛津杯,向孔中添加50 μL芽孢杆菌上清液。每组进行4个重复,并以无菌LB液体培养基为阴性对照,氨苄西林为阳性对照。37 ℃培养24 h,观察抑菌结果,测量并记录抑菌圈直径。
1.4 产酶能力检测分别配制仅含5%脱脂奶粉和2%琼脂的固体培养基、仅含1%可溶性淀粉和2%琼脂的固体培养基、仅含1%羧甲基纤维素钠和2%琼脂的固体培养基,1×105 Pa高压灭菌15 min,冷却后,将培养基倾倒在放有牛津杯的培养皿中,待培养基凝固后,取出牛津杯。挑取鸡源暹罗芽孢杆菌CML548单菌落接种于5 mL无菌LB肉汤中,37 ℃、200 r/min培养12 h后8 000 r/min离心10 min,将100 μL芽孢杆菌发酵上清液加入孔内,每组进行5个重复,并以无菌LB液体培养基为空白对照,37 ℃培养24 h。检测产蛋白酶的培养基直接观察是否有水解透明圈;向检测产淀粉酶的培养基中加入2 mL碘液染色,观察是否有水解透明圈;向检测产纤维素酶的培养基中加入2 mL 0.2%刚果红溶液,显色30 min,倒出刚果红溶液,加入3 mL 1 mol/L NaCl溶液,洗脱15 min,观察是否有水解透明圈。测量并记录水解透明圈直径。
1.5 耐受性检测用HCl配制pH值为3.0、5.0的酸性LB液体培养基,用猪胆盐配制胆盐浓度为0.3% (质量体积分数)和0.5% (质量体积分数)的LB液体培养基。将鸡源暹罗芽孢杆菌CML548培养液按1% (体积分数)的量分别接种于上述培养基中,37 ℃、200 r/min培养2 h。取培养前和培养后的菌液涂板,并在37 ℃培养12 h后计数。通过处理前后活菌数的平均计数结果计算菌株的存活率。每组设置3个重复。存活率公式[11-13]为:

|
(1) |
将在−80 ℃低温冰箱保存的芽孢杆菌CML548划线接种于固体LB培养基上,37 ℃培养24 h,挑取单菌落接种于15 mL LB肉汤培养基,37 ℃培养24 h,取2 mL菌液于离心管中送至中国科学院微生物研究所测序。使用QIAamp DNA Mini试剂盒提取基因组DNA,使用Illumina HiSeq 2500测序平台进行全基因组测序。
1.7 基因组组装及基因组注释利用Unicycler[14] (V0.4.8)对clean data进行组装,得到基因组草图;然后使用Prokka[15] (V1.14.6)对基因组中的编码序列、tRNA、rRNA等进行注释。
1.8 系统发育树分析根据菌株的种属信息在NCBI上查找并下载同种菌株的基因组序列文件,利用kSNP[16] (V3.1)根据核心单核苷酸多态性(single nucleotide polymorphism, SNP)构建系统发育树。
1.9 基因功能分析利用BLAST将基因组序列在gene ontology (GO)[17-19]、Kyoto encyclopedia of genes and genomes (KEGG)[20]、cluster of orthologous groups of proteins (COG)[21]等数据库中进行功能注释。预测所得蛋白质通过dbCAN2 meta server[22] (https://bcb.unl.edu/dbCAN2/index.php)在carbohydrate-active enzymes (CAZy)数据库中比对得到B. siamensis CML548中可能的碳水化合物活性酶。从蛋白质序列注释文件中挑选出氨基酸数量小于100的蛋白质序列,利用抗菌肽数据库database of antimicrobial activity and structure of peptides (DBAASP)[23] (https://dbaasp.org/)预测基因组中潜在的抗菌肽编码基因。通过comprehensive antibiotic resistance database (CARD)数据库[24]注释耐药性相关基因;通过virulence factors of pathogenic bacteria (VFDB)数据库[25]注释菌株的毒力基因。
1.10 次级代谢产物预测利用antiSMASH V6.0.1[26] [antiSMASH bacterial version (secondarymetabolites.org)]在线软件,在细菌分析模块提交CML548菌株基因组序列,选择relaxed预测模式对菌株的次级代谢产物生物合成基因簇进行预测注释,参数选用默认值。
1.11 数据统计及可视化采用描述性统计和单因素方差分析(ANOVA)对数据进行统计;通过GraphPad Prism 9和ChiPlot (https://www.chiplot.online/)对实验数据和基因功能注释结果进行可视化处理;利用iTOL[27] (https://itol.embl.de/)进行发育树可视化;利用CGView sever[28] (https://cgview.ca/)绘制全基因组图谱。
2 结果与分析 2.1 抑菌及产酶能力采用双层板抑菌试验对52株鸡源芽孢杆菌的体外抑菌能力进行了筛选。相较而言,菌株CML548对沙门氏菌、大肠杆菌、产气荚膜梭菌均有较好的抑菌活性(表 1,图 1)。平板产酶实验结果表明,菌株CML548能够同时产生蛋白酶、纤维素酶和淀粉酶,其产生淀粉酶的能力最强(表 2,图 2)。
| Item | Inhibition zone (mm) | ||
| E. coli | S. Typhimurium | C. perfringens | |
| B. siamensis CML548 | ++ | ++ | ++ |
| LB (control) | − | − | − |
| AMP (control) | +++ | +++ | +++ |
| LB:LB液体培养基;AMP:氨苄;+:抑菌圈直径 < 10 mm;++:15 mm > 抑菌圈直径≥10 mm;+++:抑菌圈直径≥15 mm;−:无抑菌圈 LB: LB broth; AMP: Ampicillin; +: < 10 mm of inhibition; ++: Between 10 and 15 mm of inhibition; +++: 15 mm of inhibition and above; –: No inhibition. |
|||

|
| 图 1 鸡源暹罗芽孢杆菌CML548体外抑菌结果 Figure 1 In vitro antibacterial test results of Bacillus siamensis strain CML548 isolated from chicken. A:大肠杆菌. B:鼠伤寒沙门氏菌. C:产气荚膜梭菌. b:空白对照;CML:CML548抑菌效果 A: Escherichia coli. B: Salmonella Typhimurium. C: Clostridium perfringens. b: Blank control; CML: The antibacterial effect of CML548. |
|
|
| Type | Diameter of hydrolyzing zone (mm) |
| Protease | 14.4±0.42 |
| Cellulase | 12.0±0.1 |
| Amylase | 19.4±0.2 |
| LB broth | − |
| −:无水解圈 –: No hydrolyzing zone. |
|

|
| 图 2 鸡源暹罗芽孢杆菌CML548产酶结果 Figure 2 Capacity of producing enzyme of Bacillus siamensis strain CML548 isolated from chicken. A:蛋白酶. B:纤维素酶. C:淀粉酶. b:空白对照;CML:CML548产酶能力 A: Protease. B: Cellulase. C: Amylase. b: Blank control; CML: The enzyme production capacity of CML548. |
|
|
菌株CML548在不同pH值LB肉汤培养基中的生长情况如图 3所示。菌株在pH 3.0和pH 5.0环境中的存活率均大于90%,说明菌株CML548在酸性条件下的生长几乎不受影响。

|
| 图 3 鸡源暹罗芽孢杆菌CML548耐酸性实验结果 Figure 3 Acid tolerance test results of Bacillus siamensis strain CML548 isolated from chicken. |
|
|
CML548在不同胆盐浓度LB肉汤培养基中的存活情况如图 4所示。CML548在0.3%胆盐浓度下存活率高达95.25%,在0.5%胆盐浓度下存活率为89.10%,表明其具有较高的胆盐耐受能力。

|
| 图 4 鸡源暹罗芽孢杆菌CML548耐胆盐实验结果 Figure 4 Bile salt tolerance test results of Bacillus siamensis strain CML548 isolated from chicken. |
|
|
综上所述,CML548综合能力较为优良,在体外对于大肠杆菌、鼠伤寒沙门氏菌、产气荚膜梭菌具有良好的抑制效果,对酸性、胆盐有较高的耐受能力,并且能够产生蛋白酶、淀粉酶和纤维素酶。
2.3 鸡源暹罗芽孢杆菌CML548基因组的一般特征在生物学特性研究的基础上,进一步对其基因组进行了测序分析,以探究菌株CML548潜在的益生机制。
对样品测序结果进行组装和注释,得到CML548的全基因组图谱(图 5)。CML548菌株的全基因组序列全长为4 061 741 bp,GC含量为46.07%,预测其编码基因个数为3 961个,含有rRNA基因4个(23S rRNA基因1个、16S rRNA基因1个、5S rRNA基因2个),tRNA基因82个,tmRNA基因1个。

|
| 图 5 鸡源暹罗芽孢杆菌CML548的全基因组圈图 Figure 5 Whole genome map of Bacillus siamensis strain CML548 isolated from chicken. 由内到外分别是基因组序列位置坐标、GC含量、GC skew值、基因组、RNA(+)、RNA(−)、CDS(+)、CDS(−). 基因组GC含量:以窗口(染色体长度/1 000) bp,步长(染色体长度/1 000) bp来统计GC含量;向内的部分表示该区域GC含量低于全基因组平均GC含量,向外的部分与之相反,而且峰值越高表示与平均GC含量差值越大的基因组. GC skew值:窗口(染色体长度/1 000) bp,步长(染色体长度/1 000) bp,具体算法为(G−C)/(G+C);向内的粉色部分表示该区域G的含量低于C的含量,向外的绿色部分与之相反. 不同颜色的CDS即表示不同的COG类别 From the inside to the outside, the circles are position marker, GC content, GC skew value, genome, RNA(+), RNA(−), CDS(+), CDS(−). Genome GC content is calculated by window (chromosome length/1 000) bp and step size (chromosome length/1 000) bp; The inward part indicates that the GC content of the region is lower than the average GC content of the whole genome, while the outward part is the opposite, and the higher the peak value, the greater the difference from the average GC content. GC skew value: Window (chromosome length/1 000) bp, step-size (chromosome length/1 000) bp, the specific algorithm is (G−C)/(G+C); The inward pink part indicates that the content of G is lower than that of C in this region, and the outward green part is the opposite. Different color of CDSs represent different COG categories. |
|
|
根据基因组数据库分类法GTDB对菌株进行分类,菌株CML548归属于厚壁菌门(Firmicutes)暹罗芽孢杆菌(B. siamensis)。为明确其与已知暹罗芽孢杆菌菌株的进化关系,对来自NCBI的7株暹罗芽孢杆菌和菌株CML548构建基于核心SNP的系统发育树(图 6)。与CML548进化关系最接近的菌株是分离自海底沉积物的B. siamensis SCSIO 05746[28]。

|
| 图 6 鸡源暹罗芽孢杆菌CML548基于核心SNP基因序列构建的系统发育树 Figure 6 Phylogenetic tree of Bacillus siamensis strain CML548 isolated from chicken based on core SNP gene sequences. 括号内为各菌株在GenBank中的序列号;分支上数字表示进化自展支持率;比例尺显示水平线的长度,代表碱基替换数 In brackets is the sequence number of each strain in GenBank; The number on the branch indicates bootstrap; The scale bar represents the horizontal branch lengths and the number of substitutions per nucleotide position. |
|
|
菌株CML548基因组中分别有1 693、2 704、3 413、186、67、1、5个基因被GO、KEGG、COG、CAZy、DBAASP、CARD、VFDB数据库注释;通过antiSMASH预测到16个与次级代谢产物合成相关的基因簇;此外,基因比对分析发现该菌株基因组中含有tasA基因。
2.5.1 GO数据库注释结果在GO功能分析中,分别有1 464、1 440和1 112个基因注释到生物过程(biological process)、分子功能(molecular function)和细胞组分(cellular component)。其中,孢子形成(GO:0030435,174个基因)、碳水化合物代谢(GO:0005975,128个基因)、磷酸化作用(GO:0016310,102个基因)是生物过程中涉及的主要功能。在分子功能上,最主要的生物活性有ATP结合(GO:0005524;266个基因)、氧化还原酶活性(GO:0016491,187个基因)、转移酶活性和转移含磷基团(GO:0016772,140个基因)、过渡金属离子结合(GO:0046914,103个基因)、裂解酶活性(GO:0016829,102个基因)。膜组分(GO:0016021,319个基因)是细胞组分中的主要功能单元(图 7)。此外,在CML548基因组中还注释到多个与酸(34个基因)、胆盐(3个基因)、压力(12个基因)等应激耐受相关基因。
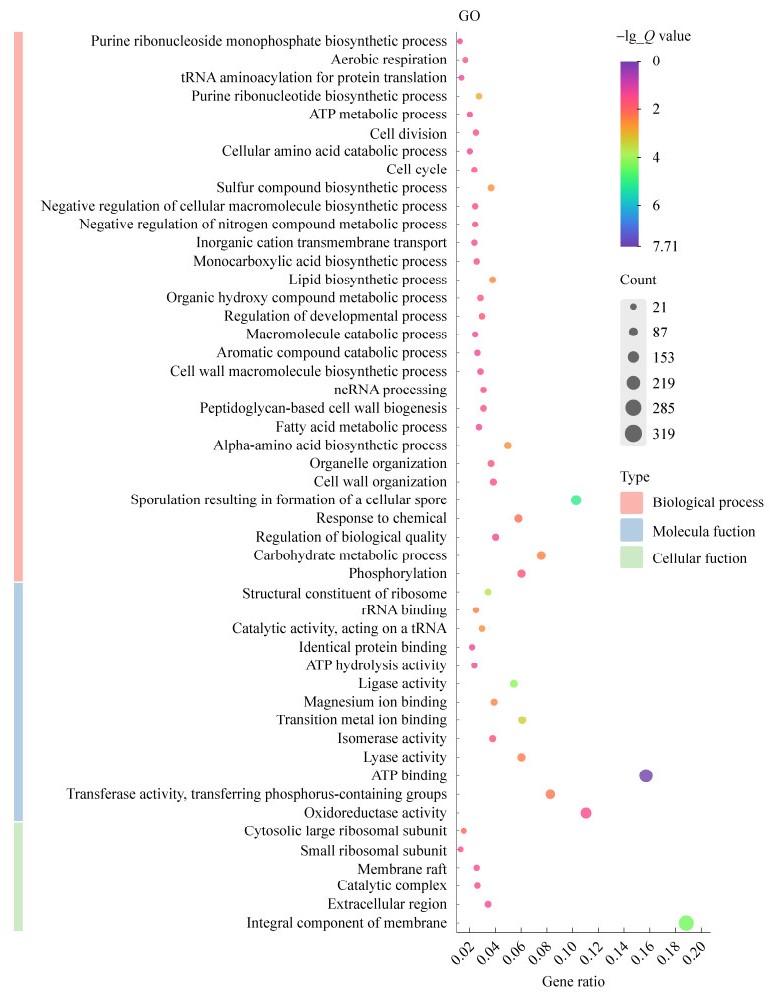
|
| 图 7 鸡源暹罗芽孢杆菌CML548基因组的GO功能分类 Figure 7 GO functional classification of Bacillus siamensis strain CML548 isolated from chicken genome. |
|
|
菌株CML548在KEGG数据库中共有2 704个基因分别在细胞过程、环境信息处理、遗传信息处理、人类疾病和新陈代谢五大功能的21条通路上得到注释(图 8)。其中参与新陈代谢的基因最多,占全部基因的92.79% (2 509个),12条代谢通路中碳水化合物代谢和氨基酸代谢的占比较高,分别占代谢通路注释基因(2 509个)的12.28%和10.88%。
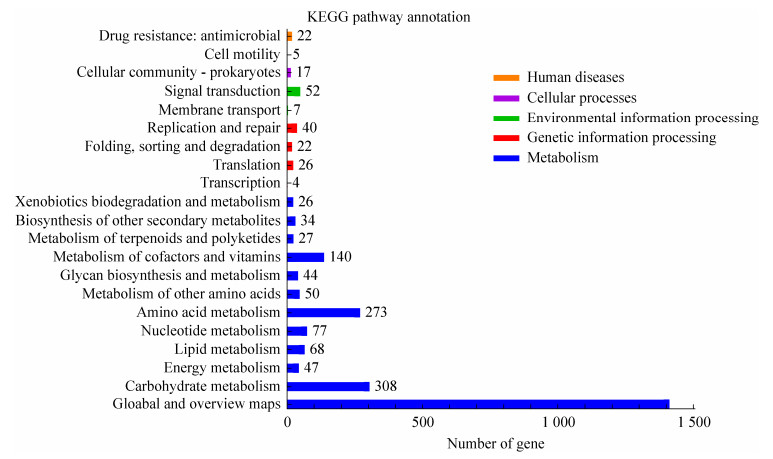
|
| 图 8 鸡源暹罗芽孢杆菌CML548基因组的KEGG代谢通路分类图 Figure 8 KEGG metabolic pathway classification of Bacillus siamensis strain CML548 isolated from chicken genome. |
|
|
将菌株CML548基因组中的蛋白编码基因进行COG注释,共有3 413个蛋白基因得到注释(图 9)。其中,未知功能基因(S: function unknown)的占比最高,共有867个(25.40%)。此外,含有基因数量较多的功能分类依次为:306个(8.97%)基因参与转录(K: transcription),265个(7.76%)基因与氨基酸转运与代谢(E: amino acid transport and metabolism)相关,233个(6.83%)基因参与能量生产和转换(C: energy production and conversion)、227个(6.65%)基因涉及碳水化合物运输和代谢(G: carbohydrate transport and metabolism)。
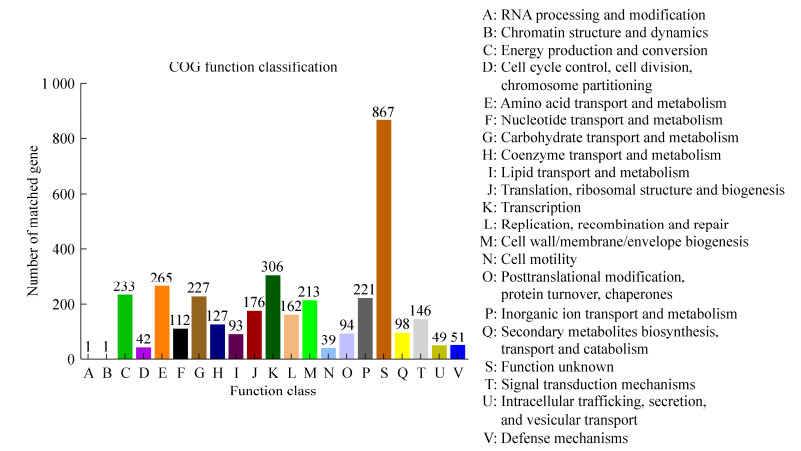
|
| 图 9 鸡源暹罗芽孢杆菌CML548基因组的COG功能分类 Figure 9 COG functional classification of Bacillus siamensis strain CML548 isolated from chicken genome. |
|
|
CAZy数据库是碳水化合物酶相关的专业数据库。通过CAZy数据库注释发现CML548菌株中共有186个基因能够编码碳水化合物酶(表 3),其中糖苷转移酶的基因最多(43.01%),糖苷水解酶次之(40.86%)。在糖苷水解酶中,纤维素酶基因(GH5家族)共有2个,淀粉酶基因(GH13家族)有13个。此外,有38个基因注释到碳水化合物结合结构域,其中28个注释到CBM50家族,该家族与降解肽聚糖和几丁质有关。
| Functional classification | Number of genes |
| 糖苷水解酶Glycoside hydrolases (GHs) | 76 |
| 糖基转移酶Glycosyl transferases (GTs) | 80 |
| 多糖裂解酶Polysaccharide lyases (PLs) | 3 |
| 糖类酯解酶Carbohydrate esterases (CEs) | 20 |
| 氧化还原酶Auxiliary activities (AAs) | 6 |
| 碳水化合物结合结构域 Carbohydrate-binding module (CBM) |
38 |
从蛋白质序列注释文件中挑选出氨基酸数量小于100的蛋白质序列,利用抗菌肽数据库(DBAASP)分析基因组中的抗菌肽编码基因,共预测到67个抗菌肽相关编码基因,其中37个基因的预测值(predictive value)≥85%,对E. coli、S. Typhimurium和C. perfringens具有潜在抑菌活性的抗菌肽基因分别有22、14和1个。
2.5.6 耐药基因分析使用CARD数据库,以identity≥80%为条件,发现菌株CML548基因组中仅含有1个耐药基因Cfr。Cfr隶属于Cfr 23S核糖体RNA甲基转移酶(Cfr 23S ribosomal RNA methyltransferase) AMR基因家族,对5类抗生素包括林可酰胺类抗生素(lincosamide antibiotic)、链霉素类抗生素(streptogramin antibiotic)、恶唑烷类抗生素(oxazolidinone antibiotic)、苯酚类抗生素(phenicol antibiotic)、胸膜致病菌类抗生素(pleuromutilin antibiotic)具有抗性。
2.5.7 毒力基因注释结果将CML548菌株的基因的氨基酸序列与VFDB数据库进行比对,以identity≥70%作为筛选条件,在菌株CML548中共找到5个毒力基因(表 4);其中2个基因与细菌黏附(adherence)相关,3个分别与免疫调控(immune modulation)、营养代谢因子(nutritional/metabolic factor)和应激生存(stress survival)相关。
| Sequence | Gene | Description | VF category | E-value | Identity (%) | Length (bp) |
| VFG048830 | gndA | NADP-dependent phosphogluconate dehydrogenase | Immune modulation | 0 | 70 | 468 |
| VFG012095 | groEL | Chaperonin GroEL | Adherence | 0 | 73 | 542 |
| VFG050015 | dhbE | 2, 3-dihydroxybenzoate adenylase DhbE | Nutritional/Metabolic factor | 0 | 72 | 538 |
| VFG000079 | clpC | Endopeptidase Clp ATP-binding chain C | Stress survival | 0 | 78 | 820 |
| VFG046465 | tufA | Elongation factor Tu | Adherence | 1.78×10−68 | 74 | 394 |
通过antiSMASH预测分析,在菌株CML548基因组中共检测到16个与次级代谢产物合成相关的基因簇(表 5)。这些基因簇可能编码的代谢产物包括以非核糖体途径(non-ribosomalpeptide synthetase, NRPS)合成的丰霉素(fengycin)、表面活性素(surfactin)、多肽类细菌素儿茶酚型杆菌巴汀(bacillibactin)等;以PKS途径(polyketide biosynthase, PKS)合成的大环内酯类化合物大环内酰亚胺H (macrolactin H)、多烯类化合物杆菌烯(bacillaene)、丁酰苷菌素A/丁酰苷菌素B (butirosin A/butirosin B)及艰难菌素(difficidin)等;这些代谢产物与抗菌和螯合铁离子等功能密切相关。其中,bacilysin、macrolactin H、fengycin、bacillaene、surfactin和bacillibactin的预测合成基因簇与已知基因簇的相似性分别为100%、100%、86%、100%、82%和100%。
| Region | Gene cluster type | Length (bp) | Most similar known cluster | Identity (%) |
| 1.1 | Other | 302 249−343 667 | Bacilysin | 100 |
| 2.1 | transAT-PKS | 20 466−108 677 | Macrolactin H | 100 |
| 2.2 | Lanthipeptide-class-ii | 272 631−301 520 | − | − |
| 2.3 | Terpene | 420 223−440 963 | − | − |
| 2.4 | PKS-like | 522 986−564 230 | Butirosin A/Butirosin B | 7 |
| 3.1 | transAT-PKS | 530 939−564 796 | Difficidin | 46 |
| 4.1 | NRPS, betalactone, transAT-PKS | 1−97 712 | Fengycin | 86 |
| 4.2 | transAT-PKS, NRPS, T3PKS | 161 379−264 019 | Bacillaene | 100 |
| 5.1 | NRPS, transAT-PKS | 23 264−100 950 | Locillomycin | 35 |
| 5.2 | NRPS | 170 537−235 944 | Surfactin | 82 |
| 6.1 | Terpene | 44 082−65 965 | − | − |
| 6.2 | T3PKS | 134 609−175 709 | − | − |
| 7.1 | transAT-PKS-like | 1−45 748 | Difficidin | 53 |
| 9.1 | NRPS, RiPP-like | 73 637−125 430 | Bacillibactin | 100 |
| 13.1 | transAT-PKS-like | 1−22 783 | Difficidin | 26 |
| 15.1 | NRPS | 1−12 962 | Fengycin | 20 |
| −:未匹配到最相似的已知基因簇 −: No similar known gene clusters are matched. |
||||
芽孢杆菌中普遍存在TasA蛋白,具有广谱抑菌作用,是芽孢杆菌属微生物重要的抑菌活性蛋白。通过基因比对分析,暹罗芽孢杆菌CML548基因组中含有tasA基因,位于基因组起始位置470 011 bp,终止位置470 796 bp,基因全长786 bp,编码261个氨基酸。其编码蛋白与来源于枯草芽孢杆菌(B. subtilis) strain 168 TasA蛋白的相似性为83.9%。
3 讨论与结论近年来,益生菌作为抗生素的替代品受到广泛关注[29]。研究发现,芽孢杆菌具有良好的抗逆特性[30],便于在动物肠道中定殖;能够分泌多种酶类,提高饲料利用率[31];能减少肠道中有害微生物的数量[32],调节动物肠道菌群,具有良好的益生潜力。
本研究从广东省云浮市矮项鸡中分离获得一株芽孢杆菌CML548,经16S rRNA基因鉴定为暹罗芽孢杆菌(B. siamensis),后对其抑菌能力和生物学特性进行研究,发现其对于大肠杆菌、鼠伤寒沙门氏菌、产气荚膜梭菌均具有良好的抑制效果。与我们的研究结果类似,从牛角瓜中分离到的B. siamensis AUC的提取物对大肠杆菌、伤寒沙门氏菌具有抗菌活性[33];从韩国发酵泡菜分离得到的B. siamensis B28体外对大肠杆菌和沙门氏菌的生长有抑制作用[34];分离自泰国泡菜B. siamensis B44v对革兰氏阳性菌和阴性菌具有广谱的抑菌活性[5]。益生菌在动物体内发挥益生作用的重要前提是对胃酸和胆盐具有一定的耐受能力[35]。此前研究表明,暹罗芽孢杆菌能够在NaCl浓度为0%−14%、pH值范围为4.5−9.0的环境下正常生长[4]。我们的结果同样显示菌株CML548对酸和胆盐均具有较高的耐受性,并且能够产生多种胞外酶,如蛋白酶、淀粉酶和纤维素酶,这些生物学性质说明该菌株是一个潜在的具有重要应用价值的新菌株。
全基因组测序分析有助于了解菌株所具有的潜在益生功能及相关分子机制[36]。本研究采用二代测序技术对菌株CML548进行全基因组测序,组装后获得基因组草图。Oliveira等[37]报道,ATP合酶(ATP synthase)、GTP焦磷酸激酶和丙酮酸激酶(GTP pyrophosphokinase and pyruvate kinase)、CTP合酶(CTP synthase)、伴侣蛋白(chaperone protein)等在细菌耐酸或耐碱过程中发挥着重要作用。与益生菌Bacillus coagulans HS243[38]和Bacillus velezensis ZBG17[39]相同,CML548基因组中同样注释到相应蛋白的编码基因。此外,在GO功能分析中174个基因富集到孢子形成的生物过程中。这些结果为菌株CML548的耐酸、耐胆盐和抵抗不利环境的能力提供了依据。COGs分类注释结果表明该菌株基因组中参与代谢的基因(1 376个,39.06%)最多,另有很大一部分基因的功能未知,有待进一步研究。B. siamensis CML548分离自鸡的盲肠食糜,食糜中含有包括蛋白和膳食纤维在内的多种营养物质;而菌株CML548含有多个参与氨基酸的运输和代谢途径、碳水化合物的运输和代谢途径相关的基因,可能为其在肠道环境中对这些营养物质的利用提供了生存优势。CAZy数据库注释发现该菌株含有13个淀粉酶基因和2个纤维素酶基因,为菌株CML548降解淀粉和纤维素的表型提供了分子基础。此外,发现其基因组中含有28个与降解几丁质和肽聚糖有关的基因,可能与该菌株破坏病原菌的细胞壁有关。值得注意的是,DBAASP数据库分析发现CML548基因组中携带大量抗菌肽相关编码基因(67个)。同时,菌株CML548含有TasA蛋白编码基因tasA。TasA蛋白是芽孢杆菌属中普遍存在的一种蛋白质,最早从B. subtilis strain 168中被分离鉴定;该蛋白对于多种革兰氏阳性菌和革兰氏阴性菌具有抑菌活性[40]。众多抗菌肽编码基因及TasA蛋白编码基因的存在,可能是菌株CML548对多种病原菌具有抑菌活性的重要原因。
另外,CML548具有抑制病原菌的能力也可能是因为其基因组中多个与抗菌活性相关的次级代谢产物(环肽类、多烯类等化合物)编码基因簇的存在(16个基因簇)。其中,3个基因簇与已报道的B. velezensis FZB42[41]合成bacilysin、macrolactin H、bacillaene基因簇的相似性完全一致;另有2个基因簇与该菌株合成surfactin、fengycin基因簇具有82%、86%的相似性。此外,CML548含有bacillibactin的合成基因簇,与B. subtilis 168[42]所含基因簇的相似性为100%。已知环脂肽类化合物表面活性素(surfactin)、多烯类化合物杆菌烯(bacillaene)、大环内酯类化合物大环内酰亚胺(macrolactin)和艰难菌素(difficidin),以及多肽类细菌素儿茶酚型杆菌巴汀(bacillibactin)、二肽类环状结构化合物杆菌溶素(bacilysin)、环状肽结构广谱细菌素淀粉环霉素(amylocyclicin)和噁唑/噻唑型窄谱抗线虫细菌素车前唑霉素(plantazolicin)等物质均具有抗细菌活性[43],因此可以推测菌株CML548可能通过合成上述化合物从而发挥对多种病原菌的抑菌作用。
耐药基因分析结果显示菌株CML548仅含有耐药基因Cfr,该基因编码甲基化酶,催化23S rRNA亚基位点8的腺嘌呤-2503甲基化,从而引起抗生素靶点改变。23S rRNA在该位点的甲基化使其对多种抗生素产生耐药性,包括链霉素、氯霉素、氟苯尼醇、利奈唑类和克林霉素[44]。未发现菌株CML548基因组中含有可移动耐药基因,说明其传播耐药基因的可能性较低。此外,虽然VFDB数据库注释结果显示该菌含有5个潜在的毒力因子编码基因,但这些基因与已知毒力基因的相似性均低于80%,可能并非真正的毒力基因。上述结果说明菌株CML548具有较高的安全性。
综上所述,菌株CML548具有优良的益生特性,其基因组中含有大量的酶基因、与抗菌活性相关的基因和基因簇;该菌株有望成为饲用益生菌制剂的重要候选菌株。
致谢: 衷心感谢动物营养学国家重点实验室提供设备与技术支持。| [1] |
YANG JC, ZHOU J, BIAN Y, MA HF, LI FW, LI GM. Trend and thinking of poultry industry development in China[J]. China Animal Industry, 2018(13): 26-29. (in Chinese) 杨景晁, 周键, 边媛, 马红芳, 李福伟, 李桂明. 我国家禽业发展的趋势与思考[J]. 中国畜牧业, 2018(13): 26-29. DOI:10.3969/j.issn.2095-2473.2018.13.014 |
| [2] |
CHAO D, SHI YM, YUAN JB, YANG M. Advance and prospect of growth-promoting feed additives after prohibiting addition of antibiotics[J]. Advances in Microbiology, 2015, 4(2): 36-43. DOI:10.12677/AMB.2015.42006 |
| [3] |
HAN M, WANG CM, LIU P, LI DF, LI Y, MA X. Dietary fiber gap and host gut microbiota[J]. Protein and Peptide Letters, 2017, 24(5): 388-396. DOI:10.2174/0929866524666170220113312 |
| [4] |
SUMPAVAPOL P, TONGYONK L, TANASUPAWAT S, CHOKESAJJAWATEE N, LUXANANIL P, VISESSANGUAN W. Bacillus siamensis sp. nov., isolated from salted crab (poo-khem) in Thailand[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(Pt 10): 2364-2370. |
| [5] |
MEIDONG R, DOOLGINDACHBAPORN S, JAMJAN W, SAKAI KJ, TASHIRO Y, OKUGAWA Y, TONGPIM S. A novel probiotic Bacillus siamensis B44v isolated from Thai pickled vegetables (Phak-Dong) for potential use as a feed supplement in aquaculture[J]. The Journal of General and Applied Microbiology, 2017, 63(4): 246-253. DOI:10.2323/jgam.2016.12.002 |
| [6] |
LI D, WANG JH, NIU HH, MIAO XY, CHI YP, JIANG YY, LIU XY, KANG LN, LI YQ. Feed containing bacillus siamensis: China, CN108208316A[P]. 2021-02-02 (in Chinese). 李达, 王景会, 牛红红, 苗欣宇, 迟燕平, 姜媛媛, 刘香英, 康立宁, 李玉秋. 含有暹罗芽孢杆菌的饲料: 中国, CN108208316A[P]. 2021-02-02. |
| [7] |
XU BH, YE ZW, ZHENG QW, WEI T, LIN JF, GUO LQ. Isolation and characterization of cyclic lipopeptides with broad-spectrum antimicrobial activity from Bacillus siamensis JFL15[J]. 3 Biotech, 2018, 8(10): 444. DOI:10.1007/s13205-018-1443-4 |
| [8] |
GORAI PS, GHOSH R, MANDAL S, GHOSH S, CHATTERJEE S, GOND SK, MANDAL NC. Bacillus siamensis CNE6- a multifaceted plant growth promoting endophyte of Cicer arietinum L. having broad spectrum antifungal activities and host colonizing potential[J]. Microbiological Research, 2021, 252: 126859. DOI:10.1016/j.micres.2021.126859 |
| [9] |
GAO CH, CHEN XQ, YU L, JIANG L, PAN DJ, JIANG S, GAN YM, LIU YH, YI XX. New 24-membered macrolactins isolated from marine bacteria Bacillus siamensis as potent fungal inhibitors against sugarcane smut[J]. Journal of Agricultural and Food Chemistry, 2021, 69(15): 4392-4401. DOI:10.1021/acs.jafc.0c07415 |
| [10] |
HUANG YH, ZHANG XR, XU H, ZHANG F, ZHANG XL, YAN YH, HE LZ, LIU JJ. Isolation of lipopeptide antibiotics from Bacillus siamensis: a potential biocontrol agent for Fusarium graminearum[J]. Canadian Journal of Microbiology, 2022, 68(6): 403-411. DOI:10.1139/cjm-2021-0312 |
| [11] |
SHEHATA MG, EL SOHAIMY SA, EL-SAHN MA, YOUSSEF MM. Screening of isolated potential probiotic lactic acid bacteria for cholesterol lowering property and bile salt hydrolase activity[J]. Annals of Agricultural Sciences, 2016, 61(1): 65-75. DOI:10.1016/j.aoas.2016.03.001 |
| [12] |
XU C, WEI FX, YANG XY, FENG YQ, LIU D, HU YF. Lactobacillus salivarius CML352 isolated from Chinese local breed chicken modulates the gut microbiota and improves intestinal health and egg quality in late-phase laying hens[J]. Microorganisms, 2022, 10(4): 726. DOI:10.3390/microorganisms10040726 |
| [13] |
LIU WJ, CHEN MX, DUO LN, WANG JC, GUO S, SUN HT, MENGHE B, ZHANG HP. Characterization of potentially probiotic lactic acid bacteria and bifidobacteria isolated from human colostrum[J]. Journal of Dairy Science, 2020, 103(5): 4013-4025. DOI:10.3168/jds.2019-17602 |
| [14] |
WICK RR, JUDD LM, GORRIE CL, HOLT KE. Unicycler: Resolving bacterial genome assemblies from short and long sequencing reads[J]. PLoS Computational Biology, 2017, 13(6): e1005595. DOI:10.1371/journal.pcbi.1005595 |
| [15] |
SEEMANN T. Prokka: rapid prokaryotic genome annotation[J]. Bioinformatics, 2014, 30(14): 2068-2069. DOI:10.1093/bioinformatics/btu153 |
| [16] |
GARDNER SN, SLEZAK T, HALL BG. kSNP3.0: SNP detection and phylogenetic analysis of genomes without genome alignment or reference genome[J]. Bioinformatics, 2015, 31(17): 2877-2878. DOI:10.1093/bioinformatics/btv271 |
| [17] |
MI HY, MURUGANUJAN A, EBERT D, HUANG XS, THOMAS PD. PANTHER version 14: more genomes, a new PANTHER GO-slim and improvements in enrichment analysis tools[J]. Nucleic Acids Research, 2019, 47(D1): D419-D426. DOI:10.1093/nar/gky1038 |
| [18] |
ASHBURNER M, BALL CA, BLAKE JA, BOTSTEIN D, BUTLER H, CHERRY JM, DAVIS AP, DOLINSKI K, DWIGHT SS, EPPIG JT, HARRIS MA, HILL DP, ISSEL-TARVER L, KASARSKIS A, LEWIS S, MATESE JC, RICHARDSON JE, RINGWALD M, RUBIN GM, SHERLOCK G. Gene Ontology: tool for the unification of biology[J]. Nature Genetics, 2000, 25(1): 25-29. DOI:10.1038/75556 |
| [19] |
CONSORTIUM GO. The gene ontology resource: enriching a GOld mine[J]. Nucleic Acids Research, 2021, 49(D1): D325-D334. DOI:10.1093/nar/gkaa1113 |
| [20] |
KANEHISA M, FURUMICHI M, SATO Y, ISHIGURO-WATANABE M, TANABE M. KEGG: integrating viruses and cellular organisms[J]. Nucleic Acids Research, 2021, 49(D1): D545-D551. DOI:10.1093/nar/gkaa970 |
| [21] |
HUERTA-CEPAS J, SZKLARCZYK D, HELLER D, HERNÁNDEZ-PLAZA A, FORSLUND SK, COOK H, MENDE DR, LETUNIC I, RATTEI T, JENSEN LJ, von MERING C, BORK P. eggNOG 5.0: a hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses[J]. Nucleic Acids Research, 2019, 47(D1): D309-D314. DOI:10.1093/nar/gky1085 |
| [22] |
ZHANG H, YOHE T, HUANG L, ENTWISTLE S, WU PZ, YANG ZL, BUSK PK, XU Y, YIN YB. dbCAN2: a meta server for automated carbohydrate-active enzyme annotation[J]. Nucleic Acids Research, 2018, 46(W1): W95-W101. DOI:10.1093/nar/gky418 |
| [23] |
PIRTSKHALAVA M, AMSTRONG AA, GRIGOLAVA M, CHUBINIDZE M, ALIMBARASHVILI E, VISHNEPOLSKY B, GABRIELIAN A, ROSENTHAL A, HURT DE, TARTAKOVSKY M. DBAASP v3: database of antimicrobial/cytotoxic activity and structure of peptides as a resource for development of new therapeutics[J]. Nucleic Acids Research, 2021, 49(D1): D288-D297. DOI:10.1093/nar/gkaa991 |
| [24] |
ALCOCK BP, RAPHENYA AR, LAU TTY, TSANG KK, BOUCHARD M, EDALATMAND A, HUYNH W, NGUYEN AL V, CHENG AA, LIU SH, MIN SY, MIROSHNICHENKO A, TRAN HK, WERFALLI RE, NASIR JA, OLONI M, SPEICHER DJ, FLORESCU A, SINGH B, FALTYN M, et al. CARD 2020: antibiotic resistome surveillance with the comprehensive antibiotic resistance database[J]. Nucleic Acids Research, 2020, 48(D1): D517-D525. |
| [25] |
LIU B, ZHENG DD, JIN Q, CHEN LH, YANG J. VFDB 2019: a comparative pathogenomic platform with an interactive web interface[J]. Nucleic Acids Research, 2019, 47(D1): D687-D692. DOI:10.1093/nar/gky1080 |
| [26] |
BLIN K, SHAW S, KLOOSTERMAN AM, CHARLOP-POWERS Z, van WEZEL GP, MEDEMA MH, WEBER T. antiSMASH 6.0: improving cluster detection and comparison capabilities[J]. Nucleic Acids Research, 2021, 49(W1): W29-W35. DOI:10.1093/nar/gkab335 |
| [27] |
LETUNIC I, BORK P. Interactive Tree Of Life (iTOL): an online tool for phylogenetic tree display and annotation[J]. Bioinformatics, 2007, 23(1): 127-128. DOI:10.1093/bioinformatics/btl529 |
| [28] |
STOTHARD P, WISHART DS. Circular genome visualization and exploration using CGView[J]. Bioinformatics, 2005, 21(4): 537-539. DOI:10.1093/bioinformatics/bti054 |
| [29] |
WANG AW. How should breeders deal with future intestinal problems after feed resistance is banned?[J]. Animals Breeding and Feed, 2020, 19(10): 101-102. (in Chinese) 王安文. 饲料禁抗后, 养殖者该如何应对未来的肠道问题[J]. 养殖与饲料, 2020, 19(10): 101-102. DOI:10.3969/j.issn.1671-427X.2020.10.049 |
| [30] |
ZHANG F, HAN SM, LI Q, SUN SY, LI JC, WEI GS, LI YB. Application of microbial feed additives in modern livestock and poultry breeding[J]. Animal Husbandry and Feed Science, 2019, 40(1): 60-62. (in Chinese) 张帆, 韩淑敏, 李琦, 孙思怡, 李井春, 魏国生, 李雁冰. 微生物饲料添加剂在现代畜禽养殖中的应用[J]. 畜牧与饲料科学, 2019, 40(1): 60-62. DOI:10.16003/j.cnki.issn1672-5190.2019.01.015 |
| [31] |
CHEN M, ZHANG X, ZHU AP. Study on technological parameters of Bacillus coagulans culture and industrial production[J]. Heilongjiang Animal Science and Veterinary Medicine, 2017(24): 183-185, 297. (in Chinese) 陈明, 张欣, 朱爱萍. 凝结芽孢杆菌培养及工业化生产工艺参数研究[J]. 黑龙江畜牧兽医, 2017(24): 183-185, 297. DOI:10.13881/j.cnki.hljxmsy.2017.2108 |
| [32] |
XU P, DONG XF, TONG JM. Microbial feed additives: major functions and its research advances[J]. Chinese Journal of Animal Nutrition, 2012, 24(8): 1397-1403. (in Chinese) 徐鹏, 董晓芳, 佟建明. 微生物饲料添加剂的主要功能及其研究进展[J]. 动物营养学报, 2012, 24(8): 1397-1403. DOI:10.3969/j.issn.1006-267x.2012.08.002 |
| [33] |
HAGAGGI NSA, MOHAMED AAA. Plant-bacterial endophyte secondary metabolite matching: a case study[J]. Archives of Microbiology, 2020, 202(10): 2679-2687. DOI:10.1007/s00203-020-01989-7 |
| [34] |
HEO S, KIM JH, KWAK MS, JEONG DW, SUNG MH. Functional genomic insights into probiotic Bacillus siamensis strain B28 from traditional Korean fermented Kimchi[J]. Foods (Basel, Switzerland), 2021, 10(8): 1906. |
| [35] |
FENG YY, QIAO L, YAO HM, LIU R, JIE LX, GAO CB. Research progress on the evaluation methods of safety and efficacy of probiotics[J]. China Animal Husbandry & Veterinary Medicine, 2017, 44(7): 2022-2032. (in Chinese) 冯媛媛, 乔琳, 姚宏明, 刘蕊, 介琳霞, 高长斌. 益生菌安全性和有效性评价方法的研究进展[J]. 中国畜牧兽医, 2017, 44(7): 2022-2032. DOI:10.16431/j.cnki.1671-7236.2017.07.018 |
| [36] |
BECK BR, PARK GS, LEE YH, IM S, JEONG DY, KANG J. Whole genome analysis of Lactobacillus plantarum strains isolated from kimchi and determination of probiotic properties to treat mucosal infections by Candida albicans and Gardnerella vaginalis[J]. Frontiers in Microbiology, 2019, 10: 433. DOI:10.3389/fmicb.2019.00433 |
| [37] |
OLIVEIRA LC, SARAIVA TDL, SILVA WM, PEREIRA UP, CAMPOS BC, BENEVIDES LJ, ROCHA FS, FIGUEIREDO HCP, AZEVEDO V, SOARES SC. Analyses of the probiotic property and stress resistance-related genes of Lactococcus lactis subsp. lactis NCDO 2118 through comparative genomics and in vitro assays[J]. PLoS One, 2017, 12(4): e0175116. DOI:10.1371/journal.pone.0175116 |
| [38] |
KAPSE NG, ENGINEER AS, GOWDAMAN V, WAGH S, DHAKEPHALKAR PK. Functional annotation of the genome unravels probiotic potential of Bacillus coagulans HS243[J]. Genomics, 2019, 111(4): 921-929. DOI:10.1016/j.ygeno.2018.05.022 |
| [39] |
SONI R, KEHARIA H, BOSE A, PANDIT N, DOSHI J, RAO SVR, PAUL SS, RAJU MVLN. Genome assisted probiotic characterization and application of Bacillus velezensis ZBG17 as an alternative to antibiotic growth promoters in broiler chickens[J]. Genomics, 2021, 113(6): 4061-4074. DOI:10.1016/j.ygeno.2021.10.012 |
| [40] |
STÖVER AG, DRIKS A. Secretion, localization, and antibacterial activity of TasA, a Bacillus subtilis spore-associated protein[J]. Journal of Bacteriology, 1999, 181(5): 1664-1672. DOI:10.1128/JB.181.5.1664-1672.1999 |
| [41] |
KOUMOUTSI A, CHEN XH, HENNE A, LIESEGANG H, HITZEROTH G, FRANKE P, VATER J, BORRISS R. Structural and functional characterization of gene clusters directing nonribosomal synthesis of bioactive cyclic lipopeptides in Bacillus amyloliquefaciens strain FZB42[J]. Journal of Bacteriology, 2004, 186(4): 1084-1096. DOI:10.1128/JB.186.4.1084-1096.2004 |
| [42] |
BARBE V, CRUVEILLER S, KUNST F, LENOBLE P, MEURICE G, SEKOWSKA A, VALLENET D, WANG TZ, MOSZER I, MÉDIGUE C, DANCHIN A. From a consortium sequence to a unified sequence: the Bacillus subtilis 168 reference genome a decade later[J]. Microbiology (Reading, England), 2009, 155(Pt 6): 1758-1775. |
| [43] |
LI XX, GAO XJ, ZHANG SM, JIANG ZY, YANG H, LIU XD, JIANG Q, ZHANG XJ. Characterization of a Bacillus velezensis with antibacterial activity and inhibitory effect on common aquatic pathogens[J]. Aquaculture, 2020, 523: 735165. DOI:10.1016/j.aquaculture.2020.735165 |
| [44] |
HANSEN LH, PLANELLAS MH, LONG KS, VESTER B. The order Bacillales hosts functional homologs of the worrisome cfr antibiotic resistance gene[J]. Antimicrobial Agents and Chemotherapy, 2012, 56(7): 3563-3567. DOI:10.1128/AAC.00673-12 |
 2023, Vol. 50
2023, Vol. 50




