扩展功能
文章信息
- 彭方, 彭晓娅, 方贻霄
- PENG Fang, PENG Xiaoya, FANG Yixiao
- 极地微生物资源的研究现状及挑战
- Research status and challenges of polar microbial resources
- 微生物学通报, 2023, 50(12): 5505-5517
- Microbiology China, 2023, 50(12): 5505-5517
- DOI: 10.13344/j.microbiol.china.230158
-
文章历史
- 收稿日期: 2023-03-01
- 接受日期: 2023-07-12
- 网络首发日期: 2023-08-29
北极是一个被陆地包围的海洋,南极洲则是一个被海洋包围的大陆,两极是地球上地理位置最遥远的区域,也是最荒凉和被探索最少的环境。在极地低温、干燥、强辐照和低养分的环境中形成了独特的微生物生态系统,这不仅为研究原始(即人类活动影响有限)区域内的微生物生态和进化提供了极好的机会,也十分有利于筛选和获得独特的微生物资源。南极半岛是地球上变暖最快的地区之一,一些北极地区也正在经历气温的快速上升;这些环境变化会影响地球的气候,并导致未被发现的生物多样性的丧失;在这种情况下,对极地微生物的生物勘探具有重要意义[1]。
随着生物技术和基因技术的进步,在对极地的生物勘探中人们对寻找和利用新的(有价值的)微生物材料和遗传资源越来越感兴趣。通过对极地苔原、雪、冰川、冻土、海洋、淡水、湖泊等环境的微生物调查,发现即使在−15 ℃的永冻土中也能检测到矿化现象,无论是海冰高盐的卤水管还是营养贫瘠的冰尘穴中都存在着活跃的微生物种群[2-4]。而且,由于极地特殊的地质气候和罕至的人为干扰,微生物的多样性在小尺度地域范围内就显示出了明显差异[5-6]。最近的宏观和微观多样性研究表明,极地微生物群落具有丰富的多样性,并不遵循一般生物的纬度多样性模式(多样性向两极减少)[7]。通过对北极和南极地区28个地点不同深度海水样本的宏基因组研究,发现极地海水中19%的操作分类单元(operational taxonomic unit, OTU)在Tara Oceans数据库(全球海洋微生物宏基因组数据库)中无法识别,3 900 000多个蛋白质编码基因找不到同源序列;对214个宏基因组组装基因组(metagenome assembled genomes, MAGs)进行分析发现,极地地区流行的菌株在温带海水中几乎无法检测到[8]。尽管南极和北极的地理位置相距甚远且相互隔离,南极和北极样本中的病毒类群却显示两极仍然存在功能关联性[9]。从南极分离获得的一株新的假单胞菌噬菌体被发现在北极阿拉斯加冻土的宏基因组中有完全相同的基因组序列[10]。北极和南极微生物群的功能有明显差异,北极的抗生素抗性基因丰富,而南极的DNA重组等功能基因丰富[8-11]。这意味着极地无论是陆地还是海洋都具有非常丰富和独特的微生物资源。
极地微生物资源主要包括微生物菌株、代谢产物、酶和基因资源,生物勘探则是获得生物资源的过程。本文将对极地微生物资源的现况进行概述并对生物勘探中的挑战进行探讨。
1 极地微生物的功能产物极地微生物在极端环境中采取了独特的生存策略,在恶劣的条件下生长和繁殖,生物合成了一系列生物分子,可以应用于生物技术、制药、化工、食品加工和生物修复。本章节将对具有生物技术潜力的南极和北极细菌生物制品进行概述。
1.1 低温酶低温酶在低温下具有高催化效率和极大的分子灵活性。灵活性的增加通常是由于氢键和盐桥数量的减少造成的[12-13]。对南极菌株(Oleispira antarctica)冷适应酶晶体结构的研究发现其Glu+Asp/Arg+Lys比值高于中温酶,使蛋白表面疏水性和负电荷较高[14]。此外,蛋白中脯氨酸和精氨酸含量的降低导致了更高的分子熵,增加了活性位点的可及性,为底物和辅助因子的结合提供了更大的灵活性。
研究者们从极地嗜冷细菌中筛选获得了大量低温酶,van den Burg[15]和Bruno等[16]对极地微生物的低温酶做了很好的概述:极地微生物产生的低温酶可以应用于很多领域,如加工食品(半乳糖苷酶、淀粉酶、蛋白酶和脂肪酶)、纺织品(木聚糖酶)、冷洗洗涤剂(纤维素酶、酯酶)、废水处理、水产养殖(植酸酶)、寒冷气候下的生物修复和分子生物学(肽链内切酶、碱性磷酸酶和核酸酶)、化学合成(S-甲酰谷胱甘肽水解酶)、制药(环氧化物水解酶、亮氨酸脱氢酶)以及化妆品(超氧化物歧化酶)等。
低温酶在低温下具有较高的催化效率,因此所需的用量较低,节省能源,减少成本。在食品工业中可以用于不耐热食品的加工,在较低的温度(大约10‒15 ℃)下催化反应,一方面有助于保持食品的风味;另一方面还能够降低工业生产中外源微生物生长的污染。如果在生产中通过改变温度对酶进行灭活,就可以避免添加化学试剂,便于产物的收集与分离,减少了工业成本。此外,新颖的酶基因序列为目前的酶工程改造提供了原材料和新思路,比如将低温酶的中温失活特性用于活疫苗的设计:Duplantis等[17]通过将新凶手弗朗西丝氏菌(Francisella novicida) (引起小鼠土拉菌血症)生长必需酶的编码基因替换为北极细菌的酶基因,完全改变了F. novicida的生存方式;将中温病原体中的关键酶改造成温度敏感的低温酶,这种工程菌株在哺乳动物体温条件下失活,失去致病性,但保留其全部抗原性。将同样的方法应用于肠道沙门菌(Salmonella enterica)、革兰氏阳性分枝杆菌[18]、肠链球菌菌株和结核分枝杆菌(Mycobacterium tuberculosis)并证明是安全的,不会引起人类严重疾病,但仍然能够刺激保护性免疫反应[19]。
1.2 多糖极地微生物的多糖可为细胞提供结构支持,同时也参与多种生化反应,主要包括荚膜多糖、脂多糖和胞外多糖3类[20]。胞外多糖的乳化活性、冷冻保护和重金属结合等功能对细胞具有低温保护作用,而且胞外多糖有利于细胞聚集、形成生物膜、吸收营养物质和微量元素并且耐受干燥,在极地不利环境条件下对微生物的生存至关重要[21]。对南极来源的嗜盐假单胞菌(Pseudoalteromonas haloplanktis) TAC125的脂多糖和胞外多糖成分分析发现,在不同生长温度条件下其化学结构会发生变化,随着温度的升高磷酸化程度增加[22]。然而对极地来源红球菌(Rhodococcus sp.) JG3和极单胞菌(Polaromonas sp.) Eur3 1.2.1的研究中也发现低温能够刺激荚膜的合成[23],一些南极分离获得的细菌新种具有合成荚膜的能力[24-25],但是其功能、适应性以及应用尚未见相关报道。从南极洲分离出的3株革兰氏阴性菌其细胞膜上的脂多糖不饱和程度高且酰基链短,在低温环境下可以更好地维持细胞膜流动性[26]。还有研究发现南极超微细菌(Alteromonas stellipolaris) AT52和AT82脂多糖有望成为梭子蟹的免疫刺激剂抗细菌性疾病[27]。相较于荚膜和脂多糖,极地微生物胞外多糖(黏液多糖)的应用研究更加广泛。从南极洲帝企鹅粪便分离出的一株产胞外多糖的放线菌,胞外多糖可作为生物絮凝剂使用,并在胞外形成生物膜[28]。从北极海湾分离出的菌株(Polaribacter sp.) SM1127,其胞外多糖可以帮助皮肤抵抗UVB辐射和减轻冻伤伤害[29]。从南极一株隐球酵母(Crytococcus heimaeyensis) S20中提取的胞外多糖具有抗多种肿瘤的活性,而且对其他正常细胞的毒害小[30]。多糖在食品、农业、医疗和工业中应用广泛,有着很高的商业价值。极地微生物多糖因其特殊结构从而更抗低温和氧化,还可作为防冻和防腐材料用于建材行业。
1.3 代谢产物目前极地微生物具有应用价值的代谢产物主要包括抗生素、表面活性剂、色素、多肽和聚酮化合物等,其主要可应用于医疗卫生行业,是新型药物的重要来源。dos Santos等[31]报道在2018‒2020年这3年中发现了北极和南极微生物产生的96种新化合物,真菌提供77种化合物,27种来自北极物种(15种新化合物),50种来自南极物种(18种种新化合物);而来自北极的细菌提供了10种化合物(2种新化合物);来自南极的细菌提供了17种化合物(6种新分子);对这些化合物研究得最多的是其细胞毒性和抗菌潜力,另外还有抗寄生虫、抗氧化、抗炎、免疫调节和光反应潜力的研究。这些新的极地微生物化合物(抗生素)结构更新颖、功能更独特且更为稀有,对于产生耐药性的多种病原菌可能具有更好的抑制作用。除了抗生素,研究还发现北极抗氧化细菌(Paracoccus sp.) Arc7-R13产生的纳米银[32]和南极分离出的一株细菌产生的紫色色素也具有抗菌活性[33]。从北极分离获得一株新的肌病毒科噬菌体,其裂解酶基因的重组表达产物对革兰氏阴性菌株表现出广谱的溶菌活性[34]。
寒冷地区的微生物大多会产生色素用于抵抗氧化应激并保持细胞膜的流动性,极地微生物色素种类繁多,除了常见的类胡萝卜素、吲哚类、含氧杂环类和醌类等,还包括生物碱、柔红素等[35]。从南极的某些蓝藻中分离出一种脂溶性的生物碱,呈黄褐色或暗红色,可帮助蓝藻抵抗紫外辐射[36]。这些具有抗氧化活性的色素可以作为光保护剂或者防辐射材料应用于化妆品、食品行业等。
南极和北极都发现一些菌株能够产生生物表面活性剂(biosurfactants, BSs)[37],包括糖蛋白、糖脂、鼠李糖脂、海藻糖脂质和脂肽等。这些表面活性剂可以防止水结晶,并在蛋白质周围形成保护罩以减缓低温对细胞的破坏[38-39]。在低温和不需要加热的情况下能保持功能的表面活性剂不仅低毒、具有高降解活性,而且还节能,有利于降低生产成本。
另外,我们发现从北极花朵中分离出的酵母产生的挥发性有机物(醇类、醛类、酯类和有机酸等)可以促进植物的生长[40],寄生在海鞘内的某种未能培养的微生物能产生帕默内酯,可能有助于治疗人类黑色素瘤[41],还有大量极地微生物产生的萜烯和萜类化合物,具有药用前景[42]。
2 极地微生物资源的保藏鉴于极地微生物资源的重要性,日本、美国、俄罗斯、德国和澳大利亚等国家先后启动了各自的极地微生物研究计划,以抢先揭示极端生命形式的奥秘,研究其特殊生理机制与特殊代谢产物。来自北美、挪威、冰岛、芬兰、瑞典、丹麦和英国的公司也积极开发基于在北极发现的遗传资源的新生物技术,现在有40多家公司参与北极的生物勘探[42]。挪威北极海洋生物勘探项目MabCent就是由科研机构和企业联合承担,4家单位分工协作:海洋生物库Marbank负责收集和保存海洋资源/生物,高通量分析实验室Marbio负责海洋代谢物的分离、鉴定和活性评估,SmallStruct和挪威结构生物学中心NorStruct测定分子的结构,商业合作伙伴Lytix Biopharma AS则进一步开发这些化合物[43]。
2.1 极地微生物保藏概况毫无疑问,在极地微生物勘探中菌种资源平台提供了重要的支持作用。一些极地勘探国建有专门的菌种保藏中心并通过菌种资源的信息化来推进极地微生物的共享利用。例如澳大利亚南极微生物保藏中心(Australian Collection of Antarctic Microorganisms, ACAM)和设在美国俄勒冈大学的极端微生物保藏中心(Culture Collection of Microorganisms from Extreme Environments, CCMEE)[44];意大利的图西亚大学环境科学系建立的“极端环境真菌保藏中心” (The Culture Collection of Fungi from Extreme Environments, CCFEE)收集了从南极岩石、土壤、苔藓、地衣和其他基质中的真菌[45]。
在美国,由政府提供资金支持的非营利性组织保藏着包括极地微生物在内的10万株微生物;欧盟保存着将近10万株微生物;德国微生物和细胞培养物保藏中心(Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH, DSMZ)在1999−2001年参加了由第4个欧洲框架计划资助、针对南极微生物多样性及新化合物研究的Micromat;比利时联合菌种保藏中心(Belgian Coordinated Collections of Microorganisms, BCCM)在2005−2008年参加了极地微生物收集计划;在亚洲,日本通产省宣布从2002年开始,投资60亿日元、由约30位专业研究员负责收集和保存新发现的微生物,其数量到2010年将达10万株;我国对极地微生物资源的调查始于1986‒1987年中国第3次南极考察期间,但一直到1997年,部分研究机构才开始了较为系统的极地微生物资源收集工作[46]。
由于各菌种保藏中心极地菌株数量未公开,以下统计数据均参考Web of Science数据库中的极地模式菌株文献。
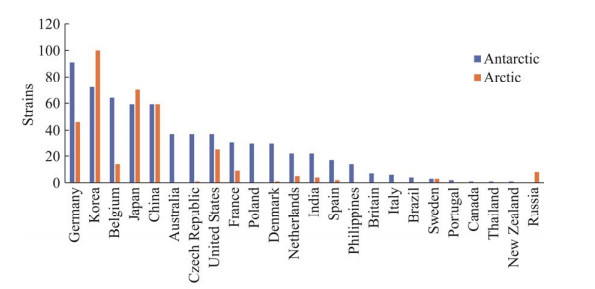
2.2 极地模式菌株种类和数量统计模式菌株(type strain)是在发表一个新种时指定的最能反映这个种特征的菌株,因此,来源于极地的模式菌株即表示一个从极地分离出来的微生物新物种。模式菌株要求通过Web of Science数据库文献统计,截至目前,来源于极地的模式菌株共520株,其中来源于南极的极地模式菌株326株,来源于北极的极地模式菌株194株。按种类统计,来源于南极的极地模式菌株中细菌266株、蓝细菌3株、真菌共57株(包括地衣、酵母和丝状真菌等)。来源于北极的极地模式菌株中细菌158株、真菌共36株(包括地衣、酵母和丝状真菌等)。细菌新种在发表获得国际承认时须将其模式菌株存放于国际承认的微生物保藏机构,因此,查阅文献中其保藏编号即可知道极地分离获得的新种保藏在了哪个国家的哪个机构。如图 1所示,保藏南极模式菌株(新种)数量最多的国家是德国,共91株,主要保藏在德国的微生物和细胞培养物保藏中心(DSMZ)。保藏北极模式菌株(新种)数量最多的国家是韩国,共100株,也是极地新种保藏量整体最多的国家,主要保藏在韩国典型菌种保藏中心(Korean Collection for Type Cultures, KCTC)和韩国农业培养物保藏中心(Korean Agricultural Culture Collection, KACC)两个保藏机构。我国来源于南北极的新种保藏量均为59株,主要保藏在中国典型培养物保藏中心(China Center for Type Culture Collection, CCTCC),也是目前我国极地微生物保藏量最多的机构,约7 000株。
|
| 图 1 南北极模式菌株保藏量 Figure 1 Preservation amount of type strains from Arctic and Antarctic. |
|
|
鉴于极地微生物低温酶、多糖和代谢产物的强大功能和应用前景,其中一些极地来源的乳糖酶或β-半乳糖苷酶获得了专利,并由比利时纽三拉布公司(Bekkevoort)大量生产;美国纽英伦生物技术有限公司(New England Biolabs, Inc.)对从南极嗜冷细菌中分离出一种冷适应的尿苷-DNA糖基化酶和一种碱性磷酸酶进行商业化生产应用于分子生物学技术;日本宝生物公司(TaKaRa-Clontech)也从南极嗜冷细菌Shewanella sp. Ac10中分离出一种冷活性酶[47]。
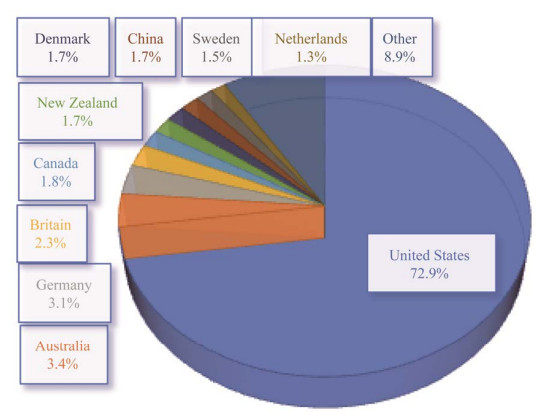
根据Oldham等[48]2013年的数据,有439种南极物种注册了专利。本文通过SOOPAT网站(http://www.soopat.com/)对1988‒2022年间在中国和世界申请的南北极微生物相关专利进行搜索,搜索到中国专利373项,世界专利1 083项。在世界专利中以美国789项占绝对优势(73%),而中国申请的极地微生物相关专利为18项,占1.7%排在第八(图 2)。申请世界专利最多的机构是美国的生物技术公司Diversa Corporation (酶制剂、微生物酶的检测和高通量筛选技术),其他前五的是美国马泰克生物科学有限公司Martek Biosciences Corporation、美国麻省理工学院Massachusetts Institute of Technology (微生物的酶和代谢产物)、The Regents of The University of California (酶制剂)、美国哈佛-麻省理工的博德研究所Broad Institute, INC. (微生物酶及代谢产物基因组医学)。我国极地微生物专利申请前五的单位分别是中国海洋大学、国家海洋局第一海洋研究所、中国水产科学研究院黄海水产研究所、浙江海洋大学和广东越群海洋生物研究开发有限公司,以科研院所为主。国内外极地微生物专利大部分都与微生物的酶和代谢产物有关,但是美国更多地偏重化工方向,主要是用于医药和基因工程的酶制剂,而我国的专利更偏重农业,在农作物、食品、兽医和水产保护防治方面的专利较多。我国极地微生物专利数量逐年增加,在2017年之后趋于平稳。但是世界极地微生物专利数量在2014年达到高峰后逐渐明显地下降,可能是到了一定的瓶颈期,需要新技术和思路的突破。
|
| 图 2 南北极微生物各国世界专利申请比例图 Figure 2 Proportion of world patent applications for polar microorganisms. |
|
|
极地微生物被证实具有很强的次级代谢产物的合成潜力,且很多都是暂时未被发现的新物质,在药物开发、食品和卫生医疗等方面都有很大的应用前景。但极地微生物存在未培养的难题,而且低温生长缓慢,极地一些区域难以到达和回溯,生物类样品(微生物的宿主)难以保存、运输等导致极地微生物资源的研究充满挑战。高通量测序已经广泛应用于获得特定环境中不可培养微生物的信息,可以从核糖体RNA基因和序列、宏基因组和宏转录组数据中详细分析各种生态系统中微生物的群落结构、多样性和功能等信息(包括极地)[49]。这无疑加速了新型生物活性分子的发现,为解决微生物分离或培养的困境带来了新的技术手段,但是基因功能预测的准确性、表达的成功率等仍然存在诸多挑战。
4.1 可培养性的挑战目前研究认为一个环境样本中只能培养出约10%‒20%的原核生物物种[50-51]。通过对波兰北极Hornsund站区(挪威Spitsbergen)附近的土壤进行测试,发现与高通量测序结果相比,只能培养获得6.37%的细菌属和20%的真菌属;另外,也有68个培养获得的属不在高通量测序结果中;这可能源于PCR扩增的偏好性以及V3‒V4 16S rRNA基因测序的局限性[52]。
通过高通量测序获得的极地土壤微生物多样性、宏基因组组装的基因组和功能注释信息,可以设计培养条件获得目的培养物。Dziurzynski等[52]还建立了一种与众不同的方法,将北极土壤样品进行多种培养基培养后,将平板上的菌苔冲洗下来进行高通量测序,获得可培养微生物的多样性信息;根据这些信息在数据库中找到对应物种的基因组序列,然后评估这些序列中耐受重金属和能够降解石油烃的基因以及可能的培养条件,设计培养基获得了具有这些功能的耐冷菌株。
尽管测序技术取得了不可否认的进展,但分子方法还不足以进行适用于生物勘探领域的详尽分析。新菌株的收集仍然是最重要的基础,一方面是为了扩充基因组序列和信息的辨识;另一方面是为了提高对冷适应性微生物表型和生理学的研究。Zarins-Tutt等[53]尝试通过共培养试验激活抗生素产生菌株在纯培养物中不表达的沉默小分子生物合成基因簇(biosynthetic gene clusters, BGC)。Marcolefas等[54]设计并在北极安装了cryo-iPlate原位培养装置,图 3装置尺寸为15 cm×12 cm,有160个0.5 cm宽和深的孔(之前的原型就是移液吸头盒子的隔板);在部署到现场之前,中央板的孔中充满了2% (质量体积分数)结冷胶,在现场将10 μL含有约1‒10个微生物细胞的土壤稀释液加入孔中,在顶层和底层粘贴上无菌半透性0.03 μm孔径聚碳酸酯膜;将装置固定在土壤中10 d后,在实验室5 ℃孵育3个月再取出结冷胶进行分离培养。

|
| 图 3 原位培养cryo-iPlate装置 Figure 3 In situ cryo-iPlate cultivation. |
|
|
另外,Bode、Kristoffersen等[55-57]通过改变培养条件或添加特殊底物(如鼠李糖脂前体、葵花籽油和硝酸甘蔗汁)激活极地菌株不同的代谢途径,得到多种次级代谢物或提高其产量。又或者设置比较严苛的培养条件,比如低于最佳培养温度进行培养、添加重金属、增加压力和盐度等刺激拮抗活性物质的产生或胞外多糖(extracellular polysaccharide substances, EPS)的产量[58]。
4.2 宏基因组方法的挑战通过文献检索,在2004‒2022年间300余篇文献中利用宏基因组对南北极样品进行了研究。其中组装了1 706个基因组(metagenome-assembled genomes, MAGs) (基本上是2021、2022年发表的论文为16、20篇,其他一篇2019,3篇2020年)。通过对(宏)基因组DNA进行序列分析,然后预测酶的功能,从而提高了低温酶的发现率。基于序列的宏基因组方法是一种强大的工具,通过减少要进行功能测试的靶点数量,避免了与经典筛选相关的密集、昂贵和耗时的实验室工作。
宏基因组方法仅局限于发现与已知序列具有高度相似性的基因,这使得发现新的酶功能变得困难[59],而且预测的功能也未必准确。近年来,通过对宏基因组或宏转录组中的基因序列进行批量体外重组表达,然后筛选克隆子的代谢产物以获得新结构的化学分子或酶。但是在中温生长的宿主如大肠杆菌(Escherichia coli)中,低温酶通常形成无活性的聚集体。因此有必要开发新的低温表达系统,包括鉴定新的宿主生物和相关的遗传操作工具。虽然盐浮游假交替单胞菌(Pseudoalteromonas haloplanotis)、希瓦氏菌(Shewanella)、节杆菌(Arthrobacter)和红红球菌(Rhodococcus rhodopolis)[60]都被认为是此类应用的候选菌株,但目前尚未作为有效表达系统进行广泛应用。Söderberg等构建了以北极细菌(Aliivibrio wodanis)为宿主的低温酶异源表达体系,能够成功获得之前不能在大肠杆菌中表达的低温酶[61]。另一种策略则是在大肠杆菌中共表达来自南极细菌(Oleispira antarctica)的伴侣蛋白Cpn60和Cpn10,该系统在非常低的温度下(4‒12 ℃)可以高效表达外源基因,提高蛋白的活性和可溶性重组蛋白的产量,这个被称为“Arctic Express”的工程菌已经商业化并得到广泛应用[62]。
随着宏基因组数据的日渐积累和完善,极地的原位生物多样性将有可能逐步转化为数据,极地微生物资源的研究和应用对材料样本的依赖程度降低,这将对生物勘探的管理和惠益分享提出挑战。
4.3 大数据的挑战随着基因测序、数据存储和挖掘软件以及基因编辑和组装技术的快速发展,生物勘探发生了巨大变化,作为研究对象的生物体和表型越来越多地让位于遗传、基因组和宏基因组信息,研究方法也从显微镜、化学分析和多相分类试验转向关注基因序列结构和生物信息学分析[63]。这意味着在生物多样性研究中需要重新考虑观察方式、分类方式[64],甚至收集生物多样性的方式[65]。因为小样本(比如一滴海水)中包含的所有DNA信息都可以一次提取出来,这不仅减少了对大样本的依赖,而且还减少了保存样本的需要,一旦序列信息被提取并存储在数据库中,就无须返回原始样本提取站点获取新样本。
关于生物资源数字化后未来在实验室组装的生命形式是否是其原始状态,脱离环境的生物资源能否被称为生物多样性等问题必定存在争论。但其发展趋势必定是生物勘探实现完全独立于原始生物体,即无须获得极地样本或者微生物即可通过数据进行研究和应用,从而开启生物勘探的新机遇。当然,其前提是数据流的开放和共享协作。这就涉及惠益分享制度的建立和数据的监管。其核心问题在于数字序列信息(digital sequence information, DSI)是否算作“遗传材料”被纳入“名古屋议定书” (保障资源主权国分享资源开放的利益)的监管。通常生物技术产业发达的国家倾向于反对将DSI视为遗传材料,从而脱离生物多样性公约(Convention on Biological Diversity, CBD)和名古屋议定书监管;而资源输出国则认为,遗传材料与其数字对应物必须等同权益[66]。
对于南极大陆资源相关管理尤其混乱,一方面由于主权不明而不在“名古屋议定书”监管范围,而且《南极条约》规定:“来自南极的科学观测和结果应交换和免费提供”,另一方面极地事务参与国根据《与贸易有关的知识产权协定》(Agreement on Trade-Related Aspects of Intellectual Property Rights, TRIPS)通过各国的专利制度对资源进行法律保护[67]。那么,数字序列信息(DSI)是否纳入国际可承认的专利保护体系对于极地的生物勘探非常关键。另外一个问题是序列的可溯源性。由于微生物学中“万物无处不在” (所有微生物无处不在)的理论,导致“相同序列可以在不同地点找到”,从而很难“证明”序列的来源。而数据库在数据获得、溯源以及惠益分享方面的作用越来越重要,对于极地这样主权不明的区域统一管理各国生物勘探的生物样品或生物资源不现实,但是资源数据化的趋势对于极地微生物资源的统一监管却是一个机遇。
| [1] |
TIMLIN U, MEYER A, NORDSTRÖM T, RAUTIO A. Permafrost thaw challenges and life in Svalbard[J]. Current Research in Environmental Sustainability, 2022, 4: 100122. DOI:10.1016/j.crsust.2021.100122 |
| [2] |
de JONG AEE, IN'T ZANDT MH, MEISEL OH, JETTEN MSM, DEAN JF, RASIGRAF O, WELTE CU. Increases in temperature and nutrient availability positively affect methane-cycling microorganisms in Arctic thermokarst lake sediments[J]. Environmental Microbiology, 2018, 20(12): 4314-4327. DOI:10.1111/1462-2920.14345 |
| [3] |
HAYWARD MK, DEWEY ED, SHAFFER KN, HUNTINGTON AM, BURCHELL BM, STOKES LM, ALEXANDER BC, GEORGE JE, KEMPHER ML, JOYE SB, MADIGAN MT, SATTLEY WM. Cultivation and characterization of snowbound microorganisms from the South Pole[J]. Extremophiles, 2021, 25(2): 159-172. DOI:10.1007/s00792-021-01218-z |
| [4] |
CAVICCHIOLI R, SIDDIQUI KS, ANDREWS D, SOWERS KR. Low-temperature extremophiles and their applications[J]. Current Opinion in Biotechnology, 2002, 13(3): 253-261. DOI:10.1016/S0958-1669(02)00317-8 |
| [5] |
ZHANG YM, LU L, CHANG XL, JIANG F, GAO XD, YAO YF, LI CS, CAO SN, ZHOU QM, PENG F. Small-scale soil microbial community heterogeneity linked to landform historical events on King George Island, maritime Antarctica[J]. Frontiers in Microbiology, 2018, 9: 3065. DOI:10.3389/fmicb.2018.03065 |
| [6] |
NOPNAKORN P, ZHANG YM, YANG L, PENG F. Antarctic Ardley Island Terrace—an ideal place to study the marine to terrestrial succession of microbial communities[J]. Frontiers in Microbiology, 2023, 14: 942428. DOI:10.3389/fmicb.2023.942428 |
| [7] |
GREGORY AC, ZAYED AA, CONCEIÇÃO-NETO N, TEMPERTON B, BOLDUC B, ALBERTI A, ARDYNA M, ARKHIPOVA K, CARMICHAEL M, CRUAUD C, DIMIER C, DOMÍNGUEZ-HUERTA G, FERLAND J, KANDELS S, LIU YX, MAREC C, PESANT S, PICHERAL M, PISAREV S, POULAIN J, et al. Marine DNA viral macro- and microdiversity from pole to pole[J]. Cell, 2019, 177(5): 1109-1123.e14. DOI:10.1016/j.cell.2019.03.040 |
| [8] |
CAO SN, ZHANG WP, DING W, WANG M, FAN S, YANG B, MCMINN A, WANG M, XIE BB, QIN QL, CHEN XL, HE JF, ZHANG YZ. Structure and function of the Arctic and Antarctic marine microbiota as revealed by metagenomics[J]. Microbiome, 2020, 8(1): 47. DOI:10.1186/s40168-020-00826-9 |
| [9] |
WANG J, XIAO J, ZHU Z, WANG SY, ZHANG L, FAN ZJ, DENG YL, HU ZH, PENG F, SHEN S, DENG F. Diverse viromes in polar regions: a retrospective study of metagenomic data from Antarctic animal feces and Arctic frozen soil in 2012‒2014[J]. Virologica Sinica, 2022, 37(6): 883-893. DOI:10.1016/j.virs.2022.08.006 |
| [10] |
LIU ZY, JIANG WH, KIM C, PENG XY, FAN C, WU YL, XIE ZX, PENG F. A Pseudomonas lysogenic bacteriophage crossing the antarctic and arctic, representing a new genus of Autographiviridae[J]. International Journal of Molecular Sciences, 2023, 24(8): 7662. DOI:10.3390/ijms24087662 |
| [11] |
AMBROSINI R, MUSITELLI F, NAVARRA F, TAGLIAFERRI I, GANDOLFI I, BESTETTI G, MAYER C, MINORA U, AZZONI RS, DIOLAIUTI G, SMIRAGLIA C, FRANZETTI A. Diversity and assembling processes of bacterial communities in cryoconite holes of a Karakoram glacier[J]. Microbial Ecology, 2017, 73(4): 827-837. DOI:10.1007/s00248-016-0914-6 |
| [12] |
FELLER G, GERDAY C. Psychrophilic enzymes: hot topics in cold adaptation[J]. Nature Reviews Microbiology, 2003, 1(3): 200-208. DOI:10.1038/nrmicro773 |
| [13] |
GIORDANO D, COPPOLA D, RUSSO R, TINAJERO-TREJO M, di PRISCO G, LAURO F, ASCENZI P, VERDE C. The globins of cold-adapted Pseudoalteromonas haloplanktis TAC125: from the structure to the physiological functions[M]. Advances in Microbial Physiology. Amsterdam: Elsevier, 2013: 329-389.
|
| [14] |
KUBE M, CHERNIKOVA TN, AL-RAMAHI Y, BELOQUI A, LOPEZ-CORTEZ N, GUAZZARONI ME, HEIPIEPER HJ, KLAGES S, KOTSYURBENKO OR, LANGER I, NECHITAYLO TY, LÜNSDORF H, FERNÁNDEZ M, JUÁREZ S, CIORDIA S, SINGER A, KAGAN O, EGOROVA O, PETIT PA, STOGIOS P, et al. Genome sequence and functional genomic analysis of the oil-degrading bacterium Oleispira antarctica[J]. Nature Communications, 2013, 4: 2156. DOI:10.1038/ncomms3156 |
| [15] |
van den BURG B. Extremophiles as a source for novel enzymes[J]. Current Opinion in Microbiology, 2003, 6(3): 213-218. DOI:10.1016/S1369-5274(03)00060-2 |
| [16] |
BRUNO S, COPPOLA D, Di PRISCO G, GIORDANO D, VERDE C. Enzymes from marine polar regions and their biotechnological applications[J]. Marine Drugs, 2019, 17(10): 544. DOI:10.3390/md17100544 |
| [17] |
DUPLANTIS BN, OSUSKY M, SCHMERK CL, ROSS DR, BOSIO CM, NANO FE. Essential genes from Arctic bacteria used to construct stable, temperature-sensitive bacterial vaccines[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(30): 13456-13460. |
| [18] |
PINTO CT, NANO FE. Stable, temperature-sensitive recombinant strain of Mycobacterium smegmatis generated through the substitution of a psychrophilic ligA gene[J]. FEMS Microbiology Letters, 2015, 362(18): fnv152. DOI:10.1093/femsle/fnv152 |
| [19] |
DUPLANTIS BN, PUCKETT SM, ROSEY EL, AMEISS KA, HARTMAN AD, PEARCE SC, NANO FE. Temperature-sensitive Salmonella enterica serovar enteritidis PT13a expressing essential proteins of psychrophilic bacteria[J]. Applied and Environmental Microbiology, 2015, 81(19): 6757-6766. DOI:10.1128/AEM.01953-15 |
| [20] |
SUN ZM, LIU T, GUO N, GUO ZW. Biological characteristics of polar microbial polysaccharides and corresponding mechanism of metal corrosion[J]. Surface Technology, 2022, 51(9): 65-73. (in Chinese) 孙振美, 刘涛, 郭娜, 郭章伟. 极地微生物多糖的生物学特征及对金属的腐蚀影响机制[J]. 表面技术, 2022, 51(9): 65-73. DOI:10.16490/j.cnki.issn.1001-3660.2022.09.006 |
| [21] |
CARUSO C, RIZZO C, MANGANO S, POLI A, Di DONATO P, NICOLAUS B, Di MARCO G, MICHAUD L, GIUDICE AL. Extracellular polymeric substances with metal adsorption capacity produced by Pseudoalteromonas sp. MER144 from Antarctic seawater[J]. Environmental Science and Pollution Research, 2018, 25(5): 4667-4677. DOI:10.1007/s11356-017-0851-z |
| [22] |
CORSARO MM, LANZETTA R, PARRILLI E, PARRILLI M, TUTINO ML, UMMARINO S. Influence of growth temperature on lipid and phosphate contents of surface polysaccharides from the antarctic bacterium Pseudoalteromonas haloplanktis TAC 125[J]. Journal of Bacteriology, 2004, 186(1): 29-34. DOI:10.1128/JB.186.1.29-34.2004 |
| [23] |
RAYMOND-BOUCHARD I, TREMBLAY J, ALTSHULER I, GREER CW, WHYTE LG. Comparative transcriptomics of cold growth and adaptive features of a eury- and steno-psychrophile[J]. Frontiers in Microbiology, 2018, 9: 1565. DOI:10.3389/fmicb.2018.01565 |
| [24] |
KIM CS, LIU ZY, PENG XY, QIN K, HUANG J, NIU JJ, LIU YX, LIU J, SUN MJ, PENG F. Paraconexibacter antarcticus sp. nov., a novel actinobacterium isolated from antarctic tundra soil[J]. International Journal of Systematic and Evolutionary Microbiology, 2022, 72(12): 5647. |
| [25] |
ZHANG YM, JIANG F, CHANG XL, QIU X, REN LZ, QU ZH, DENG SS, DA XY, FANG CX, PENG F. Flavobacterium collinsense sp. nov., isolated from a till sample of an Antarctic glacier[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(1): 172-177. DOI:10.1099/ijsem.0.000688 |
| [26] |
DI LORENZO F, CRISAFI F, La CONO V, YAKIMOV MM, MOLINARO A, SILIPO A. The structure of the lipid A of gram-negative cold-adapted bacteria isolated from Antarctic environments[J]. Marine Drugs, 2020, 18(12): 592. DOI:10.3390/md18120592 |
| [27] |
YANG JF, GUO LY, CHEN FS, WANG HL. The effects of lipopolysaccharides from two strains of oligotrophic bacteria from Antarctic Ocean Alteromonas stellipolaris on non-specific immunity of the swimming crab Portunus trituberculatus[J]. Oceanologia et Limnologia Sinica, 2011, 42(2): 294-299. (in Chinese) 杨季芳, 郭卢云, 陈福生, 王海丽. 2株南极海洋寡营养细菌(Alteromonas stellipolaris)脂多糖对三疣梭子蟹(Portunus trituberculatus)非特异性免疫活性的影响[J]. 海洋与湖沼, 2011, 42(2): 294-299. |
| [28] |
GAO HM, XIE PF, ZHANG XL, YANG Q. Isolation, phylogenetic and gephyromycin metabolites characterization of new exopolysaccharides-bearing antarctic actinobacterium from feces of emperor penguin[J]. Marine Drugs, 2021, 19(8): 458. DOI:10.3390/md19080458 |
| [29] |
SUN ML. Discovery of a novel marine bacteriophage MfV, and potential evaluation of the exopolysaccharide from marine bacterium Polaribacter sp. SM1127[D]. Jinan: Doctoral Dissertation of Shandong University, 2020 (in Chinese). 孙美玲. 海洋新型噬菌体MfV的发现及海洋细菌Polaribacter sp. SM1127胞外多糖的应用潜力评价[D]. 济南: 山东大学博士学位论文, 2020. |
| [30] |
PENG F, SHEN C, ZHEN CY, HUANG Y, HAO Y, LIN DQ. Application of Cryptococcosis heimaeyensis and its exopolysaccharide: China, CN201910211986. X[P]. 2020-10-13 (in Chinese). 彭方, 沈超, 郑从义, 黄瑶, 郝瑶, 林丹青. 一株隐球酵母及其胞外多糖与应用: CN201910211986. X[P]. 2020-10-13. |
| [31] |
dos SANTOS GS, TEIXEIRA TR, COLEPICOLO P, DEBONSI HM. Natural products from the poles: structural diversity and biological activities[J]. Revista Brasileira de Farmacognosia, 2021, 31(5): 531-560. DOI:10.1007/s43450-021-00203-z |
| [32] |
ZHANG Z, LI S, GU XQ, LI J, LIN XZ. Biosynthesis, characterization and antibacterial activity of silver nanoparticles by the Arctic anti-oxidative bacterium Paracoccus sp. Arc7-R13[J]. Artificial Cells, Nanomedicine, and Biotechnology, 2019, 47(1): 1488-1495. DOI:10.1080/21691401.2019.1601631 |
| [33] |
ATALAH J, BLAMEY L, MUÑOZ-IBACACHE S, GUTIERREZ F, URZUA M, ENCINAS MV, PÁEZ M, SUN JS, BLAMEY JM. Isolation and characterization of violacein from an antarctic Iodobacter: a non-pathogenic psychrotolerant microorganism[J]. Extremophiles, 2020, 24(1): 43-52. DOI:10.1007/s00792-019-01111-w |
| [34] |
DENG SS, XU Q, FU YJ, LIANG LQ, WU Y, PENG F, GAO MY. Genomic analysis of a novel phage infecting the Turkey pathogen Escherichia coli APEC O78 and its endolysin activity[J]. Viruses, 2021, 13(6): 1034. DOI:10.3390/v13061034 |
| [35] |
SILVA TRE, SILVA LCF, de QUEIROZ AC, ALEXANDRE MOREIRA MS, de CARVALHO FRAGA CA, de MENEZES GCA, ROSA LH, BICAS J, de OLIVEIRA VM, DUARTE AWF. Pigments from Antarctic bacteria and their biotechnological applications[J]. Critical Reviews in Biotechnology, 2021, 41(6): 809-826. DOI:10.1080/07388551.2021.1888068 |
| [36] |
DEMAY J, BERNARD C, REINHARDT A, MARIE B. Natural products from cyanobacteria: focus on beneficial activities[J]. Marine Drugs, 2019, 17(6): 320. DOI:10.3390/md17060320 |
| [37] |
VOLLÚ RE, JURELEVICIUS D, RAMOS LR, PEIXOTO RS, ROSADO AS, SELDIN L. Aerobic endospore-forming bacteria isolated from Antarctic soils as producers of bioactive compounds of industrial interest[J]. Polar Biology, 2014, 37(8): 1121-1131. DOI:10.1007/s00300-014-1505-y |
| [38] |
JADHAV VV, YADAV A, SHOUCHE YS, APHALE S, MOGHE A, PILLAI S, ARORA A, BHADEKAR RK. Studies on biosurfactant from Oceanobacillus sp. BRI 10 isolated from Antarctic Sea water[J]. Desalination, 2013, 318: 64-71. DOI:10.1016/j.desal.2013.03.017 |
| [39] |
CORRADINI D, STREKALOVA EG, STANLEY HE, GALLO P. Microscopic mechanism of protein cryopreservation in an aqueous solution with trehalose[J]. Scientific Reports, 2013, 3: 1218. DOI:10.1038/srep01218 |
| [40] |
NIU JJ, LI XH, ZHANG SY, YAO YF, ZHANG YP, LIU YX, PENG XY, HUANG J, PENG F. Identification and functional studies of microbial volatile organic compounds produced by Arctic flower yeasts[J]. Frontiers in Plant Science, 2023, 13: 941929. DOI:10.3389/fpls.2022.941929 |
| [41] |
DIYABALANAGE T, AMSLER CD, MCCLINTOCK JB, BAKER BJ. Palmerolide A, a cytotoxic macrolide from the Antarctic tunicate Synoicum adareanum[J]. Journal of the American Chemical Society, 2006, 128(17): 5630-5631. DOI:10.1021/ja0588508 |
| [42] |
NÚÑEZ-PONS L, SHILLING A, VERDE C, BAKER BJ, GIORDANO D. Marine terpenoids from polar latitudes and their potential applications in biotechnology[J]. Marine Drugs, 2020, 18(8): 401. DOI:10.3390/md18080401 |
| [43] |
SVENSON J. MabCent: arctic marine bioprospecting in Norway[J]. Phytochemistry Reviews, 2013, 12(3): 567-578. DOI:10.1007/s11101-012-9239-3 |
| [44] |
LING XL, YU Y, ZHU JG, CHEN B, XIA MY, CHENG WF. Design and function on database of microorganisms from polar environments[J]. Chinese Journal of Polar Research, 2009, 21(1): 69-75. (in Chinese) 凌晓良, 俞勇, 朱建钢, 陈波, 夏明一, 程文芳. 极地微生物菌种数据库的设计及其功能简述[J]. 极地研究, 2009, 21(1): 69-75. |
| [45] |
ONOFRI S, FENICE M, CICALINI AR, TOSI S, MAGRINO A, PAGANO S, SELBMANN L, ZUCCONI L, VISHNIAC HS, OCAMPO-FRIEDMANN R, FRIEDMANN EI. Ecology and biology of microfungi from antarctic rocks and soils[J]. Italian Journal of Zoology, 2000, 67(sup1): 163-167. DOI:10.1080/11250000009356372 |
| [46] |
ZENG YX, CHEN B, ZOU Y, ZHENG TL. Polar microorganisms, a potential source for new natural medicines—a review[J]. Acta Microbiologica Sinica, 2008, 48(5): 695-700. (in Chinese) 曾胤新, 陈波, 邹扬, 郑天凌. 极地微生物—新天然药物的潜在来源[J]. 微生物学报, 2008, 48(5): 695-700. |
| [47] |
DANIELA G. Bioactive molecules from extreme environments[J]. Marine Drugs, 2020, 18: 640. DOI:10.3390/md18120640 |
| [48] |
OLDHAM P, HALL S, FORERO O. Biological diversity in the patent system[J]. PLoS One, 2013, 8(11): e78737. DOI:10.1371/journal.pone.0078737 |
| [49] |
HORGAN RP, KENNY LC. 'Omic' technologies: genomics, transcriptomics, proteomics and metabolomics[J]. The Obstetrician & Gynaecologist, 2011, 13(3): 189-195. |
| [50] |
LLOYD KG, STEEN AD, LADAU J, YIN JQ, CROSBY L. Phylogenetically novel uncultured microbial cells dominate earth microbiomes[J]. mSystems, 2018, 3(5): e00055-18. |
| [51] |
STEEN AD, CRITS-CHRISTOPH A, CARINI P, DEANGELIS KM, FIERER N, LLOYD KG, CAMERON THRASH J. High proportions of bacteria and archaea across most biomes remain uncultured[J]. The ISME Journal, 2019, 13(12): 3126-3130. DOI:10.1038/s41396-019-0484-y |
| [52] |
DZIURZYNSKI M, GORECKI A, PAWLOWSKA J, ISTEL L, DECEWICZ P, GOLEC P, STYCZYNSKI M, POSZYTEK K, ROKOWSKA A, GORNIAK D, DZIEWIT L. Revealing the diversity of bacteria and fungi in the active layer of permafrost at Spitsbergen Island (arctic)-combining classical microbiology and metabarcoding for ecological and bioprospecting exploration[J]. Science of the Total Environment, 2023, 856: 159072. DOI:10.1016/j.scitotenv.2022.159072 |
| [53] |
ZARINS-TUTT JS, BARBERI TT, GAO H, MEARNS-SPRAGG A, ZHANG LX, NEWMAN DJ, GOSS RJM. Prospecting for new bacterial metabolites: a glossary of approaches for inducing, activating and upregulating the biosynthesis of bacterial cryptic or silent natural products[J]. Natural Product Reports, 2016, 33(1): 54-72. DOI:10.1039/C5NP00111K |
| [54] |
MARCOLEFAS E, LEUNG T, OKSHEVSKY M, McKAY G, HIGNETT E, HAMEL J, AGUIRRE G, BLENNER-HASSETT O, BOYLE B, LÉVESQUE RC, NGUYEN D, GRUENHEID S, WHYTE L. Culture-dependent bioprospecting of bacterial isolates from the Canadian high Arctic displaying antibacterial activity[J]. Frontiers in Microbiology, 2019, 10: 1836. DOI:10.3389/fmicb.2019.01836 |
| [55] |
BODE HB, BETHE B, HÖFS R, ZEECK A. Big effects from small changes: possible ways to explore nature's chemical diversity[J]. Chembiochem, 2002, 3(7): 619-627. DOI:10.1002/1439-7633(20020703)3:7<619::AID-CBIC619>3.0.CO;2-9 |
| [56] |
KRISTOFFERSEN V, RÄMÄ T, ISAKSSON J, ANDERSEN JH, GERWICK WH, HANSEN E. Characterization of rhamnolipids produced by an Arctic marine bacterium from the Pseudomonas fluorescence group[J]. Marine Drugs, 2018, 16(5): 163. DOI:10.3390/md16050163 |
| [57] |
TISO T, SABELHAUS P, BEHRENS B, WITTGENS A, ROSENAU F, HAYEN H, BLANK LM. Creating metabolic demand as an engineering strategy in Pseudomonas putida-Rhamnolipid synthesis as an example[J]. Metabolic Engineering Communications, 2016, 3: 234-244. DOI:10.1016/j.meteno.2016.08.002 |
| [58] |
MARX JG, CARPENTER SD, DEMING JW. Production of cryoprotectant extracellular polysaccharide substances (EPS) by the marine psychrophilic bacterium Colwellia psychrerythraea strain 34H under extreme conditions[J]. Canadian Journal of Microbiology, 2009, 55(1): 63-72. DOI:10.1139/W08-130 |
| [59] |
BEHRENS GA, HUMMEL A, PADHI SK, SCHÄTZLE S, BORNSCHEUER UT. Discovery and protein engineering of biocatalysts for organic synthesis[J]. Advanced Synthesis & Catalysis, 2011, 353(13): 2191-2215. |
| [60] |
MITEVA V, LANTZ S, BRENCHLEY J. Characterization of a cryptic plasmid from a Greenland ice core Arthrobacter isolate and construction of a shuttle vector that replicates in psychrophilic high G+C Gram-positive recipients[J]. Extremophiles, 2008, 12(3): 441-449. DOI:10.1007/s00792-008-0149-7 |
| [61] |
SÖDERBERG JJ, GRGIC M, HJERDE E, HAUGEN P. Aliivibrio wodanis as a production host: development of genetic tools for expression of cold-active enzymes[J]. Microbial Cell Factories, 2019, 18(1): 197. DOI:10.1186/s12934-019-1247-1 |
| [62] |
FERRER M, CHERNIKOVA TN, YAKIMOV MM, GOLYSHIN PN, TIMMIS KN. Chaperonins govern growth of Escherichia coli at low temperatures[J]. Nature Biotechnology, 2003, 21(11): 1266-1267. |
| [63] |
DELGADO A. Microbial extractions: sequence-based bioprospecting, augmented promises, and elusive politics[J]. Science, Technology, & Human Values, 2021: 01622439211055693.
|
| [64] |
BOWKER GC. Book review: barcoding nature: shifting cultures of taxonomy in an age of biodiversity loss[J]. Science, Technology, & Human Values, 2014, 39(5): 759-761. |
| [65] |
BOWKER GC. Mapping biodiversity[J]. International Journal of Geographical Information Science, 2000, 14(8): 739-754. DOI:10.1080/136588100750022769 |
| [66] |
BOND MR, SCOTT D. Digital biopiracy and the (dis)assembling of the Nagoya protocol[J]. Geoforum, 2020, 117: 24-32. DOI:10.1016/j.geoforum.2020.09.001 |
| [67] |
BOWKER GC. Mapping biodiversity[J]. Patent law and bioprospecting in Antarctica[J]. Polar Record, 2011, 47(240): 46-55.
|
 2023, Vol. 50
2023, Vol. 50