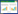扩展功能
文章信息
- 桂仁跃, 洪宇, 余晓斌, 罗玮
- GUI Renyue, HONG Yu, YU Xiaobin, LUO Wei
- 三孢布拉霉crgA启动子的克隆和活性分析
- Cloning and activity of the crgA promoter from Blakeslea trispora
- 微生物学通报, 2023, 50(12): 5275-5285
- Microbiology China, 2023, 50(12): 5275-5285
- DOI: 10.13344/j.microbiol.china.230388
-
文章历史
- 收稿日期: 2023-05-11
- 接受日期: 2023-06-10
- 网络首发日期: 2023-07-25
三孢布拉霉(Blakeslea trispora, Bt)是目前工业化生产β-胡萝卜素的主要微生物之一。三孢布拉霉不仅生长速度快,β-胡萝卜素的产量也很高,其合成β-胡萝卜素受到负调控因子CrgA的调控[1]。CrgA被发现于卷枝毛霉中,作为关键负调控因子发挥作用[2-4],同时在藤仓镰孢、尖孢镰刀菌[5]、三孢布拉霉和布拉克须霉[6]等丝状真菌中也都存在crgA直系同源基因。crgA缺陷型的卷枝毛霉在整合三孢布拉霉来源的crgA后,表现出和野生型卷枝毛霉类似的表型[7]。在三孢布拉霉crgA缺陷菌株中,参与β-胡萝卜素合成途径上的相关结构基因转录水平明显增加,β-胡萝卜素的合成量较野生型也提高了31.2%,表明其对β-胡萝卜素的合成起到负调控作用;同时,在光照条件下,crgA的转录也体现出“光适应”和“光激活”现象[8-9],但其表达水平与β-胡萝卜素合成水平之间的相互关系尚未得到充分解析。为研究CrgA在三孢布拉霉体内受到光刺激时的表达状态,本研究以crgA启动子结构和活性研究为切入点,为CrgA调控β-胡萝卜素合成提供一些理论依据。
启动子一般是位于结构基因5′端上游的一段DNA序列,RNA聚合酶可通过序列的特异性来识别和结合启动序列,可调控下游基因的表达,通过调控转录水平进而影响下游基因的表达部位、水平以及方式[10]。在反式作用调控的研究中,对启动子功能区进行缺失突变分析是常用的方法,为之后研究相关转录因子的DNA和蛋白质相互作用试验奠定基础[11]。
目前对三孢布拉霉CrgA调控β-胡萝卜素合成过程及机制解析的研究非常有限,本研究旨在分析该基因启动子中光响应元件功能及其调控crgA的表达模式。首先以btcrgA上游2 000 bp序列为研究对象[12],克隆并对其序列中的顺式作用元件进行预测,逐渐截短其5′端序列构建突变载体,通过测定β-d-葡萄糖苷酸酶(β-d-glucuronidase, GUS)的活性,确定各个截短启动子驱动外源表达基因的能力,并设置不同光照模式探究btcrgA的转录是否受到光照调控及其调控模式,以及btcrgA启动子受光调控顺式作用元件所在的区域,了解btcrgA上游启动子的核心序列位置[13-15]。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒三孢布拉霉(Blakeslea trispora) NRRL2896和大肠杆菌(Escherichia coli) DH5α由本实验室保藏;农杆菌LBA4404,酷来博科技有限公司;pCAMBIA1303由本实验室保藏。
1.1.2 主要试剂和仪器RNA pure Plant Kit和5′-RACE试剂盒,生工生物工程(上海)股份有限公司;蛋白浓度测定试剂盒,上海碧云天生物技术有限公司;GUS报告基因定量检测试剂盒,酷来博科技有限公司;Clone® Plus Multi One Step Cloning Kit,Hifair® Ⅲ 1st Strand cDNA Synthesis SuperMix for qPCR (gDNA digester plus)和Heffi® qPCR SYBR Green Master Mix,翊圣生物科技公司;蜗牛酶、纤维素酶、溶壁酶和溶菌酶,索莱宝生物科技有限公司;引物合成与测序等,苏州金唯智生物科技有限公司。荧光定量PCR仪,Bio-Rad公司;植物基因组DNA提取试剂盒,天根生化科技有限公司;2×Rapid Taq Master Mix PC,南京诺维赞生物科技股份有限公司;Prime STAR® Max DNA Polymerase,宝日医生物技术有限公司;多功能酶标仪,伯腾仪器有限公司。
1.1.3 培养基麦汁固体培养基:5°的麦汁加入20 g/L琼脂粉;种子培养基(g/L):玉米粉30.0,黄豆粉50.0,KH2PO4 1.5,MgSO4·7H2O 0.5 g/L;LB培养基(g/L):酵母提取物5.0,蛋白胨10.0,NaCl 10.0;酵母浸粉蛋白胨培养基(yeast extract peptone medium, YEP) (g/L):牛肉浸膏10.0,酵母提取物10.0,NaCl 5.0;土豆葡萄糖培养基(potato dextrose medium, PD)培养基(g/L):土豆200.0,葡萄糖20.0,KH2PO4 1.0,MgSO4·7H2O 0.1。
1.2 光照处理三孢布拉霉三孢布拉霉NRRL2896在添加有200 μg/mL头孢菌素的麦汁固体培养基上25 ℃黑暗培养5 d,随后在25 ℃环境下光照培养,从黑暗培养挪到光照培养时间点为0 min,设置持续光照(15 W/m2白光),并在2、5、10、30、60、180和360 min取各自平板上的三孢布拉霉菌丝体用液氮快速冷冻,保存于−80 ℃冰箱用于后续试验。
分别将含有pCAMBIA1303、pCAMBIA1303-procrgAF、F1、F2和F3质粒的农杆菌用于侵染三孢布拉霉,试验得到的三孢布拉霉转化子转接到含200 μg/mL头孢菌素、75 μg/mL潮霉素的麦汁培养基上,设置两组试验样本,一组为15 W/m2的白光照射条件;另一组为黑暗条件,温度均为25 ℃,分别继续培养3 d,挑取菌丝−80 ℃冰箱保存。
1.3 相对转录水平的分析取适量三孢布拉霉菌丝,放在研钵中用液氮充分研磨,后续按照RNA pure Plant Kit说明书提取纯化RNA,并使用琼脂糖凝胶电泳对提取的RNA质量进行检测。对质量良好的RNA进行反转录试验,根据Hifair® Ⅲ 1st Strand cDNA Synthesis SuperMix for qPCR (gDNA digester plus)试剂盒步骤,反转录得到相应的cDNA,按照试剂盒Heffi® qPCR SYBR Green Master Mix说明书步骤加入所需cDNA和其他成分。实时荧光定量PCR反应体系(50 μL):模板cDNA (0.1 g/L) 2 μL,上、下游引物(10 μmol/L)各1 μL,2×qPCR buffer 25 μL,ddH2O 21 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 10 s,60 ℃ 30 s,60 ℃ 30 s,4个循环;溶解曲线为仪器默认,试验中所用到的引物序列见表 1。通过实时荧光定量PCR仪Step One Plus上机检测得到相应数据,采用相对定量2−ΔΔCt法计算各个目的基因和内参基因tef1的相对转录水平,分析数据并用Origin作图。
| Primer name | Primer sequences (5′→3′) | Products size (bp) |
| pcrgA-R | TAGTCAGATCTACCATTGTTGATAATTCTCAAAAGAAAAAAAAAAAAAAGAAAAAGAATTGAAGC | 2 186 |
| pcrgA-F3 | AGCCAATACGCAAACCGGGGTTGATTTGTCGATCACAAG | |
| pcrgA-R | TAGTCAGATCTACCATTGTTGATAATTCTCAAAAGAAAAAAAAAAAAAAGAAAAAGAATTGAAGC | 1 499 |
| pcrgA-F2 | AGCCAATACGCAAACCCTTTGATAGGTAATGTATGTCGGTGTTGG | |
| pcrgA-R | TAGTCAGATCTACCATTGTTGATAATTCTCAAAAGAAAAAAAAAAAAAAGAAAAAGAATTGAAGC | 989 |
| pcrgA-F1 | AGCCAATACGCAAACCACGACTGGATTGGGCACTTAC | |
| pcrgA-R | TAGTCAGATCTACCATTGTTGATAATTCTCAAAAGAAAAAAAAAAAAAAGAAAAAGAATTGAAGC | 632 |
| pcrgA-F | AGCCAATACGCAAACCTTGCCAACAAGTTAGAGCAAT | |
| 1303-F | CTGCTCTAGCCAATACGCAAACCG | 2 166 |
| 1303-R | GCTTTCCCACCAACGCTGATC | |
| GUS-F | ATGGTAGATCTGACTAGTTTACGTCCTG | 11 336 |
| GUS-R | GGTTTGCGTATTGGCTAGAGCAG | |
| At-16s-F | GATCCTGGCTCAGAACGAACG | 236 |
| At-16s-R | CCCTACGGCTACCTTGTTACGAC | |
| q-crgA-F | GTCCTCCACCAGCCAAAATCT | 152 |
| q-crgA-R | CCAATCAACAAGGGAACACGG | |
| tef1-F | AACTCGGTAAGGGTTCCTTCAAG | 129 |
| tef1-R | CGGGAGCATCAATAACGGTAAC |
取适量三孢布拉霉菌丝于研钵中,倒入液氮充分研磨,使用植物基因组DNA提取试剂盒提取三孢布拉霉基因组用于btcrgA上游启动子的扩增。根据JGI数据库已有的Blakeslea trispora NRRL2456 V1.0基因组,设计引物扩增btcrgA翻译起始位点上游2 000 bp左右作为启动子序列,同源重组整合进pUC57载体中,转化到感受态E. coli DH5α中,37 ℃培养过夜,挑取菌落通过PCR和琼脂糖凝胶电泳验证并测序,得到的btcrgA启动子序列用Plant CARE分析该序列的顺式调控元件和BDGP预测可能的转录起始位点[16]。
1.5 btcrgA启动子截短载体构建和转化以三孢布拉霉基因组为模板设计相应引物,根据预测的启动子转录起始位点,且不破坏序列上的顺式调控元件基础上,从5′端逐渐截短crgA上游启动子序列,分别扩增不同长度的btcrgA启动子序列,取代原有的CaMV35S启动子,构建pCAMBIA1303-procrgAF、F1、F2和F3。使用重组酶Clone® Plus Multi One Step Cloning Kit连接到pCAMBIA-1303载体上,并转化到大肠杆菌感受态DH5α中,用2×Rapid Taq Master Mix PCR验证并提取质粒,测序结果正确后,通过电转化试验将构建好的质粒转化到农杆菌LBA4404中,并涂布在YEP固体培养基上,30 ℃培养48 h,挑取单菌落溶于ddH2O,并用高保真酶对特异片段进行PCR扩增,PCR反应体系(50 μL):溶于ddH2O的农杆菌1 μL,上、下游引物(10 μmol/L)各1 μL,2×PrimeSTAR Max Premix 25 μL,ddH2O 22 μL。PCR反应条件:95 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,共30个循环;72 ℃ 10 min。PCR产物用于测序,以确定构建的正确序列的质粒转入到了农杆菌中。
1.6 三孢布拉霉原生质体制作吸取适量三孢布拉霉孢子溶液涂布于麦汁固体培养基上,25 ℃培养5 d,刮取5 cm2的菌苔接种于种子液中,25 ℃、180 r/min摇床培养60 h后,取上述培养的野生型三孢布拉氏酶种子液于8 000 r/min离心10 min;用无菌水洗涤菌丝体和培养基混合物两次(8 000 r/min, 10 min),加入之前配制好的酶解液(2%溶菌酶、0.5%溶壁酶、0.25%蜗牛酶和0.25%纤维素酶溶解于0.6 mol/L NaCl溶液中)混匀,28 ℃、75 r/min避光孵育4 h[17]。待酶解完成,将三孢布拉霉菌丝用三层擦镜纸过滤,用0.6 mol/L NaCl溶液洗涤原生质体,4 000 r/min离心10 min,在光学显微镜下观察并用血球计数板计数,稀释原生质体到1×106个/mL备用。
1.7 农杆菌侵染三孢布拉霉将含有pCAMBIA1303-procrgA-F、F1、F2和F3载体菌株(LBA4404)分别接种于50 mL的YEP液体培养基中,在28 ℃、75 r/min条件下培养,使OD600约为0.4−0.6之间[18-20];吸取400 mL上述活化的农杆菌培养液与200 mL的三孢布拉霉原生质体混匀,并且加入终浓度为200 μmol/L的乙酰丁香酮,28 ℃、180 r/min避光诱导培养48 h,再加入800 μL PD液体培养基,继续在28 ℃、180 r/min条件下复苏24 h,8 000 r/min离心10 min,吸取剩余200 μL左右的培养液混匀涂布于含有200 μg/mL头孢菌素、75 μg/mL潮霉素的麦汁培养基上,25 ℃培养5 d,挑取菌落保存于−80 ℃冰箱,用于提取基因组和蛋白。
1.8 mGFP5蛋白荧光观察和GUS活性检测mGFP5是将野生型绿色荧光蛋白(green fluorescent protein, GFP)的Ser65替换为Thr,荧光强度较野生型的GFP提高4−6倍,且较GFP对生物毒害性减弱。挑取适量麦汁培养基上农杆菌侵染的三孢布拉霉转化子于PBS溶液中洗涤并8 000 r/min离心10 min,重复2次,取少量菌丝体到载玻片上置于倒置荧光显微镜上观察。取适量的新鲜三孢布拉霉菌丝体在研钵中充分研磨,按照GUS报告基因定量检测试剂盒提取植物总蛋白,−80 ℃保存,使用Bradford蛋白浓度测定试剂盒制作蛋白浓度标准曲线,并测定提取的三孢布拉霉菌丝总蛋白浓度。GUS蛋白可以将4-甲基-7-乙酰氧基香豆素-β-d-葡萄糖醛酸苷(4-methyl-2-oxo-2H-1-benzopyran-7-yl-β-d-glucopyranosiduronic acid, 4-MUG)催化为4-甲基伞形酮(4-methylumbelliferon, 4-MU),用反应终止液配制4-MU的1‒10 nmol/L稀释液作为标准液绘制标准曲线。4-MU的激发波长为356 nm,使用多功能酶标仪测定样品在456 nm处的吸光值。根据标准曲线公式计算各个反应样品4-MU生成浓度,GUS的活性用反应结果生成的4-MU量与总蛋白量和时间的比值[pmol 4-MU/(μg·min)]表示。
2 结果与讨论 2.1 光照对三孢布拉霉crgA转录的影响对不同光照时间培养条件下的三孢布拉霉btcrgA相对转录水平的分析(图 1)表明,光照显著影响了btcrgA在体内的转录水平,在10 min时达到较高的相对转录水平,是对照组黑暗处理条件下(0 min)的30多倍,二者间差异达到显著水平(P < 0.05);随着光照处理时间的增加,相对转录水平也逐渐降低,6 h后的btcrgA相对转录水平约为对照组的2倍,说明随着光照处理时间的延长,btcrgA的转录会受到光照的抑制作用。因此,在光照条件下,btcrgA的表达存在“光激活”和“光适应”现象[21]。
|
| 图 1 btcrgA持续光处理条件下各个时间点的相对转录水平 Figure 1 Relative transcription levels of btcrgA gene at various time points under continuous light treatment. 不同小写字母表示差异显著 Different lowercase letters indicate significant difference. |
|
|
启动子是转录水平上的一个重要调控元件,决定着基因的表达模式和表达强度,在对btcrgA翻译起始位点上游2 000 bp序列进行分析后,发现序列中靠近btcrgA起始密码子部分包含多个TATA-box元件,整个序列上分布着很多启动子和增强区域相关的顺式作用元件CAAT-box;此外还包括光响应元件、昼夜节律响应元件、创伤诱导元件、厌氧感应元件、低温诱导元件、脱落酸响应元件、干旱响应元件和茉莉酸甲酯响应元件等(表 2)。这表明btcrgA的表达可能受到光照、低温、脱落酸和干旱等外在因素的影响[22]。btcrgA启动子上存在多个光响应元件,如Sp1、LAMP-element、TCT-motif、G-box、Box 4和AE-box等,说明在光照条件下,btcrgA表达可能受到上游启动子光响应元件的调控。
| Name of cis-acting element | Sequence (5′→3′) | Function | Location | Number |
| TGACG-motif | TGACG/CGTCA | Involved in the MeJA-responsiveness | 484 (+), 1 450 (+) | 2 |
| WUN-motif | AAATTTCCT | Wound-responsive element | 1 822 (+) | 1 |
| ABRE | TACGGTC/ACGTG | Involved in the abscisic acid responsiveness | 678 (+), 957 (+) | 2 |
| AE-box | AGAAACAA | Part of a module for light response | 595 (+) | 1 |
| MBS | CAACTG | Involved in drought-inducibility | 731 (‒) | 1 |
| ARE | AAACCA | Essential for the anaerobic induction | 10 (‒), 90 (+), 893 (+), 1 050 (‒), 1 141 (+), 1 435 (+) | 6 |
| Box4 | ATTAAT | Part of a conserved DNA module involved in light responsiveness | 1 459 (+) | 1 |
| G-box | CACGTC | Involved in light responsiveness | 956 (+) | 1 |
| LAMP-element | CTTTATCA | Part of a light responsive element | 158 (+) | 1 |
| TCA-element | CCATCTTTTT | Involved in salicylic acid responsiveness | 1 828 (+) | 1 |
| LTR | CCGAAA | Involved in low-temperature responsiveness | 152 (+), 753 (+), 1 015 (+) | 3 |
| TCT-motif | TCTTAC | Part of a light responsive element | 627 (‒) | 1 |
| Circadian | CAAAGATATC | Involved in circadian control | 360 (‒) | 1 |
| Sp1 | GGGCGG | Light responsive element | 870 (+), 1 370 (+) | 2 |
| TGA-element | AACGAC | Auxin-responsive element | 505 (+), 601 (+) | 2 |
| O2-site | GATGATGTGG | Involved in zein metabolism regulation | 93 (‒) | 1 |
| (+):该顺式作用元件位于正链;(‒):该顺式作用元件位于互补链 (+): The cis-acting regulatory elements locate on the positive chain; (‒): The cis-acting regulatory elements locate on the complementary chain. |
||||
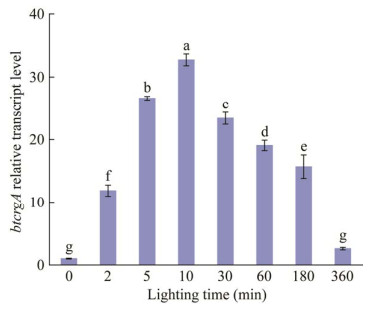
btcrgA上游启动子序列经过BDGP预测,得到一个位于启动子下游且分值为0.98的启动子转录起始区序列(5′-TTCTCTCTCAAAAAAAAAAGGGCTTTTTTAACGCTTGATCGATTCTTTTT-3′)。经过5′ cDNA末端快速扩增(rapid-amplification of cDNA ends, 5′ RACE)试验表明,btcrgA的起始转录位点为GTG上游的278 bp处。根据转录因子结合位点和启动子转录起始区在启动子序列上的分布位置,在不切断转录因子结合位点的原则下,以btcrgA翻译起始位点至上游632 bp处为最小的启动子截短序列,将这2 000 bp设计了4个缺失突变片段(图 2A),分别为procrgAF (2 186 bp)、procrgAF1 (1 499 bp)、procrgAF2 (989 bp)和procrgAF3 (632 bp),通过后续试验来确定btcrgA启动子的核心区域和起主要光调控作用的光响应元件所在区域[23]。
|
| 图 2 截短突变的btcrgA启动子片段及载体构建图 Figure 2 The different length of btcrgA promoters and vectors build legend. A:不同截短长度的procrgAFs. B:btcrgA启动子取代CaMV35S启动子位置结构图 A: procrgAFs with different truncated lengths. B: btcrgA promoter replaces CaMV35S promoter position structure legend. |
|
|
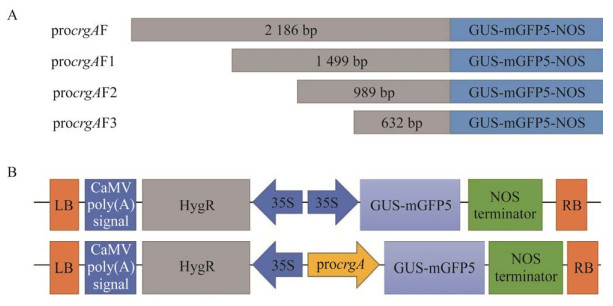
通过设计不同的引物(表 1),以三孢布拉霉基因组DNA为模板,经过PCR特异性扩增得到图 2A中不同大小的序列。以pCAMBIA1303质粒为出发载体(图 2B),构建pCAMBIA1303-procrgAF、pCAMBIA1303-procrgAF1、pCAMBIA1303-procrgAF2和pCAMBIA1303-procrgAF3 (以pCAMBIA1303载体作为阳性对照)。经过大肠杆菌DH5α扩增得到所需的质粒,电转进入农杆菌LBA4404,使用引物1303-F/R扩增特定序列经琼脂糖凝胶电泳(图 3)检测后送金唯智公司测序,并和JGI基因组序列对比,序列相似度为98%,说明获得了含有各个启动子序列缺失载体的LBA4404菌株。
|
| 图 3 重组载体菌落PCR验证 Figure 3 PCR products of recombinant vector colonies. M: DL5000 Marker; 1: pCAMBIA1303-procrgAF; 2: pCAMBIA1303-procrgAF1; 3: pCAMBIA1303-procrgAF2; 4: pCAMBIA1303-procrgAF3. |
|
|
构建的pCAMBIA1303、pCAMBIA1303-procrgAF、pCAMBIA1303-procrgAF1、pCAMBIA1303-procrgAF2和pCAMBIA1303-procrgAF3等5个载体分别转入农杆菌,并以它们侵染三孢布拉霉原生质体,得到的三孢布拉霉转化子和阴性对照(不携带质粒的农杆菌LBA4404侵染的三孢布拉霉)一同分别在倒置荧光显微镜下观察,每个视野都对明场(bright field, BF)和mGFP5 [异硫氰酸荧光素场(fluorescein isothiocyanate isomer, FITC) field]进行了观察,并设置了BF和FITC的融合视野(图 4),结果表明mGFP5在三孢布拉霉中基本不存在本底水平表达,其他均观察到了荧光信号,CaMV35S可以驱动GUS-mGFP5融合蛋白在三孢布拉霉中表达,且构建的4个截短的btcrgA启动子均可使外源基因在三孢布拉霉中表达。
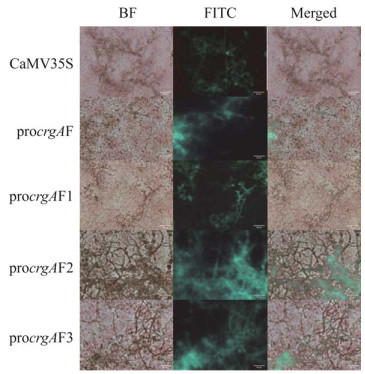
|
| 图 4 三孢布拉霉mGFP5荧光观察结果 Figure 4 Fluorescence observation of mGFP5 in Blakeslea trispora (40×). |
|
|
为测定CaMV35S和不同截短长度的btcrgA启动子驱动GUS表达的能力,在OD595和OD456条件下,绘制了牛血清白蛋白(bovine serum albumin, BSA)和4-MU标准曲线,它们分别为y=0.929 61x+0.523 82 (R2=0.990 53)和y=5 700.509 09x+1 503.727 27 (R2=0.998 0),符合测定要求。经过对GUS酶活性的数据统计,结果表明野生型三孢布拉霉基本无催化4-MUG的活性,而CaMV35S和不同截短的btcrgA启动子对应菌株都具有一定的催化4-MUG的能力(图 5),这间接表明了不同启动子相应驱动GUS表达的能力。在光照条件下,不同启动子驱动GUS表达的能力由强到弱依次为:procrgAF3 > procrgAF2 > procrgAF1 > CaMV35S。另外,启动子procrgAF1和procrgAF在驱动GUS表达的能力上无显著差异,procrgAF3则在驱动GUS表达能力上约为procrgAF2的2倍,两者之间差异显著(P < 0.05)。不同种类、数量的顺式作用元件和转录因子结合位点的不同,最后都会影响启动子驱动基因的表达结果,启动子片段procrgAF3比procrgAF2强,在启动子序列的989−632 bp区间内可能有较强的转录抑制元件。同样地,在1 499−989 bp区间也含有转录抑制元件,而procrgAF1和procrgAF启动子片段驱动GUS表达的能力相近。但通过启动子序列的分析结果表明,在这一区间内有多个和增强子相关的CAAT-box元件,说明在2 186−1 499 bp之间可能存在能够抵消CAAT-box带来的增强转录水平的抑制元件。通过对黑暗条件和光照条件下各个启动子活性的对比发现,光照条件下procrgAF和procrgAF1的启动子活性与黑暗条件下procrgAF和procrgAF1的启动子活性之间差异显著,即在启动子的1 499−989 bp之间存在依赖光激活的响应元件发挥作用。对启动子该处区域进行分析,发现该区域包含了AE Box、G Box和SP1等光响应元件。procrgAF2和procrgAF3启动子分别在黑暗和光照条件下的活性都无明显变化,但在procrgAF3的632 bp序列内有一个Box 4顺式作用元件(参与光响应的保守DNA模块的部分元件),Box 4未发挥光调控可能是由于其需要和其他的元件共同作用[24]。另外,procrgAF3驱动GUS的表达能力为在三孢布拉霉体内表达外源基因提供了内源性的强启动子,对激活元件和抑制元件在btcrgA启动子上的具体定位提供了研究基础。
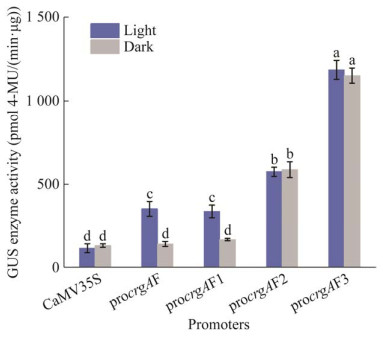
|
| 图 5 CaMV35S启动子和截短突变的btcrgA启动子片段三孢布拉霉表达GUS酶活性 Figure 5 GUS enzyme activity expressed by Blakeslea trispora with different length promoter fragments. 不同小写字母表示差异显著 Different lowercase letters indicate significant difference. |
|
|
本研究克隆并分析了btcrgA上游启动子2 000 bp序列的顺式作用元件,其中Sp1、LAMP-element、TCT-motif、G-box、Box4和AE-box等顺式作用元件均和光照有关。本研究根据预测的启动子中元件组成,构建了4个不同长度的启动子截短突变片段,驱动下游GUS-mGFP5融合蛋白的三孢布拉霉体内表达载体;通过荧光信号观察和GUS酶活检测,对构建的截短启动子活性进行了定性和定量分析,结果表明crgAF3启动子驱动的GUS表达的酶活性最高,btcrgA启动子的核心区位于crgAF3截短序列中,且4个截短启动子在三孢布拉霉体内驱动外源基因表达的能力都高于CaMV35S启动子的活性;黑暗和光照条件下GUS酶活结果表明,btcrgA启动子的1 499−989 bp序列之间存在依赖光激活的顺式作用元件,对下游基因的表达具有显著的调控作用。
| [1] |
NAVARRO E, RUIZ-PÉREZ VL, TORRES-MARTÍNEZ S. Overexpression of the crgA gene abolishes light requirement for carotenoid biosynthesis in Mucor circinelloides[J]. European Journal of Biochemistry, 2000, 267(3): 800-807. DOI:10.1046/j.1432-1327.2000.01058.x |
| [2] |
LORCA-PASCUAL JM, MURCIA-FLORES L, GARRE V, TORRES-MARTÍNEZ S, RUIZ-VÁZQUEZ RM. The RING-finger domain of the fungal repressor crgA is essential for accurate light regulation of carotenogenesis[J]. Molecular Microbiology, 2004, 52(5): 1463-1474. DOI:10.1111/j.1365-2958.2004.04070.x |
| [3] |
MURCIA-FLORES L, LORCA-PASCUAL JM, GARRE V, TORRES-MARTÍNEZ S, RUIZ-VÁZQUEZ RM. Non-AUG translation initiation of a fungal RING finger repressor involved in photocarotenogenesis[J]. The Journal of Biological Chemistry, 2007, 282(21): 15394-15403. DOI:10.1074/jbc.M610366200 |
| [4] |
NICOLÁS-MOLINA FE, NAVARRO E, RUIZ-VÁZQUEZ RM. Lycopene over-accumulation by disruption of the negative regulator gene crgA in Mucor circinelloides[J]. Applied Microbiology and Biotechnology, 2008, 78(1): 131-137. DOI:10.1007/s00253-007-1281-5 |
| [5] |
RODRÍGUEZ-ORTIZ R, MICHIELSE C, REP M, LIMÓN MC, AVALOS J. Genetic basis of carotenoid overproduction in Fusarium oxysporum[J]. Fungal Genetics and Biology: FG & B, 2012, 49(9): 684-696. |
| [6] |
TAGUA VG, NAVARRO E, GUTIÉRREZ G, GARRE V, CORROCHANO LM. Light regulates a Phycomyces blakesleeanus gene family similar to the carotenogenic repressor gene of Mucor circinelloides[J]. Fungal Biology, 2020, 124(5): 338-351. DOI:10.1016/j.funbio.2019.10.007 |
| [7] |
GONG ZY, LUO W, DU Y, YU XB. Regulation effect of crgA on carotenoid production in Blakeslea trispora[J]. Acta Microbiologica Sinica, 2017, 57(10): 1527-1535. (in Chinese) 巩尊洋, 罗玮, 杜瑶, 余晓斌. crgA调控三孢布拉霉合成类胡萝卜素[J]. 微生物学报, 2017, 57(10): 1527-1535. |
| [8] |
QUILES-ROSILLO MD, RUIZ-VÁZQUEZ RM, TORRES-MARTÍNEZ S, GARRE V. Light induction of the carotenoid biosynthesis pathway in Blakeslea trispora[J]. Fungal Genetics and Biology, 2005, 42(2): 141-153. DOI:10.1016/j.fgb.2004.10.008 |
| [9] |
LUO W, GONG ZY, LI N, ZHAO YZ, ZHANG HL, YANG X, LIU YT, RAO ZM, YU XB. A negative regulator of carotenogenesis in Blakeslea trispora[J]. Applied and Environmental Microbiology, 2020, 86(6): e02462-19. |
| [10] |
HU YB, PI CY, ZHANG Z, XIANG BY, XIA LQ. Recent advances in protein expression system of filamentous fungi[J]. China Biotechnology, 2020, 40(5): 94-104. (in Chinese) 胡益波, 皮畅钰, 张哲, 向柏宇, 夏立秋. 丝状真菌蛋白表达系统研究进展[J]. 中国生物工程杂志, 2020, 40(5): 94-104. |
| [11] |
YIN JY, WANG Y, XU LX, ZHU L, WANG C, LIU WB, MIAO WG. Cloning and expression analysis of different-length fragments of Oidium heveae (HO-73) promoter WY172[J]. Biotechnology Bulletin, 2020, 36(1): 29-36. (in Chinese) 殷金瑶, 王义, 徐良向, 朱利, 王晨, 刘文波, 缪卫国. 橡胶树白粉菌(HO-73)启动子WY172不同长度片段的克隆及表达活性分析[J]. 生物技术通报, 2020, 36(1): 29-36. |
| [12] |
GOLDBERG H, HELAAKOSKI T, GARRETT LA, KARSENTY G, PELLEGRINO A, LOZANO G, MAITY S, de CROMBRUGGHE B. Tissue-specific expression of the mouse alpha 2(I) collagen promoter. Studies in transgenic mice and in tissue culture cells[J]. Journal of Biological Chemistry, 1992, 267(27): 19622-19630. DOI:10.1016/S0021-9258(18)41820-0 |
| [13] |
HUANG XN, LU XF, LI JJ. Cloning, characterization and application of a glyceraldehyde-3-phosphate dehydrogenase promoter from Aspergillus[J]. Journal of Industrial Microbiology & Biotechnology, 2014, 41(3): 585-592. |
| [14] |
CEN YF, ZHU MZ, YE W, LI SN, ZHONG GH, ZHANG WM. Cloning and functional identification of trichothecene mycotoxin biosynthesis gene promoter from Paramyrothecium roridum[J]. Biotechnology Bulletin, 2021, 37(8): 85-94. (in Chinese) 岑由飞, 朱牧孜, 叶伟, 李赛妮, 钟国华, 章卫民. Paramyrothecium roridum中单端孢霉烯毒素生物合成基因启动子的克隆和功能鉴定[J]. 生物技术通报, 2021, 37(8): 85-94. |
| [15] |
BHURIA M, GOEL P, KUMAR S, SINGH AK. The promoter of AtUSP is co-regulated by phytohormones and abiotic stresses in Arabidopsis thaliana[J]. Frontiers in Plant Science, 2016, 7: 1957. |
| [16] |
WANG Y, WANG C, RAJAOFERA MJN, ZHU L, LIU WB, ZHENG FC, MIAO WG. WY7 is a newly identified promoter from the rubber powdery mildew pathogen that regulates exogenous gene expression in both monocots and dicots[J]. PLoS One, 2020, 15(6): e0233911. |
| [17] |
WANG YL, WANG YC, CHEN X, GAO N, WU Y, ZHANG HF. Protoplast fusion between Blakeslea trispora 14 271 (+) and 14 272 (−) enhanced the yield of lycopene and β-carotene[J]. World Journal of Microbiology and Biotechnology, 2021, 37(4): 58. |
| [18] |
FRANDSEN RJN. A guide to binary vectors and strategies for targeted genome modification in fungi using Agrobacterium tumefaciens-mediated transformation[J]. Journal of Microbiological Methods, 2011, 87(3): 247-262. |
| [19] |
LI DD, TANG Y, LIN J, CAI WW. Methods for genetic transformation of filamentous fungi[J]. Microbial Cell Factories, 2017, 16(1): 168. |
| [20] |
MICHIELSE CB, HOOYKAAS PJJ, van den HONDEL CAMJJ, RAM AFJ. Agrobacterium-mediated transformation as a tool for functional genomics in fungi[J]. Current Genetics, 2005, 48(1): 1-17. |
| [21] |
SCHMOLL M. Assessing the relevance of light for fungi: Implications and insights into the network of signal transmission[J]. Advances in applied microbiology, 2011, 76: 27-78. |
| [22] |
GAO Y, ZAN XL, WU XF, YAO L, CHEN YL, JIA SW, ZHAO KJ. Identification of fungus-responsive cis-acting element in the promoter of Brassica juncea chitinase gene, BjCHI1[J]. Plant Science, 2014, 215/216: 190-198. |
| [23] |
KWON BR, KIM MJ, PARK JA, CHUNG HJ, KIM JM, PARK SM, YUN SH, YANG MS, KIM DH. Assessment of the core cryparin promoter from Cryphonectria parasitica for heterologous expression in filamentous fungi[J]. Applied Microbiology and Biotechnology, 2009, 83(2): 339-348. |
| [24] |
LI ZX, CHEN XB. Research advances on plant inducible promoters and related cis-acting elements[J]. Biotechnology Bulletin, 2015, 31(10): 8-15. (in Chinese) 李濯雪, 陈信波. 植物诱导型启动子及相关顺式作用元件研究进展[J]. 生物技术通报, 2015, 31(10): 8-15. |
 2023, Vol. 50
2023, Vol. 50