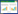扩展功能
文章信息
- 高亚男, 吴珊珊, 王鹏, 李春阳
- GAO Yanan, WU Shanshan, WANG Peng, LI Chunyang
- 微生物二甲基巯基丙酸内盐(DMSP)合成途径中关键酶的研究进展
- Research progress of key enzymes involved in the microbial dimethylsulfoniopropionate (DMSP) synthesis pathways
- 微生物学通报, 2022, 49(9): 3945-3954
- Microbiology China, 2022, 49(9): 3945-3954
- DOI: 10.13344/j.microbiol.china.220009
-
文章历史
- 收稿日期: 2022-01-03
- 接受日期: 2022-04-18
- 网络首发日期: 2022-05-17
硫元素作为生物必需的大量元素之一,在地球上广泛存在,约占地壳成分的0.03%[1]。硫元素在海洋中以不同价态的化合态存在,通过不同价态的有机硫和无机硫的相互转化参与全球硫循环,将陆地、海洋及大气生态系统联系在一起,实现全球硫循环[2]。二甲基巯基丙酸内盐(dimethysulfoniopropionate,DMSP)作为地球上含量最丰富的有机硫分子,每年产量约109 t,是海洋细菌重要的营养物质,对全球硫循环、信号传递和调节气候变化等方面具有非常重要的作用[3-5]。在细胞内,DMSP还发挥很多重要的生理功能,可以作为抗氧化剂、渗透保护剂、相容性溶质及自由基清除剂等[6-12]。此外,DMSP还是二甲基硫(dimethylsulfide,DMS)的主要前体物质。DMS是一种挥发性气体,每年通过微生物对DMSP的裂解作用可以产生多达3×108 t的DMS,其是大气中硫的最大生物来源[13-15]。DMS从海洋扩散到大气中后,会在光氧化作用下生成二甲基亚砜或硫酸盐气溶胶;DMS及其氧化产物可以通过形成云凝结核参与云的形成,导致云量增加,从而增加对照射到地球表面的阳光的反射,进而影响全球气候;大气中的DMS及其氧化产物还可以通过降水、降雪过程回到陆地环境中,从而完成从海洋到陆地的硫循环过程[2, 7-8]。
DMSP在海洋环境中广泛分布,其最早是在1948年从多管藻属的红藻中被鉴定出[16]。DMSP的产生者主要是海洋大型藻类(如红藻、绿藻)和浮游植物(如甲藻、硅藻),尤其在甲藻中DMSP的产量较高,甲藻的丰度与DMSP的产量呈正相关关系,随着甲藻的增加,DMSP在海水中的浓度明显升高[6-7]。海洋表层海水中DMSP的浓度通常比深层海水的浓度高,DMSP的浓度也有较强的季节变化规律,春季浓度开始上升,夏季浓度最高,秋季和冬季再逐渐下降[2, 6, 12, 17]。目前,人们对海洋细菌分解代谢DMSP的过程及机制开展了大量研究[18-19],DMSP代谢过程中多个酶的催化过程及机制得到了阐明[20-25]。与DMSP分解代谢的研究相比,人们对DMSP合成过程及相关酶催化机制的研究相对较少。DMSP合成途径中的第一个酶由Curson等在2017年从一株海洋细菌中鉴定获得[12],随后,他们从浮游植物中鉴定出DMSP合成途径中关键酶的基因[11]。2019年,Williams等报道了在近海沉积物中细菌是DMSP的主要产生者[26]。2020年,Zheng等发现了马里亚纳海沟里的细菌可以产生DMSP[9];同年,Trottmann等发现伯克霍尔德氏菌(Burkholderia)在产生细菌毒力因子的过程中会产生DMSP[27]。
DMSP的生产者以甲硫氨酸为前体物质,通过甲基化反应、转氨反应和脱羧反应等催化过程在体内合成DMSP[26]。目前已报道的DMSP合成途径有3条(图 1),根据第一步反应类型的不同,这3条代谢途径分别被称为甲基化途径、转氨途径和脱羧途径。甲基化途径是部分高等植物和一些海洋异养细菌合成DMSP的途径,在该途径中,甲硫氨酸首先发生甲基化反应产生S-甲基-甲硫氨酸(S-methyl-methionine,SMM),SMM再经过脱羧、氧化过程产生DMSP-乙醛(DMSP-aldehyde)后,脱氢产生DMSP (图 1);在DMSP转氨合成途径中,甲硫氨酸先发生转氨反应产生4-甲基巯基-2-氧丁酸(4-methylthio-2-oxobutyrate,MTOB),再经过还原过程和甲基化反应产生4-二甲基亚砜-2-羟基丁酸(4-dimethylsulfonio-2-hydroxybutyrate,DMSHB),最后通过脱羧反应产生DMSP,转氨途径主要存在于大型藻类、硅藻、珊瑚及多种海洋α变形菌纲细菌中(图 1);DMSP的脱羧合成途径存在于甲藻和鞭毛虫体内,该途径的第一步是甲硫氨酸通过脱羧反应产生3-甲基巯基丙胺(3-methylthiopropylamine,MTPA),该途径的后续反应步骤尚待证实,推测会先生成巯基丙酸甲酯(methylmercaptopropionate,MMPA)再最终产生DMSP (图 1)。在DMSP的合成过程中,硫原子的甲基化过程是DMSP合成的关键步骤[14, 28]。
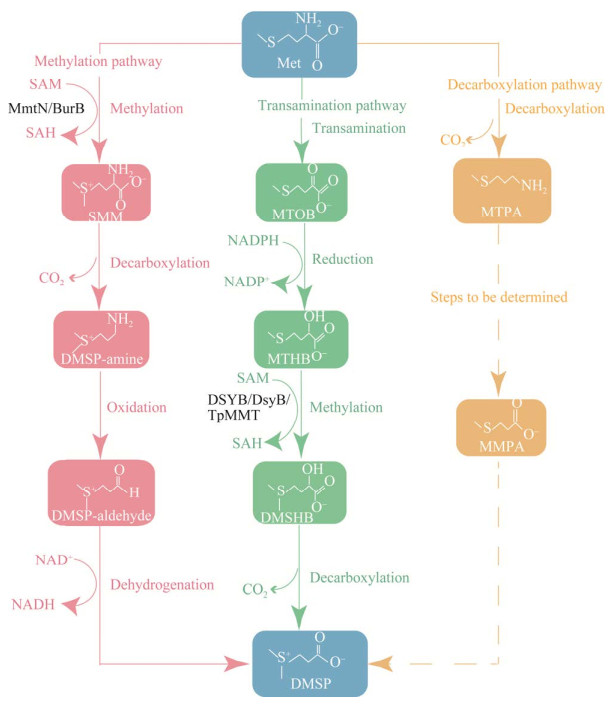
|
| 图 1 DMSP的3种生物合成途径 Figure 1 Three biosynthetic pathways of DMSP. 红色的代表DMSP合成的甲基化途径,绿色的代表转氨途径,黄色的代表脱羧途径;虚线表示脱羧途径中尚待证实的反应过程;目前已经鉴定出的DMSP合成途径中的关键酶用黑色表示 The methylation pathway for DMSP synthesis is depicted in red, the transamination pathway in green, and decarboxylation pathway in yellow. Dotted lines represent the unconfirmed steps of the decarboxylation pathway. Key enzymes identified in DMSP synthesis pathways are highlighted in black. |
|
|
截至目前,已经报道的参与DMSP合成的关键酶有5种,包括DsyB[12]、DSYB[11]、TpMMT[10]、MmtN[26]和BurB[27]。这5种酶均属于甲基转移酶类,其中来源于细菌的DsyB和来源于藻类的DSYB、TpMMT是转氨合成途径中的关键酶,催化MTHB甲基化生成DMSHB,而细菌来源的MmtN和BurB则在DMSP合成的甲基化途径中发挥作用,催化甲硫氨酸生成SMM[10-12, 26-27]。
DMSP是全球硫循环的重要参与者。DMSP不仅在海洋生物中发挥着重要的生理功能,其裂解产物DMS还可以通过参与云凝结核的形成对全球气候产生影响,因此,研究DMSP的合成机制具有重要的意义。目前已经有3条DMSP合成途径被报道,其中有5种DMSP合成关键酶被鉴定获得,但是尚无关于DMSP合成关键酶的晶体结构及催化机制的研究。本文对目前已报道的DMSP合成关键酶的研究进展进行系统阐述,梳理DMSP合成酶之间的联系与区别并进行讨论和展望,以期增进人们对DMSP产生过程及全球硫循环的理解。
1 DMSP转氨合成途径中的关键酶 1.1 海洋细菌中MTHB甲基化转移酶DsyB第一个被鉴定的DMSP合成基因dsyB来源于海洋异养细菌Labrenzia aggregata LZB033;Curson等对中国东海分离出的细菌菌株进行筛选,发现菌株LZB033可以裂解DMSP产生DMS;然而在未添加外源DMSP时该菌株仍然能产生DMS,表明菌株LZB033可能具有合成DMSP的能力;进一步的气相色谱和液相色谱-质谱联用结果表明该菌株确实能合成DMSP,这是第一个海洋细菌合成DMSP的报道[12]。当该菌株在DMSP转氨合成途径的中间产物存在的条件下生长时,DMSP产量显著提高,说明其可能通过转氨途径合成DMSP;从该菌株的细胞提取物中能检测到甲硫氨酸氨基转移酶和MTOB还原酶的活性,表明该菌株可以利用甲硫氨酸合成MTHB (图 1)[12]。通过构建基因组文库并对功能基因进行筛选和测序,Curson等鉴定出基因dsyB是菌株LZB033合成DMSP过程中的关键甲基化酶,其可以催化MTHB生成DMSHB;基因敲除的结果表明dsyB是该菌株中DMSP合成所必需的基因,其编码的DsyB蛋白属于S-腺苷甲硫氨酸(S-adenosylmethionine,SAM)依赖的甲基转移酶家族(Pfam family 00891)[12]。
Curson等的研究进一步发现,基因组中含有dsyB同源基因的海洋细菌均有产生DMSP的能力,表明dsyB可以作为一个标记基因预测细菌合成DMSP的能力[12]。根据宏基因组分析,在Tara Oceans和全球海洋样品(global ocean sampling)宏基因组数据库中,约0.5%的海洋细菌含有dsyB的同源基因,并且dsyB多存在于海洋α变形菌纲的细菌中,而在陆地宏基因组中几乎找不到dsyB的同源基因,这个结果与DMSP主要来源于海洋的观点一致[11-12]。
1.2 浮游植物中的MTHB甲基转移酶DSYB海洋浮游植物和大型海藻等被认为是DMSP的重要生产者,不同种群细胞中的DMSP浓度差异较大[3-4, 11, 17]。基于海洋细菌中dsyB的研究结果,Curson等从真核藻类中鉴定出编码DMSP合成蛋白的关键基因DSYB[11]。DSYB是细菌中DsyB的同源蛋白,也是一种SAM依赖的甲基转移酶,需要SAM的参与才能表现出其甲基化活性;DSYB与DsyB是同功酶,均催化MTHB生成DMSHB;体外酶活实验表明,在大肠杆菌中异源表达的DSYB无酶活,需要添加热变性的藻体细胞裂解物后才表现出体外活性,表明DSYB的活性可能需要藻类细胞裂解物中存在的辅助因子[11]。
虽然DSYB是DsyB的同源蛋白,系统发育分析表明DSYB来源于真核生物而DsyB来源于海洋α变形菌纲细菌,两者各自独立成支,这可能与微生物在进化中的基因复制与丢失有关[11]。目前在海洋植物、珊瑚、部分硅藻及大多数甲藻中均有DSYB同源基因的存在,其中珊瑚中的DSYB可能是甲藻中DSYB基因水平转移的结果[11, 17]。通过对北太平洋表层海水宏转录组的分析,Curson等发现真核生物中DSYB的转录本数量大约是原核生物中dsyB转录本数量的2倍,表明在北太平洋表层海水中,相比于海洋细菌,真核生物应该是DMSP更主要的产生者[11]。
1.3 硅藻中的MTHB甲基化转移酶TpMMT硅藻在全球广泛分布,是海洋浮游植物中的一个重要类群,DMSP在一些海洋硅藻中可以积累到较高的胞内浓度[10]。Kageyama等以硅藻拟南海链藻(Thalassiosira pseudonana) CCMP1335为研究对象,对其基因组进行分析,搜索可能与DMSP合成相关的基因;通过基因组分析、体内活性和cDNA产物的功能研究,他们找到了硅藻中编码DMSP合成途径关键酶的候选基因(TpMT1和TpMT2),为了检测候选基因表达的蛋白是否具有酶活,将其克隆至大肠杆菌中表达并纯化蛋白,纯化后的重组蛋白TpMT2表现出MTHB甲基转移酶活性,能将MTHB转化为DMSHB,他们将TpMT2命名为TpMMT[10]。
重组表达的TpMMT催化反应的最适温度为25 ℃,最适酶活pH值为8.0,高浓度的氯化钠或氯化钾均会抑制TpMMT的酶活;TpMMT与DSYB/DsyB属于同功酶,但其与DSYB/DsyB的蛋白序列相似度很低[10]。目前TpMMT基因只在硅藻中发现,在其他生物中未找到其同源基因。
2 DMSP甲基化合成途径中的关键酶 2.1 海洋细菌中甲硫氨酸甲基转移酶MmtN除海水外,海洋沉积物中也有DMSP的积累。盐沼池塘、河口和深海的表层沉积物中的DMSP浓度(5−128 nmol/g)比沉积物上层水体中的DMSP浓度(0.01−0.70 nmol/mL)高出大约3个数量级[26]。Williams等发现从盐沼沉积物分离的细菌菌株中有1/4均能产生DMSP,这些细菌大多数含有基因dsyB,暗示着它们可以利用转氨途径合成DMSP;另一部分则不含有dsyB,表明这部分细菌可能具有一个未知的合成DMSP的基因或途径[26]。Williams等选择了新鞘脂菌属的一株细菌Novosphingobium sp. BW1进行了进一步研究,在菌株BW1的培养液中添加DMSP三种合成途径的不同中间产物,发现当添加甲硫氨酸和SMM时DMSP的产生量显著提高[26]。同时在反应产物中检测到甲基化途径的另一种中间产物DMSP-amine,确定菌株BW1应该利用甲基化途径合成DMSP;对比基因文库发现了一个参与DMSP合成的关键基因mmtN,其异源表达纯化后的蛋白MmtN具有SAM依赖的甲硫氨酸甲基转移酶活性,可以催化甲硫氨酸和SAM反应生成SMM,MmtN对SAM的Km值为1.00 mmol/L,对甲硫氨酸的Km值为2.02 mmol/L (图 2),产生的SMM再经过脱羧、氧化等反应产生DMSP[26](图 1)。底物特异性分析表明,MmtN只对甲硫氨酸有催化活性,而对DMSP转氨合成途径中的中间产物MTHB和DMSP脱羧合成途径中的中间产物MMPA及另一种氨基酸——甘氨酸均无催化活性(图 3),表明MmtN具有相对专一的底物特异性,基因mmtN主要存在于一些α变形菌类群中[26]。
在本研究选取的海水和沉积物样本中,含有mmtN的细菌数量低于含有dsyB的细菌,然而富集培养后的样品中含有mmtN的细菌数量更高,这表明富集培养条件下DMSP的产生更依赖含有mmtN的细菌[26]。细菌MmtN蛋白与植物中甲硫氨酸甲基转移酶MMT的N-末端甲基转移酶结构域有约30%的序列一致性,植物中的MMT还有一个额外的C端氨基转移酶结构域;MMT在高等植物中催化甲硫氨酸产生SMM,但无证据表明具有MMT的植物可以产生DMSP;细菌MmtN和植物MMT的序列长度和结构域的差异可能导致细菌MmtN的Km值较高,高达12.80 mmol/L,大约是植物MMT的10倍[26]。编码植物MMT的基因也存在于一些细菌基因组中,其中大多数是δ变形菌纲的细菌;大多数含有MMT编码基因的细菌不能产生DMSP,表明该基因可能不直接参与DMSP的合成,而是和细菌中的甲硫氨酸代谢有关。系统发育分析表明,细菌mmtN和植物的MMT基因之间序列差异较大,这可能是通过古老的水平基因转移产生的[26]。
2.2 细菌中甲硫氨酸甲基转移酶BurB伯克霍尔德氏菌属(Burkholderia)细菌是一种广泛存在于土壤、水体和人体中的革兰氏阴性菌。有些伯克霍尔德氏菌属细菌可以引起严重的传染病,如鼻疽和类鼻疽,严重时可能致命;马利基酚(malleicyprols)是伯克霍尔德菌的一种重要细菌毒力因子,其作用的发挥依赖分子中环丙醇活性基团[27]。Trottmann等以伯克霍尔德氏菌属中的模式菌株Burkholderia thailandensis为研究对象,在探究环丙醇活性基团的生物合成时发现了DMSP是一种中间代谢产物,表明该菌可以合成DMSP,通过基因失活实验确定了基因簇bur与DMSP的合成相关[27]。伯克霍尔德菌能以甲硫氨酸作为前体物质,利用甲基化途径合成DMSP,参与反应的酶包括甲基转移酶(BurB)、脱羧酶(BurI)、转氨酶(BurD)和脱氢酶(BurE),其中BurB为催化甲硫氨酸形成SMM的甲基转移酶,其与MmtN为同功酶[27]。序列比对表明BurB属于第Ⅴ类甲基转移酶,具有一个SET结构域;在之前的报道中,只有第Ⅰ类和第Ⅲ类甲基转移酶包含SET结构域[26-27]。在大肠杆菌中异源表达的BurB具有体外活性,可以在SAM存在的情况下将甲硫氨酸转变为SMM[26-27]。目前,BurB在海洋细菌中的分布情况尚未被报道,但BurB的发现为后续鉴定能够合成DMSP的菌株提供了新的可能。
3 展望目前已经报道的5种DMSP合成酶中(表 1),转氨途径的DSYB和DsyB在藻类和细菌中较常见[11-12],TpMMT目前仅在硅藻中发现,转氨途径被认为是表层海水中DMSP的主要产生途径,与海水及光照密切相关;在目前已报道的3条DMSP合成途径中,转氨途径对全球DMSP的产量贡献最大[10];甲基化途径也有2种酶(MmtN和BurB)在细菌中被报道,mmtN在海水环境中的丰度远远低于dsyB,而在沉积物中的相对丰度更高,因此甲基化途径可能更多存在于海洋沉积物环境中[26-27]。在DMSP的脱羧合成途径中,目前尚无任何一个酶被鉴定获得,这一合成途径中DMSP合成关键酶仍值得关注和探究。目前仅在甲藻和鞭毛虫体内发现了DMSP通过脱羧途径合成,该条途径对全球DMSP产量的贡献尚不清楚。虽然DMSP转氨合成途径和甲基化合成途径中的5种甲基转移酶已经被鉴定获得,但是这些酶的催化机制尚未见报道,阐明关键酶的催化机制将有助于更好地理解DMSP的代谢和全球硫循环过程。
| 酶 Enzyme |
来源 Source |
功能 Function |
分子质量 Molecular mass (kDa) |
所属途径 Pathway |
主要分布 Main distribution |
| DsyB | Labrenzia aggregata LZB033 | 催化MTHB生成DMSHB的SAM依赖的甲基转移酶 SAM-dependent methyltransferase that catalyzes MTHB to produce DMSHB |
~37.2 | 转氨途径 Transamination pathway |
海水环境中的α变形菌纲细菌 Marine alphaproteobacteria |
| DSYB | Prymnesium parvum CCAP946 |
催化MTHB生成DMSHB的SAM依赖的甲基转移酶 SAM-dependent methyltransferase that catalyzes MTHB to produce DMSHB |
~40.0 | 转氨途径 Transamination pathway |
海洋植物、珊瑚、部分硅藻及大多数甲藻 Marine plants, corals, some diatoms and most dinoflagellates |
| TpMMT | Thalassiosira pseudonana CCMP1335 | 催化MTHB生成DMSHB的SAM依赖的甲基转移酶 SAM-dependent methyltransferase that catalyzes MTHB to produce DMSHB |
~36.4 | 转氨途径 Transamination pathway |
部分硅藻 Some diatoms |
| MmtN | Novosphingobium sp. BW1 | 催化甲硫氨酸生成SMM的SAM依赖的甲基转移酶 SAM-dependent methyltransferase that catalyzes Met to produce SMM |
~33.1 | 甲基化途径 Methylation pathway |
海洋沉积物中的α变形菌纲细菌 Marine alphaproteobacteria in marine sediments |
| BurB | Burkholderia thailandensis | 催化甲硫氨酸生成SMM的SAM依赖的甲基转移酶 SAM-dependent methyltransferase that catalyzes Met to produce SMM |
~19.1 | 甲基化途径 Methylation pathway |
伯克霍尔德菌属 Burkholderia |
过去认为只有光合真核生物才能产生DMSP,所以DMSP只产生于海洋真光层,浮游植物是该环境中DMSP的主要生产者;但Zheng等的研究表明在无光的马里亚纳海沟中也有DMSP的产生,而细菌则是此环境中DMSP的重要产生者[9]。浮游植物和细菌采用不同的营养方式,两者在DMSP合成过程中的异同是一个值得关注的问题。在目前已经报道的能够产生DMSP的细菌中,很多细菌基因组中不含有已被鉴定的DMSP合成关键基因;此外,最近的生物信息学分析表明,在海洋尤其在深海中,目前已报道的DMSP合成基因的丰度远低于DMSP分解代谢基因的丰度[29],表明在海洋细菌中尚有未被发现的DMSP合成关键酶或合成途径,对DMSP合成过程中新酶和新途径的鉴定将会成为未来研究的热点。DMSP具有重要的生理功能,在生态系统中也发挥着重要的作用,对DMSP生物合成、降解过程及代谢机制的研究将有助于更好地理解全球硫循环过程。
| [1] |
Li L, Wang P, Peng MJ, Li YG. Advances on studies of marine dimethyl sulfide producing bacteria and its functional genes[J]. Journal of Huazhong Agricultural University, 2013, 32(5): 20-28. (in Chinese) 李立, 汪鹏, 彭梦珺, 李友国. 海洋二甲基硫产生菌及其功能基因研究进展[J]. 华中农业大学学报, 2013, 32(5): 20-28. DOI:10.3969/j.issn.1000-2421.2013.05.004 |
| [2] |
Yoch DC. Dimethylsulfoniopropionate: its sources, role in the marine food web, and biological degradation to dimethylsulfide[J]. Applied and Environmental Microbiology, 2002, 68(12): 5804-5815. DOI:10.1128/AEM.68.12.5804-5815.2002 |
| [3] |
Yu J, Lai JG, Tian JY, Xu R, Wang XD, Xiong J, Yang GP. Research progress on DMSP source in the marine planktonic ecosystem and its transfer and transformation through the food chain[J]. Periodical of Ocean University of China, 2021, 51(4): 80-89. (in Chinese) 于娟, 来敬广, 田继远, 许瑞, 王雪丹, 熊婕, 杨桂朋. 海洋浮游生态系统中DMSP的来源及在食物链中的传递和转化研究进展[J]. 中国海洋大学学报(自然科学版), 2021, 51(4): 80-89. |
| [4] |
Li SF, Chen YF, Zhang HP, Yang XW, Chen MC, Chen HR. Research progress of DMSP in marine algae-bacteria interaction[J]. Marine Environmental Science, 2020, 39(5): 809-816. (in Chinese) 黎双飞, 陈俞妃, 张惠萍, 杨雪薇, 陈敏纯, 陈辉蓉. DMSP在海洋藻-菌互作中的作用研究进展[J]. 海洋环境科学, 2020, 39(5): 809-816. |
| [5] |
Curson ARJ, Todd JD, Sullivan MJ, Johnston AWB. Catabolism of dimethylsulphoniopropionate: microorganisms, enzymes and genes[J]. Nature Reviews Microbiology, 2011, 9(12): 849-859. DOI:10.1038/nrmicro2653 |
| [6] |
Zhang XH, Liu J, Liu JL, Yang GP, Xue CX, Curson ARJ, Todd JD. Biogenic production of DMSP and its degradation to DMS-their roles in the global sulfur cycle[J]. Science China Life Sciences, 2019, 62(10): 1296-1319. DOI:10.1007/s11427-018-9524-y |
| [7] |
Liao CS, Seebeck FP. In vitro reconstitution of bacterial DMSP biosynthesis[J]. Angewandte Chemie International Edition, 2019, 58(11): 3553-3556. DOI:10.1002/anie.201814662 |
| [8] |
Dickschat JS, Rabe P, Citron CA. The chemical biology of dimethylsulfoniopropionate[J]. Organic & Biomolecular Chemistry, 2015, 13(7): 1954-1968. |
| [9] |
Zheng YF, Wang JY, Zhou S, Zhang YH, Liu J, Xue CX, Williams BT, Zhao XX, Zhao L, Zhu XY, et al. Bacteria are important dimethylsulfoniopropionate producers in marine aphotic and high-pressure environments[J]. Nature Communications, 2020, 11: 4658. DOI:10.1038/s41467-020-18434-4 |
| [10] |
Kageyama H, Tanaka Y, Shibata A, Waditee-Sirisattha R, Takabe T. Dimethylsulfoniopropionate biosynthesis in a diatom Thalassiosira pseudonana: identification of a gene encoding MTHB-methyltransferase[J]. Archives of Biochemistry and Biophysics, 2018, 645: 100-106. DOI:10.1016/j.abb.2018.03.019 |
| [11] |
Curson ARJ, Williams BT, Pinchbeck BJ, Sims LP, Martínez AB, Rivera PPL, Kumaresan D, Mercadé E, Spurgin LG, Carrión O, et al. DSYB catalyses the key step of dimethylsulfoniopropionate biosynthesis in many phytoplankton[J]. Nature Microbiology, 2018, 3(4): 430-439. DOI:10.1038/s41564-018-0119-5 |
| [12] |
Curson ARJ, Liu J, Bermejo Martínez A, Green RT, Chan Y, Carrión O, Williams BT, Zhang SH, Yang GP, Bulman Page PC, et al. Dimethylsulfoniopropionate biosynthesis in marine bacteria and identification of the key gene in this process[J]. Nature Microbiology, 2017, 2: 17009. DOI:10.1038/nmicrobiol.2017.9 |
| [13] |
Zhang XH, Liu J, Liu JL, Zheng YF. Biosynthesis and cleavage of DMSP and their roles in global sulfur cycle[J]. Bulletin of National Natural Science Foundation of China, 2018, 32(5): 471-478. (in Chinese) 张晓华, 刘骥, 柳敬丽, 郑艳芬. DMSP的生物合成与裂解及其在硫循环中的作用[J]. 中国科学基金, 2018, 32(5): 471-478. |
| [14] |
Gage DA, Cooper AJL, Hanson AD. S-methylmethionine conversion to dimethylsulfoniopropionate: evidence for an unusual transamination reaction[J]. Plant Physiology, 1997, 115(4): 1541-1548. DOI:10.1104/pp.115.4.1541 |
| [15] |
Gage DA, Rhodes D, Nolte KD, Hicks WA, Leustek T, Cooper AJL, Hanson AD. A new route for synthesis of dimethylsulphoniopropionate in marine algae[J]. Nature, 1997, 387(6636): 891-894. DOI:10.1038/43160 |
| [16] |
Challenger F, Simpson MI. A precursor of the dimethyl sulphide evolved by Polysiphonia fastigiata; dimethyl- beta-propiothetine (dimethyl-beta-carboxythylsulphonium hydroxide) and its salts[J]. The Biochemical Journal, 1947, 41(3): xl. |
| [17] |
Raina JB, Tapiolas DM, Forêt S, Lutz A, Abrego D, Ceh J, Seneca FO, Clode PL, Bourne DG, Willis BL, et al. DMSP biosynthesis by an animal and its role in coral thermal stress response[J]. Nature, 2013, 502(7473): 677-680. DOI:10.1038/nature12677 |
| [18] |
Chen Y, Schäfer H. Towards a systematic understanding of structure-function relationship of dimethylsulfoniopropionate-catabolizing enzymes[J]. Molecular Microbiology, 2019, 111(6): 1399-1403. DOI:10.1111/mmi.14230 |
| [19] |
Johnston AW, Green RT, Todd JD. Enzymatic breakage of dimethylsulfoniopropionate—a signature molecule for life at sea[J]. Current Opinion in Chemical Biology, 2016, 31: 58-65. DOI:10.1016/j.cbpa.2016.01.011 |
| [20] |
Li CY, Wei TD, Zhang SH, Chen XL, Gao X, Wang P, Xie BB, Su HN, Qin QL, Zhang XY, et al. Molecular insight into bacterial cleavage of oceanic dimethylsulfoniopropionate into dimethyl sulfide[J]. PNAS, 2014, 111(3): 1026-1031. DOI:10.1073/pnas.1312354111 |
| [21] |
Wang P, Chen XL, Li CY, Gao X, Zhu DY, Xie BB, Qin QL, Zhang XY, Su HN, Zhou BC, et al. Structural and molecular basis for the novel catalytic mechanism and evolution of DddP, an abundant peptidase-like bacterial Dimethylsulfoniopropionate lyase: a new enzyme from an old fold[J]. Molecular Microbiology, 2015, 98(2): 289-301. DOI:10.1111/mmi.13119 |
| [22] |
Li CY, Zhang D, Chen XL, Wang P, Shi WL, Li PY, Zhang XY, Qin QL, Todd JD, Zhang YZ. Mechanistic insights into dimethylsulfoniopropionate lyase DddY, a new member of the cupin superfamily[J]. Journal of Molecular Biology, 2017, 429(24): 3850-3862. DOI:10.1016/j.jmb.2017.10.022 |
| [23] |
Peng M, Chen XL, Zhang D, Wang XJ, Wang N, Wang P, Todd JD, Zhang YZ, Li CY. Structure-function analysis indicates that an active-site water molecule participates in dimethylsulfoniopropionate cleavage by DddK[J]. Applied and Environmental Microbiology, 2019, 85(8): e03127-e03118. |
| [24] |
Shao X, Cao HY, Zhao F, Peng M, Wang P, Li CY, Shi WL, Wei TD, Yuan ZL, Zhang XH, et al. Mechanistic insight into 3-methylmercaptopropionate metabolism and kinetical regulation of demethylation pathway in marine dimethylsulfoniopropionate-catabolizing bacteria[J]. Molecular Microbiology, 2019, 111(4): 1057-1073. DOI:10.1111/mmi.14211 |
| [25] |
Li CY, Wang XJ, Chen XL, Sheng Q, Zhang S, Wang P, Quareshy M, Rihtman B, Shao X, Gao C, et al. A novel ATP dependent dimethylsulfoniopropionate lyase in bacteria that releases dimethyl sulfide and acryloyl-CoA[J]. eLife, 2021, 10: e64045. DOI:10.7554/eLife.64045 |
| [26] |
Williams BT, Cowles K, Bermejo Martínez A, Curson ARJ, Zheng YF, Liu JL, Newton-Payne S, Hind AJ, Li CY, Rivera PPL, et al. Bacteria are important dimethylsulfoniopropionate producers in coastal sediments[J]. Nature Microbiology, 2019, 4(11): 1815-1825. DOI:10.1038/s41564-019-0527-1 |
| [27] |
Trottmann F, Ishida K, Franke J, Stanišić A, Ishida-Ito M, Kries H, Pohnert G, Hertweck C. Sulfonium acids loaded onto an unusual thiotemplate assembly line construct the cyclopropanol warhead of a Burkholderia virulence factor[J]. Angewandte Chemie International Edition, 2020, 59(32): 13511-13515. DOI:10.1002/anie.202003958 |
| [28] |
Bullock HA, Luo HW, Whitman WB. Evolution of dimethylsulfoniopropionate metabolism in marine phytoplankton and bacteria[J]. Frontiers in Microbiology, 2017, 8: 637. |
| [29] |
Teng ZJ, Qin QL, Zhang WP, Li J, Fu HH, Wang P, Lan MS, Luo GF, He JF, McMinn A, et al. Biogeographic traits of dimethyl sulfide and dimethylsulfoniopropionate cycling in polar oceans[J]. Microbiome, 2021, 9(1): 207. DOI:10.1186/s40168-021-01153-3 |
 2022, Vol. 49
2022, Vol. 49