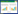扩展功能
文章信息
- 申丹丹, 兰佳鑫, 李诚昊, 陆健, 李晓敏
- SHEN Dandan, LAN Jiaxin, LI Chenghao, LU Jian, LI Xiaomin
- 食源性致病性大肠杆菌O157:H7和O55:H7特异性噬菌体的分离与鉴定
- Isolation and identification of phages of food-borne pathogenic Escherichia coli O157:H7 and O55:H7
- 微生物学通报, 2022, 49(8): 3358-3372
- Microbiology China, 2022, 49(8): 3358-3372
- DOI: 10.13344/j.microbiol.china.211224
-
文章历史
- 收稿日期: 2021-12-24
- 接受日期: 2022-03-10
- 网络首发日期: 2022-04-05
2. 江南大学粮食发酵与食品生物制造国家工程研究中心, 江苏 无锡 214122;
3. 江南大学江苏省生物活性制品加工工程技术研究中心, 江苏 无锡 214122
2. National Engineering Research Center for Cereal Fermentation and Food Biomanufacturing, Jiangnan University, Wuxi 214122, Jiangsu, China;
3. Jiangsu Provincial Engineering Research Center for Bioactive Product Processing, Jiangnan University, Wuxi 214122, Jiangsu, China
大肠杆菌(Escherichia coli)是人体肠道中的正常菌群之一,部分菌株携带致病基因,可诱发不同程度的腹泻及肠道外感染[1-2]。其中,肠出血性大肠杆菌(enterohemorrhagic E. coli,EHEC)感染会导致出血性肠炎、血性腹泻和溶血性尿毒症综合征,最常见的血清型[3]是O157:H7[4];肠致病性大肠杆菌(enteropathogenic E. coli,EPEC)是儿童死亡和发病的主要原因之一[5],血清型O55:H7于2003年在德国分离,是首先完成基因组测序分析的2株EPEC之一,O55:H7也被证明是O157:H7的最新前体[6]。在治疗EHEC和EPEC的感染中,抗生素的大量使用导致多重耐药菌株的出现,已发展成为全球公共卫生问题。因此,新型抗菌制剂的研发成为目前的研究热点。
噬菌体是感染细菌细胞的病毒,只有在其宿主细菌存在的情况下才能复制,因此不会感染非特异性细菌、动物、人类或植物[7]。裂解性噬菌体可在新噬菌体颗粒复制后诱导宿主细菌细胞裂解,对宿主菌以外的生命体不具有毒性作用,可用作控制细菌病原体的生物保护剂。近年来,关于噬菌体控制食源性致病菌的研究及应用多有报道[8-9],针对沙门氏菌[10]、单核细胞性李斯特菌[11]、发酵乳杆菌[12]和金黄色葡萄球菌[13]的特异性噬菌体制剂已用于食品工业,而且前两者已获得美国食品和药物管理局批准用于食品工业和噬菌体治疗[14]。
本研究分别以EHEC O157:H7 EDL933和EPEC O55:H7 CB9615为宿主菌,从污水中分离出2株噬菌体vB_EcoM_P251和vB_EcoM_P255 (以下简称P251和P255),对其形态、感染复数、感染特征、宿主范围进行鉴定,并进一步对其全基因组进行测序和生物信息学分析,对探索致病性大肠杆菌防控的抗生素替代方法、减少致病菌耐药性提高的风险、促进人类健康具有参考意义。
1 材料与方法 1.1 菌株大肠杆菌O157:H7 EDL933 (ATCC 43895)、O103:K: H8、O78:K80和O78:H11等购自美国典型培养物保藏中心(American Type Culture Collection,ATCC),大肠杆菌O55:H7 CB9615、O128:H2、O114:H2、O145:H28和O111:H8等购自中国普通微生物菌种保藏管理中心(China General Microbiological Culture Collection Center,CGMCC)。噬菌体P251和P255为本研究分离。
1.2 培养基和主要试剂、仪器LB液体培养基(g/L):NaCl 10.0,蛋白胨10.0,酵母粉5.0。LB固体培养基:含20.0 g/L琼脂的LB液体培养基。LB半固体培养基:含5.0 g/L琼脂的LB液体培养基。
SM缓冲液:NaCl 5.8 g,MgSO4·7H2O 2.0 g,1 mol/L Tris-HCl 50 mL,2%明胶5 mL,溶于1 L蒸馏水中,1×105 Pa灭菌20 min,常温保存备用。
小型冷冻高速离心机,艾本德公司;透射电子显微镜,日立株式会社;恒温恒湿培养箱,上海一恒科学仪器有限公司。
1.3 方法 1.3.1 噬菌体的分离与优化参照Bujak等的方法[15],将采集的污水(青岛)经过滤纸过滤后12 000 r/min离心6 min,并通过0.22 μm滤膜过滤除菌。将滤液与LB液体培养基、对数期(OD600为0.6)宿主菌液混合,37 ℃、160 r/min培养过夜,培养液经过12 000 r/min离心6 min及0.22 μm滤膜过滤除菌,收集的滤液即为样品液。
吸取100 μL宿主菌液涂布于LB平板上,吸取10 μL上述样品液滴于其上,晾干后37 ℃倒置培养12 h。挑取平板上的单个噬菌斑,加入1 mL SM缓冲液并涡旋,静置后上清即为噬菌体悬液。重复上述步骤1次,并采用双层平板法继续纯化3−5次,获得纯噬菌体,于−80 ℃保存(与30%甘油等比例混匀)。使用噬斑试验和10倍倍比系列稀释测定噬菌体效价[16]。
1.3.2 噬菌体的形态测定将连续稀释的噬菌体涂布于含有其宿主细菌的LB平板上,37 ℃培养12 h后使用游标卡尺测量噬菌斑直径(mm)。
参照Yang等的方法[17]利用PEG法浓缩噬菌体,用2%磷钨酸钠(pH 6.7)进行负染色,利用透射电子显微镜在100 kV电压下观察。选取4个相同的噬菌体颗粒估计噬菌体的形态结构和平均大小。
1.3.3 噬菌体的宿主谱测定本研究的测试菌株包括大肠杆菌O157:H7、O55:H7、O111:K58:H21、O103:H8、O78:K80、O78:H11、O128:H2、O114:H2、O145:H28、O26:H11、O111:H8等常见食源性致病性大肠杆菌和模式菌株DH5α、JM109、K12,及金黄色葡萄球菌、枯草芽孢杆菌、地衣芽孢杆菌、酵母菌等。
参照Han等的方法[18],通过潜在宿主平板上的斑点试验确定待测噬菌体宿主谱。
1.3.4 噬菌体的最佳感染复数(multiplicity of infection, MOI)和体外裂解动力的测定将噬菌体与对数期宿主细菌(108 CFU/mL)以100、10、1、0.1和0.01 PFU/CFU的比例混合,37 ℃孵育15 min,8 000 r/min离心2 min,用6 mL的LB液体培养基重悬沉淀,37 ℃、220 r/min培养3 h,分装于1.5 mL EP管,12 000 r/min离心10 min,经0.22 μm滤膜过滤;取100 μL滤液梯度稀释,每个稀释梯度3个重复,用双层平板法测定噬菌体滴度,最终噬菌体滴度为3个平行实验的平均值,其中效价最高的MOI值为该噬菌体的最佳感染复数。
将O157:H7和O55:H7培养至OD600为1.0 (浓度约为1×109 CFU/mL),分别按MOI为0.01、0.1、1、10加入对应噬菌体,对照组为1 mL菌液与等体积SM Buffer混合液。以上5组分别用LB调整至OD600为0.5且体积相同,37 ℃、160 r/min培养,每隔30 min (0−300 min)取样测定OD600。每组3个重复。
1.3.5 噬菌体的吸附速率和一步生长曲线测定参照Yıldirim等的方法[19],按MOI为0.01分别将P251和P255加入O157:H7和O55:H7对数期培养液中(感染复数MOI为0.01),室温孵育5、10和15 min时各收集100 µL样品,5 000×g离心1 min去除噬菌体吸附的细胞,未吸附噬菌体滴度通过双层板滴定法测定,根据未吸附噬菌体的初始滴度和测试滴度之间的比率确定吸附的噬菌体数量。数据来自3次重复实验。
按最佳感染复数比例分别将P251和P255加入OD600为1.0的O157:H7和O55:H7培养液中,37 ℃吸附15 min,8 000×g离心15 min,用LB液体培养基洗涤沉淀2次,去除未吸附的噬菌体颗粒。加入100 mL LB液体培养基,37 ℃孵育,每5 min取样一次,用双层平板法测定每个时间点的噬菌体效价。绘制一步生长曲线,计算噬菌体潜伏期和暴发量。数据来自3次重复实验。
1.3.6 噬菌体的温度和pH敏感性测定将200 μL噬菌体原液分别于37、50、60和70 ℃孵育2 h[16],每20 min取100 μL样品梯度稀释,双层平板法测定效价。每个温度3次重复。
取900 μL LB液体培养基(pH 1.0−13.0)分别与100 μL噬菌体原液混合,37 ℃孵育2 h,取100 μL样品梯度稀释,双层平板法测定效价。每个pH值3次重复。
1.3.7 噬菌体的基因组分析采用苯酚-氯仿法提取噬菌体P251和P255的基因组[20]。对提取的基因组进行核酸类型鉴定:将提取的核酸分别与DNase I (20 U/µg)和RNase A (5 U/µg)混合并孵育;对P251基因组采用限制性内切酶Sma I、对P255基因组采用ApaL I酶切并进行琼脂糖凝胶电泳检测。DNA文库构建使用Illumina TruSeq™ Nano DNA Sample Prep Kit完成。利用上海美吉生物医药科技有限公司的MiSeq PE300测序仪完成序列测定。基因组序列拼装结果由上海美吉生物公司提供,通过RAST和Prokka综合确定的开放阅读框的结果,并根据BLASTp对功能注释结果进行了补充[21]。核苷酸起始编号被设置在编码末端酶大亚基的基因起点的上游[22]。利用SnapGene制作全基因组图谱[23]。选择末端酶大亚基蛋白,采用MEGA 6.0软件neighbor-joining方法构建系统发育树。
2 结果与分析 2.1 噬菌体的分离与优化两株噬菌体是独立分离的。分别以大肠杆菌O157:H7和O55:H7为宿主,从污水样品中分离出2株噬菌体,参照Adriaenssens等[24]提出的分类命名规则,分别命名为大肠杆菌噬菌体vB_EcoM_P251和大肠杆菌噬菌体vB_EcoM_P255。两株噬菌体在平板上对其宿主细胞形成不同大小的圆形透明区。P251噬菌斑经游标卡尺测量的直径大小为(0.48±0.03) mm,边缘无晕环(图 1A);P255噬菌斑直径大小为(0.90±0.03) mm,边缘有晕环(图 1B)。
|
| 图 1 P251 (A)和P255 (B)的噬菌斑 Figure 1 Plaque of phage P251 (A) and P255 (B). |
|
|
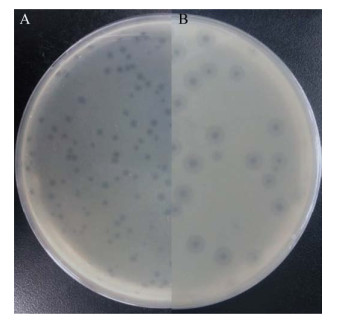
使用透射电子显微镜(transmission electronmicroscope,TEM)观察噬菌体样品。P251噬菌体头部为二十面体结构,尾部由一个可伸缩的外层尾鞘和一个内部尾管组成,头部直径约为(88.00±2.11) nm,尾部结构约长(52.00±2.44) nm,该病毒在形态上类似于肌尾病毒科(Myoviridae)的噬菌体(图 2A)。P255噬菌体具有典型的二十面体头部结构和长的可收缩的尾巴,包含长径为(90.00±2.11) nm、横径为(61.00±1.95) nm的头部和(109.00±1.53) nm长度、(18.00±1.62) nm厚度的尾部,具有肌尾病毒科(Myoviridae)的特点(图 2B)。
|
| 图 2 噬菌体P251 (A)和P255 (B)在TEM下的形态 Figure 2 Morphology of bacteriophages P251 (A) and P255 (B) under TEM. |
|
|
针对不同血清型的大肠杆菌,评估了2株噬菌体的宿主范围。P251噬菌体仅对测试的2株大肠杆菌菌株O157:H7和O78:H11具有裂解活性。P255噬菌体的宿主覆盖了EHEC和EPEC的多个菌株,包括大肠杆菌O157:H7、O55:H7、O78:H11、O128:H2、O26:H11、O111:H8等不同血清型,P255对它们均具有裂解活性。
此外,P255噬菌体对大肠杆菌DH5α、JM109和K12也有感染性。两株噬菌体对于其他种属的菌株,如金黄色葡萄球菌、地衣芽孢杆菌、枯草芽孢杆菌、沙门氏菌、酵母菌均不具有裂解活性(表 1)。
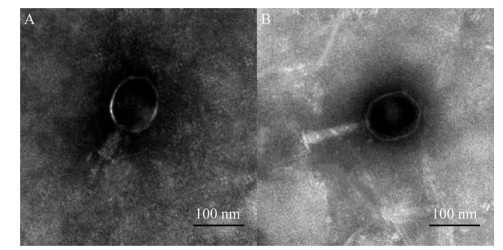
2.2.3 噬菌体的最佳感染复数(MOI)MOI值是感染性噬菌体与宿主细胞的比率,最佳MOI值被认为是以最大水平产生噬菌体的MOI值。最终的菌体滴度为3个平行实验的平均值,其中效价最高的MOI值为该噬菌体的最佳感染复数。如表 2所示,随着感染复数比例的提高,噬菌体活性呈增长趋势,但并非完全正相关,噬菌体与宿主菌比例达到一定程度后趋于稳定,甚至出现下降趋势。当MOI为1时,P251和P255噬菌体的效价均达到最大,说明2株噬菌体的最佳感染复数均为1。
如图 3所示,噬菌体P251与宿主菌在不同感染复数的混合培养中,120 min前培养液OD600缓慢增加;120 min后实验各组培养液的OD600均开始下降,表明噬菌体P251可在不同MOI下裂解细菌,MOI为1时裂解效果最好;180 min后细菌基本被裂解完全。对噬菌体P255而言,OD600一直缓慢增加。
|
| 图 3 噬菌体P251 (A)和P255 (B)的体外裂解动力 Figure 3 In vitro lysis kinetics of bacteriophages P251 (A) and P255 (B). |
|
|
| Strains | Source or No. | P251 | P255 |
| E. coli O157:H7 | ATCC 43895 | + | + |
| E. coli O55:H7 | CB9615 | − | + |
| E. coli O157:H7 | ATCC 35150 | − | + |
| E. coli O157:H7 | ATCC 700728 | − | + |
| E. coli O111:K58:H21 | ATCC 29552 | − | − |
| E. coli O103:H8 | ATCC 11229 | − | − |
| E. coli O78:K80 | CICC 10421 | − | − |
| E. coli O78:H11 | ATCC 35401 | + | + |
| E. coli K12 | ATCC 23631 | − | + |
| ETEC control bacteria | CMCC(B)44817 | − | − |
| E. coli O128:H2 | JN1095 | − | + |
| E. coli O114:H2 | JN1316 | − | − |
| E. coli O145:H28 | CB9723 | − | − |
| E. coli O26:H11 | CB8962 | − | + |
| E. coli O111:H8 | CB7322 | − | + |
| E. coli DH5α | Thermo | − | + |
| E. coli JM109 | Thermo | − | + |
| Staphylococcus aureus | ATCC 25923 | − | − |
| Bacillus subtilis | ATCC 6633 | − | − |
| Salmonella enterica subsp. enterica | ATCC 14901 | − | − |
| Bacillus licheniformis | ATCC 14580 | − | − |
| Blastobotrys adeninivorans | CICC 33224 | − | − |
| 注:+:有裂解性;−:无裂解性 Note: +: Cracking capacity; −: No cracking capacity. |
|||
| 感染复数 MOI |
宿主菌浓度 Host bacteria concentration (CFU/mL) |
噬菌体浓度 Phage concentration (PFU/mL) |
P251效价 Titer of P251 (PFU/mL) |
P255效价 Titer of P255 (PFU/mL) |
| 100 | 104 | 106 | 7.80×107 | 8.66×107 |
| 10 | 105 | 106 | 2.16×109 | 2.54×109 |
| 1 | 106 | 106 | 2.56×1010 | 2.86×1010 |
| 0.1 | 107 | 106 | 1.04×109 | 9.40×108 |
| 0.01 | 108 | 106 | 1.55×109 | 1.48×107 |
噬菌体吸附到宿主细胞的速率、裂解后释放的噬菌体数量和暴发大小是任何病毒的基本生物学特性,也是噬菌体-宿主的特征值。噬菌体吸附实验显示,在培养的前5 min,游离噬菌体含量均降低到20%以下;作用15 min后游离噬菌体含量分别降低到8.1%和9.2%,吸附速率达到91.9%和90.8% (图 4)。
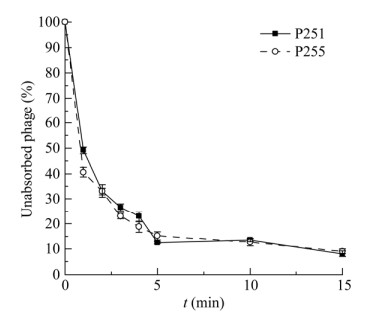
|
| 图 4 噬菌体P251和P255的吸附曲线 Figure 4 Adsorption rate curves of bacteriophages P251 and P255. |
|
|
对2株噬菌体进行了一步生长曲线测定,以确定其潜伏期和噬菌体暴发量。P251和P255的潜伏期为20 min和15 min,此后释放的病毒颗粒数量逐渐增加,上升期分别为20 min和30 min,分别在35 min和45 min后进入平台期(图 5)。
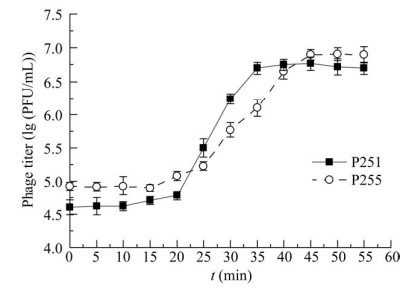
|
| 图 5 噬菌体P251和P255的一步生长曲线 Figure 5 One-step growth curve of bacteriophages P251 and P255. |
|
|
噬菌体的酸碱度和热稳定性是噬菌体评估的重要指标。两株噬菌体对温度都有一定的稳定性,P251在37、50和60 ℃作用120 min效价基本保持稳定,70 ℃作用60 min后效价开始下降,80 min后效价急剧下降,实验结束时,噬菌体效价下降了4.6 lg (PFU/mL) (图 6A)。P255在37、50和60 ℃作用120 min效价基本保持稳定,无下降趋势;70 ℃作用40 min后效价开始持续下降;120 min后效价为0 (图 6B)。
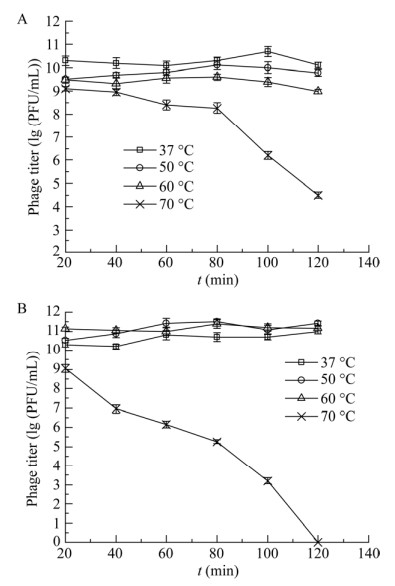
|
| 图 6 噬菌体P251 (A)和P255 (B)在不同温度下的生物稳定性 Figure 6 Biological stability of P251 (A) and P255 (B) under different temperature. |
|
|
噬菌体P251和P255分别在pH 3.0−11.0和pH 4.0−11.0可保持裂解活性,超出该范围时噬菌体效价有所降低。在pH 2.0时,P251和P255的效价分别降低2.44、5.29 lg (PFU/mL);在pH 12.0时,其效价滴度分别下降了3.91、2.68 lg (PFU/mL);当pH为1.0或13.0时,2株噬菌体效价均为0 (图 7)。
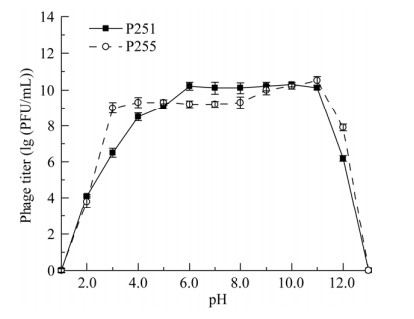
|
| 图 7 噬菌体P251和P255在不同pH条件下的生物稳定性 Figure 7 Biological stability of P251 and P255 under different pH. |
|
|
如图 8A、8B所示,2株噬菌体基因组均仅能被DNase I消化,对RNase A具有抗性,表明其核酸类型为DNA。选用的限制性内切酶均可作用于噬菌体基因组,说明其遗传物质是双链DNA (dsDNA)。经测序,P251基因组存在3个Sma I酶切位点,在图 8C电泳图中显示4个条带,P255基因组存在2个ApaL I酶切位点,在图 8C电泳图中显示2个条带且条带大小相符。因此,P251基因组为线性dsDNA,P255基因组为环状dsDNA。
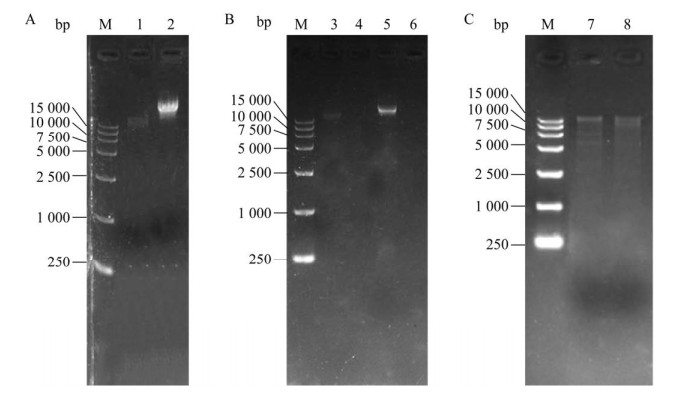
|
| 图 8 噬菌体核酸类型分析 Figure 8 Analysis of the nucleic acid type. A:1:P251基因组;2:P255基因组。B:3:P251基因组RNA酶酶切;4:P251基因组DNA酶酶切;5:P255基因组RNA酶酶切;6:P255基因组DNA酶酶切。C:7:Sma I酶酶切;8:ApaL I酶酶切。M:15000 DNA Marker A: 1: P251 genome; 2: P255 genome. B: 3: p251 genomic RNA enzyme digestion; 4: P251 genomic DNA enzyme digestion; 5: P255 genomic RNA enzyme digestion; 6: P255 genomic DNA enzyme digestion. C: 7: Sma I enzyme digestion; 8: ApaL I enzyme digestion. M: 15000 DNA Marker. |
|
|
通过RAST和Prokka综合确定开放阅读框,并利用BLASTp对功能注释结果进行了补充。核苷酸起始编号被设置在编码末端酶小亚基的基因起点的上游。
P251基因组全长为136 254 bp,GC含量为37%,含227个开放阅读框,具有明确功能的ORF有80个,而且具有2个tRNA编码区,具有一般双链噬菌体共有的4个结构单元:核苷酸代谢和复制,结构,裂解,以及DNA包装和代谢,不包括假定蛋白。核苷酸代谢模块主要包括DNA核酸外切酶(ORF154,ORF156)、核酸内切酶(ORF222,ORF61)、重组修复蛋白(ORF140)、核糖核苷酸还原酶(ORF64)等,它们共同提供合成噬菌体基因组的原材料。DNA连接酶(ORF37)、DNA聚合酶(ORF147,ORF151)、DNA解旋酶(ORF22,ORF23)等催化合成噬菌体DNA。RNA聚合酶(ORF161)等可以修饰宿主RNA聚合酶并识别噬菌体基因组的启动子区域,实现噬菌体基因表达。在DNA包装模块中,ORF1和ORF2分别被注释为末端酶大亚基和小亚基,共同组成了噬菌体末端酶,大亚基是多功能酶,在包装中起主要作用;小亚基与基因组特异性结合,引导包装进程的进行;ORF6被注释为门户蛋白,是噬菌体侵染过程中的DNA介导通道。此外,也发现了如ORF89的穿孔素表达基因,以及ORF224等与裂解有关的基因。在ORF84−89之间的尾部相关基因模块,hhpred结构域注释显示可能与受体识别和溶菌酶基因相关;同时还注释到了ORF10−12等与衣壳结构有关的蛋白模组。
P255基因组全长为111 068 bp,GC含量为35%,含有173个ORF,其中有明确功能的有73个,未找到编码tRNA的区域。注释了DNA包装模块和门户蛋白如ORF1、ORF2、ORF5,衣壳蛋白如ORF8−11,结构蛋白模块,核酸解旋酶模块如ORF17、ORF18,以及核苷酸还原酶如ORF68。另外,ORF84−90的尾部相关模块及穿孔素蛋白反映了P255的裂解模块。同样地,可以找到核酸代谢模块和DNA包装模块等聚集在一个区域的功能模块。
利用snapgene绘制噬菌体P251 (图 9)和P255 (图 10)的全基因组图谱。ORF分别用蓝色(噬菌体结构蛋白)、红色(宿主裂解)、绿色(DNA装配与复制)及白色(假定蛋白)三角箭头表示。
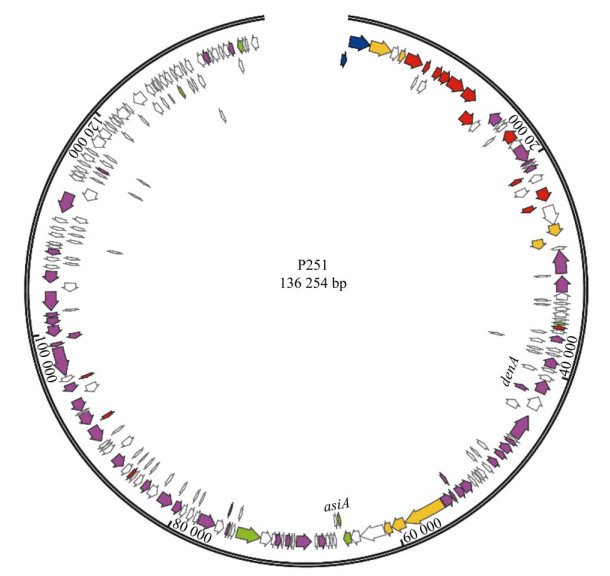
|
| 图 9 噬菌体P251全基因组图谱 Figure 9 The genome-wide map of bacteriophages P251. |
|
|
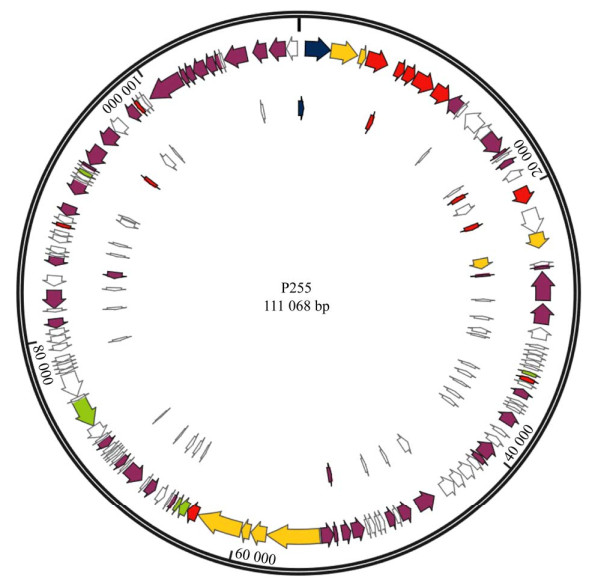
|
| 图 10 噬菌体P255全基因组图谱 Figure 10 The genome-wide map of bacteriophages P255. |
|
|
比较基因组显示,噬菌体P251和P255基因组在其48%的长度上共享72.24%的核苷酸同一性。91个ORF共享氨基酸同一性,包括核苷酸代谢和复制、结构、裂解、DNA包装和代谢等不同方面。
2.3.3 比较基因组分析和进化分析对噬菌体基因组核苷酸序列的BLASTn分析显示,在噬菌体基因组数据库中与其最相似的噬菌体是AR1 (NCBI ID:11034)。为更好地分析噬菌体之间的氨基酸统一性,使用Easyfig将P251、P255和AR1的开放阅读框进行比较(图 11)。
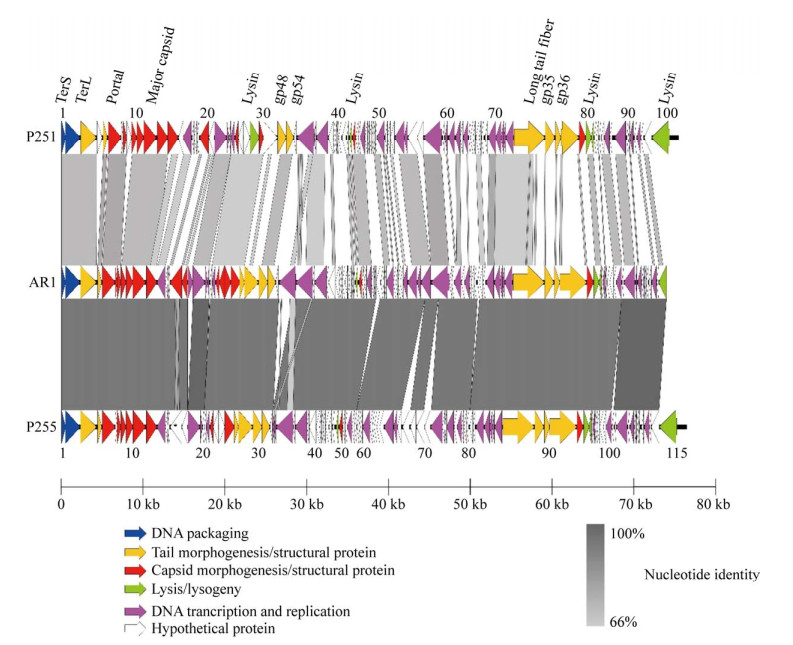
|
| 图 11 大肠杆菌噬菌体P251和P255与大肠杆菌噬菌体AR1的成对BLASTn比较 Figure 11 Paired BLASTn comparison of P251 and P255 and AR1. 图中标注了选定基因和编码保守的蛋白质。TerS:终止小亚单位;TerL:终止子大亚基;TMP:尾卷尺测量蛋白质;DIT:远端尾蛋白;Tal:尾部相关赖氨酸 The selected genes and proteins encoding conserved genes are marked in the figure. TerS: Termination of small subunit; TerL: Terminator large subunit; TMP: Tail tape to measure protein; DIT: Distal tail protein; Tal: Tail associated lysine. |
|
|
P251和AR1在55%的长度上具有72.5%的相似度,包括末端酶及衣壳、门户蛋白质等结构蛋白。对于裂解相关模块和尾部相关模块,P251-ORF45与AR1-ORF217具有76%的氨基酸相似度,而且均被注释为裂解抑制辅助蛋白。穿孔素蛋白P251-ORF45和AR1-ORF254具有64%的氨基酸相似度。P251-ORF84与AR1-ORF255共享57%的氨基酸相似度,均被标注为尾部纤维蛋白。利用hppred在AR1和P251的氨基酸序列中检测到了代号为PF00959的phage lysozyme的结构域。BLASTp结果显示被标记为远端亚基的P251-ORF87蛋白与AR1-ORF258具有一定相似度,均含有参与病毒释放的endo-N-acetylneuraminidase编码区。P251-ORF88和AR1-ORF259中同时发现了gp38这个和受体识别有关的基因区域,将P251-ORF89进一步注释为受体识别蛋白。
P255和AR1在94%的长度上具有92.82%的相似度,而且在噬菌体进化中的保守序列如末端酶和衣壳蛋白等氨基酸序列相似度都在95%及以上。与P251类似,AR1中被注释为和裂解模块及尾部相关模块的蛋白同样可以在P255中找到类似的对应区域,而且与P251相比二者氨基酸序列相似度更高。
如图 12所示,系统发育树显示噬菌体P251和CC31处于同一分支,亲缘关系最近;噬菌体P255和Enterobacteria phage RB53亲缘关系最近。噬菌体CC31和RB33均属于肌尾噬菌体科,结合电镜结果,可以基本确定噬菌体P251和P255属于肌尾噬菌体科。
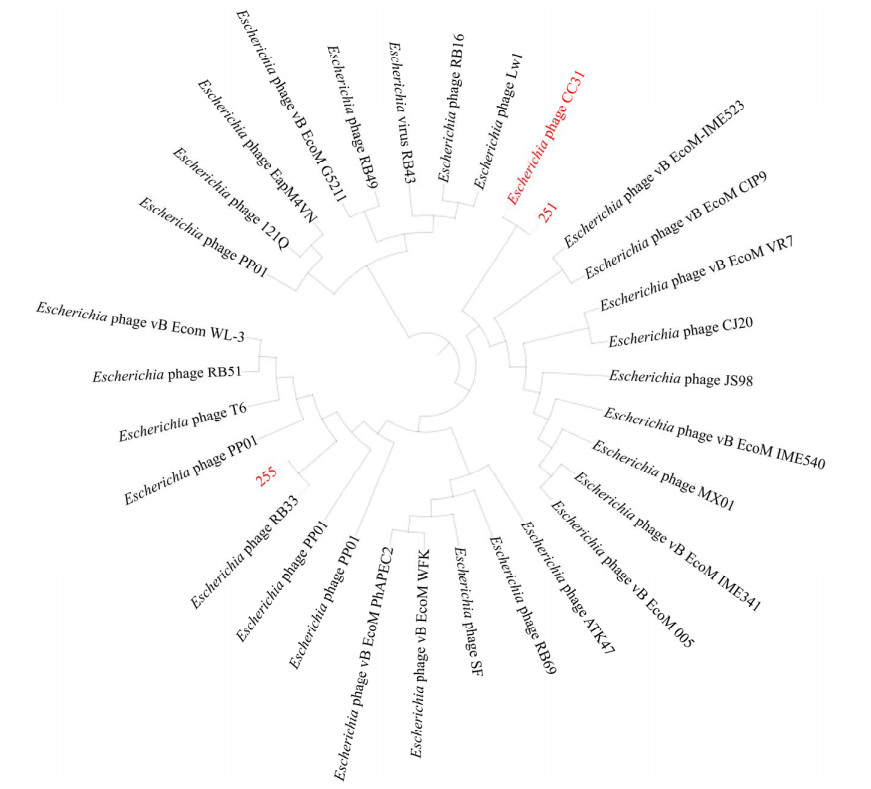
|
| 图 12 同源进化树 Figure 12 Evolutionary tree. |
|
|
本研究分别以EHEC O157:H7和EPEC O55:H7为宿主菌,从污水样品中分离出2株特异性噬菌体vB_EcoM_P251和vB_EcoM_P255,并对其形态、最佳感染系数、温度和pH敏感性、宿主谱等生物学特性进行了研究。结果表明,P251直径为(0.48±0.03) mm,边缘无晕环,效价为2.6×108 PFU/mL;P255直径为(0.90±0.03) mm,边缘有晕环,效价为6.1×108 PFU/mL。透射电子显微镜显示噬菌体P251和P255具有典型的二十面体头部和可收缩尾部,均属于肌尾病毒科(Myoviridae)。P251和P255的最佳感染复数均为1且具有较高的吸附效率。Yıldirim等[19]筛选的10株噬菌体在前5 min均表现出90%以上的吸附速率;Topka等[25]报道中称50%的噬菌体在孵育2 min内吸附到其宿主细胞上,与本文筛选的噬菌体表现出了近似特征。噬菌体P251和P255在37−60 ℃、pH 4.0−11.0条件下保持稳定的生物学活性,因此具有较宽的温度和酸碱性适用范围,其表现出的耐热、耐酸碱能力为其实际应用提供了参考。与感染大肠杆菌的其他噬菌体相比,P251和P255具有相对较高的暴发量、较短的潜伏期和较长的暴发时间,表明其对宿主细胞具有较高的感染效果和较强的溶解活性,在实践中可用于快速生产应用。Zhao等[26]观察到大多数肌尾病毒科的潜伏期和暴发量分别在21−120 min和50−100 PFU/cell之间,本研究中P251和P255作为肌尾病毒科符合这一规律;Litt等[27]观察到的7种噬菌体的上升期和暴发量分别在19−40 min和89−631 PFU/cell之间变化,与本研究结果相似。在本研究的测试宿主中,P251仅对O157:H7和O78:H11具有感染性,P255对11种不同血清型的EHEC和EPEC大肠杆菌均具有感染性,表现出对致病性大肠杆菌的广泛裂解能力。此外,P251和P255基因组均为双链DNA,其基因组全长分别为136 254 bp和111 068 bp,GC含量分别为37%和35%,分别含有227个和173个开放阅读框,其中分别有80个和73个ORF与已知功能的蛋白质具有显著相似性。P251和P255基因组在其48%的长度上共享72.24%的核苷酸同一性;91个开放阅读框共享氨基酸同一性,包括核苷酸代谢和复制、结构、裂解、DNA包装和代谢等不同方面。系统发育树进一步证实了P251和P255均属于肌尾噬菌体科。
新分离的O157:H7和O55:H7特异性噬菌体vB_EcoM_P251和vB_EcoM_P255具有短潜伏期、高暴发量和高吸附率的特点,表现出较强的温度稳定性和酸碱稳定性。这些特性使新分离的噬菌体成为食品工业、兽医和医学中抗O157:H7、O55:H7、O78:H11、O128:H2、O26:H11、O111:H8等致病性大肠杆菌的潜在理想生物活性剂,同时对于控制食源性致病菌的流行及减少致病菌耐药性提高的风险具有重要的意义。
| [1] |
Safwat Mohamed D, Farouk Ahmed E, Mohamed Mahmoud A, Abd El-Baky RM, John J. Isolation and evaluation of cocktail phages for the control of multidrug-resistant Escherichia coli serotype O104: H4 and E. coli O157: H7 isolates causing diarrhea[J]. FEMS Microbiology Letters, 2017, 365(2): fnx275. |
| [2] |
Kyle JL, Cummings CA, Parker CT, Quiñones B, Vatta P, Newton E, Huynh S, Swimley M, Degoricija L, Barker M, et al. Escherichia coli serotype O55: H7 diversity supports parallel acquisition of bacteriophage at shiga toxin phage insertion sites during evolution of the O157: H7 lineage[J]. Journal of Bacteriology, 2012, 194(8): 1885-1896. DOI:10.1128/JB.00120-12 |
| [3] |
Sharma M, Ryu JH, Beuchat LR. Inactivation of Escherichia coli O157: H7 in biofilm on stainless steel by treatment with an alkaline cleaner and a bacteriophage[J]. Journal of Applied Microbiology, 2005, 99(3): 449-459. DOI:10.1111/j.1365-2672.2005.02659.x |
| [4] |
Tozzoli R, Grande L, Michelacci V, Fioravanti R, Gally D, Xu XF, La Ragione R, Anjum M, Wu GH, Caprioli A, et al. Identification and characterization of a peculiar vtx2-converting phage frequently present in verocytotoxin-producing Escherichia coli O157 isolated from human infections[J]. Infection and Immunity, 2014, 82(7): 3023-3032. DOI:10.1128/IAI.01836-14 |
| [5] |
Weiss-Muszkat M, Shakh D, Zhou YZ, Pinto R, Belausov E, Chapman MR, Sela S. Biofilm formation by and multicellular behavior of Escherichia coli O55: H7, an atypical enteropathogenic strain[J]. Applied and Environmental Microbiology, 2010, 76(5): 1545-1554. DOI:10.1128/AEM.01395-09 |
| [6] |
Zhou ZM, Li XM, Liu B, Beutin L, Xu JG, Ren Y, Feng L, Lan RT, Reeves PR, Wang L. Derivation of Escherichia coli O157: H7 from its O55: H7 precursor[J]. PLoS One, 2010, 5(1): e8700. DOI:10.1371/journal.pone.0008700 |
| [7] |
Sulakvelidze A. Phage therapy: an attractive option for dealing with antibiotic-resistant bacterial infections[J]. Drug Discovery Today, 2005, 10(12): 807-809. DOI:10.1016/S1359-6446(05)03441-0 |
| [8] |
Cieplak T, Soffer N, Sulakvelidze A, Nielsen DS. A bacteriophage cocktail targeting Escherichia coli reduces E. coli in simulated gut conditions, while preserving a non-targeted representative commensal normal microbiota[J]. Gut Microbes, 2018, 9(5): 391-399. |
| [9] |
Dissanayake U, Ukhanova M, Moye ZD, Sulakvelidze A, Mai V. Bacteriophages reduce pathogenic Escherichia coli counts in mice without distorting gut microbiota[J]. Frontiers in Microbiology, 2019, 10: 1984. DOI:10.3389/fmicb.2019.01984 |
| [10] |
Yi Y, Abdelhamid AG, Xu YM, Yousef AE. Characterization of broad-host lytic Salmonella phages isolated from livestock farms and application against Salmonella enteritidis in liquid whole egg[J]. LWT, 2021, 144: 111269. DOI:10.1016/j.lwt.2021.111269 |
| [11] |
Sanlibaba P, TEZEL BU. Isolation of phages infecting Listeria monocytogenes[J]. 2019. DOI: 10.15237/gida.GD19036
|
| [12] |
Liu M, Bischoff KM, Gill JJ, Mire-Criscione MD, Berry JD, Young R, Summer EJ. Bacteriophage application restores ethanol fermentation characteristics disrupted by Lactobacillus fermentum[J]. Biotechnology for Biofuels, 2015, 8: 132. DOI:10.1186/s13068-015-0325-9 |
| [13] |
Horiuk Y, Horiuk V, Kukhtyn M, Tsvihun A, Kernychnyi S. Characterization of lytic activity of phage SAvB14 on Staphylococcus aureus variant bovis[J]. Journal of Advanced Veterinary and Animal Research, 2020, 7(3): 509-513. DOI:10.5455/javar.2020.g447 |
| [14] |
Guo HR, Xu JY, Zhou Y, Li ZQ, Dong XX, Li JQ. Extensive distribution of bacteriophage and the safety of oral application[J]. Journal of Chinese Institute of Food Science and Technology, 2021, 21(2): 349-357. (in Chinese) 郭浩然, 许菁彦, 周洋, 李子齐, 董星星, 李锦铨. 噬菌体的分布广泛性以及口服安全性[J]. 中国食品学报, 2021, 21(2): 349-357. |
| [15] |
Bujak K, Decewicz P, Kaminski J, Radlinska M. Identification, characterization, and genomic analysis of novel Serratia temperate phages from a gold mine[J]. International Journal of Molecular Sciences, 2020, 21(18): 6709. DOI:10.3390/ijms21186709 |
| [16] |
Litt PK, Saha J, Jaroni D. Characterization of bacteriophages targeting non-O157 shiga toxigenic Escherichia coli[J]. Journal of Food Protection, 2018, 81(5): 785-794. DOI:10.4315/0362-028X.JFP-17-460 |
| [17] |
Yang HJ, Liang L, Lin SX, Jia SR. Isolation and characterization of a virulent bacteriophage AB1 of Acinetobacter baumannii[J]. BMC Microbiology, 2010, 10: 131. DOI:10.1186/1471-2180-10-131 |
| [18] |
Han H, Wei XT, Wei Y, Zhang XF, Li XM, Jiang JZ, Wang R. Isolation, characterization, and bioinformatic analyses of lytic Salmonella enteritidis phages and tests of their antibacterial activity in food[J]. Current Microbiology, 2017, 74(2): 175-183. DOI:10.1007/s00284-016-1169-7 |
| [19] |
Yıldirim Z, Sakin T, Akçelik M, Akçelik N. Identification and characterization of lytic bacteriophages specific to foodborne pathogenic Escherichia coli O157: H7[J]. Food Science and Technology International, 2021, 27(1): 56-72. DOI:10.1177/1082013220929836 |
| [20] |
Summer EJ, Gonzalez CF, Bomer M, Carlile T, Embry A, Kucherka AM, Lee J, Mebane L, Morrison WC, Mark L, et al. Divergence and mosaicism among virulent soil phages of the Burkholderia cepacia complex[J]. Journal of Bacteriology, 2006, 188(1): 255-268. DOI:10.1128/JB.188.1.255-268.2006 |
| [21] |
Yuan YH, Gao MY, Peng Q, Wu DD, Liu PM, Wu Y. Genomic analysis of a phage and prophage from a Bacillus thuringiensis strain[J]. The Journal of General Virology, 2014, 95(Pt 3): 751-761. |
| [22] |
Lu SG, Le S, Tan YL, Zhu JM, Li M, Rao XC, Zou LY, Li S, Wang J, Jin XL, et al. Genomic and proteomic analyses of the terminally redundant genome of the Pseudomonas aeruginosa phage PaP1: establishment of genus PaP1-like phages[J]. PLoS One, 2013, 8(5): e62933. DOI:10.1371/journal.pone.0062933 |
| [23] |
Philippe C, Levesque S, Dion MB, Tremblay DM, Horvath P, Lüth N, Cambillau C, Franz C, Neve H, Fremaux C, et al. Novel genus of phages infecting Streptococcus thermophilus: genomic and morphological characterization[J]. Applied and Environmental Microbiology, 2020, 86(13): e00227-e00220. |
| [24] |
Adriaenssens E, Brister JR. How to name and classify your phage: an informal guide[J]. Viruses, 2017, 9(4): 70. DOI:10.3390/v9040070 |
| [25] |
Topka G, Bloch S, Nejman-Faleńczyk B, Gąsior T, Jurczak-Kurek A, Necel A, Dydecka A, Richert M, Węgrzyn G, Węgrzyn A. Characterization of bacteriophage vB-EcoS-95, isolated from urban sewage and revealing extremely rapid lytic development[J]. Frontiers in Microbiology, 2019, 9: 3326. DOI:10.3389/fmicb.2018.03326 |
| [26] |
Zhao FY, Sun HZ, Zhou XY, Liu GQ, Li ML, Wang C, Liu S, Zhuang YT, Tong YG, Ren HY. Characterization and genome analysis of a novel bacteriophage vB_SpuP_Spp16 that infects Salmonella enterica serovar pullorum[J]. Virus Genes, 2019, 55(4): 532-540. DOI:10.1007/s11262-019-01664-0 |
| [27] |
Litt PK, Jaroni D. Isolation and physiomorphological characterization of Escherichia coli O157: H7-infecting bacteriophages recovered from beef cattle operations[J]. International Journal of Microbiology, 2017, 2017: 7013236. |
 2022, Vol. 49
2022, Vol. 49