扩展功能
文章信息
- 杨青山, 黄罗冬, 吴宜婷, 张茜, 申佩弘
- YANG Qingshan, HUANG Luodong, HUANG Luodong, ZHANG Xi, SHEN Peihong
- 甲基营养菌MB200中mutS的缺失及高浓度甲醇和甲醛诱变
- Mutation of mutS gene in Methylobacterium sp. MB200 and induction of strains resistant to high concentration of methanol and formaldehyde
- 微生物学通报, 2022, 49(7): 2730-2740
- Microbiology China, 2022, 49(7): 2730-2740
- DOI: 10.13344/j.microbiol.china.211088
-
文章历史
- 收稿日期: 2021-11-17
- 接受日期: 2021-12-22
- 网络首发日期: 2022-01-27
2. 亚热带农业生物资源保护与利用国家重点实验室,广西 南宁 530004
2. State Key Laboratory for Conservation and Utilization of Subtropical Agro-Bioresources, Nanning 530004, Guangxi, China
甲基营养菌(Methylobacterium)是一类具有专门的甲基代谢途径的细菌,可利用单碳或非C−C键的低碳化合物(如甲烷、甲醇、甲醛等)进行生长,由于这类细菌多为杆状,也称为甲基杆菌[1-2]。截至目前,研究最广泛的是Methylobacterium extorquens AM1,该菌是一种多功能的甲基营养细菌,可利用多种碳基质包括单碳化合物(如甲醇、甲胺和甲酸盐)和多碳化合物(如丙酮酸、琥珀酸、乳酸和醋酸盐等)进行生长[3]。目前菌株AM1已被设计用于生产各种代谢产物,包括衣康酸[4]、甲戊酸[5]、甲基延胡索酸、甲基琥珀酸[6]和巴豆酸[7]等。
使用甲醇作为唯一碳源进行发酵可降低实验污染风险,也可以降低下游加工成本,甲醇是一种很有前途的低成本可再生原料,已被用作微生物生产代谢的原料来生产各种增值化合物[8]。然而以甲醇作为唯一底物也面临许多问题,如甲醇耐受性问题,甲醇浓度低时菌株生长量低下,但甲醇浓度高时会抑制菌株生长甚至会导致菌株死亡[9],并且随着甲基营养菌以甲醇为底物的生长代谢中会产生抑制细胞生长的甲醛,尤其是在高浓度甲醇条件下会有大量的甲醛在细胞内积累而使细菌生长停滞[10]。
DNA错配修复系统(mismatch repair,MMR)是细胞进化上一种高度保守的分子机制,其能够去除DNA复制或者损伤过程中引起的突变,从而维持遗传复制的准确性[11-13]。该系统包含MutS、MutL和MutH蛋白[14-15],错配修复系统MMR的启动先利用MutS二聚体的DNA错配识别功能识别异常碱基对,随后在ATP的协助下与MutL结合成MutS-DNA复合物[16],激活与MutH相关的核酸内切酶,定位在错配处两侧[17],然后招募UvrD解旋酶(DNA解旋酶II)从链断裂到错配的有利方向解开异源双链,DNA聚合酶III填补缺口,由DNA连接酶连接;最后,甲基化酶Dam迅速使子链甲基化完成错配修复[18]。在只有DNA聚合酶的校对能力下,错配依然会发生在DNA复制过程中,其细菌的自发突变频率为10−7;而在DNA错配修复系统的作用下,将保真度提高50−1 000倍,进而保持低突变率1×10−9–1×10−10以保持基因组完整性[19-20]。Kang等对野生型大肠杆菌MG1655和mutS缺失的大肠杆菌在乳酸或甘油培养基上平行进化了800代后进行全基因组测序,结果发现ΔmutS中的突变更快,比野生型高出4倍以上,并且ΔmutS菌株中积累了大量随机突变[21]。Zhang等在工业DNA合成中开发了一种蛋白酶eMutS结合纤维素柱的过程,通过基因工程和蛋白质工程工艺的结合将eMutS的全活性保质期从7 d延长至63 d,而且碱基复制准确度提高了37.6倍,比野生型eMutS更有效、更稳健[22];Hu等研究发现,大肠杆菌中的mutS缺失使菌株产生的突变频率高于mutH的缺失,并且发现甲基化酶dam基因和mutS双缺失比dam或mutS单个缺失菌株的突变频率更高[23];Qiu在动物研究中发现,MutS的同源物MSH2缺乏会增加遗传性非息肉性结肠直肠癌的易感性,MSH2可能在癌症的发生、发展和免疫浸润中起重要作用,MSH2可以成为癌症诊断和预后的潜在生物标志物[24]。
本研究对甲基营养菌(Methylobacterium sp.) MB200的mutS基因进行插入缺失突变,破坏菌株中的错配修复系统,改造该菌株成为高频突变子,在唯一碳源分别为甲醇和甲醛的条件下进行定向诱导突变,以期得到高耐受性甲醇或甲醛的菌株,为甲基营养菌的进一步研究奠定基础并提供具有应用潜力的菌种资源。
1 材料与方法 1.1 菌种与质粒Methylobacterium sp. MB200 (萘啶酮酸抗性,Nmr)、大肠杆菌(Escherichia coli) DH5α、表达载体pCM80 (四环素抗性,Tcr)、帮助质粒pRK2073 (卡那霉素抗性,Kmr)和自杀质粒pK18mod (卡那霉素抗性,Kmr)均为本实验室保存;克隆载体pEASY-T1 Cloning Kit (氨苄青霉素抗性,Ampr),北京全式金生物技术有限公司;限制性内切酶、T4 DNA连接酶和DNA聚合酶,Thermo公司;DNA提取试剂盒、质粒提取试剂盒和DNA纯化试剂盒,Qiagen公司。
1.2 培养基大肠杆菌培养使用LB液体培养基:胰蛋白胨10.0 g,酵母提取物5.0 g,NaCl 10.0 g,加水定容至1.0 L,pH 7.0。菌株MB200基础培养基(MM)[25]:(NH4)2HPO4 3.0 g/L,K2HPO4 2.0 g/L,NaCl 1.0 g/L,MgSO4·7H2O 0.2 g/L,FeSO4·7H2O 0.010 g/L,MnSO4·6H2O 0.005 g/L,维生素B1 10 μg/L,烟酸20 μg/L,泛酸钙20 μg/100 mL,维生素B2 20 μg/L,生物素10 μg/L,对氨基苯甲酸10 μg/L。甲基营养菌在30 ℃的MM基础培养基中生长,并根据需要添加不同浓度的甲醇和甲醛作为唯一碳源,大肠杆菌DH5α在37 ℃的LB培养基中培养。根据需要向培养基中按以下终浓度添加相应的抗生素:氨苄青霉素(Amp) 50 μg/mL,卡那霉素(Km) 25 μg/mL,四环素(Tc) 75 μg/mL,萘啶酮酸(Nm) 50 μg/mL。
1.3 方法 1.3.1 DNA分子操作按照试剂盒说明书进行分离提取染色体DNA、质粒和基因组DNA,依照酶的说明书进行DNA的PCR、酶切、连接、转化,DNA的纯化、回收、酶切、连接、转化等其他DNA操作通过标准程序完成[26]。
1.3.2 构建mutS突变株根据MB200的基因组序列,借助软件Vector NTI设计引物MutS-F和MutS-R,以MB200的基因组DNA为模板PCR扩增目的基因mutS;再以扩增出的基因mutS为模板,以MutSps-F和MutSps-R为引物扩增mutS基因的部分片段mutSps;以Km抗性基因的引物对(Km-F/Km-R)和Tc抗性基因的引物对(Tc-F/ Tc-R) PCR扩增筛选缺失和回补菌株。所有引物序列见表 1。
| Primer name | Sequence (5′→3′) | Usage |
| MutS-F MutS-R |
CATAAGCTTCAGGCGGCGGATACG CGCCTGCAGATCAGCGACGCTCTATA |
Clone mutS gene |
| MutSps-F MutSps-R |
TATAAGCTTACGCTCGCCATCGACGCG TATCTGCAGGGCGATGAAGGCTTCGCC |
Cloning part of the mutS gene |
| Km-F Km-R |
ATGATTGAACAAGATGGATTGCACCGC TCAGAAGAACTCGTCAAGAAGGCGAT |
Validation of mutant strains of mutS gene |
| Tc-F Tc-R |
TTCCTGACGGGCTGTTTCCTTTTGC ATGCCCACCCGTTCCACGTTGTTAT |
Validation of mutS gene complementation |
| 注:下划线为酶切位点 Note: Underline indicate the enzyme recognition site. | ||
PCR反应体系(25 μL):DNA (0.50–1.00 μg/μL) 1 μL、上、下游引物(10‒100 pmol/μL)各1 μL、酶12.5 μL和水9.5 μL。mutS的PCR反应条件:95 ℃ 2 min;95 ℃ 30 s,58 ℃ 150 s,72 ℃ 60 s,30次循环;72 ℃ 5 min。再以mutS基因作为模板,采用引物MutSps-F和MutSps-R进行mutSps的PCR,将得到的PCR产物和pEASY-T1连接命名为pEASY-T1-mutsps。导入大肠杆菌DH5α,在含有Amp的LB平板上进行蓝白斑筛选并提取质粒,用Pst I与Hind III酶切后测序验证。
利用Pst I与Hind III对重组质粒pEASY- T1-mutsps进行双酶切,回收mutSps片段,与同样利用Pst I与Hind III双酶切的pK18mob载体连接,得到重组质粒pK18mod-mutsps,用热激法转化到大肠杆菌DH5α中,命名为pK18mod-mutsps/DH5α。接下来采用三亲本接合方法[27]将受体菌株MB200 (Nm,50 μg/mL,30 ℃)、供体菌株pK18mod-mutsps/DH5α (Km,25 μg/mL,37 ℃)和帮助菌pRK2073/DH5α (Km,25 μg/mL,37 ℃)在相应的抗生素和温度条件下培养到对数期(OD600为0.8−1.0),按照DH5α/ pK18mod-mutsps: MB200:DH5α/pRK2073的菌体湿重比例为1:2:1 (以1 mL的菌体量为1),4 ℃、6 000 r/min离心10 min收集菌体,用生理盐水洗涤3次后将混合菌体转移到含5% LB的MM固体培养基的醋酸纤维薄膜上,在32 ℃的培养箱中培养3−4 d,然后稀释涂布于含有卡那霉素(Km)的MM固体培养基进行筛选、鉴定,并经(Km-F/Km-R)进行PCR扩增检测,获得的重组突变株命名为MB200sTB。
1.4 野生菌株MB200的甲醇和甲醛耐受将野生菌株MB200划线培养,得到的对数期(OD600为0.8−1.0)的种子液按照1%的接种量接种至甲醇浓度分别为4、8、12、16、20 g/L或甲醛浓度分别为0.2、0.4、0.6、0.8、1.0、1.2 g/L的MM培养基中,在30 ℃、200 r/min条件下培养,测量野生菌株的生长曲线及菌体量OD600。
1.5 突变株MB200sTB的定向诱导 1.5.1 突变株MB200sTB的甲醇诱导根据野生菌在甲醇培养基上的生长曲线,从最适甲醇浓度开始进行定向诱导突变。将MB200sTB菌株划线挑单菌落接入指型瓶中,于30 ℃、200 r/min条件下培养,将培养得到的对数期(OD600为0.8−1.0,下同)种子液以5%的接种量接入100 mL的MM培养基中,继续以30 ℃、200 r/min培养36−48 h达到对数期;之后依然以5%的接种量转接到更高甲醇浓度(甲醇浓度增加4 g/L)的MM培养基中,30 ℃、200 r/min培养36−48 h,待生长达到对数期后继续上述操作。当培养到甲醇浓度过高时,由于菌株受到的甲醇抑制程度增大,菌株生长受到影响时,减少甲醇的增加梯度,降为2 g/L。每一个浓度保存一次菌株,当菌株无法生长时,重新从保存的上一个浓度的菌液接种培养,最终经过不断地传代诱导、筛选后可获得耐高浓度甲醇的菌株。同时以野生菌MB200为对照组进行同样的定向诱导。
1.5.2 突变株MB200sTB的甲醛诱导将MB200sTB菌株划线挑单菌落接入指型瓶中,于30 ℃、200 r/min条件下培养,将培养4−5 d的稳定期种子液以5%的接种量接入100 mL甲醛浓度为0.4 g/L的MM培养基中,于30 ℃、200 r/min培养4−5 d达到稳定期,每轮培养依次增加0.05 g/L的甲醛。同样以野生菌MB200为对照组进行同样的定向诱导。
1.6 构建回补菌株MB200sHBc和MB200sHBq 1.6.1 表达载体pCM80-mutS的构建使用Pst I和Hind III进行双酶切克隆载体pEASY-T1-muts,胶回收mutS目的片段,与同样双酶切的pCM80连接,转化至E. coliDH5α,在四环素抗性筛选培养基上筛选获得阳性重组子,测序验证正确后命名为E. coli DH5α/pCM80-muts。
1.6.2 构建菌株MB200sHBc和MB200sHBq参考Faber等[28]的电转化方法,制作菌株MB200sTBc和MB200sTBq感受态,利用电转化将表达载体pCM80-muts转化到菌株MB200sTBc和MB200sTBq中,并在含Tc和Nm的MM固体培养基中筛选,命名为菌株MB200sHBq和MB200sHBc。
1.7 菌株MB200sHBc和MB200sHBq的生长情况分析将菌株MB200、MB200sHBc和MB200sHBq划线接种于MM固体培养基,30 ℃培养72 h后挑单菌落接种至对应的碳源培养液中,30 ℃、200 r/min培养36 h。将MB200、MB200sHBc菌液按照1%的接种量接种到100 mL含甲醇或甲醛(诱导的最高浓度)的MM液体培养基进行培养,每隔8 h检测一次OD600,每个组取3个平行检测菌株的生长情况。
2 结果与分析 2.1 菌株MB200的生长情况 2.1.1 菌株MB200在不同甲醇浓度下的生长情况菌株MB200在甲醇浓度分别为4、8、12、16、20 g/L的MM液体培养基中的生长状况如图 1所示,菌株MB200在8 g/L的甲醇条件下生长最快,菌株的生长量也最大;但随着甲醇浓度的进一步提高,菌株的生长速度变慢且生长量也减少,并且当甲醇浓度增加为16 g/L时菌株基本不生长,所以得到菌株MB200生长的最适甲醇浓度为8 g/L。
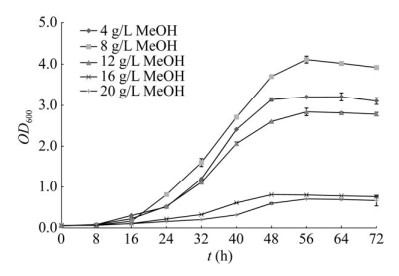
|
| 图 1 野生型菌株MB200在不同甲醇浓度下的生长曲线 Figure 1 Growth curve of wild-type strain MB200 at different methanol concentrations. |
|
|
菌株MB200在甲醛浓度分别为0.2、0.4、0.6、0.8、1.0、1.2 g/L的MM液体培养基中的生长状况如图 2所示,菌株MB200最高可以在0.4 g/L的甲醛浓度条件下生长,而且在甲醛浓度为0.6 g/L时菌株就无法生长了,证明菌株MB200的最高甲醛耐受性为0.4 g/L,生长的OD600最高只有0.4左右,表明在甲醛存在的条件下菌株MB200的生长状况差,以甲醛作为唯一碳源时生长代谢比较困难。
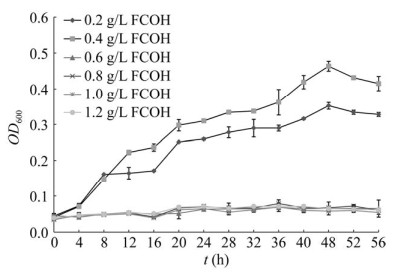
|
| 图 2 野生型菌株MB200在不同甲醛浓度下的生长曲线 Figure 2 The growth curve of wild-type strain MB200 at different concentrations of formaldehyde. |
|
|
将mutSps与载体pK18mob酶切连接后整体转化至E. coli DH5α中,获得pK18mod-mutsps/ DH5α菌株,提取质粒进行酶切验证(图 3A)。利用三亲本结合方法,将质粒pK18mod-mutsps导入菌株MB200中,经抗生素抗性筛选,并对Km基因进行PCR扩增验证(图 3B),获得mutS基因缺失的突变株,命名为MB200sTB。
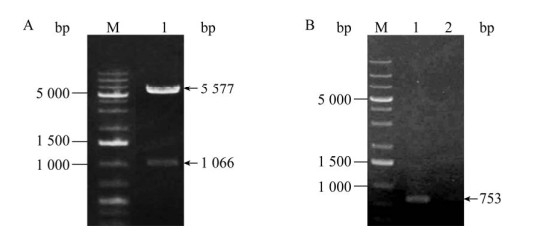
|
| 图 3 重组质粒pK18mob-mutsps酶切验证(A)和MB200sTB突变体的PCR鉴定(B) Figure 3 Recombinant plasmid pK18mob-mutsps restriction digestion (A) and PCR identification results of MB200sTB mutant (B). M:GeneRuler 1 kb Plus DNA Ladder。A:1:为重组质粒pK18mob-mutsps的Pst I与Hind III双酶切。B:1:突变菌株MB200sTB中Km基因的PCR扩增;2:MB200的Km基因扩增 M: GeneRuler 1 kb Plus DNA Ladder. A: 1: Recombinant plasmid pK18mob-mutsps digested by Pst I and Hind III. B: 1: PCR products of Km resistance gene from mutant strain MB200sTB; 2: PCR products of Km resistance gene from wild strain MB200. |
|
|
突变菌株MB200sTB经过3个月的定向诱导,在以甲醇为碳源时,其甲醇耐受性提升到44 g/L,提高了近6倍,而野生菌MB200的最高甲醇耐受性只提升到24 g/L;以甲醛为碳源时,突变菌株MB200sTB诱导后的耐受性并不高,经过3个月的诱导,该菌株最高可以耐受0.45 g/L的甲醛浓度,但生长量比野生型有所增加,野生菌MB200的甲醛耐受性无变化。
2.4 回补菌株MB200sHBc和MB200sHBq的构建将mutS基因片段与表达载体pCM80连接,并转化至大肠杆菌E. coli DH5α感受态细胞中,提取质粒酶切验证(图 4A)后,通过三亲本结合将pCM80-muts导入相应的耐受性菌株MB200sTB中,将耐受高浓度甲醇和甲醛的回补菌株分别命名为MB200sHBc和MB200sHBq,并通过Tc基因的PCR扩增进行验证(图 4B)。
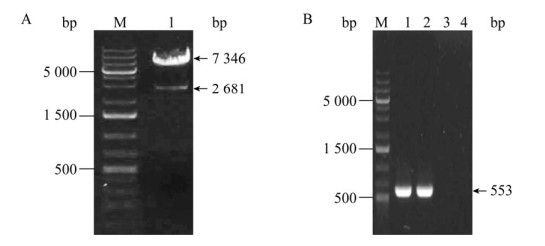
|
| 图 4 表达载体pCM80-muts酶切验证(A)和MB200sHBc和MB200sHBq回补菌株中Tc基因的PCR鉴定(B) Figure 4 The expression vector pCM80-muts restriction digestion verification (A) and the PCR identification results of MB200sHBc and MB200sHBq replenishing strains (B). M:GeneRuler 1 kb Plus DNA Ladder。A:1:重组质粒pCM80-muts的Pst I与Hind III酶切。B:1:回补菌株MB200sHBc中含有Tc抗性基因;2:回补菌株MB200sHBq中含有Tc抗性基因;3、4:MB200中未发现Tc抗性基因 M: GeneRuler 1 kb Plus DNA Ladder. A: 1: Restriction digestion verification of recombinant plasmid pCM80-muts digested by Pst I and Hind III. B: 1: Tc resistance gene was contained in the complementing strain MB200sHBc; 2: Tc resistance gene was contained in the complementing strain MB200sHBq; 3, 4: MB200 was not found Tc resistance gene. |
|
|
将菌株MB200sHBc接种到甲醇浓度逐步提高的培养基中进行诱导培养,经过定向诱导得到了最高甲醇耐受性为44 g/L的菌株,继续传代培养8代验证其稳定性并保存,对生长状况优异的菌株MB200sHBc,在44 g/L的甲醇条件下进行生长曲线测定,由图 5可以明显看出,经过甲醇定向诱导得到的菌株MB200sHBc在44 g/L的条件下生长良好,结合图 1可以看出,菌株MB200sHBc在44 g/L条件下的最高生长量可以达到野生菌株MB200在甲醇浓度为8 g/L的水平,最高的OD600可以达到4.2左右,MB200sHBc的甲醇耐受性比野生型提高5.5倍。在甲醇低浓度的条件下野生型菌株MB200比定向诱导的菌株MB200sHBc先达到对数期,MB200在40 h左右达到对数期,MB200sHBc推迟了36 h左右达到对数期,说明高浓度的甲醇对菌株生长起到了一定的抑制作用。
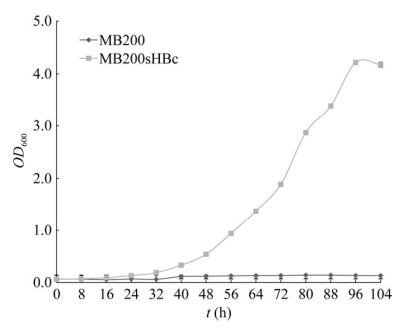
|
| 图 5 菌株MB200sHBc与MB200在44 g/L甲醇条件下的生长曲线 Figure 5 Growth curve of strain MB200sHBc and MB200 under 44 g/L methanol condition. |
|
|
将菌株MB200sHBq接种到逐步提高甲醛浓度的培养基中,发现菌株最高甲醛耐受性是0.45 g/L,对其传代培养8代验证其稳定性并保存。菌株MB200sHBq在0.45 g/L甲醛条件下的生长情况如图 6所示,可以看出经过甲醛定向诱导,得到了最高可耐受0.45 g/L甲醛的菌株MB200sHBq,并且通过其生长曲线可以看出菌株的生长量得到了较大提高,达到了野生型菌株MB200的1.69倍,说明菌株的甲醛耐受性得到了比较好的提升。然而从生长量来看,以甲醛为单碳源的条件下诱导效果并不理想,甲醛对菌株生长具有较大的抑制性。
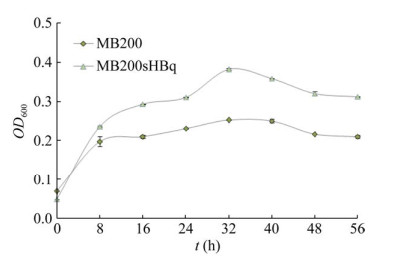
|
| 图 6 菌株MB200sHBq与MB200在0.45 g/L的甲醛条件下的生长曲线 Figure 6 Growth curves of strains MB200sHBq and MB200 under 0.45 g/L formaldehyde conditions. |
|
|
甲基营养菌能以甲基化合物为唯一碳源进行生产和发酵培养,生产许多有价值的代谢产物。因此,对甲基营养菌进行改进和改造使其成为具有商业价值的工程菌株具有重要意义。然而随着研究的深入出现了比较明显的瓶颈,比如在甲醇作为唯一底物时,甲醇浓度低时不能满足菌株的生长需要,而当甲醇浓度高时菌株又受甲醇毒性影响,生长受限;并且菌株以甲醇为底物的生长代谢过程中会产生毒性极强的甲醛,对菌株起到毒害作用,导致菌株无法正常生长而死亡。针对以上问题,除了从自然界中筛选寻找高耐受性的甲基营养菌菌株,也可从实验室对现有菌株进行分子改造和定向诱导。国内外已有通过各种方式提高甲基营养菌的甲醇、甲醛耐受能力的报道,结果如表 2所示。
| Strain | Method | Trait change | References |
| Methylobacterium extorquens AM1 | Plasma mutagenesis combined with laboratory self-evolution breeding | Up to 40 g/L methanol tolerance, significantly improved mevalonate MEV production and cell growth | [29] |
| Methylobacterium extorquens TK 0001 and AM1 | Laboratory continuous culture technology | The highest methanol tolerance is 80 g/L, and the production of lactic acid is significantly improved | [30] |
| Methylobacterium sp. MB200 | Mutation and laboratory-directed mutagenesis of the mutL gene | The highest methanol tolerance is 56 g/L, and the yield of L-serine is improved | [31] |
| Methylobacterium extorquens AM1 | Laboratory basic training | The minimum tolerance level of cells in formaldehyde tolerance is between 0−0.24 g/L | [32] |
| Methylobacterium sp. XJLW | Domestication in the laboratory | The highest formaldehyde tolerance of the strain can be increased to 1.2 g/L | [33] |
| Industrial synthetic methylotrophic host (Escherichia coli) | Adaptive lab evolution | Growth rate and biomass of cloned strains increased by > 50% in the presence of high methanol concentrations | [34] |
本研究以MB200菌株为出发菌株,通过插入缺失错配修复基因mutS,得到了高突变子MB200sTB菌株,在甲醇与甲醛的条件下定向诱导。与野生型相比,突变体的甲醇耐受性从8 g/L的浓度提升到了44 g/L,而且最高菌株生长量不受影响。突变体甲醛的定向诱导结果与野生型相比其耐受性和生长量都有所提高,但与甲醇为碳源的诱导效果相比差距较大,推测是因为甲醛对菌株的毒性较大,在应用上可通过结合其他碳源,如在以丁二酸为碳源的情况下补加甲醛的办法实现甲醛的生物转化。下一步研究将从转录组和代谢组的角度分析菌株在高浓度甲醇条件下对甲醇的代谢情况,包括代谢途径的分析,为甲基营养菌的进一步应用奠定基础。
| [1] |
Amaratunga K, Goodwin PM, O'Connor CD, Anthony C. The methanol oxidation genes mxaFJGIR(S)ACKLD in Methylobacterium extorquens[J]. FEMS Microbiology Letters, 1997, 146(1): 31-38. DOI:10.1111/j.1574-6968.1997.tb10167.x |
| [2] |
Arps PJ, Fulton GF, Minnich EC, Lidstrom ME. Genetics of serine pathway enzymes in Methylobacterium extorquens AM1:phosphoenolpyruvate carboxylase and malyl coenzyme A lyase[J]. Journal of Bacteriology, 1993, 175(12): 3776-3783. DOI:10.1128/jb.175.12.3776-3783.1993 |
| [3] |
Zhang WM, Song M, Yang Q, Dai ZX, Zhang SJ, Xin FX, Dong WL, Ma JF, Jiang M. Current advance in bioconversion of methanol to chemicals[J]. Biotechnology for Biofuels, 2018, 11: 260. DOI:10.1186/s13068-018-1265-y |
| [4] |
Orita I, Nishikawa K, Nakamura S, Fukui T. Biosynthesis of polyhydroxyalkanoate copolymers from methanol by Methylobacterium extorquens AM1 and the engineered strains under cobalt-deficient conditions[J]. Applied Microbiology & Biotechnology, 2014, 98(8): 3715-3725. |
| [5] |
Zhu WL, Cui JY, Cui LY, Liang WF, Yang S, Zhang C, Xing XH. Bioconversion of methanol to value-added mevalonate by engineered Methylobacterium extorquens AM1 containing an optimized mevalonate pathway[J]. Applied Microbiology and Biotechnology, 2016, 100(5): 2171-2182. DOI:10.1007/s00253-015-7078-z |
| [6] |
Sonntag F, Müller JEN, Kiefer P, Vorholt JA, Schrader J, Buchhaupt M. High-level production of ethylmalonyl-CoA pathway-derived dicarboxylic acids by Methylobacterium extorquens under cobalt-deficient conditions and by polyhydroxybutyrate negative strains[J]. Applied Microbiology and Biotechnology, 2015, 99(8): 3407-3419. DOI:10.1007/s00253-015-6418-3 |
| [7] |
Von Borzyskowski LS, Sonntag F, Pöschel L, Vorholt JA, Schrader J, Erb TJ, Buchhaupt M. Replacing the ethylmalonyl-CoA pathway with the glyoxylate shunt provides metabolic flexibility in the central carbon metabolism of Methylobacterium extorquens AM1[J]. ACS Synthetic Biology, 2018, 7(1): 86-97. DOI:10.1021/acssynbio.7b00229 |
| [8] |
Schrader J, Schilling M, Holtmann D, Sell D, Filho MV, Marx A, Vorholt JA. Methanol-based industrial biotechnology: current status and future perspectives of methylotrophic bacteria[J]. Trends in Biotechnology, 2009, 27(2): 107-115. DOI:10.1016/j.tibtech.2008.10.009 |
| [9] |
Delaney NF, Kaczmarek ME, Ward LM, Swanson PK, Lee MC, Marx CJ. Development of an optimized medium, strain and high-throughput culturing methods for Methylobacterium extorquens[J]. PLoS One, 2013, 8(4): e62957. DOI:10.1371/journal.pone.0062957 |
| [10] |
Pluschkell SB, Flickinger MC. Dissimilation of [(13)C]methanol by continuous cultures of Bacillus methanolicus MGA3 at 50 degrees C studied by (13)C NMR and isotope-ratio mass spectrometry[J]. Microbiology, 2002, 148(Pt 10): 3223-3233. |
| [11] |
Schofield MJ, Nayak S, Scott TH, Du CW, Hsieh P. Interaction of Escherichia coli MutS and MutL at a DNA mismatch[J]. Journal of Biological Chemistry, 2001, 276(30): 28291-28299. DOI:10.1074/jbc.M103148200 |
| [12] |
Marinus MG, Poteete A, Arraj JA. Correlation of DNA adenine methylase activity with spontaneous mutability in Escherichia coli K-12[J]. Gene, 1984, 28(1): 123-125. DOI:10.1016/0378-1119(84)90095-7 |
| [13] |
Lichten M, Goyon C, Schultes NP, Treco D, Szostak JW, Haber JE, Nicolas A. Detection of heteroduplex DNA molecules among the products of Saccharomyces cerevisiae meiosis[J]. PNAS, 1990, 87(19): 7653-7657. DOI:10.1073/pnas.87.19.7653 |
| [14] |
Marti TM, Kunz C, Fleck O. DNA mismatch repair and mutation avoidance pathways[J]. Journal of Cellular Physiology, 2002, 191(1): 28-41. DOI:10.1002/jcp.10077 |
| [15] |
Štambuk S, Radman M. Mechanism and control of interspecies recombination in Escherichia coli. I. mismatch repair, methylation, recombination and replication functions[J]. Genetics, 1998, 150(2): 533-542. DOI:10.1093/genetics/150.2.533 |
| [16] |
Robertson AB, Pattishall SR, Gibbons EA, Matson SW. MutL-catalyzed ATP hydrolysis is required at a post-UvrD loading step in methyl-directed mismatch repair[J]. Journal of Biological Chemistry, 2006, 281(29): 19949-19959. DOI:10.1074/jbc.M601604200 |
| [17] |
Grilley M, Griffith J, Modrich P. Bidirectional excision in methyl-directed mismatch repair[J]. Journal of Biological Chemistry, 1993, 268(16): 11830-11837. DOI:10.1016/S0021-9258(19)50275-7 |
| [18] |
Burdett V, Baitinger C, Viswanathan M, Lovett ST, Modrich P. In vivo requirement for RecJ, ExoVⅡ, ExoI, and ExoX in methyl-directed mismatch repair[J]. PNAS, 2001, 98(12): 6765-6770. DOI:10.1073/pnas.121183298 |
| [19] |
Larrea AA, Lujan SA, Kunkel TA. SnapShot: DNA mismatch repair[J]. Cell, 2010, 141(4): 730-730. DOI:10.1016/j.cell.2010.05.002 |
| [20] |
Iyer RR, Pluciennik A, Burdett V, Modrich PL. DNA mismatch repair: functions and mechanisms[J]. Chemical Reviews, 2006, 106(2): 302-323. DOI:10.1021/cr0404794 |
| [21] |
Kang M, Kim K, Choe D, Cho S, Kim SC, Palsson B, Cho BK. Inactivation of a mismatch-repair system diversifies genotypic landscape of Escherichia coli during adaptive laboratory evolution[J]. Frontiers in Microbiology, 2019, 10: 1845. DOI:10.3389/fmicb.2019.01845 |
| [22] |
Zhang J, Wang YF, Chai BH, Wang JC, Li LL, Liu M, Zhao G, Yao LS, Gao XL, Yin YF, et al. Efficient and low-cost error removal in DNA synthesis by a high-durability MutS[J]. ACS Synthetic Biology, 2020, 9(4): 940-952. DOI:10.1021/acssynbio.0c00079 |
| [23] |
Hu CK, Zhao YQ, Sun HY, Yang YX. Synergism of Dam, MutH, and MutS in methylation-directed mismatch repair in Escherichia coli[J]. Mutation Research, 2017, 795: 31-33. DOI:10.1016/j.mrfmmm.2016.12.002 |
| [24] |
Qiu WL, Ding K, Liao LS, Ling YC, Luo XQ, Wang JL. Analysis of the expression and prognostic value of MSH2 in Pan-cancer based on bioinformatics[J]. BioMed Research International, 2021, 2021: 9485273. |
| [25] |
Marx CJ, Bringel F, Chistoserdova L, Moulin L, Farhan Ul Haque M, Fleischman DE, Gruffaz C, Jourand P, Knief C, Lee MC, et al. Complete genome sequences of six strains of the genus Methylobacterium[J]. Journal of Bacteriology, 2012, 194(17): 4746-4748. DOI:10.1128/JB.01009-12 |
| [26] |
Izumi Y, Yoshida T, Miyazaki SS, Mitsunaga T, Ohshiro T, Shimao M, Miyata A, Tanabe T. L-serine production by a methylotroph and its related enzymes[J]. Applied Microbiology and Biotechnology, 1993, 39(4/5): 427-432. |
| [27] |
Turner P, Barber C, Daniels M. Behaviour of the transposons Tn5 and Tn7 in Xanthomonas campestris pv. campestris[J]. Molecular and General Genetics MGG, 1984, 195(1/2): 101-107. |
| [28] |
Faber KN, Haima P, Harder W, Veenhuis M, Ab G. Highly-efficient electrotransformation of the yeast Hansenula polymorpha[J]. Current Genetics, 1994, 25(4): 305-310. DOI:10.1007/BF00351482 |
| [29] |
Cui LY, Wang SS, Guan CG, Liang WF, Xue ZL, Zhang C, Xing XH. Breeding of methanol-tolerant Methylobacterium extorquens AM1 by atmospheric and room temperature plasma mutagenesis combined with adaptive laboratory evolution[J]. Biotechnology Journal, 2018, 13(6): e1700679. |
| [30] |
Belkhelfa S, Roche D, Dubois I, Berger A, Delmas VA, Cattolico L, Perret A, Labadie K, Perdereau AC, Darii E, et al. Continuous culture adaptation of Methylobacterium extorquens AM1 and TK 0001 to very high methanol concentrations[J]. Frontiers in Microbiology, 2019, 10: 1313. DOI:10.3389/fmicb.2019.01313 |
| [31] |
Wen DM, Huang LD, Yang QS, Shen PH. Mutation of mutL gene in methylotrophic bacteria MB200 and screening of highly methanol-tolerant strains[J]. Industrial Microbiology, Iyer RR, 2020, 50(4): 15-20. (in Chinese) 闻东明, 黄罗冬, 杨青山, 申佩弘. 甲基营养菌MB200中mutL基因的突变及高耐甲醇菌株的筛选[J]. 工业微生物, 2020, 50(4): 15-20. |
| [32] |
Lee JA, Riazi S, Nemati S, Bazurto JV, Vasdekis AE, Ridenhour BJ, Remien CH, Marx CJ. Microbial phenotypic heterogeneity in response to a metabolic toxin: continuous, dynamically shifting distribution of formaldehyde tolerance in Methylobacterium extorquens populations[J]. PLoS Genetics, 2019, 15(11): e1008458. DOI:10.1371/journal.pgen.1008458 |
| [33] |
Qiu LQ, Chen WW, Zhong L, Wu WX, Wu SJ, Chen JM, Zhang FM, Zhong WH. Formaldehyde biodegradation by immobilized Methylobacterium sp. XJLW cells in a three-phase fluidized bed reactor[J]. Bioprocess and Biosystems Engineering, 2014, 37(7): 1377-1384. DOI:10.1007/s00449-013-1110-4 |
| [34] |
Bennett RK, Gregory GJ, Gonzalez JE, Har JRG, Antoniewicz MR, Papoutsakis ET. Improving the methanol tolerance of an Escherichia coli methylotroph via adaptive laboratory evolution enhances synthetic methanol utilization[J]. Frontiers in Microbiology, 2021, 12: 638426. DOI:10.3389/fmicb.2021.638426 |
 2022, Vol. 49
2022, Vol. 49




