扩展功能
文章信息
- 张明阳, 任彪, 贾燕涛
- ZHANG Mingyang, REN Biao, JIA Yantao
- 细菌与噬菌体相互抵抗机制研究进展
- Research progress on the mutual resistance mechanisms between bacteria and bacteriophages
- 微生物学通报, 2021, 48(9): 3293-3304
- Microbiology China, 2021, 48(9): 3293-3304
- DOI: 10.13344/j.microbiol.china.210495
-
文章历史
- 收稿日期: 2021-06-01
- 接受日期: 2021-07-30
- 网络首发日期: 2021-08-12
噬菌体是地球上最丰富的生物之一,作为细菌的“克星”其能够特异性识别和裂解宿主细菌。在细菌与噬菌体长期共同进化过程中,寄生-宿主的对抗关系导致二者都不断完善自身的策略以维持个体生存。噬菌体自1915年被发现以来,一直被认为是应对细菌感染的重要制剂,尤其是近年来细菌对抗生素耐药性问题的突显,开发替代抗生素的抗菌剂已引起越来越多研究者的关注。对细菌-噬菌体相互作用分子机制的研究,有助于我们从不同角度理解噬菌体如何成功侵染细菌,以期为噬菌体作为抗菌剂的应用提供参考。
1 吸附抑制 1.1 噬菌体的生活周期噬菌体是感染细菌、真菌、放线菌或螺旋体的病毒,其结构简单,主要由蛋白外壳和遗传物质核酸组成。从噬菌体进入细菌开始到引起细菌裂解并释放出子代噬菌体为止,为一个增殖周期,一般为15-20 min。根据是否进入裂解期,可将噬菌体分为烈性噬菌体和温和噬菌体,其中烈性噬菌体应用价值更高。烈性噬菌体的繁殖周期一般分为吸附、入侵、增殖、组装和裂解5个阶段。噬菌体通过吸附蛋白与宿主细菌表面的受体特异性结合,将其遗传物质注入宿主细胞,并在宿主细胞内进行核酸复制、蛋白质合成和后代噬菌体的组装,实现增殖。噬菌体与细菌的相互抵抗发生在噬菌体繁殖周期的不同阶段。
1.2 细菌对噬菌体的吸附抑制吸附是噬菌体与细菌表面受体结合的过程,是噬菌体感染的最初环节,也是决定噬菌体感染成功与否的关键环节。大多数噬菌体通过尾部结构吸附细菌,分为可逆吸附和不可逆吸附。噬菌体Bp7的受体结合蛋白gp38与大肠杆菌(Escherichia coli) K-12的外膜蛋白LamB和OmpC结合,这一过程是可逆的;而噬菌体Bp7还可以与细菌多糖内核的HepI不可逆结合[1]。肺炎克雷伯菌(Klebsiella pneumoniae)的噬菌体NJS1通过其侧纤维蛋白(Side Fiber Protein,SFP)可逆地附着到细菌O抗原上后,噬菌体NJS1的中心纤维蛋白(Central Fiber Protein,CFP)可与细菌外膜蛋白FepA发生不可逆吸附,从而触发噬菌体DNA注入宿主细胞[2]。
吸附抑制是细菌抵御噬菌体感染的第一步,细菌阻断噬菌体吸附的机制可分为:噬菌体受体的突变或修饰、产生胞外基质来阻断受体、受体竞争性抑制剂等。细菌基因突变导致其细胞表面受体的数量、结构改变,或者表达其他蛋白对受体进行修饰,从而减少或阻断噬菌体的吸附。例如,铜绿假单胞菌(Pseudomonas aeruginosa)的噬菌体OMKO1利用多药外排系统MexAB和MexXY的外膜孔蛋白M (OprM)作为受体结合位点,细菌OprM的缺失导致其对噬菌体OMKO1产生抗性[3]。粪肠球菌(Enterococcus faecalis)多糖抗原(Epa)对于噬菌体的吸附至关重要,可作为噬菌体受体,粪肠球菌通过突变Epa阻断噬菌体感染[4]。参与黄色短杆菌(Brevibacterium flavum)噬菌体BFK20受体合成的MFS蛋白和糖基转移酶基因被破坏后,噬菌体BFK20的吸附率显著降低[5]。细菌荚膜或胞外聚合物不仅可以提高细菌在恶劣环境中的存活率,还可以作为细菌表面的受体的物理屏障,阻断噬菌体的吸附[6]。假单胞菌(Pseudomonas)、棕色固氮菌(Azotobacter vinelandii)可以产生海藻酸盐和透明质酸等胞外多糖(Exopolysaccharides,EPSs),这些多糖可以与噬菌体竞争性结合,从而保护细菌免受噬菌体侵染[7]。益生菌大肠杆菌菌株EcN K5的多糖荚膜已经被证实可以保护其免受T4噬菌体感染[8]。肺炎克雷伯菌(Klebsiella Pneumoniae)也能产生胞外多糖来隐藏其脂多糖受体,使FC3-10噬菌体不能吸附在细菌表面[9]。在某些情况下细菌产生小分子多肽来竞争噬菌体受体的活性位点,并干扰噬菌体对细菌的吸附。如在营养缺陷条件下,大肠杆菌合成一种含有21个氨基酸的抗菌肽微霉素J25,竞争性结合噬菌体受体——铁转运蛋白FhuA,从而阻断噬菌体的吸附作用[10]。除此之外,细菌还可以分泌外膜囊泡充当诱饵来“欺骗”噬菌体,如大肠杆菌被T4噬菌体感染后,产生带有噬菌体结合受体的外膜囊泡,造成细菌被噬菌体感染的假象,从而阻止噬菌体对细菌的进一步吸附[11]。
1.3 噬菌体在吸附阶段对抗细菌的策略噬菌体在吸附阶段对抗细菌的策略主要有3种:识别新受体、降解受体掩蔽、不同受体的随机识别。
当细菌表面特异性受体结构改变时,噬菌体可以通过改变自身受体结合蛋白的结构来吸附新受体。如λ噬菌体的受体结合蛋白J能与宿主表面受体LamB结合,当lamB基因的表达受到抑制时,噬菌体通过改变蛋白J的末端结构,并通过结合新的受体蛋白OmpF来完成后续的感染过程[12]。此外,噬菌体还可以通过自身突变来识别细菌表面结构发生改变的新受体。如当大肠杆菌脂多糖(Lipopolysaccharide,LPS)合成途径中的基因突变后,使脂多糖受体的多糖链更短,T7噬菌体无法识别新受体,而基因突变的T7-Re噬菌体可以识别正常与突变的2种LPS受体,从而可以侵染野生型与LPS突变后的大肠杆菌,而且毒力更强[13]。
当细菌表面受体被修饰掩盖时,噬菌体可以通过对其进行降解,使受体重新暴露。如噬菌体Sb-1可以降解胞外多糖并靶向金黄色葡萄球菌(Staphylococcus aureus),促进噬菌体吸附到细菌表面[14]。鲍曼不动杆菌(Acinetobacter baumannii)噬菌体IME200编码的荚膜解聚酶Dpo48可以降解细菌表面的荚膜多糖,有利于噬菌体对细菌的吸附[15]。噬菌体IME205的2种荚膜解聚酶(Dpo42和Dpo43)可以降解肺炎克雷伯菌(Klebsiella pneumoniae)表面多糖荚膜以降低细菌毒力并使其暴露于宿主免疫攻击[16]。
对于细菌表面受体的阶段性变化,一些噬菌体可以通过编码不同的受体结合蛋白感染处于不同状态的宿主。如一些博德特氏菌(Bordetella pertussis)具有双组分系统——BvgAS。在Bvg+阶段以毒力相关的黏附素和毒素的表达为特征,BvgAS活性高,细菌的噬菌体受体黏附素自转运体(Pertectin,PRN)仅在该阶段表达[17]。在Bvg-阶段,BvgAS不活跃,毒力基因和PRN受体基因不表达;噬菌体BPP-1能够识别细菌的PRN受体,在Bvg+期的细胞对噬菌体BPP-1感染的敏感性是Bvg-期细胞的百万倍;尽管Bvg-阶段博德特氏菌中不表达噬菌体受体PRN,但BPP-1噬菌体能够通过修饰自身受体结合蛋白而感染宿主细菌;博德特氏菌噬菌体基因mtd编码主要趋向性决定蛋白,具有宿主识别的作用;而一些博德特氏菌噬菌体利用差异性反转录因子介导的模板依赖逆转录作用,在mtd基因的可变区引入核苷酸替换,引起主要趋向性决定蛋白的末端变异,使其能够与细菌表面其他受体结合,逆转因为细菌表面受体变化而引起的吸附抑制作用[18]。
1.4 噬菌体的分离与吸附效率检测噬菌体广泛存在于海水、生活污水、土壤、动物或人类排泄物等环境中,可以通过离心去除样品中的杂质,经滤膜过滤后获得无菌样品原液。将过滤后的液体与目标菌液按一定比例混合,双层平板法培养并观察是否有噬菌斑出现[19]。当水样中噬菌体检出效率较低时,可用氯化钠或聚乙二醇对样品中的噬菌体颗粒进行富集沉淀,以提高噬菌体的分离效率[20]。细菌对噬菌体的吸附抑制可以通过比较噬菌体对野生型菌株与抗性菌株的吸附效率来判断。将细菌与噬菌体混合静置后,用双层琼脂平板法测定上清液中残留噬菌体的滴度。噬菌体吸附效率=[(噬菌体初始滴度-上清液中的残留滴度)/初始滴度]×100%,抗性菌株对噬菌体的吸附效率较野生型菌株低[21]。
2 阻止噬菌体DNA进入 2.1 超感染排斥系统当噬菌体吸附到细菌表面,其尾部释放水解蛋白局部降解细胞膜肽聚糖层,将DNA注入到宿主细胞[22-23]。遗传物质穿入阻滞是细菌抵抗噬菌体的第二道防线,通过阻止噬菌体DNA进入细胞内达到防御噬菌体的目的。超感染排斥(Superinfection Exclusion,Sie)系统是细菌阻止噬菌体DNA进入细胞的主要机制。Sie系统是在溶源性噬菌体中发现的,可以阻止特异噬菌体的再次侵染。如Coliphage T4噬菌体可编码由Imm以及Sp蛋白组成的Sie系统,可以阻止其他T偶数噬菌体的侵染。在细菌细胞内Imm通过改变注射位点的构象直接阻断噬菌体DNA进入[24];而Sp蛋白抑制噬菌体编码的T4溶菌酶活性,阻止肽聚糖降解,将噬菌体DNA限制在细胞膜外膜与肽聚糖层之间,抑制DNA进入细胞质,防止其他噬菌体感染[25]。Sie系统在几种大肠杆菌中均有发现[26]。在革兰氏阳性细菌乳酸乳球菌(Lactococcus lactis)的p335型温和噬菌体的膜蛋白Sie2009,也能够排斥936型噬菌体侵染[27]。嗜热链球菌(Streptococcus thermophilus)的温和噬菌体TP-J34编码一种脂蛋白Ltp,对于阻止DNA进入细胞至关重要[28]。Ltp直接与噬菌体尾管中的TMP蛋白相互作用,阻断噬菌体DNA进入细菌,从而抑制DNA注射[28]。
2.2 超感染排斥系统抵抗噬菌体的检测许多温和噬菌体利用超感染排斥系统阻止其他噬菌体的侵染,使细菌保持溶源状态。通常用紫外线照射或丝裂霉素C对溶源菌进行处理,使噬菌体颗粒释放出来[29]。许多原噬菌体存在超感染排斥相关基因,以乳杆菌为例,其编码的Sie基因能够抑制外源噬菌体的侵染。携带Sie基因重组质粒的宿主菌能够产生噬菌体抗性,无噬菌斑出现,同时菌株正常生长,表明该基因参与噬菌体抵抗过程[30]。为确定菌株的噬菌体抗性是由于噬菌体感染早期(噬菌体吸附或注射)还是后期(噬菌体DNA复制期间或者之后)受到抑制,可检测Sie基因是否影响噬菌体吸附效率,并利用Southern印迹杂交检测Sie基因表达菌株是否抑制外源噬菌体DNA的复制,而质粒转导实验可进一步检测该基因是否影响噬菌体DNA进入细菌细胞。若噬菌体吸附效率与对照组差异不大,同时质粒转导效率低且噬菌体DNA未进行复制,则细菌的噬菌体抗性是由于噬菌体DNA进入细菌过程受到抑制导致的[31]。
3 切割噬菌体基因组噬菌体将自身基因组注射到宿主细菌细胞内,在宿主细胞中复制、翻译并组装新的噬菌体后代。细菌也进化出限制噬菌体DNA的策略,目前研究较多的是限制-修饰系统(Restriction- Modification Systems,RM Systems)、CRISPR-Cas适应性免疫系统等。
3.1 细菌限制-修饰系统1962年,Arber等首次利用大肠杆菌λ噬菌体解释了RM Systems的作用原理,表明噬菌体DNA决定宿主细菌的识别特异性[31],目前发现,大约90%的细菌基因组中都存在这种限制-修饰系统[32]。该系统由2种酶组成,分别为甲基转移酶(Methyltransferase,MTase)和限制性内切酶(Restriction Endonuclease,REase)。其中MTase可以对细菌基因组中特定的DNA序列进行甲基化修饰,以区分自身DNA与外源入侵DNA;REase则特异性识别未被甲基化的外源DNA,并对其磷酸二酯键进行切割。经典限制-修饰系统根据其亚基的组成、识别的系列位点以及切割位置等分为I-IV这4种类型[33]。
与经典的限制-修饰系统中的碱基修饰不同,细菌还存在一种DNA硫代磷酸酯(Phosphorothioate,PT)修饰,这种修饰分别由DndABCDE或SspABCD完成将DNA双链或单链中的氧取代为硫。在很多细菌中DndABCDE以序列选择性和RP立体定向方式催化硫代反应,硫代磷酸酯修饰的DNA避免被核酸酶降解。催化单链DNA硫代磷酸酯修饰的SspABCD与SspE结合抵御噬菌体的侵染,其中SspE具有NTPase活性,SspE通过使噬菌体DNA复制时产生切口损伤抑制噬菌体的繁殖[34]。然而,SspABCD-SspFGH系统的作用模式与SspE不同[35],SspFGH破坏非PT修饰的DNA,并通过抑制噬菌体DNA复制发挥抗噬菌体功能。尽管SspFGH与SspE的防御机制不同,但能够同时与SspABCD组合,大大增强了对噬菌体的抵抗作用[35]。
除此之外,天蓝色链霉菌(Streptomyces coelicolor)中存在一种类似限制-修饰系统的噬菌体生长限制系统(Phage Growth Limitation System,Pgl),该系统由4个核心基因pglW、pglX、pglY和pglZ组成,其中pglX编码一种MTase,pglZ编码一种REase,修饰并切割噬菌体基因组DNA[36]。噬菌体在侵染具有Pgl系统的细菌后,后代噬菌体DNA被甲基化修饰。当噬菌体再次侵染细菌时,会被细菌的Pgl系统识别为外源DNA,并对其进行切割。此外一种类似限制-修饰系统的防御岛(Defence Island Associated with Restriction-Modification,DISARM)广泛分布于整个微生物界中,主要包含3个核心基因(drmA、drmB和drmC)、甲基转移酶基因(drmMI或drmMII)和一个附加基因(drmD或drmE),DISARM能够抑制噬菌体复制[37]。枯草芽孢杆菌(Bacillus subtilis)与大肠杆菌中还有一种类似Pgl的噬菌体排斥系统BREX (Bacteriophage Exclusion)。BREX系统与限制-修饰系统相似,除了pglX和pglZ外,还包括brxA/B/C/L噬菌体排斥系统,BREX也是在噬菌体DNA注射到宿主细胞后发挥作用,可以抑制噬菌体DNA的复制,阻止噬菌体DNA整合到宿主染色体上[38]。
3.2 噬菌体逃避RM系统的策略噬菌体也进化出了一系列防御机制逃避宿主的限制-修饰。例如噬菌体DNA在进入细菌细胞后,可以利用宿主的MTase对自身进行甲基化修饰,以逃避REase的切割,这种机制只能暂时躲避宿主的防御,不能遗传给后代。噬菌体也可以将REase识别位点突变,或者对其进行糖基化、羟甲基化等修饰,使这些位点不能被REase识别。大肠杆菌P1噬菌体利用抗限制系统(Defence Against Restriction System,Dar System)阻止自身基因组的降解[39]。另外,噬菌体还可以抑制宿主限制性内切酶REase活性,逃避限制-修饰系统的切割[40]。
3.3 限制-修饰系统抑制噬菌体的检测目前通常采用次级感染的方法,筛选在自然状态下产生的噬菌体抗性菌株[41]。将野生型菌株与噬菌体按照一定比例接种到新鲜液体培养基中,培养至菌体完全裂解,之后继续培养48 h,将裂解液涂布到固体培养基上培养过夜,获得具有噬菌体抗性候选菌株,候选菌株与噬菌体共培养,传代3次均能正常生长,则该菌株确定为抗噬菌体菌株。通过检测噬菌体对抗性菌株的吸附效率,排除抗性菌株通过吸附阶段影响的噬菌体侵染。抗性菌中限制-修饰系统的检测方法:利用双层琼脂平板法检测噬菌体与野生型、抗性菌株分别培养后的成斑率(Efficiency of Plating,EOP) (EOP=等量噬菌体以抗性菌株为指示菌时形成的噬菌斑数目/等量噬菌体以野生型为指示菌时形成的噬菌斑数目),记为EOP1;从抗性菌平板上挑取1-2个噬菌斑接种到抗性菌株的过夜培养物中,以允许潜在修饰的噬菌体扩增,将得到的裂解液与野生型、抗性菌株分别混合均匀,检测成斑率,记为EOP2;从野生型菌株平板上挑取单个噬菌斑接种于野生型菌株中,测定所得到的裂解液与2种菌株培养后的成斑率,记为EOP3。如果EOP3与EOP1相近,则表明抗性菌株中可能存在限制-修饰系统[30]。利用REBASE数据库,预测细菌基因组DNA中编码限制-修饰系统基因的位点[42],构建带有限制-修饰系统基因的质粒,并转化到噬菌体敏感菌株内,通过检测成斑率的变化,判断敏感菌株是否获得噬菌体抗性,而将抗性菌株的限制-修饰系统敲除后,使细菌对噬菌体的抗性消失。通过Southern杂交实验检测感染细胞内噬菌体的复制,缺乏限制-修饰系统的菌株中噬菌体DNA复制不受影响,而存在限制-修饰系统的菌株内噬菌体DNA的复制明显受到抑制[30]。
3.4 CRISPR-Cas系统CRISPR-Cas系统是一种适应性免疫机制,由一些成簇的规则间隔的短回文序列以及具有核酸加工功能的Cas蛋白(CRISPR-Associated Proteins)组成。这种免疫记忆的分子基础是细菌将入侵者基因组中30-40个碱基对作为一种短的间隔序列存储在自身的基因组中,CRISPR-Cas系统的发现就始于这种短的重复序列,该序列特点是由许多较短且保守的重复序列(Repeats)及两侧间隔区序列(Spacers)组成[43]。研究发现,间隔序列可以与噬菌体DNA序列片段互补[44],并首次在嗜热链球菌中证实CRISPR-Cas系统是一种抵御噬菌体侵染的防御机制[45]。目前已知在大约40%已测序的细菌和90%的古菌中都存在这种特殊的基因序列[44]。
细菌将噬菌体DNA中一段较短的序列作为CRISPR间隔子储存在自身的基因组中,当噬菌体再次侵染时,这种免疫记忆就会被激活,CRISPR-Cas识别并切割噬菌体DNA。Cas蛋白介导的免疫过程主要分为3个阶段,当噬菌体首次侵染宿主细菌时,外源入侵DNA的特征序列被加工整合到2个重复序列之间形成非重复的间隔序列,经过转录形成成熟的CRISPR RNA (crRNA);之后Cas蛋白在crRNA指导下精确切割外源基因特定位点,阻止噬菌体DNA在宿主细胞内复制[46]。目前已知的CRISPR-Cas系统有I-VI型[47],其中I、III、IV型系统的Cas蛋白由多亚基复合物组成,这类CRISPR-Cas系统主要参与噬菌体抵抗;而II、V、VI型系统Cas蛋白是由单亚基组成,II型系统中Cas9蛋白切割双链DNA,在分子生物学领域得到了广泛应用[48-49]。
3.5 噬菌体抵抗CRISPR-Cas的机制噬菌体通过对自身的基因位点进行突变来逃避宿主CRISPR-Cas的切割,但由于CRISPR-Cas系统具有自适应性,可以重新获得新的免疫能力,因此噬菌体仅靠基因突变并不能完美逃避宿主细菌的制约。2013年,在铜绿假单胞菌的噬菌体中发现了一种抗CRISPR蛋白,截至2019年,已经有40种抗CRISPR蛋白被发现[50]。抗CRISPR蛋白可以通过阻断Csy复合体与噬菌体目标DNA结合,使噬菌体不被降解[51-52]。抗CRISPR蛋白AcrE1抑制Cas3核酸酶活性,将内源TYPE I-E CRISPR系统转化为靶标基因的转录抑制因子,阻止噬菌体基因表达,而噬菌体也可以利用该系统抑制宿主细菌的基因表达[53]。
3.6 CRISPR-Cas抑制噬菌体的检测细菌CRISPR-Cas系统的演化过程是从外源DNA中获取新的间隔子,并将其添加到CRISPR序列中。由于CRISPR序列间保守性低,可通过多种方法,如CRISPR Finder、PILER-CR工具、CRISPRDetect、CRISPRdisco等进行生物信息学分析[54]。将细菌基因组中的CRISPR重复序列和间隔序列与噬菌体基因组序列进行比对,以判断细菌CRISPR-Cas系统是否具有抵抗噬菌体侵染的潜能,自然状态下产生噬菌体抗性的菌株CRISPR序列与野生型菌株相比有明显差异。为了证明间隔区序列是否与噬菌体抗性有关,通过删除或添加新的间隔区改变CRISPR序列,并观察菌株对噬菌体的敏感性的变化[45]。Cas蛋白是一种依赖金属离子的DNA特异性核酸内切酶,通过敲除抗性菌株的cas基因,菌株失去噬菌体抗性,说明cas基因也参与了噬菌体抵抗过程[55]。
4 流产感染 4.1 流产感染与噬菌体互作机制流产感染(Abortive Infection,Abi)是指在噬菌体将自身DNA注入到宿主细胞后,宿主细菌阻断后代噬菌体的繁殖,导致只有少量噬菌体释放的过程[56]。流产感染的结果也伴随着被侵染宿主的过早死亡,这是细菌个体保护周围种群的自我牺牲机制,革兰氏阴性菌和革兰氏阳性菌中都存在这种利他机制。目前研究较多的是大肠杆菌和乳酸菌,T4噬菌体衣壳蛋白的Gol多肽激活大肠杆菌K12的Lit蛋白酶,Lit切割核糖体延伸因子EF-Tu,抑制宿主及噬菌体基因的翻译[57]。大肠杆菌还有一种RexAB介导的RII排斥机制,由RexAB与RIIAB以及一系列宿主蛋白参与,通过改变细胞膜的通透性,降低膜电位,导致细胞死亡[58]。乳酸菌中也发现了20多种Abi系统,乳酸菌中的Abi系统由Abi蛋白介导,抑制噬菌体DNA复制[59]。此外,葡萄球菌中也发现了一种由Stk激酶(Serine/Threonine Kinase)介导的流产感染系统。噬菌体蛋白激活Stk激酶后,诱导流产感染的发生,从而防止噬菌体的传播[60]。
流产感染系统还可以通过毒素-抗毒素(Toxin-Antitoxin,TA)机制发挥作用,该系统由共转录的毒素和抗毒素组成,毒素靶向抑制细菌重要的细胞过程,导致细菌休眠或者死亡[61]。根据毒素与抗毒素因子的类型,TA系统分为蛋白质与蛋白质、蛋白质与RNA、RNA与RNA三类[26]。正常生长条件下,细菌的抗毒素与毒素因子结合,抑制毒素因子活性;当噬菌体侵染细菌后,抗毒素因子被抑制或者降解,毒素与抗毒素因子的平衡被打破,毒素释放,导致细菌死亡[62]。这种TA机制介导的流产感染首次在黑腐果胶杆菌(Pectobacterium atrosepticum)中发现,黑腐果胶杆菌的ToxIN系统由toxI与toxN组成,分别编码抗毒素因子与毒素因子;ToxN抑制细菌生长,ToxI RNA中和ToxN毒性,在噬菌体感染细菌后,毒素与抗毒素比例失衡,释放ToxN,诱发流产感染[63]。
噬菌体也可以表达抗病毒因子,逃避细菌的毒素-抗毒素系统的制约。例如T4噬菌体编码一种Dmd抗毒素因子,可以直接抑制大肠杆菌的毒素因子,阻断流产感染[64]。T7噬菌体则是通过合成一种抑制抗毒素降解的蛋白,从而使宿主毒素因子保持稳定;此外噬菌体可以合成一种类似toxI的序列伪装成抗病毒因子,抑制毒素因子以保护噬菌体后代[65]。除了上述这些机制外,逆转录子(Retrons)介导的流产感染机制也参与细菌抵抗噬菌体过程,包括逆转录酶(Reverse Transcriptase)、非编码RNA (Non-Coding RNA)及效应蛋白,噬菌体蛋白对RecBCD的抑制会激活逆转录子,导致流产感染和细胞死亡[66]。最近的研究发现,I型CRISPR-Cas基因簇内部还存在一对RNA的毒素-抗毒素系统——CreT/A,CRISPR-Cas系统可利用RNA抗毒素CreA控制RNA毒素CreT的表达,使宿主菌无法丢失其CRISPR-Cas系统(对其“上瘾”)。一旦CRISPR-Cas组分被破坏,就会诱导CreT毒素的表达,从而抑制或杀死该宿主菌,使得CRISPR-Cas系统能够在细胞群体中稳定存在[67]。
4.2 流产感染参与噬菌体抵抗的检测典型的流产感染机制的特征是在感染后期细胞过早裂解,释放未组装的噬菌体颗粒。以乳酸乳球菌为例,噬菌体抗性菌株中包含多个质粒,将质粒转入噬菌体敏感菌株,成斑率‘EOP’明显下降,通过构建基因突变体,确定AbiQ基因与噬菌体抵抗相关[68]。噬菌体吸附效率检测显示敏感菌株和抗性菌株的吸附效率差异不大,同时利用Southern实验监测不同侵染时间内抗性菌株与敏感菌株中噬菌体DNA复制情况,在AbiQ存在时噬菌体DNA能正常复制[68]。利用单克隆抗体检测噬菌体蛋白衣壳的表达,AbiQ不影响噬菌体蛋白衣壳的合成,抗性菌株上清液中噬菌体蛋白衣壳较敏感菌株明显减少,表明噬菌体抗性是由于AbiQ阻断了噬菌体的裂解周期,造成流产感染[68]。
5 细菌群体感应 5.1 细菌群体感应与噬菌体互作机制群体感应(Quorums Sensing,QS)是一种依赖于细菌密度的调控机制,当细菌密度达到一定阈值,胞外的信号分子介导基因的表达调控,从而抵御噬菌体侵染。鳗弧菌(Vibrio anguillarum)通过群体感应系统降低噬菌体的受体OmpK表达水平,而对噬菌体不敏感,群体感应信号分子N-酰基高丝氨酸内酯(Acyl Homoserine Lactone,AHL)与OmpK的mRNA水平呈明显的负相关[69]。群体感应也可以协同CRISPR-Cas系统抵御噬菌体侵染,如沙雷氏菌(Serratia)的群体感应使TYPE I-E、I-F和III-A这3种CRISPR-Cas系统的表达上调,增强细菌对噬菌体的抵抗作用[70]。细菌可以利用群体感应来调控生物膜的形成,而生物膜中的蛋白质水解酶和纤维素内切酶可以使噬菌体失活[71],并且一些噬菌体受体也可能被生物膜遮蔽,从而避免了噬菌体的侵染[72],这也是一种细菌通过群体感应抵抗噬菌体的方式。
在噬菌体侵染细菌的过程中,群体感应可以参与细菌抵抗噬菌体,同时噬菌体也可以利用群体感应来抵抗细菌。在细菌和噬菌体长期共进化的过程中,噬菌体有裂解性和溶源性2种状态。大肠杆菌AI-2型群体感应系统可以介导大肠杆菌噬菌体T1由溶源态到裂解态的转变,而噬菌体的裂解则进一步受到细菌的cAMP受体蛋白所介导的细胞代谢状态的调控[73]。群体感应介导的噬菌体由溶源态到裂解态的转变有利于噬菌体的侵染。群体感应的信号分子AHLs能提高T4噬菌体对大肠杆菌BL21(DE3)的裂解效率[74],而噬菌体可以诱导群体猝灭(Quorum Quenching,QQ)内酯酶的合成,通过这种酶水解AHL和抑制群体感应系统活性来减少铜绿假单胞菌生物膜的形成,从而提高噬菌体的侵染效率[75]。
5.2 群体感应调控噬菌体侵染的检测大肠杆菌由于缺乏LuxI型AHL合酶,不产生AHL信号分子,但可以通过LuxR型转录调节因子SdiA响应外源AHL信号分子。在添加或不添加AHL信号分子的情况下,将放射性标记的λ噬菌体与大肠杆菌野生型菌株或群体感应转录因子SdiA缺失突变体共培养,测定吸附噬菌体的放射量换算为吸附率。添加AHL使噬菌体对野生型菌株的吸附率降低,而对SdiA缺失突变体的吸附率没有显著变化[76]。在沙雷氏菌(Serratia sp. ATCC39006)中,将lacZ报告基因与cas基因或CRISPR序列融合构建报告菌株,通过检测β-半乳糖苷酶的活性,判断AHL对CRISPR-Cas系统表达的影响,发现AHL可以诱导I-E、I-F和III-A这3种CRISPR-Cas系统的表达,增强细菌对噬菌体的抵抗[70]。用群体感应信号分子淬灭酶处理铜绿假单胞菌菌株,测定绿脓素、蛋白酶和弹性蛋白酶等毒力因子的活性,群体感应淬灭酶可以降低菌株毒力因子的表达,同时细菌对所有噬菌体的敏感性提高[77]。
6 展望噬菌体的发现已有100多年的历史,由于抗生素耐药菌的出现,噬菌体的应用才重新受到广泛关注,对于噬菌体的研究也有了很多突破性进展。更好地了解噬菌体与宿主细菌之间的相互作用,尤其是二者相互抵抗机制(图 1),有利于人们充分认识噬菌体的抗菌潜力,这对于噬菌体疗法的应用具有重要意义。细菌和噬菌体共同进化,展现了二者多样的相互抵抗策略,也是维持噬菌体-宿主平衡的重要动力。随着对细菌-噬菌体相互作用分子机制认识的深入,有望在扩展噬菌体宿主范围及二者平衡的人工干预方面开辟新的应用途径。
|
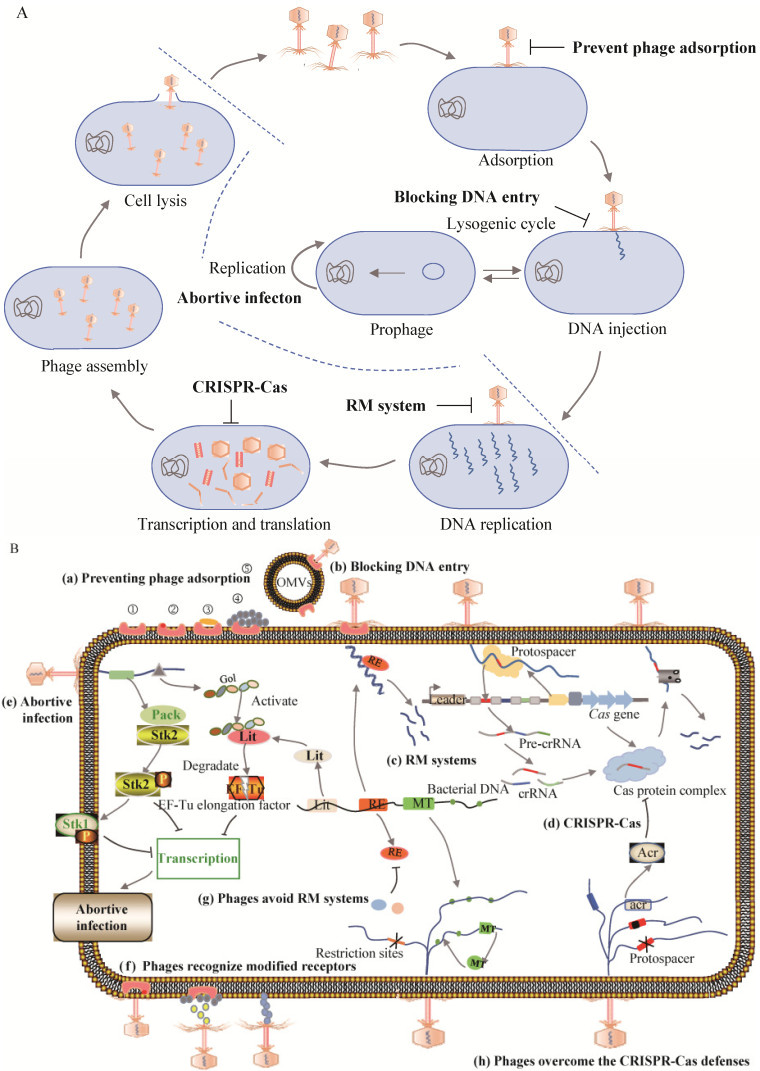
| 图 1 细菌抵抗噬菌体机制以及噬菌体的反抑制策略 Figure 1 The mechanism of bacterial resistance to phages and the counter-attacks used by phages to defend bacterial hosts 注:A:噬菌体复制周期。烈性噬菌体通过裂解周期进行复制,温和噬菌体可以通过选择进入溶源或裂解周期进行复制,细菌抵抗噬菌体侵染的机制可以在不同的复制阶段中发挥功能。B:细菌抵抗噬菌体侵染的机制。(a):吸附抑制。j至n依次表示正常的噬菌体受体、基因突变的受体、蛋白修饰后受体、受体被多糖荚膜包被、外膜囊泡OMVs;受体突变或修饰抑制噬菌体吸附,OMVs作为诱饵阻断噬菌体吸附;(b):阻止DNA进入;(c):限制-修饰系统。RE为限制性内切酶、MT为甲基转移酶;(d):CRISPR-Cas系统。细菌识别噬菌体DNA的原间隔序列,将其储存在CRISPR序列中,当噬菌体再次侵染细菌时,在crRNA引导下,Cas蛋白复合体的作用切割外源DNA;(e):细菌流产感染机制。细菌在受到噬菌体侵染后,噬菌体蛋白或Gol多肽通过一系列作用,引发流产感染;(f):噬菌体应对吸附抑制。当细菌表面的受体发生突变或者修饰时,噬菌体通过识别突变受体、降解受体的多糖荚膜或者重新识别新的受体吸附到细菌表面;(g):噬菌体应对限制-修饰系统。噬菌体通过删除DNA上限制内切酶识别位点、合成甲基转移酶或者利用宿主的甲基转移酶对自身DNA进行修饰进行甲基化修饰,逃避限制-修饰系统;(h):噬菌体应对CRISPR-Cas系统。噬菌体产生新的原间隔序列(突变或删除)、合成Acr蛋白抑制Cas复合物切割自身DNA类抵抗细菌的CRISPR-Cas系统 Note: A: The phage replication cycle; Virulent phages replicate through the lytic cycle, whereas temperate phages may replicate through either the lytic or the lysogenic cycle. Bacteria have numerous anti-phage mechanisms that function at different stages of the phage replication cycle to prevent phage replication. B: Anti-phage mechanisms act at different stages. (a): Adsorption inhibition. j to n indicate normal phage receptor, receptor mutations, protein modified receptor, and receptor coated with polysaccharide capsule, outer membrane vesicle (OMVs); Mutations or modifications of receptors inhibit phage adsorption, and OMVs act as bait to block phage adsorption. (b): Blocking phage DNA entry into the bacterial cells. (c): Restricted modification systems. RE is a restriction endonuclease and MT is a methyltransferase. (d): CRISPR-Cas systems. Bacteria recognize the protospacers of phage DNA and store them in the CRISPR array. When the phages infect the bacteria again, the Cas protein complexes cut the foreign DNA. (e): The mechanism of bacterial abortion infection. Bacterial abortion infection caused by the phage proteins. (f): Bacteriophages counteract bacterial adsorption inhibition. When the receptors are mutated or modified, the phages recognize a new receptor, or degrade the receptor's polysaccharide capsule. (g): Phages avoid restriction modification systems. Phages evade the RM systems by deletion the restriction sites, encoding methyltransferase or using the host's methyltransferase to modify its DNA. (h): Phages respond to the CRISPR-Cas systems. Phages produce a new protospacer (mutation or deletion), synthesize the Acr protein to inactivate CRISPR-Cas systems |
|
|
| [1] |
Chen PP, Sun HZ, Ren HY, Liu WH, Li GM, Zhang C. LamB, OmpC, and the core lipopolysaccharide of Escherichia coli K-12 function as receptors of bacteriophage Bp7[J]. Journal of Virology, 2020, 94(12): e00325-20. |
| [2] |
Hao GJ, Yuan CQ, Shu RD, Jia YQ, Zhao SQ, Xie SJ, Liu M, Zhou HJ, Sun SH, Wang H. O-antigen serves as a two-faced host factor for bacteriophage NJS1 infecting nonmucoid Klebsiella pneumoniae[J]. Microbial Pathogenesis, 2021, 155: 104897. DOI:10.1016/j.micpath.2021.104897 |
| [3] |
Chan BK, Sistrom M, Wertz JE, Kortright KE, Narayan D, Turner PE. Phage selection restores antibiotic sensitivity in MDR Pseudomonas aeruginosa[J]. Scientific Reports, 2016, 6: 26717. DOI:10.1038/srep26717 |
| [4] |
Chatterjee A, Johnson CN, Luong P, Hullahalli K, McBride SW, Schubert AM, Palmer KL, Carlson PE, Duerkop BA. Bacteriophage resistance alters antibiotic mediated intestinal expansion of enterococci[J]. Infection and Immunity, 2019, 87(6): e00085-00019. |
| [5] |
Tkacova A, Orieskova M, Halgasova N, Bocanova L, Bukovska G. Identification of Brevibacterium flavum genes related to receptors involved in bacteriophage BFK20 adsorption[J]. Virus Research, 2019, 274: 197775. DOI:10.1016/j.virusres.2019.197775 |
| [6] |
Hanlon GW, Denyer SP, Olliff CJ, Ibrahim LJ. Reduction in exopolysaccharide viscosity as an aid to bacteriophage penetration through Pseudomonas aeruginosa biofilms[J]. Applied and Environmental Microbiology, 2001, 67(6): 2746-2753. DOI:10.1128/AEM.67.6.2746-2753.2001 |
| [7] |
Ge HJ, Hu MZ, Zhao G, Du Y, Xu NN, Chen X, Jiao XA. The "fighting wisdom and bravery" of tailed phage and host in the process of adsorption[J]. Microbiological Research, 2020, 230: 126344. DOI:10.1016/j.micres.2019.126344 |
| [8] |
Soundararajan M, Von Bünau R, Oelschlaeger TA. K5 capsule and lipopolysaccharide are important in resistance to T4 phage attack in probiotic E. coli strain Nissle 1917[J]. Frontiers in Microbiology, 2019, 10: 2783. DOI:10.3389/fmicb.2019.02783 |
| [9] |
Camprubí S, Merino S, Benedí VJ, Tomás JM. Isolation and characterization of bacteriophage FC3-10 from Klebsiella spp[J]. FEMS Microbiology Letters, 1991, 83(3): 291-297. DOI:10.1111/j.1574-6968.1991.tb04479.x |
| [10] |
Destoumieux-Garzón D, Duquesne S, Peduzzi J, Goulard C, Desmadril M, Letellier L, Rebuffat S, Boulanger P. The iron—siderophore transporter FhuA is the receptor for the antimicrobial peptide microcin J25: role of the microcin Val(11)-Pro(16) beta-hairpin region in the recognition mechanism[J]. The Biochemical Journal, 2005, 389(Pt3): 869-876. |
| [11] |
Manning AJ, Kuehn MJ. Contribution of bacterial outer membrane vesicles to innate bacterial defense[J]. BMC Microbiology, 2011, 11: 258. DOI:10.1186/1471-2180-11-258 |
| [12] |
Meyer JR, Dobias DT, Weitz JS, Barrick JE, Quick RT, Lenski RE. Repeatability and contingency in the evolution of a key innovation in phage lambda[J]. Science, 2012, 335(6067): 428-432. DOI:10.1126/science.1214449 |
| [13] |
Hu B, Margolin W, Molineux IJ, Liu J. The bacteriophage T7 virion undergoes extensive structural remodeling during infection[J]. Science, 2013, 339(6119): 576-579. DOI:10.1126/science.1231887 |
| [14] |
Tkhilaishvili T, Lombardi L, Klatt AB, Trampuz A, Di Luca M. Bacteriophage Sb-1 enhances antibiotic activity against biofilm, degrades exopolysaccharide matrix and targets persisters of Staphylococcus aureus[J]. International Journal of Antimicrobial Agents, 2018, 52(6): 842-853. DOI:10.1016/j.ijantimicag.2018.09.006 |
| [15] |
Liu YN, Mi ZQ, Mi LY, Huang Y, Li PY, Liu HY, Yuan X, Niu WK, Jiang N, Bai CQ, et al. Identification and characterization of capsule depolymerase Dpo48 from Acinetobacter baumannii phage IME200[J]. PeerJ, 2019, 7: e6173. DOI:10.7717/peerj.6173 |
| [16] |
Liu YN, Leung SSY, Huang Y, Guo YT, Jiang N, Li PY, Chen JC, Wang RT, Bai CQ, Mi ZQ, et al. Identification of two depolymerases from phage IME205 and their antivirulent functions on K47 capsule of Klebsiella pneumoniae[J]. Frontiers in Microbiology, 2020, 11: 218. DOI:10.3389/fmicb.2020.00218 |
| [17] |
Decker KB, James TD, Stibitz S, Hinton DM. The Bordetella pertussis model of exquisite gene control by the global transcription factor BvgA[J]. Microbiology: Reading, England, 2012, 158(Pt 7): 1665-1676. |
| [18] |
Liu M, Deora R, Doulatov SR, Gingery M, Eiserling FA, Preston A, Maskell DJ, Simons RW, Cotter PA, Parkhill J, et al. Reverse transcriptase-mediated tropism switching in Bordetella bacteriophage[J]. Science, 2002, 295(5562): 2091-2094. DOI:10.1126/science.1067467 |
| [19] |
Van Twest R, Kropinski AM. Bacteriophage enrichment from water and soil[J]. Methods in Molecular Biology, 2009, 501: 15-21. |
| [20] |
Gill JJ, Hyman P. Phage choice, isolation, and preparation for phage therapy[J]. Current Pharmaceutical Biotechnology, 2010, 11(1): 2-14. DOI:10.2174/138920110790725311 |
| [21] |
Le S, He XS, Tan YL, Huang GT, Zhang L, Lux R, Shi WY, Hu FQ. Mapping the tail fiber as the receptor binding protein responsible for differential host specificity of Pseudomonas aeruginosa bacteriophages PaP1 and JG004[J]. PLoS One, 2013, 8(7): e68562. DOI:10.1371/journal.pone.0068562 |
| [22] |
Mosig G, Lin GW, Franklin J, Fan WH. Functional relationships and structural determinants of two bacteriophage T4 lysozymes: a soluble (gene e) and a baseplate-associated (gene 5) protein[J]. The New Biologist, 1989, 1(2): 171-179. |
| [23] |
Moak M, Molineux IJ. Peptidoglycan hydrolytic activities associated with bacteriophage virions[J]. Molecular Microbiology, 2004, 51(4): 1169-1183. DOI:10.1046/j.1365-2958.2003.03894.x |
| [24] |
Lu MJ, Stierhof YD, Henning U. Location and unusual membrane topology of the immunity protein of the Escherichia coli phage T4[J]. Journal of Virology, 1993, 67(8): 4905-4913. DOI:10.1128/jvi.67.8.4905-4913.1993 |
| [25] |
Lu MJ, Henning U. Superinfection exclusion by T-even-type coliphages[J]. Trends in Microbiology, 1994, 2(4): 137-139. DOI:10.1016/0966-842X(94)90601-7 |
| [26] |
Labrie SJ, Samson JE, Moineau S. Bacteriophage resistance mechanisms[J]. Nature Reviews Microbiology, 2010, 8(5): 317-327. DOI:10.1038/nrmicro2315 |
| [27] |
Sun XM, Göhler A, Heller KJ, Neve H. The ltp gene of temperate Streptococcus thermophilus phage TP-J34 confers superinfection exclusion to Streptococcus thermophilus and Lactococcus lactis[J]. Virology, 2006, 350(1): 146-157. DOI:10.1016/j.virol.2006.03.001 |
| [28] |
Bebeacua C, Lorenzo Fajardo JC, Blangy S, Spinelli S, Bollmann S, Neve H, Cambillau C, Heller KJ. X-ray structure of a superinfection exclusion lipoprotein from phage TP-J34 and identification of the tape measure protein as its target[J]. Molecular Microbiology, 2013, 89(1): 152-165. DOI:10.1111/mmi.12267 |
| [29] |
Ruiz-Cruz S, Parlindungan E, Erazo Garzon A, Alqarni M, Lugli GA, Ventura M, Van Sinderen D, Mahony J. Lysogenization of a lactococcal host with three distinct temperate phages provides homologous and heterologous phage resistance[J]. Microorganisms, 2020, 8(11): 1685. DOI:10.3390/microorganisms8111685 |
| [30] |
McGrath S, Fitzgerald GF, Van Sinderen D. Identification and characterization of phage-resistance genes in temperate lactococcal bacteriophages[J]. Molecular Microbiology, 2002, 43(2): 509-520. DOI:10.1046/j.1365-2958.2002.02763.x |
| [31] |
Arber W, Dussoix D. Host specificity of DNA produced by Escherichia coli: I. Host controlled modification of bacteriophage λ[J]. Journal of Molecular Biology, 1962, 5(1): 18-36. DOI:10.1016/S0022-2836(62)80058-8 |
| [32] |
Vasu K, Nagaraja V. Diverse functions of restriction-modification systems in addition to cellular defense[J]. Microbiology and Molecular Biology Reviews, 2013, 77(1): 53-72. DOI:10.1128/MMBR.00044-12 |
| [33] |
Roberts RJ, Belfort M, Bestor T, Bhagwat AS, Bickle TA, Bitinaite J, Blumenthal RM, Degtyarev SK, Dryden DTF, Dybvig K, et al. A nomenclature for restriction enzymes, DNA methyltransferases, homing endonucleases, and their genes[J]. Nucleic Acids Research, 2003, 31(7): 1805-1812. DOI:10.1093/nar/gkg274 |
| [34] |
Xiong XL, Wu G, Wei Y, Liu LQ, Zhang YB, Su R, Jiang XY, Li MX, Gao HY, Tian XH, et al. SspABCD-SspE is a phosphorothioation-sensing bacterial defence system with broad anti-phage activities[J]. Nature Microbiology, 2020, 5(7): 917-928. DOI:10.1038/s41564-020-0700-6 |
| [35] |
Wang SW, Wan MP, Huang RL, Zhang YJ, Xie YQ, Wei Y, Ahmad M, Wu D, Hong Y, Deng ZX, et al. SspABCD-SspFGH constitutes a new type of DNA phosphorothioate-based bacterial defense system[J]. mBio, 2021, 12(2): e00613-21. |
| [36] |
Hoskisson PA, Sumby P, Smith MCM. The phage growth limitation system in Streptomyces coelicolor A(3)2 is a toxin/antitoxin system, comprising enzymes with DNA methyltransferase, protein kinase and ATPase activity[J]. Virology, 2015, 477: 100-109. DOI:10.1016/j.virol.2014.12.036 |
| [37] |
Ofir G, Melamed S, Sberro H, Mukamel Z, Silverman S, Yaakov G, Doron S, Sorek R. DISARM is a widespread bacterial defence system with broad anti-phage activities[J]. Nature Microbiology, 2018, 3(1): 90-98. DOI:10.1038/s41564-017-0051-0 |
| [38] |
Barrangou R, Van Der Oost J. Bacteriophage exclusion, a new defense system[J]. The EMBO Journal, 2015, 34(2): 134-135. DOI:10.15252/embj.201490620 |
| [39] |
Piya D, Vara L, Russell WK, Young R, Gill JJ. The multicomponent antirestriction system of phage P1 is linked to capsid morphogenesis[J]. Molecular Microbiology, 2017, 105(3): 399-412. DOI:10.1111/mmi.13705 |
| [40] |
Atanasiu C, Su TJ, Sturrock SS, Dryden DTF. Interaction of the ocr gene 0.3 protein of bacteriophage T7 with EcoKI restriction/modification enzyme[J]. Nucleic Acids Research, 2002, 30(10): 3936-3944. |
| [41] |
Quiberoni A, Reinheimer JA, Suárez VB. Performance of Lactobacillus helveticus spontaneous phage-resistant mutants in hard cheese production[J]. International Dairy Journal, 1998, 8(12): 941-949. DOI:10.1016/S0958-6946(98)00126-5 |
| [42] |
Roberts RJ, Vincze T, Posfai J, Macelis D. REBASE—a database for DNA restriction and modification: enzymes, genes and genomes[J]. Nucleic Acids Research, 2015, 43(D1): D298-D299. DOI:10.1093/nar/gku1046 |
| [43] |
Koonin EV, Makarova KS. Mobile genetic elements and evolution of CRISPR-Cas systems: all the way there and back[J]. Genome Biology and Evolution, 2017, 9(10): 2812-2825. DOI:10.1093/gbe/evx192 |
| [44] |
Makarova KS, Wolf YI, Alkhnbashi OS, Costa F, Shah SA, Saunders SJ, Barrangou R, Brouns SJJ, Charpentier E, Haft DH, et al. An updated evolutionary classification of CRISPR-Cas systems[J]. Nature Reviews Microbiology, 2015, 13(11): 722-736. DOI:10.1038/nrmicro3569 |
| [45] |
Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science, 2007, 315(5819): 1709-1712. DOI:10.1126/science.1138140 |
| [46] |
Frank H, Hagen R, Pey WS, Majda B, Sarah R, Emmanuelle C. The biology of CRISPR-Cas: backward and forward[J]. Cell, 2018, 172(6): 1239-1259. DOI:10.1016/j.cell.2017.11.032 |
| [47] |
Koonin EV, Makarova KS, Zhang F. Diversity, classification and evolution of CRISPR-Cas systems[J]. Current Opinion in Microbiology, 2017, 37: 67-78. DOI:10.1016/j.mib.2017.05.008 |
| [48] |
Brouns SJJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJH, Snijders APL, Dickman MJ, Makarova KS, Koonin EV, Van Der Oost J. Small CRISPR RNAs guide antiviral defense in prokaryotes[J]. Science, 2008, 321(5891): 960-964. DOI:10.1126/science.1159689 |
| [49] |
Strotskaya A, Savitskaya E, Metlitskaya A, Morozova N, Datsenko KA, Semenova E, Severinov K. The action of Escherichia coli CRISPR-Cas system on lytic bacteriophages with different lifestyles and development strategies[J]. Nucleic Acids Research, 2017, 45(4): 1946-1957. |
| [50] |
Bondy-Denomy J, Pawluk A, Maxwell KL, Davidson AR. Bacteriophage genes that inactivate the CRISPR/Cas bacterial immune system[J]. Nature, 2013, 493(7432): 429-432. DOI:10.1038/nature11723 |
| [51] |
Chowdhury S, Carter J, Rollins MF, Golden SM, Jackson RN, Hoffmann C, Nosaka L, Bondy-Denomy J, Maxwell KL, Davidson AR, et al. Structure reveals mechanisms of viral suppressors that intercept a CRISPR RNA-guided surveillance complex[J]. Cell, 2017, 169(1): 47-57. DOI:10.1016/j.cell.2017.03.012 |
| [52] |
Guo TW, Bartesaghi A, Yang H, Falconieri V, Rao P, Merk A, Eng ET, Raczkowski AM, Fox T, Earl LA, et al. Cryo-EM structures reveal mechanism and inhibition of DNA targeting by a CRISPR-cas surveillance complex[J]. Cell, 2017, 171(2): 414-426. DOI:10.1016/j.cell.2017.09.006 |
| [53] |
Pawluk A, Shah M, Mejdani M, Calmettes C, Moraes TF, Davidson AR, Maxwell KL. Disabling a type I-E CRISPR-Cas nuclease with a bacteriophage-encoded anti-CRISPR protein[J]. mBio, 2017, 8(6): 01751-17. |
| [54] |
Alkhnbashi OS, Meier T, Mitrofanov A, Backofen R, Voß B. CRISPR-Cas bioinformatics[J]. Methods: San Diego, Calif, 2020, 172: 3-11. DOI:10.1016/j.ymeth.2019.07.013 |
| [55] |
Wiedenheft B, Zhou KH, Jinek M, Coyle SM, Ma W, Doudna JA. Structural basis for DNase activity of a conserved protein implicated in CRISPR-mediated genome defense[J]. Structure, 2009, 17(6): 904-912. DOI:10.1016/j.str.2009.03.019 |
| [56] |
Lopatina A, Tal N, Sorek R. Abortive infection: bacterial suicide as an antiviral immune strategy[J]. Annual Review of Virology, 2020, 7(1): 371-384. DOI:10.1146/annurev-virology-011620-040628 |
| [57] |
Bingham R, Ekunwe SIN, Falk S, Snyder L, Kleanthous C. The major head protein of bacteriophage T4 binds specifically to elongation factor Tu[J]. Journal of Biological Chemistry, 2000, 275(30): 23219-23226. DOI:10.1074/jbc.M002546200 |
| [58] |
Wong S, Alattas H, Slavcev RA. A snapshot of the λ T4rII exclusion (Rex) phenotype in Escherichia coli[J]. Current Genetics, 2021. DOI:10.1007/s00294-021-01183-2 |
| [59] |
Chopin MC, Chopin A, Bidnenko E. Phage abortive infection in lactococci: variations on a theme[J]. Current Opinion in Microbiology, 2005, 8(4): 473-479. DOI:10.1016/j.mib.2005.06.006 |
| [60] |
Depardieu F, Didier JP, Bernheim A, Sherlock A, Molina H, Duclos B, Bikard D. A eukaryotic-like serine/threonine kinase protects Staphylococci against phages[J]. Cell Host Microbe, 2016, 20(4): 471-481. DOI:10.1016/j.chom.2016.08.010 |
| [61] |
Harms A, Brodersen DE, Mitarai N, Gerdes K. Toxins, targets, and triggers: an overview of toxin-antitoxin biology[J]. Molecular Cell, 2018, 70(5): 768-784. DOI:10.1016/j.molcel.2018.01.003 |
| [62] |
Srivastava A, Pati S, Kaushik H, Singh S, Garg LC. Toxin-antitoxin systems and their medical applications: current status and future perspective[J]. Applied Microbiology and Biotechnology, 2021, 105(5): 1803-1821. DOI:10.1007/s00253-021-11134-z |
| [63] |
Fineran PC, Blower TR, Foulds IJ, Humphreys DP, Lilley KS, Salmond GP. The phage abortive infection system, ToxIN, functions as a protein-RNA toxin-antitoxin pair[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(3): 894-899. DOI:10.1073/pnas.0808832106 |
| [64] |
Otsuka Y, Yonesaki T. Dmd of bacteriophage T4 functions as an antitoxin against Escherichia coli LsoA and RnlA toxins[J]. Molecular Microbiology, 2012, 83(4): 669-681. DOI:10.1111/j.1365-2958.2012.07975.x |
| [65] |
Blower TR, Evans TJ, Przybilski R, Fineran PC, Salmond GPC. Viral evasion of a bacterial suicide system by RNA-based molecular mimicry enables infectious altruism[J]. PLoS Genetics, 2012, 8(10): e1003023. DOI:10.1371/journal.pgen.1003023 |
| [66] |
Millman A, Bernheim A, Stokar-Avihail A, Fedorenko T, Voichek M, Leavitt A, Oppenheimer-Shaanan Y, Sorek R. Bacterial retrons function in anti-phage defense[J]. Cell, 2020, 183(6): 1551-1561. DOI:10.1016/j.cell.2020.09.065 |
| [67] |
Li M, Gong LY, Cheng FY, Yu HY, Zhao DH, Wang R, Wang T, Zhang SJ, Zhou J, Shmakov SA, et al. Toxin-antitoxin RNA pairs safeguard CRISPR-Cas systems[J]. Science, 2021, 372(6541): eabe5601. DOI:10.1126/science.abe5601 |
| [68] |
Emond E, Dion E, Walker SA, Vedamuthu ER, Kondo JK, Moineau S. AbiQ, an abortive infection mechanism from Lactococcus lactis[J]. Applied and Environmental Microbiology, 1998, 64(12): 4748-4756. DOI:10.1128/AEM.64.12.4748-4756.1998 |
| [69] |
Tan DM, Svenningsen SL, Middelboe M. Quorum sensing determines the choice of antiphage defense strategy in Vibrio anguillarum[J]. mBio, 2015, 6(3): e00627. |
| [70] |
Patterson AG, Jackson SA, Taylor C, Evans GB, Salmond GPC, Przybilski R, Staals RHJ, Fineran PC. Quorum sensing controls adaptive immunity through the regulation of multiple CRISPR-cas systems[J]. Molecular Cell, 2016, 64(6): 1102-1108. DOI:10.1016/j.molcel.2016.11.012 |
| [71] |
Azeredo J, Sutherland IW. The use of phages for the removal of infectious biofilms[J]. Current Pharmaceutical Biotechnology, 2008, 9(4): 261-266. DOI:10.2174/138920108785161604 |
| [72] |
Rickard AH, Gilbert P, High NJ, Kolenbrander PE, Handley PS. Bacterial coaggregation: an integral process in the development of multi-species biofilms[J]. Trends in Microbiology, 2003, 11(2): 94-100. DOI:10.1016/S0966-842X(02)00034-3 |
| [73] |
Laganenka L, Sander T, Lagonenko A, Chen Y, Link H, Sourjik V. Quorum sensing and metabolic state of the host control lysogeny-lysis switch of bacteriophage T1[J]. mBio, 2019, 10(5): e01884-19. |
| [74] |
Taj MK, Bing LL, Zhang Q, Ling JX, Taj I, Hassani TM, Samreen Z, Mangle A, Wei YL. Quorum sensing molecules acyl-homoserine lactones and indole effect on T4 bacteriophage production and lysis activity[J]. Pakistan Veterinary Journal, 2014, 34(3): 397-399. |
| [75] |
Pei RT, Lamas-Samanamud GR. Inhibition of biofilm formation by T7 bacteriophages producing quorum-quenching enzymes[J]. Applied and Environmental Microbiology, 2014, 80(17): 5340-5348. DOI:10.1128/AEM.01434-14 |
| [76] |
Høyland-Kroghsbo NM, Mærkedahl RB, Svenningsen SL. A quorum-sensing-induced bacteriophage defense mechanism[J]. mBio, 2013, 4(1): e00362-12. |
| [77] |
Mion S, Rémy B, Plener L, Brégeon F, Chabrière E, Daudé D. Quorum quenching lactonase strengthens bacteriophage and antibiotic arsenal against Pseudomonas aeruginosa clinical isolates[J]. Frontiers in Microbiology, 2019, 10: 2049. DOI:10.3389/fmicb.2019.02049 |
 2021, Vol. 48
2021, Vol. 48