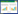扩展功能
文章信息
- 李瑞娟, 赵晓雨, 杨润雨, 刘洋, 颜富, 王海龙, 张友明, 符军
- LI Ruijuan, ZHAO Xiaoyu, YANG Runyu, LIU Yang, YAN Fu, WANG Hailong, ZHANG Youming, FU Jun
- 噬菌体重组酶介导的DNA同源重组工程
- Recombineering mediated by bacteriophage recombinases
- 微生物学通报, 2021, 48(9): 3230-3248
- Microbiology China, 2021, 48(9): 3230-3248
- DOI: 10.13344/j.microbiol.china.210402
-
文章历史
- 收稿日期: 2021-05-07
- 接受日期: 2021-06-04
- 网络首发日期: 2021-07-22
在基因工程中,来源于噬菌体(Bacteriophage)的遗传操作工具具有非常重要的地位,主要包括位点特异性重组酶、柯斯质粒文库以及同源重组酶等。
溶源性噬菌体在宿主菌染色体上的整合和切离大部分是由噬菌体编码的整合酶完成的。噬菌体整合酶属于位点特异性重组酶(Site-Specific Recombinases)类[1]。根据氨基酸同源性及催化残基的不同,位点特异性重组酶主要分为2个家族:酪氨酸家族和丝氨酸家族[2-3]。酪氨酸家族的位点特异性重组酶主要包括Lambda整合酶(Lambda Integrase)[4]、E. coli P1噬菌体Cre重组酶[5]、E. coli D6噬菌体Dre重组酶[6]、酿酒酵母的FLP重组酶[7]及E. coli的XerC蛋白[8]等。丝氨酸家族的位点特异性重组酶主要包括链霉菌ΦC31重组酶[9]、链霉菌ΦBT1重组酶[10]和肺炎克雷伯菌Tn3重组酶[11]等。其中,Lambda整合酶能够介导attP/attB位点间的重组[4]。目前,该系统已经被Invitrogen公司开发成Gateway®克隆试剂盒,主要用于高通量质粒的构建[12]。Cre重组酶可以使2个LoxP位点之间发生特异性重组[5]。目前,Cre/lox系统通过位点特异性重组可以实现基因敲除与敲入、替换、缺失突变、染色体结构改造及基因表达调控等多种基因工程操作,在原核以及真核生物的遗传工程中得到了非常广泛的应用[13]。ΦC31重组酶介导attP/attB位点间的重组,主要用于将外源DNA整合到染色体上,具有高效、不需要辅因子和插入片段大等优点[14]。
由人工构建的含有质粒复制子colEI和Lambda噬菌体cos序列的质粒载体,称为柯斯质粒(Cosmid),又叫黏粒。其含有Lambda噬菌体cos位点和质粒复制起始位点,可以选择性地将37-50 kb的DNA包裹进噬菌体头部,兼具了噬菌体载体和质粒载体的优点,常用于高通量DNA文库的构建。
大肠杆菌中早期的同源重组工程依赖于RecA蛋白,需要大于500 bp的同源臂,从而需要中间载体的构建。1998年,Zhang等发现在携带Rac原噬菌体的E. coli中,重组酶RecE/RecT能够促进携带短同源臂的线性DNA分子进行同源重组[15]。1999年,Muyrers等发现Lambda噬菌体的Red操纵子编码的重组酶Redα/Redβ也能够在体内介导高效同源重组[16]。基于噬菌体同源重组酶Redα/Redβ和RecE/RecT建立的同源重组系统分别为Red系统和RecET系统,统称为DNA同源重组工程(Recombineering),有文献也称Red/ET同源重组工程(Red/ET Recombineering)[17-24]。与传统的RecA同源重组系统相比,Red/ET同源重组工程只需要35–50 bp的短同源臂就可以实现高效率的同源重组,对靶标DNA分子进行快速、精准、高效的修饰,因为不受限制性内切酶识别位点和DNA分子大小的限制,极大地提高了目的基因修饰的效率,已经广泛地应用于生命科学研究的各个领域。
本文主要综述了噬菌体同源重组酶及其作用机制、同源重组工程在E. coli及其他细菌中的研究进展,以及同源重组工程在细菌次级代谢产物挖掘、动植物转基因、病毒基因组DNA的克隆及修饰等方面的应用。
1 噬菌体同源重组酶 1.1 Redαβγ重组酶在大肠杆菌recA突变株中,Lambda噬菌体red突变株缺失了同源重组的能力,从而确定了Red操纵子的功能[25]。在Red操纵子里,redα、redβ和redγ被pL启动子转录,而pL启动子受温度敏感的cI857所抑制[26]。
Redα分子量为24 kD,包含226个氨基酸,是一种不依赖于ATP但需要Mg2+的5′→3′双链DNA核酸外切酶,其结合在磷酸化的双链DNA(Double Strand DNA,dsDNA)末端,将dsDNA降解为3′单链悬突的DNA[27]。Redα只会结合在dsDNA的末端,不会作用在dsDNA的缺口(Nicks or Gaps)上[28]。在催化核酸外切反应时,Redα形成一个环形的同源三聚体,具有一个中心漏斗形通道,通道的入口可以容纳一个dsDNA进入,而出口的直径仅有单链DNA(Single Strand DNA,ssDNA)的大小[29]。因此,Redα的同源三聚体通道入口端与dsDNA末端结合后,dsDNA的5′末端被三聚体的活性位点降解,使DNA分子的3′末端变成单链悬突(3′-ssDNA Overhang),从同源三聚体通道的出口端退出[29]。Redα降解DNA的速度大约为每秒12个核苷酸[30]。DNA的GC含量不同,Redα降解速度会有所变化,因为限速步骤是氢键断裂[31]。在酶切过程中,当Redα遇到5′-GGCGA-3′序列时会出现长时间的停顿(3.5-11.2 s)[32]。
Redβ分子量为25.8 kD,包含261个氨基酸,是ssDNA退火蛋白,能够与Redα产生的ssDNA结合形成一个ssDNA-Redb复合体,保护暴露出来的ssDNA免受单链核酸酶的攻击,并且可以寻找到ssDNA的同源序列[33]。Redβ能够与长度大于35 bp的ssDNA稳定结合,在催化退火互补形成dsDNA后,其仍然与退火的dsDNA紧密结合在一起,这种退火的dsDNA-Redβ复合物能够抵抗DNAase I,比ssDNA-Redβ复合物更加稳定[33]。当Redβ从细胞里分离出来的时候,其和Redα、S1核糖体蛋白以及NusA转录延伸因子形成一个复合体[34]。Caldwell等发现Redβ可以与大肠杆菌ssDNA结合蛋白SSB相互作用,并证明了SSB羧基端的末端肽在相互作用中的重要性[35]。最近,Filsinger等发现Redβ与SSB的相互作用主要依赖于对SSB羧基端7个氨基酸的识别,并且这种相互作用在一定程度上决定了Redβ的宿主特异性[36]。有些Redβ的突变体不仅影响其本身的功能,也影响Redα的核酸外切酶活性[25]。这暗示着Red操纵子的功能不仅来源于Redα和Redβ之间的相互作用,而且与宿主中存在的因子相关。在电子显微镜下,当没有ssDNA时,Redβ形成一个大约由12个亚基组成的无活性小环;当ssDNA存在时,Redβ则会形成一个大约由15-18个亚基组成的有活性大环;退火后的dsDNA-Redβ复合体则是一个左手螺旋结构[37]。原子力显微镜观察发现,没有DNA的时候,Redβ会形成一个带有右手螺旋结构的缺口椭圆,ssDNA的存在则破坏了这种结构,当加入第2个互补的ssDNA时,退火的产物形成一个左手螺旋结构[38]。在最新的研究中,研究者用高分辨率质谱分析发现,在不含DNA的情况下,Redβ会形成12个亚基的低聚物;在38 bp的ssDNA存在时,4-14个Redβ会与之形成复合物;在38 bp的退火dsDNA上,会有11个亚基与之形成更稳定的复合物[39]。
redγ会从不同的翻译起始位点编码16.1 kD的GamL和11.4 kD的GamS。大肠杆菌核酸酶RecBCD复合体虽然在一个细胞内只有10个拷贝,但是其对线性DNA的破坏力极强,可以清除大多数线性dsDNA[40]。GamS二聚体可以与RecBCD形成RecBCD-Redγ复合体,从而抑制其核酸外切酶和解旋酶的活性[41]。Redγ晶体结构显示其二聚体形似于DNA的双螺旋,并且外围的α螺旋富含负电荷,可以模拟DNA与RecBCD结合,抑制其核酸外切酶活性对外源DNA的降解,从而能够提高Redα/Redβ的DNA同源重组效率[42]。Redγ并没有完全抑制RecBCD的活性,因为表达Redγ的野生菌株和recBC突变菌株的表型是不同的,表达Redγ的细菌仍然保持很高水平的DNA同源重组活性,而DNA双链断裂的修复在RecBCD敲除菌株中则完全没有[41]。这说明,Redγ只是改变了RecBCD的活性而不是一个完全的RecBCD抑制蛋白,这种活性的改变需要RecF途径的参与[9, 43]。
1.2 RecET重组酶在一些大肠杆菌菌株的基因组中发现了类似Lambda的Rac前噬菌体,Rac前噬菌体包含编码同源重组酶功能的recE和recT,这些基因最初被认为是属于RecBCD和RecF途径之外的第3种大肠杆菌内源性重组途径,因此也被称为RecE途径[44]。在recBC的缺陷株中,sbcA的突变可以激活recE和recT基因的表达,从而提高了重组水平[44]。RecE在功能上类似于Redα,而RecT在功能上类似于Redβ[45]。
RecE也被称为ExoVIII DNA核酸外切酶,与dsDNA末端结合,可以非常迅速地从5′端进行降解,形成3′端单链DNA悬突[46]。全长的RecE是由866个氨基酸组成的96.4 kD蛋白,大小约是Redα的4倍,然而最初的一些研究认为其氨基端大约600个氨基酸是没有功能的,而羧基端264个氨基酸具有全长RecE的所有活性[27, 46]。2012年,Fu等发现全长的RecE有利于线性DNA之间的重组,而氨基端564或者602个残基缺失的RecE,对于线性DNA之间的重组效率显著降低,而对于环状DNA与线性DNA之间的重组效率显著提升,但是RecE氨基端的功能目前仍然不清楚[47]。全长的RecE没有获得晶体结构,羧基端晶体结构显示,它形成一个环形的同源四聚体,与Redα的三聚体类似,中央有一个锥形通道,其宽度足以在入口端结合dsDNA末端,但在出口端仅允许3′端单链DNA通过[33]。
RecT的分子量为29.7 kD,是一个ssDNA退火蛋白,在电镜下观察RecT蛋白以及RecT蛋白和DNA的复合物,发现在没有DNA的时候RecT蛋白形成棒状聚合物,与ssDNA组成的复合物成丝状螺旋结构[48]。与Redβ不同的是,RecT可以稳定地结合ssDNA和dsDNA;但是,RecT可以促进ssDNA的聚集,而不能促进dsDNA的聚集[48]。对RecT促进配对的体外研究表明,RecT可以介导同源ssDNA或者线性dsDNA的同源单链末端入侵超螺旋DNA,带有同源单链末端的线性dsDNA类似于RecE核酸外切酶消化后的DNA底物,这个结果支持了RecT可以直接参与RecE途径中DNA双链断裂修复的假说[49]。
免疫共沉淀实验表明,RecE和RecT之间存在特定的蛋白质-蛋白质相互作用,这与Redα和Redβ相似。因此,这表明重组需要核酸外切酶与相应的DNA退火蛋白之间的协同功能,Redα与RecT,或者Redβ与RecE之间没有协同作用,所以他们的组合也没有类似的重组功能[27, 50]。
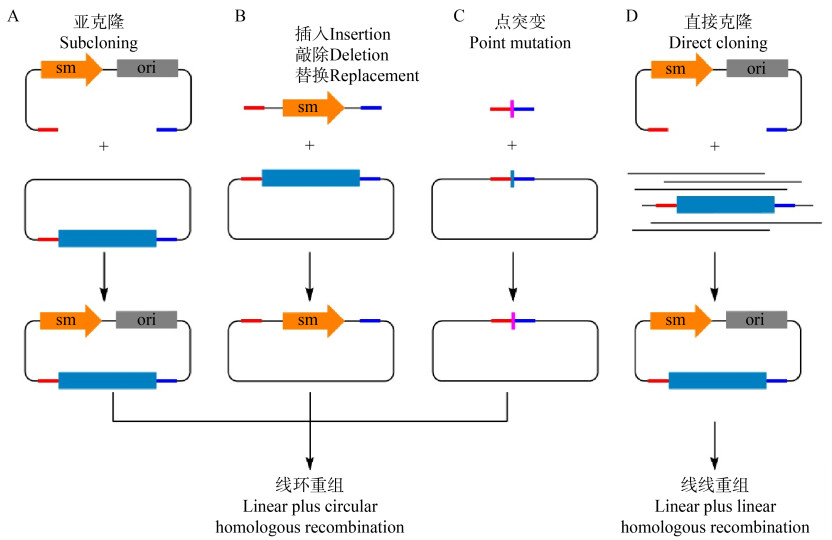
2 噬菌体同源重组酶作用机制噬菌体蛋白Redα/Redβ和RecE/RecT发挥同源重组作用,具有5′→3′双链DNA核酸外切酶活性的Redα或者RecE首先将dsDNA分子从5′端开始进行降解,使DNA分子的3′末端变成单链悬突,或者使较短的dsDNA底物形成一个ssDNA分子。ssDNA退火蛋白Redβ或者RecT与暴露的ssDNA结合形成复合物,防止ssDNA分子被降解,并且该复合物会在Redβ或者RecT的介导下寻找同源序列,最终完成DNA重组(图 1)。
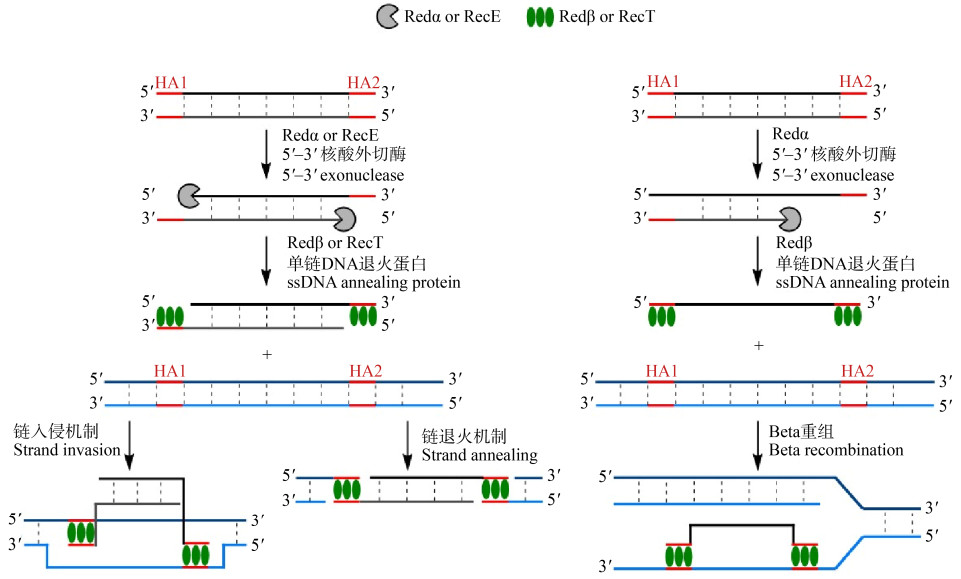
|
| 图 1 噬菌体同源重组酶Redα/Redβ和RecE/RecT作用机制 Figure 1 Models for Red/ET mediated recombineering |
|
|
在同源重组工程中,线性DNA片段和环形DNA分子之间的重组被称为线环重组(Linear Plus Circular Homologous Recombination,LCHR)(图 2A),2个线性DNA分子之间的重组被称为线线重组(Linear Plus Linear Homologous Recombination,LLHR)(图 2B);Redγ只存在于Red操纵子中,但是将其与RecE/RecT共表达,能极大地提高其重组效率;研究还证实,Red系统介导线环重组更有效,全长的RecET系统介导线线重组更有效[47]。线环重组和线线重组的机理不同,线环重组更倾向于链入侵(Strand Invasion)机制或者Beta重组(Beta Recombination)[51-52],依赖于DNA的复制;线线重组更倾向于链退火(Strand Annealing)机制,其重组的完成不依赖于DNA的复制(图 1)。实验证明,在线环重组中,DNA在复制叉处入侵到滞后链的效率比先导链高。以通过PCR得到的dsDNA作为底物时,Redα可能仅从dsDNA的一端开始消化,产生ssDNA中间体,该中间体将在复制叉处形成单链异双螺旋。因为Redα核酸外切酶偏好于具有5′磷酸化末端的dsDNA,而不能降解巯基化脱氧核糖核酸存在时形成的硫代硫酸酯键,所以在设计扩增dsDNA底物的引物时可以进行不对称修饰,使Redα在体内定向地消化产生与滞后链互补的ssDNA,从而提高重组效率[51]。外源线性DNA分子进行线环重组时作为冈崎片段参与DNA的复制是唯一通过实验验证的机理模型,称为Beta重组[51]。
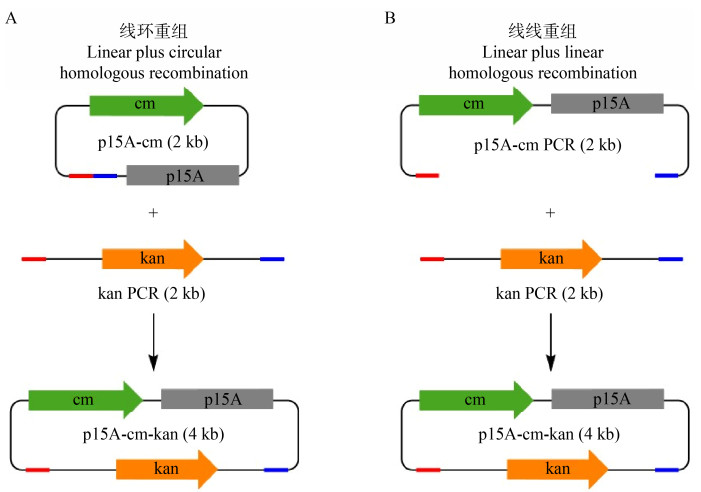
将含有质粒复制位点、选择性标记基因以及相应同源臂的线性dsDNA片段转入表达了Red系统或者RecET系统重组酶的E. coli中,通过线环同源重组线性dsDNA片段可以与细胞内的质粒、细菌人工染色体(Bacterial Artificial Chromosomes,BAC)或者E. coli染色体上的目的DNA形成环状质粒,此技术称为亚克隆(Subcloning)或者缺口修补(Gap Repair)[22, 53](图 3A)。
1998年,Zhang等利用同源重组工程在E. coli中实现了对高拷贝质粒、含有小鼠Hoxa基因的P1载体(> 76 kb)以及E. coli染色体进行修饰,包括目的DNA的插入(Insertion)、敲除(Deletion)以及替换(Replacement)等[15](图 3B)。1999年,Muyrers等利用同源重组工程在E. coli中实现了对BAC的修饰[16](图 3B)。利用同源重组工程结合SacB介导的反向筛选技术,Zhang等实现了对高拷贝质粒的点突变(Point Mutation)修饰[15],Muyrers等实现了对BAC的点突变修饰[24](图 3C)。
1998年,Zhang等将含有质粒复制位点、选择性标记基因以及相应同源臂的线性dsDNA片段和来源于大肠杆菌、酵母或者小鼠胚胎干细胞的包含目的片段的基因组DNA共转化到表达了RecET系统重组酶的E. coli中,通过线线同源重组,实现了对目的DNA片段的直接克隆(Direct Cloning)[22](图 3D)。
在进行DNA同源重组时,recA突变的大肠杆菌有利于质粒的稳定,但是Wang等发现在电转化前瞬时表达RecA可以提高电击后细胞的存活率,从而提高重组效率[54]。
3.2 直接克隆技术的发展2012年,Fu等利用全长的RecE和RecT,加上Redγ和RecA构建了一个新的直接克隆系统,对Photorhabdus luminescens中的10个次级代谢基因簇进行了直接克隆并在异源宿主中尝试表达,建立了一个通过直接克隆和异源表达进行生物活性次级代谢产物挖掘的新方法[47]。2015年,Wang等创建了RecET直接克隆和Red重组技术的一体化方案,在温敏型质粒上通过阿拉伯糖诱导表达RecET重组酶,在染色体上通过鼠李糖诱导表达Red重组酶,并对克隆载体和基因平行转移元件进行了标准化,构建了一个系统的工具盒,极大地便利了直接克隆技术在微生物基因组挖掘中的应用[55]。RecET介导的直接克隆技术避免了基因文库的构建和筛选,省时省力,但是对于原核基因组中的大于50 kb的DNA片段或者真核基因组中特定基因的克隆效率不高。2018年,Wang等在RecET直接克隆技术的基础上开发了ExoCET技术[56]。ExoCET技术的机理是首先利用T4 DNA聚合酶的核酸外切酶活性,在体外对2个dsDNA的末端进行降解,形成ssDNA并退火,随后在体内,通过RecET同源重组形成重组子,实现复杂基因组的高效直接克隆。在此过程中,虽然可以形成完全环化分子的数量是有限的,但是部分分子之间可以实现一端退火,把2个分子组装成一个分子,突破了2个分子共转化效率低的瓶颈。目前,T4 DNA聚合酶和RecET的协同作用是在大肠杆菌中进行直接克隆最高效的方法[56]。
3.3 DNA多片段组装技术Wang等开发的ExoCET技术还可以用于对多个DNA片段进行组装。与酿酒酵母的TAR克隆技术相比,ExoCET技术只需要3 d时间就能实现多个DNA片段的组装,而TAR克隆则需要至少2周;与商业化的Gibson克隆技术相比,ExoCET技术可以一次性将20个DNA片段组装成一个50 kb的质粒,而Gibson则最多只能组装5个DNA片段;而且ExoCET对于DNA序列的GC含量和重复序列不敏感,适应性更好[56]。Song等利用ExoCET技术对多杀菌素的生物合成基因簇进行人工设计以及体外多片段组装,人工重构的多杀菌素基因簇在异源宿主中的表达产量显著提高[57]。
3.4 DNA无痕修饰技术联合使用反向筛选和同源重组工程可以对DNA序列进行无痕修饰,包括敲除、替换和点突变等。本文主要介绍利用sacB、rpsL和ccdB反向筛选标记基因结合同源重组工程进行无痕修饰技术的发展。
来自Bacillus subtilis的sacB基因是最早用于同源重组工程的反向筛选标记基因[15, 24]。在E. coli细胞内,sacB基因编码的胞外果聚糖蔗糖转移酶可以将蔗糖水解成葡萄糖和果聚糖,而果聚糖的富集对细菌会产生毒性。因为在没有蔗糖的培养基中,SacB已经对细菌产生一定的毒性,从而导致sacB基因或者细菌染色体DNA的自发突变来逃逸毒性,因此产生大量选择本底。很多基因文库是用pBACe3.6载体和pTARBAC载体构建,其本身含有sacB基因,因此,sacB不能用来修饰这些BAC[24]。
rpsL基因编码的核糖体蛋白S12亚基是链霉素的作用靶标。2011年,Bird等在E. coli细胞内利用rpsL-neo对BAC进行反向筛选修饰的时候,发现Redα的表达增加了分子内重复序列之间的重组概率,造成极高的筛选本底,而且仅表达Redβ和Redγ,没有Redα也可以较低效率地完成dsDNA(rpsL-neo PCR)和BAC的重组;继而在仅表达Redβ的情况下,用ssDNA(合成的寡核苷酸)替换BAC上的反向筛选元件,可以有效解决重复序列的影响[58]。但是rpsL反向标记本身也具有3个缺点:(1)链霉素对光敏感导致选择不良,在选择的菌落下常有本底生长;(2)由于其需要伴随正向选择抗性基因,因此在插入反向选择基因组件后,需要对反向选择基因进行功能检测,排除突变;(3)rpsL基因的自然突变概率为10-6,平板选择有明显的时间窗口,没有经验的技术人员往往会错过最佳时间。
2014年,Wang等将ccdB反向筛选与同源重组工程相结合,建立了新型无痕修饰系统,可对多拷贝质粒、BAC和E. coli染色体等进行点突变和模块替换等无痕修饰[59]。ccdB反向筛选具有的良好选择性源于其编码的CcdB毒性蛋白能够攻击DNA旋转酶A亚单位基因(gyrA),从而阻止DNA的复制,并且该系统不需要任何特殊的培养基或者试剂。因此,基于ccdB反向筛选与同源重组工程相结合的DNA无痕修饰技术,为科学工作者对DNA分子进行点突变和模块替换等修饰提供了有力的工具。然而有些DNA存在大量重复序列,如聚酮类和非核糖体多肽类化合物的生物合成基因簇,导致利用上述无痕修饰技术进行基因点突变和结构域替换等操作极具挑战性。2020年,Song等开发了RedEx基因编辑技术,该技术是将Redαβ蛋白介导的线环重组、CcdB介导的反向筛选和核酸外切酶介导的体外同源重组结合到一起:首先,使用Redαβ蛋白介导线环重组,将设计的DNA片段与侧翼有独特限制性位点的正反向选择标记(抗生素抗性基因和ccdB反向筛选标记基因)一起插入基因簇的靶位点;然后,用限制性内切酶将载体线性化,以暴露两端20 bp同源臂;最后,通过核酸外切酶介导的体外退火,将末端重叠的线性DNA分子环化[60]。Song等利用该技术成功对绿色生物农药多杀菌素聚酮合酶基因进行了异源结构域插入,获得了表达杀虫性能更好的丁烯基多杀菌素的生物合成基因簇[60]。
4 噬菌体同源重组酶在其他细菌中的应用和开发来源于Lambda噬菌体的同源重组酶Redα/Redβ或来源于Rac原噬菌体的同源重组酶RecE/RecT,不仅能在E. coli中介导高效率的同源重组,而且能在与E. coli亲缘关系较近的菌株中发挥一定的重组功能(表 1)。
| Species | Proteins | References | |||
| 5′→3′ exonuclease | ssDNA annealing protein | Host exonuclease inhibitor | ssDNA binding protein | ||
| Shigella flexneri | Redα(phage lambda) | Redβ(phage lambda) | Redγ(phage lambda) | [61] | |
| Yersinia pseudotuberculosis | Redα(phage lambda) | Redβ(phage lambda) | Redγ(phage lambda) | [62] | |
| Serratia marcescens | Redα(phage lambda) | Redβ(phage lambda) | Redγ(phage lambda) | [63] | |
| Salmonella enterica | Redα(phage lambda) | Redβ(phage lambda) | Redγ(phage lambda) | [64] | |
| Pseudomonas aeruginosa | Redα(phage lambda) | Redβ(phage lambda) | Redγ(phage lambda) | [65] | |
| Burkholderia thailandensis | Redα(phage lambda) | Redβ(phage lambda) | Redγ(phage lambda) | [66] | |
| B. pseudomallei | Redα(phage lambda) | Redβ(phage lambda) | Redγ(phage lambda) | [66] | |
| Klebsiella pneumoniae | Redα(phage lambda) | Redβ(phage lambda) | Redγ(phage lambda) | [68] | |
| Agrobacterium tumefaciens | Redα(phage lambda) | Redβ(phage lambda) | Redγ(phage lambda) | [68] | |
| Escherichia albertii | Redα(phage lambda) | Redβ(phage lambda) | Redγ(phage lambda) | [69] | |
| Mycobacterium tuberculosis | Gp60(phage Che9c) | Gp61(phage Che9c) | [70-71] | ||
| Pseudomonas syringae | RecEPsy(P. syringae) | RecTPsy(P. syringae) | [72] | ||
| Lactococcus reuteri | RecTl(L. reuteri) | [73-74] | |||
| Lactococcus lactis | RecTl(L. reuteri) | [73-74] | |||
| Clostridium acetobutylicum | Cpf0939 (C. perfringens) | [75] | |||
| Bacillus subtilis | GP35(phage SSP1) | [76] | |||
| Photorhabdus luminescens | Plu2936(P. luminescens) | Plu2935(P. luminescens) | Plu2934(P. luminescens) | [77] | |
| Xenorhabdus stockiae | Plu2936(P. luminescens) | Plu2935(P. luminescens) | Plu2934(P. luminescens) | [77] | |
| Lactobacillus plantarum | Lp_0642(L. plantarum) | Lp_0641(L. plantarum) | Lp_0640(L. plantarum) | [78-79] | |
| Schlegelella brevitalea | Redα7029(S. brevitalea) | Redβ7029(S. brevitalea) | [80-81] | ||
| Pseudomonas aeruginosa | A(phage Ab31) | B(phage Ab31) | Redγ(phage lambda) | S(phage Ab31) | [82] |
| Pseudomonas putida | A(phage Ab31) | B(phage Ab31) | Redγ(phage lambda) | S(phage Ab31) | [82] |
| Pseudomonas fluorescens | A(phage Ab31) | B(phage Ab31) | Redγ(phage lambda) | S(phage Ab31) | [82] |
| Pseudomonas syringae | A(phage Ab31) | B(phage Ab31) | Redγ(phage lambda) | S(phage Ab31) | [82] |
| Burkholderia glumae | RecEYI23 (B. cordobensis YI23) | RecTYI23 (B. cordobensis YI23) | [83] | ||
| Burkholderia plantarii | RecEYI23 (B. cordobensis YI23) | RecTYI23 (B. cordobensis YI23) | [83] | ||
2003年,胡堃等将表达Lambda Red重组系统的质粒转入Shigella flexneri中,并且利用带有50 bp同源臂的抗性基因替换掉其染色体上的alkA基因[61]。2003年,Derbise等将Lambda Red重组系统转入Yersinia pseudotuberculosis中,利用带有500 bp同源臂的抗性基因失活了其染色体上的目的基因[62]。利用Lambda Red重组系统进行遗传操作的菌株还涉及Serratia marcescens[63]、Salmonella enterica Serovar Typhimurium[64]、Pseudomonas aeruginosa[65]、Burkholderia thailandensis[66]、B. pseudomallei[66]、Klebsiella pneumoniae[67]、Agrobacterium tumefaciens[68]以及Escherichia albertii[68]。
Lambda Red重组系统可以在与E. coli亲缘关系比较近的菌株中进行重组,但是在与E. coli亲缘关系较远的菌株中一般不能发挥重组功能。因此,寻找细菌本身的噬菌体同源重组酶,并建立其自身同源重组系统进而开发具有种属特异性的同源重组工程成为近年来的研究趋势(表 1)。
2007年,Van Kessel等在Mycobacterium tuberculosis的噬菌体Che9c基因组中找到了RecE/RecT同源蛋白对Gp60/Gp61,并在此基础上建立了结合分枝杆菌的同源重组工程系统[70-71]。用同样的方法开发了重组系统的菌株,涉及P. syringae pv. tomato DC3000[72]、Lactococcus reuteri[73-74]、L. lactis[73-74]、Clostridium acetobutylicum[75]、B. subtilis[76]、Photorhabdus luminescens[77]、Xenorhabdus stockiae[77]、Lactobacillus plantarum WCFS1[78-79]、Schlegelella brevitalea DSM 7029[80-81]、P. aeruginosa[82]、B. glumae[83]和B. plantarii[83]。
5 基于噬菌体同源重组酶的DNA重组工程技术的应用 5.1 噬菌体同源重组酶在微生物中用于次级代谢产物的挖掘微生物次级代谢产物一直以来都是新药发现的重要来源。近年来,随着基因测序技术、基因组学和生物信息学的快速发展,在许多微生物的基因组中发现了大量沉默次级代谢产物的生物合成基因簇(Biosynthetic Gene Clusters,BGCs),其在实验室生长条件下无法表达或者表达量很低。因此,通过基因组挖掘来发现更多新颖的微生物次级代谢产物用于新药先导化合物的筛选成为一个研究热点。对于沉默生物合成基因簇的基因组挖掘主要有2个策略:第1个是在原宿主菌中靶向激活沉默生物合成基因簇,这种策略的前提是原宿主菌具有遗传操作系统或者能够在原宿主菌中构建一套遗传操作系统,然后通过启动子和调控元件修饰将沉默生物合成基因簇进行激活表达;第2个是将沉默生物合成基因簇克隆至载体上,然后将其稳定地转移到一个合适的异源底盘宿主中进行表达(图 4)。
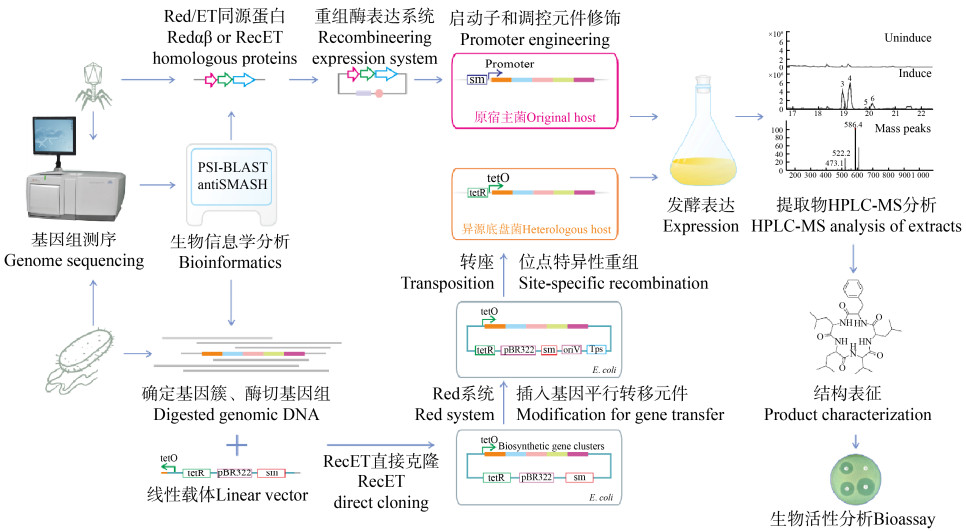
|
| 图 4 微生物沉默生物合成基因簇的基因组挖掘策略 Figure 4 Strategy of genome mining of silent biosynthetic gene clusters of microorganisms |
|
|
2015年,Yin等在P. luminescens和X. stockiae中构建了一套基于其原噬菌体同源重组酶的重组工程系统,并利用该系统在非核糖体多肽合成酶基因簇plu2670的前面插入了四环素诱导型启动子,进而成功地激活了该基因簇[77]。2018年,Wang等利用在Schlegelella brevitalea DSM 7029中构建的同源重组工程系统成功地激活了5个沉默的生物合成基因簇[81]。Li等在Burkholderia中构建了一套同源重组工程系统,并且利用该系统在伯克氏菌中成功激活了3个生物合成基因簇,并鉴定得到了一系列新的脂肽类化合物[83]。在细菌中构建同源重组工程系统,并将其应用于同属的菌株中对沉默生物合成基因簇进行激活,从而发现新的次级代谢产物已经成为基因组挖掘的一个新的研究方向(图 4)。
目前,还有很多微生物没有可以利用的遗传操作系统,或者有的微生物存在培养非常困难的问题。因此,通过基因组序列分析发现的新的沉默生物合成基因簇只能通过异源表达的方式进行激活。此策略主要是通过直接克隆或者化学合成的方式得到生物合成基因簇,然后将生物合成基因簇克隆到表达载体上,并将其稳定转移到合适的底盘宿主中进行表达。Fu等报道了全长RecE和RecT能够对较大的DNA片段进行直接克隆,利用这一技术,Fu等从发光杆菌中直接克隆得到了10个沉默基因簇(10-50 kb),其中2个基因簇plu3263(15.6 kb)和plu1881–plu1877(18.3 kb)在大肠杆菌中实现了异源表达,得到了Luminmides和Luminmycin这2种化合物[47]。对于比较大的基因簇,可以采取分段克隆的手段先得到较小的片段,然后再利用同源重组工程将其组装到一个表达载体上。例如,Yin等首先利用直接克隆技术从白色链霉菌(Streptomyces albus)DSM41398中得到盐霉素生物合成基因簇(106 kb)的3个片段(F1,43 kb;F2,33 kb;F3,30 kb),然后利用DNA组装技术将3个片段组装到一个表达载体上,最终得到了盐霉素的完整生物合成基因簇并在天蓝色链霉菌(S. coelicolor)A3(2)中实现了异源表达[85]。Xu等在济南游动放线菌(Actinoplanes tsinanensis)中利用同源重组工程克隆了创新霉素的生物合成基因簇(11 kb),并且在天蓝色链霉菌(S. coelicolor)A3(2)中实现了异源表达[86]。在直接克隆技术开发之前,获得大片段次级代谢产物生物合成基因簇的DNA需要柯斯质粒文库的构建,然后通过Red重组系统缝合2个柯斯质粒,在一个载体上获得完整的基因簇,从而完成DNA平行转移和异源表达[87-89],整个工作费时费力。
同源重组工程能够用于微生物中次级代谢产物的挖掘,通过对生物合成基因簇进行改造,尤其是启动子工程改造,可以极大地提高次级代谢产物的产量[90]。除此之外,利用同源重组工程开发的无痕修饰技术可以对次级代谢产物生物合成基因簇的模块、结构域进行替换或者点突变,改变次级代谢产物的结构,发掘新的衍生物[60]。
5.2 动物和植物转基因载体的构建在人类基因组计划完成以后,生命科学进入后基因组时代,其以揭示蛋白编码基因的功能以及调控机制为主要目标,其中重要的技术手段就是构建基因修饰的小鼠模型。基因打靶是定向改变生物活体遗传信息的技术,包括基因失活、缺失突变和点突变及外源基因定点引入等,使修饰后的遗传信息在生物活体内遗传并表达突变性状,从而研究基因功能,并提供相关疾病治疗和药物筛选评价模型[91]。目前,在胚胎干细胞中进行同源重组的技术已于2007年获得诺贝尔生理学或医学奖,该技术首先需要在大肠杆菌内构建基因打靶载体[92],目前已经成为对小鼠染色体进行基因打靶的常规技术。
2003年,Testa等首先利用噬菌体重组酶介导的同源重组工程构建基因打靶载体,获得了混合性白血病Mll基因的条件敲除小鼠[18]。2010年,Fu等建立了利用同源重组工程高通量构建条件基因打靶载体的技术流程,从含有小鼠基因的BAC修饰开始到获得最终的基因打靶载体(图 5),需要4步Redα/Redβ介导的DNA同源重组,整个实验可以在9 d内完成,重组子的选择在液体培养基里进行,因此可以扩展到96孔板的高通量模式[53]。欧洲条件性基因突变小鼠项目(European Conditional Mouse Mutagenesis Consortium,EUCOMM)利用该技术,构建了12 000个基因打靶载体,获得了9 000个条件性基因突变细胞系,建立了小鼠胚胎干细胞条件敲除突变资源库[93]。2016年,Baker等利用RecET介导的同源重组工程,一步将4个片段或者5个片段的DNA组装成一个质粒,快速构建基因打靶载体[94]。2017年,Baker等利用Red同源重组工程构建鼠源序列和人源序列嵌合BAC载体,在Cas9人工核酸酶的辅助下进行基因打靶,实现了42 kb人源基因序列和小鼠同源基因序列的原位替换,构建了KMT2D人源化小鼠模型[95]。
人和小鼠的BAC文库是含有人或小鼠基因随机片段的重组DNA克隆群体,BAC克隆一般插入150-220 kb的外源DNA,对于大部分人或小鼠基因,可以筛选到含有完整编码序列以及调控元件的BAC克隆,是转基因技术的重要资源。2012年,Rostovskaya等利用Redα/Redβ介导的DNA同源重组将PiggyBac和Sleeping Beauty转座酶识别的反向重复序列(Inverted Repeats,IR)精准插入BAC载体,将修饰的BAC载体与转座酶表达质粒共转染,首次实现人胚胎干细胞的BAC转基因[96]。利用BAC转座载体与PiggyBac转座酶mRNA一起进行小鼠受精卵的原核显微注射,可以快速获得转基因小鼠[97]。
对于植物转基因,噬菌体重组酶介导的DNA同源重组工程对于高效构建转基因载体也发挥了积极的作用。2020年,Brumos等开发了用于高通量植物转基因载体构建的DNA同源重组工程工具盒,他们以BAC载体为基础,利用Redα/Redβ介导的DNA同源重组,对62个与生长素相关的基因融合了蛋白标签,随后通过Flp介导的盒式交换技术(Recombinase Mediated Cassette Exchange,RMCE)将BAC载体替换为双元载体(Binary Vectors),最终通过农杆菌介导的植物转基因技术获得拟南芥突变株(图 6)[98]。
5.3 病毒基因组的克隆和修饰病毒反向遗传学是采用病毒的遗传材料在培养的细胞中重新拯救出活病毒,该技术一般需要在细菌质粒中克隆病毒基因组的DNA拷贝或者cDNA序列,构建感染性克隆能够用于病毒基因功能研究,开发病毒载体疫苗或者构建基因治疗载体。
病毒基因组的大小可以超过200 kb,例如疱疹病毒的基因组在105-235 kb之间。DNA同源重组工程成功突破了大型病毒基因组修饰的局限性,牛痘病毒、疱疹病毒和杆状病毒的基因组在BAC上可以直接修饰。随后,将BAC转染到哺乳动物细胞中进行病毒拯救,这样可以直接获得纯净的重组病毒,然而在细胞内通过基因打靶获得的重组病毒需要烦琐的纯化步骤。
2017年,Zhang等[98]利用RecET直接克隆技术构建了首个人腺病毒DNA文库,包括32个基因型。RecET重组酶可以将完整的人腺病毒DNA直接克隆在p15A质粒上,利用Red重组酶在E3区克隆CAG启动子表达绿色荧光蛋白和荧光素酶基因,拯救出带有双报告基因的病毒,感染小鼠并进行活体成像,示踪不同基因型病毒的组织特异性[99](图 7)。由于RecET直接克隆的高效性,可以从临床样本中直接将腺病毒DNA克隆到p15A质粒上进行分析,为快速病例诊断提供了新方法[99]。
对噬菌体的遗传改造不仅是为了研究其基因功能,开发人工噬菌体用于细菌的精准控制、检测和诊断,作为人、动物、植物及食物的生物抑菌剂,而且人工噬菌体可以超越其抗菌作用,作为药物的载体或者疫苗。噬菌体的遗传操作包括体外改造和体内改造,其中体外改造包括人工合成全长噬菌体核酸[100]、在酵母里进行噬菌体基因的组装[101],以及体外转录和翻译的无细胞体系里进行噬菌体的同源重组[102]。噬菌体的体内改造可以利用细菌自身的同源重组系统[103],CRISPR-Cas系统可以作为反向筛选降解未被遗传修饰的噬菌体[104],来源于噬菌体的重组酶也被用来进行噬菌体的同源重组。
利用噬菌体重组酶介导的同源重组工程对噬菌体进行遗传改造,目前主要是针对大肠杆菌和沙门氏菌的噬菌体,其方法是将RecE/RecT或Redα/Redβ在细菌里进行调控表达,然后将噬菌体DNA和用于同源重组的DNA共同电转化。2012年,Fehér等利用RecE/RecT同源重组工程删除P1噬菌体的转座子IS1,发现删除IS1的噬菌体生物学特性没有改变,从而构建了一个没有潜在转座子污染的大肠杆菌基因工程载体[105]。2014年,Shin等用RecE/RecT同源重组工程对沙门氏菌温和噬菌体SPN9CC进行遗传修饰,发现cl敲除的噬菌体由温和转为烈性,为人工将温和噬菌体改造成用于生物防治的烈性噬菌体提供了新方法[106]。2004年,Oppenheim等应用Redα/Redβ重组系统电转化人工合成的寡核苷酸,通过80个核苷酸的ssDNA在Lambda噬菌体上实现了点突变,同时也尝试了基因删除和置换,验证了Red重组系统对于噬菌体修饰的有效性[107]。
6 总结基于噬菌体同源重组酶Redα/Redβ和RecE/RecT开发的DNA同源重组工程在1998年被开发出来,是分子生物学技术的一个里程碑。该技术能够对靶标DNA分子进行快速、精准、高效的修饰,包括直接克隆、亚克隆、插入、删除、替换、点突变等,具有不受限制性内切酶识别位点和DNA分子大小限制的优势。同源重组工程不仅仅在大肠杆菌中对DNA进行修饰,在其他细菌中也开发了基于具有种属特异性的噬菌体同源重组酶的高效遗传操作系统,促进了生物功能基因的研究。
近年来,DNA同源重组工程延伸开发了一系列技术,包括可以对DNA大片段进行直接克隆以及可以用于多片段DNA组装的ExoCET技术,可以对DNA尤其是具有大量重复序列的生物合成基因簇进行无痕修饰的RedEx基因编辑技术等。这为微生物基因组挖掘以及次级代谢产物生物合成机理的研究提供了有力的工具。作为分子生物学源头技术,同源重组工程在生命科学领域的方方面面都发挥了重要作用,尤其是在动植物转基因以及病毒基因组克隆和修饰方面取得了显著成效。
| [1] |
Groth AC, Calos MP. Phage integrases: biology and applications[J]. Journal of Molecular Biology, 2004, 335(3): 667-678. DOI:10.1016/j.jmb.2003.09.082 |
| [2] |
Smith MCM, Thorpe HM. Diversity in the serine recombinases[J]. Molecular Microbiology, 2002, 44(2): 299-307. DOI:10.1046/j.1365-2958.2002.02891.x |
| [3] |
Esposito D, Scocca JJ. The integrase family of tyrosine recombinases: evolution of a conserved active site domain[J]. Nucleic Acids Research, 1997, 25(18): 3605-3614. DOI:10.1093/nar/25.18.3605 |
| [4] |
Enquist LW, Kikuchi A, Weisberg RA. The role of λ integrase in integration and excision[J]. Cold Spring Harbor Symposia on Quantitative Biology, 1979, 43: 1115-1120. DOI:10.1101/SQB.1979.043.01.124 |
| [5] |
Austin S, Ziese M, Sternberg N. A novel role for site-specific recombination in maintenance of bacterial replicons[J]. Cell, 1981, 25(3): 729-736. DOI:10.1016/0092-8674(81)90180-X |
| [6] |
Anastassiadis K, Fu J, Patsch C, Hu SB, Weidlich S, Duerschke K, Buchholz F, Edenhofer F, Stewart AF. Dre recombinase, like Cre, is a highly efficient site-specific recombinase in E. coli, mammalian cells and mice[J]. Disease Models & Mechanisms, 2009, 2(9/10): 508-515. |
| [7] |
Broach JR, Guarascio VR, Jayaram M. Recombination within the yeast plasmid 2μ circle is site-specific[J]. Cell, 1982, 29(1): 227-234. DOI:10.1016/0092-8674(82)90107-6 |
| [8] |
Colloms SD, Sykora P, Szatmari G, Sherratt DJ. Recombination at ColE1 cer requires the Escherichia coli xerC gene product, a member of the lambda integrase family of site-specific recombinases[J]. Journal of Bacteriology, 1990, 172(12): 6973-6980. DOI:10.1128/jb.172.12.6973-6980.1990 |
| [9] |
Kuhstoss S, Rao RN. Analysis of the integration function of the streptomycete bacteriophage φC31[J]. Journal of Molecular Biology, 1991, 222(4): 897-908. DOI:10.1016/0022-2836(91)90584-S |
| [10] |
Zhang L, Ou XJ, Zhao GP, Ding XM. Highly efficient in vitro site-specific recombination system based on Streptomyces phage phiBT1 integrase[J]. Journal of Bacteriology, 2008, 190(19): 6392-6397. DOI:10.1128/JB.00777-08 |
| [11] |
Krasnow MA, Cozzarelli NR. Site-specific relaxation and recombination by the Tn3 resolvase: recognition of the DNA path between oriented res sites[J]. Cell, 1983, 32(4): 1313-1324. DOI:10.1016/0092-8674(83)90312-4 |
| [12] |
Katzen F. Gateway® recombinational cloning: a biological operating system[J]. Expert Opinion on Drug Discovery, 2007, 2(4): 571-589. DOI:10.1517/17460441.2.4.571 |
| [13] |
Long DP, Tan B, Zhao AC, Xu LX, Xiang ZH. Progress in Cre/lox site-specific recombination system in higher eukaryotes[J]. Hereditas, 2012, 34(2): 177-189. (in Chinese) 龙定沛, 谭兵, 赵爱春, 许龙霞, 向仲怀. Cre/lox位点特异性重组系统在高等真核生物中的研究进展[J]. 遗传, 2012, 34(2): 177-189. |
| [14] |
Thomason LC, Calendar R, Ow DW. Gene insertion and replacement in Schizosaccharomyces pombe mediated by the Streptomyces bacteriophage phiC31 site-specific recombination system[J]. Molecular Genetics and Genomics, 2001, 265(6): 1031-1038. DOI:10.1007/s004380100498 |
| [15] |
Zhang YM, Buchholz F, Muyrers JPP, Francis Stewart A. A new logic for DNA engineering using recombination in Escherichia coli[J]. Nature Genetics, 1998, 20(2): 123-128. DOI:10.1038/2417 |
| [16] |
Muyrers JPP, Zhang YM, Testa G, Stewart AF. Rapid modification of bacterial artificial chromosomes by ET-recombination[J]. Nucleic Acids Research, 1999, 27(6): 1555-1557. DOI:10.1093/nar/27.6.1555 |
| [17] |
Zhang Y, Yin YB, Chen YJ, Gao G, Yu P, Luo JC, Jiang Y. PCAS: a precomputed proteome annotation database resource[J]. BMC Genomics, 2003, 4(1): 42. DOI:10.1186/1471-2164-4-42 |
| [18] |
Testa G, Zhang YM, Vintersten K, Benes V, Pijnappel WWMP, Chambers I, Smith AJH, Smith AG, Stewart AF. Engineering the mouse genome with bacterial artificial chromosomes to create multipurpose alleles[J]. Nature Biotechnology, 2003, 21(4): 443-447. DOI:10.1038/nbt804 |
| [19] |
Muyrers JPP, Zhang YM, Stewart AF. Techniques: recombinogenic engineering-new options for cloning and manipulating DNA[J]. Trends in Biochemical Sciences, 2001, 26(5): 325-331. DOI:10.1016/S0968-0004(00)01757-6 |
| [20] |
Ellis HM, Yu D, DiTizio T, Court DL. High efficiency mutagenesis, repair, and engineering of chromosomal DNA using single-stranded oligonucleotides[J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(12): 6742-6746. DOI:10.1073/pnas.121164898 |
| [21] |
Copeland NG, Jenkins NA, Court DL. Recombineering: a powerful new tool for mouse functional genomics[J]. Nature Reviews Genetics, 2001, 2(10): 769-779. DOI:10.1038/35093556 |
| [22] |
Zhang YM, Muyrers JPP, Testa G, Francis Stewart A. DNA cloning by homologous recombination in Escherichia coli[J]. Nature Biotechnology, 2000, 18(12): 1314-1317. DOI:10.1038/82449 |
| [23] |
Muyrers JP, Zhang Y, Buchholz F, Stewart AF. RecE/RecT and Redαlpha/Redbeta initiate double-stranded break repair by specifically interacting with their respective partners[J]. Genes & Development, 2000, 14(15): 1971-1982. |
| [24] |
Muyrers JP, Zhang Y, Benes V, Testa G, Ansorge W, Stewart AF. Point mutation of bacterial artificial chromosomes by ET recombination[J]. EMBO Reports, 2000, 1(3): 239-243. DOI:10.1093/embo-reports/kvd049 |
| [25] |
Shulman MJ, Hallick LM, Echols H, Signer ER. Properties of recombination-deficient mutants of bacteriophage lambda[J]. Journal of Molecular Biology, 1970, 52(3): 501-520. DOI:10.1016/0022-2836(70)90416-X |
| [26] |
Remaut E, Stanssens P, Fiers W. Plasmid vectors for high-efficiency expression controlled by the PL promoter of coliphage lambda[J]. Gene, 1981, 15(1): 81-93. DOI:10.1016/0378-1119(81)90106-2 |
| [27] |
Little JW, Lehman IR, Kaiser AD. An exonuclease induced by bacteriophage Λ[J]. Journal of Biological Chemistry, 1967, 242(4): 672-678. DOI:10.1016/S0021-9258(18)96257-5 |
| [28] |
Masamune Y, Fleischman RA, Richardson CC. Enzymatic removal and replacement of nucleotides at single strand breaks in deoxyribonucleic acid[J]. The Journal of Biological Chemistry, 1971, 246(8): 2680-2691. DOI:10.1016/S0021-9258(18)62340-3 |
| [29] |
Zhang JJ, McCabe KA, Bell CE. Crystal structures of lambda exonuclease in complex with DNA suggest an electrostatic ratchet mechanism for processivity[J]. Proceedings of the National Academy of Academy of Sciences of the United States of America, 2011, 108(29): 11872-11877. |
| [30] |
Subramanian K, Rutvisuttinunt W, Scott W, Myers RS. The enzymatic basis of processivity in lambda exonuclease[J]. Nucleic Acids Research, 2003, 31(6): 1585-1596. DOI:10.1093/nar/gkg266 |
| [31] |
Van Oijen AM. Single-molecule kinetics of exonuclease reveal base dependence and dynamic disorder[J]. Science, 2003, 301(5637): 1235-1238. DOI:10.1126/science.1084387 |
| [32] |
Perkins TT, Dalal RV, Mitsis PG, Block SM. Sequence-dependent pausing of single lambda exonuclease molecules[J]. Science, 2003, 301(5641): 1914-1918. DOI:10.1126/science.1088047 |
| [33] |
Karakousis G, Ye N, Li Z, Chiu SK, Reddy G, Radding CM. The beta protein of phage λ binds preferentially to an intermediate in DNA renaturation[J]. Journal of Molecular Biology, 1998, 276(4): 721-731. DOI:10.1006/jmbi.1997.1573 |
| [34] |
Venkatesh TV, Radding CM. Ribosomal protein S1 and NusA protein complexed to recombination protein beta of phage lambda[J]. Journal of Bacteriology, 1993, 175(6): 1844-1846. DOI:10.1128/jb.175.6.1844-1846.1993 |
| [35] |
Caldwell BJ, Zakharova E, Filsinger GT, Wannier TM, Hempfling JP, Chun-Der L, Pei DH, Church GM, Bell CE. Crystal structure of the Redβ C-terminal domain in complex with λ exonuclease reveals an unexpected homology with λ Orf and an interaction with Escherichia coli single stranded DNA binding protein[J]. Nucleic Acids Research, 2019, 47(4): 1950-1963. DOI:10.1093/nar/gky1309 |
| [36] |
Filsinger GT, Wannier TM, Pedersen FB, Lutz ID, Zhang JL, Stork DA, Debnath A, Gozzi K, Kuchwara H, Volf V, et al. Characterizing the portability of phage-encoded homologous recombination proteins[J]. Nature Chemical Biology, 2021, 17(4): 394-402. DOI:10.1038/s41589-020-00710-5 |
| [37] |
Passy SI, Yu X, Li Z, Radding CM, Egelman EH. Rings and filaments of protein from bacteriophage suggest a superfamily of recombination proteins[J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(8): 4279-4284. DOI:10.1073/pnas.96.8.4279 |
| [38] |
Erler A, Wegmann S, Elie-Caille C, Bradshaw CR, Maresca M, Seidel R, Habermann B, Muller DJ, Stewart AF. Conformational adaptability of Redbeta during DNA annealing and implications for its structural relationship with Rad52[J]. Journal of Molecular Biology, 2009, 391(3): 586-598. DOI:10.1016/j.jmb.2009.06.030 |
| [39] |
Caldwell BJ, Norris A, Zakharova E, Smith CE, Wheat CT, Choudhary D, Sotomayor M, Wysocki VH, Bell CE. Oligomeric complexes formed by Redβ single strand annealing protein in its different DNA bound states[J]. Nucleic Acids Research, 2021, 49(6): 3441-3460. DOI:10.1093/nar/gkab125 |
| [40] |
Taylor A, Smith GR. Unwinding and rewinding of DNA by the RecBC enzyme[J]. Cell, 1980, 22(2): 447-457. DOI:10.1016/0092-8674(80)90355-4 |
| [41] |
Murphy KC. Lambda Gam protein inhibits the helicase and Chi-stimulated recombination activities of Escherichia coli RecBCD enzyme[J]. Journal of Bacteriology, 1991, 173(18): 5808-5821. DOI:10.1128/jb.173.18.5808-5821.1991 |
| [42] |
Murphy KC. The λ gam protein inhibits RecBCD binding to dsDNA ends[J]. Journal of Molecular Biology, 2007, 371(1): 19-24. DOI:10.1016/j.jmb.2007.05.085 |
| [43] |
Paškvan I, Salaj-Šmic E, Ivančić-Baće I, Zahradka K, Trgovčević Ž, Brčić-Kostić K. The genetic dependence of RecBCD-Gam mediated double strand end repair in Escherichia coli[J]. FEMS Microbiology Letters, 2001, 205(2): 299-303. DOI:10.1111/j.1574-6968.2001.tb10964.x |
| [44] |
Barbour SD, Nagaishi H, Templin A, Clark AJ. Biochemical and genetic studies of recombination proficiency in Escherichia coli, II. rec+ revertants caused by indirect suppression of rec-mutations[J]. Proceedings of the National Academy of Sciences of the United States of America, 1970, 67(1): 128-135. DOI:10.1073/pnas.67.1.128 |
| [45] |
Joseph JW, Kolodner R. Exonuclease VIII of Escherichia coli. II. Mechanism of action[J]. Journal of Biological Chemistry, 1983, 258(17): 10418-10424. DOI:10.1016/S0021-9258(17)44473-5 |
| [46] |
Chang HW, Julin DA. Structure and function of the Escherichia coli RecE protein, a member of the RecB nuclease domain family[J]. Journal of Biological Chemistry, 2001, 276(49): 46004-46010. DOI:10.1074/jbc.M108627200 |
| [47] |
Fu J, Bian XY, Hu S, Wang HL, Huang F, Seibert PM, Plaza A, Xia LQ, Müller R, Francis Stewart A, et al. Full-length RecE enhances linear-linear homologous recombination and facilitates direct cloning for bioprospecting[J]. Nature Biotechnology, 2012, 30(5): 440-446. DOI:10.1038/nbt.2183 |
| [48] |
Thresher RJ, Makhov AM, Hall SD, Kolodner R, Griffith JD. Electron microscopic visualization of RecT protein and its complexes with DNA[J]. Journal of Molecular Biology, 1995, 254(3): 364-371. DOI:10.1006/jmbi.1995.0623 |
| [49] |
Noirot P, Gupta RC, Radding CM, Kolodner RD. Hallmarks of homology recognition by RecA-like recombinases are exhibited by the unrelated Escherichia coli RecT protein[J]. The EMBO Journal, 2003, 22(2): 324-334. DOI:10.1093/emboj/cdg027 |
| [50] |
Muniyappa K, Radding CM. The homologous recombination system of phage lambda. Pairing activities of beta protein[J]. Journal of Biological Chemistry, 1986, 261(16): 7472-7478. DOI:10.1016/S0021-9258(17)38416-8 |
| [51] |
Maresca M, Erler A, Fu J, Friedrich A, Zhang YM, Stewart AF. Single-stranded heteroduplex intermediates in lambda Red homologous recombination[J]. BMC Molecular Biology, 2010, 11: 54. DOI:10.1186/1471-2199-11-54 |
| [52] |
Lim SI, Min BE, Jung GY. Lagging strand-biased initiation of red recombination by linear double-stranded DNAs[J]. Journal of Molecular Biology, 2008, 384(5): 1098-1105. DOI:10.1016/j.jmb.2008.10.047 |
| [53] |
Fu J, Teucher M, Anastassiadis K, Skarnes W, Stewart AF. A recombineering pipeline to make conditional targeting constructs[J]. Methods in Enzymology, 2010, 477: 125-144. |
| [54] |
Wang JP, Sarov M, Rientjes J, Hu J, Hollak H, Kranz H, Xie Y, Stewart AF, Zhang YM. An improved recombineering approach by adding RecA to λ Red recombination[J]. Molecular Biotechnology, 2006, 32(1): 43-53. DOI:10.1385/MB:32:1:043 |
| [55] |
Wang HL, Li Z, Jia RN, Hou Y, Yin J, Bian XY, Li AY, Müller R, Stewart AF, Fu J, et al. RecET direct cloning and Redαβ recombineering of biosynthetic gene clusters, large operons or single genes for heterologous expression[J]. Nature Protocols, 2016, 11(7): 1175-1190. DOI:10.1038/nprot.2016.054 |
| [56] |
Wang HL, Li Z, Jia RN, Yin J, Li AY, Xia LQ, Yin YL, Müller R, Fu J, Stewart AF, et al. ExoCET: exonuclease in vitro assembly combined with RecET recombination for highly efficient direct DNA cloning from complex genomes[J]. Nucleic Acids Research, 2018, 46(5): e28. DOI:10.1093/nar/gkx1249 |
| [57] |
Song CY, Luan J, Cui QW, Duan QY, Li Z, Gao YS, Li RJ, Li AY, Shen YM, Li YZ, et al. Enhanced heterologous spinosad production from a 79-kb synthetic multioperon assembly[J]. ACS Synthetic Biology, 2019, 8(1): 137-147. DOI:10.1021/acssynbio.8b00402 |
| [58] |
Bird AW, Erler A, Fu J, Hériché JK, Maresca M, Zhang YM, Hyman AA, Francis Stewart A. High-efficiency counterselection recombineering for site-directed mutagenesis in bacterial artificial chromosomes[J]. Nature Methods, 2012, 9(1): 103-109. DOI:10.1038/nmeth.1803 |
| [59] |
Wang HL, Bian XY, Xia LQ, Ding XZ, Müller R, Zhang YM, Fu J, Stewart AF. Improved seamless mutagenesis by recombineering using ccdB for counterselection[J]. Nucleic Acids Research, 2014, 42(5): e37. DOI:10.1093/nar/gkt1339 |
| [60] |
Song CY, Luan J, Li RJ, Jiang CJ, Hou Y, Cui QW, Cui TQ, Tan L, Ma ZC, Tang YJ, et al. RedEx: a method for seamless DNA insertion and deletion in large multimodular polyketide synthase gene clusters[J]. Nucleic Acids Research, 2020, 48(22): e130. DOI:10.1093/nar/gkaa956 |
| [61] |
Hu K, Shi ZX, Wang HL, Feng EL, Huang LY. Study on gene knockout using red system in Shigella flexneri[J]. Acta Microbiologica Sinica, 2003, 43(6): 740-746. (in Chinese) 胡堃, 史兆兴, 王恒樑, 冯尔玲, 黄留玉. Red重组系统在痢疾杆菌基因敲除中的应用研究[J]. 微生物学报, 2003, 43(6): 740-746. DOI:10.3321/j.issn:0001-6209.2003.06.009 |
| [62] |
Derbise A, Lesic B, Dacheux D, Ghigo JM, Carniel E. A rapid and simple method for inactivating chromosomal genes in Yersinia[J]. FEMS Immunology and Medical Microbiology, 2003, 38(2): 113-116. DOI:10.1016/S0928-8244(03)00181-0 |
| [63] |
Rossi MS, Paquelin A, Ghigo JM, Wandersman C. Haemophore-mediated signal transduction across the bacterial cell envelope in Serratia marcescens: the inducer and the transported substrate are different molecules[J]. Molecular Microbiology, 2003, 48(6): 1467-1480. DOI:10.1046/j.1365-2958.2003.03516.x |
| [64] |
Karlinsey JE, Hughes KT. Genetic transplantation: Salmonella enterica serovar typhimurium as a host to study Sigma factor and anti-Sigma factor interactions in genetically intractable systems[J]. Journal of Bacteriology, 2006, 188(1): 103-114. DOI:10.1128/JB.188.1.103-114.2006 |
| [65] |
Lesic B, Rahme LG. Use of the lambda Red recombinase system to rapidly generate mutants in Pseudomonas aeruginosa[J]. BMC Molecular Biology, 2008, 9: 20. DOI:10.1186/1471-2199-9-20 |
| [66] |
Kang Y, Norris MH, Wilcox BA, Tuanyok A, Keim PS, Hoang TT. Knockout and pullout recombineering for naturally transformable Burkholderia thailandensis and Burkholderia pseudomallei[J]. Nature Protocols, 2011, 6(8): 1085-1104. DOI:10.1038/nprot.2011.346 |
| [67] |
Wei D, Wang M, Shi JP, Hao J. Red recombinase assisted gene replacement in Klebsiella pneumoniae[J]. Journal of Industrial Microbiology & Biotechnology, 2012, 39(8): 1219-1226. |
| [68] |
Hu SB, Fu J, Huang F, Ding XZ, Stewart AF, Xia LQ, Zhang YM. Genome engineering of Agrobacterium tumefaciens using the lambda Red recombination system[J]. Applied Microbiology and Biotechnology, 2014, 98(5): 2165-2172. DOI:10.1007/s00253-013-5412-x |
| [69] |
Egan M, Ramirez J, Xander C, Upreti C, Bhatt S. Lambda red-mediated recombineering in the attaching and effacing pathogen Escherichia albertii[J]. Biological Procedures Online, 2016, 18(1): 3. DOI:10.1186/s12575-015-0032-8 |
| [70] |
Van Kessel JC, Hatfull GF. Efficient point mutagenesis in Mycobacteria using single-stranded DNA recombineering: characterization of antimycobacterial drug targets[J]. Molecular Microbiology, 2008, 67(5): 1094-1107. DOI:10.1111/j.1365-2958.2008.06109.x |
| [71] |
Van Kessel JC, Hatfull GF. Recombineering in Mycobacterium tuberculosis[J]. Nature Methods, 2007, 4(2): 147-152. DOI:10.1038/nmeth996 |
| [72] |
Swingle B, Bao ZM, Markel E, Chambers A, Cartinhour S. Recombineering using RecTE from Pseudomonas syringae[J]. Applied and Environmental Microbiology, 2010, 76(15): 4960-4968. DOI:10.1128/AEM.00911-10 |
| [73] |
Van Pijkeren JP, Neoh KM, Sirias D, Findley AS, Britton RA. Exploring optimization parameters to increase ssDNA recombineering in Lactococcus lactis and Lactobacillus reuteri[J]. Bioengineered, 2012, 3(4): 209-217. DOI:10.4161/bioe.21049 |
| [74] |
Van Pijkeren JP, Britton RA. High efficiency recombineering in lactic acid bacteria[J]. Nucleic Acids Research, 2012, 40(10): e76. DOI:10.1093/nar/gks147 |
| [75] |
Dong HJ, Tao WW, Gong FY, Li Y, Zhang YP. A functional recT gene for recombineering of Clostridium[J]. Journal of Biotechnology, 2014, 173: 65-67. DOI:10.1016/j.jbiotec.2013.12.011 |
| [76] |
Sun ZP, Deng AH, Hu T, Wu J, Sun QY, Bai H, Zhang GQ, Wen TY. A high-efficiency recombineering system with PCR-based ssDNA in Bacillus subtilis mediated by the native phage recombinase GP35[J]. Applied Microbiology and Biotechnology, 2015, 99(12): 5151-5162. DOI:10.1007/s00253-015-6485-5 |
| [77] |
Yin J, Zhu HB, Xia LQ, Ding XZ, Hoffmann T, Hoffmann M, Bian XY, Müller R, Fu J, Stewart AF, et al. A new recombineering system for Photorhabdus and Xenorhabdus[J]. Nucleic Acids Research, 2015, 43(6): e36. DOI:10.1093/nar/gku1336 |
| [78] |
Ventura M, Canchaya C, Kleerebezem M, De Vos WM, Siezen RJ, Brüssow H. The prophage sequences of Lactobacillus plantarum strain WCFS1[J]. Virology, 2003, 316(2): 245-255. DOI:10.1016/j.virol.2003.08.019 |
| [79] |
Yang P, Wang J, Qi QS. Prophage recombinases-mediated genome engineering in Lactobacillus plantarum[J]. Microbial Cell Factories, 2015, 14(1): 154. DOI:10.1186/s12934-015-0344-z |
| [80] |
Tang B, Yu YC, Liang JH, Zhang YM, Bian XY, Zhi XY, Ding XM. Reclassification of 'Polyangium brachysporum' DSM 7029 as Schlegelella brevitalea sp. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 2019, 69(9): 2877-2883. DOI:10.1099/ijsem.0.003571 |
| [81] |
Wang X, Zhou HB, Chen HN, Jing XS, Zheng WT, Li RJ, Sun T, Liu JQ, Fu J, Huo LJ, et al. Discovery of recombinases enables genome mining of cryptic biosynthetic gene clusters in Burkholderiales species[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(18): E4255-E4263. DOI:10.1073/pnas.1720941115 |
| [82] |
Yin J, Zheng WT, Gao YS, Jiang CJ, Shi HB, Diao XT, Li SS, Chen HN, Wang HL, Li RJ, et al. Single-stranded DNA-binding protein and exogenous RecBCD inhibitors enhance phage-derived homologous recombination in Pseudomonas[J]. iScience, 2019, 14: 1-14. DOI:10.1016/j.isci.2019.03.007 |
| [83] |
Li RJ, Shi HB, Zhao XY, Liu XQ, Duan Q, Song CY, Chen HN, Zheng WT, Shen QY, Wang MQ, et al. Development and application of an efficient recombineering system for Burkholderia glumae and Burkholderia plantarii[J]. Microbial Biotechnology, 2021, 14(4): 1809-1826. DOI:10.1111/1751-7915.13840 |
| [84] |
Bian XY, Plaza A, Zhang YM, Müller R. Luminmycins A–C, cryptic natural products from Photorhabdus luminescens identified by heterologous expression in Escherichia coli[J]. Journal of Natural Products, 2012, 75(9): 1652-1655. DOI:10.1021/np300444e |
| [85] |
Yin J, Hoffmann M, Bian XY, Tu Q, Yan F, Xia LQ, Ding XZ, Francis Stewart A, Muller R, Fu J, et al. Direct cloning and heterologous expression of the salinomycin biosynthetic gene cluster from Streptomyces albus DSM41398 in Streptomyces coelicolor A3(2)[J]. Scientific Reports, 2015, 5: 15081. DOI:10.1038/srep15081 |
| [86] |
Xu XK, Zhou HB, Liu Y, Liu XT, Fu J, Li AY, Li YZ, Shen YM, Bian XY, Zhang YM. Heterologous expression guides identification of the biosynthetic gene cluster of chuangxinmycin, an indole alkaloid antibiotic[J]. Journal of Natural Products, 2018, 81(4): 1060-1064. DOI:10.1021/acs.jnatprod.7b00835 |
| [87] |
Fu J, Wenzel SC, Perlova O, Wang JP, Gross F, Tang ZR, Yin YL, Stewart AF, Müller R, Zhang YM. Efficient transfer of two large secondary metabolite pathway gene clusters into heterologous hosts by transposition[J]. Nucleic Acids Research, 2008, 36(17): e113. DOI:10.1093/nar/gkn499 |
| [88] |
Perlova O, Fu J, Kuhlmann S, Krug D, Stewart AF, Zhang YM, Müller R. Reconstitution of the myxothiazol biosynthetic gene cluster by Red/ET recombination and heterologous expression in Myxococcus xanthus[J]. Applied and Environmental Microbiology, 2006, 72(12): 7485-7494. DOI:10.1128/AEM.01503-06 |
| [89] |
Wenzel SC, Gross F, Zhang YM, Fu J, Stewart AF, Müller R. Heterologous expression of a myxobacterial natural products assembly line in pseudomonads via Red/ET recombineering[J]. Chemistry & Biology, 2005, 12(3): 349-356. |
| [90] |
Tu Q, Herrmann J, Hu SB, Raju R, Bian XY, Zhang YM, Müller R. Genetic engineering and heterologous expression of the disorazol biosynthetic gene cluster via Red/ET recombineering[J]. Scientific Reports, 2016, 6: 21066. DOI:10.1038/srep21066 |
| [91] |
Gurumurthy CB, Saunders T, Ohtsuka M. Designing and generating a mouse model: frequently asked questions[J]. The Journal of Biomedical Research, 2021, 35: 76-90. DOI:10.7555/JBR.35.20200197 |
| [92] |
Thomas KR, Capecchi MR. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells[J]. Cell, 1987, 51(3): 503-512. DOI:10.1016/0092-8674(87)90646-5 |
| [93] |
Skarnes WC, Rosen B, West AP, Koutsourakis M, Bushell W, Iyer V, Mujica AO, Thomas M, Harrow J, Cox T, et al. A conditional knockout resource for the genome-wide study of mouse gene function[J]. Nature, 2011, 474(7351): 337-342. DOI:10.1038/nature10163 |
| [94] |
Baker O, Gupta A, Obst M, Zhang YM, Anastassiadis K, Fu J, Stewart AF. RAC-tagging: recombineering and Cas9-assisted targeting for protein tagging and conditional analyses[J]. Scientific Reports, 2016, 6: 25529. DOI:10.1038/srep25529 |
| [95] |
Baker O, Tsurkan S, Fu J, Klink B, Rump A, Obst M, Kranz A, Schröck E, Anastassiadis K, Stewart AF. The contribution of homology arms to nuclease-assisted genome engineering[J]. Nucleic Acids Research, 2017, 45(13): 8105-8115. DOI:10.1093/nar/gkx497 |
| [96] |
Rostovskaya M, Fu J, Obst M, Baer I, Weidlich S, Wang HL, Smith AJH, Anastassiadis K, Stewart AF. Transposon-mediated BAC transgenesis in human ES cells[J]. Nucleic Acids Research, 2012, 40(19): e150. DOI:10.1093/nar/gks643 |
| [97] |
Rostovskaya M, Naumann R, Fu J, Obst M, Mueller D, Stewart AF, Anastassiadis K. Transposon mediated BAC transgenesis via pronuclear injection of mouse zygotes[J]. Genesis, 2013, 51(2): 135-141. DOI:10.1002/dvg.22362 |
| [98] |
Brumos J, Zhao C, Gong Y, Soriano D, Patel AP, Perez-Amador MA, Stepanova AN, Alonso JM. An improved recombineering toolset for plants[J]. Plant Cell, 2020, 32(1): 100-122. DOI:10.1105/tpc.19.00431 |
| [99] |
Zhang WL, Fu J, Liu J, Wang HL, Schiwon M, Janz S, Schaffarczyk L, Von Der Goltz L, Ehrke-Schulz E, Dörner J, et al. An engineered virus library as a resource for the spectrum-wide exploration of virus and vector diversity[J]. Cell Reports, 2017, 19(8): 1698-1709. DOI:10.1016/j.celrep.2017.05.008 |
| [100] |
Smith HO, Hutchison CA, Pfannkoch C, Venter JC. Generating a synthetic genome by whole genome assembly: X174 bacteriophage from synthetic oligonucleotides[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(26): 15440-15445. DOI:10.1073/pnas.2237126100 |
| [101] |
Ando H, Lemire S, Pires DP, Lu TK. Engineering modular viral scaffolds for targeted bacterial population editing[J]. Cell Systems, 2015, 1(3): 187-196. DOI:10.1016/j.cels.2015.08.013 |
| [102] |
Shin J, Jardine P, Noireaux V. Genome replication, synthesis, and assembly of the bacteriophage T7 in a single cell-free reaction[J]. ACS Synthetic Biology, 2012, 1(9): 408-413. DOI:10.1021/sb300049p |
| [103] |
Loessner MJ, Rees CE, Stewart GS, Scherer S. Construction of luciferase reporter bacteriophage A511: for rapid and sensitive detection of viable Listeria cells[J]. Applied and Environmental Microbiology, 1996, 62(4): 1133-1140. DOI:10.1128/aem.62.4.1133-1140.1996 |
| [104] |
Kiro R, Shitrit D, Qimron U. Efficient engineering of a bacteriophage genome using the type I-E CRISPR-Cas system[J]. RNA Biology, 2014, 11(1): 42-44. DOI:10.4161/rna.27766 |
| [105] |
Fehér T, Karcagi I, Blattner FR, Pósfai G. Bacteriophage recombineering in the lytic state using the lambda red recombinases[J]. Microbial Biotechnology, 2012, 5(4): 466-476. DOI:10.1111/j.1751-7915.2011.00292.x |
| [106] |
Shin H, Lee JH, Yoon H, Kang DH, Ryu S. Genomic investigation of lysogen formation and host lysis systems of the Salmonella temperate bacteriophage SPN9CC[J]. Applied and Environmental Microbiology, 2014, 80(1): 374-384. DOI:10.1128/AEM.02279-13 |
| [107] |
Oppenheim AB, Rattray AJ, Bubunenko M, Thomason LC, Court DL. In vivo recombineering of bacteriophage λ by PCR fragments and single-strand oligonucleotides[J]. Virology, 2004, 319(2): 185-189. DOI:10.1016/j.virol.2003.11.007 |
 2021, Vol. 48
2021, Vol. 48