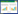扩展功能
文章信息
- 田真, 杜牧云, 纪孟志, 崔润博, 赵云迎, 樊祥宇
- TIAN Zhen, DU Muyun, JI Mengzhi, CUI Runbo, ZHAO Yunying, FAN Xiangyu
- PhageGT:dsDNA噬菌体基因组末端分析软件
- PhageGT: dsDNA phage genome termini analysis software
- 微生物学通报, 2021, 48(9): 3130-3140
- Microbiology China, 2021, 48(9): 3130-3140
- DOI: 10.13344/j.microbiol.china.210341
-
文章历史
- 收稿日期: 2021-04-14
- 接受日期: 2021-06-04
- 网络首发日期: 2021-07-01
2. 济南大学生物科学与技术学院 山东 济南 250022;
3. 中国气象局武汉暴雨研究所 湖北 武汉 430205
2. School of Biological Science and Technology, University of Jinan, Jinan, Shandong 250022, China;
3. Wuhan Institute of Heavy Rain, China Meteorological Administration, Wuhan, Hubei 430205, China
噬菌体作为细菌的病毒,具有感染并杀伤宿主菌的能力。目前,抗生素滥用情况日益严重,导致耐药菌不断增多。为了遏制耐药菌的传播,噬菌体疗法正逐步受到中外科学家的关注和应用[1]。随着噬菌体疗法的发展,针对不同宿主菌的新噬菌体分离和其基因组测序工作势在必行。在基因组水平上,噬菌体分为4类:双链DNA (dsDNA)噬菌体、单链DNA (ssDNA)噬菌体、双链RNA (dsRNA)噬菌体和单链RNA (ssRNA)噬菌体。在分离出的噬菌体中,以dsDNA噬菌体最为常见,也应用最广。dsDNA噬菌体的基因组分为环状和线状2种,其中线状基因组的末端类型多种多样[2]。
dsDNA噬菌体基因组的末端类型和其包装机制相关。噬菌体的包装机制相对保守。在dsDNA噬菌体感染周期接近尾声时,其基因组通常会形成串联体,在包装过程中被终止酶切割,形成成熟的染色体。噬菌体使用4种主要机制来识别它们自己的DNA并启动和终止其包装[3]:(1) 终止酶识别交错切割的特定位点(cos位点),从而产生具有黏性末端的固定DNA末端,该末端具有5′或3′突出部分(例如Lambda、HK97)。(2) 从噬菌体DNA上识别出一个固定位置,通过延伸合成将在此产生直接末端重复序列(Direct Terminal Repeats,DTR)。这些DTR的大小可以从一百多个碱基(例如T3、T7)到超过一万个碱基(例如T5、Spo1)不等。(3) 终止酶可以在特定的包装位点(pac位点)启动对噬菌体串联体的包装。当噬菌体头部被填满后,随后的切割将在不同位置进行。这导致衣壳含有冗余末端且循环排列的基因组,其注射到宿主细胞后可通过重组使噬菌体基因组成环。(4) 类T4噬菌体使用这种头部包装机制的变体。在这种机制中不识别pac位点,包装是随机启动的。这些噬菌体通常会降解宿主DNA,确保只包装病毒DNA。
除此之外还至少有3种不常见的噬菌体基因组末端包装策略[3]:(1) 噬菌体P2携带cos位点,但包装底物是环形dsDNA。(2) 噬菌体Mu通过转座在宿主基因组中复制,并携带宿主DNA片段作为其末端。(3) 噬菌体phi29在其DNA末端携带共价结合蛋白。当考虑到只有少量的噬菌体精确研究了其基因组末端包装机制,很可能在自然界中还存在其他包装机制。因此判断出噬菌体的末端类型有助于辅助新分离噬菌体的分类。
随着近几年高通量测序技术的不断发展以及价格的不断降低,大部分噬菌体基因组测序已经应用高通量测序技术来完成。在高通量测序技术大规模应用之前,dsDNA噬菌体的末端序列主要利用大引物(Mega-Primer) PCR的方法进行检测[4]。现在针对dsDNA噬菌体基因组的高通量测序结果,中外科学家已经建立了相对标准的流程来验证dsDNA噬菌体的基因组末端[3, 5-6]。其中Zhang等提出了利用噬菌体基因组高通量测序的高频序列判定噬菌体基因组末端的方法,这一方法指出,如噬菌体的基因组有末端,那么匹配到此末端序列的Reads就会表现出高频的特点[7]。在这一思想的指导下,为了判定噬菌体的末端序列,研究者首先应统计高通量测序结果中的高频Reads序列。
Zhang等给出了统计高频Reads序列的方法以及在Linux系统下的命令[7]。但鉴于大部分的病毒学或微生物学工作者对Linux操作系统不熟,使得这一方法和命令的应用受到了限制。在这一背景下,我们对Zhang等提出的统计高频Reads序列的方法进行了优化,设计出了在Windows操作系统下统计高通量测序数据中高频Reads的软件PhageGT。
1 材料与方法 1.1 所需材料与开发环境 1.1.1 材料选取我们课题组以前发表的2个病毒(SRT6[8]和SRT7[9])和在NCBI数据库随机下载的8个病毒(SRR13926753、SRR13783533、SRR10882832、SRR10882831、SRR10882571、SRR10760098、SRR14121700、SRR10210268),利用其原始的测序文件(20个,分别为2个病毒组双末端测序所得的正向和反向2个文件)进行软件测试及验证。其中文件中数据的细节如图 1所示。
|
| 图 1 高通量测序结果文件 Figure 1 High-throughput sequencing result file |
|
|
Windows 10、Visual Studio 2019。
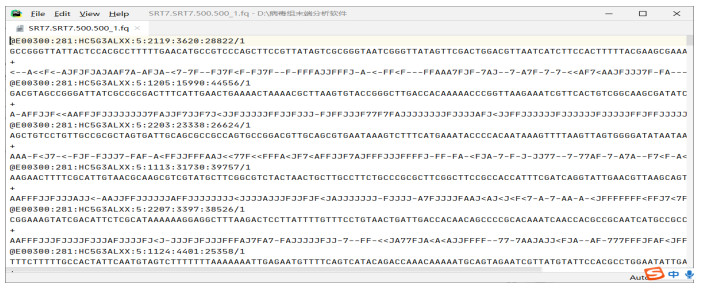
1.2 软件界面设计为了让更多的科研工作者使用起来简单、便捷,此软件的界面设计较为简单,软件界面如图 2和图 3所示。软件全自动化执行,用户只需输入2个特定的初始值并点击“open a file”按钮选择需要操作的序列文件,然后等待软件自动运行即可。软件运行完毕可自动弹出并保存数据文件及展示软件运行情况。
|
| 图 2 软件主界面 Figure 2 Main interface of the software |
|
|
|
| 图 3 关于界面 Figure 3 On the interface |
|
|
目前高通量测序已经成为研究病毒基因组的首选实验方案。随着测序价格的降低,现在高通量测序的数据量通常较为庞大,结果文件大多为Gb数量级。我们开发的PhageGT软件可以在庞大的序列文件中进行序列筛选,然后提取出文件中的所有序列,而且对重复序列进行计数。
1.3.2 计算频率此软件可以计算每个序列出现的频率。
1.3.3 根据频率将序列从大到小进行排列每个序列出现的频率大小不一,其中末端序列出现的次数最为频繁,其频率也比其他序列高。利用此种方法,可以筛选出高频序列,进而对噬菌体基因组的末端序列进行分析。
1.3.4 R值计算R值代表病毒组测序原始序列中Reads的最高频率和Reads平均频率的比值。可根据R值简单地推出此病毒组是否存在末端。
1.4 主要算法 1.4.1 界面设计在Visual Studio 2019中按照文件丨新建丨项目丨MFC应用的顺序新建基于对话框的桌面应用程序。然后在工具箱中添加编辑框及“open a file” “drop out”等按钮,并删除“确定” “取消”等按钮。新建模态类型的关于对话框并在“about”对话框中添加软件信息,当用户点击“about”按钮时将跳转到关于对话框。
在主对话框中提示用户应导入的文件类型,在打开文件对话框中将用户导入的文件格式强制为.fq丨.txt和所有文件2种方式,目的是防止用户导入错误的文件格式,但如果用户有其他类型的测序文件时还可以在所有文件选项中导入测序文件。
基于测序进度添加必要的文字提示,例如“Please import the virus group high-throughput sequencing file (**.fq)” “File is being read, please wait patiently...” “Sorting data...” “Calculating R value...” “File is exported to the root directory of Disk D”。
1.4.2 自定义所取碱基个数对多组序列进行测试后发现对于不同病毒基因组的不同数据,所需碱基数不同结果会有一定的差异,所以在PhageGT软件中设定了自定义读取Reads中碱基个数的功能。建议碱基个数的设定数值为20,这也是Zhang等所建议的数值[7]。发现选取的碱基个数越多其准确率越高,但随着选取碱基个数的增加,软件运行时间将会越长。当选取碱基个数接近单行序列总长度时,准确率会降低,因为测序时存在的微小误差会造成同一序列不同Reads的个别碱基不同。如果选取长度过长会将此类Reads归为不同类。所以选取合适碱基数量尤为重要。
获取用户输入的字符串,然后将其类型转换为int类型,目的是后面字符读取数量时避免因数字类型不同造成不必要的错误。
1.4.3 序列筛选因每个序列前都有一个符号“/”,所以可将其作为标识符,遍历序列文件中的每个字符,当遍历到“/”符号时再向后遍历的下一行就是基因序列。新建一个结构体,结构体中私有成员为基因序列和其出现的次数。当第一次遍历到基因序列时将其存入已经新建的数组中,当第二次遍历到基因序列时将其与存入数组中的序列进行比较,若数组中已经存在此序列则继续进行遍历,否则将其存入数组。由此可以得到筛选出的不重复序列。
1.4.4 序列排序定义comp()函数,此函数的功能为比较2个序列的出现次数大小。然后利用sort()函数进行排序,排序方法为comp()。
1.4.5 序列重复次数及频率计算在将得到的序列与数组中的序列进行对比时若此序列在数组中已经存在则将其成员变量num加一,最后程序运行完毕时每个变量其成员num为其出现的次数。
设置一个变量记录遍历过的基因序列的总数,然后用每个序列其出现的次数除以总数即是对应基因序列的频率。
1.4.6 R值计算[7]R=文件中序列的最高频率/序列的平均频率。最高频率=最高频Reads的频率,平均频率=序列文件中Reads总数/(2×基因组长度)。R < 30,说明没有末端或者末端很少;30 < R < 60,说明具有部分末端;R > 60,说明末端存在。
1.4.7 运行时间利用colcok()函数记录程序运行的初始时间和结束时间,然后两者相减即为程序运行时间。因其所得结果以秒为单位,因此要对所得结果进行判断若结果小于60则不需进行处理;若结果大于60小于3 600则结果应除以60转换为分钟计时方式;若结果大于3 600则结果应除以3 600转换为小时计时方式。
1.5 使用方法 1.5.1 打开软件双击打开PhageGT.exe可执行文件。
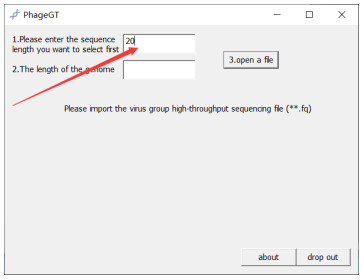
1.5.2 自定义要读取的碱基个数在第一个编辑框中输入要选择的碱基个数,如图 4所示。
|
| 图 4 碱基个数输入 Figure 4 Number of bases |
|
|
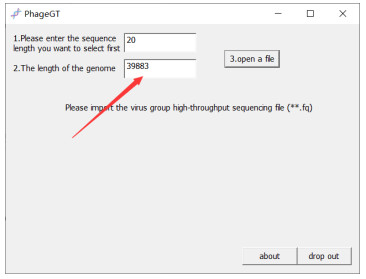
在第二个编辑框中输入基因组的长度,如图 5所示,其中基因组的长度由CLC Genomics Workbench 20软件将一组正反序列拼接后获得。
|
| 图 5 基因组长度输入 Figure 5 Length of the genome |
|
|
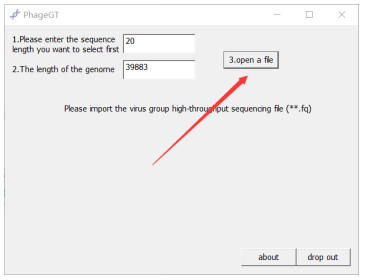
点击打开文件进行文件浏览,如图 6所示。
|
| 图 6 打开文件按钮 Figure 6 Open the file button |
|
|
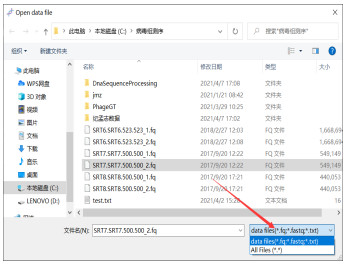
在文件浏览对话框中选择要导入的文件类型,如图 7所示。
|
| 图 7 文件类型选择 |
|
|
选中要导入的文件,然后点击打开导入文件,如图 8所示。
|
| 图 8 选择文件 Figure 8 Select the file |
|
|
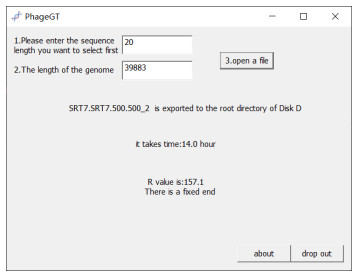
根据软件进度提示:数据读取,数据排序,数据导出,数据导出完毕,如图 9、图 10、图 11所示,等待测序完成,需等待时间决定机器配置。若读取序列文件较大可能会出现软件未响应,这属于正常现象,软件实际仍在运行,耐心等待即可。
|
| 图 9 数据读取 Figure 9 Data reading |
|
|
|
| 图 10 数据导出完毕 Figure 10 Data export is completed |
|
|

|
| 图 11 数据处理结果 Figure 11 Data processing results |
|
|
同一病毒正向和反向高通量测序的结果,经此软件处理后输出的前几个碱基序列应是相同的,并且其出现次数与频率也应大致相同,如表 1、表 2、表 3、表 4所示。
| 序列Sequence | 碱基序列Base sequence (5′→3′) | 出现次数Occurrences | 频率Frequency (%) |
| Sequence 1 | AACCACGAACCCACTAGGCA | 4 676 | 0.095 7 |
| Sequence 2 | AGCGACAGCTTACACCCGTT | 2 699 | 0.055 2 |
| Sequence 3 | GACAGCTTACACCCGTTCCC | 749 | 0.015 3 |
| Sequence 4 | GAACCCACTAGGCATACCTA | 446 | 0.009 1 |
| Sequence 5 | GGGTGGCCGTGGTCTTTCCT | 380 | 0.007 8 |
| 序列Sequence | 碱基序列Base sequence (5′→3′) | 出现次数Occurrences | 频率Frequency (%) |
| Sequence 1 | AACCACGAACCCACTAGGCA | 4 389 | 0.089 8 |
| Sequence 2 | AGCGACAGCTTACACCCGTT | 2 479 | 0.051 1 |
| Sequence 3 | CCCCCCCCCCCCCCCCCCCC | 901 | 0.018 4 |
| Sequence 4 | GACAGCTTACACCCGTTCCC | 732 | 0.015 0 |
| Sequence 5 | GAACCCACTAGGCATACCTA | 442 | 0.009 0 |
| 序列Sequence | 碱基序列Base sequence (5′→3′) | 出现次数Occurrences | 频率Frequency (%) |
| Sequence 1 | AGGGACTGAGGCATAACCAA | 2 889 | 0.222 2 |
| Sequence 2 | TCTCAAAGATACAACTCCCA | 2 062 | 0.158 6 |
| Sequence 3 | ATCGTCGTCGTAGGCTGCTC | 1 353 | 0.104 0 |
| Sequence 4 | GTATCGCTGGACACTGGACC | 1 129 | 0.086 8 |
| Sequence 5 | AACCTCACGCGGTGGACTCA | 913 | 0.070 2 |
| 序列Sequence | 碱基序列Base sequence (5′→3′) | 出现次数Occurrences | 频率Frequency (%) |
| Sequence 1 | TCTCAAAGATACAACTCCCA | 2 513 | 0.193 2 |
| Sequence 2 | AGGGACTGAGGCATAACCAA | 2 436 | 0.187 3 |
| Sequence 3 | CTCAAAGATACAACTCCCAA | 1 853 | 0.142 5 |
| Sequence 4 | ATCGTCGTCGTAGGCTGCTC | 1 312 | 0.100 9 |
| Sequence 5 | AACCTCACGCGGTGGACTCA | 987 | 0.075 9 |
由表 5可得对于同一组序列其R值大小接近,都可以判断是否存在末端,但是随着序列文件大小的增大处理时间也随之升高。对于不同电脑配置其软件运行时间也是不同的,电脑配置越高其处理时间越短。
| 序列文件 The sequence file |
R值大小 R value size |
是否存在末端 Whether there is an end |
文件大小 File size (Mb) |
用时 Use time (h) |
| STR6 positive direction | 178.13 | Yes | 1 628 | 24.4 |
| STR6 opposite direction | 167.20 | Yes | 1 628 | 29.0 |
| STR7 positive direction | 177.24 | Yes | 429 | 7.1 |
| STR7 opposite direction | 154.17 | Yes | 429 | 14.1 |
由表 5可得2个噬菌体基因组均存在末端,由表 1、表 2、表 3、表 4可知噬菌体SRT6、噬菌体SRT7分别存在2个主要的高频序列5′-AACCACGAACCCACTAGGCA-3′和5′-AGCGA CAGCTTACACCCGTT-3′、5′-AGGGACTGAGGC ATAACCAA-3′和5′-TCTCAAAGATACAACTCC CA-3′。如表 5所示,末端序列与一般序列出现频率的比值分别为178.13 (噬菌体STR6的正向)、167.20 (噬菌体STR6的反向)、177.24 (噬菌体STR7的正向)、154.17 (噬菌体STR7的反向)。根据SRT6和SRT7的全基因组序列以及已报道的计算方法[7]可计算出噬菌体基因组分别存在有1 165 bp (F19968,R21132)和175 bp (F1,R175)的直接重复末端。
2.4 软件通用性和准确性分析将STR6、STR7和NCBI数据库中随机选取的8个病毒(20个序列文件)同时在Windows系统下使用此软件(碱基个数设定数值为20)和在Linux系统下使用命令“awk 'NR%4==2' XXX.fastq|sort|uniq- c -w20|sort -g -r -o XXX.Freq”进行分析。
根据表 6可得此软件的计算结果与在Linux系统下使用命令行界面计算的结果完全相同,所以可以判断出此软件具有较高的准确性。因为选取的序列是在NCBI数据库中随机选取的,所以可以判断出此软件具有较高的通用性。
| 序列 Sequence |
碱基序列 Base sequence (5′→3′) |
次数 Occurrences |
|||
| Windows | Linux | Windows | Linux | ||
| STR6 positive direction 1 | AACCACGAACCCACTAGGCA | AACCACGAACCCACTAGGCA | 4 676 | 4 676 | |
| STR6 opposite direction 1 | AACCACGAACCCACTAGGCA | AACCACGAACCCACTAGGCA | 4 389 | 4 389 | |
| STR6 positive direction 2 | AGCGACAGCTTACACCCGTT | AGCGACAGCTTACACCCGTT | 2 699 | 2 699 | |
| STR6 opposite direction 2 | AGCGACAGCTTACACCCGTT | AGCGACAGCTTACACCCGTT | 2 479 | 2 479 | |
| STR6 positive direction 3 | GACAGCTTACACCCGTTCC | GACAGCTTACACCCGTTCC | 749 | 749 | |
| STR6 opposite direction 3 | CCCCCCCCCCCCCCCCCCCCC | CCCCCCCCCCCCCCCCCCCCC | 901 | 901 | |
| STR7 positive direction 1 | AGGGACTGAGGCATAACCAA | AGGGACTGAGGCATAACCAA | 2 889 | 2 889 | |
| STR7 opposite direction 1 | TCTCAAAGATACAACTCCCA | TCTCAAAGATACAACTCCCA | 2 513 | 2 513 | |
| STR7 positive direction 2 | TCTCAAAGATACAACTCCCA | TCTCAAAGATACAACTCCCA | 2 062 | 2 062 | |
| STR7 opposite direction 2 | AGGGACTGAGGCATAACCAA | AGGGACTGAGGCATAACCAA | 2 436 | 2 436 | |
| STR7 positive direction 3 | ATCGTCGTCGTAGGCTGCTC | ATCGTCGTCGTAGGCTGCTC | 1 353 | 1 353 | |
| STR7 opposite direction 3 | CTCAAAGATACAACTCCCAA | CTCAAAGATACAACTCCCAA | 1 853 | 1 853 | |
| SRR13926753 positive direction 1 | GATCGGAAGAGCACACGTCT | GATCGGAAGAGCACACGTCT | 22 564 | 22 564 | |
| SRR13926753 opposite direction 1 | GATCGGAAGAGCGTCGTGTA | GATCGGAAGAGCGTCGTGTA | 21 134 | 21 134 | |
| SRR13926753 positive direction 2 | CCAAACGCAACAATCGAAGC | CCAAACGCAACAATCGAAGC | 415 | 415 | |
| SRR13926753 opposite direction 2 | CCAAACGCAACAATCGAAGC | CCAAACGCAACAATCGAAGC | 324 | 324 | |
| SRR13926753 positive direction 3 | ACCAAACGCAACAATCGAAG | ACCAAACGCAACAATCGAAG | 314 | 314 | |
| SRR13926753 opposite direction 3 | CACCAAACGCAACAATCGAA | CACCAAACGCAACAATCGAA | 239 | 239 | |
| SRR13783533 positive direction 1 | GATCGGAAGAGCACACGTCT | GATCGGAAGAGCACACGTCT | 1 291 | 1 291 | |
| SRR13783533 opposite direction 1 | CTAAACAAGGCACACACAAG | CTAAACAAGGCACACACAAG | 855 | 855 | |
| SRR13783533 positive direction 2 | CTAAACAAGGCACACACAAG | CTAAACAAGGCACACACAAG | 976 | 976 | |
| SRR13783533 opposite direction 2 | GTCGCGCGCCCCGCCCGCCC | GTCGCGCGCCCCGCCCGCCC | 710 | 710 | |
| SRR13783533 positive direction 3 | GTCGCGCGCCCCGCCCGCCC | GTCGCGCGCCCCGCCCGCCC | 835 | 835 | |
| SRR13783533 opposite direction 3 | CCGACCTGTAACCAAGACGT | CCGACCTGTAACCAAGACGT | 53 | 53 | |
| SRR10882832 positive direction 1 | GTAGCTCCAGTTTTGCCCAG | GTAGCTCCAGTTTTGCCCAG | 276 | 276 | |
| SRR10882832 opposite direction 1 | CGTCAAAAGGCGACACTTTC | CGTCAAAAGGCGACACTTTC | 263 | 263 | |
| SRR10882832 positive direction 2 | CCAAACGCAACAATCGAAGC | CCAAACGCAACAATCGAAGC | 273 | 273 | |
| SRR10882832 opposite direction 2 | GAACGACATGGCTACGATCC | GAACGACATGGCTACGATCC | 260 | 260 | |
| SRR10882832 positive direction 3 | CGTCAAAAGGCGACACTTTC | CGTCAAAAGGCGACACTTTC | 267 | 267 | |
| SRR10882832 opposite direction 3 | GTTCCTCAATTTCCTCTTTG | GTTCCTCAATTTCCTCTTTG | 234 | 234 | |
| SRR10882831 positive direction 1 | CCAAACGCAACAATCGAAGC | CCAAACGCAACAATCGAAGC | 337 | 337 | |
| SRR10882831 opposite direction 1 | AGTTCGCGCACTCGACGTAA | AGTTCGCGCACTCGACGTAA | 255 | 255 | |
| SRR10882831 positive direction 2 | AGTACCCTGATTACGTCGAG | AGTACCCTGATTACGTCGAG | 287 | 287 | |
| SRR10882831 opposite direction 2 | AGTACCCTGATTACGTCGAG | AGTACCCTGATTACGTCGAG | 221 | 221 | |
| SRR10882831 positive direction 3 | AGTTACACATGTGGAAGGGG | AGTTACACATGTGGAAGGGG | 261 | 261 | |
| SRR10882831 opposite direction 3 | GTCTTTGTATTTCTGTAGCT | GTCTTTGTATTTCTGTAGCT | 219 | 219 | |
| SRR10882571 positive direction 1 | GTGAAACCTTCCCCTCTTGC | GTGAAACCTTCCCCTCTTGC | 2 020 | 2 020 | |
| SRR10882571 opposite direction 1 | GTGAAACCTTCCCCTCTTGC | GTGAAACCTTCCCCTCTTGC | 1 430 | 1 430 | |
| SRR10882571 positive direction 2 | AATAGCACTTTTTGTTAAAA | AATAGCACTTTTTGTTAAAA | 550 | 550 | |
| SRR10882571 opposite direction 2 | AATAGCACTTTTTGTTAAAA | AATAGCACTTTTTGTTAAAA | 540 | 540 | |
| SRR10882571 positive direction 3 | GTTTTAACAAAAAGTGCTAT | GTTTTAACAAAAAGTGCTAT | 433 | 433 | |
| SRR10882571 opposite direction 3 | AAATAGCACTTTTTGTTAAA | AAATAGCACTTTTTGTTAAA | 391 | 391 | |
| SRR10760098 positive direction 1 | AATAAAGACCATGCGACTTT | AATAAAGACCATGCGACTTT | 340 | 340 | |
| SRR10760098 opposite direction 1 | AATAAAGACCATGCGACTTT | AATAAAGACCATGCGACTTT | 296 | 296 | |
| SRR10760098 positive direction 2 | GTTATAAGGTTGTCCCATTT | GTTATAAGGTTGTCCCATTT | 250 | 250 | |
| SRR10760098 opposite direction 2 | GTATTAGTAGCAATATTAAA | GTATTAGTAGCAATATTAAA | 267 | 267 | |
| SRR10760098 positive direction 3 | CTCCCACACTGAGCGCTTAG | CTCCCACACTGAGCGCTTAG | 255 | 255 | |
| SRR10760098 opposite direction 3 | GTTATAAGGTTGTCCCATTT | GTTATAAGGTTGTCCCATTT | 264 | 264 | |
| SRR14121700 positive direction 1 | GTCCTGGACCTCGTAGGGGC | GTCCTGGACCTCGTAGGGGC | 43 | 43 | |
| SRR14121700 opposite direction 1 | GTACTGGAACAGGCCGGTGG | GTACTGGAACAGGCCGGTGG | 50 | 50 | |
| SRR14121700 positive direction 2 | GTGCAATGGGTGTCCGGGAC | GTGCAATGGGTGTCCGGGAC | 42 | 42 | |
| SRR14121700 opposite direction 2 | GTGCTGGCCAAGGGCCGCGG | GTGCTGGCCAAGGGCCGCGG | 47 | 47 | |
| SRR14121700 positive direction 3 | CCCATTGCACCGTGCAATCG | CCCATTGCACCGTGCAATCG | 41 | 41 | |
| SRR14121700 opposite direction 3 | GTGTACGACTGCACCTGCAG | GTGTACGACTGCACCTGCAG | 34 | 34 | |
| SRR10210268 positive direction 1 | CTCCTACACCGTTCGTTAAA | CTCCTACACCGTTCGTTAAA | 3 688 | 3 688 | |
| SRR10210268 opposite direction 1 | CTCCTACACCGTTCGTTAAA | CTCCTACACCGTTCGTTAAA | 3 534 | 3 534 | |
| SRR10210268 positive direction 2 | GTGTAGGAGAAGTGTTTGCC | GTGTAGGAGAAGTGTTTGCC | 3 133 | 3 133 | |
| SRR10210268 opposite direction 2 | TCGTTATACAGTGCCTCATA | TCGTTATACAGTGCCTCATA | 2 875 | 2 875 | |
| SRR10210268 positive direction 3 | TCGTTATACAGTGCCTCATA | TCGTTATACAGTGCCTCATA | 2 912 | 2 912 | |
| SRR10210268 opposite direction 3 | GTGTAGGAGAAGTGTTTGCC | GTGTAGGAGAAGTGTTTGCC | 2 840 | 2 840 | |
在表 7中发现Windows系统下此软件的计算时间比在Linux系统下使用命令操作高了数倍,所以此软件在运行时间上不如Linux命令操作有优势。但此软件可以直接得出R值并判断是否具有末端,并且采用Windows系统下的图形化界面,使操作简洁、直观,方便了不熟悉Linux系统的工作人员。
| 序列 Sequence |
文件大小 File size (Mb) |
用时Use time (h) | 用时Use time (min) | |
| Windows | Linux | |||
| STR6 positive direction | 1 628 | 10.2 | 8 | |
| STR6 opposite direction | 1 628 | 11.3 | 8 | |
| STR7 positive direction | 429 | 8.2 | 2 | |
| STR7 opposite direction | 429 | 6.3 | 2 | |
| SRR13926753 positive direction | 602 | 3.2 | 2 | |
| SRR13926753 opposite direction | 602 | 3.3 | 2 | |
| SRR13783533 positive direction | 517 | 2.9 | 2 | |
| SRR13783533 opposite direction | 517 | 2.8 | 2 | |
| SRR10882832 positive direction | 1 310 | 6.2 | 5 | |
| SRR10882832 opposite direction | 1 310 | 6.5 | 5 | |
| SRR10882831 positive direction | 1 290 | 7.2 | 4 | |
| SRR10882831 opposite direction | 1 290 | 7.1 | 5 | |
| SRR10882571 positive direction | 1 290 | 6.8 | 6 | |
| SRR10882571 opposite direction | 1 290 | 6.5 | 5 | |
| SRR10760098 positive direction | 423 | 4.2 | 3 | |
| SRR10760098 opposite direction | 423 | 4.1 | 3 | |
| SRR14121700 positive direction | 21 | 20 | 2 | |
| SRR14121700 opposite direction | 21 | 25 | 2 | |
| SRR10210268 positive direction | 2 028 | 12.1 | 10 | |
| SRR10210268 positive direction | 2 028 | 11.5 | 10 |
高通量测序技术的发展与使用,使科研人员需要处理的基因序列信息越来越大。针对噬菌体,已有科学家提出可利用基因组高通量测序的数据分析得到其基因组末端信息。但具体的分析过程需要相应的序列处理软件。与人工手动处理这些序列相比借助于计算机处理更节省时间,科研人员可以将更多的时间投入到更有价值的方向。将此前发布的Linux系统下噬菌体基因组高通量测序结果分析的方法[7],在Windows系统下设计相应软件,可以让不熟悉Linux系统的科研工作者省去了学习Linux系统的时间,使用熟悉的Windows系统的应用软件即可简单地进行数据处理。
当前挖掘各种生物数据之间的关系变得尤为重要,计算机技术的进步更是推动了这一生物信息学领域的发展,为相应科研人员提供了一个更好的平台,让他们可以借助于计算机快捷、简单地进行数据处理。根据我们测试的2个噬菌体基因组序列末端的验证结果可以看出此软件确实可行,可以较为简单地得到各种我们期望的结果。但是由于目前不同公司给出高通量测序的序列文件的细节可能有所不同,针对不同数据,软件也应相应的进行修改,需要开发与之相匹配的处理软件。在后续版本中我们将会对软件进行升级,以提供可以处理尽可能全部公司原始测序数据的软件。
所有数据集和软件均可从https://zenodo.org/record/4674231#.YHADb-gzZxc免费获得。
| [1] |
Kortright KE, Chan BK, Koff JL, Turner PE. Phage therapy: a renewed approach to combat antibiotic-resistant bacteria[J]. Cell Host & Microbe, 2019, 25(2): 219-232. |
| [2] |
Reardon S. Phage therapy gets revitalized[J]. Nature, 2014, 510(7503): 15-16. DOI:10.1038/510015a |
| [3] |
Garneau JR, Depardieu F, Fortier LC, Bikard D, Monot M. PhageTerm: a tool for fast and accurate determination of phage termini and packaging mechanism using next-generation sequencing data[J]. Scientific Reports, 2017, 7: 8292. DOI:10.1038/s41598-017-07910-5 |
| [4] |
Fan XY, Yan JL, Xie LX, Zeng LY, Young RF, Xie JP. Genomic and proteomic features of mycobacteriophage SWU1 isolated from China soil[J]. Gene, 2015, 561(1): 45-53. DOI:10.1016/j.gene.2015.02.053 |
| [5] |
Li SS, Fan H, An XP, Fan HH, Jiang HH, Chen YB, Tong YG. Scrutinizing virus genome termini by high-throughput sequencing[J]. PLoS One, 2014, 9(1): e85806. DOI:10.1371/journal.pone.0085806 |
| [6] |
Zhang X, Wang YH, Li SS, An XP, Pei GQ, Huang Y, Fan H, Mi ZQ, Zhang ZY, Wang W, et al. A novel termini analysis theory using HTS data alone for the identification of Enterococcus phage EF4-like genome termini[J]. BMC Genomics, 2015, 16: 414. DOI:10.1186/s12864-015-1612-3 |
| [7] |
Zhang X, Wang YH, Tong YG. Analyzing genome termini of bacteriophage through high-throughput sequencing[J]. Bacteriophages, 2018, 139-163. |
| [8] |
Liu ZC, Li Q, Zhang FK, Zhang CL, Ma HR, Yi TX, Gao XM, Fan XY. Isolation and genome sequence analysis of a novel Pseudomonas aeruginosa SRT6[J]. Journal of Liaocheng University: Natural Science Edition, 2018, 31(3): 67-78. (in Chinese) 刘子辰, 李骑, 张福康, 张春龙, 马鸿芮, 伊廷旭, 高晓萌, 樊祥宇. 一株新铜绿假单胞菌噬菌体SRT6的分离以及全基因组序列分析[J]. 聊城大学学报(自然科学版), 2018, 31(3): 67-78. |
| [9] |
Zhao KL, Song SK, Zhao ZP, Liu ZC, Ji Y, Gu PF, Fan XY, Li Q. The complete genome sequence of Escherichia phage SRT7, a novel T7-like phage[J]. Archives of Virology, 2019, 164(4): 1217-1219. DOI:10.1007/s00705-019-04182-4 |
 2021, Vol. 48
2021, Vol. 48