扩展功能
文章信息
- 王杰, 王晨, 杜燕, 徐晶玉, 班睿
- WANG Jie, WANG Chen, DU Yan, XU Jingyu, BAN Rui
- 枯草芽孢杆菌表达和分泌异源蛋白的研究进展
- Advances in heterologous protein expression and secretion of Bacillus subtilis
- 微生物学通报, 2021, 48(8): 2815-2826
- Microbiology China, 2021, 48(8): 2815-2826
- DOI: 10.13344/j.microbiol.china.200895
-
文章历史
- 收稿日期: 2020-09-05
- 接受日期: 2021-01-15
- 网络首发日期: 2021-03-17
枯草芽孢杆菌是土壤中广泛存在的一种产芽孢革兰氏阳性菌,作为模式菌株已被深入研究,其基因组已完成全序列测序以及部分基因解析,代谢机理和遗传背景相对清晰。枯草芽孢杆菌不分泌外毒素,也不含内毒素,被美国食品药品监督管理局(Food and Drug Administration,FDA)认定为生物安全菌株(Generally Regarded as Safe,GRAS),在发酵工业中被用于生产各类发酵产品[1],例如重组蛋白[2]、γ-聚谷氨酸[3]、透明质酸[4]、D-核糖[5]、核黄素[6]和尿苷[7]等,在食品和药品的发酵应用上,枯草芽孢杆菌更有安全性的优势。
枯草芽孢杆菌作为宿主菌生产异源蛋白已被证明是一个可行且具有良好前景的方案,其不仅具有高效的蛋白分泌途径,而且含有与分泌途径相关的分子伴侣,能够使分泌的蛋白质具有天然构象和生物活性[8]。在发酵工业中,枯草芽孢杆菌的蛋白分泌能力被用来生产异源蛋白,部分产品已成功实现工业化生产,例如α-淀粉酶[9]、脂肪酶[10]、β-甘露聚糖酶[11]、人类白介素-3[12]等。尽管如此,用枯草芽孢杆菌生产某些异源蛋白仍存在较大困难(尤其是来源于真核生物的蛋白),例如存在基因表达水平过低、分泌蛋白错误折叠、分泌蛋白在胞内外被降解等问题。为了解决异源蛋白在枯草芽孢杆菌中表达和分泌的瓶颈问题,研究者主要在6个方面进行了研究:(1) 提高基因转录水平和稳定性;(2) 优化分泌途径,提高异源蛋白分泌效率;(3) 过表达分子伴侣,提高异源蛋白折叠效率;(4) 对细胞壁、细胞膜以及相关蛋白进行改造,增强异源蛋白易位效率;(5) 敲除胞外蛋白酶,防止异源蛋白在胞外被降解;(6) 开发新型策略,例如优化全局碳氮代谢调控、基因组精简等。关于枯草芽孢杆菌表达和分泌异源蛋白的策略如表 1所示。本文对近年来枯草芽孢杆菌生产异源蛋白的相关研究进行综述,并对此提出评价以及相应的解决方案。
| Classifications | Strategies | Heterologous protein | Superiorities | References |
| Transcription | Optimizinsg promoter | β-galactosidase and green fluorescent protein | 26 and 195-fold improvement of enzyme activity | [13] |
| Promoter screening | α-amylase; hydantoinase | 2.10-fold improvement of production; 60.55-fold improvement of transcriptional level | [14-15] | |
| Dual promoters | 4-α-glucanotransferase; β-cyclodextrin glycosyltransferase; aminopeptidase | 11 to 12-fold improvement of enzyme activity; the maximum enzyme activity of 30.5 U/mL; the maximum production of 8 g/L | [16-18] | |
| Optimization of gene copy numbers | Green fluorescent protein | 3.6-fold improvement of production | [19] | |
| Improving RNA stability | Human FGF21; β-galactosidase | Approximately 3.0-fold improvement of production; increasing the half-life of mRNAs to more than 60 min | [20-21] | |
| Chaperone | Overexpression of GroESL | β-mannanase | Improvement of β-mannanase activity | [11] |
| Overexpression of PrsA | Lipase; α-amylase; human interferon-β; lipoxygenase | 1.6 to 4.0-fold improvement of enzyme activity or production | [10, 22-26] | |
| Secretory pathway | Optimization of signal peptide | Aminopeptidase; α-amylase | The maximum production of 8 g/L; 3.5-fold improvement of enzyme activity | [18, 27] |
| Overexpression of SecYEG | α-amylase | 10% improvement of production | [28] | |
| Overexpression of SecDF | Lipase | 28% improvement of production | [10] | |
| Overexpression of SecA, SecYEG, SecDF, SRP and FstY | α-amylase | Enhanced production of α-amylase | [24] | |
| Overexpression of TatAdCd | Lipase | 1.4-fold improvement of enzyme activity | [29] | |
| Overexpression of SipT | α-amylase | Enhanced production of α-amylase | [30] | |
| Overexpression of SipS and SipT | β-mannanase | Improvement of β-mannanase activity | [11] | |
| Protease | Overexpression of RasP | Serine protease; α-amylase | 3.0-fold improvement of production; 10.0-fold improvement of production | [31] |
| Deletion of nprE, nprB, mpr, vpr, epr, bpr, wprA and aprE | Antidigoxin single-chain antibody | Production increased by 10 to 15 mg/L | [32] | |
| Deletion of aprX | α-amylase | 8.0-fold improvement of production | [33] | |
| Deletion of clpxp | α-amylase | 2.0-fold increase in pre-protein | [30] | |
| Cell surface engineering | Deletion of dlt operon | α-amylase | Enhanced efficiency of translocation | [34] |
| Deletion of pssA and clsA | α-amylase | Enhanced production of α-amylase | [35] | |
| Global transcriptional regulators | Random mutagenesis of ccpA and codY | β-galactosidase | 2.9-fold improvement of enzyme activity | [36] |
| Genome streamlining | Deleting 874 kb fragment of genomic | Cellulase | Enhanced production of cellulase | [37] |
| Cell autolysis | Deletion of skfA, sdpC, xpf and lytC | β-galactosidase | 1.72-fold improvement of production | [38] |
基因表达水平是决定相应蛋白质合成速率及胞内或胞外水平的首要因素。因此,利用枯草芽孢杆菌生产异源蛋白,首先要解决的问题就是编码基因的高水平表达。研究者一般从启动子工程、优化基因拷贝数和提高mRNA稳定性3个方面解决基因表达水平问题。
1.1 启动子工程启动子是基因转录调控的重要表达元件,其决定转录的方式、时间和强度。传统的策略是通过优化启动子或筛选强启动子的方式来提高基因转录水平[39]。典型原核启动子的特征包括保守区域–35区(TTGACA)和–10区(TATAAT)、–35区和–10区间的间隔区域以及转录起始位点(TTS)[40]。通过定向修饰启动子的保守区域、TTS或者–35区上游的UP元件等方法可以明显提高启动子转录效率。Zhou等[13]通过将–35区和–10区的原序列均替换成共有序列来增强Pylb的活性,并将rrnB P1启动子上游UP元件替换原有UP元件,最终使2个报道蛋白(β-半乳糖苷酶和绿色荧光蛋白)的活性均大幅度提高,分别为野生型的26倍和195倍。除了对启动子进行优化以外,筛选强启动子能够明显提高异源基因的转录水平。Song等[14]以β-半乳糖苷酶对枯草芽孢杆菌中84种不同启动子进行筛选,证明PtrnQ、PsigX、PgroES启动子的转录强度高于P43,利用PtrnQ表达异源α-淀粉酶,与P43相比,其产量提高了2.1倍。李法彬等[15]探究4个不同启动子(P43、PspoVG、PlytR、PAE)对异源D-氨甲酰水解酶活性的影响,结果发现人工构建的组成型启动子PAE可以大幅提升目的基因转录水平(其转录水平是P43-adc的60.55倍)。然而,即便同一个启动子对不同的编码基因以及不同生物环境表现出的转录强度也不一样。因此,在一定条件下对启动子的活性作出标准化的定义是至关重要的,方便科研人员对启动子进行精准的比较和筛选。
利用串联启动子提高异源蛋白的表达量是近年来的研究热点之一。在自然界中,串联启动子广泛应用于调控某些芽孢杆菌的致病基因或者其他生理功能相关基因,例如调控枯草芽孢杆菌的鞭毛合成基因[41]。受到这类调控机制启发,近年来大量研究人员致力于构建和筛选串联启动子的方式来增强目的基因转录水平[16-18]。Kang等[16]构建串联启动子PHpaII-PamyR2和PHpaII-PBLMA分别过量生产异源4-α-葡聚糖转移酶,与单启动子PHpaII表达系统相比,目的蛋白的酶活分别提高了11倍和12倍。Zhang等[17]以β-环糊精糖基转移酶(β-CGTase)为报道蛋白,比较9种公认的强启动子的表达强度(Psrf、Pxyl′、PgsiB、Pxyl、PHpaII、PamyQ′、PaprE、PnprE和PamyQ′),并将其中6种转录水平更高的启动子分别与PamyQ′′串联构建6种不同的串联启动子,最终筛选得到串联启动子PHpaII-PamyQ′′,与单启动子PamyQ′表达系统相比,目的蛋白的酶活提高了1.27倍,达到30.5 U/mL。使用串联启动子来提高异源蛋白产量已被证明是一个优良的策略,然而目前有关研究中缺乏对串联启动子下基因绝对转录水平的表征。此外,关于串联启动子如何提高基因转录水平的机理研究仍处于空白,需要研究人员进一步探究。
1.2 优化基因拷贝数目的基因的拷贝数,即基因剂量是影响基因转录水平的重要因素。通常可利用多拷贝表达质粒或者基因组上多位点整合表达来增加基因剂量,进而提高异源蛋白的产量。在一定范围内基因拷贝数与蛋白成正比,但过高的基因拷贝数会导致细胞代谢负荷过大,影响菌体的正常生长,使宿主菌的遗传稳定性明显下降。因此,只有通过合理地提高基因拷贝数才能有效地提高异源蛋白产量。Zhang等[42]以谷氨酰胺酶(Mglu)作为报道蛋白,分别使用染色体多位点整合表达以及高拷贝质粒的方法来提高基因拷贝数,最终获得的最佳拷贝数为7-8拷贝,目的蛋白的酶活可达到357.6 U/mL。Nadler等[19]将带有同源臂片段、复制起始位点(ori)和抗性基因的线性化低拷贝质粒融合到枯草芽孢杆菌中,使其染色体同源臂之间的靶基因复制到质粒上并重新环化,从而构建CopySwitch系统;此方法不仅可以在菌体内快速简捷地优化基因拷贝数,而且能够克服传统环状质粒转化技术的困难;利用该系统使得绿色荧光蛋白的表达量提高了3.6倍,成熟磷脂酶C的表达量提高了1.3倍。
1.3 增强mRNA稳定性在枯草芽孢杆菌中,不同mRNA的半衰期差异很大,短的可小于1 min,长的可高达60 min。mRNA半衰期的长短对蛋白翻译效率起着重要作用,通过提高目的基因mRNA的半衰期是增加异源蛋白产量的重要手段。RNase J1和RNase Y是枯草芽孢杆菌主要的mRNA降解酶,其中RNase J1不仅具有核酸内切酶的活性,而且具有5′-端至3′-端的核酸外切酶活性[43]。研究表明,5′-非翻译区(5′-UTR)末端的二级结构是影响mRNA半衰期的决定性因素;另外,标准的核糖体结合位点(Ribosomebinding Site,RBS)序列能够提高其与核糖体结合效率,进而提高mRNA半衰期[43]。Li等[20]在目的基因rhFG21上游插入不同来源的微型顺反子盒(gfp、glvA、rdpE、gsiB)来提高mRNA的稳定性和翻译效率,最终发现插入顺反子盒(gisB)具有积极作用,使得目的蛋白的表达量提升了约3倍。Phan等[21]以β-半乳糖苷酶作为报道蛋白,详细分析了不同的mRNA稳定元件对异源蛋白产量的影响,包括3′-茎环、RBS、5′-茎环以及5′-茎环与RBS之间的间隔区,并将2种mRNA稳定元件(Escherichia coli lac操纵子的lacO元件与gsiB的RBS序列)相组合,融合于groE启动子,最终使目的基因mRNA的半衰期延长至60 min以上,其酶活性提高5倍以上。未来研究可以集中于开发新型天然茎环结构或者构建人工茎环结构,结合于目的基因的5′-非翻译区或者3′-非翻译区以达到mRNA稳定作用。
2 异源蛋白在枯草芽孢杆菌中分泌优化策略 2.1 异源蛋白分泌过程及途径枯草芽孢杆菌分泌蛋白过程如图 1所示,一般可分为4个过程:(1) 分泌早期阶段:分泌前体蛋白与锚定因子SRP结合,并与分泌转运酶结合形成复合物,而在胞内发生错误折叠的分泌蛋白会在胞内蛋白酶的作用下降解;(2) 分泌中期阶段:复合物通过转位酶跨越细胞膜完成易位;(3) 分泌晚期阶段:蛋白质从转位酶中释放、折叠、去除信号肽以及通过细胞壁[44];(4) 完成分泌后阶段:分泌后的蛋白质可能被胞外蛋白酶所降解,因此胞外蛋白酶是影响异源蛋白最终产量的重要因素[45]。枯草芽孢杆菌分泌胞外蛋白的具体途径主要有经典蛋白分泌途径(Sec分泌途径)和双精氨酸分泌途径(Tat分泌途径),另外也有部分蛋白通过ATP结合转运途径(ABC途径)或者假纤毛转运途径分泌至胞外。
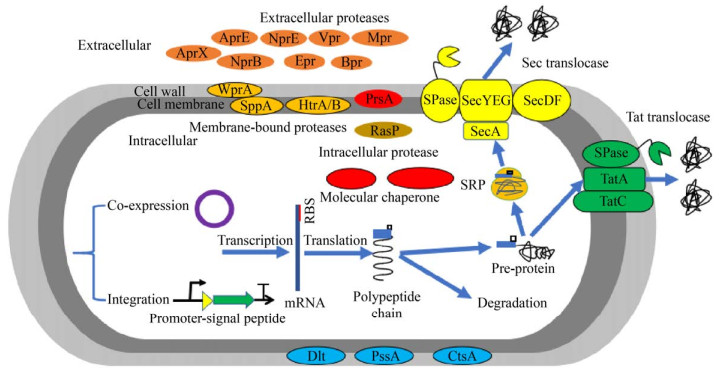
|
| 图 1 枯草芽孢杆菌异源蛋白表达和分泌过程 Figure 1 The processes of expression and secretion of heterologous proteins in Bacillus subtilis |
|
|
Sec分泌途径主要负责未折叠蛋白质的转运,转运元件包括转运动力蛋白SecA及易位通道蛋白SecYEG和SecDF,其他元件还包括细胞识别因子SRP、膜相关受体FtsY、胞内分子伴侣以及信号肽酶等。Sec分泌途径包括3个过程:分选锚定;易位转运;折叠释放。首先前体蛋白信号肽的高度疏水区域N端被SRP识别并锚定构成复合物,SecA则与细胞膜上的SecYEG结合,SecA识别复合物并引导至SecYEG通道;然后SecA与ATP绑定,SecA的C端穿过SecYEG透过细胞膜,前体蛋白的前端进入SecYEG易位通道,在ATP水解产生的动力驱动和质子动力势的作用下,前体蛋白被运输出细胞;最后在PrsA的作用下完成折叠,信号肽酶切除信号肽,完成分泌[46]。近年来,对于Sec途径分泌机制提出新的观点:前体蛋白成熟域中存在的某些关键碱基(Mature Domain Targeting Signals,MTS)对分泌过程起了重要作用[47]。
Tat分泌途径主要转运一些不能通过Sec分泌途径进行紧密折叠的蛋白质或多亚基酶复合物,其转运元件主要包括一个小的膜蛋白TatA和一个大的膜蛋白TatC;其中,TatA有3个亚蛋白,TatC有2个亚蛋白,组合构成2条平行的转运途径——TatAy-TatCy和TatAd-TatCd,这2条途径分别介导不同的底物蛋白;枯草芽孢杆菌中发现的依赖Tat转运途径的分泌蛋白有PhoD、YwbN、QcrA、YkuE等[48]。
2.2 蛋白质折叠细胞中的异源蛋白若无法正确折叠,则会形成不溶性聚集体并滞留在细胞内,最终被细胞壁结合蛋白酶HtrA/B清除。分子伴侣可以在蛋白分泌过程中辅助其折叠和装配,避免被蛋白酶降解。因此,枯草芽孢杆菌中分子伴侣的表达量和活性是影响异源蛋白产量的关键因素。枯草芽孢杆菌中存在2种分子伴侣:胞内分子伴侣(GroES/GroEL、DnaK/DnaJ)与胞质外分子伴侣(PrsA)。
胞内分子伴侣GroES/GroEL和DnaK/DnaJ分别由groESL操纵子和dnaK操纵子编码,2个操纵子构成枯草芽孢杆菌CIRCE调节子[49]。2个操纵子均受到阻遏蛋白HrcA的负调控,当hrcA缺陷时,2个操纵子所编码的分子伴侣则可获得组成型表达。Song等[11]通过系统基因过表达探究异源蛋白的分泌瓶颈,结果表明当过表达groESL时,β-甘露聚糖酶的酶活可提升1.25倍,但过表达dnaK对目的蛋白分泌并未产生积极影响。
目前,枯草芽孢杆菌中已知胞质外分子伴侣仅有PrsA。PrsA不仅可以辅助蛋白质在细胞膜外折叠,形成具有天然结构的活性蛋白,而且可以防止分泌蛋白在胞外被蛋白酶降解,PrsA的表达量可能是枯草芽孢杆菌生产异源蛋白的瓶颈[50]。大量研究表明过表达PrsA可以使异源蛋白的产量提高2倍及以上(脂肪酶[10]、α-淀粉酶[9, 22-24]、人类β-干扰素[25]、脂氧合酶[26])。Kakeshita等[25]在研究枯草芽孢杆菌中表达人类β-干扰素(hIFN-β)过程中发现:当仅过表达PrsA时,重组hIFN-β的总活性提高了2.5倍;而在hIFN-β编码区上游共表达AmyE的前肽,并同时过表达PrsA,重组hIFN-β的总活性可提高4.9倍。从该研究中可推测PrsA的促蛋白折叠机制可能与前肽有关。通过过表达同源分子伴侣能够显著提升异源蛋白的产量,然而目前仍无法突破真核基因在枯草芽孢杆菌中的表达瓶颈。寻找与靶基因相匹配的异源分子伴侣,并在枯草芽孢杆菌共表达可能是突破该瓶颈的核心路线。
2.3 分泌途径优化 2.3.1 筛选信号肽信号肽是辅助前体蛋白折叠以及引导前体蛋白跨膜易位的重要元件,是影响异源蛋白分泌效率以及产量的限制性因素。信号肽有3个公认的结构域:(1) 位于氨基末端带正电的N-域,该区域至少含有一个精氨酸或赖氨酸残基,该区域可与细胞膜中带负电的磷脂相互作用,决定信号肽的运动方向;(2) 位于N-域之后的H-域,其由一串疏水的氨基酸残基组成,可在细胞膜处形成α-螺旋结构,便于信号肽嵌入细胞膜中完成介导作用;(3) 位于H-域之后的C-域,能够在易位后被信号肽酶识别和切割,释放成熟蛋白[51]。通过逐一筛选或者高通量筛选信号肽,可以实现异源蛋白的高效分泌,增加目的蛋白产量[11, 18, 27, 52]。Guan等[18]以氨肽酶作为报道蛋白,用串联启动子PgsiB-PHpaII作为表达元件,通过比较氨肽酶的胞外活性,从19个信号肽中筛选出最优信号肽SPyncM,使得氨肽酶产量提高到8 g/L。Yao等[27]通过构建文库和高通量筛选的方法,将枯草芽孢杆菌中173种信号肽分别与异源α-淀粉酶融合,获得并鉴定出3种合适信号肽(SPYojL、SPRpmG和SPAspB),最终筛选出最优信号肽SPYojL,使得酶活提高了3.5倍。虽然目前对信号肽的氨基酸构成、基础结构以及作用机制已经有了详细的报道,但是关于信号肽与目标蛋白的适用机制仍不明确[18, 52]。截至目前,生物界尚未开发出能够精确预测最优信号肽的算法,导致信号肽的选择只能局限于高通量筛选。通过生物信息学的方法整理与分析枯草芽孢杆菌中上百种信号肽,并依据不同结构特征对其进行科学分类可能是探究信号肽与目标蛋白适用机制的良好策略。
2.3.2 过表达分泌途径相关蛋白枯草芽孢杆菌中95%以上的分泌蛋白通过Sec分泌途径分泌至胞外,通过提高Sec途径相关蛋白(SecA、SecYEG、SecDF、Ffh和FtsY)的表达量可以增强异源蛋白分泌效率[10, 11, 24, 28, 53]。SecA蛋白是转运动力蛋白,在异源蛋白分泌过程起关键调节作用。Kakeshita等[53]分别在SecA的C末端区域删除22个氨基酸残基和61个氨基酸残基,成功提高了异源蛋白在枯草芽孢杆菌中的分泌。除SecA蛋白外,转运酶的缺失会导致异源蛋白在细胞膜上发生堵塞;相反,提高转运酶的表达量可以提高异源蛋白的分泌效率。Mulder等[28]首次构建了人工木糖诱导启动子Pxyl控制的secYEG操纵子,并整合到枯草芽孢杆菌中,最终异源α-淀粉酶产量提高了10%。Ma等[10]通过将secDF与prsA共过表达,使脂肪酶的产量提高了59%,达到287.8 U/mL。
Sec途径担负了上百种胞外蛋白的分泌工作,因此Sec途径可能会处于“拥堵”和“竞争”状态。部分信号肽(例如SPlip)不仅能够通过Sec途径,同时也符合Tat途径信号肽特征[54]。基于这类信号肽的特征,可以利用2个蛋白分泌途径来提高异源蛋白产量。白雷雷等[29]通过过表达TatAdCd转位酶相关基因,使得脂肪酶活提升了1.4倍,达到243 U/mL,为增强异源蛋白分泌效率提供新思路。
2.3.3 过表达信号肽酶当异源蛋白完成转运和易位后,信号肽被信号肽酶识别并切割,从而形成成熟蛋白质。枯草芽孢杆菌中信号肽酶分为2种:Ⅰ型信号肽酶(SipS、SipT、SipU、SipV、SipW)和Ⅱ型信号肽酶(LspA)。Ⅰ型信号肽酶可针对大部分的分泌蛋白进行切割;Ⅱ型信号肽酶针对脂蛋白的信号肽进行切割。SipS和SipT是主要的信号肽酶,对蛋白质的分泌具有重要意义,这2个信号肽酶足以处理大部分前体蛋白的成熟,而其余多种信号肽酶则可以保证细胞处于各种条件下进行信号肽切割。研究表明,在枯草芽孢杆菌中过表达SipS或SipT可以增加异源蛋白的产量;相反,缺陷SipS则会导致多种前体蛋白的加工速率降低[11, 30, 55-56]。Pummi等[30]发现蛋白酶ClpxP的突变失活可以将SipS、SipT、SipV的转录水平上调2-5倍,导致异源α-淀粉酶的前体蛋白胞内积累,从而提高其产量。
2.4 蛋白质易位 2.4.1 细胞质量控制系统对分泌蛋白的影响枯草芽孢杆菌中的双组分调节系统CssR-CssS能够感知热胁迫或分泌胁迫,从而激活包括htrA和htrB在内的相关基因表达[57]。这些细胞壁结合蛋白酶(HtrA/HtrB和WprA)构成枯草芽孢杆菌中的细胞质量控制系统,它们负责降解易位过程中错误折叠或者异常的蛋白质,以避免这些蛋白质堵塞细胞孔道。研究发现,这些蛋白酶也能够降解正常分泌的未折叠蛋白质,这可能是导致异源蛋白产量下降的原因之一[58]。Pohl等[59]构建了htrA、htrB和wprA不同组合缺陷型的突变体并进行蛋白组学分析,结果表明虽然缺陷htrA和htrB菌株生长状况基本没有受到影响,但能够激活同工酶HtrC的大量表达,继续执行胞内质量控制的功能;在敲除wprA后,报道蛋白重组炭疽保护性抗原(rPA)的产量明显提升,但当继续敲除htrA和htrB后,其产量并未提升。Vitikainen等[60]敲除了枯草芽孢杆菌的cssS或cssR,并未在敲除株中观察到异源α-淀粉酶和肺炎球菌溶血素产量的提高,反而导致菌体的生长受损;但当cssRS系统正常表达时,HtrA和HtrB降解异源蛋白(百日咳毒素),上清液中出现大量降解产物。目前,直接敲除cssR-cssS或者htrA/B来提高异源蛋白产量的策略均未实现,通过温和的基因修饰手段来调节htrA和htrB的表达水平,达到既不影响细胞的生长又能提升异源蛋白产量的平衡态可能是个更优的选择;其次,由于这些质量控制因子能够感知分泌压力发生应激反应,可用于检测异源蛋白的表达量,这为表征异源蛋白产量提供了新手段。
2.4.2 细胞表面工程细胞膜与细胞壁在异源蛋白折叠与易位的过程中起着重要作用,利用细胞表面工程来提高异源蛋白产量是具有广泛前景的新型策略。RasP是属于Site-2蛋白酶(S2P)家族的膜内蛋白酶,可切割完整膜蛋白的跨膜结构域。Neef等[31, 61]研究表明,在枯草芽孢杆菌中过表达RasP,使丝氨酸蛋白酶产量提升3倍,α-淀粉酶的产量提升了10倍;当在枯草芽孢杆菌中敲除rasP,丝氨酸蛋白酶和α-淀粉酶的产量则严重下降。Neef等对RasP的作用机制提供了3种猜测:(1) RasP可能在细胞膜上有助于信号肽的切除;(2) RasP可以清除细胞膜上分泌失调的前体蛋白;(3) RasP可能会调节膜蛋白的表达[31]。Neef等的研究似乎证明RasP可能是枯草芽孢杆菌表达异源蛋白的瓶颈,但是RasP对异源蛋白作用的普适性尚未确定,以及对真核蛋白是否有效亟待证明[31]。
枯草芽孢杆菌的细胞壁是多层网状结构,由肽聚糖、阴离子聚合物(磷壁酸与脂磷壁酸)及蛋白质所形成的共聚物组成。细胞壁的负电荷密度受磷壁酸侧链上的D-丙氨酸酰化程度调节。敲除dlt操纵子可以降低磷壁酸的丙氨酸酰化程度,增加细胞壁的负电荷密度,可以提高异源蛋白分泌效率[34-35, 62]。Cao等[35]在枯草芽孢杆菌中依次敲除与细胞表面电荷密度相关的6个基因(pssA、tagO、clsA、dltA、tuaA、dacA),结果显示,敲除CDP-二酰基甘油-丝氨酸O-磷脂酰转移酶基因(pssA)和心磷脂合成酶基因(clsA),能够增加细胞表面负电荷密度,增强异源α-淀粉酶的折叠效率和稳定性,使产量提升了47%;此外,他们的研究结果表明异源蛋白的等电点也是影响蛋白分泌效率的因素之一。
2.5 胞外蛋白酶枯草芽孢杆菌作为宿主生产异源蛋白的另一限制因素是其能够分泌多种胞外蛋白酶。枯草芽孢杆菌分泌的异源蛋白常常在胞外被胞外蛋白酶所降解,因此敲除胞外蛋白酶相关基因可以提高异源蛋白的产量。目前,工业中已构建多种胞外蛋白酶缺失菌株用于高效表达异源蛋白。
枯草芽孢杆菌的胞外蛋白酶包括碱性蛋白酶(AprE)、中型蛋白酶(NprE和NprB)、金属蛋白酶(Mpr)、丝氨酸蛋白酶(Vpr、Epr和Bpr)和细胞壁结合蛋白酶(WprA),其中AprE和NprE的胞外蛋白酶活性在95%以上。即便如此,剩余6种蛋白酶的存在足以对异源蛋白的产量构成瓶颈[63]。Wu等[64]敲除枯草芽孢杆菌中6个胞外蛋白酶基因构建WB600,其胞外蛋白酶活性只有野生型的0.32%;结果表明,使用P43-sacY盒和WB600组合生产β-内酰胺酶,其产量提高了18倍。Wu等[32]在WB600的基础上敲除WprA和Vpr进而构建WB800,与WB700相比,报道蛋白(抗地高辛单链抗体)的产量提高10-15 mg/L。Kodama等[33]在WB800的基础上敲除了胞内蛋白酶AprX进而构建菌株Dpr9,与先前报道的KA8AX菌株相比,α-淀粉酶的产量提高了8倍,约为80 mg/L。敲除8种胞外蛋白酶固然可以有效提高异源蛋白的产量,但同时对细胞的氮源利用效率也会产生不利影响,部分胞外蛋白酶对特定的异源蛋白分泌甚至有积极作用。通过对胞外蛋白酶的选择性敲除构建第二代蛋白酶缺陷型菌株,可以提高异源蛋白的产量[65]。
3 讨论与展望相较于其他异源蛋白表达系统,枯草芽孢杆菌具有遗传操作简单、菌株安全、遗传背景清晰等诸多优势,是生产异源蛋白的优良宿主。基于对枯草芽孢杆菌表达和分泌异源蛋白的机理研究,目前已经开发多种策略用于解决异源蛋白产量过低的问题,包括筛选启动子[14-15]、提高mRNA稳定性[20-21]、筛选信号肽[18, 27]、分泌途径的优化(转运蛋白[28-29]、信号肽酶[11, 30])、过表达伴侣蛋白[22-26]、细胞壁改造[34-35]以及胞外蛋白酶敲除[32-33]等。除了上述热点较高的策略之外,其他新型策略正在逐步被开发,例如:改善全局碳、氮代谢网络调控[36],对细胞基因组进行精简[37],敲除细胞自裂解的相关基因来提高菌株发酵密度[38],均被证明是提高异源蛋白产量的有效方法。
虽然,在枯草芽孢杆菌中表达和分泌异源蛋白已经取得了巨大成就,然而高水平生产真核生物蛋白仍具有较大挑战,例如存在表达质粒不稳定、真核基因转录水平低、蛋白折叠效率低以及蛋白转运效率低等问题。未来,我们建议可着眼于以下3个方面对真核基因的分泌表达瓶颈进行探究:(1) 启动子活性的定义:目前对启动子的研究主要集中于启动子的筛选和优化以及串联启动子,如果能够对启动子的活性有精确定义,有利于研究人员快速便捷地选择最适启动子。(2) 信号肽与编码区的匹配机制:不同异源基因的最适信号肽均不相同,而且两者之间的匹配机制仍尚未清楚。通过生物信息学的方法将所有信号肽进行合理归纳,探究信号肽与编码区的匹配机制,或许可以突破真核基因分泌表达的瓶颈。(3) 异源分子伴侣和目的基因共表达:寻找与目的蛋白相匹配的异源分子伴侣并在枯草芽孢杆菌中共表达,或许可以大幅度提高蛋白折叠效率,防止其在胞内被降解,从而提高异源蛋白产量。同时,可以将新兴技术应用于枯草芽孢杆菌分泌表达异源蛋白这一领域中,例如基因编辑技术:CRISPR/Cas9技术作为靶向基因编辑有力工具,能够在枯草芽孢杆菌中实现多位点基因编辑、多途径精确调控,是未来菌株改造的有效手段[66];孢子表面展示技术:孢子表面展示技术在生产异源蛋白领域具有诸多优势,例如融合蛋白不需要通过任何膜,大大提高分泌效率,然而该技术仍存在困难需要研究人员克服,例如孢子上表达的蛋白数量过低、展示蛋白的活性较低等[67]。
随着蛋白组学和生物信息学的蓬勃发展,对枯草芽孢杆菌的基础研究将会更加深入,表达和分泌异源蛋白的瓶颈有望得到突破。枯草芽孢杆菌将在工业酶、食品酶、药物蛋白生产领域展现出更大潜力,最终实现细胞工厂化。
| [1] |
Dong HN, Zhang DW. Current development in genetic engineering strategies of Bacillus species[J]. Microbial Cell Factories, 2014, 13(1): 63. DOI:10.1186/1475-2859-13-63 |
| [2] |
Zweers JC, Barák I, Becher D, Driessen AJ, Hecker M, Kontinen VP, Saller MJ, Vavrová L, Van Dijl JM. Towards the development of Bacillus subtilis as a cell factory for membrane proteins and protein complexes[J]. Microbial Cell Factories, 2008, 7(1): 10. DOI:10.1186/1475-2859-7-10 |
| [3] |
Xu H, Jiang M, Li H, Lu DQ, Ouyang PK. Efficient production of poly(γ-glutamic acid) by newly isolated Bacillus subtilis NX-2[J]. Process Biochemistry, 2005, 40(2): 519-523. DOI:10.1016/j.procbio.2003.09.025 |
| [4] |
Westbrook AW, Ren X, Moo-Young M, Chou CP. Engineering of cell membrane to enhance heterologous production of hyaluronic acid in Bacillus subtilis[J]. Biotechnology and Bioengineering, 2018, 115(1): 216-231. DOI:10.1002/bit.26459 |
| [5] |
Cheng J, Zhuang W, Li NN, Tang CL, Ying HJ. Efficient biosynthesis of D-ribose using a novel co-feeding strategy in Bacillus subtilis without acid formation[J]. Letters in Applied Microbiology, 2017, 64(1): 73-78. DOI:10.1111/lam.12685 |
| [6] |
Zhang X, Ban R, Liu L, Zhang R. Riboflavin production by a genetically modified Bacillus subtilis[J]. Microbiology China, 2017, 44(1): 59-67. (in Chinese) 张续, 班睿, 刘露, 张然. 枯草芽孢杆菌基因修饰生产核黄素[J]. 微生物学通报, 2017, 44(1): 59-67. |
| [7] |
Wang YH, Ma RJ, Liu L, He L, Ban R. Improvement of uridine production in Bacillus subtilis by metabolic engineering[J]. Biotechnology Letter, 2018, 40(1): 151-155. DOI:10.1007/s10529-017-2453-8 |
| [8] |
Gu Y, Xu XH, Wu YK, Niu TF, Liu YF, Li JH, Du GC, Liu L. Advances and prospects of Bacillus subtilis cellular factories: from rational design to industrial applications[J]. Metabolic Engineering, 2018, 50: 109-121. DOI:10.1016/j.ymben.2018.05.006 |
| [9] |
Chen JQ, Gai YM, Fu G, Zhou WJ, Zhang DW, Wen JP. Enhanced extracellular production of α-amylase in Bacillus subtilis by optimization of regulatory elements and over-expression of PrsA lipoprotein[J]. Biotechnology Letters, 2015, 37(4): 899-906. DOI:10.1007/s10529-014-1755-3 |
| [10] |
Ma RJ, Wang YH, Liu L, Bai LL, Ban R. Production enhancement of the extracellular lipase LipA in Bacillus subtilis: effects of expression system and Sec pathway components[J]. Protein Expression and Purification, 2018, 142: 81-87. DOI:10.1016/j.pep.2017.09.011 |
| [11] |
Song YF, Fu G, Dong HN, Li JJ, Du YG, Zhang DW. High-efficiency secretion of β-mannanase in Bacillus subtilis through protein synthesis and secretion optimization[J]. Journal of Agricultural and Food Chemistry, 2017, 65(12): 2540-2548. DOI:10.1021/acs.jafc.6b05528 |
| [12] |
Westers L, Dijkstra DS, Westers H, Van Dijl JM, Quax WJ. Secretion of functional human interleukin-3 from Bacillus subtilis[J]. Journal of Biotechnology, 2006, 123(2): 211-224. DOI:10.1016/j.jbiotec.2005.11.007 |
| [13] |
Zhou CY, Ye B, Cheng S, Zhao LZ, Liu YX, Jiang JD, Yan X. Promoter engineering enables overproduction of foreign proteins from a single copy expression cassette in Bacillus subtilis[J]. Microbial Cell Factories, 2019, 18(1): 111. DOI:10.1186/s12934-019-1159-0 |
| [14] |
Song YF, Nikoloff JM, Fu G, Chen JQ, Li QG, Xie NZ, Zheng P, Sun JB, Zhang DW. Promoter screening from Bacillus subtilis in various conditions hunting for synthetic biology and industrial applications[J]. PLoS One, 2016, 11(7): e0158447. DOI:10.1371/journal.pone.0158447 |
| [15] |
Li FB, Liu L, Du Y, Ban R. Construction of recombinant Bacillus subtilis as catalyst for preparing D-p-hydroxyphenylglycine[J]. China Biotechnology, 2019, 39(3): 75-86. (in Chinese) 李法彬, 刘露, 杜燕, 班睿. 构建重组枯草芽孢杆菌催化制备D-对羟基苯甘氨酸[J]. 中国生物工程杂志, 2019, 39(3): 75-86. |
| [16] |
Kang HK, Jang JH, Shim HJ, Park JT, Kim YW, Park KH. Efficient constitutive expression of thermostable 4-α-glucanotransferase in Bacillus subtilis using dual promoters[J]. World Journal of Microbiology and Biotechnology, 2010, 26(10): 1915-1918. DOI:10.1007/s11274-010-0351-5 |
| [17] |
Zhang K, Su LQ, Duan XG, Liu LN, Wu J. High-level extracellular protein production in Bacillus subtilis using an optimized dual-promoter expression system[J]. Microbial Cell Factories, 2017, 16(1): 32. DOI:10.1186/s12934-017-0649-1 |
| [18] |
Guan CR, Cui WJ, Cheng JT, Liu R, Liu ZM, Zhou L, Zhou ZM. Construction of a highly active secretory expression system via an engineered dual promoter and a highly efficient signal peptide in Bacillus subtilis[J]. New Biotechnology, 2016, 33(3): 372-379. DOI:10.1016/j.nbt.2016.01.005 |
| [19] |
Nadler F, Bracharz F, Kabisch J. CopySwitch-in vivo optimization of gene copy numbers for heterologous gene expression in Bacillus subtilis[J]. Frontiers in Bioengineering and Biotechnology, 2019, 6: 207. DOI:10.3389/fbioe.2018.00207 |
| [20] |
Li DD, Fu G, Tu R, Jin ZX, Zhang DW. High-efficiency expression and secretion of human FGF21 in Bacillus subtilis by intercalation of a mini-cistron cassette and combinatorial optimization of cell regulatory components[J]. Microbial Cell Factories, 2019, 18(1): 17. DOI:10.1186/s12934-019-1066-4 |
| [21] |
Phan TTP, Nguyen HD, Schumann W. Construction of a 5'-controllable stabilizing element (CoSE) for over-production of heterologous proteins at high levels in Bacillus subtilis[J]. Journal of Biotechnology, 2013, 168(1): 32-39. DOI:10.1016/j.jbiotec.2013.07.031 |
| [22] |
Quesada-Ganuza A, Antelo-Varela M, Mouritzen JC, Bartel J, Becher D, Gjermansen M, Hallin PF, Appel KF, Kilstrup M, Rasmussen M, et al. Identification and optimization of PrsA in Bacillus subtilis for improved yield of amylase[J]. Microbial Cell Factories, 2019, 18(1): 158. DOI:10.1186/s12934-019-1203-0 |
| [23] |
Lindholm A, Ellmén U, Tolonen-Martikainen M, Palva A. Heterologous protein secretion in Lactococcus lactis is enhanced by the Bacillus subtilis chaperone-like protein PrsA[J]. Applied Microbiology and Biotechnology, 2006, 73(4): 904-914. DOI:10.1007/s00253-006-0551-y |
| [24] |
Chen JQ, Fu G, Gai YM, Zheng P, Zhang DW, Wen JP. Combinatorial Sec pathway analysis for improved heterologous protein secretion in Bacillus subtilis: identification of bottlenecks by systematic gene overexpression[J]. Microbial Cell Factories, 2015, 14(1): 92. DOI:10.1186/s12934-015-0282-9 |
| [25] |
Kakeshita H, Kageyama Y, Endo K, Tohata M, Ara K, Ozaki K, Nakamura K. Secretion of biologically-active human interferon-β by Bacillus subtilis[J]. Biotechnology Letters, 2011, 33(9): 1847-1852. DOI:10.1007/s10529-011-0636-2 |
| [26] |
Zhang C, Tao TT, Ying Q, Zhang DL, Lu FX, Bie XM, Lu ZX. Extracellular production of lipoxygenase from Anabaena sp. PCC 7120 in Bacillus subtilis and its effect on wheat protein[J]. Applied Microbiology and Biotechnology, 2012, 94(4): 949-958. DOI:10.1007/s00253-012-3895-5 |
| [27] |
Yao DB, Su LQ, Li N, Wu J. Enhanced extracellular expression of Bacillus stearothermophilus α-amylase in Bacillus subtilis through signal peptide optimization, chaperone overexpression and α-amylase mutant selection[J]. Microbial Cell Factories, 2019, 18(1): 69. DOI:10.1186/s12934-019-1119-8 |
| [28] |
Mulder KCL, Bandola J, Schumann W. Construction of an artificial SecYEG operon allowing high level secretion of α-amylase[J]. Protein Expression and Purification, 2013, 89(1): 92-96. DOI:10.1016/j.pep.2013.02.008 |
| [29] |
Bai LL, Ma RJ, Yang SM, Ban R. Effect of TatAdCd translocases overexpression on the secretion of lipase in Bacillus subtilis[J]. Microbiology China, 2016, 43(1): 2-8. (in Chinese) 白雷雷, 马然静, 杨绍梅, 班睿. 过表达TatAdCd转位酶对枯草芽孢杆菌脂肪酶分泌的影响[J]. 微生物学通报, 2016, 43(1): 2-8. |
| [30] |
Pummi T, Leskelä S, Wahlström E, Gerth U, Tjalsma H, Hecker M, Sarvas M, Kontinen VP. ClpXP protease regulates the signal peptide cleavage of secretory preproteins in Bacillus subtilis with a mechanism distinct from that of the Ecs ABC transporter[J]. Journal of Bacteriology, 2002, 184(4): 1010-1018. DOI:10.1128/jb.184.4.1010-1018.2002 |
| [31] |
Neef J, Bongiorni C, Goosens VJ, Schmidt B, Van Dijl JM. Intramembrane protease RasP boosts protein production in Bacillus[J]. Microbial Cell Factories, 2017, 16(1): 57. DOI:10.1186/s12934-017-0673-1 |
| [32] |
Wu SC, Yeung JC, Duan YJ, Ye RQ, Szarka SJ, Habibi HR, Wong SL. Functional production and characterization of a fibrin-specific single-chain antibody fragment from Bacillus subtilis: effects of molecular chaperones and a wall-bound protease on antibody fragment production[J]. Applied and Environmental Microbiology, 2002, 68(7): 3261-3269. DOI:10.1128/AEM.68.7.3261-3269.2002 |
| [33] |
Kodama T, Endo K, Sawada K, Ara K, Ozaki K, Kakeshita H, Yamane K, Sekiguchi J. Bacillus subtilis AprX involved in degradation of a heterologous protein during the late stationary growth phase[J]. Journal of Bioscience and Bioengineering, 2007, 104(2): 135-143. DOI:10.1263/jbb.104.135 |
| [34] |
Hyyryläinen HL, Vitikainen M, Thwaite J, Wu HY, Sarvas M, Harwood CR, Kontinen VP, Stephenson K. D-Alanine substitution of teichoic acids as a modulator of protein folding and stability at the cytoplasmic membrane/cell wall interface of Bacillus subtilis[J]. Journal of Biological Chemistry, 2000, 275(35): 26696-26703. DOI:10.1016/S0021-9258(19)61432-8 |
| [35] |
Cao HJ, Van Heel AJ, Ahmed H, Mols M, Kuipers OP. Cell surface engineering of Bacillus subtilis improves production yields of heterologously expressed α-amylases[J]. Microbial Cell Factories, 2017, 16(1): 56. DOI:10.1186/s12934-017-0674-0 |
| [36] |
Cao HJ, Villatoro-Hernandez J, Weme RDO, Frenzel E, Kuipers OP. Boosting heterologous protein production yield by adjusting global nitrogen and carbon metabolic regulatory networks in Bacillus subtilis[J]. Metabolic Engineering, 2018, 49: 143-152. DOI:10.1016/j.ymben.2018.08.001 |
| [37] |
Toya Y, Hirasawa T, Morimoto T, Masuda K, Kageyama Y, Ozaki K, Ogasawara N, Shimizu H. 13C-metabolic flux analysis in heterologous cellulase production by Bacillus subtilis genome-reduced strain[J]. Journal of Biotechnology, 2014, 179: 42-49. DOI:10.1016/j.jbiotec.2014.03.025 |
| [38] |
Wang Y, Chen ZM, Zhao RL, Jin TT, Zhang XM, Chen XD. Deleting multiple lytic genes enhances biomass yield and production of recombinant proteins by Bacillus subtilis[J]. Microbial Cell Factories, 2014, 13(1): 129. |
| [39] |
Cui WJ, Han LC, Suo FY, Liu ZM, Zhou L, Zhou ZM. Exploitation of Bacillus subtilis as a robust workhorse for production of heterologous proteins and beyond[J]. World Journal of Microbiology and Biotechnology, 2018, 34(10): 145. DOI:10.1007/s11274-018-2531-7 |
| [40] |
Xiong HT, Wei YT. Research progress of Bacillus subtilis expression system and its promoter regulatory elements[J]. Guangxi Sciences, 2018, 25(3): 233-241. (in Chinese) 熊海涛, 韦宇拓. 枯草芽孢杆菌表达系统及其启动子的研究进展[J]. 广西科学, 2018, 25(3): 233-241. |
| [41] |
Estacio W, Anna-Arriola SS, Adedipe M, Márquez-Magaña LM. Dual promoters are responsible for transcription initiation of the fla/che operon in Bacillus subtilis[J]. Journal of Bacteriology, 1998, 180(14): 3548-3555. DOI:10.1128/JB.180.14.3548-3555.1998 |
| [42] |
Zhang X, Xu ZY, Liu S, Qian K, Xu MJ, Yang TW, Xu JZ, Rao ZM. Improving the production of salt-tolerant glutaminase by integrating multiple copies of Mglu into the protease and 16S rDNA genes of Bacillus subtilis 168[J]. Molecules, 2019, 24(3): 592. DOI:10.3390/molecules24030592 |
| [43] |
Lehnik-Habrink M, Lewis RJ, Mäder U, Stülke J. RNA degradation in Bacillus subtilis: an interplay of essential endo- and exoribonucleases[J]. Molecular Microbiology, 2012, 84(6): 1005-1017. DOI:10.1111/j.1365-2958.2012.08072.x |
| [44] |
Song YF, Nikoloff JM, Zhang DW. Improving protein production on the level of regulation of both expression and secretion pathways in Bacillus subtilis[J]. Journal of Microbiology and Biotechnology, 2015, 25(7): 963-977. DOI:10.4014/jmb.1501.01028 |
| [45] |
Wong SL. Advances in the use of Bacillus subtilis for the expression and secretion of heterologous proteins[J]. Current Opinion in Biotechnology, 1995, 6(5): 517-522. DOI:10.1016/0958-1669(95)80085-9 |
| [46] |
Zhang DW, Kang Q. Development and prospect of protein expression and secretion systems in Bacillus subtilis[J]. Journal of Microbiology, 2019, 39(1): 1-10. (in Chinese) 张大伟, 康倩. 枯草芽胞杆菌蛋白质表达分泌系统发展及展望[J]. 微生物学杂志, 2019, 39(1): 1-10. DOI:10.3969/j.issn.1005-7021.2019.01.001 |
| [47] |
Chatzi KE, Sardis MF, Tsirigotaki A, Koukaki M, Šoštarić N, Konijnenberg A, Sobott F, Kalodimos CG, Karamanou S. Preprotein mature domains contain translocase targeting signals that are essential for secretion[J]. Journal of Cell Biology, 2017, 216(5): 1357-1369. DOI:10.1083/jcb.201609022 |
| [48] |
Goosens VJ, Monteferrante CG, Van Dijl JM. The tat system of Gram-positive bacteria[J]. Biochimica et Biophysica Acta (BBA)-Molecular Cell Research, 2014, 1843(8): 1698-1706. DOI:10.1016/j.bbamcr.2013.10.008 |
| [49] |
Mogk A, Völker A, Engelmann S, Hecker M, Schumann W, Völker U. Nonnative proteins induce expression of the Bacillus subtilis CIRCE regulon[J]. Journal of Bacteriology, 1998, 180(11): 2895-2900. DOI:10.1128/JB.180.11.2895-2900.1998 |
| [50] |
Kontinen VP, Sarvas M. The PrsA lipoprotein is essential for protein secretion in Bacillus subtilis and sets a limit for high-level secretion[J]. Molecular Microbiology, 1993, 8(4): 727-737. DOI:10.1111/j.1365-2958.1993.tb01616.x |
| [51] |
Freudl R. Signal peptides for recombinant protein secretion in bacterial expression systems[J]. Microbial Cell Factories, 2018, 17(1): 52. DOI:10.1186/s12934-018-0901-3 |
| [52] |
Mu DD, Lu JJ, Qiao MQ, Kuiper OP, Zhu J, Li XJ, Yang PZ, Zhao YY, Luo SZ, Wu XF, et al. Heterologous signal peptides-directing secretion of Streptomyces mobaraensis transglutaminase by Bacillus subtilis[J]. Applied Microbiology and Biotechnology, 2018, 102(13): 5533-5543. DOI:10.1007/s00253-018-9000-y |
| [53] |
Kakeshita H, Kageyama Y, Ara K, Ozaki K, Nakamura K. Enhanced extracellular production of heterologous proteins in Bacillus subtilis by deleting the C-terminal region of the SecA secretory machinery[J]. Molecular Biotechnology, 2010, 46(3): 250-257. DOI:10.1007/s12033-010-9295-0 |
| [54] |
Kouwen TRHM, Van Der Ploeg R, Antelmann H, Hecker M, Homuth G, Mäder U, Van Dijl JM. Overflow of a hyper-produced secretory protein from the Bacillus Sec pathway into the Tat pathway for protein secretion as revealed by proteogenomics[J]. Proteomics, 2009, 9(4): 1018-1032. DOI:10.1002/pmic.200800580 |
| [55] |
Bolhuis A, Sorokin A, Azevedo V, Ehrlich SD, Braun PG, De Jong A, Venema G, Bron S, Van Dijl MJ. Bacillus subtilis can modulate its capacity and specificity for protein secretion through temporally controlled expression of the sipS gene for signal peptidase I[J]. Molecular Microbiology, 1996, 22(4): 605-618. DOI:10.1046/j.1365-2958.1996.d01-4676.x |
| [56] |
Vitikainen M, Pummi T, Airaksinen U, Wahlstrom E, Wu HY, Sarvas M, Kontinen VP. Quantitation of the capacity of the secretion apparatus and requirement for PrsA in growth and secretion of α-amylase in Bacillus subtilis[J]. Journal of Bacteriology, 2001, 183(6): 1881-1890. DOI:10.1128/JB.183.6.1881-1890.2001 |
| [57] |
Ploss TN, Reilman E, Monteferrante CG, Denham EL, Piersma S, Lingner A, Vehmaanperä J, Lorenz P, Van Dijl JM. Homogeneity and heterogeneity in amylase production by Bacillus subtilis under different growth conditions[J]. Microbial Cell Factories, 2016, 15(1): 57. DOI:10.1186/s12934-016-0455-1 |
| [58] |
Yan SM, Wu G. Proteases HtrA and HtrB for α-amylase secreted from Bacillus subtilis in secretion stress[J]. Cell Stress and Chaperones, 2019, 24(3): 493-502. DOI:10.1007/s12192-019-00985-1 |
| [59] |
Pohl S, Bhavsar G, Hulme J, Bloor AE, Misirli G, Leckenby MW, Radford DS, Smith W, Wipat A, Williamson ED, et al. Proteomic analysis of Bacillus subtilis strains engineered for improved production of heterologous proteins[J]. Proteomics, 2013, 13(22): 3298-3308. DOI:10.1002/pmic.201300183 |
| [60] |
Vitikainen M, Hyyryläinen HL, Kivimäki A, Kontinen VP, Sarvas M. Secretion of heterologous proteins in Bacillus subtilis can be improved by engineering cell components affecting post‐translocational protein folding and degradation[J]. Journal of Applied Microbiology, 2005, 99(2): 363-375. DOI:10.1111/j.1365-2672.2005.02572.x |
| [61] |
Neef J, Bongiorni C, Schmidt B, Goosens VJ, Van Dijl JM. Relative contributions of non-essential Sec pathway components and cell envelope-associated proteases to high-level enzyme secretion by Bacillus subtilis[J]. Microbial Cell Factories, 2020, 19(1): 52. DOI:10.1186/s12934-020-01315-2 |
| [62] |
Chen YZ, Cai DB, He PH, Mo F, Zhang Q, Ma X, Chen SW. Enhanced production of heterologous proteins by Bacillus licheniformis with defective D-alanylation of lipoteichoic acid[J]. World Journal of Microbiology and Biotechnology, 2018, 34(9): 135. DOI:10.1007/s11274-018-2520-x |
| [63] |
Krishnappa L, Dreisbach A, Otto A, Goosens VJ, Cranenburgh RM, Harwood CR, Becher D, Van Dijl JM. Extracytoplasmic proteases determining the cleavage and release of secreted proteins, lipoproteins, and membrane proteins in Bacillus subtilis[J]. Journal of Proteome Research, 2013, 12(9): 4101-4110. DOI:10.1021/pr400433h |
| [64] |
Wu XC, Lee W, Tran L, Wong SL. Engineering a Bacillus subtilis expression-secretion system with a strain deficient in six extracellular proteases[J]. Journal of Bacteriology, 1991, 173(16): 4952-4958. DOI:10.1128/jb.173.16.4952-4958.1991 |
| [65] |
Zhao LZ, Ye B, Zhang Q, Cheng D, Zhou CY, Cheng S, Yan X. Construction of second generation protease-deficient hosts of Bacillus subtilis for secretion of foreign proteins[J]. Biotechnology and Bioengineering, 2019, 116(8): 2052-2060. DOI:10.1002/bit.26992 |
| [66] |
Watzlawick H, Altenbuchner J. Multiple integration of the gene ganA into the Bacillus subtilis chromosome for enhanced β-galactosidase production using the CRISPR/Cas9 system[J]. AMB Express, 2019, 9(1): 158. DOI:10.1186/s13568-019-0884-4 |
| [67] |
Wang H, Wang YX, Yang RJ. Recent progress in Bacillus subtilis spore-surface display: concept, progress, and future[J]. Applied Microbiology and Biotechnology, 2017, 101(3): 933-949. DOI:10.1007/s00253-016-8080-9 |
 2021, Vol. 48
2021, Vol. 48