扩展功能
文章信息
- 尚玉婷, 叶青华, 刘振杰, 吴清平, 张菊梅
- SHANG Yuting, YE Qinghua, LIU Zhenjie, WU Qingping, ZHANG Jumei
- 微生物高通量快速检测技术研究进展
- Research progress on high-throughput rapid detection technology of microorganism
- 微生物学通报, 2021, 48(8): 2801-2814
- Microbiology China, 2021, 48(8): 2801-2814
- DOI: 10.13344/j.microbiol.china.201178
-
文章历史
- 收稿日期: 2020-12-23
- 接受日期: 2021-02-05
- 网络首发日期: 2021-03-19
2. 江南大学食品学院 食品科学与技术国家重点实验室 国家功能食品工程技术研究中心 食品安全与质量控制协同创新中心 江苏 无锡 214122
2. State Key Laboratory of Food Science and Technology, National Engineering Research Center for Functional Food, Synergetic Innovation Center of Food Safety and Quality Control, School of Food Science and Technology, Jiangnan University, Wuxi, Jiangsu 214122, China
微生物在大自然中广泛存在,和我们的生产、生活、环境有着密切的关系。其中由食源性致病菌(主要包括大肠杆菌[1]、沙门氏菌[2]、金黄色葡萄球菌[3]、单增李斯特菌[4]、副溶血性弧菌[5]、蜡样芽孢杆菌[6]和阪崎肠杆菌[7]等)引起的食物中毒和食源性疾病,严重威胁着人类的健康。据统计,在美国每年约4 800万人次患食源性疾病,12万多人住院治疗,3 000人死亡[8]。在我国每年由食品中致病菌引起的食源性疾病报告病例数约占全部报告的40%-50%[9]。由此可见,食源性致病菌严重威胁着人类的健康和国家经济的发展。虽然传统的微生物检测方法被视为“金标准”,但其操作步骤烦琐、工作量大、耗时较长,难以满足突发性食品安全事件和基层日常监管的需求,特别是在我国食品需求量大、生产规模较小、零散化等特点的现状下,提高抽样量是十分必要的。如何高通量快速地检测出微生物的存在,特别是食源性致病菌显得尤为重要。
高通量快速检测技术是指一次可检测多个样品或在同一反应体系中同时完成多个检测目的的技术。首先要快速准确地识别目标菌,主要包括双功能抗体、核酸适配体筛选和噬菌体鉴定等方法。其中双功能抗体是一种新兴的技术,在性能等方面优于抗体分子,已广泛应用于医学,在微生物学检测领域具有一定的发展潜力。然而核酸适配体筛选和噬菌体鉴定是目前研究的热点,其可以快速准确地识别目标菌、产量高、成本低,可替代传统的抗体分子。其次在多目标检测方面,主要包括多重PCR、基质辅助激光解吸电离飞行时间质谱和生物芯片等方法。多重PCR应用较广,但检测能力相对有限。基质辅助激光解吸电离飞行时间质谱可实现对病原菌的非靶向检测,但属于精密仪器,体积较大,不利于实现现场化操作。生物芯片,具有样品用量少、灵敏度高、自动化、检测结果快速直观等优点,结合上述快速识别分子,可高效准确地实现对目标菌的检测。本文重点阐述各方法的原理、优缺点及研究现状,为今后我国微生物学领域检测技术的快速发展提供参考。
1 目标菌快速识别技术 1.1 双功能抗体技术双功能抗体(Bispecific Antibodies,BsAbs)技术,又称双特异性或双价抗体,可以同时识别2个完全不相关的抗原表位,由于其特异性和双功能性,现已广泛应用于肿瘤免疫治疗及自身免疫性疾病等领域。双功能性抗体在自然状态下不存在,只能通过人工制备,制备方法包括:(1) 化学交联法。利用重链和轻链之间的二硫键经还原和再氧化,将2种不同特异性抗体的半分子结合在一起。该方法简单易行,但在连接过程中可能会导致合成的BsAbs失活、聚集或解折叠;(2) 杂化杂交瘤技术。将2个或3个不同的细胞系进行融合,形成二源或三源杂交瘤细胞,从而产生BsAbs。优点是融合成本低、效率高,缺点是产量低、产物需不断筛选、费时费力且瘤株染色体容易丢失,导致分泌抗体情况不稳定;(3) 基因工程法。利用DNA重组技术设计产生BsAbs。抗体均一性、特异性强,交叉反应少,可工业化生产,但技术难度大,费用高。BsAbs在结构上有多种设计策略,主要分为不含Fc区的小型抗体(由单链抗体或Fab区组成)和含有Fc区的全抗体2类[10-11]。近几年,国内外已对BsAbs的制备及其应用进行了大量的研究,尤其在肿瘤靶向治疗方面,揭示了BsAbs可通过介导免疫细胞杀伤、双靶点信号阻断或促进蛋白形成功能性复合体等机制发挥作用[12],但与病原菌的检测相关研究报道较少。Owais等于2014年首次将BsAbs应用于食品中单增李斯特菌的检测,其原理是,首先分别制备红细胞识别抗体和单增李斯特菌识别抗体,通过化学交联法,将2种抗体进行交联,得到可同时识别两者的BsAbs。然后在待检样品中加入红细胞和BsAbs,样品中的单增李斯特菌会与BsAbs结合,诱导与抗体结合的红细胞在20-30 min发生聚集,产生血凝现象,这种血凝现象可直接用肉眼观察,从而实现了对单增李斯特菌的检测。该方法的优点在于不需要对样品进行增菌培养,而且灵敏度在102-103 CFU/mL之间,大大提高了检测效率[13]。由于BsAbs的制备仍面临着一些挑战,因此BsAbs在病原菌检测方面目前仍处在发展初期。
1.2 适配体筛选技术核酸适配体(Aptamer)是运用指数富集配体系统筛选技术(Systematic Evolution of Ligands by Exponential Enrichment,SELEX),即通过连续多轮筛选,从大量适配体中(适配体库)以指数衰减的方式获得少数对靶分子有高亲和力的配体。核酸适配体一般是单链寡核苷酸(ssDNA或RNA),由20-80个碱基组成,可形成多种三维空间结构(发夹、茎环、假结、G-四链体等二级结构),通过静电、氢键、范德华力、碱基堆积力及构象互补等方式,与靶分子特异性结合,从而实现对金属离子、有机小分子、多肽、蛋白质甚至完整细胞和微生物的检测。相比于抗体,核酸适配体具有多种优点(表 1),被誉为“化学抗体”。适配体SELEX筛选步骤主要包括:(1) 单链寡核苷酸文库的构建。首先构建一个容量约为1013-1015的随机文库,寡核苷酸分子的两端是固定序列(20-30个碱基,含有PCR扩增引物结合序列和限制性核酸内切酶作用位点),中间是随机序列(20-60个碱基,与靶分子特异性结合);(2) 适配体的分离、筛选富集。将随机寡核苷酸文库与靶分子混合孵育,利用特定的方法将结合与未结合靶分子的寡核苷酸分离,然后洗脱结合的寡核苷酸,并以其为模板进行PCR扩增得到新文库,如此循环6-20次,即可获得与靶分子高特异性结合的适配体。常见的分离方法主要有:硝酸纤维素膜过滤法、亲和层析法、磁珠分离法、高速离心法、毛细管电泳法和氧化石墨烯吸附法等。此外,半自动或自动化和高通量的筛选系统也已建立;(3) 适配体的序列分析。将经过筛选富集得到的适配体进行碱基测序,空间立体结构模拟和亲和力、特异性、稳定性等特性分析,从而实现大批量生产。新兴的筛选方法有很多,如反向SELEX、消减SELEX、光敏SELEX、混合SELEX、切换SELEX、表达盒SELEX、复合靶SELEX、基因SELEX和细胞SELEX等,这些方法大大缩短了筛选周期,提高了筛选效率[14]。针对致病菌的适配体筛选,一般以全细胞或菌体表面分子(蛋白质、脂多糖等)为靶标,2种方式各有优势。前者无需准确了解菌体的表面分子及其结构信息,筛选后的适配体可直接用于实际样品的检测,省去了分离、纯化菌体表面分子等步骤,缩短了实验时间[15];后者降低了以活细胞为靶标带来的生物安全隐患,可清楚地了解与致病菌的结合位点,有利于机制研究[16]。目前,基于适配体作为识别元件的检测方法可分为荧光法、比色法、电化学法和表面等离子共振(Surface Plamon Resonace,SPR)、表面增强拉曼散射(Surface Enhanced Raman Scattering,SERS)等无标记法。Xu等用PEG水凝胶作为显色探针,修饰适配体后,实现了对大肠杆菌和金黄色葡萄球菌2种病原菌的同时检测,检测限为100 CFU/mL[17]。利用相似的方法,Kurt等用上转化材料和量子点分别标记鼠伤寒沙门氏菌和金黄色葡萄球菌的适配体,由于2种发光材料激发波长不同,荧光发射波长之间无重叠现象,因此可分别在980 nm和325 nm激发光下实现对相应菌株的检测,检测限为28 CFU/mL和16 CFU/mL[18]。碳纳米材料和氧化石墨烯可通过π-π堆积作用将荧光标记的适配体吸附于表面,导致荧光淬灭。加入目标菌后,适配体与靶标相结合使荧光信号再度恢复,从而快速有效地检测病原菌。Duan等用碳纳米材料和3种荧光标记的适配体,实现了对3种病原菌的同时检测[19]。Zuo等将氧化石墨烯和适配体整合于微流控芯片上,在不同反应池中修饰不同适配体,可实现对多种病原菌的检测[20]。适配体还可以与纳米金相结合,当目标菌存在时,适配体从纳米金表面脱落,导致纳米金团聚,颜色由红色变为蓝色。Wu等运用该原理,实现了鼠伤寒沙门氏菌和大肠杆菌O157:H7的检测[21]。此外,将适配体修饰在电极表面,可以通过检测电流、电阻等信号的变化来鉴定病原菌。Sheikhzadeh等通过共聚物分子将鼠伤寒沙门氏菌适配体连接于金电极表面,运用电化学阻抗谱法鉴定病原菌,定量限为102 CFU/mL,检测限为3 CFU/mL[22]。无标记检测无需任何标记分子,具有简单便捷、灵敏度高等优点,近几年得到了迅速发展。Yoo等将巯基化适配体与贵金属粒子(Ag和Au)包被的芯片表面相连接,通过SPR原理,实现对3种病原菌的检测[23]。Zhang等用SERS适配体传感器实现了对鼠伤寒沙门氏菌和金黄色葡萄球菌的同时检测,检测限分别为35 CFU/mL和15 CFU/mL;在纳米磁珠表面同时固定2种菌的适配体作为捕获探针,然后将拉曼探针分子巯基苯甲酸(Mercaptobenzoic Anzoic,MBA)与鼠伤寒沙门氏菌适配体、5, 5′-二硫代双(2-硝基苯甲酸)[(5, 5′-Dithiobis (2-Nitrobenzoic Acid)),DNTB]与金黄色葡萄球菌适配体分别修饰在金纳米粒子表面,作为信号探针,采用“夹心法”实现对菌株的鉴定[24]。其今后的发展重点包括:(1) 新型高效筛选方法的突破,增加适配体种类,特别是在真菌类;(2) 通过生物信息学手段,揭示适配体与靶分子相互作用机制;(3) 建立更多可同时检测多种食源性致病菌的适配体传感器,实现其向高通量、集成化、自动化方向发展。
| Characteristic | Aptamer | Antibody |
| Screening method | Simple and easy to perform, without experimental animals | Animal immune response, complicated operation |
| Screening time | 2-3 months, minimum 2 weeks | 3–6 months |
| Application | Wide, more than 200 targets | Narrow, limited in number |
| Purity | High, little variation between individuals | Low, easy to produce non-specific antibodies |
| Affinity | High, up to picomolar level | Relatively low, micromolar or nanomolar level |
| Stability | High temperature resistance, acid and alkali resistance, room temperature storage | Environmentally sensitive, low temperature storage |
| Modification | Terminal is easily modified with various active groups | Proteins are easily inactivated after modification |
| Cost | Low | High |
噬菌体(Bacteriophage)是一类可以感染细菌的病毒,主要由核酸(ssDNA、dsDNA或RNA遗传物质)和蛋白质衣壳组成,不能独立生存,必须依赖于宿主细胞。凡是有细菌的地方,都有相应噬菌体的存在。据估计,噬菌体的数量是细菌的10倍,可达到1031,目前已用电镜观察到的超过5 500种[25]。根据噬菌体的生活周期不同,可将其分为烈性噬菌体(λ、T4和T7噬菌体)和温和噬菌体(M13和fd噬菌体)。鉴于噬菌体具有结构简单、繁殖速度快,寄主专一性强等特点,现已广泛应用于食源性致病菌的快速检测及控制。噬菌体检测方法的原理涉及噬菌体繁殖全过程(吸附、侵入、增殖、装配和裂解),大致可分以下5种:
(1) 基于噬菌体的传统检测方法
由于烈性噬菌体能够高度专一性地裂解宿主菌,并在培养基上形成肉眼可见的噬菌斑,因此根据噬菌斑的数目可以检测样品中病原菌的种类和数量。根据此原理可对细菌进行分型,相比于传统的方法,其操作简单、省时、成本低。当样品中待检细菌数量较少时,不利于噬菌斑的形成,但可通过富集培养和噬菌体扩增法这2种途径来解决。噬菌体扩增法是利用辅助菌配合噬菌体对细菌的裂解,从而在双层平板中形成明显的噬菌斑[26]。Duc等利用此法在2 h内成功检测出食品中1.3 lg (CFU/mL)的鼠伤寒沙门氏菌和大肠杆菌O157:H7[27]。但是该方法对噬菌体的浓度、感染时间、辅助菌的选择等条件都有严格的要求,实验室必须配备一整套分型所用的噬菌体及对照株,而且单独使用难以达到很高的准确度,因此其常与其他技术联合使用。Banu等使用T7噬菌体扩增法结合LC-MRM-MS/MS技术,成功检测了LB培养基、椰子汁和苹果汁中的大肠杆菌,检测限分别为3.0×103、4.1×104和1.9×103 CFU/mL[28]。Cox等使用γ噬菌体扩增法结合免疫层析试纸条,在2 h内实现了对炭疽杆菌的检测,检测限为2.5×104 CFU/mL[29]。
(2) 报告噬菌体法
报告噬菌体法是利用基因工程方法将报告基因整合于噬菌体基因组上,然后通过噬菌体的识别与侵染,将核酸释放入宿主细胞内,随着报告基因的表达,目标菌便可得到检测。常用于标记的报告基因有:荧光素酶基因(lux,luc)、绿色荧光蛋白基因(gfp)、冰核蛋白基因(inaW)、β-半乳糖苷酶基因(lacZ)和糖酶基因(celB),其具体检测机理及优缺点见参考文献[30]。Kim等构建了luxCDABE报告噬菌体,在40 min内实现了对纯培养大肠杆菌O157:H7的检测,检测限为1 CFU/mL,在莴苣、苹果汁和牛肉等食物样品中的检测限分别为10 CFU/cm2、13 CFU/mL和17 CFU/g[31]。但该方法目前还未被广泛应用,主要归因于对噬菌体的遗传学特性研究还不够清晰,很难对其进行基因改造,因此在很大程度上限制了其发展。
(3) 噬菌体与发光试剂联用
基于噬菌体最初对宿主菌的识别和吸附,将噬菌体标记上荧光或发光材料,从而作为一种标签去检测目标菌。例如通过基因改造噬菌体的衣壳蛋白,使其表面具有生物素或四半胱氨酸标记的多肽,然后分别与链霉亲和素化的量子点、双砷染料相结合,形成发光噬菌体。Yim等利用量子点标记的λ噬菌体检测大肠杆菌,证明了λ噬菌体与量子点的结合率高于T7噬菌体,使λ噬菌体更有利于提高检测的灵敏度[32]。Imai等用SiO2@AuNP标记噬菌体,实现了对金黄色葡萄球菌的检测[33]。此外,可使用商业化的核酸染料(YOYO-1、SRBY、SYTO)直接标记噬菌体的DNA或RNA分子。Mosier-Boss等用SYBR标记P22噬菌体的dsDNA,然后在荧光显微镜下成功检测到低于100 CFU/mL的沙门氏菌[34]。
生物发光法是利用噬菌体裂解宿主菌,释放胞内物质(ATP、AK、NADH等),然后在荧光素酶等物质作用下发光,通过测定荧光强度,推算出菌落总数。ATP、AK和NADH的生物发光反应机理分别如下式(1)、(2)、(3)所示[35]:
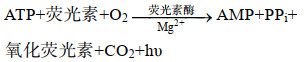
|
(1) |

|
(2) |
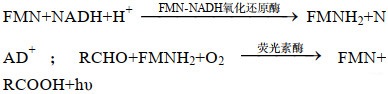
|
(3) |
Minikh等利用ATP生物发光法在2 h内检测出样品中的大肠杆菌,检测限为6×103 CFU/mL[36]。但由于食品或环境样品中ATP本底值的影响,导致灵敏度较低。Wu等利用AK生物发光法,提高了样品发光强度,可在2 h内检测出低于103 CFU/mL的大肠杆菌和沙门氏菌[37]。Mei等利用NADH生物发光法检测出肺炎克雷伯氏菌[38]。噬菌体与发光试剂联用方法操作简单,省时省力。基于此,目前Sample6公司推出了一种基于噬菌体和生物荧光素酶结合的商业化检测系统,但该方法仍需要专业检测设备,如流式细胞仪、荧光显微镜、扫描电子显微镜等,其结果重现性不佳,标记染料和催化酶价格昂贵,在实际应用推广时受到了限制。
(4) 噬菌体与生物传感器技术联用
将噬菌体作为识别元件的生物传感器近几年得到了迅速发展,其可应用于电化学传感器、光学传感器、离子敏场效应传感器、压电晶体传感器、磁传感器等方面,大量文献对其进行了归纳总结[39-40]。噬菌体传感器操作简单、分析速度快,但其抗干扰能力差、可靠性低,目前仍不能取代传统的生物传感器(以抗体、酶、核酸等作为识别原件),噬菌体在表面的结合取向和比率,是决定该方法检测灵敏度的一个重要决定因素。
此外,细菌在生长繁殖过程中,会将培养基中大分子电惰性物质转化成具有电活性的小分子产物,使培养基的导电性增加,而噬菌体的加入会抑制病原菌的生长,因此可以通过测量培养基电阻抗的变化值,鉴定样品中的目标菌。噬菌体对目标菌裂解产生的胞内酶,如β-D-半乳糖苷酶、α-葡萄糖苷酶等,也可通过循环伏安法(Cyclic Voltammetry,CV)、差分脉冲伏安法(Differential Pulse Voltammetry,DPV)、方波伏安法(Square Wave Voltammetry,SWV)等电化学测量方法来检测。Farooq等在电极表面修饰噬菌体,用DPV法检测金黄色葡萄球菌[41]。Neufeld等用CV法监测细胞释放的β-D-半乳糖苷酶,也成功检测出大肠杆菌[42]。但并不是所有的细菌都能够代谢产生电活性物质或具有特殊的胞内酶,因此该方法检测范围具有一定的局限性。
(5) 其他方法
噬菌体在适当的条件下,以200n进行增殖,即一个裂解周期内,每个噬菌体产生200个子代噬菌体。这就意味着可通过HPLC-MS、MALDI-TOF MS、PCR等技术来监测样品中噬菌体蛋白质或核酸的变化情况,从而实现对目标菌的检测。
除噬菌体整体应用外,噬菌体的某些结构成分或表达成分也可单独使用,如尾丝蛋白(Tail Spike Proteins,TSP)、内溶素的细胞壁结合区域(Cell Wall Binding Domain,CBD)、噬菌体展示技术等。内溶素是噬菌体在感染细菌后期表达产生的一种溶菌酶,其CBD结构域可高度识别并结合宿主细胞壁上的特异位点,从而替代全噬菌体的使用。如CBD与免疫磁珠结合用于病原菌的分离和富集,标记荧光蛋白用于细菌分型,作为识别元件构建生物传感器[43]。噬菌体展示技术是将外源基因所编码的多肽或蛋白以融合蛋白形式表达于噬菌体表面,从而实现基因型与表观型之间的偶联。目前,通过噬菌体展示技术制备的特异性噬菌体、抗体、多肽等常作为探针应用于食源性致病菌的检测方法中[44]。利用噬菌体对食源性致病菌进行检测具有良好的发展前景,但应注意到其使用的安全性,比如温和性噬菌体的溶源现象可能会改变宿主的表型甚至毒性。此外,如何快速筛选出特异性噬菌体、拓宽其抑菌谱、制备高纯度制剂等方面也将成为今后学术研究应关注的重点。
2 多目标物检测技术 2.1 多重PCR技术多重PCR (Multiplex PCR,mPCR)技术是指在同一核酸反应体系中,加入2对或2对以上的特异性引物,分别扩增不同目的基因,从而实现对某一病原菌的多个基因或多种病原菌的同时检测。其反应原理与传统PCR基本相同,既保留了传统PCR特异性强、灵敏性高的特点,又节约了反应时间和试剂用量,是一种既经济又高效的高通量检测方法[45]。
在利用多重PCR开展检测过程中,引物设计是反应成功的关键,不合理的设计极易引起引物二聚体或非特异性扩增产物的形成,造成假阳性结果。在遵循引物设计主要原则的基础上,各引物对之间的最佳退火温度应尽可能相同且无任何交互反应。在检测数量方面,mPCR通常可实现一个病原菌2-4个基因或2-4个目标菌的同时检测,甚至更多。Zhou等基于cigR基因,实现了对沙门氏菌属和属内鸡白痢/鸡伤寒沙门氏菌血清型的检测[46]。Meena等开发了可同时检测粪肠球菌3种主要毒力基因(gelE、hyl和asaI)的多重PCR试剂盒[47]。Latha等实现了对肉制品中鼠伤寒沙门氏菌、金黄色葡萄球菌、单增李斯特菌和小肠结肠炎耶尔森氏菌4种致病菌的检测[48],而Villamizar-Rodríguez等实现了对5种不同类型食品中9种致病菌的检测[49]。但是由于食品基质成分复杂(个别微生物含量低或含有扩增抑制因子),容易导致检测灵敏度降低,出现假阴性结果。因此与富集培养、免疫磁珠吸附、二氧化硅吸附膜等样品前处理技术相结合或加入扩增内标(Internal Amplification Control,IAC)可有效提高检测的效率和准确度。肉制品中的沙门氏菌在BPW中经过12 h富集培养后,其检测限为104 CFU/mL[50]。经过免疫磁分离后,沙门氏菌在纯菌中的检测限可达101 CFU/mL,在肉制品中为102 CFU/g[51]。将16S rRNA基因作为IAC,实现了对大肠杆菌O157:H7、沙门氏菌和单增李斯特菌的精确检测,检测限为102 CFU/mL[52]。
其次,实时荧光定量mPCR可对样品初始拷贝数进行定量分析,省去扩增后处理步骤,有效避免交叉污染问题。Alía等向反应体系中加入TaqMan探针,实现了对单核增生李斯特菌不同血清型的检测,4种血清型的特异性基因和16S rRNA作为扩增基因[53]。Skerniškytė等用同样的方法实现了对4种病原菌的同时检测,通过绘制扩增曲线,计算得出每个菌株的Ct值[54]。但由于该方法需要专门的仪器设备,特异性探针合成成本较高,目前仍处于发展阶段。
mPCR与传统PCR一样存在着难以区分死菌和活菌的缺陷,为了克服这一缺陷,可向核酸提取液中加入核酸交联剂,如叠氮溴化乙锭(Ethidium Monoazide,EMA)、叠氮溴化丙锭(Propidium Monoazide,PMA)等。PMA不能透过完整的细胞膜,但能与细胞膜受损后暴露的DNA分子结合,形成共价交联,使死菌的DNA分子在扩增时被抑制,从而提高mPCR检测活菌的准确率[55]。Liang等用PMA对目标菌进行处理,实现了对食品中沙门氏菌、大肠杆菌、大肠杆菌O157:H7、金黄色葡萄球菌和志贺氏菌的检测。添加脱氧胆酸钠(Sodium Deoxycholate,SD)可进一步破坏受损菌的细胞膜,促进PMA进入死菌细胞内[56]。此外,SD和PMA还可应用于实时荧光定量mPCR中,如Qin等用实时荧光定量mPCR结合SD和PMA处理,实现了对牛奶中单增李斯特菌、阪崎克罗诺杆菌、金黄色葡萄球菌和沙门氏菌活菌的检测[57]。mPCR所具备的这些优势,必将使其成为高通量快速检测食源性致病菌的重要方法之一。通过不断优化mPCR的反应条件,并与其他技术相结合,如基因芯片、变性梯度凝胶电泳等,可以营造一个更完整的体系,进一步提升其在检测过程中的有效性与可行性。
2.2 基质辅助激光解吸电离飞行时间质谱技术基质辅助激光解吸电离飞行时间质谱(Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry,MALDI-TOF MS)技术是近年发展起来的一种软电离质谱手段,其主要工作原理和流程为:将样品与基质的混合液加在样品靶板上,待溶剂挥发后形成结晶体,通过激光轰击结晶体,基质分子吸收能量后与样品解吸附并使其电离,样品离子在电场的作用下加速通过飞行管道,由于离子的质荷比与飞行时间成正比,根据到达检测器的飞行时间不同测定质荷比,经过数据的采集与记录,即可获得病原菌特征性的指纹图谱。然后利用相关软件将获得的指纹图谱与数据库中的标准图谱进行比对,根据比对分值的高低,鉴定待测微生物的属、种乃至亚种的水平。通常认为匹配分值大于2.0鉴定到种;1.7-2.0之间鉴定到属;小于1.7鉴定结果不可信[58-59]。由于蛋白质约占细胞干重的50%,其表达由遗传性状决定,受外界环境影响较小,因此成为MALDI-TOF MS检测病原菌的主要生物标志,峰值范围在2-20 kD m/z之间,大部分峰来自核糖体蛋白,少部分来自冷休克蛋白和DNA结合蛋白。常用基质主要包括:α-氰基-4-羟基肉桂酸(Alpha-Cyano-4-Hydroxycinnamic Acid,CHCA)、3, 5-二甲氧基-4-羟基肉桂酸(Sinapinic Acid,SA)、2, 5-二羟基苯甲酸(2, 5-Dihydroxybenzoic Acid,DHB)、2-巯基苯并噻唑(2-Mercaptobenzothiazole,MBT)和2-巯基-5-氯苯并噻唑(5-Chloro-2-Mercaptobenzothiazole,CMBT)等。MALDI-TOF MS系统主要有4种:MALDI Biotyper (Bruker Daltonics)、VITEK MS (BioMérieux)、AXIMA @ SARAMIS (AnagnosTec)和Andromas SAS (Andromas),其中前2种系统已获得中国食品和药品监督管理局许可证,可用于临床检测[60]。MALDI-TOF MS技术凭借多种优势(表 2),已成功运用于细菌和真菌的快速鉴定、耐药性筛查及临床标本的检测。
| Advantages | Disadvantages |
| 1. Rapid turnaround time, within 30 min | 1. Unable to differentiate between certain highly related organisms |
| 2. Low cost per test, 17%–32% of the cost of conventional methods | 2. High initial instrument cost |
| 3. High-throughput | 3. Requires pure culture or well-isolated colonies |
| 4. Reliable and reproducible | 4. Certain organisms (e.g., fungi) require protein extraction before analysis |
| 5. Minimal isolate required (as little as a single colony) | 5. Ability to detect antibiotic resistance is variable |
| 6. Ability to identify a broad range of microorganisms on a single platform | 6. Unable to perform quantitation |
| 7. Easy to use with respect to sample preparation, running, and data analysis | 7. The number of species in the database is insufficient to meet the needs of special identification |
| 8. According to the protein fingerprint spectrum for serovar analysis[61-62] | 8. Microbial culture conditions and protein extraction methods may affect the identification results |
| 9. Absence of practice guidelines for validation implementation, or reporting of results | |
| 10. At the time of writing, neither commercially available platform has Food and Drug Administration clearance for in vitro |
在细菌鉴定方面,大部分革兰氏阳性菌和革兰氏阴性菌都能准确鉴定,如Jadhav等通过选择性富集培养,检测了牛奶、奶酪和鸡肉中的单增李斯特菌[63]。Xu等对北京市200份蔬菜样品中的大肠杆菌、鼠伤寒沙门氏菌、金黄色葡萄球菌和单增李斯特菌进行了检测[64]。通常阴性菌鉴定准确率高于阳性菌,需氧菌高于厌氧菌。但也存在一些缺陷,如在革兰氏阴性菌中,由于大肠埃希菌和志贺菌的指纹图谱很相似,因此两者很难区分;在革兰氏阳性菌中,链球菌无法准确鉴定,尤其是亲缘关系比较相近的肺炎链球菌和缓症链球菌等;厌氧菌对培养和鉴定条件要求苛刻,参考谱图还比较少,仍需不断完善;分枝杆菌生长缓慢、培养周期长,在鉴定时需经特殊蛋白提取步骤,以提高准确率,但目前仍不能区分结核分枝杆菌复合群和一些密切相关的种属[65]。
在真菌鉴定方面,通常酵母菌的鉴定正确率要高于霉菌。一些真菌无法鉴定或鉴定错误,主要原因是数据库不全面,尤其是丝状真菌,由于具有多种形态,如孢子、子实体、表面菌丝和基内菌丝体等,使其蛋白谱图可能因生长状况和取样部位的不同而发生变化,影响谱图的再现性[66-67]。
此外,MALDI-TOF MS技术也可应用于对耐药菌株的检测与鉴定,主要是通过寻找耐药菌株与敏感菌株指纹图谱峰的改变,或将耐药菌株与抗生素共培养,然后检测耐药酶或抗生素的分解产物。目前研究较多的是关于革兰氏阴性杆菌对β-内酰胺类药物产生的耐药性,主要通过MALDI-TOF MS检测β-内酰胺类药物的水解产物,或直接检测β-内酰胺酶分子、肽段和其相关蛋白[68]。Alksne等应用MALDI-TOF MS技术区分了耐甲氧西林金黄色葡萄球菌和甲氧西林敏感金黄色葡萄球菌[69];同样,Giordano等用该技术鉴定了耐多粘菌素肺炎克雷伯菌,进行了暴发流行病学的调查[70]。但是,该方法对于其他细菌及真菌耐药机制的研究尚不明确。
目前,MALDI-TOF MS技术已成功应用于临床标本的检测,如血培养标本、尿标本和脑髓液等。样本经过离心、过滤、溶解血细胞等预处理即可上机检测,大大缩短了检测时间[71-73]。
MALDI-TOF MS技术的发展与应用,将会成为微生物检测领域一个划时代的变革,但仍有许多问题亟待改进与解决,特别是作为该技术核心部分的数据库,仍需不断地完善和改进,以及如何及时整合不同实验室的资源,针对不同型号的设备建立一套标准化的工作流程,也将是今后需要努力的方向之一。
2.3 生物芯片生物芯片技术是指将特定的探针(基因片段或抗体)固定于载体(玻璃片、硅片、尼龙膜或高分子聚合物等)上,在一定条件下,通过碱基互补配对或抗原抗体特异性免疫的原理与待测样品进行杂交,用同位素法、化学荧光法、化学发光法、酶标法对结果进行显示,然后通过检测系统的扫描和计算机的整合分析,从而实现对DNA、RNA、多肽或蛋白质的检测[74-75]。其具有高度的平行化、自动化和微型化等特点,可同时在一张芯片上进行多个目标物的检测,化学试剂消耗量少,是一种环境友好型的技术。目前以基因芯片和液相芯片在食源性致病菌检测中应用较多。
将已知的寡核苷酸或cDNA片段按序列排列在载体上,构成了基因芯片,其工作流程主要包括4个步骤:芯片制备、样品制备与标记、杂交反应、信号检测和分析[74]。(1) 芯片制备。主要有原位合成法和直接点样法,高密度芯片通常采用原位合成法,低密度芯片则采用直接点样法。该步骤需要大量已知的DNA或cDNA片段信息,充分利用这些信息才能使芯片技术实现集成化,但目前尚缺乏公开可得的、准确的基因序列。芯片的制作系统较为昂贵,限制了其广泛的应用;(2) 样品制备与标记。样品的制备与标记是一个重要环节,细胞或组织中提取出的DNA或RNA,经过逆转录、PCR扩增后进行末端标记,标记方法有荧光标记、生物素标记、同位素标记和新兴的纳米材料标记等。通常使用荧光标记,如Suo等结合mPCR,用荧光染料Cy3进行末端标记,实现了对12个样品中大肠杆菌O157:H7、沙门氏菌、单增李斯特菌和空肠弯曲菌的同时检测,检测限达到1×10-4 ng/μL[76]。Brennan等用非对称PCR进行扩增,Cy5标记,可用于大肠杆菌ssrA基因的筛选[77]。但荧光染料容易发生光漂白,而且缺乏足够数量的荧光染料来标记DNA,而纳米材料的开发与应用在一定程度上避免了上述问题。Huang等利用量子点作为荧光标记物,实现了对10种病原菌的同时检测,检测限为10 CFU/mL[78]。Wang等用金标银染可视化方法,实现了对8种病原菌的同时检测,检测限在3.3–85 CFU/mL之间[79]。虽然目前已存在多种样品标记方法,但其操作过程仍比较复杂,而且各研究机构未形成统一标准,资源数据未能共享;(3) 杂交反应。芯片与样品的杂交属于固-液相反应,反应体系中盐浓度、探针长度和所带电荷、待检基因的质量等因素都会影响结果的准确性和重复性,导致不同的实验室或实验平台得到的结果可能各不相同,因此该技术真正应用到实际操作中仍需要进一步规范标准化;(4) 信号检测和分析。目前商业化的芯片荧光扫描仪有激光共聚焦扫描仪和CCD荧光扫描仪两大类,但其价格昂贵,不利于推广。可视化方法作为一项突破性技术,不再需要光学仪器检测系统,而且使用便捷。Li等将碱性磷酸酶与底物之间的酶学显色反应引入检测体系,使芯片可视化,检测了4种病原菌,检测限为102 CFU/mL[80]。Zhang等则应用辣根过氧化物酶的显色反应,实现了对3种病原菌的同时检测,检测限为5 copies/30 fg[81]。
液相芯片是由美国Luminex公司基于多重分析(Flexible Multi-Analyte Profiling,xMAP)技术而研发的新一代生物芯片技术平台,是继基因芯片、蛋白芯片之后的又一高通量分子检测方法。其原理是用荧光染料编码聚苯乙烯微球,使每种荧光微球都具有一个独特的编号,一个编号的微球偶联一种探针,该探针可捕获互补的待测核酸或蛋白,检测时加入样品和报告分子藻红蛋白,样品中的目标分子能够与探针和报告分子发生特异性结合,从而形成微球-目标分子-报告分子三元复合物。然后运用流式细胞技术和激光技术,使微球逐个通过检测通道。红色激光激发微球本身的荧光编码,从而鉴定各个不同的反应类型(即定性),绿色激光激发目标分子上的藻红蛋白,从而确定微球上结合目标分子的数量(即定量)。目前已推出Luminex 100、Luminex 200和Flexmap 3D这3种液相芯片检测系统。Luminex 100运用2种荧光染料编码100种微球,可同时对1个样本中的100种不同目标分子进行检测。Luminex 200可根据需要调节探针高度,增强检测的准确性。Flexmap 3D加入第3种荧光染料,使微球扩充到500种,可同时对96个样本中的500种不同目标分子进行检测[82]。该技术的优点包括[83]:(1) 检测通量高。最高可达500种,而且使用者只需增减探针和报告分子即可满足不同的检测需要;(2) 特异性强。检测系统只记录同时出现红、绿色的荧光信号,未结合的荧光信号不被记录,所以无交叉荧光信号产生;(3) 检测灵敏度高且速度快。由于微球悬浮于液体中,增加了反应面积,从而提高了结合效率。Lusk Pfefer等利用液相芯片技术鉴定了布鲁氏菌的8个种,此平台可实现对96个样本中100种不同目标分子的同时检测[84]。Gand等结合多重寡核苷酸连接-PCR方法,实现了对沙门氏菌6种血清型的同时检测,准确率高达99.7%[85]。Sun等结合mPCR,实现了对6种病原菌的同时检测,检测限为1.6×10-6 mmol/L,检测灵敏度比传统PCR高5倍[86]。虽然液相芯片大大缩短了检测时间,但应用于多种核酸检测时,需要进行mPCR扩增,mPCR扩增个数的能力,限制了其高通量的检测。目前商品化的试剂盒覆盖率不高,仍需自行设计引物和探针,而且荧光微球基本上依赖进口,拥有自主知识产权的荧光微球还有待开发。
虽然生物芯片技术仍面临着一系列亟待解决的问题,但相信随着基因组学的深入和芯片技术的完善,其今后必将会有更广阔的发展空间。
3 展望与传统方法相比,高通量快速检测技术具有明显的优势,并成为微生物领域的研究热点。综上所述,在不同的情况下,各检测方法均有利弊,仍处于不断完善的阶段。在今后该技术的不断发展过程中,许多问题尚待进一步改进:
(1) 前增菌问题。由于目标菌含量低,食品基质成分干扰等因素,往往需要对样品进行增菌处理,但培养时间较长,因此建立一种能够同时选择性富集多种目标菌的多重培养基,是实现高通量检测亟须解决的问题;
(2) 假阳(阴)性结果。在目标菌检测过程中,死亡的菌株已不可繁殖,但仍会被分子生物学技术检测到,造成假阳性结果。亚致死细胞不能通过培养得到损伤修复,导致无法识别,造成假阴性结果。因此,需要改进方法以能够有效区别死活菌,构建检测内标,从而避免假性结果的出现,提高检测的准确性;
(3) 绝对定量分析。大部分检测方法仅能进行半定量或相对定量分析,而实现目标菌的快速绝对定量分析十分重要。相信随着各种检测方法的不断发展与完善,未来必将实现对微生物的高通量快速检测。
| [1] |
Zhang SH, Wu QP, Zhang JM, Zhu XM. Occurrence and characterization of enteropathogenic Escherichia coli (EPEC) in retail ready-to-eat foods in China[J]. Foodborne Pathogens and Disease, 2016, 13(1): 49-55. DOI:10.1089/fpd.2015.2020 |
| [2] |
Chen L, Zhang JM, Yang XJ, Wu QP, Xu MF. Prevalence and characterization of Salmonella spp. from foods in South China[J]. Acta Microbiologica Sinica, 2013, 53(12): 1326-1333. (in Chinese) 陈玲, 张菊梅, 杨小鹃, 吴清平, 徐明芳. 南方食品中沙门氏菌污染调查及分型[J]. 微生物学报, 2013, 53(12): 1326-1333. |
| [3] |
Yang XJ, Zhang JM, Yu SB, Wu QP, Guo WP, Huang JH, Cai SZ. Prevalence of Staphylococcus aureus and methicillin-resistant Staphylococcus aureus in retail ready-to-eat foods in China[J]. Frontiers in Microbiology, 2016, 7: 816. |
| [4] |
Wu S, Wu QP, Zhang JM, Chen MT, Zéan Y. Prevalence, antibiotic resistance and genetic diversity of Listeria monocytogenes isolated from retail ready-to-eat foods in China[J]. Food Control, 2015, 47: 340-347. DOI:10.1016/j.foodcont.2014.07.028 |
| [5] |
Xu XK, Wu QP, Zhang JM, Cheng JH, Zhang SH, Wu K. Prevalence, pathogenicity, and serotypes of Vibrio parahaemolyticus in shrimp from Chinese retail markets[J]. Food Control, 2014, 46: 81-85. DOI:10.1016/j.foodcont.2014.04.042 |
| [6] |
Yu PF, Yu SB, Wang J, Guo H, Zhang Y, Liao XY, Zhang JH, Wu S, Gu QH, Xue L, et al. Bacillus cereus isolated from vegetables in China: incidence, genetic diversity, virulence genes, and antimicrobial resistance[J]. Frontiers in Microbiology, 2019, 10: 948. DOI:10.3389/fmicb.2019.00948 |
| [7] |
Xu XK, Li CS, Wu QP, Zhang JM, Huang JH, Yang GZ. Prevalence, molecular characterization, and antibiotic susceptibility of Cronobacter spp. in Chinese ready-to-eat foods[J]. International Journal of Food Microbiology, 2015, 204: 17-23. DOI:10.1016/j.ijfoodmicro.2015.03.003 |
| [8] |
Estimates of Foodborne Illness in the United States[EB/OL]. https://www.cdc.gov/foodsafety/foodborne-germs.html
|
| [9] |
Chen XM, Yang H, Gui GH, Dai BL, Wang PP, Xiao YP. Analysis of food poisoning situation in China from 2008 to 2015[J]. China Food Safety Magazine, 2017(25): 69-73. (in Chinese) 陈小敏, 杨华, 桂国弘, 戴宝玲, 王佩佩, 肖英平. 2008-2015年全国食物中毒情况分析[J]. 食品安全导刊, 2017(25): 69-73. DOI:10.3969/j.issn.1674-0270.2017.25.028 |
| [10] |
Wu XF, Demarest SJ. Building blocks for bispecific and trispecific antibodies[J]. Methods, 2019, 154: 3-9. DOI:10.1016/j.ymeth.2018.08.010 |
| [11] |
Hosseini SS, Khalili S, Baradaran B, Bidar N, Shahbazi MA, Mosafer J, Hashemzaei M, Mokhtarzadeh A, Hamblin MR. Bispecific monoclonal antibodies for targeted immunotherapy of solid tumors: recent advances and clinical trials[J]. International Journal of Biological Macromolecules, 2021, 167: 1030-1047. DOI:10.1016/j.ijbiomac.2020.11.058 |
| [12] |
Suurs FV, Lub-de Hooge MN, De Vries EGE, De Groot DJA. A review of bispecific antibodies and antibody constructs in oncology and clinical challenges[J]. Pharmacology & Therapeutics, 2019, 201: 103-119. |
| [13] |
Owais M, Kazmi S, Tufail S, Zubair S. An alternative chemical redox method for the production of bispecific antibodies: implication in rapid detection of food borne pathogens[J]. PLoS One, 2014, 9(3): e91255. DOI:10.1371/journal.pone.0091255 |
| [14] |
Ning Y, Hu J, Lu FG. Aptamers used for biosensors and targeted therapy[J]. Biomedicine & Pharmacotherapy, 2020, 132: 110902. |
| [15] |
Liu PD, Qu F. Research advances of aptamer selection for whole cell[J]. Chinese Journal of Chromatography, 2016, 34(4): 382-388. (in Chinese) 刘品多, 屈锋. 全细胞的核酸适配体筛选的研究进展[J]. 色谱, 2016, 34(4): 382-388. |
| [16] |
Xie PY, Zhu LJ, Xu WT. Application progress of aptamers in the detection of food-borne pathogenic bacteria[J]. Biotechnology Bulletin, 2016, 32(4): 48-62. (in Chinese) 解沛燕, 朱龙佼, 许文涛. 适配体在食源性致病菌检测中的应用进展[J]. 生物技术通报, 2016, 32(4): 48-62. |
| [17] |
Xu YS, Wang H, Luan CX, Liu YX, Chen BA, Zhao YJ. Aptamer-based hydrogel barcodes for the capture and detection of multiple types of pathogenic bacteria[J]. Biosensors and Bioelectronics, 2018, 100: 404-410. DOI:10.1016/j.bios.2017.09.032 |
| [18] |
Kurt H, Yüce M, Hussain B, Budak H. Dual-excitation upconverting nanoparticle and quantum dot aptasensor for multiplexed food pathogen detection[J]. Biosensors and Bioelectronics, 2016, 81: 280-286. DOI:10.1016/j.bios.2016.03.005 |
| [19] |
Duan N, Gong WH, Wang ZP, Wu SJ. An aptasensor based on fluorescence resonance energy transfer for multiplexed pathogenic bacteria determination[J]. Analytical Methods, 2016, 8(6): 1390-1395. DOI:10.1039/C5AY02608C |
| [20] |
Zuo P, Li XJ, Dominguez DC, Ye BC. A PDMS/paper/glass hybrid microfluidic biochip integrated with aptamer-functionalized graphene oxide nano-biosensors for one-step multiplexed pathogen detection[J]. Lab on a Chip, 2013, 13(19): 3921-3928. DOI:10.1039/c3lc50654a |
| [21] |
Wu WH, Li M, Wang Y, Ouyang HX, Wang L, Li CX, Cao YC, Meng QH, Lu JX. Aptasensors for rapid detection of Escherichia coli O157:H7 and Salmonella typhimurium[J]. Nanoscale Research Letters, 2012, 7(1): 658. DOI:10.1186/1556-276X-7-658 |
| [22] |
Sheikhzadeh E, Chamsaz M, Turner APF, Jager EWH, Beni V. Label-free impedimetric biosensor for Salmonella Typhimurium detection based on poly[pyrrole-co-3-carboxyl-pyrrole] copolymer supported aptamer[J]. Biosensors and Bioelectronics, 2016, 80: 194-200. DOI:10.1016/j.bios.2016.01.057 |
| [23] |
Yoo SM, Kim DK, Lee SY. Aptamer-functionalized localized surface plasmon resonance sensor for the multiplexed detection of different bacterial species[J]. Talanta, 2015, 132: 112-117. DOI:10.1016/j.talanta.2014.09.003 |
| [24] |
Zhang H, Ma XY, Liu Y, Duan N, Wu SJ, Wang ZP, Xu BC. Gold nanoparticles enhanced SERS aptasensor for the simultaneous detection of Salmonella typhimurium and Staphylococcus aureus[J]. Biosensors and Bioelectronics, 2015, 74: 872-877. DOI:10.1016/j.bios.2015.07.033 |
| [25] |
Ackermann HW. 5500 Phages examined in the electron microscope[J]. Archives of Virology, 2007, 152(2): 227-243. DOI:10.1007/s00705-006-0849-1 |
| [26] |
Van Der Merwe RG, Van Helden PD, Warren RM, Sampson SL, Van Pittius NCG. Phage-based detection of bacterial pathogens[J]. The Analyst, 2014, 139(11): 2617-2626. DOI:10.1039/C4AN00208C |
| [27] |
Duc HM, Son HM, Yi HPS, Sato J, Ngan PH, Masuda Y, Honjoh KI, Miyamoto T. Isolation, characterization and application of a polyvalent phage capable of controlling Salmonella and Escherichia coli O157:H7 in different food matrices[J]. Food Research International, 2020, 131: 108977. DOI:10.1016/j.foodres.2020.108977 |
| [28] |
Banu M, Ng D, Zheng L, Goh LT, Bi XZ, Ow DSW. Rapid quantification of Escherichia coli in food and media using bacteriophage T7 amplification and liquid chromatography-multiple reaction monitoring tandem mass spectrometry[J]. Journal of Biotechnology, 2014, 192: 50-58. DOI:10.1016/j.jbiotec.2014.10.017 |
| [29] |
Cox CR, Jensen KR, Mondesire RR, Voorhees KJ. Rapid detection of Bacillus anthracis by γ phage amplification and lateral flow immunochromatography[J]. Journal of Microbiological Methods, 2015, 118: 51-56. DOI:10.1016/j.mimet.2015.08.011 |
| [30] |
Schmelcher M, Loessner MJ. Application of bacteriophages for detection of foodborne pathogens[J]. Bacteriophage, 2014, 4(2): e28137. DOI:10.4161/bact.28137 |
| [31] |
Kim J, Kim M, Kim S, Ryu S. Sensitive detection of viable Escherichia coli O157:H7 from foods using a luciferase-reporter phage phiV10lux[J]. International Journal of Food Microbiology, 2017, 254: 11-17. DOI:10.1016/j.ijfoodmicro.2017.05.002 |
| [32] |
Yim PB, Clarke ML, McKinstry M, De Paoli Lacerda SH, Pease LF Ⅲ, Dobrovolskaia MA, Kang H, Read TD, Sozhamannan S, Hwang J. Quantitative characterization of quantum dot-labeled lambda phage for Escherichia coli detection[J]. Biotechnology and Bioengineering, 2009, 104(6): 1059-1067. DOI:10.1002/bit.22488 |
| [33] |
Imai M, Mine KH, Tomonari H, Uchiyama J, Matuzaki S, Niko Y, Hadano S, Watanabe S. Dark-field microscopic detection of bacteria using bacteriophage-immobilized SiO2@AuNP core-shell nanoparticles[J]. Analytical Chemistry, 2019, 91(19): 12352-12357. DOI:10.1021/acs.analchem.9b02715 |
| [34] |
Mosier-Boss PA, Lieberman SH, Andrews JM, Rohwer FL, Wegley LE, Breitbart M. Use of fluorescently labeled phage in the detection and identification of bacterial species[J]. Applied Spectroscopy, 2003, 57(9): 1138-1144. DOI:10.1366/00037020360696008 |
| [35] |
Li M, Wang JX, Lin H. Research progress of the use of bacteriophage to detect foodborne pathogenic bacteria[J]. Food Science, 2010, 31(23): 439-446. (in Chinese) 李萌, 王静雪, 林洪. 噬菌体检测食源性致病菌的研究进展[J]. 食品科学, 2010, 31(23): 439-446. |
| [36] |
Minikh O, Tolba M, Brovko LY, Griffiths MW. Bacteriophage-based biosorbents coupled with bioluminescent ATP assay for rapid concentration and detection of Escherichia coli[J]. Journal of Microbiological Methods, 2010, 82(2): 177-183. DOI:10.1016/j.mimet.2010.05.013 |
| [37] |
Wu Y, Brovko L, Griffiths MW. Influence of phage population on the phage-mediated bioluminescent adenylate kinase (AK) assay for detection of bacteria[J]. Letters in Applied Microbiology, 2001, 33(4): 311-315. DOI:10.1046/j.1472-765X.2001.01002.x |
| [38] |
Mei CX, Wang JX, Lin H, Li M, Cui LQ. Rapid detection of Klesiella by bacterial luciferase system combined with the bacteriophage lysis[C]. International Conference of National Product and Traditional Medicine, 2009
|
| [39] |
Farooq U, Yang QL, Ullah MW, Wang SQ. Bacterial biosensing: recent advances in phage-based bioassays and biosensors[J]. Biosensors and Bioelectronics, 2018, 118: 204-216. DOI:10.1016/j.bios.2018.07.058 |
| [40] |
Singh S, Dhanjal DS, Sonali, Thotapalli S, Kumar V, Datta S, Kumar V, Kumar M, Singh J. An insight in bacteriophage based biosensors with focus on their detection methods and recent advancements[J]. Environmental Technology & Innovation, 2020, 20: 101081. |
| [41] |
Farooq U, Ullah MW, Yang QL, Aziz A, Xu JJ, Zhou L, Wang SQ. High-density phage particles immobilization in surface-modified bacterial cellulose for ultra-sensitive and selective electrochemical detection of Staphylococcus aureus[J]. Biosensors and Bioelectronics, 2020, 157: 112163. DOI:10.1016/j.bios.2020.112163 |
| [42] |
Neufeld T, Schwartz-Mittelmann A, Biran D, Ron EZ, Rishpon J. Combined phage typing and amperometric detection of released enzymatic activity for the specific identification and quantification of bacteria[J]. Analytical Chemistry, 2003, 75(3): 580-585. DOI:10.1021/ac026083e |
| [43] |
Kong M, Sim J, Kang T, Nguyen HH, Park HK, Chung BH, Ryu S. A novel and highly specific phage endolysin cell wall binding domain for detection of Bacillus cereus[J]. European Biophysics Journal, 2015, 44(6): 437-446. DOI:10.1007/s00249-015-1044-7 |
| [44] |
Zhao YY, Zhao SM, Liang Y, Liu XJ, Zhang H, Li HB, Tu K. Application of phage display technology in food safety[J]. Science and Technology of Food Industry, 2017, 38(14): 342-346. (in Chinese) 赵岩岩, 赵圣明, 梁颖, 刘贤金, 张浩, 李红波, 屠康. 噬菌体展示技术在食品安全分析中的应用[J]. 食品工业科技, 2017, 38(14): 342-346. |
| [45] |
Zhong ZC, Wang J, Zhang SY. Advances in multiple PCR technology studies[J]. Chinese Journal of Biotechnology, 2020, 36(2): 171-179. (in Chinese) 钟泽澄, 王进, 张师音. 多重PCR技术研究进展[J]. 生物工程学报, 2020, 36(2): 171-179. |
| [46] |
Zhou YY, Kang XL, Meng C, Xiong D, Xu Y, Geng SZ, Pan ZM, Jiao XN. Multiple PCR assay based on the cigR gene for detection of Salmonella spp. and Salmonella Pullorum/Gallinarum identification[J]. Poultry Science, 2020, 99(11): 5991-5998. DOI:10.1016/j.psj.2020.07.026 |
| [47] |
Meena B, Anburajan L, Varma KS, Vinithkumar NV, Kirubagaran R, Dharani G. A multiplex PCR kit for the detection of three major virulent genes in Enterococcus faecalis[J]. Journal of Microbiological Methods, 2020, 177: 106061. DOI:10.1016/j.mimet.2020.106061 |
| [48] |
Latha C, Anu CJ, Ajaykumar VJ, Sunil B. Prevalence of Listeria monocytogenes, Yersinia enterocolitica, Staphylococcus aureus, and Salmonella enterica Typhimurium in meat and meat products using multiplex polymerase chain reaction[J]. Veterinary World, 2017, 10(8): 927-931. DOI:10.14202/vetworld.2017.927-931 |
| [49] |
Villamizar-Rodríguez G, Fernández J, Marín L, Muñiz J, González I, Lombó F. Multiplex detection of nine food-borne pathogens by mPCR and capillary electrophoresis after using a universal pre-enrichment medium[J]. Frontiers in Microbiology, 2015, 6: 1194. |
| [50] |
Chin WH, Sun Y, Høgberg J, Quyen TL, Engelsmann P, Wolff A, Bang DD. Direct PCR: a rapid method for multiplexed detection of different serotypes of Salmonella in enriched pork meat samples[J]. Molecular and Cellular Probes, 2017, 32: 24-32. DOI:10.1016/j.mcp.2016.11.004 |
| [51] |
Li F, Li FL, Chen BL, Zhou BQ, Yu P, Yu S, Lai WH, Xu HY. Sextuplex PCR combined with immunomagnetic separation and PMA treatment for rapid detection and specific identification of viable Salmonella spp., Salmonella enterica serovars Paratyphi B, Salmonella Typhimurium, and Salmonella Enteritidis in raw meat[J]. Food Control, 2017, 73: 587-594. DOI:10.1016/j.foodcont.2016.09.009 |
| [52] |
Nguyen TT, Van Giau V, Vo TK. Multiplex PCR for simultaneous identification of E. coli O157:H7, Salmonella spp. and L. monocytogenes in food[J]. 3 Biotech, 2016, 6(2): 1-9. |
| [53] |
Alía A, Andrade MJ, Córdoba JJ, Martín I, Rodríguez A. Development of a multiplex real-time PCR to differentiate the four major Listeria monocytogenes serotypes in isolates from meat processing plants[J]. Food Microbiology, 2020, 87: 103367. DOI:10.1016/j.fm.2019.103367 |
| [54] |
Skerniškytė J, Armalytė J, Kvietkauskaitė R, Šeputienė V, Povilonis J, Sužiedėlienė E. Detection of Salmonella spp., Yersinia enterocolitica, Listeria monocytogenes and Campylobacter spp. by real-time multiplex PCR using amplicon DNA melting analysis and probe-based assay[J]. International Journal of Food Science and Technology, 2016, 51(2): 519-529. DOI:10.1111/ijfs.12990 |
| [55] |
Nocker A, Cheung CY, Camper AK. Comparison of propidium monoazide with ethidium monoazide for differentiation of live vs. dead bacteria by selective removal of DNA from dead cells[J]. Journal of Microbiological Methods, 2006, 67(2): 310-320. DOI:10.1016/j.mimet.2006.04.015 |
| [56] |
Liang TB, Zhou P, Zhou BQ, Xu Q, Zhou ZQ, Wu X, Aguilar ZP, Xu HY. Simultaneous quantitative detection of viable Escherichia coli O157:H7, Cronobacter spp., and Salmonella spp. using sodium deoxycholate-propidium monoazide with multiplex real-time PCR[J]. Journal of Dairy Science, 2019, 102(4): 2954-2965. DOI:10.3168/jds.2018-15736 |
| [57] |
Qin H, Shi XQ, Yu L, Li K, Wang JW, Chen JH, Yang F, Xu HY, Xu HL. Multiplex real-time PCR coupled with sodium dodecyl sulphate and propidium monoazide for the simultaneous detection of viable Listeria monocytogenes, Cronobacter sakazakii, Staphylococcus aureus and Salmonella spp. in milk[J]. International Dairy Journal, 2020, 108: 104739. DOI:10.1016/j.idairyj.2020.104739 |
| [58] |
Angeletti S. Matrix assisted laser desorption time of flight mass spectrometry (MALDI-TOF MS) in clinical microbiology[J]. Journal of Microbiological Methods, 2017, 138: 20-29. DOI:10.1016/j.mimet.2016.09.003 |
| [59] |
Gu CH. MALDI-TOF MS technique and its application challenges in food microbiology detection[J]. China Brewing, 2019, 38(9): 24-27. (in Chinese) 顾春华. MALDI-TOF MS技术及其在食品微生物检测方面的应用[J]. 中国酿造, 2019, 38(9): 24-27. |
| [60] |
Sauget M, Valot B, Bertrand X, Hocquet D. Can MALDI-TOF mass spectrometry reasonably type bacteria?[J]. Trends in Microbiology, 2017, 25(6): 447-455. DOI:10.1016/j.tim.2016.12.006 |
| [61] |
Mangmee S, Reamtong O, Kalambaheti T, Roytrakul S, Sonthayanon P. MALDI-TOF mass spectrometry typing for predominant serovars of non-typhoidal Salmonella in a Thai broiler industry[J]. Food Control, 2020, 113: 107188. DOI:10.1016/j.foodcont.2020.107188 |
| [62] |
Kyritsi MA, Kristo I, Hadjichristodoulou C. Serotyping and detection of pathogenecity loci of environmental isolates of Legionella pneumophila using MALDI-TOF MS[J]. International Journal of Hygiene and Environmental Health, 2020, 224: 113441. DOI:10.1016/j.ijheh.2019.113441 |
| [63] |
Jadhav S, Sevior D, Bhave M, Palombo EA. Detection of Listeria monocytogenes from selective enrichment broth using MALDI-TOF mass spectrometry[J]. Journal of Proteomics, 2014, 97: 100-106. DOI:10.1016/j.jprot.2013.09.014 |
| [64] |
Xu XM, Liu GY, Huang XD, Li LY, Lin H, Xu DH. MALDI-TOF MS-based identification of bacteria and a survey of fresh vegetables with pathogenic bacteria in Beijing, China[J]. Food Bioscience, 2021, 41: 100746. DOI:10.1016/j.fbio.2020.100746 |
| [65] |
Hou TY, Chiang-Ni C, Teng SH. Current status of MALDI-TOF mass spectrometry in clinical microbiology[J]. Journal of Food and Drug Analysis, 2019, 27(2): 404-414. DOI:10.1016/j.jfda.2019.01.001 |
| [66] |
Chalupová J, Raus M, Sedlářová M, Šebela M. Identification of fungal microorganisms by MALDI-TOF mass spectrometry[J]. Biotechnology Advances, 2014, 32(1): 230-241. DOI:10.1016/j.biotechadv.2013.11.002 |
| [67] |
Quéro L, Courault P, Cellière B, Lorber S, Jany JL, Puel O, Girard V, Vasseur V, Nodet P, Mounier J. Application of MALDI-TOF MS to species complex differentiation and strain typing of food related fungi: case studies with Aspergillus section Flavi species and Penicillium roqueforti isolates[J]. Food Microbiology, 2020, 86: 103311. DOI:10.1016/j.fm.2019.103311 |
| [68] |
Yu JJ, Yu J, Liu Y. Research progresses of applying MALDI-TOF mass spectrometry in the detection of β-lactamase[J]. Journal of Shanghai Jiao Tong University: Medical Science, 2017, 37(4): 566-570. (in Chinese) 余佳佳, 俞静, 刘瑛. 基质辅助激光解析电离飞行时间质谱检测β-内酰胺酶的研究进展[J]. 上海交通大学学报: 医学版, 2017, 37(4): 566-570. DOI:10.3969/j.issn.1674-8115.2017.04.029 |
| [69] |
Alksne L, Makarova S, Avsejenko J, Cibrovska A, Trofimova J, Valciņa O. Determination of methicillin-resistant Staphylococcus aureus and Staphylococcus epidermidis by MALDI-TOF MS in clinical isolates from Latvia[J]. Clinical Mass Spectrometry, 2020, 16: 33-39. DOI:10.1016/j.clinms.2020.03.001 |
| [70] |
Giordano C, Barnini S. Rapid detection of colistin-resistant Klebsiella pneumoniae using MALDI-TOF MS peak-based assay[J]. Journal of Microbiological Methods, 2018, 155: 27-33. DOI:10.1016/j.mimet.2018.11.008 |
| [71] |
Fang C, Zhou Z, Li JP, Chen XJ, Zhou MM. Rapid identification of microorganisms from positive blood cultures in pediatric patients by MALDI-TOF MS: sepsityper kit versus short-term subculture[J]. Journal of Microbiological Methods, 2020, 172: 105894. DOI:10.1016/j.mimet.2020.105894 |
| [72] |
Curtoni A, Cipriani R, Marra ES, Barbui AM, Cavallo R, Costa C. Rapid identification of microorganisms from positive blood culture by MALDI-TOF MS after short-term incubation on solid medium[J]. Current Microbiology, 2017, 74(1): 97-102. DOI:10.1007/s00284-016-1161-2 |
| [73] |
March Rosselló GA, Gutiérrez Rodríguez MP, Ortiz De Lejarazu Leonardo R, Orduña Domingo A, Bratos Pérez MÁ. New procedure for rapid identification of microorganisms causing urinary tract infection from urine samples by mass spectrometry (MALDI-TOF)[J]. Enfermedades Infecciosas y Microbiología Clínica, 2015, 33(2): 89-94. DOI:10.1016/j.eimc.2014.02.022 |
| [74] |
Su HB, Zhang Y, Peng HW. Progress in applications of biochip in food safety and detection[J]. Journal of Food Safety & Quality, 2018, 9(11): 2756-2761. (in Chinese) 苏焕斌, 张燕, 彭宏威. 生物芯片在食品安全检测中的应用研究进展[J]. 食品安全质量检测学报, 2018, 9(11): 2756-2761. DOI:10.3969/j.issn.2095-0381.2018.11.035 |
| [75] |
Kant K, Shahbazi MA, Dave VP, Ngo TA, Chidambara VA, Than LQ, Bang DD, Wolff A. Microfluidic devices for sample preparation and rapid detection of foodborne pathogens[J]. Biotechnology Advances, 2018, 36(4): 1003-1024. DOI:10.1016/j.biotechadv.2018.03.002 |
| [76] |
Suo B, He YP, Paoli G, Gehring A, Tu S, Shi XM. Development of an oligonucleotide-based microarray to detect multiple foodborne pathogens[J]. Molecular and Cellular Probes, 2010, 24(2): 77-86. DOI:10.1016/j.mcp.2009.10.005 |
| [77] |
Brennan D, Glynn B, Keegan G, McDonagh C, Barry T, Galvin P. Incorporating asymmetric PCR and microarray hybridization protocols onto an integrated microfluidic device, screening for the Escherichia coli ssrA gene[J]. Sensors and Actuators B: Chemical, 2018, 261: 325-334. DOI:10.1016/j.snb.2018.01.148 |
| [78] |
Huang AH, Qiu ZG, Jin M, Shen ZQ, Chen ZL, Wang XW, Li JW. High-throughput detection of food-borne pathogenic bacteria using oligonucleotide microarray with quantum dots as fluorescent labels[J]. International Journal of Food Microbiology, 2014, 185: 27-32. DOI:10.1016/j.ijfoodmicro.2014.05.012 |
| [79] |
Wang XQ, Ying SS, Wei XG, Yuan J. Development of a gold nanoparticle-based universal oligonucleotide microarray for multiplex and low-cost detection of foodborne pathogens[J]. International Journal of Food Microbiology, 2017, 253: 66-74. DOI:10.1016/j.ijfoodmicro.2017.05.005 |
| [80] |
Li YJ. Establishment and application of a visual DNA microarray for the detection of food-borne pathogens[J]. Analytical Sciences, 2016, 32(2): 215-218. DOI:10.2116/analsci.32.215 |
| [81] |
Zhang H, Zhang YL, Lin YK, Liang TW, Chen ZH, Li JF, Yue ZF, Lv J, Jiang Q, Yi CQ. Ultrasensitive detection and rapid identification of multiple foodborne pathogens with the naked eyes[J]. Biosensors and Bioelectronics, 2015, 71: 186-193. DOI:10.1016/j.bios.2015.04.034 |
| [82] |
Wang SL, Lv JZ, Huang LH, Ma SM, Huang XD, Liu HL, Zhao F, Hong XL. Research progress of liquid chip technology in the field of food safety[J]. Journal of Food Safety & Quality, 2016, 7(11): 4550-4556. (in Chinese) 王舒乐, 吕敬章, 黄李华, 马淑棉, 黄欣迪, 刘慧玲, 赵芳, 洪小柳. 液相芯片技术在食品安全领域的研究进展[J]. 食品安全质量检测学报, 2016, 7(11): 4550-4556. |
| [83] |
Dang YL, Zhou TY, Yao CY, Yu Y. Application progress on the high-flux and rapid detection technolgy of the suspension array for the food borne[J]. Food Research and Development, 2017, 38(8): 192-195. (in Chinese) 党亚丽, 周亭屹, 么春艳, 俞盈. 食源性致病菌液相芯片高通量快速检测技术的应用进展[J]. 食品研究与开发, 2017, 38(8): 192-195. DOI:10.3969/j.issn.1005-6521.2017.08.045 |
| [84] |
Lusk Pfefer TS, Timme R, Kase JA. Identification of Brucella genus and eight Brucella species by Luminex bead-based suspension array[J]. Food Microbiology, 2018, 70: 113-119. DOI:10.1016/j.fm.2017.09.015 |
| [85] |
Gand M, Mattheus W, Roosens NHC, Dierick K, Marchal K, De Keersmaecker SCJ, Bertrand S. A multiplex oligonucleotide ligation-PCR method for the genoserotyping of common Salmonella using a liquid bead suspension assay[J]. Food Microbiology, 2020, 87: 103394. DOI:10.1016/j.fm.2019.103394 |
| [86] |
Sun ZY, Peng Y, Zhang MC, Wang K, Bai JL, Li XL, Ning BA, Gao ZX. Simultaneous and highly sensitive detection of six different foodborne pathogens by high-throughput suspension array technology[J]. Food Control, 2014, 40: 300-309. DOI:10.1016/j.foodcont.2013.11.028 |
 2021, Vol. 48
2021, Vol. 48