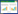扩展功能
文章信息
- 王粟萍, 陈学梅, 魏云林, 张琦, 季秀玲
- WANG Suping, CHEN Xuemei, WEI Yunlin, ZHANG Qi, JI Xiuling
- 纳帕海高原湿地噬藻体g20基因系统发育多样性分析
- Phylogenetic diversity of cyanophages g20 gene in Napahai plateau wetland in China
- 微生物学通报, 2021, 48(8): 2574-2582
- Microbiology China, 2021, 48(8): 2574-2582
- DOI: 10.13344/j.microbiol.china.201022
-
文章历史
- 收稿日期: 2020-10-24
- 接受日期: 2021-01-24
- 网络首发日期: 2021-03-15
病毒是世界上最丰富的“生命形式”[1-2],在生物地球化学循环、遗传多样性以及微生物群落调控等方面具有重要的生态功能。由于其高丰度和特殊的多样性,病毒成为地球上最大的基因库。噬藻体是感染原核藻类的病毒,介导水体中蓝藻种群的密度和多样性,并在微食物环中扮演重要角色[3]。目前鉴定的噬藻体大都属于双链DNA病毒,从形态上可分为3个家族:长尾病毒科(Siphoviridae)、肌尾病毒科(Myoviridae)和短尾病毒科(Podoviridae)[4],而且大部分都是肌尾病毒科。蓝藻具有固氮和光合作用,也是一种很重要的初级生产者。蓝藻分布在全球生态中,不论是在陆生系统还是水生系统中,几乎都能检测到蓝藻的存在[5]。近年来,随着病毒基因组学的不断发展,表明病毒亚群相对保守。衣壳蛋白g20基因已被广泛应用于环境评价、噬藻体遗传多样性和系统发育等方面的分子标记研究[6]。通过聚合酶链反应(polymerase chain reaction,PCR)从海洋[7]、淡水环境[8]、水稻水域[9-10]和水稻土壤[11]中扩增得到了各种各样的g20基因片段。相对而言,目前人们对自然湿地中噬藻体的遗传多样性研究较缺乏。
高原湿地是生物多样性较为丰富的地区,在环境保护中具有很高的价值,因此也被称为是“地球之肾”。纳帕海高原湿地位于青藏高原延伸的横断山脉,是金沙江流域高海拔、低纬度的季节性沼泽湿地[12-13]。纳帕海湿地不仅调节着河流水量和地表水流,防止土壤侵蚀,对长江下游的水位和水量均衡也有重要的影响,同时也因其天然牧场的重要地理地位和特殊的生物多样性,为当地居民的生活和生存带来了充足的自然资源,具有较高的社会价值。
因此,选择自然湿地——纳帕海高原湿地作为本次研究的对象,基于噬藻体外壳组装蛋白g20基因的多样性,通过PCR扩增测序,将其与其他环境进行比较分析,有助于评价高原湿地病毒群落,研究噬藻体与宿主的相互作用,以期为更加深入了解高原湿地以及维持其生态平衡奠定一定的理论基础。
1 材料与方法 1.1 主要试剂和仪器琼脂糖,康润生物科技有限公司;Taq DNA聚合酶,宝日医生物技术有限公司;氨苄青霉素,索莱宝科技有限公司;病毒基因组提取试剂盒,OMEGA公司。Mini柱,Omega公司;PCR仪,北京六一生物科技有限公司;紫外凝胶系统,GE Amersham公司;真空冷冻干燥机,兄弟仪器设备公司。
1.2 水样的采集与处理2013年12月从纳帕海高原湿地的雨季采用5点取样法收集不同样点的水样。水样采集后立即送回实验室,利用超/微滤实验装置(滤膜孔径0.22 μm)进行浓缩预处理,浓缩液分装后于-20 ℃保存备用。
1.3 噬藻体DNA提取取预处理过的水样20 mL加入到50 mL离心管中,在-80 ℃下冻存8 h,利用冻干机冻干成粉末,用病毒全基因组提取试剂盒提取水体样品中的全基因组DNA:加入250 μL Elution Buffer、10 μL OB Protease和250 mL Buffer BL,漩涡振荡15 s,将其置于65 ℃恒温水浴锅中10 min,加入260 μL无水乙醇,漩涡振荡20 s,取Mini柱放入2 mL收集管中,将上一步收集液全部吸入Mini柱中,8 000×g离心1 min,倒掉废液,将Mini柱放入新的收集管中,加入500 μL Buffer HB冲洗,8 000×g离心1 min,弃离心液,加入700 μL DNA Wash Buffer至Mini柱中,8 000×g离心1 min,15 000×g空柱离心2 min,将离心后的mini柱放入1.5 mL的离心管,加入75 μL的ddH2O洗柱,室温放置5 min后8 000×g离心1 min,所得的基因组保存于-20 ℃。
1.4 g20基因片段的PCR扩增利用PhiSiGns软件设计合成针对噬藻体g20基因的保守引物Cps1 (5′-GTAGWATTTTCTACAT TGAYGTTGG-3′)和Cps8 (5′-AAATAYTTNCCAA CAWATGGA-3′)进行PCR扩增[14]。50 μL的PCR反应体系:10×Taq Buffer 5 μL,0.002 5 mol/L dNTPs 4 μL,Taq DNA聚合酶0.4 μL,10 μmol/L引物Cps1-F、Cps8-R各0.8 μL,DNA模板5 ng。PCR反应条件:94 ℃ 5 min;94 ℃ 30 s,35.2 ℃ 30 s,72 ℃ 60 s,32个循环;72 ℃ 10 min。
1.5 PCR产物胶回收、TA克隆及测序PCR的扩增产物经琼脂糖凝胶电泳回收后,连接到pMD-18T载体上,转入Escherichia coli DH5α感受态细胞,随机挑选阳性克隆子,菌落PCR验证正确后进行测序。
1.6 g20基因翻译成氨基酸利用Editseq软件去除引物,并用Mothur V1.13软件筛选序列[15],将筛选得到的基因序列提交到NCBI数据库进行Blast比对,获得g20基因的GenBank登录号及同源性序列,通过Editseq软件将基因序列翻译成氨基酸序列。
1.7 序列分析系统发育分析:通过MEGA 6.06软件的邻接法构建来自4种不同环境、10个不同地区的689条序列(表 1)与采样得到的有效基因序列的整体系统发育树,同时单独构建纳帕海高原湿地的有效序列的系统发育树。
| 生境 Biological environment |
GenBank登录号 GenBank accession No. |
序列数 Sequences number |
文献 References |
| 马尾藻海 Sargasso Sea | AAK29755–29767 AAK31643–31775 |
13 133 |
[16] |
| 切萨皮克海湾 Chesapeake Bay | AAO13199–13213 | 15 | [17] |
| 望山海湾 Mount Hope Bay | AAP83536–83575 | 40 | [18] |
| 克兰芝水库 Kranji Reservoir | AGL61417–61501 | 85 | [19] |
| 布尔热湖 Lake Bourget | AAR10291–10337 | 47 | [20] |
| 安纳西湖和布尔热湖 Lake Annecy and Bourget | AGN88749–88858 | 110 | [21] |
| 日本稻田水 Japan Paddy water | BAG85049–85125 | 77 | [22] |
| 中国东北稻田水 NE China Paddy water | AGT78138–78191 | 54 | [23] |
| 日本稻田土壤 Paddy soil of Japan | BAJ07464–07533 | 70 | [24] |
| 伊利湖 Lake Erie | DQ318388–318432 | 45 | [25] |
Unifrac分析:选取上述已报道的9个不同区域的54条氨基酸参考序列,每个区域均选取6条序列,利用Unifrac在线程序对纳帕海湿地序列进行主坐标分析(Principal Coordinates Analysis,PCoA)[26]。
2 结果与分析 2.1 g20基因PCR扩增及测序以纳帕海高原湿地g20基因的简并引物对其进行PCR扩增,在最适退火温度35.2 ℃下,得到大小约600 bp的目的片段。通过测序共得到47条纳帕海高原湿地水体(Napahai,NPH)样品g20基因的核苷酸序列,经过序列拼接、去除两端引物和重复序列后翻译成氨基酸序列,获得26条不同的序列。
2.2 Unifrac分析通过Unifrac在线程序,将26条g20有效氨基酸序列与淡水湖泊、海洋、稻田水和土壤进行PCoA分析(图 1)。从图 1中可以看出,纳帕海高原湿地水体样品g20序列分布在第二、三象限,而且它们的位置相距对较近;两簇水稻水和三簇水稻土位置相距较近,因此,在相似的环境中,噬藻体群落组成彼此相似[27]。虽然纳帕海高原湿地的噬藻体g20基因与中国东北和日本的稻田较为接近,但噬藻体群落仍存在差异。结果表明,来自海洋的5个簇位置相距较近,与其他生境相隔都较远,这与东北稻田水中T4型噬菌体的g20序列的结果相一致。为进一步对纳帕海高原湿地噬藻体基因的遗传多样性进行研究分析,选取4种不同环境、10个不同地区的600多条氨基酸序列与本研究获取的26条序列进行系统发育树分析。
|
| 图 1 纳帕海高原湿地g20基因序列与其他生境序列UniFrac分析 Figure 1 UniFrac analysis of g20 gene sequence and other biological environment sequences in Napahai plateau wetland |
|
|
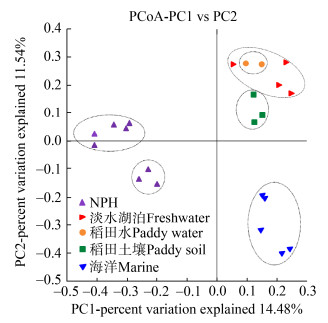
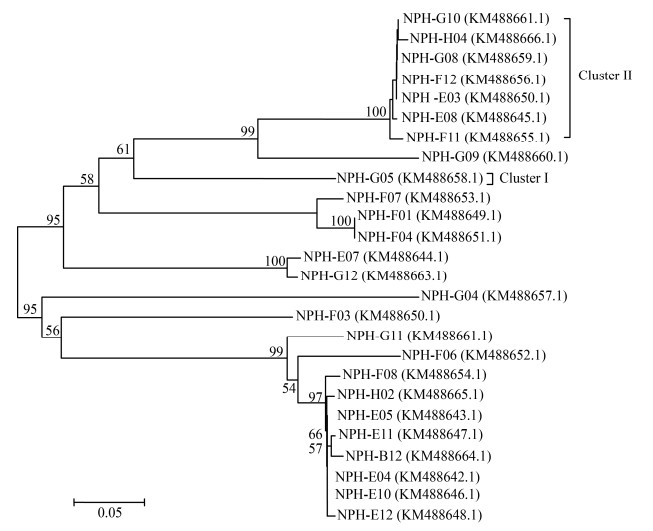
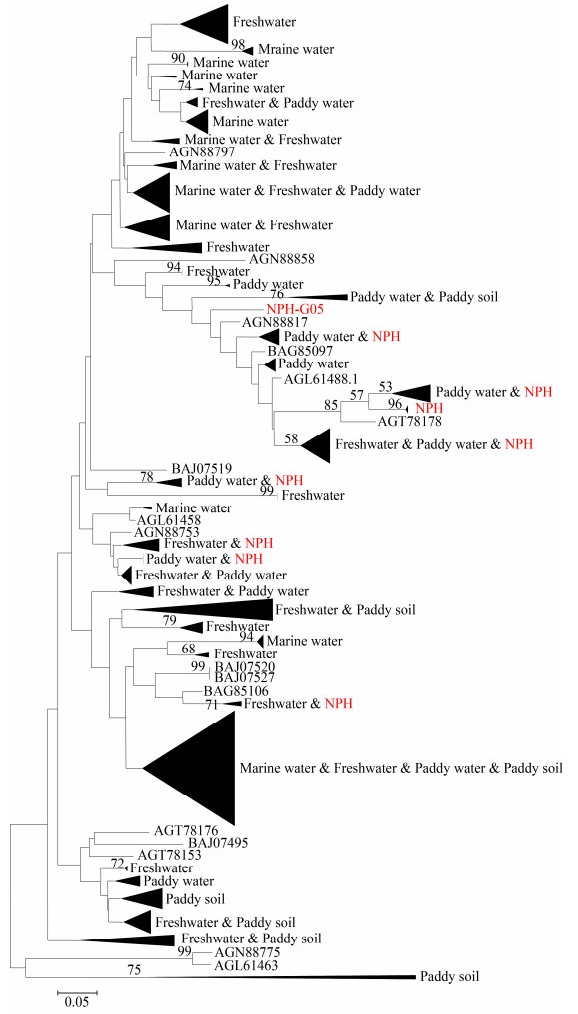
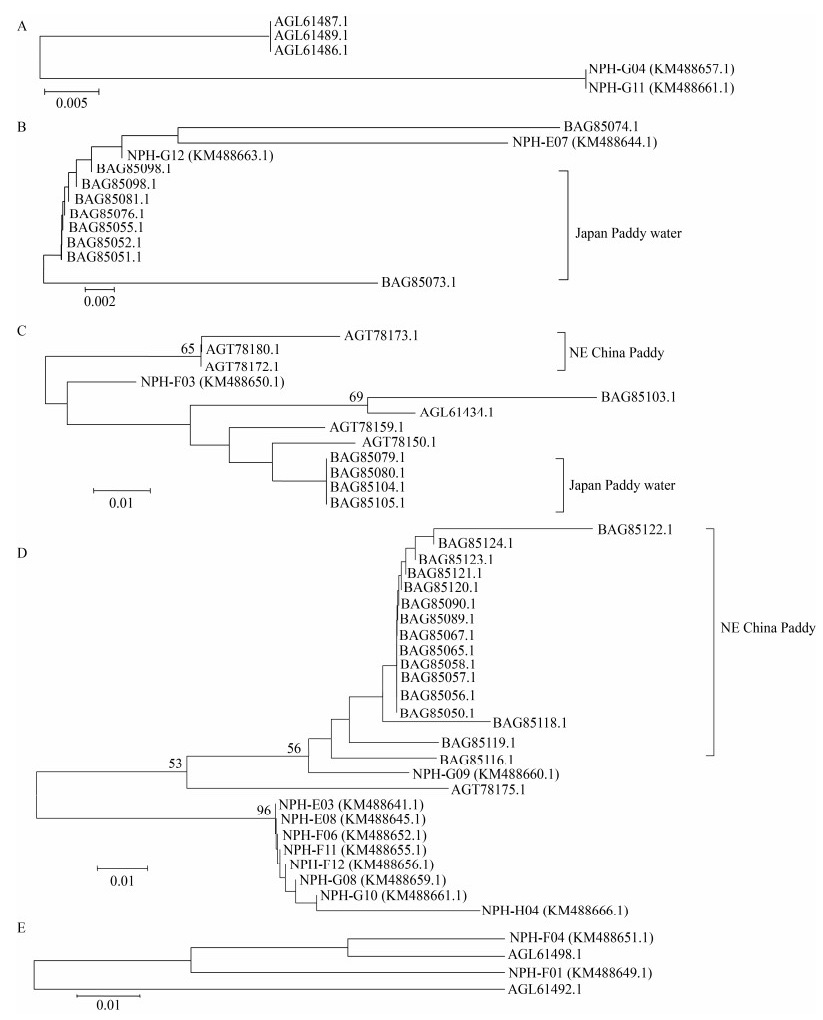
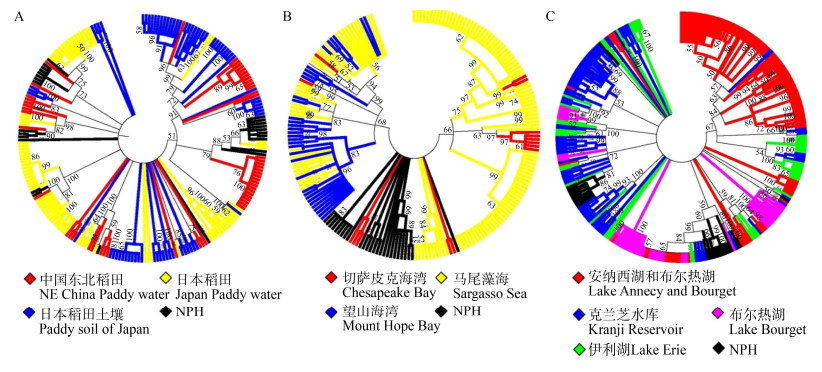
将26条不同的氨基酸序列经NCBI数据库Blast比对后,运用MEGA 6.0软件中的邻接(Neighbor-Joining,NJ)法——单独构建系统发育树(图 2),从不同生境中选取g20基因序列,将得到的26条序列与选取的稻田、淡水湖泊、海洋序列构建系统发育树总图(图 3)、局部放大图(图 4)和环形系统发育树(图 5)。
|
| 图 2 纳帕海高原湿地g20基因氨基酸序列系统发育树 Figure 2 Neighbor-joining phylogenetic tree of g20 gene sequences obtained in Napahai plateau wetland 注:邻接法构建系统发育树,节点上数字表示Bootstrap值,括号内表示g20基因的GenBank登录号,标尺代表进化距离。下同 Note: The phylogenetic tree was constructed by neighbor-joining model. The number on the node indicating the bootstrap value, the GenBank entry number of the g20 gene in parentheses, and the ruler represents the evolutionary distance. Same as below |
|
|
|
| 图 3 纳帕海湿地g20基因序列与其他生境序列整体系统发育树 Figure 3 Neighbor-joining whole phylogenetic tree of g20 gene sequences obtained in Napahai wetland and other biological environment |
|
|
|
| 图 4 纳帕海湿地g20基因氨基酸序列与其他生境序列局部系统发育树 Figure 4 Neighbor-joining local phylogenetic tree of g20 gene sequences obtained in Napahai wetland and other biological environment |
|
|
|
| 图 5 纳帕海湿地g20基因氨基酸序列与其他生境序列的环形系统发育分析 Figure 5 Circular phylogentic analysis of g20 amino acids sequences obtained in the Napahai wetland and other biological environment sequences |
|
|
从系统发育树可看出,纳帕海高原湿地得到的26条氨基酸序列分别属于8个簇(图 2),其中NPH-F01、NPH-F04、NPH-F07、NPH-G04、NPH-G11与克兰芝水库序列聚为一簇(图 4A、图 4E);NPH-G09、NPH-G12、NPH-E (04、05、07、10、11、12)、NPH-F08、NPH-H02、NPH-B12与日本稻田g20序列聚为一簇(图 4B);NPH-F03与日本稻田、克兰芝水库和中国东北稻田序列聚为一簇(图 4C)。另外有2个簇为纳帕海高原湿地所独有,分别为ClusterⅡ (NPH-E03、NPH-E08、NPH-F06、NPH-F11、NPH-F12、NPH-G08、NPH-G10、NPH-H04),ClusterⅠ (NPH-G05) (图 2),推测这2簇为纳帕海高原湿地的独有序列。
从环形系统发育树中可看出(图 5),8个主要类群的噬藻体序列与中国东北稻田和日本稻田序列在遗传上的亲缘关系较近,4个主要类群的噬藻体序列与克兰芝水库的淡水序列相似,1个主要类群的噬藻体序列与伊利湖的淡水序列相似,本研究获得的几乎所有序列都与海洋序列相距较远。这表明陆生水环境中的g20基因与海洋环境中的不同,噬藻体在湿地系统中的重要性可能与在海洋系统中的重要性相同。总体而言,本研究中得到的噬藻体序列与稻田序列的亲缘关系相比,与其他海洋和淡水噬藻体序列的亲缘关系更加接近。由于纳帕海高原湿地与中国东北和日本平原湿地的纬度、海拔、温度、金属元素和铁等物理、化学和生物特征不同,纳帕海高原湿地也具有独特的g20序列。
3 讨论与结论蓝藻是稻田、土壤、海洋、淡水湖泊等生态系统的重要成员,所以对噬藻体的研究一直是热点课题。1998年,Fuller等[28]首次设计引物Cps1/Cps2对海洋噬藻体g20基因进行了扩增,并对水体环境中的蓝藻病毒丰度进行了定量监测。
2002年,Zhong等[29]通过设计引物Cps1/Cps8对海洋中分离的噬藻体及自然环境中噬藻体集合体的g20基因进行系统发育分析,在得到的9个簇中,仅有3个簇包括已分离获得的噬藻体(ClusterⅠ、Ⅱ和Ⅲ),其余6个簇(ClusterA、B、C、D、E和F)均来自环境样品,显示出海洋噬藻体基因具有丰富的多样性。2008年,Sullivan等[30]设计引物对Cps1.1/Cps8.1,对分离的38株海洋噬藻体的g20基因研究发现这些菌株均属于CSP类群。Yan等[6]以Cps1/Cps4为引物,采用PCR-RFLP法对青岛海域噬藻体g20基因进行分析发现,获得的序列可以分为2个部分:一部分属于Cluster Ⅲ,另外一部分是首次在青岛海域发现的噬藻体簇ClusterW。Li等[31]用2对引物Cps1/Cps4和Cps1/Cps8从噬藻体中扩增出g20基因,通过RFLP分析和测序鉴定得到61条独特的序列,经过系统发育分析表明该地区样品中至少含有8个不同的簇,说明汕头近岸海域病毒群落的多样性比较丰富。通过以上研究可以表明,在不同的海域中都有独特的噬藻体类群存在。
Wang等[9]设计了引物Cps1/Cps8从日本稻田水样中扩增出g20基因序列,通过系统发育树分析发现这些基因序列与来自湖泊和海洋水体中g20基因聚类为5个主要的进化簇,其中9个克隆在Cluster δ中形成2个独立的Subcluster;2011年,Wang等[11]进一步通过g20基因的多样性研究表明:噬藻体的类群特征在稻田水体和其土壤之间均有着较为显著的差异。Li等[32]利用PCR技术对g20基因序列进行了扩增,系统发育树分析表明该g20基因序列是可培养噬藻体类群(Cluster δ)。Jing等[33]为阐明中国东北稻田水域噬菌体的遗传多样性,使用引物Cps1/Cps8扩增了5个洪水样品的g20基因,研究显示g20的分布与日本稻田中的序列有所不同,而且确定了3个新的小型群集(类似于Ⅸ的PFW-Ⅶ)。Yeo等[34]收集了新加坡克兰芝水库3个站点的样本,采用Cps1/Cps8引物对进行扩增,系统发育研究表明克兰芝水库的噬藻体群落表现出丰富的多样性,并发现其与海洋环境的序列关系更密切。总之,稻田中大多数g20基因形成几个独特的分支,而且与淡水环境亲缘关系较接近,与海洋环境序列亲缘关系较远。
纳帕海高原湿地地处3 200 m以上的海拔,地理位置独特,是我国独有的低纬度、高海拔、季节性高原沼泽湿地,常年受到西南季风气候影响,干湿季分明。本研究以纳帕海高原湿地的g20基因为探针,通过构建系统发育树和Unifrac在线软件综合分析显示:大部分基因序列与稻田和淡水湖泊序列较为相似,可能纳帕海高原湿地也是淡水地域,而且近年来该地区多受到人类生活、生产等活动干扰,从而影响了该地区的生物群落结构,进而使噬藻体类群及其多样性也发生了一定的变化;小部分序列是纳帕海高原湿地特有的噬藻体类群序列,原因可能是纳帕海高原湿地拥有独特的气候条件和地理位置[35]。总体而言,纳帕海高原湿地的g20序列与其他生境产生了较大差异,而且该地区拥有部分独有的序列,因此说明该地区拥有丰富的噬藻体。
| [1] |
Suttle CA. Viruses in the sea[J]. Nature, 2005, 437(7057): 356-361. DOI:10.1038/nature04160 |
| [2] |
Pujato SA, Quiberoni A, Mercanti DJ. Bacteriophages on dairy foods[J]. Journal of Applied Microbiology, 2019, 126(1): 14-30. DOI:10.1111/jam.14062 |
| [3] |
Zhang QY, Gui JF. Diversity, evolutionary contribution and ecological roles of aquatic viruses[J]. Science China Life Sciences, 2018, 61(12): 1486-1502. DOI:10.1007/s11427-018-9414-7 |
| [4] |
Suttle CA. Cyanophages and their role in the ecology of cyanobacteria[J]. The Ecology of Cyanobacteria, 2002. DOI:10.1007/0-306-46855-7_20 |
| [5] |
Rasool A, Ali S, Ali W, Eldesoky GE, Shafeeque M, Ahmad I, Akram M, Tahir M, Muhammad S, Nasim W. Microbial diversity and community structure in alpine stream soil[J]. Geomicrobiology Journal, 2021, 38(3): 210-219. DOI:10.1080/01490451.2020.1824039 |
| [6] |
Yan Q, Wang M, Bai X, Sun J, Liang Y, Wang F, Yang L, Liu G, Lu L. New phylogenetically distinct cyanophages found in the coastal Yellow Sea by Qingdao[J]. Acta Virologica, 2010, 54(4): 255-260. |
| [7] |
Sieradzki ET, Cesar Ignacio-Espinoza J, Needham DM, Fichot EB, Fuhrman JA. Dynamic marine viral infections and major contribution to photosynthetic processes shown by spatiotemporal picoplankton metatranscriptomes[J]. Nature Communications, 2019, 10: 1169. DOI:10.1038/s41467-019-09106-z |
| [8] |
Zhang D, You F, He YL, Te SH, Gin KYH. Isolation and characterization of the first freshwater cyanophage infecting Pseudanabaena[J]. Journal of Virology, 2020. DOI:10.1128/jvi.00682-20 |
| [9] |
Wang GH, Murase J, Asakawa S, Kimura M. Unique viral capsid assembly protein gene (g20) of cyanophages in the floodwater of a Japanese paddy field[J]. Biology and Fertility of Soils, 2010, 46(2): 93-102. DOI:10.1007/s00374-009-0410-y |
| [10] |
Wang XZ, Liu JJ, Yu ZH, Jin J, Liu XB, Wang GH. Novel groups and unique distribution of phage phoH genes in paddy waters in northeast China[J]. Scientific Reports, 2016, 6: 38428. DOI:10.1038/srep38428 |
| [11] |
Wang GH, Asakawa S, Kimura M. Spatial and temporal changes of cyanophage communities in paddy field soils as revealed by the capsid assembly protein gene g20[J]. FEMS Microbiology Ecology, 2011, 76(2): 352-359. DOI:10.1111/j.1574-6941.2011.01052.x |
| [12] |
J L, M HJ, C L. Boundary data of napahai wetland[J]. Journal of Global Change Data & Discovery, 2017, 1(3): 369. |
| [13] |
Feng CH, He ZP, Tian K, Guan DX, Liu ZY, Sun M. Comparative study of functional adaptability of Scirpus validus under different elevations[J]. Journal of Southwest Forestry University: Natural Sciences, 2019, 39(1): 166-171. (in Chinese) 冯春慧, 何照棚, 田昆, 管东旭, 刘振亚, 孙梅. 不同海拔生长的水葱功能适应性对比研究[J]. 西南林业大学学报(自然科学), 2019, 39(1): 166-171. |
| [14] |
Sun C. Spatial and temporal distribution of virioplankton and genetic diversity of g20 gene in napahai wetland[D]. Kunming: Master's Thesis of Kunming University of Science and Technology, 2015(in Chinese) 孙策. 纳帕海浮游病毒时空分布及g20基因多样性研究[D]. 昆明: 昆明理工大学硕士学位论文, 2015 |
| [15] |
Cui ZH, Li S, Qin KH, Zhang Q, Wei YL, Tang B, Ji XL. Preliminary study on genetic diversity of mazG in the Napahai plateau wetland[J]. Genomics and Applied Biology, 2020, 39(5): 2148-2153. (in Chinese) 崔自红, 李珊, 秦堃豪, 张琦, 魏云林, 唐兵, 季秀玲. 纳帕海高原湿地mazG遗传多样性初步研究[J]. 基因组学与应用生物学, 2020, 39(5): 2148-2153. |
| [16] |
Chen YY, Lu XG. The wetland function and research tendency of wetland science[J]. Wetland Science, 2003, 1(1): 7-11. (in Chinese) 陈宜瑜, 吕宪国. 湿地功能与湿地科学的研究方向[J]. 湿地科学, 2003, 1(1): 7-11. DOI:10.3969/j.issn.1672-5948.2003.01.003 |
| [17] |
Mitsch WJ, Mitsch RH, Turner RE. Wetlands of the old and new worlds: ecology and management[A]//Mitsch WJ. Global Wetlands[M]. Amsterdam: Elsevier, 1994
|
| [18] |
Wilen BO, Tiner RW. Wetlands of the United States[A]//Whigham DF, Dykyjová D, Hejný S. Wetlands of the World: Inventory, Ecology and Management Volume I[M]. Dordrecht: Springer, 1993: 515-636
|
| [19] |
Cowardin LM, Carter V, Golet FC, LaRoe ET. Classification of wetlands and deepwater habitats of the United States[R]. Washington: US Department of the Interior, Fish and Wildlife Service, 1979
|
| [20] |
Brinson MM. A hydrogeomorphic classification for wetlands[R]. Vicksburg, MS: U.S. Army Corps of Engineers, 1993
|
| [21] |
Mitsch WJ, Gosselink JG. Wetlands[M]. New York: Van Nostrand Reinhold, 1986.
|
| [22] |
Mitsch WJ, Gosselink JG. Wetlands[M]. New York: Van Nostrand Reinhold, 2000.
|
| [23] |
Thormann MN, Bayley SE. Aboveground plant production and nutrient content of the vegetation in six peatlands in Alberta, Canada[J]. Plant Ecology, 1997, 131(1): 1-16. DOI:10.1023/A:1009736005824 |
| [24] |
Sundareshwar PV, Morris JT. Phosphorus sorption characteristics of intertidal marsh sediments along an estuarine salinity gradient[J]. Limnology and Oceanography, 1999, 44(7): 1693-1701. DOI:10.4319/lo.1999.44.7.1693 |
| [25] |
Wilhelm SW, Carberry MJ, Eldridge ML, Poorvin L, Saxton MA, Doblin MA. Marine and freshwater cyanophages in a Laurentian Great Lake: evidence from infectivity assays and molecular analyses of g20 genes[J]. Applied and Environental Microbiology, 2006, 72(7): 4957-4963. DOI:10.1128/AEM.00349-06 |
| [26] |
Lozupone C, Knight R. UniFrac: a new phylogenetic method for comparing microbial communities[J]. Applied and Environmental Microbiology, 2005, 71(12): 8228-8235. DOI:10.1128/AEM.71.12.8228-8235.2005 |
| [27] |
Wang XZ, Liu JJ, Yu ZH, Jin J, Liu XB, Wang GH. Novel groups of cyanobacterial podovirus DNA polymerase (pol) genes exist in paddy waters in northeast China[J]. FEMS Microbiology Ecology, 2016, 92(12): fiw192. DOI:10.1093/femsec/fiw192 |
| [28] |
Fuller NJ, Wilson WH, Joint IR, Mann NH. Occurrence of a sequence in marine cyanophages similar to that of T4g20 and its application to PCR-based detection and quantification techniques[J]. Applied and Environmental Microbiology, 1998, 64(6): 2051-2060. DOI:10.1128/AEM.64.6.2051-2060.1998 |
| [29] |
Zhong Y, Chen F, Wilhelm SW, Poorvin L, Hodson RE. Phylogenetic diversity of marine cyanophage isolates and natural virus communities as revealed by sequences of viral capsid assembly protein gene g20[J]. Applied and Environmental Microbiology, 2002, 68(4): 1576-1584. DOI:10.1128/AEM.68.4.1576-1584.2002 |
| [30] |
Sullivan MB, Coleman ML, Quinlivan V, Rosenkrantz JE, DeFrancesco AS, Tan G, Fu R, Lee JA, Waterbury JB, Bielawski JP, et al. Portal protein diversity and phage ecology[J]. Environmental Microbiology, 2008, 10(10): 2810-2823. DOI:10.1111/j.1462-2920.2008.01702.x |
| [31] |
Li CB, Ding J, Zhou LZ, Zhang Z, Li SK, Liu WH, Wen XB. Survey of cyanomyovirus abundance in Shantou coastal waters by g20[J]. Chinese Journal of Oceanology and Limnology, 2015, 33(3): 604-615. DOI:10.1007/s00343-015-4168-9 |
| [32] |
Li W, Li RR, Jing RY, Zhang HJ, Hu PF, Wang LY. Isolation and phylogenetic analysis of related genes of cultivable cyanophages in Daqing Wetland[J]. Acta Microbiologica Sinica, 2016, 56(5): 824-834. (in Chinese) 李伟, 李睿瑞, 荆瑞勇, 张海军, 胡鹏飞, 王丽艳. 大庆湿地可培养噬藻体的分离及其相关基因的系统进化分析[J]. 微生物学报, 2016, 56(5): 824-834. |
| [33] |
Jing RY, Liu JJ, Yu ZH, Liu XB, Wang GH. Phylogenetic distribution of the capsid assembly protein gene (g20) of cyanophages in paddy floodwaters in Northeast China[J]. PLoS One, 2014, 9(2): e88634. DOI:10.1371/journal.pone.0088634 |
| [34] |
Yeo BH, Gin KYH. Population dynamics of cyanomyovirus in a tropical eutrophic reservoir[J]. Microbes and Environments, 2015, 30(1): 12-20. DOI:10.1264/jsme2.ME14039 |
| [35] |
Dong Y, Tian K, Guo XH, Xiao DR, Guo XL. Impacts of regional climate change on chlorophyll fluorescence characteristics of plateau wetland plants in Napahai, Yunnan China[J]. Ecology and Environmental Sciences, 2013, 22(4): 588-594. (in Chinese) 董瑜, 田昆, 郭绪虎, 肖德荣, 郭雪莲. 不同区域气候条件影响下的纳帕海湿地植物叶绿素荧光特性[J]. 生态环境学报, 2013, 22(4): 588-594. DOI:10.3969/j.issn.1674-5906.2013.04.007 |
 2021, Vol. 48
2021, Vol. 48