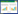扩展功能
文章信息
- 陈小蓉, 潘海学, 唐功利
- CHEN Xiaorong, PAN Haixue, TANG Gongli
- 天然产物产生菌自抗性中DNA损伤修复的研究进展
- Research progress on DNA damage repair in self-resistance of natural product producing bacteria
- 微生物学通报, 2021, 48(7): 2454-2466
- Microbiology China, 2021, 48(7): 2454-2466
- DOI: 10.13344/j.microbiol.china.210108
-
文章历史
- 收稿日期: 2021-01-27
- 接受日期: 2021-04-04
- 网络首发日期: 2021-04-25
2. 中国科学院上海有机化学研究所 生命有机化学国家重点实验室 上海 200032
2. State Key Laboratory of Bio-Organic and Natural Products Chemistry, Shanghai Institute of Organic Chemistry, Chinese Academy of Sciences, Shanghai 200032, China
天然产物因其结构多样性以及广谱的抑菌活性,自发现以来一直是抗生素类药物开发的重要源泉,广泛应用于人类医学和农业生产等方面[1-2]。据统计,在过去的短短100多年里,抗生素在现代医学上的应用使人类平均寿命延长了23年[3]。天然产物结构复杂、种类繁多,根据作用靶标的不同大致可以分为抑制细胞壁合成类、抑制蛋白合成类、抑制RNA和DNA合成类等。其中,作用于DNA类的天然产物由于能够中断DNA复制、转录或引起DNA骨架链的断裂导致细胞死亡,快速繁殖的肿瘤细胞对于它们尤为敏感[4-5]。因此,除了治疗感染性疾病,这类天然产物及其衍生物的使用也促进了癌症治疗的进一步发展。例如Mitomycin C已作为临床化疗药物治疗非小细胞肺癌、前列腺癌等多种癌症,Bleomycin也被用于治疗淋巴瘤等[6-7]。然而,随着抗生素的广泛使用,随之而产生的抗生素耐药性问题已成为人类未来几十年所需面临的最大健康威胁之一。研究表明,如果不采取行动,到2050年,每年将有1 000万人死于耐药细菌的感染[8]。因此,抗生素抗性机制的解析不仅可以揭示抗生素抗性的起源,为临床上抗生素耐药性问题的解决提供新的线索,同时,还能利用同源抗性基因挖掘新型天然产物、鉴定生物合成基因簇、预测相关天然产物的生物活性[9-10]。
临床上使用的抗生素大多是由微生物产生,这类化合物在产生菌中往往作为防御武器抵御其他微生物群落的入侵。然而,作用于其他微生物的同时,抗生素在合成过程中也对产生菌本身具有细胞毒性。因此,自抗性是抗生素合成的先决条件,而且抗性基因通常与其生物合成基因簇成簇或串联排列[11]。根据功能的不同,抗性机制大致可以分为药物隔离(Sequestration)、外排(Efflux)、修饰(Modification)、靶标保护(Target Protection)以及损伤修复(Damage Repair)[12-14]。抗生素产生菌通常采用其中的一种或多种抗性机制实现自我保护。随着生物技术的发展,越来越多的抗性蛋白逐渐被发现。其中最让人惊奇的是多种DNA损伤修复类蛋白也参与到抗生素的自抗性中[15-17]。因此,本文着重介绍了DNA损伤抗生素(Genotoxic Antibiotics)产生菌中DNA损伤修复机制的研究进展,并对目前所存在的问题及DNA损伤修复类抗性因子的应用进行简要讨论。
1 DNA损伤抗生素简介DNA损伤抗生素(Genotoxic Antibiotics),顾名思义,泛指能够与基因组DNA相互作用、引起DNA损伤的一类化合物。这类化合物具有强烈的细胞毒性,能够中断DNA的复制、转录,诱导DNA链的断裂,最终导致细胞死亡,表现出显著的抑菌和抗癌活性[18-19]。因此,这类化合物是抗肿瘤药物的重要来源[20-21]。按照与DNA作用模式的不同,DNA损伤类抗生素可分为与DNA共价结合和非共价结合2类化合物。
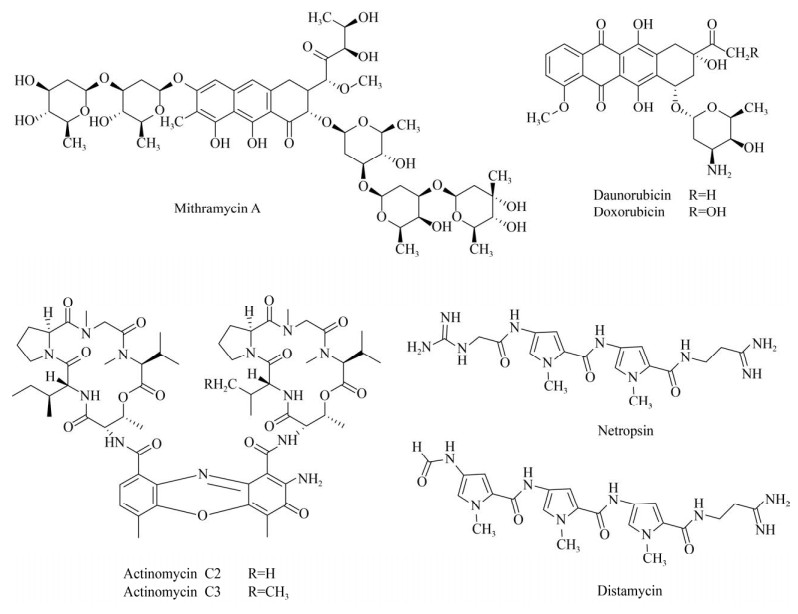
1.1 DNA非共价结合类非共价结合是指药物分子与DNA通过静电、沟槽结合(DNA大沟或小沟区)或嵌插结合的方式相互作用[22]。大多数DNA结合药物都是通过与DNA形成非共价键而发挥作用(图 1)。以Netropsin为例,该化合物最初分离于链霉菌Streptomyces netropsis,能够与DNA结合,具有强烈的细胞毒性;其作用机制是通过药物小分子上的胍基基团中亚胺基NH与DNA骨架链上腺嘌呤碱基中N-3上的氮和胸腺嘧啶碱基C-2的羰基氧形成氢键,结合至DNA小沟中富含AT区域,从而阻止DNA的复制和转录,最终导致细胞死亡,表现出抑菌和抗肿瘤活性[23-25]。蒽环类(Anthracylines)和放线菌素类(Actinomycins)抗生素则是通过嵌插的方式与DNA结合。例如放线菌素中的苯氧嗪环基团以嵌插的方式插入到富含GC的DNA链中,五肽内酯环则与DNA小沟中碱基形成氢键,最终阻止DNA的转录,中断RNA的合成[26]。该类化合物抗瘤谱广,而且对于不同生长周期的肿瘤细胞均有效,因此作为临床药物广泛应用于治疗包括急性白血病、淋巴瘤、乳腺癌在内的多种恶性肿瘤[27-28]。
|
| 图 1 部分与DNA非共价结合的天然产物 Figure 1 Several natural products non-covalently bound with DNA |
|
|
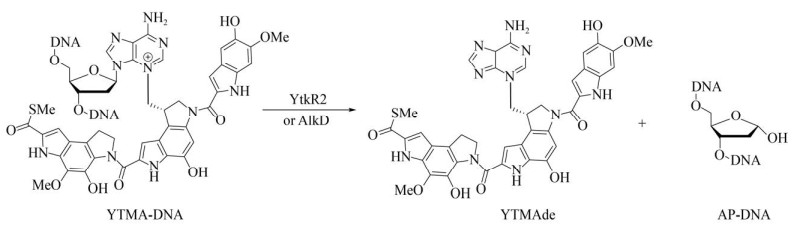
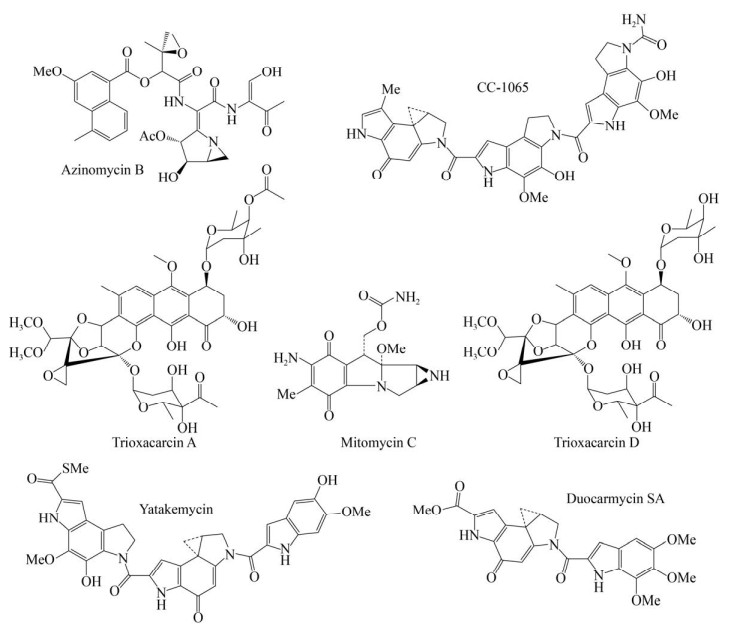
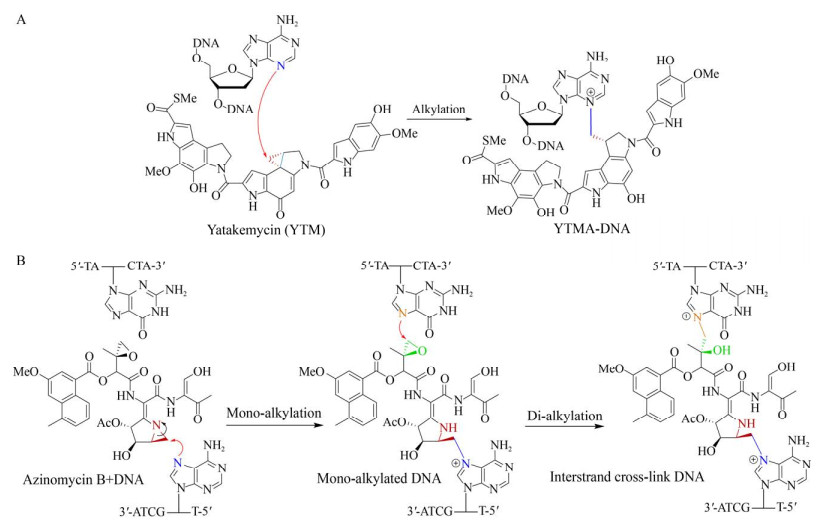
相比于非共价结合,抗生素与DNA发生共价结合则是通过药物分子对DNA进行烷基化、链间交联或链内交联的方式来完成。这类化合物包括芳香聚酮类的三欣卡辛家族(Trioxacarcins)、含有苯醌基团的丝裂霉素类(Mitomycins)、含有萘甲酸基团的阿嗪霉素类(Azinomycins)、苯并双吡咯家族的CC-1065、多卡霉素(Duocarmycin)和Yatakemycin等(图 2)。以Yatakemycin为例,该化合物由2个吡咯吲哚环和1个吲哚基团通过酰胺键连接而成,能够选择性地识别DNA链上的AT序列,腺嘌呤碱基N-3上的氮亲核进攻Yatakemycin分子上的环丙基,与其发生烷基化反应,从而阻断DNA的复制与转录,表现出显著的抗真菌和抗肿瘤活性(对细胞株L1210的半数抑制浓度IC50为3 pmol) (图 3A)[29]。然而阿嗪霉素类和丝裂霉素类抗生素则是通过与DNA形成交联结构发挥抗菌和抗癌活性。以阿嗪霉素B (Azinomycin B)为代表,该化合物能够特异性地识别双链DNA中大沟内嘌呤碱基,随后DNA序列5′-d(GNPy)-3′中嘌呤碱基的N7位亲核攻击药物分子氮杂双环上的C10位,形成单烷基化产物,随后药物分子上的环氧基团与互补链的5′端嘌呤碱基之间发生二次烷基化,最终形成链间交联(图 3B)[30-32]。
|
| 图 2 部分与DNA共价结合的天然产物 Figure 2 Several natural products covalently bound with DNA |
|
|
|
| 图 3 抗生素与DNA共价结合的作用模式[15, 17] Figure 3 The action mode of covalent binding of antibiotics to DNA[15, 17] 注:A:Yatakemycin与DNA共价结合形成烷基化产物的作用模式;B:Azinomycin B与DNA共价结合形成烷基化产物的作用模式 Note: A: The mode of the alkylation products formed by the covalent binding of yatakemycin with DNA; B: The mode of the alkylation products formed by the covalent binding of azinomycin B with DNA |
|
|
维持基因组的稳定性和遗传信息的完整性对于遗传密码的准确传输至关重要。然而,在DNA复制过程中不断受到来自于细胞内或环境中DNA损伤试剂的威胁,其通过氧化、烷基化、脱氨或水解产生多种损伤,包括单链或双链断裂、链间或链内交联、碱基脱离或碱基修饰[33-34]。如果这些DNA损伤没有得到正确修复,将会使得包括复制、转录和细胞周期调节在内的重要细胞过程受到损害,导致突变、染色体重排和基因组不稳定,诱导可遗传性疾病甚至导致死亡,威胁生物体的生存[34-36]。因此,细胞必须及时修复这些损伤。为了应对DNA损伤所造成的潜在危害,生物体进化出一系列完整的修复途径对DNA损伤进行监测和修复。总而言之,最常见的DNA损伤修复机制可以概况为5类,包括碱基切除修复(Base Excision Repair,BER)、核苷酸切除修复(Nucleotide Excision Repair,NER)、错配修复(Mismatch Repair,MMR)、同源重组(Homologous Recombination,HR)和非同源末端连接(Nonhomologous End Joining,NHEJ)[37-39]。细胞在对DNA损伤进行修复时通常根据损伤类型、细胞周期和生物物种选择其中的一种或多种修复途径完成损伤修复。然而在DNA损伤类抗生素产生菌中,目前发现仅有碱基切除修复和类核苷酸切除修复途径参与到了抗生素产生菌的自抗性保护系统,修复药物对靶标DNA产生的损伤。
2.1 碱基切除修复碱基切除修复是所有生物体中通用的一类DNA损伤修复途径,常用于修复DNA中包括烷基化、脱氨和氧化在内的多种DNA损伤[40-43]。该修复途径由多种酶组成,包括DNA糖基化酶(DNA Glycosylase)、APE1 (AP Endonuclease 1)、DNA聚合酶、DNA连接酶和一系列辅助蛋白[40]。碱基切除修复主要通过4步完成:(1) DNA糖基化酶催化异常碱基的切除,形成无碱基位点(AP);(2) 核酸内切酶APE1识别该位点并水解DNA骨架中的磷酸二酯键产生缺口;(3) 在DNA聚合酶的作用下,5′端的脱氧核糖磷酸基团(5′-Deoxyribose Phosphate,5′-dRP)被移除,并在3′端插入未修饰的碱基;(4) 在连接酶等酶的作用下恢复至原始DNA序列(图 4)[44-45]。
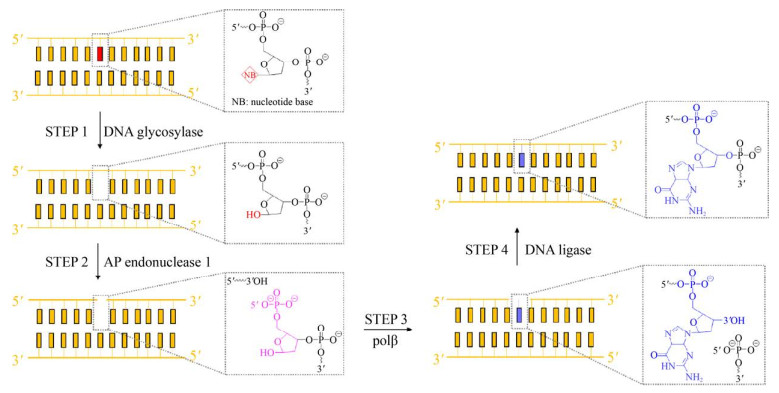
DNA糖基化酶(DNA Glycosylase)是BER途径中的关键蛋白,负责识别和切除损伤碱基,是启动BER途径的第一步[46-47]。在对损伤DNA进行切除时,这些酶通常采用碱基翻转(Base-Flipping)机制将被修饰碱基翻转至DNA双链外侧,随后翻转出的修饰碱基与位于糖基化酶表面的空腔结合,而碱基翻转形成的空隙由蛋白质上一个或多个氨基酸残基填充,从而稳定DNA构型;碱基结合空腔负责根据异常核苷酸的形状和所形成的互补电荷识别损伤,同时也负责为N-糖苷键的水解提供支架,这也就导致修饰碱基的大小受到严格限制[48-50]。鉴于与核苷酸结合的空腔通常较小,无法容纳超过1个位点修饰的碱基,因此研究者们普遍认为DNA糖基化酶不能修复大型加合物或链间交联在内的其他类型的损伤。然而,近年来研究发现,在烷基化抗生素Yatakemycin和Azinomycin B生物合成基因簇中编码的DNA糖基化酶YtkR2和AlkZ能够识别并修复Yatakemycin和Azinomycin B烷基化引起的DNA损伤[15-16, 51]。这也就说明除小型加合物外,糖基化酶还具有切除大型DNA加合物的功能。
2.1.1 BER赋予宿主对Yatakemycin抗性Yatakemycin属于苯并双吡咯家族,能够特异性地结合至DNA上富含AT区域的小沟中,与腺嘌呤碱基上的N3位发生烷基化反应[52]。此外,除了烷基化亚基外,药物分子上两侧的亚基均能与DNA通过C-H/Π相互作用形成非共价结合,从而导致DNA复制和转录过程中的解链严重受阻,同时也使得这些损伤难以通过NER途径修复[53]。研究者们在对Yatakemycin产生菌自抗性机制进行研究时,发现其生物合成基因簇中的ytkR2所编码的蛋白YtkR2属于DNA糖基化酶家族,与来自Bacillus cereus的DNA糖基化酶AlkD具有较高相似性(48%)。YtkR2的缺失将导致Yatakemycin产量下降40%,而将其异源表达后可赋予异源宿主Escherichia coli对Yatakemycin和甲基甲磺酸盐(Methyl Methanesulphonate,MMS)的抗性[15]。这也就说明YtkR2作为抗性因子可能参与Yatakemycin引起的DNA损伤修复。随后体外实验进一步证实YtkR2能够催化Yatakemycin与DNA形成的烷基化产物(3-Yatakemycinyl-2′- Deoxyadenosine,YTMA)中糖苷键的水解,生成无碱基位点(图 5)。此外,与ytkR2邻近的基因ytkR3、ytkR4和ytkR5所编码的蛋白分别与黄嘌呤磷酸核糖基转移酶、金属依赖的TatD家族的DNase和AP核酸内切酶具有一定同源性,而这些酶均参与BER途径中的碱基切除修复[54]。这也就说明Yatakemycin产生菌中含有完整的BER修复途径,糖基化酶YtkR2的确可以通过切除Yatakemycin与DNA形成的烷基化产物YTMA,诱导BER途径的发生,从而赋予宿主对Yatakemycin的抗性。
由于YtkR2自抗性功能是根据其与AlkD的相似性得出,而且AlkD同样能够切割烷基化产物YTMA[55],因此推测YtkR2可能采取与AlkD相似的机制识别并切割烷基化产物YTMA。在对损伤DNA进行切除时,大多数DNA糖基化酶都是采用碱基翻转机制移除损伤。然而在对AlkD切割损伤DNA机制进行解析时,研究发现AlkD不含有碱基结合口袋和明显的DNA嵌入氨基酸残基,而且在切割嘌呤碱基上N3和N7位烷基化损伤时,采用的是非碱基翻转(No-Base-Flipping)机制[49, 56-59]。AlkD通过与DNA磷酸骨架上脱氧核糖相互作用识别异常碱基,而且在整个反应过程中,损伤碱基仍位于DNA双链中。在这种构型下,蛋白中的氨基酸残基Trp109、Asp113和Trp187与损伤碱基对应的脱氧核糖部分作用,并催化糖苷键水解;其中Asp113用于稳定切割糖苷键后糖环上的正电荷,Trp109和Trp187则与脱氧核糖基团之间形成C-H/Π键[60]。体外酶活实验显示AlkD能够水解YTMA-DNA,然而体内实验结果表明在Yatakemycin的处理下,AlkD介导的BER修复途径仅能赋予宿主微弱抗性[55-61]。这也就说明YtkR2与AlkD在结构上存在差异。晶体结构显示,AlkD在将损伤碱基切除后仍然紧密结合在无碱基位点上,与AP-DNA和损伤碱基(Yatakemycin-Adenine,YTMAde)形成结构稳定的三联体复合物,从而阻止后续的核酸内切酶和外切酶(如EndoIV、ExoIII)靠近,导致损伤位点不能进一步被后续的BER途径修复,因此,AlkD催化的YTMA-DNA水解类似于一种自杀式酶催化反应,没有很好地形成催化循环,然而YtkR2和AlkD在与YTMAde结合位点上的氨基酸并不完全相同,这也就导致YtkR2对YTMAde的亲和性降低,从而使得三联体复合物稳定性降低,促进水解产物的解离,诱导后续BER系统进一步修复损伤;与AlkD相比,YtkR2显然更适合于识别YTMA-DNA,从而有效地启动BER系统以实现Yatakemycin生物合成过程中产生菌的自我保护[55]。
2.1.2 BER赋予宿主对Azinomycin B抗性Azinomycin B是杂合聚酮-非核糖体多肽类天然产物,含有高活性的氮杂双环和环氧基团,能够特异性地结合到富含GC序列的DNA大沟区域,形成链间交联[62-63]。DNA链间交联具有强烈的细胞毒性,能够通过阻断DNA链的分离终止DNA的复制与转录,导致染色体突变、重排、不稳定甚至细胞死亡[64]。在细菌中,DNA链间交联通常是由核苷酸切除修复途径与跨损伤合成或同源重组联合完成。然而,在研究Azinomycin B自抗性机制时,研究者发现在其生物合成基因簇中alkZ所编码的DNA糖基化酶AlkZ可以解除Azinomycin B介导的链间交联,诱导BER途径修复损伤,说明BER同样可以参与DNA链间交联的修复(图 6)[16]。在生物合成基因簇中,alkZ是Azinomycin B合成所必需基因,其编码的AlkZ属于HTH_42超家族,其缺失将导致产生菌死亡,而将其异源表达后可赋予异源宿主S. albus和S. lividans对Azinomycin B的抗性;此外,除了损伤修复外,AlkZ还可以通过与靶标DNA结合来防止交联形成,从而赋予宿主对Azinomycin B的自抗性[16]。随后对AlkZ的晶体结构进行解析时,研究发现其与DNA糖基化酶AlkD相似,都是C型结构,不含碱基结合口袋和明显的DNA嵌入氨基酸残基,而且蛋白质表面含有可与DNA结合的带正电荷的凹槽;氨基酸突变证实β-发夹(β11/β12)、Gln37和Gln39为AlkZ活性位点,紧接着通过DNA对接建模得出Gln37负责与DNA骨架上的磷酸基团相互作用,而β11/β12发卡则插入DNA小沟与DNA双链骨架和其中的一个修饰碱基相互作用,位于小沟外的Gln39则负责催化糖苷键水解;此外,对接建模指出在切除Azinomycin B诱导的DNA链间交联时,AlkZ可能以二聚体的形式存在[51]。然而,该推测还需要进一步验证。
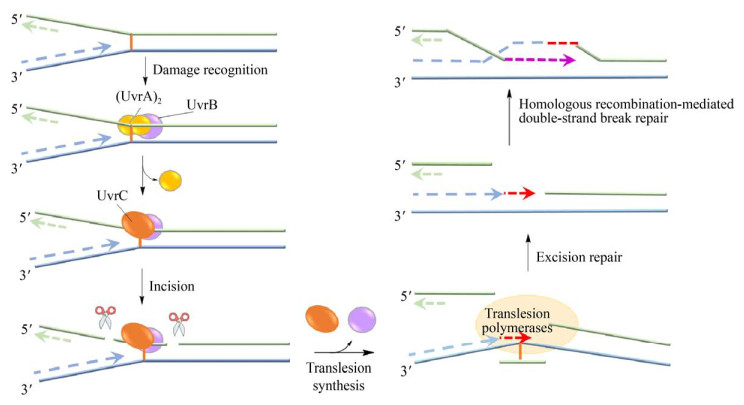
核苷酸切除修复是一种能够识别广谱DNA损伤的修复机制,对某些大范围的损伤,例如交联、大型加合物和多碱基损伤的修复尤为重要[65-66]。该修复途径主要包括3个步骤:损伤识别;损伤部位寡核苷酸的切除;DNA聚合酶合成新的片段填补缺口[67-68]。在原核生物中,寡核苷酸的切除过程通常由核酸内切酶UvrA、UvrB、UvrC所形成的蛋白复合体完成。如图 7所示,首先,UvrA和UvrB形成复合物对DNA链进行扫描,其中UvrA亚基负责通过双链螺旋结构中的扭曲识别损伤;当复合物识别出损伤位点时,UvrA亚基从复合物中解离,随后UvrC亚基与UvrB结合形成新的复合物UvrBUvrC,对损伤位点5′端和3′端的磷酸二酯键进行切割,形成12−13个核苷酸的寡核苷酸片段;最后,在DNA螺旋酶的作用下释放含损伤位点的寡核苷酸片段[69-71]。
近年研究报道发现,在DNA损伤类抗生素产生菌中,存在UvrA同源蛋白参与产生菌的自抗性保护系统,赋予宿主抗性。例如,柔红霉素(Daunorubicin,DNR)和阿霉素(Doxorubicin,DXR)生物合成基因簇中的基因drrC所编码的蛋白DrrC,该蛋白与UvrA具有较高相似性,能够识别DNR和DXR诱导的DNA损伤,释放嵌入在DNA链中的药物分子,从而修复损伤[72]。DrrC在DNR产生菌S. peucetius的自抗性系统中发挥着主要作用,基因drrC敲除只能在中断DNR产生的菌株中完成[73]。此外,色霉素A3 (Chromomycin A3)生物合成基因簇中的类UvrA蛋白CmrX同样可以赋予宿主对色霉素A3的抗性,基因cmrX的缺失导致抗生素的产量降低,而且产生菌本身对色霉素A3敏感性增加[74]。光辉霉素(Mithramycin)生物合成基因簇所编码的类UvrA蛋白MtrX同样可以赋予异源宿主对光辉霉素的抗性[75]。然而这些来自于天然产物生物合成基因簇的类UvrA蛋白并不能代替UvrA在E. coli中的功能,说明这类蛋白并不具有切割DNA的功能,而其修复机制还需要进一步研究。
目前,在抗生素生物合成基因簇中已发现的具有DNA切割能力的抗性因子仅有Azinomycin B生物合成基因簇中的alkN,该基因编码的蛋白AlkN属于Glyoxalase_2家族,与丝裂霉素(Mitomycin)结合蛋白MRD具有一定同源性[17]。在Mitomycin C产生菌中,MRD可与Mitomycin C结合屏蔽药物活性,随后与外排蛋白Mct一起形成高效的药物外排系统,抵抗来自丝裂霉素的细胞毒性[76-77]。然而,与MRD同源的AlkN虽然能与Azinomycin B结合,但并不具备屏蔽Azinomycin B药物活性的能力;相比较而言,AlkN发挥抗性功能是通过识别Azinomycin B从而结合到药物介导的交联DNA,随后对损伤位点附近的磷酸骨架进行切割,导致双链断裂,诱导后续NER途径完成损伤修复;尽管AlkN与已知核酸内切酶无序列相似性,但一系列体外实验证实了该蛋白为结构特异性核酸酶[17]。这类酶通常参与DNA复制、重组和转录等过程中出现的二级结构的修复,同时也参与DNA损伤修复[78]。DNA链间交联的修复是一个复杂的过程,涉及多种蛋白的协调作用。因此,在AlkN切割损伤DNA后,断裂的双链DNA是如何被修复还有待进一步研究。
3 总结与展望DNA损伤类抗生素具有强烈的细胞毒性,表现出显著的抗菌和抗癌活性,是药物开发的重要资源[20, 79]。在其产生菌中往往伴随着包括药物隔离、外排、修饰、靶标保护、损伤修复等在内的一种或多种抗性机制,其中DNA损伤修复机制在产生菌的自抗性保护系统中通常发挥着重要作用。然而相比于其他研究相对清楚的自抗性机制,抗生素产生菌中的DNA损伤修复机制尚不完善,目前还有很多问题有待研究。例如,新型的DNA糖基化酶YtkR2和AlkZ虽然能够通过非碱基翻转机制切除损伤碱基赋予宿主抗性,但切割产物是否能够被传统BER途径中的下游酶识别并处理,目前仍然不清楚;如果不能被识别,那么在抗生素产生菌中是否存在特异酶或其他的DNA损伤修复途径参与后续修复。其次,尽管YtkR2和AlkZ都属于DNA糖基化酶,但在序列上二者完全不具有同源性,属于2个不同家族,那么在不同类型的DNA损伤类抗生素产生菌中是否存在不同的DNA糖基化酶参与损伤修复。此外,NER途径参与修复交联DNA的机制也尚未完全研究清楚。交联DNA损伤修复过程极其复杂,需要多种酶和调控因子协同完成。虽然核酸酶AziN切割Azinomycin B双烷基化形成的交联DNA,诱导NER途径修复损伤,然而AziN与已知核酸内切酶并无同源性。传统NER途径以及下游中的酶是否能够识别AziN的切割产物并参与后续修复,或者在抗生素产生菌中是否存在其他新的核酸酶及辅因子参与损伤修复等问题都需要进一步研究。对此,可通过寻找传统修复途径中参与后续修复的同源基因、获得目标蛋白来验证传统损伤修复途径中的下游酶对DNA损伤的修复。另外,也可以通过蛋白与蛋白互作、蛋白与DNA互作等方法从DNA损伤类抗生素产生菌中筛选参与损伤修复的蛋白。相信随着基因挖掘、测序技术、分子生物学、结构生物学等技术的发展,天然产物产生菌中的DNA损伤修复自抗性机制将逐渐得到解析。
彻底了解抗生素抗性机制对于设计新的策略来应对耐药性问题至关重要。许多疾病与DNA损伤相关,如何防止和修复DNA损伤是预防和治疗疾病的重点关注方向[80-83]。抗生素产生菌中新型DNA损伤修复蛋白的发现暗示着在生物体中可能存在其他的DNA修复机制,这为研究感染性疾病和癌症等的治疗及预防提供了新的思路,同时也为利用同源抗性基因挖掘新型天然产物、鉴定生物合成基因簇、预测相关天然产物的生物活性打下了基础。此外,自抗性是抗生素产生的先决条件,因此,对于DNA损伤修复抗性机制的研究,也为基因改造构建工程菌株提高DNA损伤类抗生素产量以及利用合成生物学的方法合成新型抗生素开启了新的思路。
致谢: 2000年9月1日(UC-Davis)到2003年8月15日(UW-Madison),我很荣幸能够有机会在沈奔教授的课题组做博士后研究。近3年的时间里,我深刻体会了如何做科研,并奠定了后来我自己课题组的研究方向。我们努力在接下来的时光里将沈老师对科研热爱与追求的精神进一步发扬光大。(唐功利)| [1] |
Newman DJ, Cragg GM. Natural products as sources of new drugs from 1981 to 2014[J]. Journal of Natural Products, 2016, 79(3): 629-661. DOI:10.1021/acs.jnatprod.5b01055 |
| [2] |
Rodrigues T, Reker D, Schneider P, Schneider G. Counting on natural products for drug design[J]. Nature Chemistry, 2016, 8(6): 531-541. DOI:10.1038/nchem.2479 |
| [3] |
Hutchings MI, Truman AW, Wilkinson B. Antibiotics: past, present and future[J]. Current Opinion in Microbiology, 2019, 51: 72-80. DOI:10.1016/j.mib.2019.10.008 |
| [4] |
Liu BY, Xue QZ, Tang Y, Cao J, Guengerich FP, Zhang HD. Mechanisms of mutagenesis: DNA replication in the presence of DNA damage[J]. Mutation Research/Reviews in Mutation Research, 2016, 768: 53-67. DOI:10.1016/j.mrrev.2016.03.006 |
| [5] |
He MY, Zhou WW, Li C, Guo MX. MicroRNAs, DNA damage response, and cancer treatment[J]. International Journal of Molecular Sciences, 2016, 17(12): E2087. DOI:10.3390/ijms17122087 |
| [6] |
Wit RD, Stoter G, Kaye SB, Sleijfer DT, Jones WG, Ten Bokkel Huinink WW, Rea LA, Collette L, Sylvester R. Importance of bleomycin in combination chemotherapy for good-prognosis testicular nonseminoma: a randomized study of the European Organization for Research and Treatment of Cancer Genitourinary Tract Cancer Cooperative Group[J]. Journal of Clinical Oncology, 1997, 15(5): 1837-1843. DOI:10.1200/JCO.1997.15.5.1837 |
| [7] |
Verweij J, Pinedo HM. Mitomycin C: mechanism of action, usefulness and limitations[J]. Anti Cancer Drugs, 1990, 1(1): 5-13. DOI:10.1097/00001813-199010000-00002 |
| [8] |
Neil JO. The review on antimocronial resistance[Z/OL]. 2016. https://amr-review.org
|
| [9] |
Yan Y, Liu QK, Zang X, Yuan SG, Bat-Erdene U, Nguyen C, Gan JH, Zhou JH, Jacobsen SE, Tang Y. Resistance-gene-directed discovery of a natural-product herbicide with a new mode of action[J]. Nature, 2018, 559(7714): 415-418. DOI:10.1038/s41586-018-0319-4 |
| [10] |
Yan Y, Liu N, Tang Y. Recent developments in self-resistance gene directed natural product discovery[J]. Natural Product Reports, 2020, 37(7): 879-892. DOI:10.1039/C9NP00050J |
| [11] |
Wencewicz TA. Crossroads of antibiotic resistance and biosynthesis[J]. Journal of Molecular Biology, 2019, 431(18): 3370-3399. DOI:10.1016/j.jmb.2019.06.033 |
| [12] |
Cundliffe E, Demain AL. Avoidance of suicide in antibiotic-producing microbes[J]. Journal of Industrial Microbiology & Biotechnology, 2010, 37(7): 643-672. |
| [13] |
Tenconi E, Rigali S. Self-resistance mechanisms to DNA-damaging antitumor antibiotics in actinobacteria[J]. Current Opinion in Microbiology, 2018, 45: 100-108. DOI:10.1016/j.mib.2018.03.003 |
| [14] |
Almabruk KH, Dinh LK, Philmus B. Self-resistance of natural product producers: past, present, and future focusing on self-resistant protein variants[J]. ACS Chemical Biology, 2018, 13(6): 1426-1437. DOI:10.1021/acschembio.8b00173 |
| [15] |
Xu H, Huang W, He QL, Zhao ZX, Zhang F, Wang RX, Kang JW, Tang GL. Self-resistance to an antitumor antibiotic: a DNA glycosylase triggers the base-excision repair system in yatakemycin biosynthesis[J]. Angewandte Chemie: International Ed in English, 2012, 51(42): 10532-10536. DOI:10.1002/anie.201204109 |
| [16] |
Wang S, Liu K, Xiao L, Yang LY, Li H, Zhang FX, Lei L, Li SQ, Feng X, Li AY, et al. Characterization of a novel DNA glycosylase from S. sahachiroi involved in the reduction and repair of azinomycin B induced DNA damage[J]. Nucleic Acids Research, 2016, 44(1): 187-197. DOI:10.1093/nar/gkv949 |
| [17] |
Chen XR, Sun YD, Wang S, Ying K, Xiao L, Liu K, Zuo XL, He J. Identification of a novel structure-specific endonuclease AziN that contributes to the repair of azinomycin B-mediated DNA interstrand crosslinks[J]. Nucleic Acids Research, 2020, 48(2): 709-718. |
| [18] |
Portugal J. Challenging transcription by DNA-binding antitumor drugs[J]. Biochemical Pharmacology, 2018, 155: 336-345. DOI:10.1016/j.bcp.2018.07.030 |
| [19] |
Deslouches B, Di YP. Antimicrobial peptides with selective antitumor mechanisms: prospect for anticancer applications[J]. Oncotarget, 2017, 8(28): 46635-46651. DOI:10.18632/oncotarget.16743 |
| [20] |
Sharifi-Rad J, Ozleyen A, Boyunegmez Tumer T, Oluwaseun Adetunji C, El Omari N, Balahbib A, Taheri Y, Bouyahya A, Martorell M, Martins N, et al. Natural products and synthetic analogs as a source of antitumor drugs[J]. Biomolecules, 2019, 9(11): 679. DOI:10.3390/biom9110679 |
| [21] |
Gavande NS, Vandervere-Carozza PS, Hinshaw HD, Jalal SI, Sears CR, Pawelczak KS, Turchi JJ. DNA repair targeted therapy: the past or future of cancer treatment?[J]. Pharmacology & Therapeutics, 2016, 160: 65-83. |
| [22] |
Rehman SU, Sarwar T, Husain MA, Ishqi HM, Tabish M. Studying non-covalent drug-DNA interactions[J]. Archives of Biochemistry and Biophysics, 2015, 576: 49-60. DOI:10.1016/j.abb.2015.03.024 |
| [23] |
Durand M, Thuong NT, Maurizot JC. Binding of netropsin to a DNA triple helix[J]. Journal of Biological Chemistry, 1992, 267(34): 24394-24399. DOI:10.1016/S0021-9258(18)35779-X |
| [24] |
Kopka ML, Yoon C, Goodsell D, Pjura P, Dickerson RE. The molecular origin of DNA-drug specificity in netropsin and distamycin[J]. Proceedings of the National Academy of Sciences of the United States of America, 1985, 82(5): 1376-1380. DOI:10.1073/pnas.82.5.1376 |
| [25] |
Strekowski L, Wilson B. Noncovalent interactions with DNA: an overview[J]. Mutation Research, 2007, 623(1/2): 3-13. |
| [26] |
Goldberg IH. The interaction of actinomycin with DNA[J]. Antibiotics and Chemotherapy, 1971, 17: 67-86. DOI:10.1159/000392364 |
| [27] |
Finocchiaro G. Actinomycin D: a new opening for an old drug[J]. Neuro-Oncology, 2020, 22(9): 1235-1236. DOI:10.1093/neuonc/noaa172 |
| [28] |
Lin SQ, Zhang CY, Liu FY, Ma JH, Jia FJ, Han Z, Xie WD, Li X. Actinomycin V inhibits migration and invasion via suppressing snail/slug-mediated epithelial-mesenchymal transition progression in human breast cancer MDA-MB-231 cells in vitro[J]. Marine Drugs, 2019, 17(5): E305. DOI:10.3390/md17050305 |
| [29] |
Parrish JP, Kastrinsky DB, Wolkenberg SE, Igarashi Y, Boger DL. DNA alkylation properties of yatakemycin[J]. Journal of the American Chemical Society, 2003, 125(36): 10971-10976. DOI:10.1021/ja035984h |
| [30] |
Rajski SR, Williams RM. DNA cross-linking agents as antitumor drugs[J]. Chemical Reviews, 1998, 98(8): 2723-2796. DOI:10.1021/cr9800199 |
| [31] |
Armstrong RW, Salvati ME, Nguyen M. Novel interstrand cross-links induced by the antitumor antibiotic carzinophilin/azinomycin B[J]. Journal of the American Chemical Society, 1992, 114(8): 3144-3145. DOI:10.1021/ja00034a074 |
| [32] |
Fujiwara T, Saito I, Sugiyama H. Highly efficient DNA interstrand crosslinking induced by an antitumor antibiotic, carzinophilin[J]. Tetrahedron Letters, 1999, 40(2): 315-318. DOI:10.1016/S0040-4039(98)02339-9 |
| [33] |
Gates KS. An overview of chemical processes that damage cellular DNA: spontaneous hydrolysis, alkylation, and reactions with radicals[J]. Chemical Research in Toxicology, 2009, 22(11): 1747-1760. DOI:10.1021/tx900242k |
| [34] |
Chatterjee N, Walker GC. Mechanisms of DNA damage, repair, and mutagenesis[J]. Environmental and Molecular Mutagenesis, 2017, 58(5): 235-263. DOI:10.1002/em.22087 |
| [35] |
Terabayashi T, Hanada K. Genome instability syndromes caused by impaired DNA repair and aberrant DNA damage responses[J]. Cell Biology and Toxicology, 2018, 34(5): 337-350. DOI:10.1007/s10565-018-9429-x |
| [36] |
Kiwerska K, Szyfter K. DNA repair in cancer initiation, progression, and therapy: a double-edged sword[J]. Journal of Applied Genetics, 2019, 60(3/4): 329-334. |
| [37] |
Yi C, He C. DNA repair by reversal of DNA damage[J]. Cold Spring Harbor Perspectives in Biology, 2013, 5(1): a012575. DOI:10.1101/cshperspect.a012575 |
| [38] |
Kim JH, Ryu TH, Lee SS, Lee S, Chung BY. Ionizing radiation manifesting DNA damage response in plants: an overview of DNA damage signaling and repair mechanisms in plants[J]. Plant Science, 2019, 278: 44-53. DOI:10.1016/j.plantsci.2018.10.013 |
| [39] |
Spampinato CP. Protecting DNA from errors and damage: an overview of DNA repair mechanisms in plants compared to mammals[J]. Cellular and Molecular Life Sciences, 2017, 74(9): 1693-1709. DOI:10.1007/s00018-016-2436-2 |
| [40] |
Krokan HE, Bjoras M. Base excision repair[J]. Cold Spring Harbor Perspectives in Biology, 2013, 5(4): a012583. |
| [41] |
Kim YJ, Wilson DM. Overview of base excision repair biochemistry[J]. Current Molecular Pharmacology, 2012, 5(1): 3-13. DOI:10.2174/1874467211205010003 |
| [42] |
Wallace SS. Base excision repair: a critical player in many games[J]. DNA Repair, 2014, 19: 14-26. DOI:10.1016/j.dnarep.2014.03.030 |
| [43] |
Dizdaroglu M, Coskun E, Jaruga P. Repair of oxidatively induced DNA damage by DNA glycosylases: mechanisms of action, substrate specificities and excision kinetics[J]. Mutation Research/Reviews in Mutation Research, 2017, 771: 99-127. |
| [44] |
Schermerhorn KM, Delaney S. A chemical and kinetic perspective on base excision repair of DNA[J]. Accounts of Chemical Research, 2014, 47(4): 1238-1246. DOI:10.1021/ar400275a |
| [45] |
Beard WA, Wilson SH. DNA polymerase beta and other gap-filling enzymes in mammalian base excision repair[J]. The Enzymes, 2019, 45: 1-26. |
| [46] |
Mullins EA, Rodriguez AA, Bradley NP, Eichman BF. Emerging roles of DNA glycosylases and the base excision repair pathway[J]. Trends in Biochemical Sciences, 2019, 44(9): 765-781. DOI:10.1016/j.tibs.2019.04.006 |
| [47] |
Coey CT, Drohat AC. Kinetic methods for studying DNA glycosylases functioning in base excision repair[J]. Methods in Enzymology, 2017, 592: 357-376. |
| [48] |
Stivers JT. Site-specific DNA damage recognition by enzyme-induced base flipping[J]. Progress in Nucleic Acid Research and Molecular Biology, 2004, 77: 37-65. |
| [49] |
Brooks SC, Adhikary S, Rubinson EH, Eichman BF. Recent advances in the structural mechanisms of DNA glycosylases[J]. Biochimica et Biophysica Acta: BBA-Proteins and Proteomics, 2013, 1834(1): 247-271. DOI:10.1016/j.bbapap.2012.10.005 |
| [50] |
Slupphaug G, Mol CD, Kavli B, Arvai AS, Krokan HE, Tainer JA. A nucleotide-flipping mechanism from the structure of human uracil-DNA glycosylase bound to DNA[J]. Nature, 1996, 384(6604): 87-92. DOI:10.1038/384087a0 |
| [51] |
Mullins EA, Warren GM, Bradley NP, Eichman BF. Structure of a DNA glycosylase that unhooks interstrand cross-links[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(17): 4400-4405. DOI:10.1073/pnas.1703066114 |
| [52] |
MacMillan KS, Boger DL. Fundamental relationships between structure, reactivity, and biological activity for the duocarmycins and CC-1065[J]. Journal of Medicinal Chemistry, 2009, 52(19): 5771-5780. DOI:10.1021/jm9006214 |
| [53] |
Wu S, Jian XH, Yuan H, Jin WB, Yin Y, Wang LY, Zhao J, Tang GL. Unified biosynthetic origin of the benzodipyrrole subunits in CC-1065[J]. ACS Chemical Biology, 2017, 12(6): 1603-1610. DOI:10.1021/acschembio.7b00302 |
| [54] |
Schärer OD. Chemistry and biology of DNA repair[J]. Angewandte Chemie International Edition, 2003, 42(26): 2946-2974. DOI:10.1002/anie.200200523 |
| [55] |
Mullins EA, Shi RX, Eichman BF. Toxicity and repair of DNA adducts produced by the natural product yatakemycin[J]. Nature Chemical Biology, 2017, 13(9): 1002-1008. DOI:10.1038/nchembio.2439 |
| [56] |
Mullins EA, Shi RX, Parsons ZD, Yuen PK, David SS, Igarashi Y, Eichman BF. The DNA glycosylase AlkD uses a non-base-flipping mechanism to excise bulky lesions[J]. Nature, 2015, 527(7577): 254-258. DOI:10.1038/nature15728 |
| [57] |
Dalhus B, Helle IH, Backe PH, Alseth I, Rognes T, Bjørås M, Laerdahl JK. Structural insight into repair of alkylated DNA by a new superfamily of DNA glycosylases comprising HEAT-like repeats[J]. Nucleic Acids Research, 2007, 35(7): 2451-2459. DOI:10.1093/nar/gkm039 |
| [58] |
Rubinson EH, Metz AH, O'Quin J, Eichman BF. A new protein architecture for processing alkylation damaged DNA: the crystal structure of DNA glycosylase AlkD[J]. Journal of Molecular Biology, 2008, 381(1): 13-23. DOI:10.1016/j.jmb.2008.05.078 |
| [59] |
Rubinson EH, Prakasha Gowda AS, Spratt TE, Gold B, Eichman BF. An unprecedented nucleic acid capture mechanism for excision of DNA damage[J]. Nature, 2010, 468(7322): 406-411. DOI:10.1038/nature09428 |
| [60] |
Parsons ZD, Bland JM, Mullins EA, Eichman BF. A catalytic role for C-H/π interactions in base excision repair by Bacillus cereus DNA glycosylase AlkD[J]. Journal of the American Chemical Society, 2016, 138(36): 11485-11488. DOI:10.1021/jacs.6b07399 |
| [61] |
Stivers JT, Jiang YL. A mechanistic perspective on the chemistry of DNA repair glycosylases[J]. Chemical Reviews, 2003, 103(7): 2729-2760. DOI:10.1021/cr010219b |
| [62] |
Lawley PD, Phillips DH. DNA adducts from chemotherapeutic agents[J]. Mutation Research, 1996, 355(1/2): 13-40. |
| [63] |
Hata T, Koga F, Sano Y, Kanamori K, Matsumae A, Sugawara R, Hoshi T, Shima T, Ito S, Tomizawa S. Carzinophilin, a new tumor inhibitory substance produced by Streptomyces I[J]. The Journal of Antibiotics, 1954, 7(4): 107-112. |
| [64] |
Dronkert MLG, Kanaar R. Repair of DNA interstrand cross-links[J]. Mutation Research/DNA Repair, 2001, 486(4): 217-247. DOI:10.1016/S0921-8777(01)00092-1 |
| [65] |
Truglio JJ, Croteau DL, Van Houten B, Kisker C. Prokaryotic nucleotide excision repair: the UvrABC system[J]. Chemical Reviews, 2006, 106(2): 233-252. DOI:10.1021/cr040471u |
| [66] |
Kemp MG. Damage removal and gap filling in nucleotide excision repair[J]. The Enzymes, 2019, 45: 59-97. |
| [67] |
Sancar A. DNA excision repair[J]. Annual Review of Biochemistry, 1996, 65(1): 43-81. DOI:10.1146/annurev.bi.65.070196.000355 |
| [68] |
Wood RD. Nucleotide excision repair in mammalian cells[J]. Journal of Biological Chemistry, 1997, 272(38): 23465-23468. DOI:10.1074/jbc.272.38.23465 |
| [69] |
Kisker C, Kuper J, Van Houten B. Prokaryotic nucleotide excision repair[J]. Cold Spring Harbor Perspectives in Biology, 2013, 5(3): a012591. |
| [70] |
Jaciuk M, Swuec P, Gaur V, Kasprzak JM, Renault L, Dobrychłop M, Nirwal S, Bujnicki JM, Costa A, Nowotny M. A combined structural and biochemical approach reveals translocation and stalling of UvrB on the DNA lesion as a mechanism of damage verification in bacterial nucleotide excision repair[J]. DNA Repair, 2020, 85: 102746. DOI:10.1016/j.dnarep.2019.102746 |
| [71] |
Selby CP, Lindsey-Boltz LA, Yang YY, Sancar A. Mycobacteria excise DNA damage in 12- or 13-nucleotide-long oligomers by prokaryotic-type dual incisions and performs transcription-coupled repair[J]. Journal of Biological Chemistry, 2020, 295(50): 17374-17380. DOI:10.1074/jbc.AC120.016325 |
| [72] |
Prija F, Prasad R. DrrC protein of Streptomyces peucetius removes daunorubicin from intercalated dnrI promoter[J]. Microbiological Research, 2017, 202: 30-35. DOI:10.1016/j.micres.2017.05.002 |
| [73] |
Lomovskaya N, Hong SK, Kim SU, Fonstein L, Furuya K, Hutchinson RC. The Streptomyces peucetius drrC gene encodes a UvrA-like protein involved in daunorubicin resistance and production[J]. Journal of Bacteriology, 1996, 178(11): 3238-3245. DOI:10.1128/jb.178.11.3238-3245.1996 |
| [74] |
Menéndez N, Braña AF, Salas JA, Méndez C. Involvement of a chromomycin ABC transporter system in secretion of a deacetylated precursor during chromomycin biosynthesis[J]. Microbiology, 2007, 153(Pt 9): 3061-3070. |
| [75] |
Garcia-Bernardo J, Braña AF, Méndez C, Salas JA. Insertional inactivation of mtrX and mtrY genes from the mithramycin gene cluster affects production and growth of the producer organism Streptomyces argillaceus[J]. FEMS Microbiology Letters, 2000, 186(1): 61-65. DOI:10.1111/j.1574-6968.2000.tb09082.x |
| [76] |
Sheldon PJ, Johnson DA, August PR, Liu HW, Sherman DH. Characterization of a mitomycin-binding drug resistance mechanism from the producing organism, Streptomyces lavendulae[J]. Journal of Bacteriology, 1997, 179(5): 1796-1804. DOI:10.1128/jb.179.5.1796-1804.1997 |
| [77] |
Sheldon PJ, Mao Y, He M, Sherman DH. Mitomycin resistance in Streptomyces lavendulae includes a novel drug-binding-protein-dependent export system[J]. Journal of Bacteriology, 1999, 181(8): 2507-2512. DOI:10.1128/JB.181.8.2507-2512.1999 |
| [78] |
Dehé PM, Gaillard PHL. Control of structure-specific endonucleases to maintain genome stability[J]. Nature Reviews Molecular Cell Biology, 2017, 18(5): 315-330. |
| [79] |
Bhattacharya P, Mukherjee S, Mandal SM. Fluoroquinolone antibiotics show genotoxic effect through DNA-binding and oxidative damage[J]. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 2020, 227: 117634. DOI:10.1016/j.saa.2019.117634 |
| [80] |
Curigliano G. Targeting DNA Repair[A]//Mechanisms of Drug Resistance in Cancer Therapy[M]. Cham: Springer International Publishing, 2017: 161-180
|
| [81] |
Ferguson LR, Chen H, Collins AR, Connell M, Damia G, Dasgupta S, Malhotra M, Meeker AK, Amedei A, Amin A, et al. Genomic instability in human cancer: Molecular insights and opportunities for therapeutic attack and prevention through diet and nutrition[J]. Seminars in Cancer Biology, 2015, 35: S5-S24. |
| [82] |
Tian H, Gao Z, Li HZ, Zhang BF, Wang G, Zhang Q, Pei DS, Zheng JN. DNA damage response — a double-edged sword in cancer prevention and cancer therapy[J]. Cancer Letters, 2015, 358(1): 8-16. DOI:10.1016/j.canlet.2014.12.038 |
| [83] |
Kurthkoti K, Kumar P, Sang PB, Varshney U. Base excision repair pathways of bacteria: new promise for an old problem[J]. Future Medicinal Chemistry, 2020, 12(4): 339-355. |
 2021, Vol. 48
2021, Vol. 48