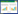扩展功能
文章信息
- 向晨晨, 周珊珊, 柴树茂, 曹明明, 王立岩, 冯治洋
- XIANG Chenchen, ZHOU Shanshan, CHAI Shumao, CAO Mingming, WANG Liyan, FENG Zhiyang
- 红树林链霉菌ZFSM1-146中抗菌活性物质的发现
- Discovery of antibacterial compounds from a mangrove Streptomyces strain ZFSM1-146
- 微生物学通报, 2021, 48(7): 2329-2340
- Microbiology China, 2021, 48(7): 2329-2340
- DOI: 10.13344/j.microbiol.china.201203
-
文章历史
- 收稿日期: 2020-12-28
- 接受日期: 2021-03-06
- 网络首发日期: 2021-04-01
2. 深圳大学生命与海洋科学学院 广东 深圳 518060
2. College of Life Science and Oceanography, Shenzhen University, Shenzhen, Guangdong 518060, China
微生物来源的天然产物及其衍生物是药物研发的重要来源[1],已广泛应用于治疗癌症、细菌和病毒感染以及免疫抑制等疾病[2]。然而,从普通陆地环境微生物中挖掘结构新颖、活性独特的次级代谢产物越来越困难,研究者们把目光投向了特殊环境,如红树林[3-4]、海洋[5]、极地[6]、沙漠[7]、热泉[8]等。
红树林是一种生长在热带和亚热带海岸潮间带、受周期性潮水浸没的独特植物群落,具有耐缺氧、高水分、高盐等特征,是蕴含丰富微生物资源的特殊生态系统[9]。放线菌是天然产物的重要来源之一[10]。截至目前,从微生物中获得的生物活性化合物中,45%是由放线菌产生的[11]。在极端环境中生长的微生物是特殊代谢产物的重要来源[12]。放线菌一般适宜干燥、偏碱性的环境,而红树林土壤中的放线菌长期生存于湿润、强酸性、高盐的环境,必然有其独特的分子适应机制,具备形成结构新颖的次级代谢产物的潜能。因此,红树林土壤是筛选和发现放线菌新物种及生物活性次级代谢产物的宝贵资源。
本研究从红树林土壤中筛选出一株抗菌活性较强的放线菌,通过16S rRNA基因序列及其基因片段构建的系统发育树分析,确定其与已知菌种的亲缘关系。通过活性追踪法,确定发酵粗提物中的抗菌活性物质;并通过质谱和核磁共振波谱手段,鉴定出抗菌活性物质的结构。本研究中抗菌活性物质生产菌的发现,为后续相关抗菌化合物的生产提供了可能的菌种资源,为后续相关化合物类似药的开发提供了物质基础,同时表明了红树林土壤放线菌资源丰富,具有挖掘新型天然产物的潜力。
1 材料与方法 1.1 材料 1.1.1 红树林土壤样品红树林土壤样品采集自广东省深圳市红树林自然保护区(22°31′21′′N,114°1′54′′E)。
1.1.2 供试菌Bacillus subtilis 168、Staphylococcus aureus ATCC 25923、Escherichia coli ATCC 25922均由本实验室保存;Methicillin resistant Staphylococcus aureus (MRSA) ATCC 43300由南京农业大学食品生物技术与酶工程研究室惠赠。
1.1.3 培养基高氏一号培养基:可溶性淀粉20.00 g,K2HPO4 0.50 g,FeSO4 0.01 g,MgSO4 0.50 g,KNO3 1.00 g,NaCl 0.50 g,琼脂20.00 g,蒸馏水定容至1 L,pH 7.3。用于放线菌的筛选。
R5培养基:蔗糖103.00 g,K2SO4 0.25 g,MgCl2·6H2O 10.12 g,葡萄糖10.00 g,酪蛋白氨基酸0.10 g,微量元素溶液2 mL (400 μL/L母液),酵母粉5.00 g,TES buffer 5.73 g,蒸馏水定容至1 L。灭菌后添加KH2PO4 (0.5%) 10 mL,CaCl2 (5 mol/L) 4 mL,L-proline (20%) 15 mL,NaOH (1 mol/L) 7 mL。用于放线菌种子培养及发酵。
ISP4培养基:可溶性淀粉10.0 g,K2HPO4 1.0 g,MgSO4 1.0 g,NaCl 1.0 g,(NH4)2SO4 2.0 g,CaCO3 2.0 g,现配现用:FeSO4·7H2O 1.0 g/30 mL (54.9 μL),MnCl2 1.0 g/50 mL (196.25 μL),ZnSO4 1.0 g/50 mL (222.5 μL),蒸馏水定容至1 L。用于放线菌种子培养及发酵。
LB培养基:NaCl 10.0 g,酵母粉5.0 g,胰蛋白胨10.0 g,蒸馏水定容至1 L。用于供试菌的培养。
MH肉汤培养基:称取21.0 g MH肉汤培养基粉末于1 L水中溶解。用于测定化合物MIC值。
TSB培养基:称取30.0 g TSB培养基粉末于1 L水中溶解。用于培养放线菌以提取基因组DNA。
MⅠ培养基:酵母粉20.0 g,KH2PO4 1.5 g,MgSO4·7H2O 0.5 g,蒸馏水定容至1 L,pH 8.0。
MⅡ培养基:大豆粉20.0 g,KH2PO4 1.5 g,MgSO4·7H2O 0.5 g,蒸馏水定容至1 L,pH 8.0。
MⅢ培养基:可溶性淀粉22.5 g,酵母粉5.0 g,KH2PO4 1.5 g,MgSO4·7H2O 0.5 g,蒸馏水定容至1 L,pH 8.0。
MⅣ培养基:葡萄糖22.5 g,酵母粉5.0 g,KH2PO4 1.5 g,MgSO4·7H2O 0.5 g,蒸馏水定容至1 L,pH 8.0。
MⅠ−MⅣ培养基用于放线菌素产量优化。
所有培养基均1×105 Pa灭菌20 min。
1.1.4 主要试剂和仪器分析纯甲醇、分析纯乙酸乙酯、分析纯二氯甲烷等有机试剂,上海凌峰化学试剂有限公司;色谱级甲醇,TEDIA公司。
细菌DNA提取试剂盒,礼美生物科技(上海)有限公司;DNA凝胶回收试剂盒,北京擎科新业生物技术有限公司。
恒温培养摇床,上海一恒科学仪器有限公司;旋转蒸发仪,上海爱朗仪器有限公司;高效液相色谱仪,岛津公司;超净工作台,苏州苏洁净化设备有限公司;立式压力蒸汽灭菌锅,上海申安医疗器械厂;恒温培养箱,上海申安医疗器械有限公司。
1.2 方法 1.2.1 红树林土壤放线菌的分离采用稀释涂布平板法:称取5 g土样于45 mL无菌水中溶解,28 ℃、225 r/min摇床振荡45 min,即终浓度为10−1 g/mL;吸取0.5 mL土壤悬液至4.5 mL无菌水中,连续进行10倍比梯度稀释,使其终浓度为10−2、10−3、10−4、10−5 g/mL,各取0.2 mL分别涂布于高氏一号培养基平板上,置恒温培养箱28 ℃培养7 d,每个稀释度做3个重复。挑出放线菌后于高氏一号培养基纯化培养3次(所有高氏一号培养基内均含有终浓度50 mg/L重铬酸钾),最后再挑取单菌落涂布于高氏一号培养基,7 d后收孢子。
1.2.2 活性放线菌初筛及复筛参照张孟[13]的初筛方法,将测试菌株分别划线于R5和ISP4固体培养基上,28 ℃培养7 d,在平板上铺一层含1%琼脂且含指示菌(B. subtilis 168和S. aureus ATCC 25923)的LB培养基,12 h后观察抑菌圈。将40 μL菌株孢子接种到含40 mL R5液体培养基的250 mL摇瓶中,28 ℃、225 r/min培养3 d,按10%的量分别转接至50 mL R5和ISP4 (含5%树脂)液体培养基,于28 ℃、225 r/min培养,每个编号做3个重复。7 d后,将R5培养基发酵液按1:1 (体积比)加入乙酸乙酯萃取,上层经旋转蒸发仪减压蒸干,溶于800 μL甲醇,得到R5粗提物;12 d后,收集ISP4液体培养基发酵液中的树脂,洗后晾干,用4 mL甲醇浸泡萃取,上清经旋转蒸发仪减压蒸干后溶于800 μL甲醇,得到ISP4粗提物;根据滤纸片法[14],分别吸取10 μL R5和ISP4粗提物于直径为6 mm的无菌滤纸片上,晾干后置于含B. subtilis 168和S. aureus ATCC 25923的LB平板上,12 h后观察抑菌结果,同时以甲醇为阴性对照。
1.2.3 菌种鉴定取菌株ZFSM1-146的孢子3 μL接种于含3 mL TSB液体培养基的玻璃管中,28 ℃、225 r/min培养3 d,使用细菌DNA提取试剂盒提取基因组DNA。以提取的基因组DNA为模板,利用扩增细菌16S rRNA基因的通用引物对27F (5′-AGAGTTT GATCCTGGCTCAG-3′)和1492R (5′-TACGGCTAC CTTGTTACGACTT-3′)[15]进行PCR扩增反应。PCR反应体系(50 μL):2×Prime STAR Mix 25 μL,27F (10 μmol/L) 2 µL,1492R (10 μmol/L) 2 μL,模板浓度(150 ng/μL) 2 μL,DMSO 2.5 μL,ddH2O 16.5 μL。PCR反应条件:95 ℃ 5 min;98 ℃ 1 min;98 ℃ 10 s,60 ℃ 5 s (每个循环后降1 ℃),72 ℃ 1 min 40 s,10个循环;98 ℃ 10 s,50 ℃ 5 s,72 ℃ 1 min 40 s,30个循环;72 ℃ 10 min。通过琼脂糖凝胶电泳和DNA凝胶回收试剂盒纯化PCR产物后,送通用(安徽)生物公司测序。
将菌株ZFSM1-146的16S rRNA基因序列上传到NCBI网站进行Blast比对,寻找相似性近的菌株,选用MEGA 5.0的ClustalW Mutiple alignment对结果中一致性高的16S rRNA基因序列和ZFSM1- 146序列进行多序列比对,采用Neighbor-joining法构建系统发育树,设定步长检验次数1 000次,位点缺失阈值50%,同时以Micromonospora inyonensis DSM 46123作为外参,进一步确定菌株ZFSM1-146与已知菌种的亲缘关系。
1.2.4 发酵产物的提取分离及抗菌活性物质的确定取菌株ZFSM1-146孢子40 μL接种到含40 mL R5液体培养基的250 mL摇瓶中,28 ℃、225 r/min培养3 d,取40 mL种子液转接至含400 mL R5液体培养基的1 L摇瓶中,于28 ℃、225 r/min发酵7 d,共发酵10 L。发酵液用等体积乙酸乙酯萃取3次后合并萃取液,蒸干后发酵粗提物以二氯甲烷: 甲醇为梯度洗脱剂进行硅胶色谱层析分离,分离组分分别用滤纸片法检测抑菌活性。发酵粗提物与分离组分利用HPLC与活性检测确定目标化合物后,利用HPLC纯化目标化合物。
HPLC检测条件:流动相:MeOH-H2O;色谱柱:Fisher Wharton Xbrige-C18,5 μm,4.6 mm× 250 mm;流速:1 mL/min。检测程序:0−30 min,20%−100% MeOH;30−35 min,100% MeOH。研究中的HPLC检测方法均保持一致。
HPLC纯化条件:流动相:MeOH-H2O;色谱柱:SHIMADZU Shim-pack GIS-C18,10 μm,10 mm×250 mm;流速:4 mL/min。检测程序:0−35 min,75% MeOH。
1.2.5 抗菌活性物质结构鉴定通过HPLC纯化,得到化合物1−3的纯品。对化合物1−3进行HR-ESI-MS检测,确定分子量并计算分子式。化合物1−3用氘代氯仿溶解后,利用NMR技术检测化合物1−3的氢谱和碳谱。
1.2.6 抗菌活性物质最小抑菌浓度的测定参照传统的微量肉汤稀释法[16]进行体外抗菌活性试验,测定化合物1−3的最小抑菌浓度(Minimum Inhibitory Concentration,MIC)。化合物1−3用DMSO溶解,配制成母液;B. subtilis 168、S. aureus ATCC 25923和MRSA ATCC 43300为供试菌时,设置的药物终浓度梯度为1.6、0.8、0.4、0.2、0.1、0.05、0.025、0.012 5、0.006、0.003、0.001 5、0.000 75 μmol/L,E. coli ATCC 25922为供试菌时,设置的药物终浓度梯度为160、80、40、20、10、5、2.5、1.25、0.625、0.312 5、0.16、0.08 μmol/L。
1.2.7 放线菌素产量优化采用MⅠ–MⅣ培养基[17]优化放线菌素XOβ、X2和D的产量,发酵条件及产物提取方法同利用R5培养基的菌种发酵与产物提取方法。
2 结果与分析 2.1 红树林土壤放线菌的分离及活性筛选通过高氏一号培养基从红树林土壤共分离得到350株放线菌。分离的放线菌先通过琼脂块法初筛,结果显示22株放线菌分别在R5和ISP4平板上对B. subtilis 168和S. aureus ATCC 25923都有抑菌活性;初筛得到的放线菌再经滤纸片法复筛,结果显示11株放线菌的R5或ISP4发酵粗提物对B. subtilis 168和S. aureus ATCC 25923有抑菌活性(表 1)。菌株ZFSM1-146发酵产物的抑菌活性最强,发酵液呈深黄色,选取该菌株做进一步研究。
| No. | R5 inhibition zone diameter (cm) | ISP4 inhibition zone diameter (cm) | |||
| B. subtilis 168 | S. aureus ATCC 25923 | B. subtilis 168 | S. aureus ATCC 25923 | ||
| ZFSF12-2 | 1.0 | 0.8 | 0.0 | 0.0 | |
| ZFSF21-28 | 0.0 | 0.0 | 2.0 | 1.8 | |
| ZFSM1-4 | 2.5 | 1.5 | 2.4 | 1.8 | |
| ZFSM1-29 | 2.0 | 2.0 | 2.0 | 1.6 | |
| ZFSM1-88 | 1.8 | 1.4 | 0.0 | 0.0 | |
| ZFSM1-92 | 1.6 | 2.0 | 1.8 | 1.8 | |
| ZFSM1-96 | 2.5 | 1.5 | 2.4 | 1.8 | |
| ZFSM1-129 | 2.5 | 1.5 | 2.4 | 1.8 | |
| ZFSM1-146 | 2.6 | 2.4 | 2.0 | 1.6 | |
| ZFSL1-82 | 0.0 | 0.0 | 2.0 | 1.8 | |
| ZFSL2-2 | 1.2 | 1.4 | 1.5 | 1.4 | |
菌株ZFSM1-146的16S rRNA基因序列经Blast比对,结果表明菌株ZFSM1-146与链霉菌属(Streptomyces)的菌株具有较高的相似性。利用MEGA 5.0,将菌株ZFSM1-146的16S rRNA基因序列与相似性相近的不同链霉菌菌株16S rRNA基因序列采用Neighbor-joining法构建系统发育树,结果见图 1。菌株ZFSM1-146与Streptomyces antibioticus NRRL B-1701在同一个分支,bootstrap值达到100%,相似性为100%。可初步确定菌株ZFSM1-146属于链霉菌属的抗生链霉菌。将菌株ZFSM1-146的16S rRNA基因序列上传至GenBank数据库,获得登录号为MW362453。
|
| 图 1 基于16S rRNA基因序列构建的菌株ZFSM1-146系统发育树 Figure 1 Phylogenetic tree of strain ZFSM1-146 based on 16S rRNA gene sequences 注:括号内表示16S rRNA基因序列在GenBank中的登录号;分支处数值表示Bootstrap值;标尺表示进化距离 Note: The GenBank accession number of 16S rRNA gene sequences are shown in the brackets; Numbers at branch nodes represent bootstrap value; Bar (0.01) represents sequence divergence |
|
|
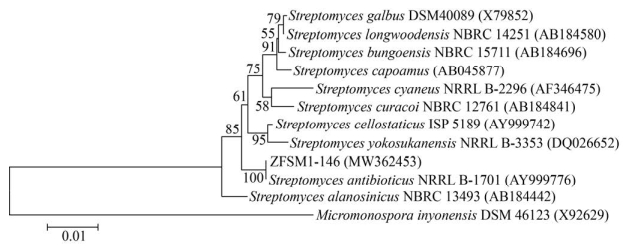
菌株ZFSM1-146发酵液经乙酸乙酯萃取、旋转蒸发仪蒸干后得到粗提物;通过硅胶色谱层析方法,粗提物以二氯甲烷-甲醇体系(100:0、99:1、98:2、97:3、96:4、95:5、94:6、93:7、90:10、85:15、0:100,体积比)梯度洗脱,得到13个硅胶组分Fr1−Fr13。Fr1−Fr13经HPLC和滤纸片法抑菌活性检测,结果显示Fr5−Fr13有抑菌活性(图 2A);比对Fr5−Fr13的HPLC结果,分别取少量Fr5和Fr7合并,利用HPLC检测条件分段收集各个峰的洗脱液,得到i、ii、iii和iv这4个组分(图 2C),再次通过滤纸片法检测抑菌活性,结果显示ii、iii和iv组分有抑菌活性(图 2B)。将ii、iii和iv组分分别通过HPLC检测,结果显示分别与粗提物中的化合物1、2和3一致。由此可以确定菌株ZFSM1-146产生的抗菌活性物质主要为化合物1−3 (图 2C)。其他具有抗菌活性的硅胶组分没有进一步分析是因为它们同样含有化合物1。通过HPLC纯化,得到化合物1−3的纯品,经计算,化合物1−3的产量分别为7.4±0.4、72.6±3.1和54.7±4.3 mg/L。
|
| 图 2 菌株ZFSM1-146发酵产物抗菌活性及HPLC分析 Figure 2 Antibacterial activity and HPLC analysis of strain ZFSM1-146 fermentation products 注:A:硅胶柱层析Fr1−Fr13的抗菌活性检测;B:Fr5+Fr7分段收集产物的抗菌活性检测;C:ZFSM1-146发酵产物及Fr5+Fr7 HPLC分析(检测波长为254 nm)。i:Fr5+Fr7中10-30 min收集的产物;ii:Fr5+Fr7中30.7−31.3 min收集的产物;iii:Fr5+Fr7中31.6-32.0 min收集的产物;iv:Fr5+Fr7中32.2-32.5 min收集的产物 Note: A: Antibacterial activity of Fr1−Fr13 of silica gel column chromatography; B: Antibacterial activity of different fractions from Fr5+Fr7; C: HPLC profiles of crude extract of ZFSM1-146 fermentation broth and Fr5+Fr7 (detection at 254 nm). i: 10−30 min HPLC fraction of Fr5+Fr7; ii: 30.7−31.3 min HPLC fraction of Fr5+Fr7; iii: 31.6−32.0 min HPLC fraction of Fr5+Fr7; iv: 32.2−32.5 min HPLC fraction of Fr5+Fr7 |
|
|
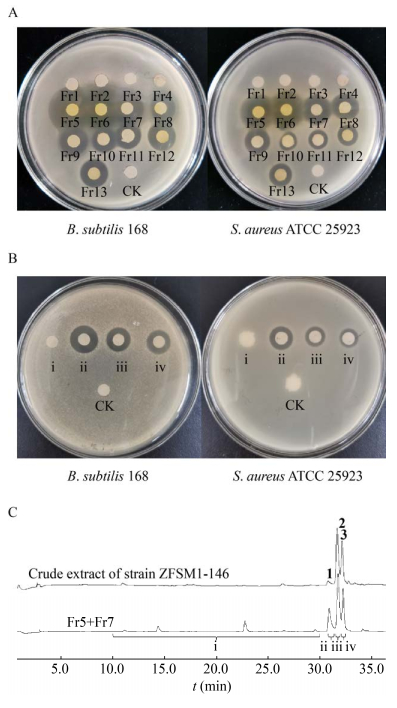
化合物1,红色粉末,通过HR-ESI-MS确定化合物1的m/z为1 271.630 8 [M+H]+,并计算出分子式为C62H86N12O17。将化合物1的碳谱数据通过微谱数据库中13C NMR库查询,该化合物与放线菌素类化合物相似度极高。进一步与文献中的放线菌素XOβ的核磁数据比对,发现化合物1与文献中报道的放线菌素XOβ的数据一致[18],因此确定化合物1为actinomycin XOβ。
化合物2,红色粉末,通过HR-ESI-MS确定化合物2的m/z为1 269.616 3 [M+H]+,并计算出分子式为C62H84N12O17,比化合物1少2个氢原子。比较化合物1与化合物2的碳谱数据,发现二者极为相似,但化合物2在低场多了一个信号C 209.04,推测化合物2的β-ring中脯氨酸C4上的羟基被氧原子取代,形成羰基,导致碳的化学位移升高。与文献[19]报道的核磁数据比对,化合物2的核磁数据与放线菌素X2的数据一致,可确定化合物2为actinomycin X2。
化合物3,红色粉末,通过HR-ESI-MS确定化合物3的m/z为1 255.638 0 [M+H]+,并计算出分子式为C62H86N12O16,比化合物1少1个氧原子;比化合物2少1个氧原子、多2个氢原子。比较化合物2与化合物3的碳谱数据,发现化合物3在高场多了一个信号C 23.01,推测化合物3的β-ring中脯氨酸C4上的羟基变成亚甲基,导致碳的化学位移下降。与文献报道的核磁数据比对[18],化合物3的核磁数据与放线菌素D的数据一致,可确定化合物3为actinomycin D。
|
| 图 3 化合物1−3的结构 Figure 3 The structures of compounds 1−3 |
|
|
| Position | 1 | 2 | 3 | ||||||
| 13C NMR (δC)a | 1H NMR (δH)b | 13C NMR (δC)c | 1H NMR (δH)d | 13C NMR (δC)a | 1H NMR (δH)b | ||||
| Chromophore | |||||||||
| 1 | 102.86, C | 101.86, C | 101.91, C | ||||||
| 2 | 147.01, C | 147.51, C | 147.72, C | ||||||
| 3 | 179.16, C | 179.21, C | 179.28, C | ||||||
| 4 | 113.67, C | 113.75, C | 113.67, C | ||||||
| 5 | 145.02, C | 146.06, C | 145.25, C | ||||||
| 6 | 140.73, C | 140.64, C | 140.63, C | ||||||
| 7 | 128.50, C | 128.03, C | 127.79, C | ||||||
| 8 | 130.46, CH | 7.36, d (7.6) | 130.47, CH | 7.36, d (7.7) | 130.42, CH | 7.36, d (7.4) | |||
| 9 | 126.18, CH | 7.64, d (7.8) | 126.31, CH | 7.61, d (7.7) | 125.90, CH | 7.62, d (7.2) | |||
| 10 | 131.36, C | 132.23, C | 132.73, C | ||||||
| 11 | 129.63, C | 129.29, C | 129.28, C | ||||||
| 12 | 146.05, C | 145.17, C | 146.03, C | ||||||
| 4-Me | 7.90, CH3 | 2.23, s | 7.93, CH3 | 2.23, s | 7.94, CH3 | 2.25, s | |||
| 7-Me | 15.26, CH3 | 2.54, s | 15.23, CH3 | 2.55, s | 15.21, CH3 | 2.55, s | |||
| 1-CO | 169.09, C | 166.65, C | 169.17, C | ||||||
| 10-CO | 166.79, C | 166.20, C | 166.67, C | ||||||
| -ring | |||||||||
| L-Thr | 1 | 168.84, C | 168.77, C | 168.67, C | |||||
| NH | 7.49, d (6.9) | 7.16, d (7.0) | 7.18, d (6.7) | ||||||
| 2 | 55.48, CH | 4.84, dd (7.0, 2.0) | 55.09, CH | 4.54, dd (5.7, 2.5) | 55.38, CH | 4.51, dd (6.7, 2.4) | |||
| 3 | 75.36, CH | 5.25, m | 74.88, CH | 5.15, m | 75.21, CH | 5.21, m | |||
| 4 | 17.79, CH3 | 1.29, d (6.1) | 17.31, CH3 | 1.13, d (6.5) | 17.92, CH3 | 1.25, d (5.0) | |||
| D-Val | 1 | 173.34, C | 173.69, C | 173.90, C | |||||
| NH | 8.20, d (4.8) | 7.67, d (4.9) | 8.00, d (5.0) | ||||||
| 2 | 58.91, CH | 3.59, m | 58.67, CH | 3.56, dd (9.2, 6.2) | 59.07, CH | 3.55, m | |||
| 3 | 31.44, CH | 2.12, m | 31.84, CH | 2.13, m | 31.97, CH | 2.15, m | |||
| 4 | 19.19, CH3 | 0.90, d (7.0) | 18.96 CH3 | 0.90, d (7.4) | 19.20 CH3 | 0.91, d (6.4) | |||
| 5 | 19.38, CH3 | 1.13, d (6.7) | 19.20, CH3 | 1.12, d (7.2) | 19.43, CH3 | 1.12, d (5.8) | |||
| L-Pro | 1 | 173.49, C | 173.28, C | 173.50, C | |||||
| 2 | 56.92, CH | 6.04, d (8.9) | 56.53, CH | 5.95, d (9.1) | 56.58, CH | 6.01, d (8.8) | |||
| 3 | 31.97, CH2 | 2.06, m | 31.14, CH2 | 1.84, m | 31.44, CH2 | 1.81, m | |||
| 2.69, m | 2.75, m | 2.67, m | |||||||
| 4 | 23.00, CH2 | 2.13, m | 23.12, CH2 | 2.07, m | 23.17, CH2 | 2.08, m | |||
| 2.18, m | 2.28, m | 2.29, m | |||||||
| 5 | 47.62, CH2 | 3.87, m | 47.58, CH2 | 3.74, m | 47.75, CH2 | 3.73, m | |||
| 3.90, m | 3.97, m | ||||||||
| Sar | 1 | 166.50, C | 166.43, C | 166.64, C | |||||
| N-Me | 35.10, CH3 | 2.94, s | 34.92, CH3 | 2.88, s | 35.12, CH3 | 2.88, s | |||
| 2 | 51.50, CH2 | 3.65, d (17.6) | 51.47, CH2 | 3.64, d (17.3) | 51.55, CH2 | 3.64, d (17.2) | |||
| 4.74, d (17.6) | 4.72, d (17.3) | 4.72, d (17.2) | |||||||
| Meval | 1 | 167.70, C | 167.64, C | 167.81, C | |||||
| N-Me | 39.42, CH3 | 2.95, s | 39.54, CH3 | 2.92, s | 39.42, CH3 | 2.90, s | |||
| 2 | 71.37, CH | 2.69, m | 71.38, CH | 2.65, m | 71.55, CH | 2.67, m | |||
| 3 | 27.26, CH | 2.69, m | 27.06, CH | 2.65, m | 27.07, CH | 2.67, m | |||
| 4 | 19.32, CH3 | 0.75, d (2.9) | 19.35, CH3 | 0.73, d (5.7) | 19.38, CH3 | 0.74, d (5.0) | |||
| 5 | 21.75, CH3 | 0.96, d (6.0) | 21.75, CH3 | 0.95, d (5.6) | 21.84, CH3 | 0.96, d (6.2) | |||
| β-ring | |||||||||
| L-Thr | 1 | 167.70, C | 169.06, C | 167.70, C | |||||
| NH | 7.44, d (6.3) | 7.67, d (4.9) | 7.75, d (5.3) | ||||||
| 2 | 54.87, CH | 4.51, dd (6.4, 2.6) | 54.89, CH | 4.48, dd (6.0, 2.5) | 54.99, CH | 4.62, dd (6.4, 2.1) | |||
| 3 | 74.75, CH | 5.25, m | 74.79, CH | 5.24, m | 75.10, CH | 5.17, m | |||
| 4 | 17.68, CH3 | 1.25, d (6.1) | 17.86, CH3 | 1.26, d (6.1) | 17.50, CH3 | 1.25, d (5.0) | |||
| D-Val | 1 | 173.03, C | 174.17, C | 173.47, C | |||||
| NH | 7.93, d (6.1) | 8.20, d (5.8) | 8.18, d (5.5) | ||||||
| 2 | 58.07, CH | 3.56, m | 57.29, CH | 3.70, dd (9.7, 6.0) | 58.89, CH | 3.55, m | |||
| 3 | 31.23, CH | 2.12, m | 31.99, CH | 2.28, m | 31.69, CH | 2.23, m | |||
| 4 | 19.02, CH3 | 0.88, d (6.9) | 19.06, CH3 | 0.89, d (7.4) | 19.12, CH3 | 0.88, d (6.2) | |||
| 5 | 19.15, CH3 | 1.11, d (6.7) | 19.23, CH3 | 1.16, d (7.0) | 19.24, CH3 | 1.12, d (5.8) | |||
| L-Pro | 1 | 173.24, C | 172.84, C | 173.41, C | |||||
| 2 | 56.57, CH | 5.98, d (9.0) | 54.40, CH | 6.56, d (10.4) | 56.40, CH | 5.95, d (8.7) | |||
| 3 | 39.69, CH2 | 2.13, m | 42.05, CH2 | 2.33, d (17.5) | 31.09, CH2 | 1.86, m | |||
| 2.16, m | 3.84, dd (17.8, 11.1) | 2.97, m | |||||||
| 4 | 69.96, CH | 4.15, d (17.1) | 209.04, C | 23.01, CH2 | 2.11, m | ||||
| 2.29, m | |||||||||
| 5 | 54.77, CH2 | 3.73, m | 53.00, CH2 | 3.97, d (19.3) | 47.50, CH2 | 3.73, m | |||
| 3.94, m | 4.56, m | 3.84, m | |||||||
| Sar | 1 | 166.20, C | 166.04, C | 166.42, C | |||||
| N-Me | 35.16, CH3 | 2.88, s | 35.06, CH3 | 2.89, s | 35.05, CH3 | 2.88, s | |||
| 2 | 51.37, CH2 | 3.63, d (17.6) | 51.44, CH2 | 3.64, d (17.4) | 51.52, CH2 | 3.61, d (17.6) | |||
| 4.58, d (17.6) | 4.60, d (17.4) | 4.79, d (17.6) | |||||||
| Meval | 1 | 166.50, C | 167.67, C | 166.51, C | |||||
| N-Me | 39.32, CH3 | 2.88, s | 39.37, CH3 | 2.93, s | 39.31, CH3 | 2.93, s | |||
| 2 | 71.36, CH | 2.69, m | 71.57, CH | 2.70, m | 71.37, CH | 2.63 m | |||
| 3 | 27.14, CH | 2.69, m | 27.10, CH | 2.70, m | 27.05, CH | 2.63, m | |||
| 4 | 19.16, CH3 | 0.74, d (2.7) | 19.39, CH3 | 0.74, d (6.2) | 19.24, CH3 | 0.74, d (5.0) | |||
| 5 | 21.72, CH3 | 0.95, d (5.8) | 21.86, CH3 | 0.97, d (5.9) | 21.73, CH3 | 0.94, d (6.0) | |||
| 注:a:数据采集自125 MHz;b:采集自500 MHz;c:采集自150 MHz;d:采集自600 MHz。碳谱数据中CH3:伯碳原子;CH2:仲碳原子;CH:叔碳原子;C:季碳原子。氢谱数据中s:单峰;d:双峰;dd:双二重峰;m:多重峰 Note: a: Data were measured at 125 MHz; b: Data were measured at 500 MHz; c: Data were measured at 150 MHz; d: Data were measured at 600 MHz. CH3: primary carbon; CH2: secondary carbon; CH: tertiary carbon; C: quaternary carbon in 13C NMR data. s: singlet; d: doublet; dd: double-doublet; m: multplet in 1H NMR |
|||||||||
表 3是3种放线菌素对不同细菌的MIC值,放线菌素XOβ、X2和D对B. subtilis 168、S. aureus ATCC 25923和MRSA ATCC 43300都具有明显的抑菌活性;放线菌素X2和D的抑菌效果明显优于放线菌素XOβ,而且放线菌素X2的抑菌活性最强。上述结果显示放线菌素X2和D在治疗由B. subtilis和S. aureus引起的传染病(尤其是MRSA引起的肺炎)方面具有潜在用途[17]。
| Strains | Actinomycin XOβ | Actinomycin X2 | ActinomycinD |
| B. subtilis 168 | 0.4 | 0.006 | 0.012 5 |
| S. aureus ATCC 25923 | 0.4 | 0.025 | 0.050 0 |
| MRSA ATCC 43300 | 0.4 | 0.025 | 0.050 0 |
| E. coli ATCC 25922 | 80.0 | 40.000 | 40.000 0 |
通过MⅠ−MⅣ这4种培养基,优化链霉菌ZFSM1-146放线菌素的产量,结果表明MⅢ培养基放线菌素的产量最高,XOβ、X2和D这3个组分分别达到8.4±0.8、136.0±5.8和39.4±1.9 mg/L (表 4),其中放线菌素X2的产量达到136.0±5.8 mg/L,约达到优化前的2倍。王聪等[20]对海洋来源放线菌S. parvulus OUCMDZ-2554产放线菌素D的发酵条件进行优化,通过单因素和正交试验,确定发酵培养基为:K2HPO4 1.5 g,MgSO4 0.5 g,酵母浸膏5 g,可溶性淀粉22.5 g,陈海水1 L,起始pH 7.5;最佳培养条件:种龄4 d,装液量150/500 mL,盐度3%,发酵时间12 d。优化后放线菌素D的产量是优化前的3.6倍,达到364 mg/L。在此基础上,Wang等[17]对能同时产放线菌素XOβ、X2和D的S. heliomycini WH1的发酵条件进行优化,他们先通过单因素试验确定培养基的盐浓度和pH值对该菌株生产放线菌素产量有一定的影响;其次选择大豆粉培养基(2%大豆粉,pH 8.0)和MⅠ−MⅣ培养基发酵,结果显示菌株用MⅡ培养基发酵生产放线菌素XOβ、X2和D的产量最高;最后选取MⅡ培养基进行探索,在盐浓度为5%、最佳pH 8.5时,放线菌素XOβ、X2和D的产量分别达到107.6±4.2、283.4±75.3、458.0±76.3 mg/L。Wang等的研究成果[17, 20]给本研究放线菌素产量优化的培养基选择提供了参考依据。MⅠ−MⅣ这4种培养基的区别主要在于碳源和氮源的组合不同,通过MⅠ−MⅣ这4种发酵培养基进行放线菌素产量优化,结果表明,菌株ZFSM1-146生长于以可溶性淀粉或葡萄糖作为碳源、酵母粉作为氮源的MⅢ、MⅣ培养基时,放线菌素产量明显高于仅以酵母粉作为碳源和氮源的MⅠ培养基的放线菌素产量;此外,可溶性淀粉作为碳源的MⅢ培养基的放线菌素产量高于葡萄糖作为碳源的MⅣ培养基的放线菌素产量。这说明在常见的微生物生长所需碳源中,链霉菌ZFSM1-146发酵时利用可溶性淀粉作为碳源则放线菌素的产量最高。
| Medium | 1 | 2 | 3 |
| R5 | 7.4±0.4 | 72.6±3.1 | 54.7±4.3 |
| MⅠ | 1.5±0.3 | 4.5±0.5 | 20.1±1.1 |
| MⅡ | 3.5±0.2 | 43.8±1.7 | 14.6±1.6 |
| MⅢ | 8.4±0.8 | 136.0±5.8 | 39.4±1.9 |
| MⅣ | 7.4±0.4 | 68.0±2.3 | 26.8±2.2 |
本研究仅对链霉菌ZFSM1-146通过尝试不同发酵培养基进行放线菌素产量的初步优化,后期可以采用MⅡ培养基针对种龄、盐浓度、pH值和发酵时间等因素探索最佳发酵条件,进一步提高放线菌素产量。链霉菌ZFSM1-146在放线菌素产量提升方面具有较大的潜力,为后续通过不同培养条件等因素优化放线菌素产量提供了宝贵的菌种资源。
3 讨论与结论放线菌素是一类环肽化合物,由2个环五肽内酯组成,分别称为α-ring和β-ring,通过酰胺键连接到一个杂三环发色团,一般呈现红色或橘红色[21]。大多数放线菌素都具有抗菌、抗病毒和抗肿瘤活性[22]。1940年,Waksman等[23]从土壤来源的抗生链霉菌中分离出第一种放线菌素。截至目前,从链霉菌属分离鉴定的天然放线菌素类化合物已经达到40多种,主要包括放线菌素D、放线菌素C、放线菌素X、放线菌素Z、放线菌素Y等[24]。放线菌素D已经被广泛研究并应用到临床上,但由于其强烈的细胞毒性,仅限于恶性肿瘤的治疗。由于化学合成方法步骤长、产量低、成本高,放线菌素D主要通过微生物发酵获得。放线菌素的生物合成主要包括3个阶段:(1) 通过犬尿氨酸途径合成3-羟基-4-甲基邻氨苯甲酸(4-MHA);(2) 在非核糖体酶的作用下合成3-羟基-4-甲基邻氨苯甲酸-环五肽内酯;(3) 2个3-羟基-4-甲基邻氨苯甲酸-环五肽内酯缩合成吩恶嗪酮发色团[25]。近年来,研究者期望通过化学全合成研究构效关系,得到抗肿瘤活性强和细胞毒性低的放线菌素D类似物,使其在临床上的广泛应用成为可能。张邦治等[26]通过保留环肽2位D-Val不变、环肽5位的MeVal替换成氨基酸侧链延长的D-Me-Leu和Me-Ile均能显著提高类似物的抗肿瘤活性。倪京满等[27]通过合成放线菌素的类似物2, 2′-双苯丙氨酸放线菌素D,在相同毒性情况下,类似物的抗肿瘤活性比放线菌素D有一定的提高。
本研究从深圳地区红树林土壤来源的放线菌出发,通过初筛和复筛筛选出具有抗菌活性的放线菌,最终选择一株放线菌菌株ZFSM1-146挖掘其产生的抗菌活性物质。基于16S rRNA基因序列及构建的系统发育树分析,可初步确定菌株ZFSM1-146属于链霉菌属的抗生链霉菌。采用有机溶剂萃取法从ZFSM1-146发酵液中萃取出抗菌活性物质,通过活性追踪法确定化合物1−3为抗菌活性物质;经HR-ESI-MS和NMR鉴定出抗菌活性物质化合物1−3都属于放线菌素类化合物,分别是放线菌素XOβ、X2和D,产量分别达到7.4±0.4、72.6±3.1和54.7±4.3 mg/L。通过培养基优化,MⅢ培养基放线菌素的产量最高,分别达到8.4±0.8、136.0±5.8和39.4±1.9 mg/L,其中放线菌素X2的产量为136.0±5.8 mg/L,约达到优化前的2倍;相较于放线菌素XOβ和D,放线菌素X2的抑菌活性最强。放线菌素X2被认为具有较放线菌素D更强的抗肿瘤活性,人们对放线菌素X2的抗肿瘤作用正进行深入的研究[28]。本研究为研究放线菌素X2抗肿瘤作用提供了物质基础,为后续通过分子遗传手段对放线菌素XOβ、X2和D进行结构修饰及产量优化以得到新颖的放线菌素和抗肿瘤活性强、细胞毒性低放线菌素提供了宝贵的菌种资源,对后续放线菌素类似药的开发具有重要意义。
| [1] |
Newman DJ, Cragg GM. Natural products as sources of new drugs from 1981 to 2014[J]. Journal of Natural Products, 2016, 79(3): 629-661. DOI:10.1021/acs.jnatprod.5b01055 |
| [2] |
Gullo VP, McAlpine J, Lam KS, Baker D, Petersen F. Drug discovery from natural products[J]. Journal of Industrial Microbiology & Biotechnology, 2006, 33(7): 523-531. |
| [3] |
Ma QQ, Ding WJ, Chen Z, Ma ZJ. Bisamides and rhamnosides from mangrove actinomycete Streptomyces sp. SZ-A15[J]. Natural Product Research, 2018, 32(7): 761-766. DOI:10.1080/14786419.2017.1315578 |
| [4] |
Tan LTH, Chan KG, Pusparajah P, Yin WF, Khan TM, Lee LH, Goh BH. Mangrove derived Streptomyces sp. MUM265 as a potential source of antioxidant and anticolon-cancer agents[J]. BMC Microbiology, 2019, 19(1): 1-16. DOI:10.1186/s12866-019-1409-7 |
| [5] |
Braddock AA, Theodorakis EA. Marine spirotetronates: biosynthetic edifices that inspire drug discovery[J]. Marine Drugs, 2019, 17(4): 232. DOI:10.3390/md17040232 |
| [6] |
Zeng YX, Chen B, Zou Y, Zheng TL. Polar microorganisms, a potential source for new natural medicines: a review[J]. Acta Microbiologica Sinica, 2008, 48(5): 695-700. (in Chinese) 曾胤新, 陈波, 邹扬, 郑天凌. 极地微生物——新天然药物的潜在来源[J]. 微生物学报, 2008, 48(5): 695-700. DOI:10.3321/j.issn:0001-6209.2008.05.022 |
| [7] |
Rateb ME, Ebel R, Jaspars M. Natural product diversity of actinobacteria in the Atacama Desert[J]. Antonie Van Leeuwenhoek, 2018, 111(8): 1467-1477. DOI:10.1007/s10482-018-1030-z |
| [8] |
Saxena R, Dhakan DB, Mittal P, Waiker P, Chowdhury A, Ghatak A, Sharma VK. Metagenomic analysis of hot springs in central India reveals hydrocarbon degrading thermophiles and pathways essential for survival in extreme environments[J]. Frontiers in Microbiology, 2016, 7: 2123. |
| [9] |
Ye JJ, Zheng HY, Wu Y, Jiang LX, Chen JH, Huang SS, Huang DL. Diversity and antimicrobial activity of actinobacteria isolated from mangrove rhizosphere soil in the Maowei Sea of Guangxi[J]. Journal of Pathogen Biology, 2018, 13(11): 1221-1226, 1231. (in Chinese) 叶景静, 郑红芸, 吴越, 蒋莲秀, 陈建宏, 黄庶识, 黄大林. 广西茅尾海红树林植物根际土壤放线菌多样性及抗菌活性研究[J]. 中国病原生物学杂志, 2018, 13(11): 1221-1226, 1231. |
| [10] |
Palazzotto E, Tong YJ, Lee SY, Weber T. Synthetic biology and metabolic engineering of actinomycetes for natural product discovery[J]. Biotechnology Advances, 2019, 37(6): 107366. |
| [11] |
Mahajan GB, Balachandran L. Antibacterial agents from actinomycetes: a review[J]. Frontiers in Bioscience: Elite Edition, 2012, 4: 240-253. |
| [12] |
Sayed AM, Hassan MHA, Alhadrami HA, Hassan HM, Goodfellow M, Rateb ME. Extreme environments: Microbiology leading to specialized metabolites[J]. Journal of Applied Microbiology, 2020, 128(3): 630-657. DOI:10.1111/jam.14386 |
| [13] |
Zhang M. Screening and identification of marine Actinomyces and study on of its antimicrobial active substances[D]. Shanghai: Master's Thesis of Shanghai Ocean University, 2017 (in Chinese) 张孟. 海洋抑菌放线菌的筛选、鉴定及其抑菌活性物质研究[D]. 上海: 上海海洋大学硕士学位论文, 2017 |
| [14] |
Lin ZT, Ye ZJ, Zhuang LP, Xu L, Zhang G, Qiang H. Isolation and structure identification of the antimicrobial compounds from a marine actinomycetes strains[J]. Acta Agriculturae Universitatis Jiangxiensis, 2017, 39(3): 559-566. (in Chinese) 林真亭, 叶子坚, 庄玲萍, 许莉, 张岗, 强华. 一株海洋放线菌抗菌活性物质的分离与结构解析[J]. 江西农业大学学报, 2017, 39(3): 559-566. |
| [15] |
Farris MH, Olson JB. Detection of Actinobacteria cultivated from environmental samples reveals bias in universal primers[J]. Letters in Applied Microbiology, 2007, 45(4): 376-381. DOI:10.1111/j.1472-765X.2007.02198.x |
| [16] |
Appendino G, Gibbons S, Giana A, Pagani A, Grassi G, Stavri M, Smith E, Rahman MM. Antibacterial cannabinoids from Cannabis sativa: a structure-activity study[J]. Journal of Natural Products, 2008, 71(8): 1427-1430. DOI:10.1021/np8002673 |
| [17] |
Wang DY, Wang C, Gui PY, Liu HS, Khalaf SMH, Elsayed EA, Wadaan MAM, Hozzein WN, Zhu WM. Identification, bioactivity, and productivity of actinomycins from the marine-derived Streptomyces heliomycini[J]. Frontiers in Microbiology, 2017, 8: 1147. DOI:10.3389/fmicb.2017.01147 |
| [18] |
Song XQ, Jiang X, Sun JB, Zhang CY, Zhang Y, Lu LC, Ju JH. Antibacterial secondary metabolites produced by mangrove-derived actinomycete stremptmeces Costaricanus SCSIO ZS0073[J]. Natural Product Research and Development, 2017, 29(3): 410-414. (in Chinese) 宋现芹, 蒋鑫, 孙建彬, 张春燕, 张云, 卢来春, 鞠建华. 红树林放线菌Stremptmeces costaricanus SCSIO ZS0073抗菌活性次级代谢产物的研究[J]. 天然产物研究与开发, 2017, 29(3): 410-414. |
| [19] |
Matsui T, Tanaka J, Namihira T, Shinzato N. Antibiotics production by an actinomycete isolated from the termite gut[J]. Journal of Basic Microbiology, 2012, 52(6): 731-735. DOI:10.1002/jobm.201100500 |
| [20] |
Wang C, Liu PP, Wang Y, Sun KL, Jia HJ, Zhu WM. Optimization of fermentation conditions for production of actinomycin D by marine-derived Streptomyces parvulus OUCMDZ-2554[J]. Chinese Journal of Marine Drugs, 2014, 33(3): 34-42. (in Chinese) 王聪, 刘培培, 王乂, 孙坤来, 贾海健, 朱伟明. 海洋来源放线菌Streptomyces parvulus OUCMDZ-2554产放线菌素D的发酵条件优化[J]. 中国海洋药物, 2014, 33(3): 34-42. |
| [21] |
Cai WL, Wang XC, Elshahawi SI, Ponomareva LV, Liu XD, McErlean MR, Cui Z, Arlinghaus AL, Thorson JS, Van Lanen SG. Antibacterial and cytotoxic actinomycins Y6-Y9 and zp from Streptomyces sp. strain gö-GS12[J]. Journal of Natural Products, 2016, 79(10): 2731-2739. |
| [22] |
Chen CX, Song FH, Wang Q, Abdel-Mageed WM, Guo H, Fu CZ, Hou WY, Dai HQ, Liu XT, Yang N, et al. A marine-derived Streptomyces sp. MS449 produces high yield of actinomycin X2 and actinomycin D with potent anti-tuberculosis activity[J]. Applied Microbiology and Biotechnology, 2012, 95(4): 919-927. DOI:10.1007/s00253-012-4079-z |
| [23] |
Waksman SA, Woodruff HB. Bacteriostatic and bactericidal substances produced by a soil Actinomyces[J]. Experimental Biology and Medicine, 1940, 45(2): 609-614. |
| [24] |
Liu MC, Jia YX, Xie YC, Zhang CY, Ma JY, Sun CL, Ju JH. Identification of the actinomycin D biosynthetic pathway from marine-derived Streptomyces costaricanus SCSIO ZS0073[J]. Marine Drugs, 2019, 17(4): 240-252. |
| [25] |
Yuan W, Jiao WH, Wang Y, Chen B, Ye BP. Progress on biosynthesis of actinomycins[J]. Amino Acids & Biotic Resources, 2014, 36(1): 1-7. (in Chinese) 袁薇, 焦伟华, 王颖, 陈彪, 叶波平. 放线菌素生物合成研究进展[J]. 氨基酸和生物资源, 2014, 36(1): 1-7. |
| [26] |
Zhang BZ, Wang KR, Wang ZZ, Li XL, Wang XL, Ni JM. Design, synthesis and in vitro antitumor activity of novel actinomycin D analogs[J]. Chemical Journal of Chinese Universities, 2010, 31(7): 1346-1352. (in Chinese) 张邦治, 王凯荣, 王则周, 李欣檑, 王小丽, 倪京满. 放线菌素D新类似物的设计、合成与体外抗肿瘤活性[J]. 高等学校化学学报, 2010, 31(7): 1346-1352. |
| [27] |
Ni JM, Wang R, Jia ZP, Pan XF, Hu XY. Studies on the synthesis and bioactivity of 5, 5'-Val2-AMD and 2, 2'- Phe2-AMD[J]. Chemical Journal of Chinese Universities, 1998, 19(2): 243-245. (in Chinese) 倪京满, 王锐, 贾正平, 潘鑫复, 胡晓愚. 抗癌药物放线菌素D类似物的全合成及生物活性研究[J]. 高等学校化学学报, 1998, 19(2): 243-245. |
| [28] |
Sun ZY, Zhang MM, Qin DH, Yang XP. Study on actinomycin X2, a secondary metabolite produced by actinomycete Z802031[J]. Journal of Capital Normal University (Natural Science Edition), 2011, 32(3): 18-22. (in Chinese) 孙肇暘, 张明明, 秦德华, 杨秀萍. 放线菌Z802031次级代谢产物放线菌素X2的研究[J]. 首都师范大学学报: 自然科学版, 2011, 32(3): 18-22. |
 2021, Vol. 48
2021, Vol. 48