扩展功能
文章信息
- 杨思芹, 王国华, 谢水波, 陈翔
- YANG Siqin, WANG Guohua, XIE Shuibo, CHEN Xiang
- 磷酸盐生物矿化处理低浓度含铀废水的研究进展
- Research advances of wastewater containing low concentration uranium by phosphate biomineralization
- 微生物学通报, 2021, 48(6): 2222-2231
- Microbiology China, 2021, 48(6): 2222-2231
- DOI: 10.13344/j.microbiol.china.201047
-
文章历史
- 收稿日期: 2020-11-05
- 接受日期: 2021-01-12
- 网络首发日期: 2021-03-26
2. 南华大学污染控制与资源化技术湖南省重点实验室 湖南 衡阳 421001
2. Hunan Provincial Key Laboratory of Pollution Control and Resources Technology, University of South China, Hengyang, Hunan 421001, China
随着我国核工业事业的飞速发展,含铀废水污染也受到了越来越多的关注。由于开采技术等原因产生的酸性含铀废水会通过食物链等途径对人体造成潜在危害,所以研究高效处理低浓度含铀废水的方法势在必行。
目前处理低浓度含铀废水的方法主要有化学沉淀法[1]、材料吸附法[2]等,这些方法应用较广,但存在成本较高、工艺复杂、废液产量大和固液分离困难的缺点[3]。酸性环境下,地下水体条件比较复杂,一定程度上限制了铀在地下的流动性,用传统处理技术处理废水中的铀极其困难。生物修复技术成本低廉且操作相对简单,其中的植物处理法[4]所需周期较长,所以不易实现。微生物处理含铀废水主要有生物吸附、生物还原、生物累积、生物矿化,其中生物矿化具有不易产生二次污染且矿化产物稳定性较好的特点,受到学者的广泛关注,但是关于生物矿化的综述还很少,本文从生物矿化简介、生物矿化的影响因素以及强化生物矿化过程几个方面,对生物矿化法处理低浓度含铀废水的修复研究进展进行综述,以期为后续生物矿化机制的进一步研究提供参考。
1 生物矿化简介生物矿化[5]是微生物代谢过程中产生的配位体(如SO42-、PO43-、CO32-和OH-等)与溶液中的金属离子形成沉淀的过程。最初是Macaskie等用Citrobacter菌株对Cd和Zn进行生物积累时发现[6],随后在含铀土壤中也发现被铀磷酸盐矿物覆盖的微生物细胞[7]。80%的微生物如芽孢杆菌属[8]、土著苏云金芽孢杆菌[5]、Idiomarina loihiensis MAH1菌株[9]等以及各种真菌可以通过体内磷酸酶活性将大分子含磷类化合物(植酸、磷灰石)分解为正磷酸盐,与环境中的铀结合生成不易被氧化的磷酸盐铀酰沉淀[10]。生物矿化中常用的微生物如表 1所示。
| 微生物名称 Organism |
磷酸酶属性 Phosphatase property |
菌属性 Bacteria property |
生长环境 Growth condition |
参考文献 References |
| Serratia strain | Alkaline phosphatase and acid phosphatase | Gram-negative | 10-36 ℃/pH 5.0-9.0; facultative anaerobic | [11] |
| Pseudomonas fluorescens | Gram-negative | 25-30 ℃/pH 7.0±0.2; chemoheterotrophic bacteria | [12] | |
| Bacillus | Acid phosphatase | Gram-positive | 37 ℃/pH 7.0 | [11] |
| Arthrobacter | Gram-positive | 20-35 ℃ | [13] | |
| Deinococcus radiodurans | Acid phosphatase | 30 ℃ | [14] | |
| Citrobacter sp. | Acid phosphatase | Gram-negative | Facultative anaerobic | [13] |
| E. coli | Acid phosphatase | Gram-negative | 37 ℃/pH 7.0; heterotrophic facultative anaerobe | [15] |
| Idiomarina loihiensis | 30 ℃/pH 2.0-7.0; aerobic bacteria | [9] | ||
| Pseudomonas sp. | Gram-negative | 30 ℃/pH 4.0-7.0; aerobic bacteria | [16] | |
| Rhodanobacter | 26 ℃/pH 5.0-6.0 | [17] | ||
| Acinetobacter sp. | 32±2 ℃/pH 7.0±0.2 | [8] | ||
| Saccharomyces cerevisiae | Fungus | 30 ℃ | [18] | |
| Aspergillus tubingensis | 30±2 ℃/pH 7.0±0.2 | [19] |
基于表达条件的差异,微生物体内的磷酸酶可以分为酸性磷酸酶和碱性磷酸酶。酸性环境下,柠檬酸细菌属(Citrobacter sp.)、耐辐射球菌(Deinococcus radiodurans)等微生物的磷酸酶活性最佳,酶解有机磷酸盐效果好。Martinez等[20]发现从铀污染土壤中分离的具有酸性磷酸酶活性的芽孢杆菌和拉恩氏菌可以与铀酰离子形成铀酰磷酸盐矿物。罗氏假单胞菌被发现具有碱性磷酸酶基因[21]。沙雷氏菌属菌株(Serratia Strain)[11]等微生物则表达出2种性质的磷酸酶,这些微生物在pH 5.0-9.0之间可以水解有机磷基质,释放出不同数量的磷酸盐对铀产生生物矿化作用,所以生物矿化可以在较宽的pH范围内发挥作用,对环境的酸碱度要求较低。
根据磷酸酶的特异性,有人利用基因工程的方法向不含磷酸酶的微生物中导入不同性质磷酸酶基因,使其具有在特异环境下分解有机磷酸盐的能力。Kulkarni等[22]向Deinococcus radiodurans和Escherichia coli这2种菌属添加PhoK和PhoN,发现这些菌属在缺乏碳酸盐的中性环境(GC1)或碳酸盐充足的碱性环境(GC2)下,都具有降解β-甘油磷酸盐的能力,与铀生成磷酸铀酰沉淀。生物矿化中常用的微生物如表 1所示。
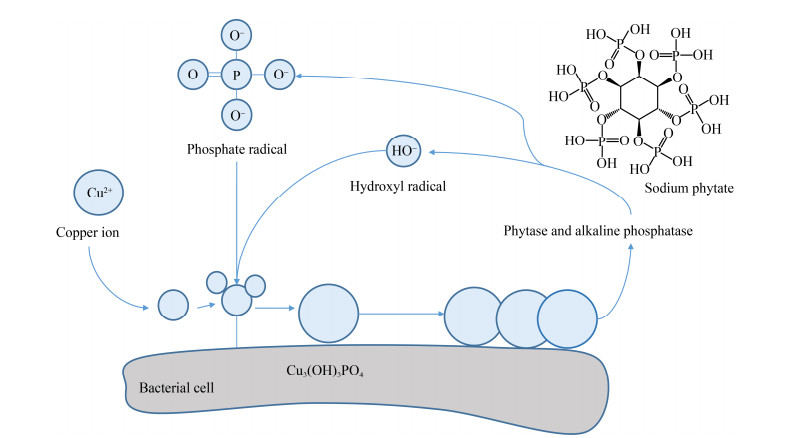
1.2 生物矿化作用机理微生物介导下,放射性核素能够以碳酸盐、磷酸盐和氢氧化物等形式在细胞表面形成沉淀[23-24]。微生物对铀的磷酸盐生物矿化可能是通过以下2种途径(图 1):(1) 从外界添加的有机磷酸盐供体利用磷酸酶活性释放无机磷酸盐来沉淀铀[25-26];(2) 微生物细胞内的多磷酸盐颗粒水解或降解,从而磷酸盐释放或外排与铀结合形成磷酸铀酰沉淀,所以在细胞表面及溶液中都发现了磷酸铀酰沉淀的存在[22]。Zhao等[27]发现Rahnella sp. LRP3可以通过自身磷酸酶降解植酸钠释放出磷酸盐,并与污染废水中的Cu在细胞表面生成Cu3(OH)3PO4晶体,如图 2所示。

|
| 图 1 微生物磷酸盐矿化U(Ⅵ)在细胞膜上的矿化机理图 Figure 1 Mineralization mechanism of microbial phosphate mineralization U(Ⅵ) on the cell membrane |
|
|
|
| 图 2 微生物降解植酸对Cu的生物矿化机理图 Figure 2 Biomineralization mechanism of Cu by microbial degradation phytic acid |
|
|
相较于其他方法,生物矿化处理低浓度含铀废水有着其独特的优势:适用范围广,对氧气和pH等条件的要求低;生物矿化产物比生物还原产物更加稳定,主要有铀酰磷酸盐矿物(Ca[UO2]2[PO4]2·2– 6H2O、Ca[UO2]2[PO4]2·10–12H2O)[12]等。
生物矿化存在的一些技术瓶颈使该技术在进一步研究与应用中面临巨大挑战:(1) 生成的沉淀可能会覆盖在微生物表面形成致密的沉淀,影响微生物的代谢活性及进一步的矿化沉淀。改变生物矿化条件可使磷酸铀酰沉淀自行脱落,但具体条件有待研究,Kulkarni等[22]发现在缺乏碳酸盐的中性环境和碳酸盐充足的碱性环境下,微生物都与U生成相同的磷酸铀酰沉淀,但TEM显示前者环境中的沉淀主要存在于微生物表面,而后者环境中产物主要为胞外磷酸盐沉淀。(2) 有机磷酸盐(甘油磷酸盐)价格较高难以大规模应用的问题。王聂颖等[19]利用廉价植酸代替甘油磷酸盐矿化含铀废水,发现植酸可以被Aspergillus tubingensis有效降解,而且磷酸矿化95.2%的铀后产生氢铀云母和变钠铀云母矿物。(3) 由于微生物的局限性,磷酸盐生物矿化难以应用于如高温、高压等较极端工况。
2 生物矿化过程的影响因素 2.1 微生物群落不同菌属的耐铀性不同,当环境中的铀浓度过高时,被污染地区的微生物群落多样性和功能会发生改变[28],铀的放射性也会导致微生物磷酸酶活性发生改变[29],最终影响铀的生物矿化效果。铀污染地区的微生物群落活性以氧化还原活性和生物吸附活性为主,而且菌种分布与未污染地区不同。Suriya等[30]发现在未污染地区的微生物群落以Bacteroidetes (27.7%)和Firmicutes (25.9%)为主,而在被污染地区的沉积物样品中以Proteobacteria (47.5%)为主,其次是Bacteroidetes (22.4%)和Firmicutes (14.6%)。利用土著菌群修复铀污染水体前需要对土著菌群结构进行分析,确定存在生物矿化相关微生物。
铀污染环境中,微生物各菌种存在竞争机制,导致各菌种的数量随时间推移而变化,有利菌种所占比例增加[31],所以微生物群落在生物矿化的不同阶段菌落结构不同[32]。不同的有机磷酸盐影响土著菌群群落的丰度[33],Crenarchaeota、Euryarchaeota、Bacteroidetes和Proteobacteria这几类菌属在有机磷(G3P和G2P)存在时,对铀的富集量最大,相对丰度增加了2倍甚至更多,分别占古菌总数的9%、50%和3%、17%[33]。
监测微生物指标对铀污染地区污染程度评价以及设计合适的生物修复策略具有极其重要的意义[9, 34]。关于放射性核素污染环境中微生物群落的宏基因组学研究表明微生物在铀污染地区中发挥极大的作用[35],而且其适应性极强[36]。
2.2 溶液化学参数 2.2.1 有机磷酸盐的种类正磷酸盐能够快速固定含铀废水中的铀离子,而且生成的磷酸铀酰离子沉淀不易溶于水,但直接注入无机磷酸盐会堵塞注入口[37],影响后续效果,所以有些学者提出注入有机磷酸盐再利用土著微生物的酶活性可以解决此问题。目前研究中使用最多的有机磷酸盐有甘油磷酸盐和植酸。
甘油磷酸钠可促进U(Ⅵ)转换成U(Ⅳ),生成稳定、不易氧化的产物。黄超等[38]证实β-甘油磷酸盐作为有机磷源的可靠性。美国能源部橡树岭沉积物中分离出来的Bacillus sp.和Rahnella sp.能从甘油-3-磷酸(G3P)中释放无机磷酸盐,矿化率分别达到73%和95%,生成产物为稳定的Ca(UO2)2(PO4)2[13]。甘油磷酸盐对铀的生物矿化效果明显,但其价格较昂贵,经济效益低,所以有些研究者将目光放到了自然界中普遍存在的植酸上。
植酸可被微生物分解成肌醇和正磷酸盐[39],与铀结合生成HUO2PO4沉淀[40]。1 mol植酸理论上可被分解产生6 mol正磷酸盐,提供更多的正磷酸盐配位体与U(Ⅵ)结合。朱定国等[41]与范黎锋等[42]利用植酸处理黑曲霉与大肠杆菌,实验发现,经植酸处理之后,黑曲霉和大肠杆菌对U(Ⅵ)的吸附效果显著增强,最终形成的络合产物沉积在微生物表面。随着研究的深入,我们发现植酸盐生产成本较低,经济可行性高;覆盖范围广,可达到污染区域最大覆盖率,使原位生物修复存在可能性;植酸盐及肌醇磷酸酯中间体在发生水解前可跟随含铀废水进行迁移,从而扩大生物修复范围[43];单靠微生物完全降解植酸所需时间较长,降解较缓慢。未来研究将着重于如何提升植酸盐降解速率,加快磷酸盐生物矿化速率及提升生物矿化产物的稳定性。
微生物种类不同或有机磷酸盐种类不同,都会导致生物矿化生成不同的最终产物。目前研究关于微生物分解有机磷酸盐生物矿化U(Ⅵ)的产物汇总如表 2所示。
| 微生物 Organism |
有机磷源 Organic phosphorus source |
最终产物 End products |
参考文献 References |
| Citrobacter sp. and Serratia sp. | Glycerol-2-phosphate | HUO2PO4 | [23] |
| Genetically altered Escherichia coli | Glycerol-2-phosphate | HUO2PO4 | [44] |
| Aspergillus niger and Paecilomyces javanicus | Glycerol-2-phosphate | Potassium uranyl phosphate hydrate, metaankoleite, uranyl phosphate hydrate, chernikovite, metaankoleite, uramphite | [45] |
| Cryptococcus filicatus, Kluyveromyces lactis, Pichia acaciae, Candida argentea, Candidasake, and Cryptococcus podzolicus | Glycerol-2-phosphate or phytic acid sodium salt hydrate | Metaankoleite, chernikovite bassetite, and uramphite | [46] |
| Escherichia coli | Phytic acid | UO2HPO4·4H2O | [40] |
| Engineered Pseudomonas veronii and Pseudomonas rhodesiae | Glycerol-3-phosphate | HUO2PO4·4H2O | [21] |
| Bacillus and Rahnella | Glycerol-3-phosphate | Ca(UO2)2(PO4)2 | [13] |
| Rahnella strain | Glycerol-3-phosphate | H2(UO2)2(PO4)2 | [47] |
| Aeromonas hydrophila, Pantoea agglomerans, and Pseudomonas rhodesiae | Glycerol-3-phosphate | Ca5(PO4)3OH | [48] |
| Pseudomonas sp. | Tributyl phosphate | HUO2PO4 | [49] |
| Recombinant Deinococcus radiodurans | β-glycerophosphate | [14] | |
| Pseudomonas aeruginosa | Cellular phosphate groups | UO2(PO3)2, (UO2)3(PO4)2H2O, U2O(PO4)2 | [50] |
| Citrobacter | NH4UO2PO4 | [51] | |
| Beauveria caledonica, Hymenoscyphus ericae, Penicillium simplicissimum, Rhizopogon rubescens, Serpula himantioides | Intracellular polyphosphates | Uramphite, chernikovite | [52-53] |
| Hymenoscyphus ericae | Intracellular polyphosphates | [54] |
酸碱度是微生物吸附过程中的重要因素,会对微生物体内的非特异性酸性磷酸酶活性[26]和生物矿化过程及产物产生影响。pH可以改变细菌细胞膜的通透性,从而影响到微生物磷酸盐生物矿化的过程。微生物在铀酰离子刺激下不仅可以通过代谢依赖的过程从细胞中释放出磷酸盐,还可以通过酶解作用分解有机磷酸盐。不同的pH条件下,微生物体内磷酸酶的表达不同,对铀的吸附能力和最终产物会发生明显的变化[55],生物矿化形成的磷酸盐-重金属沉淀不完全相同。Zheng等[56]发现在初始酸性条件下,磷酸盐和U(Ⅵ)生物矿化的最终产物为H2(UO2)2(PO4)2·8H2O,而初始碱性条件下的产物为(NH4)(UO4)PO4·3H2O。
不同pH下微生物体内表达相应的磷酸酶促进生物矿化,但生成的产物不同,pH为5.0时主要的产物为磷酸铀酰钙水合物[Ca(UO2)2(PO4)2·6H2O]和磷酸铀酰水合物[HPUO6·4H2O];pH为7.0和9.0时磷酸铀酰沉淀主要以磷酸铀酰钙[Ca(UO2)2(PO4)2·6H2O]形式存在[13]。
此外,磷酸铀酰沉淀形成的位置是由含铀污染中铀酰种类控制的,而不是磷酸酶的位置,pH值对含铀污染中铀酰种类有影响。酿酒酵母对U(Ⅵ)的生物矿化产物出现了7个不同的异构体和共存的聚合物羟基物种,可溶UO2HPO4(aq)的相对含量随pH (5.0-7.0)的增加而显著降低[18]。Nie等[57]发现溶液pH值增加到7.0时,(UO2)4(OH)7+和(UO2)3(OH)5+在沉淀中所占比例均大于90%,这些沉淀具有高电荷和大离子半径,导致U(Ⅵ)与微生物表面结合位点的空间位阻增大,导致积累量减少。
2.2.3 共存离子铀污染区域典型组分主要有硫酸盐、硝酸盐、锰和铁等,这些共存离子会与铀酰离子形成各种化合物,促进或抑制生物矿化作用,甚至影响生物矿化产物的稳定性,NH4UO2PO4的溶解度比HUO2PO4和NaUO2PO4低,证明NH4+的存在促进了铀酰磷酸盐矿物的形成[51]。Fe3+和CO32–的存在会抑制UO22+的生物矿化作用,而K+、Na+和Mg2+的存在会提高UO22+的生物矿化作用[58]。美国田纳西州的橡树岭站发现在Ca2+浓度比较高且假单胞菌存在时,添加磷酸三丁酯会形成磷酸钙铀酰沉淀,含有Ca2+时,溶液中不易被还原的Ca2UO2(CO3)3占主要成分,抑制其他形态的铀被去除[13]。刘金香等[59]研究表明金属离子对U(Ⅵ)的抑制作用从大到小为Cu2+ > Zn2+ > Ca2+。因此,在生物矿化处理低浓度含铀废水时,可以考虑如何控制溶液参数,促进沥青铀矿或者铀酰磷酸盐沉淀的生成,提高产物的稳定性。
2.2.4 氧化还原电位氧化还原电位是影响生物矿化去除低浓度含铀废水过程的重要因素,选用不同的微生物,氧化还原电位对其影响不同。一般好氧条件下,低浓度含铀废水中的氧化还原电位呈现出氧化性,生物矿化处理低浓度含铀废水的产物比较稳定,不易被氧化。这个环境下微生物的生物还原难以进行,所以微生物在外加磷酸盐源的情况下,以生物吸附、生物富集与生物矿化3种机理共同作用处理低浓度含铀废水。Zhang等[60]发现Bacillus cereus 12-2在好氧条件下微生物先将铀吸附到细胞表面,然后再进行生物矿化。
厌氧或微氧环境下,低浓度含铀废水中的氧化还原电位为负值,呈还原性。在此环境中,微生物对低浓度含铀废水中处理中4种机理都产生作用,但由于微生物比表面积较小、表面活性位点常被有机质掩盖[61],所以后期就以生物还原与生物矿化为主要作用机制。较高pH值下,氧化还原电位负值越大,U(Ⅵ)的还原速度越慢。另一项研究表明,pH值为5.5和7.0的厌氧条件下,U(Ⅵ)和磷酸盐的生物矿化效率要高于U(Ⅵ)到U(Ⅳ)的生物还原反应[43]。
3 生物矿化强化过程生物矿化法修复铀污染废水已经成为近年来的研究热点,在理论研究上已经有了一定的进展,但是在污染现场应用上还有一些困难。微生物的代谢过程比化学过程要缓慢且复杂[62]。因此,生物矿化法涉及微生物、溶液和沉积物,对生物矿化过程进行强化可以从以下3个方面入手。
3.1 微生物群落调控在天然含铀的土壤和沉积物中,微生物被铀酰磷酸盐矿物完全覆盖,说明环境微生物可通过生物矿化作用在土壤中形成铀酰矿物,导致生物衰减[7]。单一菌株对于很多复杂的生化过程难以处理,对环境的扰动适应性较差[63]。因此,寻找合适的微生物群落分解有机磷酸盐,使生物矿化含铀废水的矿化率达到最大化。
研究污染地区的土著菌群进行生物矿化修复含铀废水前,对土著菌群的群落结构通过高通量测序及功能基因分析,确定菌群中在生物矿化过程时必需的微生物。针对生物矿化过程中各个阶段的微生物群落结构及功能基因表达变化的研究,有针对性地投加不同微生物和有机磷酸盐,提高含铀污染废水的修复效果。
3.2 溶液参数调控磷酸盐生物矿化法修复含铀废水过程中,调整某些溶液参数可使微生物的磷酸酶活性达到最大化,尽可能地从有机磷酸盐释放出无机磷酸盐,并在极端pH和温度下,分离出具有高磷酸酶/植酸酶活性的微生物菌株或菌群。利用分离出的微生物菌株或菌群,在模拟实际废水环境下分析各共存离子在实际环境中对生物矿化的影响,找到一种或多种可以促进生物矿化效果的共存离子,以达到最大矿化率。
4 总结与展望磷酸盐生物矿化处理含铀废水是一种原位生物修复技术,其可操作性强,能及时限制含铀废水中的铀随水体的迁移,不易造成二次污染,所以,磷酸盐生物矿化成为目前的研究热点。但磷酸盐生物矿化也存在一些缺点,使其未能在实际含铀污染废水处理中成功应用。目前,磷酸盐生物矿化技术处理含铀废水仍处于发展阶段,实际应用中会涉及实际工况环境、微生物群落等诸多因素影响:(1) 相较于物理化学法,基于微生物的局限性,本方法难以在高温、高压等环境应用。(2) 传统微生物驯化周期长,降解有机磷酸盐的能力也不尽相同,微生物群落间共同降解磷酸盐及矿化含铀废水机制的研究较少,缺乏理论指导。(3) pH和共存离子等因素影响磷酸盐生物矿化效率的机制研究较缺乏。因此该技术有待进一步提高。
就发展趋势而言,未来关于磷酸盐生物矿化含铀废水的深入研究需注重:(1) 磷酸盐生物矿化法与其他物理化学法相互结合创新,拓宽应用范围。(2) 利用分子生物学手段优势和计算机模拟方面的理论,构建合适的人工合成群落,探究微生物降解有机磷酸盐和对含铀废水的矿化机理,优化微生物群落结构,提高有机磷酸盐降解速率和含铀废水矿化速率,形成稳定工艺,如筛选高效菌株、调配符合复配菌剂等方法。(3) 借助试验与计算机模拟相结合的手段,优化pH、共存离子等溶液参数,优化生成产物的矿物粒度、形态、矿物组成、稳定性等。以实现对污染水体的长期有效修复。(4) 在未来对磷酸盐生物矿化的研究中,微生物学可以对基团修饰等改性技术深入研究,增加微生物在实际运行过程中的机械性能、化学稳定性与热力学稳定性,增强微生物降解有机磷酸盐的能力,其研究和发展必有广阔前景。
| [1] |
Singh DK, Hareendran KN, Sreenivas T, Kain V, Dey GK. Development of a phosphate precipitation method for the recovery of uranium from lean tenor alkaline leach liquor[J]. Hydrometallurgy, 2017, 171: 228-235. DOI:10.1016/j.hydromet.2017.05.021 |
| [2] |
Wang XX, Fan QH, Yu SJ, Chen ZS, Ai YJ, Sun YB, Hobiny A, Alsaedi A, Wang XK. RETRACTED: high sorption of U(Ⅵ) on graphene oxides studied by batch experimental and theoretical calculations[J]. Chemical Engineering Journal, 2016, 287: 448-455. DOI:10.1016/j.cej.2015.11.066 |
| [3] |
Xu ZH, Li HX, Xu GL. Feasibility research on enrichment of uranium from acid solution by nano-filter membrane[J]. Uranium Mining and Metallurgy, 2018, 37(1): 37-41, 46. (in Chinese) 许智慧, 李宏星, 胥国龙. 用纳滤膜从酸性溶液中富集铀的可行性研究[J]. 铀矿冶, 2018, 37(1): 37-41, 46. |
| [4] |
Rong LS. Plant-microbial remediation of uranium- contaminated soil and the mechanism analysis[D]. Hengyang: Doctoral Dissertation of University of South China, 2015 (in Chinese) 荣丽杉. 铀污染土壤的植物-微生物修复及其机理研究[D]. 衡阳: 南华大学博士学位论文, 2015 |
| [5] |
Pan XH, Chen Z, Chen FB, Cheng YJ, Lin Z, Guan X. The mechanism of uranium transformation from U(Ⅵ) into nano-uramphite by two indigenous Bacillus thuringiensis strains[J]. Journal of Hazardous Materials, 2015, 297: 313-319. DOI:10.1016/j.jhazmat.2015.05.019 |
| [6] |
Macaskie LE, Empson RM, Cheetham AK, Grey CP, Skarnulis AJ. Uranium bioaccumulation by a Citrobacter sp. as a result of enzymically mediated growth of polycrystalline HUO2PO4[J]. Science, 1992, 257(5071): 782-784. DOI:10.1126/science.1496397 |
| [7] |
Mondani L, Benzerara K, Carrière M, Christen R, Mamindy-Pajany Y, Février L, Marmier N, Achouak W, Nardoux P, Berthomieu C, et al. Influence of uranium on bacterial communities: a comparison of natural uranium-rich soils with controls[J]. PLoS One, 2011, 6(10): e25771. DOI:10.1371/journal.pone.0025771 |
| [8] |
Sowmya S, Rekha PD, Arun AB. Uranium(Ⅵ) bioprecipitation mediated by a phosphate solubilizing Acinetobacter sp. YU-SS-SB-29 isolated from a high natural background radiation site[J]. International Biodeterioration & Biodegradation, 2014, 94: 134-140. |
| [9] |
Morcillo F, González-Muñoz MT, Reitz T, Romero- González ME, Arias JM, Merroun ML. Biosorption and biomineralization of U(Ⅵ) by the marine bacterium Idiomarina loihiensis MAH1: effect of background electrolyte and pH[J]. PLoS One, 2014, 9(3): e91305. DOI:10.1371/journal.pone.0091305 |
| [10] |
Pinto AJ, Gonçalves MA, Prazeres C, Astilleros JM, Batista MJ. Mineral replacement reactions in naturally occurring hydrated uranyl phosphates from the Tarabau deposit: examples in the Cu-Ba uranyl phosphate system[J]. Chemical Geology, 2012, 312/313: 18-26. DOI:10.1016/j.chemgeo.2012.04.004 |
| [11] |
Chandwadkar P, Misra HS, Acharya C. Uranium biomineralization induced by a metal tolerant Serratia strain under acid, alkaline and irradiated conditions[J]. Metallomics, 2018, 10(8): 1078-1088. DOI:10.1039/C8MT00061A |
| [12] |
Krawczyk-Bärsch E, Lütke L, Moll H, Bok F, Steudtner R, Rossberg A. A spectroscopic study on U(Ⅵ) biomineralization in cultivated Pseudomonas fluorescens biofilms isolated from granitic aquifers[J]. Environmental Science and Pollution Research International, 2015, 22(6): 4555-4565. DOI:10.1007/s11356-014-3671-4 |
| [13] |
Beazley MJ, Martinez RJ, Sobecky PA, Webb SM, Taillefert M. Uranium biomineralization as a result of bacterial phosphatase activity: insights from bacterial isolates from a contaminated subsurface[J]. Environmental Science & Technology, 2007, 41(16): 5701-5707. |
| [14] |
Appukuttan D, Rao AS, Apte SK. Engineering of Deinococcus radiodurans R1 for bioprecipitation of uranium from dilute nuclear waste[J]. Applied and Environmental Microbiology, 2006, 72(12): 7873-7878. DOI:10.1128/AEM.01362-06 |
| [15] |
Foulkes JM, Deplanche K, Sargent F, MacAskie LE, Lloyd JR. A novel aerobic mechanism for reductive palladium biomineralization and recovery by Escherichia coli[J]. Geomicrobiology Journal, 2016, 33(3/4): 230-236. |
| [16] |
Kazy SK, D'Souza SF, Sar P. Uranium and thorium sequestration by a Pseudomonas sp.: mechanism and chemical characterization[J]. Journal of Hazardous Materials, 2009, 163(1): 65-72. DOI:10.1016/j.jhazmat.2008.06.076 |
| [17] |
Sousa T, Chung AP, Pereira A, Piedade AP, Morais PV. Aerobic uranium immobilization by Rhodanobacter A2-61 through formation of intracellular uranium-phosphate complexes[J]. Metallomics, 2013, 5(4): 390-397. DOI:10.1039/c3mt00052d |
| [18] |
Shen YH, Zheng XY, Wang XY, Wang TS. The biomineralization process of uranium(Ⅵ) by Saccharomyces cerevisiae: transformation from amorphous U(Ⅵ) to crystalline chernikovite[J]. Applied Microbiology and Biotechnology, 2018, 102(9): 4217-4229. DOI:10.1007/s00253-018-8918-4 |
| [19] |
Wang NY, Zhang H, Sui Y, Chen YY, Hu N, Dai ZR, Ding DX. Biomineralization of U(Ⅵ)-PO43– promoted by Aspergillus tubingensis mediated phytate hydrolysis[J]. China Environmental Science, 2019, 39(5): 2161-2169. (in Chinese) 王聂颖, 张辉, 隋阳, 陈约余, 胡南, 戴仲然, 丁德馨. Aspergillus tubingensis介导植酸盐水解促进U(Ⅵ)-PO43–生物矿化[J]. 中国环境科学, 2019, 39(5): 2161-2169. DOI:10.3969/j.issn.1000-6923.2019.05.045 |
| [20] |
Martinez RJ, Beazley MJ, Taillefert M, Arakaki AK, Skolnick J, Sobecky PA. Aerobic uranium (Ⅵ) bioprecipitation by metal-resistant bacteria isolated from radionuclide- and metal-contaminated subsurface soils[J]. Environmental Microbiology, 2007, 9(12): 3122-3133. DOI:10.1111/j.1462-2920.2007.01422.x |
| [21] |
Powers LG, Mills HJ, Palumbo AV, Zhang CL, Delaney K, Sobecky PA. Introduction of a plasmid-encoded phoA gene for constitutive overproduction of alkaline phosphatase in three subsurface Pseudomonas isolates[J]. FEMS Microbiology Ecology, 2002, 41(2): 115-123. DOI:10.1111/j.1574-6941.2002.tb00972.x |
| [22] |
Kulkarni S, Misra CS, Gupta A, Ballal A, Apte SK. Interaction of uranium with bacterial cell surfaces: inferences from phosphatase-mediated uranium precipitation[J]. Applied and Environmental Microbiology, 2016, 82(16): 4965-4974. DOI:10.1128/AEM.00728-16 |
| [23] |
Macaskie LE, Bonthrone KM, Rouch DA. Phosphatase- mediated heavy metal accumulation by a Citrobacter sp. and related enterobacteria[J]. FEMS Microbiology Letters, 1994, 121(2): 141-146. DOI:10.1111/j.1574-6968.1994.tb07090.x |
| [24] |
Lloyd JR, MacAskie LE. Bioremediation of radionuclide- containing wastewaters[A]//Lovley DR. Environmental Microbe-Metal Interactions[M]. Washington, DC, USA: ASM Press, 2014: 277-327
|
| [25] |
Wufuer R, Wei YY, Lin QH, Wang HW, Song WJ, Liu W, Zhang DY, Pan XL, Gadd GM. Uranium bioreduction and biomineralization[J]. Advances in Applied Microbiology, 2017, 101: 137-168. |
| [26] |
Beazley MJ, Martinez RJ, Webb SM, Sobecky PA, Taillefert M. The effect of pH and natural microbial phosphatase activity on the speciation of uranium in subsurface soils[J]. Geochimica et Cosmochimica Acta, 2011, 75(19): 5648-5663. DOI:10.1016/j.gca.2011.07.006 |
| [27] |
Zhao XM, Do H, Zhou Y, Li Z, Zhang XF, Zhao SJ, Li MT, Wu D. Rahnella sp. LRP3 induces phosphate precipitation of Cu(Ⅱ) and its role in copper-contaminated soil remediation[J]. Journal of Hazardous Materials, 2019, 368: 133-140. DOI:10.1016/j.jhazmat.2019.01.029 |
| [28] |
Caetano AL, Marques CR, Gavina A, Carvalho F, Gonçalves F, Da Silva EF, Pereira R. Contribution for the derivation of a soil screening value (SSV) for uranium, using a natural reference soil[J]. PLoS One, 2014, 9(10): e108041. DOI:10.1371/journal.pone.0108041 |
| [29] |
Zeglin LH. Stream microbial diversity in response to environmental changes: review and synthesis of existing research[J]. Frontiers in Microbiology, 2015, 6: 454. |
| [30] |
Suriya J, Chandra Shekar M, Nathani NM, Suganya T, Bharathiraja S, Krishnan M. Assessment of bacterial community composition in response to uranium levels in sediment samples of sacred Cauvery River[J]. Applied Microbiology and Biotechnology, 2017, 101(2): 831-841. DOI:10.1007/s00253-016-7945-2 |
| [31] |
Pfennig DW, Pfennig KS. Character displacement and the origins of diversity[J]. The American Naturalist, 2010, 176(Suppl 1): S26-S44. |
| [32] |
Lopez-Fernandez M, Vilchez-Vargas R, Jroundi F, Boon N, Pieper D, Merroun ML. Microbial community changes induced by uranyl nitrate in bentonite clay microcosms[J]. Applied Clay Science, 2018, 160: 206-216. DOI:10.1016/j.clay.2017.12.034 |
| [33] |
Martinez RJ, Wu CH, Beazley MJ, Andersen GL, Conrad ME, Hazen TC, Taillefert M, Sobecky PA. Microbial community responses to organophosphate substrate additions in contaminated subsurface sediments[J]. PLoS One, 2014, 9(6): e100383. DOI:10.1371/journal.pone.0100383 |
| [34] |
Yin TT, Lin H, Dong YB, Li B, He YH, Liu CJ, Chen X. A novel constructed carbonate-mineralized functional bacterial consortium for high-efficiency cadmium biomineralization[J]. Journal of Hazardous Materials, 2021, 401: 123269. DOI:10.1016/j.jhazmat.2020.123269 |
| [35] |
Gołębiewski M, Deja-Sikora E, Cichosz M, Tretyn A, Wróbel B. 16S rDNA pyrosequencing analysis of bacterial community in heavy metals polluted soils[J]. Microbial Ecology, 2014, 67(3): 635-647. DOI:10.1007/s00248-013-0344-7 |
| [36] |
Dhal PK, Islam E, Kazy SK, Sar P. Culture-independent molecular analysis of bacterial diversity in uranium-ore/-mine waste-contaminated and non-contaminated sites from uranium mines[J]. 3 Biotech, 2011, 1(4): 261-272. DOI:10.1007/s13205-011-0034-4 |
| [37] |
Wellman DM, Icenhower JP, Owen AT. Comparative analysis of soluble phosphate amendments for the remediation of heavy metal contaminants: Effect on sediment hydraulic conductivity[J]. Environmental Chemistry, 2006, 3(3): 219-224. DOI:10.1071/EN05023 |
| [38] |
Huang C, Zhang H, Hu N, Ding DX, Li DX, Sun J. Remediation of uranium contaminated groundwater by β-glycerophosphate[J]. China Environmental Science, 2018, 38(9): 3391-3397. (in Chinese) 黄超, 张辉, 胡南, 丁德馨, 李殿鑫, 孙静. β-甘油磷酸钠修复含铀地下水[J]. 中国环境科学, 2018, 38(9): 3391-3397. DOI:10.3969/j.issn.1000-6923.2018.09.024 |
| [39] |
Hurrell RF, Reddy MB, Juillerat MA, Cook JD. Degradation of phytic acid in cereal porridges improves iron absorption by human subjects[J]. The American Journal of Clinical Nutrition, 2003, 77(5): 1213-1219. DOI:10.1093/ajcn/77.5.1213 |
| [40] |
Paterson-Beedle M, Readman JE, Hriljac JA, MacAskie LE. Biorecovery of uranium from aqueous solutions at the expense of phytic acid[J]. Hydrometallurgy, 2010, 104(3/4): 524-528. |
| [41] |
Zhu DG, Xie SB, Liu YJ, Wang Y, Si ZY, Zhu AQ. Experimental study on uranium(Ⅵ) adsorption in water by phytic acid modified Aspergillus niger[J]. Science Technology and Engineering, 2019, 19(21): 344-351. (in Chinese) 朱定国, 谢水波, 刘迎九, 王越, 司子彦, 朱奥琦. 植酸改性黑曲霉菌对水中U(Ⅵ)的吸附试验研究[J]. 科学技术与工程, 2019, 19(21): 344-351. DOI:10.3969/j.issn.1671-1815.2019.21.053 |
| [42] |
Fan LF, Xie SB, Liu YJ, Chen J. Adsorption of U(Ⅵ) containing wastewater by E. coli with phytic acid[J]. Chinese Journal of Environmental Engineering, 2016, 10(8): 4167-4171. (in Chinese) 范黎锋, 谢水波, 刘迎九, 陈婧. 大肠杆菌配合植酸对铀的吸附[J]. 环境工程学报, 2016, 10(8): 4167-4171. |
| [43] |
Salome KR, Beazley MJ, Webb SM, Sobecky PA, Taillefert M. Biomineralization of U(Ⅵ) phosphate promoted by microbially-mediated phytate hydrolysis in contaminated soils[J]. Geochimica et Cosmochimica Acta, 2017, 197: 27-42. DOI:10.1016/j.gca.2016.10.008 |
| [44] |
Basnakova G, Stephens ER, Thaller MC, Rossolini GM, MacAskie LE. The use of Escherichia coli bearing a phoN gene for the removal of uranium and nickel from aqueous flows[J]. Applied Microbiology and Biotechnology, 1998, 50(2): 266-272. DOI:10.1007/s002530051288 |
| [45] |
Liang XJ, Csetenyi L, Gadd GM. Uranium bioprecipitation mediated by yeasts utilizing organic phosphorus substrates[J]. Applied Microbiology and Biotechnology, 2016, 100(11): 5141-5151. DOI:10.1007/s00253-016-7327-9 |
| [46] |
Liang XJ, Hillier S, Pendlowski H, Gray N, Ceci A, Gadd GM. Uranium phosphate biomineralization by fungi[J]. Environmental Microbiology, 2015, 17(6): 2064-2075. DOI:10.1111/1462-2920.12771 |
| [47] |
Beazley MJ, Martinez RJ, Sobecky PA, Webb SM, Taillefert M. Nonreductive biomineralization of uranium(Ⅵ) phosphate via microbial phosphatase activity in anaerobic conditions[J]. Geomicrobiology Journal, 2009, 26(7): 431-441. DOI:10.1080/01490450903060780 |
| [48] |
Shelobolina ES, Konishi H, Xu HF, Roden EE. U(Ⅵ) sequestration in hydroxyapatite produced by microbial glycerol 3-phosphate metabolism[J]. Applied and Environmental Microbiology, 2009, 75(18): 5773-5778. DOI:10.1128/AEM.00628-09 |
| [49] |
Thomas RAP, Macaskie LE. Biodegradation of tributyl phosphate by naturally occurring microbial isolates and coupling to the removal of uranium from aqueous solution[J]. Environmental Science & Technology, 1996, 30(7): 2371-2375. |
| [50] |
Choudhary S, Sar P. Uranium biomineralization by a metal resistant Pseudomonas aeruginosa strain isolated from contaminated mine waste[J]. Journal of Hazardous Materials, 2011, 186(1): 336-343. DOI:10.1016/j.jhazmat.2010.11.004 |
| [51] |
Yong P, Macaskie LE. Enhancement of uranium bioaccumulation by a Citrobacter sp. via enzymically- mediated growth of polycrystalline NH4UO2PO4[J]. Journal of Chemical Technology and Biotechnology, 1995, 63(2): 101-108. DOI:10.1002/jctb.280630202 |
| [52] |
Fomina M, Charnock J, Bowen AD, Gadd GM. X-ray absorption spectroscopy (XAS) of toxic metal mineral transformations by fungi[J]. Environmental Microbiology, 2007, 9(2): 308-321. DOI:10.1111/j.1462-2920.2006.01139.x |
| [53] |
Fomina M, Charnock JM, Hillier S, Alvarez R, Gadd GM. Fungal transformations of uranium oxides[J]. Environmental Microbiology, 2007, 9(7): 1696-1710. DOI:10.1111/j.1462-2920.2007.01288.x |
| [54] |
Fomina M, Charnock JM, Hillier S, Alvarez R, Livens F, Gadd GM. Role of fungi in the biogeochemical fate of depleted uranium[J]. Current Biology, 2008, 18(9): R375-R377. DOI:10.1016/j.cub.2008.03.011 |
| [55] |
Wang TS, Zheng XY, Wang XY, Lu X, Shen YH. Different biosorption mechanisms of uranium(Ⅵ) by live and heat-killed Saccharomyces cerevisiae under environmentally relevant conditions[J]. Journal of Environmental Radioactivity, 2017, 167: 92-99. DOI:10.1016/j.jenvrad.2016.11.018 |
| [56] |
Zheng XY, Shen YH, Wang XY, Wang TS. Effect of pH on uranium(Ⅵ) biosorption and biomineralization by Saccharomyces cerevisiae[J]. Chemosphere, 2018, 203: 109-116. DOI:10.1016/j.chemosphere.2018.03.165 |
| [57] |
Nie XQ, Dong FQ, Liu MX, He HC, Sun SY, Bian L, Yang G, Zhang W, Qin YL, Huang R, et al. Microbially mediated stable uranium phosphate nano-biominerals[J]. Journal of Nanoscience and Nanotechnology, 2017, 17(9): 6771-6780. DOI:10.1166/jnn.2017.14463 |
| [58] |
Wei YL, Chen Z, Song H, Zhang J, Lin Z, Dang Z, Deng H. The immobilization mechanism of U(Ⅵ) induced by Bacillus thuringiensis 016 and the effects of coexisting ions[J]. Biochemical Engineering Journal, 2019, 144: 57-63. DOI:10.1016/j.bej.2019.01.013 |
| [59] |
Liu JX, Xiong F, Rong LS, Li SY, Ling H, Xie SB. Influence of coexistent metal ions on reduction of U(Ⅵ) by Shewanella oneidensis with Fe0[J]. Chinese Journal of Environmental Engineering, 2016, 10(9): 5269-5274. (in Chinese) 刘金香, 熊芬, 荣丽杉, 李仕友, 凌辉, 谢水波. 共存金属离子对Fe0-奥奈达希瓦氏菌协同还原U(Ⅵ)的影响[J]. 环境工程学报, 2016, 10(9): 5269-5274. |
| [60] |
Zhang J, Song H, Chen Z, Liu SS, Wei YL, Huang JY, Guo CL, Dang Z, Lin Z. Biomineralization mechanism of U(Ⅵ) induced by Bacillus cereus 12-2: the role of functional groups and enzymes[J]. Chemosphere, 2018, 206: 682-692. DOI:10.1016/j.chemosphere.2018.04.181 |
| [61] |
Liu JX, Ge YJ, Xie SB, Yu K. Application progress of modified microbial adsorbents for the treatment of heavymetal wastewater[J]. Microbiology China, 2020, 47(3): 941-951. (in Chinese) 刘金香, 葛玉杰, 谢水波, 俞坤. 改性微生物吸附剂在重金属废水处理中的应用进展[J]. 微生物学通报, 2020, 47(3): 941-951. |
| [62] |
Arias D, Cisternas L, Rivas M. Biomineralization mediated by ureolytic bacteria applied to water treatment: a review[J]. Crystals, 2017, 7(11): 345. DOI:10.3390/cryst7110345 |
| [63] |
Wang GH, Yang SQ, Zhou YH, Yuan HS, Li SY, Lyu YJ, Xie SB. Research progress on the bioremediation of groundwater polluted by uranium via bio-reduction[J]. Environmental Science & Technology, 2019, 42(8): 47-53. (in Chinese) 王国华, 杨思芹, 周耀辉, 袁华山, 李仕友, 吕怡锦, 谢水波. 生物还原法修复铀污染地下水的研究进展[J]. 环境科学与技术, 2019, 42(8): 47-53. |
 2021, Vol. 48
2021, Vol. 48




