扩展功能
文章信息
- 戴利铭, 李岚岚, 刘一贤, 施玉萍, 蔡志英
- DAI Liming, LI Lanlan, LIU Yixian, SHI Yuping, CAI Zhiying
- 解淀粉芽孢杆菌生防菌BS-3全基因组测序及生物信息分析
- Whole genome sequencing and genomics analysis of Bacillus amyloliquefaciens BS-3 with biocontrol activity
- 微生物学通报, 2021, 48(6): 2073-2088, 2142
- Microbiology China, 2021, 48(6): 2073-2088, 2142
- DOI: 10.13344/j.microbiol.china.200888
-
文章历史
- 收稿日期: 2020-09-04
- 接受日期: 2020-10-26
- 网络首发日期: 2020-12-24
解淀粉芽孢杆菌(Bacillus amyloliquefaciens)是一种与枯草芽孢杆菌(B. subtilis)亲缘性很近的细菌,其菌体生长快、易培养、抗逆性强,能够产生脂肽类、肽类、细菌素和抗菌蛋白等多种抗菌物质[1],这些抗菌物质种类繁多、稳定性高、抑菌谱广、抑菌活性强、不易产生抗药性,对人体和动植物的细胞安全且不会造成环境污染,是近年来植物病害生物防治、动物饲料加工、生鲜农产品防腐保鲜、医药研发、污水治理等领域研究的热点[2-3]。
近年来,有研究者采用液相色谱-质谱法分析了解淀粉芽孢杆菌纯化后的发酵产物,发现含有表面活性素(Surfactin)同系物和小分子量的抑菌肽成分[4]。对胡椒瘟病生防菌B. subtilis VD18R19全基因组测序及比较基因组学分析发现,该菌含有6个抑菌次级代谢产物合成基因簇,编码表面活性素、大侧柏素、儿茶酚型嗜铁素、杆菌溶素、聚酮化合物、羊毛硫类细菌素等抑菌物质[5]。Arrebola等从B. amyloliquefaciens发酵产物中分离出具有抑制病原真菌作用的IturinA脂肽[6]。Wong等报道从解淀粉芽孢杆菌发酵产物中分离出可抑制肝癌、结肠癌细胞生长的蛋白质[7]。An等从解淀粉芽孢杆菌的发酵产物中分离得到对食物腐败菌有抑制作用的细菌素[8]。从解淀粉芽孢杆菌中获得次级代谢产物合成基因簇和抑菌活性成分的研究备受学者青睐。
我们项目组在健康的橡胶树树根中筛选到一株内生解淀粉芽孢杆菌(BS-3),该菌对橡胶树褐根病菌(Phellinus noxius)、尖孢炭疽病菌(Colletotrichum acutatum)、小孢拟盘多毛孢(Pestalotiopsis microspora)、橡胶疫霉(Phytophthora heveae)多种病原真菌具有很好的抑菌效果。传统的试验分析和鉴定方法很难全面地分析生防菌中的抗性物质且耗时耗力,难以充分挖掘其全部抗菌基因和揭示生防作用机理[9]。为了深入研究BS-3菌株防病功能的内在原因,本研究采用第二代BGISEQ平台与第三代PacBio平台相结合的测序技术对解淀粉芽孢杆菌BS-3全基因组测序,在获得BS-3菌株全基因组序列的基础上,借助GO、KEGG、COG、NR和Swiss-Prot数据库注释BS-3菌株基因功能、预测菌株BS-3的次级代谢产物基因簇和比较基因组学分析,以期对挖掘次级代谢产物基因资源、高效开发利用BS-3和对其抑菌机理的研究提供生物信息学基础。
1 材料与方法 1.1 材料 1.1.1 菌株菌株解淀粉芽孢杆菌BS-3分离自云南省景洪市橡胶树根,对多种病原菌有抑制作用,保存于云南省热带作物科学研究所植物保护与微生物利用研究中心。
植物病原真菌:橡胶树褐根病菌(Phellinus noxius)、尖孢炭疽病菌(Colletotrichum acutatum)、小孢拟盘多毛孢(Pestalotiopsis microspora)、橡胶疫霉(Phytophthora heveae)均保存于云南省热带作物科学研究所植物保护与微生物利用研究中心。
1.1.2 主要试剂和仪器细菌基因组DNA提取试剂盒,布鲁克(北京)科技有限公司;DL2000 DNA Marker,北京天根生化科技有限公司。PCR仪,Applied Biosystems公司;电泳仪,北京六一生物科技有限公司;NanoDrop 2000,赛默飞世尔科技公司。
1.1.3 培养基LB培养基(g/L):蛋白胨10.0,酵母提取物5.0,NaCl 10.0;PDA培养基(g/L):马铃薯200.0、葡萄糖20.0。固体培养基均添加琼脂粉15 g/L。1×105 Pa灭菌30 min。
1.2 方法 1.2.1 植物病原真菌拮抗试验采用平板对峙法,在PDA平板中间分别放置橡胶树褐根病菌、尖孢炭疽病菌、小孢拟盘多毛孢、橡胶疫霉4种植物病原菌的菌块,菌块四周划线接种BS-3,以只接种病原菌的处理为对照,重复3次,28 ℃恒温箱中培养5 d,测量抑菌圈直径(对照组菌落生长直径−处理组菌落生长直径),观察抑菌情况并拍照。
1.2.2 菌株总DNA提取菌株BS-3接种于LB培养基中,在37 ℃、180 r/min条件下培养12 h,将培养液10 000×g离心5 min收集菌体,采用细菌基因组DNA提取试剂盒提取基因组,操作步骤参照试剂盒说明书。使用1%的琼脂糖凝胶电泳和NanoDrop 2000检测DNA样品的浓度、纯净度和降解程度。
1.2.3 全基因测序本次测序委托深圳华大基因股份有限公司完成。样本质检合格后,使用Covaris仪超声波打断DNA样品,进行文库构建。采用第二代BGISEQ平台与第三代PacBio平台相结合的测序技术进行测序。
1.2.4 基因组组分分析使用pb-falcon v2.2.3软件对三代Reads进行组装,并对二代数据组装结果进行2次Polish (pilon v1.23软件)。编码基因预测使用GeneMarkS软件(V4.17) (http://topaz.gatech.edu/GeneMark/)[10];重复序列的预测使用RepeatMasker (Version open-4.0.5)软件和TRF (Tandem Repeats Finder,V4.07b)[11-12];tRNA预测通过tRNAscan-SE软件(V1.3.1)[13],rRNA预测采用RNAmmer软件(V1.2)[14],sRNA确定采用cmsearch程序(V1.1rc4)[15-16]。基因岛预测采用IslandPath-DIOMB软件(V0.2)[17],前噬菌体预测通过PhiSpy软件[18](V2.3),CRISPR预测利用CRISPRdigger[19](V1.0)。
1.2.5 基因功能分析(1) 基因功能注释
使用BLAST软件与Gene Ontology (GO)[20]、Kyoto Encyclopedia of Genes and Genomes (KEGG)[21-22]、Cluster of Orthologous Groups of Proteins (COG)[23]、Non-Redundant Protein Database (NR)[24]、Carbohydrate-Active EnZymes Database (CAZy)[25]、Type Ⅲ Secretion System Effector Protein (T3SS)[26]、Virulence Factor Database (VFDB)[27]、Pathogen Host Interactions (PHI)[28]、Swiss-Prot[29]数据库进行比对,获得功能注释信息。
(2) BS-3的进化和比较基因组学分析
根据全基因组注释信息,选择gyrA基因序列及NCBI数据库比对分析结果,利用MEGA 7.0软件的Neighbor-Joining法构建系统发育树;同时将菌株BS-3的基因组序列与参考菌B. amyloliquefaciens subsp. FZB42 (CP000560)的基因组序列基于Mummer进行比对分析。
(3) 拮抗物质分析
利用antiSMASH 3.0 (https://fungismash.secondarymetabolites.org)对菌株BS-3中次级代谢产物合成基因簇进行预测[30]。结合基因注释结果和NCBI BLAST比对分析结果,分析菌株BS-3次级代谢产物中的抑菌物质及编码基因簇。
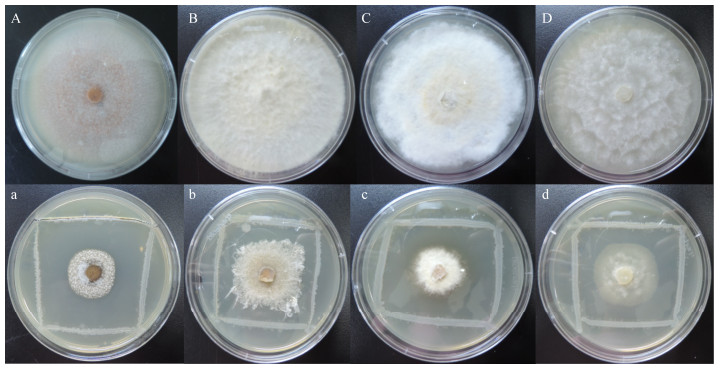
2 结果与分析 2.1 病原真菌拮抗试验拮抗试验发现,BS-3对尖孢炭疽病菌(Colletotrichum acutatum)、橡胶树褐根病菌(Phellinus noxius)、小孢拟盘多毛孢(Pestalotiopsis microspora)、橡胶疫霉(Phytophthora heveae)的生长均具有良好抑制效果,如图 1所示,抑菌圈直径分别为62.96、41.36、51.49、51.49 mm。
|
| 图 1 菌株BS-3对病原菌的拮抗作用 Figure 1 The antagonism of strain BS-3 against pathogens 注:A:尖孢炭疽病菌;B:褐根病菌;C:小孢拟盘多毛孢;D:橡胶疫霉;a:菌株BS-3对尖孢炭疽病菌的拮抗作用;b:菌株BS-3对褐根病菌的拮抗作用;c:菌株BS-3对小孢拟盘多毛孢的拮抗作用;d:菌株BS-3对橡胶疫霉的拮抗作用 Note: A: Colletotrichum acutatum; B: Phellinus noxius; C: Pestalotiopsis microspora; D: Phytophthora heveae; a: The antagonism of strain BS-3 against Colletotrichum acutatum; b: The antagonism of strain BS-3 against Phellinus noxius; c: The antagonism of strain BS-3 against Pestalotiopsis microspora; d: The antagonism of strain BS-3 against Phytophthora heveae |
|
|
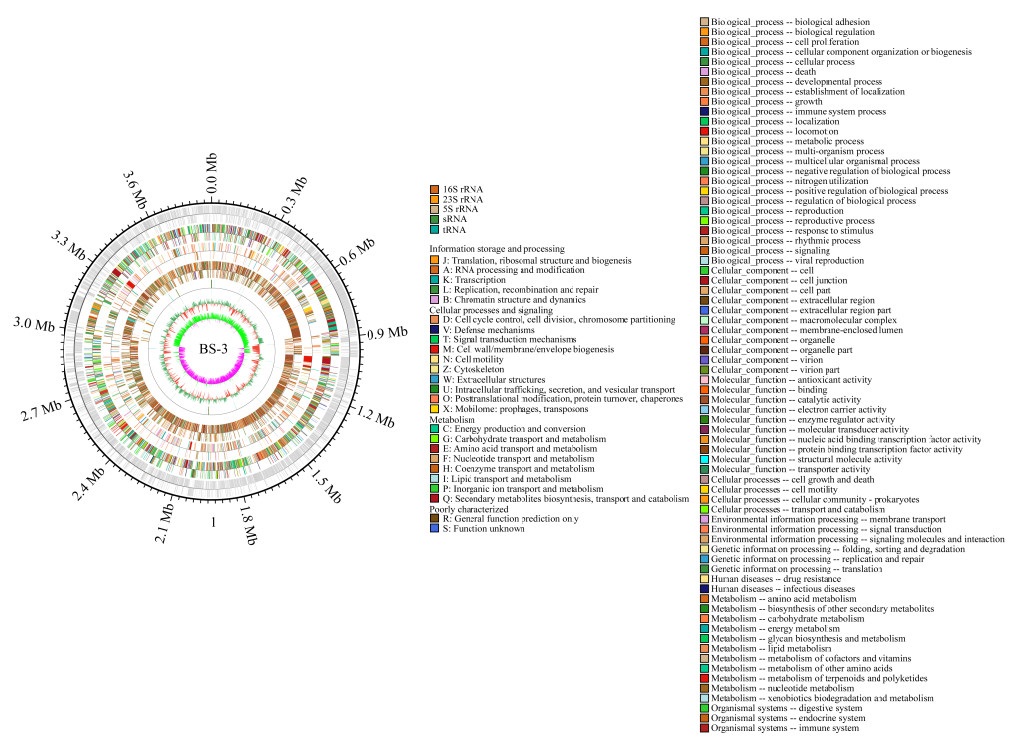
采用第二代BGISEQ平台与第三代PacBio平台相结合的测序技术,对细菌BS-3的全基因组进行测序。菌株BS-3全基因组大小为3 870 130 bp,平均GC含量为46.88%,共编码基因4 161个;所有编码基因的总长度为3 463 893 bp,编码基因的平均长度为832 bp,含有串联重复(Tandem Repeat,TR)序列122个,总长为10 580 bp,大小为6−282 bp,其中小卫星(Minisatellite) DNA 98个,总长为7 148 bp,大小为10−60 bp,微卫星(Microsatellite) DNA 2个,总长为78 bp。含有tRNA基因92个,5S rRNA 10个,16S rRNA和23S rRNA各9个,sRNA 10个。BS-3基因组测序数据提交至GenBank,登录号为CP060384,BS-3基因组图见图 2。将BS-3的全基因组信息与其他已测序的近缘芽孢杆菌比较分析[31],如表 1所示,BS-3与FZB42的大小较为接近,而GC含量的比例也较为接近,但BS-3的Protein-Coding Sequences基因数却比FZB42多468个。
|
| 图 2 B. amyloliquefaciens BS-3基因组图 Figure 2 Genomic map of the B. amyloliquefaciens BS-3 注:由外至内,第1圈表示编码基因的分布;第2圈表示COG注释基因分布;第3圈表示KEGG注释基因分布;第4圈表示GO的注释基因分布;第5圈表示ncRNA分布 Note: From the outer to inner circle, the first circle is the distribution of the coding genes; The second is the COG annotated genes; The third is the KEGG annotated ggenes; The fourth is the GO annotated genes; The fifth circle is the distribution of ncRNA |
|
|
| Items | B. amyloliquefaciens FZB42 | B. amyloliquefaciens B9601-Y2 | B. amyloliquefaciens BS-3 | B. subtilis 168 |
| Genome size (bp) | 3 918 589 | 4 238 624 | 3 870 130 | 4 214 630 |
| GC content (%) | 46.4 | 45.8 | 46.9 | 43.5 |
| Protein-coding sequences | 3 693 | 4 159 | 4 161 | 4 106 |
| Average CDS size (bp) | 933 | 890 | 832 | 895 |
| Number of tRNAs | 89 | 91 | 92 | 86 |
采用GO、KEGG、COG、NR、CAZy、T3SS、VFDB、PHI、ARDB和Swiss-Prot数据库将预测基因的蛋白序列与各功能数据库进行Diamond比对(e value≤1e-5),选取Score最高的比对结果(默认Identity≥40%,Coverage≥40%)进行注释。最终注释统计数据见表 2,发现菌株BS-3基因组共有4 161个基因得到成功比对注释。其中,在NR、Swiss-Prot、COG、GO数据库中得到功能注释的基因较多,分别为4 040、3 328、2 875、2 620个,占基因总数的97.09%、79.98%、69.09%、62.97%;而在CARD数据库中得到注释的基因最少,仅有22个,占基因总数的0.53%。
| Type | Gene number |
| NR | 4 040 |
| Swiss-Prot | 3 328 |
| KEGG | 1 885 |
| COG | 2 875 |
| TCDB | 459 |
| GO | 2 620 |
| PHI | 157 |
| VFDB | 62 |
| ARDB | 30 |
| CARD | 22 |
| Secretory | 80 |
| T3SS | 80 |
| CAZY | 137 |
将菌株BS-3氨基酸序列与GO数据库进行比对和统计分析,得到菌株功能基因的分布情况。共有2 620个基因在GO数据库中被注释到,GO数据库按照细胞组分(Cellular Component)、生物学过程(Biological Process)、分子功能(Molecular Function) 3个方面对蛋白进行注释(表 3),而生物学过程、细胞组分和分子功能分支各25、11和10个,共计46个分支。在细胞组分中,共2 450个基因得到注释,其中与细胞部分(Cell Part)、细胞(Cell)、细胞器(Organelle)有关的基因表现出最高相关性,各有914、914、198个;生物学途径类共5 967个基因被注释,涉及最多的基因与细胞内过程(Cellular Process)、代谢过程(Metabolic Process)有关,各有1 464、1 466个;分子功能分支共3 271个注释结果,其中涉及最多的基因与结合(Binding)、催化活性(Catalytic Activity)、转运活性(Transporter Activity)有关,分别为1 186、1 408、236个。
| Ontology | Class | Gene number |
| Biological process | Biological adhesion | 16 |
| Biological regulation | 516 | |
| Cell proliferation | 2 | |
| Cellular component organization or biogenesis | 143 | |
| Cellular process | 1 464 | |
| Death | 4 | |
| Developmental process | 68 | |
| Establishment of localization | 525 | |
| Growth | 4 | |
| Immune system process | 7 | |
| Localization | 553 | |
| Locomotion | 42 | |
| Metabolic process | 1 466 | |
| Multi-organism process | 49 | |
| Multicellular organismal process | 23 | |
| Negative regulation of biological process | 26 | |
| Nitrogen utilization | 1 | |
| Positive regulation of biological process | 15 | |
| Regulation of biological process | 488 | |
| Reproduction | 57 | |
| Reproductive process | 38 | |
| Response to stimulus | 275 | |
| Rhythmic process | 1 | |
| Signaling | 150 | |
| Viral reproduction | 34 | |
| Cellular component | Cell | 914 |
| Cell junction | 1 | |
| Cell part | 914 | |
| Extracellular region | 23 | |
| Extracellular region part | 23 | |
| Macromolecular complex | 179 | |
| Membrane-enclosed lumen | 35 | |
| Organelle | 198 | |
| Organelle part | 95 | |
| Virion | 34 | |
| Virion part | 34 | |
| Molecular function | Antioxidant activity | 11 |
| Binding | 1 186 | |
| Catalytic activity | 1 408 | |
| Electron carrier activity | 1 | |
| Enzyme regulator activity | 13 | |
| Molecular transducer activity | 90 | |
| Nucleic acid binding transcription factor activity | 203 | |
| Protein binding transcription factor activity | 62 | |
| Structural molecule activity | 61 | |
| Transporter activity | 236 |
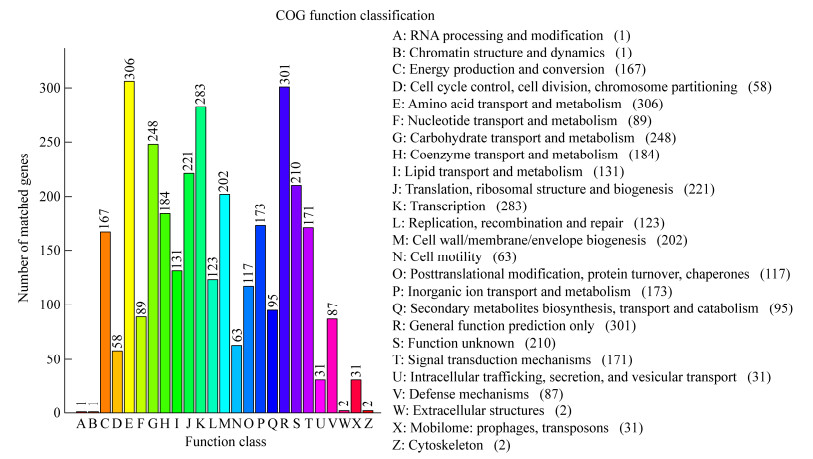
对菌株BS-3基因组中具有生物学功能的蛋白编码基因进行COG注释,结果发现共有2 875个蛋白编码基因被注释到。如图 3所示,基因功能注释结果共分为25类,分别有1、1、167、58、306、89、248、184、131、221、283、123、202、63、117、173、95、301、210、171、31、87、2、31、2个基因注释分类到A−Z,其中氨基酸代谢及转运(Amino Acid Transport and Metabolism)的注释结果最为丰富,共306个,占注释基因总数的10.64%;其次为常规功能预测(General Function Prediction Only)基因,共301个基因,占注释基因总数的10.47%,而转录(283个基因,9.84%)、碳水化合物转运和代谢(248个基因,9.88%)、翻译、核糖体结构和生物合成(221个基因,7.69%)的基因也得到较多的注释,此外还有210个(7.3%)功能未知的基因,有待今后进一步研究。
|
| 图 3 菌株BS-3基因组COG数据库比对分析结果 Figure 3 COG functional classification of the strain BS-3 |
|
|
将菌株BS-3与KEGG数据库进行比对分析,对应到KEGG Pathway的1 855个基因富集在194条代谢通路中,占菌株基因总数的44.85%;基因数大于等于20的代谢通路有24个(表 4)。KEGG富集分析显示,ABC转运蛋白(ABC Transporters)、双组分调节系统(Two-Component System)、细菌的群体行为调控机制(Quorum Sensing)是最主要的3种代谢通路,分别有120、107和70个基因注释结果;此外,嘌呤代谢(Purine Metabolism)、核糖体(Ribosome)、嘧啶代谢(Pyrimidine Metabolism)、丙酮酸代谢(Pyruvate Metabolism)、氧化磷酸化(Oxidative Phosphorylation)通路与菌株基因组相比具有较高的相关度。
| Map number | Map name | Gene number (≥20) |
| Map02010 | ABC transporters | 120 |
| Map02020 | Two-component system | 107 |
| Map02024 | Quorum sensing | 70 |
| Map00230 | Purine metabolism | 59 |
| Map03010 | Ribosome | 53 |
| Map00240 | Pyrimidine metabolism | 44 |
| Map00620 | Pyruvate metabolism | 33 |
| Map02040 | Flagellar assembly | 33 |
| Map00190 | Oxidative phosphorylation | 31 |
| Map00250 | Alanine, aspartate and glutamate metabolism | 30 |
| Map00500 | Starch and sucrose metabolism | 30 |
| Map00270 | Cysteine and methionine metabolism | 29 |
| Map00520 | Amino sugar and nucleotide sugar metabolism | 29 |
| Map00550 | Peptidoglycan biosynthesis | 28 |
| Map00970 | Aminoacyl-tRNA biosynthesis | 27 |
| Map00010 | Glycolysis / Gluconeogenesis | 23 |
| Map00260 | Glycine, serine and threonine metabolism | 23 |
| Map02030 | Bacterial chemotaxis | 22 |
| Map02060 | Phosphotransferase system (PTS) | 22 |
| Map00630 | Glyoxylate and dicarboxylate metabolism | 21 |
| Map00640 | Propanoate metabolism | 21 |
| Map00564 | Glycerophospholipid metabolism | 20 |
| Map00730 | Thiamine metabolism | 20 |
| Map00860 | Porphyrin and chlorophyll metabolism | 20 |
将基因组序列与CAZy数据库进行比对,发现菌株BS-3的基因组中共有140个基因编码的蛋白质结构域属于CAZy家族,包括20类糖苷水解酶(Glycoside Hydrolases,GHs)家族的蛋白49个、9类糖苷转移酶家族的蛋白(Glycosyl Transferases,GTs) 36个、6类碳水化合物酯酶(Carbohydrate Esterases,CEs) 17个、2类多糖裂解酶(Polysaccharide Lyases,PLs) 3个、辅助酶4个、3类碳水化合物结合组件(Carbohydrate-Binding Modules,CBMs) 31个。土壤中存在植物病残体,这些病残体物质的主要组分是糖类和蛋白质类,而菌株BS-3中含有编码内切葡聚糖酶(endo-1,4-β-D-glucanase,EC3.2.1.4)、β-葡萄糖苷酶(β-glucosidase,EC3.2.1.21)、α-淀粉酶(amyE,EC3.2.1.1)等酶的基因,从基因角度分析进一步了解菌株BS-3的生防作用机理。
2.3.5 NR数据库和Swiss-Prot数据库的注释将菌株BS-3的基因序列翻译为相应的氨基酸序列,并与NR数据库进行比对,基因组中有4 040个基因在NR数据库上得到注释,得到B. amyloliquefaciens、B. subtilis、B. velezensis、B. nakamurai、B. paralicheniformis、Streptococcus pneumoniae和Paenibacillus sp.这7类物种,数量分别为3 237、1 599、342、36、1、70和3个。Swiss-Prot是一个精选的蛋白质序列数据库,包括蛋白质功能、结构、翻译后修饰、变异等描述,菌株BS-3中有3 328个基因的蛋白序列功能在该数据库中得到有意义的注释。
2.3.6 毒力因子分析将菌株BS-3的基因组与VFDB数据库进行比对分析,在菌株BS-3的基因组发现62个毒力因子基因,其中最大基因为1 168 bp,最小基因为56 bp,具体结果如表 5所示。这说明菌株BS-3有可能释放毒力因子来抑制病原菌的生长。
| Gene_id | Align length (bp) | Gene_id | Align length (bp) | Gene_id | Align length (bp) |
| BS3_GM000167 | 331 | BS3_GM001595 | 303 | BS3_GM002704 | 388 |
| BS3_GM000364 | 156 | BS3_GM001721 | 730 | BS3_GM002705 | 148 |
| BS3_GM000444 | 305 | BS3_GM001965 | 233 | BS3_GM002706 | 389 |
| BS3_GM000527 | 688 | BS3_GM002234 | 65 | BS3_GM002746 | 251 |
| BS3_GM000632 | 493 | BS3_GM002235 | 1 154 | BS3_GM002747 | 215 |
| BS3_GM000664 | 147 | BS3_GM002236 | 293 | BS3_GM002785 | 569 |
| BS3_GM000708 | 149 | BS3_GM002237 | 529 | BS3_GM002786 | 116 |
| BS3_GM000730 | 571 | BS3_GM002239 | 253 | BS3_GM002787 | 105 |
| BS3_GM000788 | 70 | BS3_GM002390 | 253 | BS3_GM002852 | 221 |
| BS3_GM000792 | 420 | BS3_GM002427 | 250 | BS3_GM002854 | 490 |
| BS3_GM000797 | 199 | BS3_GM002509 | 384 | BS3_GM002855 | 1 168 |
| BS3_GM000804 | 217 | BS3_GM002543 | 200 | BS3_GM002919 | 396 |
| BS3_GM000805 | 78 | BS3_GM002552 | 553 | BS3_GM003021 | 336 |
| BS3_GM000807 | 346 | BS3_GM002553 | 170 | BS3_GM003162 | 330 |
| BS3_GM000808 | 652 | BS3_GM002575 | 190 | BS3_GM003166 | 221 |
| BS3_GM001060 | 301 | BS3_GM002599 | 220 | BS3_GM003174 | 231 |
| BS3_GM001153 | 197 | BS3_GM002649 | 252 | BS3_GM003336 | 805 |
| BS3_GM001300 | 119 | BS3_GM002651 | 56 | BS3_GM003832 | 548 |
| BS3_GM001301 | 265 | BS3_GM002675 | 184 | BS3_GM004007 | 257 |
| BS3_GM001552 | 274 | BS3_GM002680 | 357 | BS3_GM004103 | 284 |
| BS3_GM001578 | 192 | BS3_GM002681 | 284 |
通过ARDB数据库注释,找到菌株BS-3的基因组有耐药基因30个,耐受药物林可霉素(Lincomycin)、氯霉素(Chloramphenicol)、磷霉素(Fosfomycin)、青霉素(Penicillin)、抗菌物(na_Antimicrobials)、喹嗪(qa_Compound)、氨基糖苷(Aminoglycoside)、万古霉素(Vancomycin)、甲氧苄啶(Trimethoprim)、四环素(Tetracycline)、杆菌肽(Bacitracin)、脱氧胆酸盐(Deoxycholate)、大环内酯(Macrolide)、链霉素(Streptomycin)、膦胺霉素(Fosmidomycin)共15种,具体结果如表 6所示。
| Gene_id | Resistance type | Antibiotic resistance |
| BS3_GM000038 | lmrb | Lincomycin |
| BS3_GM000196 | blt | Chloramphenicol, doxorubicin, fluoroquinolone, puromycin |
| BS3_GM000246 | fosb | Fosfomycin |
| BS3_GM000318 | bl2a_1 | Penicillin |
| BS3_GM000350 | bl2a_iii | Penicillin |
| BS3_GM000461 | ykkc | na_antimicrobials |
| BS3_GM000462 | ykkd | na_antimicrobials |
| BS3_GM000899 | qac | qa_compound |
| BS3_GM000900 | emre | Aminoglycoside |
| BS3_GM001037 | vanrc | Vancomycin |
| BS3_GM001235 | dfra20 | Trimethoprim |
| BS3_GM001373 | vanrg | Vancomycin |
| BS3_GM001660 | tetl | Tetracycline |
| BS3_GM001965 | vanrg | Vancomycin |
| BS3_GM002153 | baca | Bacitracin |
| BS3_GM002189 | mdtg | Deoxycholate, fosfomycin |
| BS3_GM002337 | blt | Chloramphenicol, doxorubicin, fluoroquinolone, puromycin |
| BS3_GM002486 | bcra | Bacitracin |
| BS3_GM002599 | vanrg | Vancomycin |
| BS3_GM002884 | bcra | Bacitracin |
| BS3_GM003174 | vanra | Vancomycin, teicoplanin |
| BS3_GM003488 | mphb | Macrolide |
| BS3_GM003507 | lmrb | Lincomycin |
| BS3_GM003543 | mdr | – |
| BS3_GM003613 | vanrg | Vancomycin |
| BS3_GM003625 | lmrb | Lincomycin |
| BS3_GM003690 | qac | qa_compound |
| BS3_GM003809 | ykkc | na_antimicrobials |
| BS3_GM003986 | ant6ia | Streptomycin |
| BS3_GM004012 | rosa | Fosmidomycin |
| 注:−:无 Note: −: None | ||
因芽孢杆菌属内的种间亲缘关系较近,不能采用16S rRNA基因来分析芽孢杆菌属物种间的进化关系。近年来报道可以用gyrA基因区分枯草芽孢杆菌种间的差异,将菌株BS-3与9株Bacillus sp.来源的GyrA蛋白序列构建进化树,发现菌株BS-3与FZB42、UCMB5113、Y2来源的GyrA蛋白聚在一起(图 4),说明其亲缘关系非常相近,同属于解淀粉芽孢杆菌。

|
| 图 4 基于BS-3及近缘种GyrA蛋白氨基酸序列构建进化树 Figure 4 Phylogenetic tree reconstructed based on GyrA protein sequence to analyse the relationship between BS-3 and its related bacterial species 注:括号中的序号为相关菌株GenBank的登录号;分支点上的数字表示Bootstrap支持率;标尺表示1%的序列进化差异 Note: The numbers in parentheses represent the sequence accession numbers in GenBank; The numbers in each branch points denote the percentages supported by bootstrap; The scale presents 1% sequence variance |
|
|
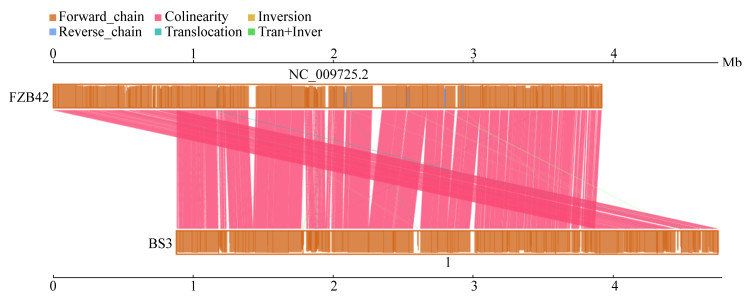
将BS-3与B. amyloliquefaciens subsp. plantarum str. FZB42的全基因组进行共线性分析,观察两者基因组之间序列的插入、缺失等信息(图 5)。从图 5中可以看出,2株基因组高度相似,但也存在翻转、易位等基因组重排。
|
| 图 5 B. amyloliquefaciens BS-3与FZB42的共线性分析 Figure 5 Synteny analysis of B. amyloliquefaciens BS-3 and FZB42 |
|
|
次级代谢产物是微生物在一定的生长时期,以初级代谢产物为前体合成的一系列大分子生长非必需物质,用anti-SMASH对菌株BS-3基因组进行次级代谢产物分析,预测得到10个次级代谢产物基因簇(表 7),并从中发现4类具有抗菌活性的物质:多烯类(Bacillaene)、丰原素(Fengycin)、儿茶酚型嗜铁素(Bacillibactin)和表面活性肽(Surfactin)。其中,Cluster 3与BGC0001089 c1来源的Bacillaene合成基因簇相似性为100%;Cluster 4与BGC0001095 c1来源的Fengycin合成基因簇相似性为93%;Cluster 7与BGC0000309 c1来源的Bacillibactin合成基因簇相似性为100%;Cluster 10与BGC0000433 c1来源的Surfactin合成基因簇相似性为86%;Cluster 8与BGC0001184 c1来源的杆菌溶素(Bacilysin)合成基因簇相似性为100%。比对发现菌株BS-3基因组中还存在4种功能未知的基因簇(Cluster 2、5、6、10),其中Lantipeptide 1种、T3PKS (Type Ⅲ PKS)类1种、萜类(Terpene) 2种,这表明菌株BS-3中可能存在合成新抑菌物质的基因簇,具有较大的农业和药业应用潜力。
| Cluster ID | Nucleotide length (bp) | Gene number | Cluster type | Ratio of genes show similarity (%) | Most similar known cluster |
| Cluster1 | 41 245 | 48 | Otherks | BGC0000693 c1(7%) | Butirosin |
| Cluster2 | 20 744 | 27 | Terpene | − | − |
| Cluster3 | 102 705 | 52 | Transatpks-NRPS | BGC0001089 c1(100%) | Bacillaene |
| Cluster4 | 110 379 | 76 | Transatpks-NRPS | BGC0001095 c1(93%) | Fengycin |
| Cluster5 | 21 884 | 24 | Terpene | − | − |
| Cluster6 | 41 107 | 55 | T3PKS | − | − |
| Cluster7 | 66 776 | 70 | Bacteriocin-NRPS | BGC0000309 c1(100%) | Bacillibactin |
| Cluster8 | 41 419 | 47 | Other | BGC0001184 c1(100%) | Bacilysin |
| Cluster9 | 65 411 | 49 | NRPS | BGC0000433 c1(86%) | Surfactin |
| Cluster10 | 22 598 | 20 | Lantipeptide | − | − |
| 注:−:无 Note: −: none | |||||
从2.4中选择3株参与gryA分析的芽孢杆菌近缘种,对基因组中已知功能的基因簇进行统计,统计结果如表 8所示,除丁酰苷菌素(Butirosin)外,其他近缘菌种基因组中均有BS-3中含有的化合物,而Butirosin与菌株BGC0000693 c1来源的基因簇相似性仅为7%,说明该基因簇可能合成新的抑菌物质,有待今后进一步研究。
| Compound | Enzyme | Y2 | BS-3 | FZB42 | B. subtilis 168 |
| Surfactin | NRPS | 1 | 1 | 1 | 1 |
| BacillomycinD | NRPS/PKS | 1 | − | 1 | − |
| Fengycin | NRPS | 1 | 1 | 1 | 1 |
| Putative peptide | NRPS | − | − | 1 | − |
| Bacillibactin | NRPS | 1 | 1 | 1 | 1 |
| Bacilysin/anticapsin | NRPS | 1 | 1 | 1 | 1 |
| Macrolactin | PKS | 1 | − | 1 | − |
| Bacillaene | NRPS/PKS | 1 | 1 | 1 | 1 |
| Difficidin | PKS | 1 | − | 1 | − |
| Amylocyclicin | RPS | 1 | − | 1 | − |
| Mersacidin | RPS | 1 | − | 1 | − |
| Butirosin | Otherks | − | 1 | − | − |
| 注:−:无 Note: −: none | |||||
据报道芽孢杆菌中的碱性丝氨酸蛋白(Alkaline Serine Protease)能够有效地杀死线虫,同时解淀粉芽孢杆菌FZB42和解淀粉芽孢杆菌Y2具有减少植物根部的根结线虫的能力,基因分析菌株BS-3中的碱性丝氨酸蛋白酶(BS3_GM000172)与解淀粉芽孢杆菌FZB42在氨基酸水平上相似性很高,这说明菌株BS-3可能有具有减少根部线虫的能力。
3 讨论随着农业的快速发展,农药用量不断增加,使用化学农药所带来的高残留、高污染及对人畜的危害等问题逐渐突显。兼具安全、绿色、高效特性的绿色防控技术成为当下研究热点,其中应用生物拮抗菌来防治各种病害被证实为一种安全有效的新方法。芽孢杆菌在生物防治领域对农作物病害有很好的防治效果,同时其在促进植物生长[32]、降解除草剂、治理水污染等方面也具有较好的作用,因而备受科研工作者关注[33]。
本团队从橡胶根部分离到了一株内生细菌BS-3,经过拮抗试验发现菌株BS-3对橡胶树褐根病菌、尖孢炭疽病菌、小孢拟盘多毛孢、橡胶疫霉病菌的生长均具有良好的抑制效果。传统的实验和鉴定方法很难全面地分析芽孢杆菌抗菌物质,也不能充分挖掘其全部抗菌基因。为了深入研究该菌,本研究利用全基因组测序和生物信息学分析技术得到了菌株BS-3的基因组序列,确定菌株基因组大小为3 870 130 bp,共编码基因4 161个,同时将基因组数据与GO、COG、KEGG、NR等数据库进行比对分析,完成菌株BS-3基因组功能注释及数据统计工作,并采用gryA基因确定了BS-3的分类地位,并从分子生物学的角度探究其抑菌作用中可能发挥作用的基因簇。
16S rRNA基因序列是种属鉴定和细菌系统发育的分类工具,但解淀粉芽孢杆菌和枯草芽孢杆菌亲缘关系很近,是形态特征相似的芽孢杆菌属,采用16S rRNA基因序列无法准确区分[34]。在基因组测序之后,解旋酶A亚基编码基因以及组氨酸激酶基因分子标记基因可以进一步确定两者的分类[35]。对菌株BS-3的全基因组信息进行分析,并与其他已测序的近缘解淀粉芽孢杆菌比较,发现菌株BS-3基因组大小与其他已测序的近缘解淀粉芽孢杆菌较为接近,GC含量也较为相似;菌株BS-3解旋酶A亚基编码基因的蛋白序列与相似物种构建进化树,发现其与解淀粉芽孢杆菌FZB42和解淀粉芽孢杆菌Y2亲缘关系非常相近,同属于解淀粉芽孢杆菌。
大多数病原真菌细胞壁的主要成分是几丁质,几丁质酶能催化几丁质水解生成N乙酰氨基葡萄糖,破坏真菌细胞壁的结构完整性,从而抑制一些真菌孢子萌发和菌丝生长[36]。在生物防治过程中,除几丁质酶外,还有葡聚糖酶、纤维素酶、蛋白酶等能够与几丁质酶协同作用,使抑菌效果更加显著[37]。本研究从菌株BS-3基因组中共获得CAZy家族的编码基因140个,包括糖苷水解酶家族的49个、糖苷转移酶家族的蛋白36个、碳水化合物酯酶17个、多糖裂解酶3个、辅助酶4个、碳水化合物结合组件31个。这些CAZy家族包含了β-1, 3-葡聚糖酶、几丁质酶等细胞壁降解酶的相关基因,推测菌株BS-3降解病原真菌的细胞壁为其抑菌机理之一。
采用antiSMASH工具预测菌株BS-3的次级代谢产物基因簇,得到脂肽类和聚酮类抗生素基因簇10个,包括4个未知功能的基因簇、5个相似度较高的抗生素合成基因簇(Bacillaene、Fengycin、Bacillibactin、Bacilysin、Surfactin)和1个相似度仅为7%的抗生素基因簇(Butirosin)。Bacillaene是一种对细菌和真菌均有抑制作用的多聚烯类抗生素,能抑制蛋白合成;Fengycin对植物病原真菌、细菌和线虫都有抗性,主要由肽链以内酯键与β-羟基脂肪酸结合而成[34]。Bacillibactin能竞争性结合环境中对病原菌生长和发挥活性必需的可溶性铁离子,是一种对铁离子具有高亲和性的螯合剂[35]。Bacilysin是目前已知芽胞杆菌合成结构最简单的肽类抗菌物质之一,由L-氨基酸抗荚膜菌素残基和L-丙氨酸残基组成[38]。Wu等报道菌株B. velezensis FZB42产生的杆菌溶素能杀死铜绿微裹藻,抑藻率达98.78%[39]。Surfactin是一种广谱抗菌物质,有很强的表面活性,而且能抗病毒和支原体[35];López等研究报道Surfactin与Fengycin同时作用于菜豆和番茄叶子部位,可显著降低灰霉病的发病率[40]。Butirosin是一种氨基糖苷类抗生素,对革兰氏阴性菌特别有效,本研究中发现的Butirosin抗生素基因簇与BGC0000693 c1来源的最高相似度仅为7%,说明该基因簇可能合成新的抑菌物质,有待今后进一步研究。此外,菌株BS-3中存在碱性丝氨酸蛋白酶基因,而有报道证明芽孢杆菌中的碱性丝氨酸蛋白能够有效杀死线虫[41],推测菌株BS-3分泌抑菌物质为其抑菌机理之一。
4 结论本文采用第二代Illumina与第三代PacBio平台相结合的测序技术,对生防菌BS-3进行全基因组测序,得到了菌株BS-3的基因组序列信息。通过比较全基因组和其中的GyrA蛋白序列,确定芽孢杆菌BS-3的分类地位为解淀粉芽孢杆菌;发现BS-3基因组中存在5种重要的次级代谢产物基因簇和一种新的抗生素基因簇(Butirosin),同时从BS-3基因组中发现能够降解病原菌细胞壁的β-1, 3-葡聚糖酶、几丁质酶和抑菌蛋白(碱性丝氨酸蛋白)的相关基因,预测菌株BS-3能够通过产生多种拮抗活性化合物、细胞壁水解酶类以及抑菌蛋白实现防病效果,在农业方面具有很大的应用潜力。
| [1] |
Yang SY, Wei J, Zheng XR. Optimization of fermentation medium for the production of antimicrobial substances by Bacillus amyloliquefaciens[J]. Food Science, 2015, 36(11): 150-156. (in Chinese) 杨胜远, 韦锦, 郑燮茹. 解淀粉芽孢杆菌抗菌物质发酵培养基的优化[J]. 食品科学, 2015, 36(11): 150-156. DOI:10.7506/spkx1002-6630-201511029 |
| [2] |
Jin Q, Xiao M. Novel antimicrobial peptides: surfactin, iturin and fengycin[J]. Journal of Microbes and Infections, 2018, 13(1): 56-64. (in Chinese) 金清, 肖明. 新型抗菌肽: 表面活性素、伊枯草菌素和丰原素[J]. 微生物与感染, 2018, 13(1): 56-64. DOI:10.3969/j.issn.1673-6184.2018.01.010 |
| [3] |
Zhou JW, McClean S, Thompson A, Zhang Y, Shaw C, Rao PF, Bjourson AJ. Purification and characterization of novel antimicrobial peptides from the skin secretion of Hylarana guentheri[J]. Peptides, 2006, 27(12): 3077-3084. DOI:10.1016/j.peptides.2006.08.007 |
| [4] |
Yao JM, Tian YP. Isolation and identification of antimicrobial peptides produced by Bacillus amyloliquefaciens and characterization of their antibacterial spectra[J]. Food Science, 2020, 41(16): 126-131. (in Chinese) 姚佳明, 田亚平. 解淀粉芽孢杆菌抑菌肽的分离鉴定及其抑菌谱表征[J]. 食品科学, 2020, 41(16): 126-131. DOI:10.7506/spkx1002-6630-20190505-020 |
| [5] |
Gao SF, Liu AQ, Sang LW, Sun SW, Gou YF, Wang Z, Meng QQ. Whole genome sequencing and comparative genomics analysis of Bacillus subtilis VD18R19 with biocontrol activity against pepper phytophtora rot disease[J]. Chinese Journal of Tropical Crops, 2018, 39(10): 2021-2027. (in Chinese) 高圣风, 刘爱勤, 桑利伟, 孙世伟, 苟亚峰, 王政, 孟倩倩. 胡椒瘟病生防菌Bacillus subtilis VD18R19全基因组测序及比较基因组学分析[J]. 热带作物学报, 2018, 39(10): 2021-2027. DOI:10.3969/j.issn.1000-2561.2018.10.019 |
| [6] |
Arrebola E, Jacobs R, Korsten L. Iturin A is the principal inhibitor in the biocontrol activity of Bacillus amyloliquefaciens PPCB004 against postharvest fungal pathogens[J]. Journal of Applied Microbiology, 2010, 108(2): 386-395. DOI:10.1111/j.1365-2672.2009.04438.x |
| [7] |
Wong JH, Hao J, Cao Z, Qiao M, Xu H, Bai Y, Ng TB. An antifungal protein from Bacillus amyloliquefaciens[J]. Journal of Applied Microbiology, 2008, 105(6): 1888-1898. DOI:10.1111/j.1365-2672.2008.03917.x |
| [8] |
An JY, Zhu WJ, Liu Y, Zhang XM, Sun LJ, Hong PZ, Wang YL, Xu CH, Xu DF, Liu HM. Purification and characterization of a novel bacteriocin CAMT2 produced by Bacillus amyloliquefaciens isolated from marine fish Epinephelus areolatus[J]. Food Control, 2015, 51: 278-282. DOI:10.1016/j.foodcont.2014.11.038 |
| [9] |
Zhang BY, Zhu TH, Han S, Wang Y, Li SJ, Qiao TM. Whole-genome sequencing and analysis of Streptomyces sampsonii KJ40[J]. Microbiology China, 2018, 45(4): 805-818. (in Chinese) 张博阳, 朱天辉, 韩珊, 王莹, 李姝江, 谯天敏. 桑氏链霉菌KJ40全基因组测序及分析[J]. 微生物学通报, 2018, 45(4): 805-818. |
| [10] |
Besemer J, Lomsadze A, Borodovsky M. GeneMarkS: a self-training method for prediction of gene starts in microbial genomes. implications for finding sequence motifs in regulatory regions[J]. Nucleic Acids Research, 2001, 29(12): 2607-2618. DOI:10.1093/nar/29.12.2607 |
| [11] |
Saha S, Bridges S, Magbanua ZV, Peterson DG. Empirical comparison of ab initio repeat finding programs[J]. Nucleic Acids Research, 2008, 36(7): 2284-2294. DOI:10.1093/nar/gkn064 |
| [12] |
Benson G. Tandem repeats finder: a program to analyze DNA sequences[J]. Nucleic Acids Research, 1999, 27(2): 573-580. DOI:10.1093/nar/27.2.573 |
| [13] |
Lowe TM, Eddy SR. tRNAscan-SE: a program for improved detection of transfer RNA genes in genomic sequence[J]. Nucleic Acids Research, 1997, 25(5): 955-964. DOI:10.1093/nar/25.5.955 |
| [14] |
Lagesen K, Hallin P, Rødland EA, Stærfeldt HH, Rognes T, Ussery DW. RNAmmer: consistent and rapid annotation of ribosomal RNA genes[J]. Nucleic Acids Research, 2007, 35(9): 3100-3108. DOI:10.1093/nar/gkm160 |
| [15] |
Gardner PP, Daub J, Tate JG, Nawrocki EP, Kolbe DL, Lindgreen S, Wilkinson AC, Finn RD, Griffiths-Jones S, Eddy SR, et al. Rfam: updates to the RNA families database[J]. Nucleic Acids Research, 2009, 37(Database issue): D136-D140. |
| [16] |
Nawrocki EP, Kolbe DL, Eddy SR. Infernal 1.0: inference of RNA alignments[J]. Bioinformatics, 2009, 25(10): 1335-1337. DOI:10.1093/bioinformatics/btp157 |
| [17] |
Hsiao W, Wan I, Jones SJ, Brinkman FSL. IslandPath: aiding detection of genomic islands in prokaryotes[J]. Bioinformatics, 2003, 19(3): 418-420. DOI:10.1093/bioinformatics/btg004 |
| [18] |
Akhter S, Aziz RK, Edwards RA. PhiSpy: a novel algorithm for finding prophages in bacterial genomes that combines similarity- and composition-based strategies[J]. Nucleic Acids Research, 2012, 40(16): e126. DOI:10.1093/nar/gks406 |
| [19] |
Ge RQ, Mai GQ, Wang P, Zhou ML, Luo YX, Cai YP, Zhou FF. CRISPRdigger: detecting CRISPRs with better direct repeat annotations[J]. Scientific Reports, 2016, 6(1): 1-10. DOI:10.1038/s41598-016-0001-8 |
| [20] |
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT, et al. Gene Ontology: tool for the unification of biology[J]. Nature Genetics, 2000, 25(1): 25-29. DOI:10.1038/75556 |
| [21] |
Kanehisa M, Goto S, Kawashima S, Okuno Y, Hattori M. The KEGG resource for deciphering the genome[J]. Nucleic Acids Research, 2004, 32(Database issue): D277-D280. |
| [22] |
Kanehisa M, Goto S, Hattori M, Aoki-Kinoshita KF, Itoh M, Kawashima S, Katayama T, Araki M, Hirakawa M. From genomics to chemical genomics: new developments in KEGG[J]. Nucleic Acids Research, 2006, 34(Database issue): D354-D357. |
| [23] |
Tatusov RL, Fedorova ND, Jackson JD, Jacobs AR, Kiryutin B, Koonin EV, Krylov DM, Mazumder R, Mekhedov SL, Nikolskaya AN, et al. The COG database: an updated version includes eukaryotes[J]. BMC Bioinformatics, 2003, 4(1): 41. DOI:10.1186/1471-2105-4-41 |
| [24] |
Li WZ, Jaroszewski L, Godzik A. Tolerating some redundancy significantly speeds up clustering of large protein databases[J]. Bioinformatics, 2002, 18(1): 77-82. DOI:10.1093/bioinformatics/18.1.77 |
| [25] |
Cantarel BL, Coutinho PM, Rancurel C, Bernard T, Lombard V, Henrissat B. The carbohydrate-active EnZymes database (CAZy): an expert resource for glycogenomics[J]. Nucleic Acids Research, 2009, 37(Database issue): D233-D238. |
| [26] |
Eichinger V, Nussbaumer T, Platzer A, Jehl MA, Arnold R, Rattei T. Effective DB-updates and novel features for a better annotation of bacterial secreted proteins and Type Ⅲ, Ⅳ, Ⅵ secretion systems[J]. Nucleic Acids Research, 2016, 44(Database issue): D669-D674. |
| [27] |
Chen LH, Xiong ZH, Sun LL, Yang J, Jin Q. VFDB 2012 update: toward the genetic diversity and molecular evolution of bacterial virulence factors[J]. Nucleic Acids Research, 2012, 40(Database issue): D641-D645. |
| [28] |
Urban M, Pant R, Raghunath A, Irvine AG, Pedro H, Hammond-Kosack KE. The Pathogen-Host Interactions database (PHI-base): additions and future developments[J]. Nucleic Acids Research, 2015, 43(Database issue): D645-D655. DOI:10.1093/nar/gku1165 |
| [29] |
Bairoch A, Apweiler R. The SWISS-PROT protein sequence database and its supplement TrEMBL in 2000[J]. Nucleic Acids Research, 2000, 28(1): 45-48. DOI:10.1093/nar/28.1.45 |
| [30] |
Medema MH, Blin K, Cimermancic P, De Jager, Zakrzewski P, Fischbach MA, Weber T, Takano E, Breitling R. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences[J]. Nucleic Acids Research, 2011, 39(Web Server issue): W339-W346. |
| [31] |
Yan YC. Genomic sequence analysis of Bacillus amyloliquefaciens PEBA20 and characteristics of the sfp gene[D]. Tai'an: Master's Thesis of Shandong Agricultural University, 2016(in Chinese) 闫永才. 解淀粉芽孢杆菌PEBA20基因组序列分析及sfp基因相关特性研究[D]. 泰安: 山东农业大学硕士学位论文, 2016 |
| [32] |
Jiang W, Wu QL, Dou X, Guan ZB, Cai YJ, Liao XR. Characterization of a phosphorus-solubilizing alkaline phosphatase AP3 cloned from Bacillus amyloliquefaciens YP6[J]. Microbiology China, 2018, 45(7): 1408-1415. (in Chinese) 江威, 吴秋兰, 窦欣, 管政兵, 蔡宇杰, 廖祥儒. 解淀粉芽孢杆菌YP6中碱性磷酸酯酶AP3的酶学性质及其溶磷作用[J]. 微生物学通报, 2018, 45(7): 1408-1415. |
| [33] |
Su ZC, Xiong RK, Luo XY. Function and product development of Bacillus amyloliquefaciens[J]. Pesticide Science and Administration, 2019, 40(6): 21-30. (in Chinese) 苏正川, 熊仁科, 罗小艳. 解淀粉芽孢杆菌的作用及其产品开发[J]. 农药科学与管理, 2019, 40(6): 21-30. DOI:10.3969/j.issn.1002-5480.2019.06.007 |
| [34] |
Jin ZX, Qin Y, Wang JL, Du NN, Li ZF, Chen WM. Classification and identification of Bacillus sp. SK007 with biocontrol activity[J]. Microbiology China, 2019, 46(10): 2591-2600. (in Chinese) 靳泽星, 秦娅, 王洁丽, 杜妮妮, 李哲斐, 陈卫民. 芽孢杆菌SK007的分类鉴定及其拮抗植物病原菌的功能分析[J]. 微生物学通报, 2019, 46(10): 2591-2600. |
| [35] |
Qi JM, Sun SS, Zhang DX, Xu ZW, Xu YP. Identification and biocontrol activity analysis of Bacillus sp. BS-6 based on genome-wide data[J]. Biotechnology Bulletin, 2019, 35(10): 111-118. (in Chinese) 戚家明, 孙杉杉, 张东旭, 徐志文, 徐延平. 芽孢杆菌BS-6基于全基因组数据的分类鉴定及拮抗能力分析[J]. 生物技术通报, 2019, 35(10): 111-118. |
| [36] |
Zhang BY, Zhu TH, Han S, Wang Y, Li SJ, Qiao TM. Cloning, expression and antibacterial functions of ChiKJ40, a chitinase gene from Streptomyces sampsonii[J]. Microbiology China, 2018, 45(5): 1016-1026. (in Chinese) 张博阳, 朱天辉, 韩珊, 王莹, 李姝江, 谯天敏. 桑氏链霉菌几丁质酶ChiKJ40基因的克隆表达及其抑菌作用[J]. 微生物学通报, 2018, 45(5): 1016-1026. |
| [37] |
Chen XY, Li JB, Lin Y, Li K, Yu L. Application of β-1, 3-glucanase and chtinase to the preservation of tropical fruit[J]. Science and Technology of Food Industry, 2008, 29(5): 294-296. (in Chinese) 陈小云, 李坚斌, 林莹, 李凯, 于兰. β-1, 3-葡聚糖酶和几丁质酶在热带水果保鲜中的应用[J]. 食品工业科技, 2008, 29(5): 294-296. |
| [38] |
Wu LM, Li X, Wu HJ, Gao XW. Research advances on bacilysin from Bacillus[J]. Journal of Nanjing Agricultural University, 2018, 41(5): 778-783. (in Chinese) 吴黎明, 李曦, 伍辉军, 高学文. 芽胞杆菌抗菌二肽溶杆菌素的研究进展[J]. 南京农业大学学报, 2018, 41(5): 778-783. |
| [39] |
Wu LM, Wu HJ, Chen LN, Xie SS, Zang HY, Borriss R, Gao XW. Bacilysin from Bacillus amyloliquefaciens FZB42 has specific bactericidal activity against harmful algal bloom species[J]. Applied and Environmental Microbiology, 2014, 80(24): 7512-7520. DOI:10.1128/AEM.02605-14 |
| [40] |
López G, Diaz-Cárdenas C, Shapiro N, Woyke T, Kyrpides NC, Alzate JD, González LN, Restrepo S, Baena S. Draft genome sequence of Pseudomonas extremaustralis strain USBA-GBX 515 isolated from Superparamo soil samples in Colombian Andes[J]. Standards in Genomic Sciences, 2017, 12: 78. DOI:10.1186/s40793-017-0292-9 |
| [41] |
Xu MZ. Comparative genomics analysis of Bacillus amyloliquefaciens TJ and LJ1[D]. Tianjin: Master's Thesis of Tianjin Agricultural University, 2015(in Chinese) 徐明珠. 解淀粉芽孢杆菌TJ与LJ1比较基因组学分析[D]. 天津: 天津农学院硕士学位论文, 2015 |
 2021, Vol. 48
2021, Vol. 48




