扩展功能
文章信息
- 张玲艳, 宋丽丽, 贾伟娟, 王学理
- ZHANG Lingyan, SONG Lili, JIA Weijuan, WANG Xueli
- 蜡样芽孢杆菌检测方法的研究进展
- Research progress on detection methods of Bacillus cereus
- 微生物学通报, 2021, 48(4): 1360-1372
- Microbiology China, 2021, 48(4): 1360-1372
- DOI: 10.13344/j.microbiol.china.200228
-
文章历史
- 收稿日期: 2020-03-13
- 接受日期: 2020-09-07
- 网络首发日期: 2020-12-04
蜡样芽孢杆菌(Bacillus cereus)是一种兼性厌氧的杆状革兰氏阳性细菌,该菌属于芽孢杆菌科蜡样芽孢杆菌属,在土壤、水、植物、饲料以及各种食品中大量存在;该菌可以产生内生孢子,对环境具有很强的适应性,能够抵抗高温、紫外线、电磁辐射和有害化学物质等不利条件;研究发现该菌与苏云金杆菌、炭疽杆菌的生物学特性十分相似,并且DNA的相似性也极高[1-2]。
近年来,大量的无毒蜡样芽孢杆菌菌株被应用于许多方面。该菌在土壤中的大量分布使其成为优势细菌,这为其在土壤定殖中的应用奠定了坚实的基础[3]。同时,该菌也是一种已知的植物促生菌(Plant Growth Promoting Rhizobacteria,PGPR)和生防菌,有些菌株可以通过自身或诱导植物合成、分泌各种有益激素,进一步提高植物的生长率[4-5]。无毒菌株作为益生菌被添加到动物饲料中可减少胃肠道疾病,增强动物自身对外界环境的抵抗能力,促进动物生长[6]。此外,蜡样芽孢杆菌分泌的可溶性胞外聚合物(Soluble Extracellular Polymeric Substances,s-EPS)作为一种新型双生物功能的缓蚀阻垢材料,具有潜在的开发价值[7]。
虽然蜡样芽孢杆菌作为有益菌被广泛使用,但其同时也是一种重要的食源性条件致病菌,给人类健康和养殖业带来了一定的影响。据国内外报道,几乎所有种类食品中都曾检测到蜡样芽孢杆菌[8-9],其为植物和动物的主要污染源,而且不同的致病菌株会引起不同疾病。产毒菌株可引起人类和动物食物中毒,中毒后还可导致人眼部发炎、心内膜炎、菌血症[10-12]等,但最常见的是引起腹泻型和呕吐型2种类型的食物中毒。在养殖过程中,该菌可使家畜感染,甚至引起死亡[13-15]。目前,动物感染该菌的病例日渐增多,在通辽地区已发现多起牛猝死病例。
本实验室从通辽市某肉牛养殖场病死牛体内分离出一株蜡样芽孢杆菌,经PCR检测该菌含有hbl、nhe、entFM等毒素基因以及管家基因groEL,而且动物致病性试验结果发现该菌也具有较强的致病性[15]。由此可见,建立快速和精准的蜡样芽孢杆菌检测方法可为该病的诊断与预防带来便利。本文对该菌的检测方法进行综述,以期为今后的检测提供参考,并为下一步研究工作奠定基础。
1 传统的检测方法传统的细菌检测方法是通过其生物学特性进行鉴定,主要包括增菌培养、分离纯化、菌落形态观察、革兰氏染色镜检、生化鉴定和生化分型几个步骤。
1.1 传统的培养方法目前美国食品药品监督管理局、国际标准化组织及美国分析化学家协会推荐MYPA和PemBA这2种标准细菌培养基来分离蜡样芽孢杆菌,但由于非典型形态的菌落产生,而且卵磷脂沉淀环的扩散重叠掩盖了部分蜡样芽孢杆菌,导致蜡样芽孢杆菌不能被准确地鉴别出来,同时菌落的交连也给该菌的鉴别和计数带来了很大的困难[16]。
为避免试验结果的不准确性,研究者已经制备出各种不同的显色培养基,可使该菌产生其特有的颜色与菌落形态,另外,显色物质只沉积在菌落上而不扩散在培养基中,因此更易观察计数。Kabir等[17]通过2种标准培养基(MYPA和Pemba)和2种显色电镀培养基(Bacara和Brilliance)对16株菌的最佳生长状态和计数进行了评价,结果表明,显色电镀培养基(Bacara and Brilliance)优于常规标准培养基(MYPA和Pemba),而且Brilliance对分离该菌具有更高的效率和选择性。滕昆仑等[18]用自己研制的蜡样芽孢杆菌显色培养基HK和其他4种从市场购买的培养基进行对比,结果显示,使用HK进行检测的效果更加显著。除了显色培养技术外,Chon等[19]采用MYPA检测60种食物中污染的蜡样芽孢杆菌后,又将添加头孢呋辛的甘露醇-卵黄-多粘菌素B琼脂(Cefu-MYPA)与原MYPA的检测性能进行了比较,结果表明Cefu-MYPA的检测结果更加显著。Kang等[20]发现在多粘菌素-蛋黄-甘露醇-溴百里香酚蓝琼脂(PemBA)中添加甲氧苄啶可以产生协同效应,同时提高对该菌的选择性。
1.2 生化检测方法生化鉴定主要是根据各种菌在生长时发生的不同生化反应来进行判断。蜡样芽孢杆菌可使过氧化氢酶试验结果呈现阳性,使明胶液化、硝酸盐还原、酪氨酸分解,不能发酵甘露醇、木糖及阿拉伯糖,但在厌氧环境下能够使葡萄糖发酵[8]。Savić等[21]在鉴定从人类粪便、食物和环境中分离的蜡样芽孢杆菌时,首先通过选择性培养基、经典生化试验进行初步鉴定,然后采用Bal基因特异性引物通过PCR检测来进一步确认,结果显示,所有菌株的经典生化检测结果均为阳性,PCR也扩增出533 bp大小的片段。该试验中使用的方法对菌种的检测结果比较精确,但是经典的生化检测方法步骤复杂,需要大量时间。
除了传统的方法外,全自动微生物鉴定系统在许多国家已经成为一种广泛使用的技术。目前经常使用的有PhoenixTM 100和VITEK2这2种。PhoenixTM 100能够对100个样本同时进行51项生化反应和药敏试验,但是仅有2种检测卡。VITEK2仅能发生30项反应,共有14种检测卡。黄晶菁等[22]使用VITEK2对16株蜡样芽孢杆菌进行检测,为确定该方法的有效性,将其检测结果和所需时间与传统方法进行对比的结果显示,使用VITEK2的准确度更高,而且检测时间也大大缩短。由此可知,使用全自动微生物鉴定系统比传统的生理生化检验方法更简单、快速、节省人力,检测结果更加精准且适合检测大量的样品。
通过上述传统的培养方法和生化检测方法可初步鉴别出蜡样芽孢杆菌,但是并不能将无毒的蜡样芽孢杆菌菌株和产毒的蜡样芽孢杆菌菌株区分,因此需要借助其他方法来进一步确定。
2 分子生物学检测方法 2.1 PCR检测近几年利用特异性靶基因的PCR检测方法被广泛应用,关于特异性靶基因的研究也逐渐增多,其中靶基因主要有16S rRNA、tuf、hblA、cytK、entFM、nheA和ces等。
通用的16S rRNA基因具有高度保守性,研究表明16S rRNA基因普遍应用于鉴别细菌,能够了解细菌之间的进化距离和相互关系[23]。然而,蜡样芽孢杆菌群的16S rRNA基因相似性非常高,只能将他们鉴定为同一个物种,并不能很好地区分蜡样芽孢杆菌群,因此需要与其他方法结合进行鉴定[24]。gyrB、rpoB、groEL、tuf均为蜡样芽孢杆菌的管家基因,其中groEL基因可编码分子伴侣蛋白,tuf为延伸因子,rpoB基因和gyrB基因分别参与RNA聚合酶β亚基和DNA旋转酶β亚基的编码[25]。卢佳琦等[26]为鉴定一株疑似蜡样芽孢杆菌的菌株,通过设计与合成16S rRNA基因通用引物、gyrB基因特异性引物,使用普通PCR方法进行检测,将其产物在测序后进行同源性比较和进化树分析,研究结果显示PCR扩增片段均与预期基因片段大小相符,16S rRNA基因片段测序大小为1 401 bp,而且进行16S rRNA基因序列分析后仅可将其归为芽孢杆菌属;gyrB基因序列分析结果与标准菌株(ATCC14579)相似性高达100%,所以结合16S rRNA基因和gyrB基因序列分析可准确鉴定该菌株为Bacillus cereus。可见,在普通PCR检测方法中,使用通用基因只能归为芽孢杆菌属,不能确定为何种菌;而使用管家基因检测则特异性强,能够实现与其他芽孢杆菌的区分和鉴定。
有毒菌株在一定的条件下能够产生呕吐毒素和腹泻型肠毒素2种毒素,因此可以通过其毒力基因进行检测。呕吐毒素(Cereulide)是一种环形十二烷基肽,其结构为[D-O-Leu-D-Ala-L-O-Val-L-Val]3[27]。ces为控制合成Cereulide的基因簇,大小为24 kb,位于pBCE4810巨型质粒上,其与炭疽芽孢杆菌pXO1质粒具有高度的相似性[28]。目前发现主要有溶血素BL (Hemolysin BL,Hbl)、非溶血性的肠毒素(Nonhaemolytic Enterotoxin,Nhe)、细胞毒素K (Cytotoxin K,cyt K)、肠毒素T (Enterotoxin,Bce T)、肠毒素II (Potential Enterotoxins Hemolysin II,HlyII)这5种肠毒素[29]。Hbl是由溶血毒素亚基L1、L2及亚基B结合而成的三组分蛋白毒素,Hbl基因由hblA、hblB、hblC和hblD这4个毒力基因组成,因未曾检测到hblB的转录现象,因此被称为假基因[30]。Nhe是三组分蛋白毒素,分别由毒力基因nheA、nheB、nheC编码相应蛋白[31]。细胞毒素K是一种由cytK基因编码的单组分蛋白毒素,bceT和hlyII分别编码肠毒素T和肠毒素II[32]。蜡样芽孢杆菌的多数毒力基因均由转录激活剂PlcR参与调控[33]。Oltuszak-Walczak等[34]针对PlcR区和cytK设计引物,成功检测出蜡样芽孢杆菌。但以毒力基因为目的基因的单重PCR检测方法存在一定的问题。Adame-Gómez等[35]为了了解从墨西哥西南部奶酪中分离出的23株蜡样芽孢杆菌的产毒特性,设计毒力基因cytK、nheA、nheB、C、hlbB、hlbC、hlbD的引物,使用普通PCR方法进行检测,最后发现每株菌检测出的毒力基因不完全相同。由此可见,鉴定某株蜡样芽孢杆菌使用普通PCR检测某一个或几个毒力基因的方法并不准确。除此之外,苏云金芽孢杆菌中也能够产生HBL毒素、NHE毒素,因此,使用普通PCR鉴定并不能准确区分种属。
普通PCR技术节约时间,操作步骤相对于其他检测方法较为简单,但是此方法的检出率低且存在漏检问题,因此需要从多个方面来进行检测。
2.2 多重PCR检测普通多重PCR检测(Multiple PCR,mPCR)指的是在同一个体系中放入多对引物进行同时扩增的一种聚合酶链式反应,其工作原理及步骤和普通PCR大致相同。
Kumar等[36]为鉴别19株蜡样芽孢杆菌群菌株,设计了hblA、nheA、cytK、cryIA和pag这5对基因的引物,同时加入了扩增内标(Internal Amplification Control,IAC),利用多重PCR与普通PCR分别进行了检测,结果发现蜡状芽孢杆菌、苏云金芽孢杆菌以及炭疽芽孢杆菌可通过mPCR方法同时检测出,而且蜡样芽孢杆菌的检出限是1×103 CFU/mL;在mPCR混合物中加入的IAC DNA虽然未改变检测的检测限,但是避免了假阴性结果的出现;将mPCR与普通PCR方法进行比较,发现使用mPCR检测不仅节约成本,其结果也更加快速、可靠。
mPCR不仅用于同时检测蜡样芽孢杆菌群的杆菌,还可用于鉴定其他不同种类的细菌。Razei等[37]通过设计nheB、nheC、hly、C基因的4对引物,采用mPCR技术同时检测出食品中的增多性李斯特菌、蜡样芽孢杆菌和空肠弯曲菌,而且检测限分别为5、4和3 pg,研究结果表明3种细菌在食物中毒中普遍存在,因此多重PCR技术可作为一种流行病学检测的鉴定方法。Yu等[38]为了能够同时检测到阪崎肠杆菌、蜡样芽孢杆菌和沙门氏菌,分别针对ompA、cesB和invA设计了3对特异性引物,建立mPCR方法;同时为了消除假阳性结果,用单叠氮丙啶(PMA)对细胞进行预处理,以选择性地消除死亡细胞的基因组DNA;此外,添加了一对基于16S rRNA基因保守区的通用引物作为内部扩增对照(Internal Amplification Control,IAC),用来排除由PCR仪、试剂问题或食品样品中的抑制剂引起的假阴性结果,结果显示在纯培养条件下,该方法对阪崎肠杆菌、肠炎沙门氏菌和蜡样芽孢杆菌的检出限分别为95、74和75 CFU/mL。采用PMA-mPCR方法和传统培养方法对3株细菌进行了检测比较,结果表明PMA-mPCR方法对同时被检测牛奶中的3种菌具有良好的敏感性和特异性[38]。
与普通PCR对比,mPCR检测技术更能够减少人力、时间,检测结果也更加准确、可靠;不仅能够同时用于检测相似性较高的菌种,还可以鉴定其他种类的细菌。然而采用mPCR检测需要同时用到多种引物或不同的DNA模板,更容易出现非特异性扩增或交叉反应。引物浓度、电泳电压、退火温度也是影响实验的3个重要因素,因此要对这3个因素进行反复优化来达到检测的最佳效果。
2.3 实时荧光定量PCR检测实时荧光定量PCR (Real-Time Fluorescent Quantitative PCR,RT-qPCR)检测技术最初是在1995年美国使用的一种核酸定量分析技术,其原理是在PCR体系内放入可被检测到的荧光化学剂,根据其产生的信号强弱来判断PCR产物的量,从而实现对该样品的定量检测。根据所使用化学制剂的不同分为荧光探针和荧光染料2种方法。
Zhan等[39]建立了PCR、催化发夹组装(CHA)和氧化石墨烯(GO)相结合的荧光检测牛奶样品中呕吐蜡样芽孢杆菌的方法,该方法是根据靶单链DNA (ssDNA)设计出H1和FAM-H2这2个互补的探针,CHA反应只能由PCR扩增产物变性产生的靶ssDNA启动,在没有靶标ssDNA的情况下,CHA反应不能被触发,导致H1和FAM-H2吸附在GO表面,表现出较低的荧光强度,反之将获得强荧光信号,结果显示该菌在牛奶样品中的检测限为62 CFU/mL。杨滴等[40]通过设计一对特异性引物和一个TaqMan探针建立了Real-Time PCR技术来检测肉类食品中的蜡样芽胞杆菌,同时用不同浓度标准菌液的DNA和其他20种不同细菌的DNA来验证该方法的有效性,实验结果表明只有蜡样芽胞杆菌显示出相应大小的曲线,而且其检出限为1×103 CFU/mL。
Forghani等[41]建立了一种同时检测蜡样芽孢杆菌、增多性李斯特菌、金黄色葡萄球菌的SYBR Green I法,该方法可针对gyrB、hly、nuc基因一次性成功地鉴别出3种菌,而且在不进行增菌的情况下测定极限为3.7×103 CFU/g,增菌后的测定极限为3.7×101 CFU/g。目前发现使用PMA-qPCR方法能够直接将活菌辨别出来,PMA可通过损伤后的细胞膜,其分解产物在光的作用下会与DNA发生交联,同时抑制死亡细胞的DNA扩增,大大降低了假阳性结果的发生。Zhou等[42]使用PMA-qPCR来检测牛奶中的活性蜡样芽孢杆菌,结果发现仅3 μg/mL的PMA就能够抑制死亡细胞的聚合酶链式反应扩增。
荧光探针PCR法可对样本进行准确定量,其结果也可直观地观察到。与荧光探针法相比,SYBR Green I染料法可与所有DNA的2条链互相结合,因此无法辨别出PCR产物、引物二聚体等,更无法对样品进行定量,会给实验结果带来误差。
2.4 微滴数字PCR技术检测作为传统定量PCR的改进技术,微滴数字PCR技术(ddPCR)是通过使用极限稀释分析和泊松分布分析来实现目标DNA拷贝数的绝对定量,其原理如图 1所示[43]。Porcellato等[44]优化并采用了一种新的ddPCR方法来鉴别牛奶中的蜡样芽孢杆菌群,并将ddPCR与标准qPCR方法进行了对比,在检测定量样品中的目标分子时,qPCR方法与新的ddPCR方法测定系数几乎一致,但使用ddPCR方法检测时检测限会比较低,检测过程中也不需要标准曲线,能够减少误差。然而,ddPCR不能够对目标丰度高的样品进行量化,而且与标准qPCR相比,ddPCR的时间及成本几乎翻了一番[44]。由此可知,新的ddPCR技术是一种很有前途的检测定量低浓度目标细菌的方法,其允许在不使用标准曲线的情况下对分子靶标进行绝对定量。
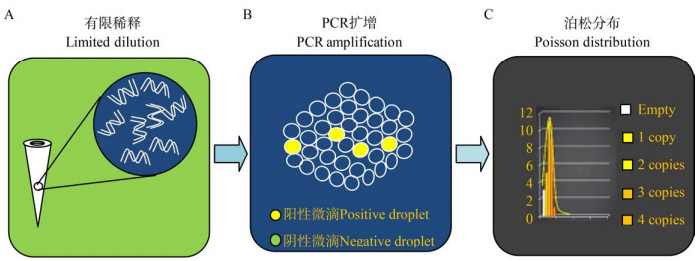
|
| 图 1 ddPCR检测技术的基本原理 Figure 1 Basic principles of ddPCR detection technology 注:A:一个待分析的PCR反应体系;B:成千上万个独立的PCR反应体系;C:阴性微滴比例推算起始靶分子的绝对量 Note: A: A PCR reaction system to be analyzed; B: Thousands of independent PCR reaction systems; C: Estimation of the absolute amount of initial target molecules by the proportion of negative droplets |
|
|
环介导等温扩增技术(LAMP)是一种恒温核酸扩增技术,不需要进行变性、退火、延伸的循环,通常使用4-6个引物和荧光试剂;阳性结果可以使用肉眼直接从浊度或颜色变化来评估,LAMP的原理如图 2、图 3所示[45]。
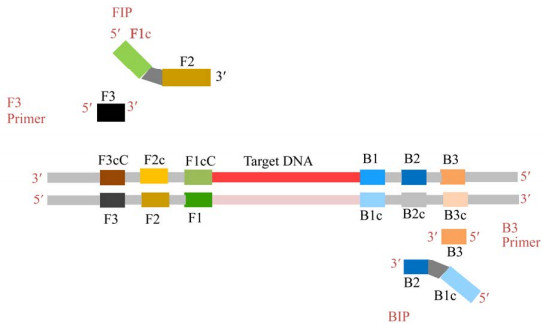
|
| 图 2 LAMP技术的引物设计 Figure 2 Primer design of LAMP |
|
|
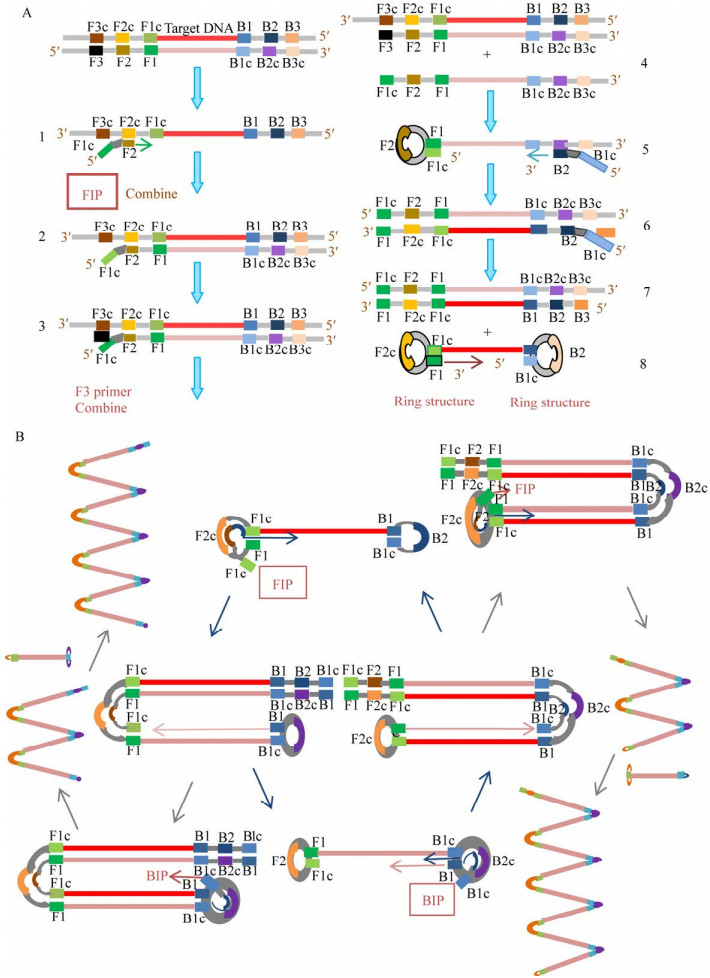
|
| 图 3 LAMP技术的基本原理 Figure 3 The basic principles of LAMP 注:A:哑铃状模板构造形成的过程;B:循环扩增阶段和延伸循环阶段 Note: A: The forming process of the dumbbell-shaped template structure; B: Cycle expansion stage and extension cycle stage |
|
|
周巍等[46]为了检测酸乳中的蜡样芽孢杆菌,根据hblA基因片段设计一套特异性引物进行LAMP检测,结果显示其检出限为6.4 CFU/mL且反应时间也较短。贾雅菁等[47]根据蜡样芽孢杆菌hblA基因序列设计内外引物后建立LAMP,并加入荧光染料SYBR Green I,用21株致病菌来验证RealAmp,并将RealAmp与普通LAMP进行比较,结果表明RealAmp对纯菌的检测浓度为8.2 CFU/mL,比普通LAMP的敏感度高出10倍,同时为检测该菌提供了新的方向。
与需要热循环和凝胶电泳的PCR方法相比,LAMP检测只需要一个简单的温度装置(通常为60-65 ℃),但是该方法不能避免反应中的假阳性结果,而且只能进行定性检测,对引物要求高。
2.6 重组酶聚合酶扩增技术重组酶聚合酶扩增技术(Recombinase Polymerase Amplification,RPA)是Piepenburg在2006年首次提出的一种新的恒温核酸扩增技术[48]。RPA技术对设备的要求非常低,该技术的原理与T4噬菌体的DNA复制机制相类似;在恒温条件下,UvsX和Gp32蛋白可与DNA双链实现特异性识别与结合,并在Bsu聚合酶的帮助下,最终实现DNA模板的指数扩增,其原理如图 4所示[49]。

|
| 图 4 RPA技术基本原理 Figure 4 Basic principles of RPA 注:A:重组酶-寡核苷酸引物复合物形成并靶向识别同源;B:SSB蛋白与被置换的链结合,使形成的D环保持稳定;C:链置换聚合酶启动合成;D:母链分开,合成继续;E:形成2个双链体。a:寡核苷酸引物;b:重组酶;c:单链DNA结合(SSB)蛋白;d:聚合酶 Note: A: The recombinase-oligonucleotide primer complex forms and targets homologous DNA; B: The SSB protein binds to the replaced chain to stabilize the formed D loop; C: Strand displacement polymerase initiates synthesis; D: The parent chain is separated and the synthesis continues; E: Form two duplexes. a: Oligonucleotide primer; b: Recombinant enzyme; c: Single stranded DNA binding (SSB) protein; d: Polymerase |
|
|
刘立兵等[50]根据蜡样芽胞杆菌的16S rRNA基因序列设计引物和exo探针,建立Real-Time RPA方法,该方法对其他芽胞杆菌和非芽胞杆菌无用,只可用来扩增蜡样芽胞杆菌,其检出限为1.0×10-3 ng/μL,而且试验表明该菌在食物中的量达到1.5×104 CFU/g时仅需6-13 min就可被检出。
RPA技术对设备要求低,特异性良好,大幅度缩短了检测时间,可广泛应用于临床和现场检测,同时也为食品的质量监测、疫病检测提供了新的方向。我们将分子生物学检测蜡样芽孢杆菌的引物汇总于表 1。
| 基因Genes | 序列Sequences (5′→3′) | 退火温度Annealing temperature (℃) | 扩增片段大小Amplicon size (bp) |
| hblA | GCAAAATCTATGAATGCCTA | 54 | 884 |
| GCATCTGTTCGTAATGTTTT | |||
| hblC | CCTATCAATACTCTCGCAA | 54 | 695 |
| TTTCCTTTGTTATACGCTGC | |||
| hblD | AATCAAGAGCTCTCACGAAT | 52 | 430 |
| CACCAATTGACCATGCTAAT | |||
| nheA | TACGCTAAGGAGGGGCA | 55 | 500 |
| GTTTTATTGCTTCATCGGCT | |||
| nheB | CAAGCTCCAGTTCATGCGG | 58 | 935 |
| GATCCCATTGTGTACCATTG | |||
| nheC | ACATCCTTTTGCAGCAGAAC | 58 | 618 |
| CCACCAGCAATGACCATATC | |||
| bceT | TTACATTACCAGGACGTGCTT | 55 | 428 |
| TGTTTGTGATTGTAATTCAGG | |||
| cytK | CGACGTCACAAGTTGTAACA | 52 | 565 |
| CGTGTGTAAATACCCCAGTT | |||
| entFM | GTTCGTTCAGGTGCTGGTAC | 55 | 486 |
| AGCTGGGCCTGTACGTACTT | |||
| Ces | GCATTTCGTGAAGCAGAGGT | 59 | 699 |
| CCCTTTATCCCCTTCGATGT | |||
| groEL | AGCTATGATTCGTGAAGGT | 54 | 236 |
| AAGTAATAACGCCGTCGT |
免疫学方法是将被检测的样品作为抗原,通过免疫反应制备得到单克隆抗体,然后通过抗原抗体之间的颜色反应或其他反应判断[51-52]。
3.1 酶联免疫吸附测定试验检测酶联免疫吸附测定试验(ELISA)检测是将抗原或者抗体固定在载体上,加入酶标抗体或者抗原使之发生反应,洗涤没有结合的物质,然后再加入酶反应底物,该底物能够被酶催化成有色物质,可以根据颜色的深浅判断样品中相关抗原或抗体的含量。ELISA既可用来直接检测菌体,也可检测该菌产生的毒素。Zhu等[53]以蜡样芽孢杆菌的全细胞为免疫原,制备了能够识别该菌表面某些成分的兔多克隆抗体和小鼠单克隆抗体;这些抗体随后被用于开发ELISA,以快速、直接和方便地测量食品中的菌体。该方法的线性检测范围约为1.0×104- 2.8×106细胞/mL,检测限为0.9×103细胞/mL;据报道,这是第一个用于直接和特异性检测蜡样芽孢杆菌细胞的夹心酶联免疫吸附试验。
ELISA是一种特异、灵敏、简便地检测大分子蛋白质、多糖和细菌的方法。该方法特异性较强且节约时间。因此,ELISA适用于常规检测蜡样芽孢杆菌,但其缺乏实时检测微生物的能力,抗体可能会对被测量的病原体或分析物的亲和力低,只有菌体达到103-105 CFU/g时才能使用免疫分析来提供实时信息。
3.2 商业化毒素检测试剂盒目前市场上售卖的多种商业试剂盒用来检测肠毒素Nhe或HBL,主要有BDE VIATM试剂盒(3M Tecra)[54]、BCET-RPLA试剂盒、Duopath(Ⓡ)试剂盒[55]。但是不同细菌可产生相同种类的毒素,因此,仅检测到某种肠毒素且不能确定为何种细菌产生时需要进一步鉴定。
4 讨论目前,抗生素的使用仍然被认为是治疗人畜感染蜡状芽孢杆菌最有效的方法。由于长期滥用抗生素或出现耐药基因,导致基因水平转移,已发现具有抗药性的蜡样芽孢杆菌菌株。特别是多耐药菌株的出现增加了感染的几率,导致抗生素治疗失败。目前我国分离出的蜡状芽孢杆菌耐药谱尚不明显,因此有必要开展相关调查,以增加对蜡样芽孢杆菌菌株抗性状况的认识,并提出有效的策略以减少这些菌株的出现。
随着关于蜡样芽孢杆菌感染病例的报道逐渐增多,越来越多相应的检测方法也迅速建立。本文中涉及的传统检测方法、分子学检测方法、免疫学检测方法都有各自的优缺点,需要根据检测的需要和条件限制进行选择,从而发挥每个检测方法的最大优势。检测蜡样芽孢杆菌方法的优缺点汇总于表 2。
| 检测方法 Methods |
优点 Advantages |
缺点 Disadvantages |
| PCR检测 PCR |
简便、节约时间 Simple and time-saving |
检测结果精确性低,并不能准确区分种属 The accuracy of the test results is low, can’t accurately distinguish between species and genera |
| 多重PCR检测 mPCR |
可同时鉴定同种类和不同种类的细菌 Identify the same and different kinds of bacteria at the same time |
容易出现非特异性扩增或交叉反应 Prone to nonspecific amplification or cross reaction |
| 实时荧光定量PCR检测 RT-qPCR |
可对样本进行准确定量,假阳性低 The sample can be quantified accurately, the false positive is low |
成本高,无法检测扩增子大小 High cost, Unable to detect the size of the extender |
| 微滴数字PCR技术检测 ddPCR |
检测限低,允许在不使用标准曲线的情况下对分子靶标进行绝对定量,可减少误差 The detection limit is low, allows absolute quantification of molecular targets without using standard curves, and the error can be reduced |
不能够对目标丰度高的样品进行量化,与qPCR相比时间及成本翻了一番 Samples with high target abundance cannot be quantified, and the time and cost are doubled compared with qPCR |
| 环介导等温扩增技术检测 LAMP |
时间短,成本低,操作方便 Short time, low cost, convenient operation |
易出现假阳性结果,只能进行定性检测 Prone to false positive results, only qualitative testing can be carried out |
| 重组酶聚合酶扩增技术检测 RPA |
不需要特殊仪器,灵敏度高,特异性性好,检测时间短 No need for special instruments, high sensitivity, the specificity is good, short detection time |
引物难设计,检测片段短 The primer is difficult to design, the detection fragment is short |
| 酶联免疫吸附测定试验检测 ELISA |
特异性较强,节约时间,成本低,不需要昂贵设备 Strong specificity, save time, low cost, no need for expensive equipment |
缺乏实时检测微生物的能力 Lack of real-time ability to detect microorganisms |
| 商业化毒素检测试剂盒 Commercial toxin test kit |
速度快 Fast |
存在漏检、误检 There are missed detection and false detection |
总而言之,蜡样芽孢杆菌给人类和动物健康都带来了潜在的威胁,然而大多数的检测方法在前期都需要增菌培养,大大延长了鉴定周期,因此,建立有效的检测方法对疾病的诊断和发病后及时治疗至关重要,但是对于致病菌的检测不应只停留在菌体上,因为菌体在高温加热情况下会被破坏,因而检测不到。毒素是菌体预先形成的,通过加热不能除去,因此还需要对其产生的毒素进行检测,从而精确地呈现检测结果。此外,对蜡样芽孢杆菌进行遗传多样性分析,可以提高对蜡样芽孢杆菌种群特征的了解,有助于建立准确、快速的检测方法,可靠地追踪蜡样芽孢杆菌并制定有针对性的防治措施。
| [1] |
Yuan XM, Lyu SJ, Zhang HQ, Hang XY, Shi WD, Liu L, Wu YL. Complete genome sequence of novel isolate SYJ15 of Bacillus cereus group, a highly lethal pathogen isolated from Chinese soft shell turtle (Pelodiscus sinensis)[J]. Archives of Microbiology, 2020, 202(1): 85-92. DOI:10.1007/s00203-019-01723-y |
| [2] |
Fayad N, Kallassy Awad M, Mahillon J. Diversity of Bacillus cereus sensu lato mobilome[J]. BMC Genomics, 2019, 20(1): 436. DOI:10.1186/s12864-019-5764-4 |
| [3] |
Wang CR, Liu ZQ, Huang YC, Zhang YN, Wang XH, Hu ZY. Cadmium-resistant rhizobacterium Bacillus cereus M4 promotes the growth and reduces cadmium accumulation in rice (Oryza sativa L.)[J]. Environmental Toxicology and Pharmacology, 2019, 72: 103265. DOI:10.1016/j.etap.2019.103265 |
| [4] |
Huang WX, Liu XY, Zhou XS, Wang XL, Liu XY, Liu HX. Calcium signaling is suppressed in Magnaporthe oryzae conidia by Bacillus cereus HS24[J]. Phytopathology, 2020, 110(2): 309-316. DOI:10.1094/PHYTO-08-18-0311-R |
| [5] |
Jiang CH, Chen Y, Yan F, Fan ZH, Guo JH. Whole-genome sequence of Bacillus cereus AR156, a potential biocontrol agent with high soilborne disease biocontrol efficacy and plant growth promotion[J]. Genome Announcements, 2017, 5(35): e00886-17. |
| [6] |
Gu XX, Zhang J, Li JJ, Wang ZH, Feng J, Li JZ, Pan KC, Ni XQ, Zeng D, Jing B, et al. Effects of Bacillus cereus PAS38 on immune-related differentially expressed genes of spleen in broilers[J]. Probiotics and Antimicrobial Proteins, 2020, 12(2): 425-438. DOI:10.1007/s12602-019-09567-0 |
| [7] |
Li SL, Qu Q, Li L, Xia K, Li Y, Zhu TT. Bacillus cereus s-EPS as a dual bio-functional corrosion and scale inhibitor in artificial seawater[J]. Water Research, 2019, 166: 115094. DOI:10.1016/j.watres.2019.115094 |
| [8] |
Cao FY, Wang P, Jiang LZ, Chen Y. Advances in methods for Bacillus cereus typing[J]. Food Science, 2017, 38(17): 286-290. (in Chinese) 曹飞扬, 王娉, 江连洲, 陈颖. 蜡样芽孢杆菌分型方法研究进展[J]. 食品科学, 2017, 38(17): 286-290. DOI:10.7506/spkx1002-6630-201717046 |
| [9] |
Zhang ZH, Feng LX, Xu HY, Liu CW, Shah NP, Wei H. Detection of viable enterotoxin-producing Bacillus cereus and analysis of toxigenicity from ready-to-eat foods and infant formula milk powder by multiplex PCR[J]. Journal of Dairy Science, 2016, 99(2): 1047-1055. DOI:10.3168/jds.2015-10147 |
| [10] |
Coburn PS, Miller FC, LaGrow AL, Parkunan SM, Randall CB, Staats RL, Callegan MC. TLR4 modulates inflammatory gene targets in the retina during Bacillus cereus endophthalmitis[J]. BMC Ophthalmology, 2018, 18: 96. DOI:10.1186/s12886-018-0764-8 |
| [11] |
Lewin A, Quach C, Rigourd V, Picaud JC, Perreault T, Frange P, Domingo MC, Lalancette C, Delage G, Germain M. Bacillus cereus infection in neonates and the absence of evidence for the role of banked human milk: case reports and literature review[J]. Infection Control & Hospital Epidemiology, 2019, 40(7): 787-793. |
| [12] |
Akamatsu R, Suzuki M, Okinaka K, Sasahara T, Yamane K, Suzuki S, Fujikura D, Furuta Y, Ohnishi N, Esaki M, et al. Novel sequence type in Bacillus cereus strains associated with nosocomial infections and bacteremia, Japan[J]. Emerging Infectious Diseases, 2019, 25(5): 883-890. DOI:10.3201/eid2505.171890 |
| [13] |
Yin XH, Zeng YB, Zhao J, Xu ZW, Zhu L. Isolation and identification of pathogenic Escherichia coli and Bacillus cereus[J]. Microbiology China, 2019, 46(4): 811-818. (in Chinese) 殷鑫欢, 曾喻兵, 赵军, 徐志文, 朱玲. 致病性大肠杆菌和蜡样芽胞杆菌的分离鉴定[J]. 微生物学通报, 2019, 46(4): 811-818. |
| [14] |
Li GN, Shi M, Zhao S, Long YH, Zhu Y. Toxicity response of silkworm intestine to Bacillus cereus SW7-1 pathogen[J]. Science of the Total Environment, 2019, 692: 1282-1290. DOI:10.1016/j.scitotenv.2019.07.349 |
| [15] |
Song LL, Zhang LY, Jia WJ, Bai ZH, Liu Y, Chen YJ, Wang XL. Isolation, identification and virulence gene detection of Bacillus cereusin cattle[J]. Acta Agriculturae Boreali-occidentalis Sinica, 2019, 28(11): 1735-1741. (in Chinese) 宋丽丽, 张玲艳, 贾伟娟, 白志恒, 刘媛, 陈云娇, 王学理. 1株牛源蜡样芽孢杆菌的分离鉴定及毒力基因检测[J]. 西北农业学报, 2019, 28(11): 1735-1741. |
| [16] |
Chon JW, Song KY, Kim H, Seo KH. Comparison of 3 selective media for enumeration of Bacillus cereus in several food matrixes[J]. Journal of Food Science, 2014, 79(12): M2480-M2484. DOI:10.1111/1750-3841.12594 |
| [17] |
Kabir MS, Hsieh YH, Simpson S, Kerdahi K, Sulaiman IM. Evaluation of two standard and two chromogenic selective media for optimal growth and enumeration of isolates of 16 unique Bacillus species[J]. Journal of Food Protection, 2017, 80(6): 952-962. DOI:10.4315/0362-028X.JFP-16-441 |
| [18] |
Teng KL, Lu MF, Cai ZH, Wu QP, Shi XF. Identifying Bacillus cereus from other food microorganisms by chromogenic media[J]. Chinese Journal of Health Laboratory Technology, 2016, 26(7): 955-958. (in Chinese) 滕昆仑, 卢勉飞, 蔡芷荷, 吴清平, 石小帆. 运用显色培养基从食品微生物中鉴别蜡样芽孢杆菌[J]. 中国卫生检验杂志, 2016, 26(7): 955-958. |
| [19] |
Chon JW, Kim YJ, Kim DH, Song KY, Kim H, Seo KH. Supplementation of modified Mannitol-Yolk-Polymyxin B agar with cefuroxime for quantitative detection of Bacillus cereus in food[J]. Journal of Food Science, 2019, 84(1): 133-137. DOI:10.1111/1750-3841.14393 |
| [20] |
Kang IB, Chon JW, Kim DH, Jeong D, Kim HS, Kim H, Seo KH. Improvement of Polymyxin-Egg Yolk-Mannitol- Bromothymol blue agar for the enumeration and isolation of Bacillus cereus in various foods[J]. Journal of Food Protection, 2017, 80(3): 502-505. DOI:10.4315/0362-028X.JFP-16-206 |
| [21] |
Savić D, Miljković-Selimović B, Lepšanović Z, Tambur Z, Konstantinović S, Stanković N, Ristanović E. Antimicrobial susceptibility and β-lactamase production in Bacillus cereus isolates from stool of patients, food and environment samples[J]. Vojnosanitetski Pregled, 2016, 73(10): 904-909. DOI:10.2298/VSP150415134S |
| [22] |
Huang JJ, Luo J, He HX. Research progress on detection methods for Bacillus cereus[J]. China Animal Husbandry & Veterinary Medicine, 2018, 45(3): 635-642. (in Chinese) 黄晶菁, 罗静, 何宏轩. 蜡样芽孢杆菌检测方法研究进展[J]. 中国畜牧兽医, 2018, 45(3): 635-642. |
| [23] |
Pomastowski P, Złoch M, Rodzik A, Ligor M, Kostrzewa M, Buszewski B. Analysis of bacteria associated with honeys of different geographical and botanical origin using two different identification approaches: MALDI-TOF MS and 16S rDNA PCR technique[J]. PLoS One, 2019, 14(5): e0217078. DOI:10.1371/journal.pone.0217078 |
| [24] |
Hakovirta JR, Prezioso S, Hodge D, Pillai SP, Weigel LM. Identification and analysis of informative single nucleotide polymorphisms in 16S rRNA gene sequences of the Bacillus cereus group[J]. Journal of Clinical Microbiology, 2016, 54(11): 2749-2756. DOI:10.1128/JCM.01267-16 |
| [25] |
Carroll LM, Kovac J, Miller RA, Wiedmann M. Rapid, high-throughput identification of anthrax-causing and emetic Bacillus cereus group genome assemblies via BTyper, a computational tool for virulence-based classification of Bacillus cereus group isolates by using nucleotide sequencing data[J]. Applied and Environmental Microbiology, 2017, 83(17): e01096-17. |
| [26] |
Lu JQ, Li SC, Wang DH, Liu YK, Chen L. Combined 16S rDNA and gyrB gene sequence to identify a strain of Bacillus cereus[J]. Journal of Henan University of Science and Technology (Natural Science), 2018, 39(1): 73-77, 83. (in Chinese) 卢佳琦, 李市场, 王大红, 刘永康, 陈龙. 16S rDNA与gyrB序列联合法鉴定一株蜡样芽孢杆菌[J]. 河南科技大学学报: 自然科学版, 2018, 39(1): 73-77, 83. |
| [27] |
Schmid D, Rademacher C, Kanitz EE, Frenzel E, Simons E, Allerberger F, Ehling-Schulz M. Elucidation of enterotoxigenic Bacillus cereus outbreaks in Austria by complementary epidemiological and microbiological investigations, 2013[J]. International Journal of Food Microbiology, 2016, 232: 80-86. DOI:10.1016/j.ijfoodmicro.2016.05.011 |
| [28] |
Bai FL, Chen S, Luo MY, Zeng BF, Tang JN. Identification and virulence genes detection of Bacillus cereus food isolates[J]. Modern Food Science and Technology, 2018, 34(10): 247-252, 204. (in Chinese) 白凤岚, 陈松, 罗梦幽, 曾宝锋, 唐俊妮. 食品中蜡样芽孢杆菌的分离及携带毒力基因的检测[J]. 现代食品科技, 2018, 34(10): 247-252, 204. |
| [29] |
Nguyen AT, Tallent SM. Screening food for Bacillus cereus toxins using whole genome sequencing[J]. Food Microbiology, 2019, 78: 164-170. DOI:10.1016/j.fm.2018.10.008 |
| [30] |
Mathur A, Feng SY, Hayward JA, Ngo C, Fox D, Atmosukarto Ⅱ, Price JD, Schauer K, Märtlbauer E, Robertson AAB, et al. A multicomponent toxin from Bacillus cereus incites inflammation and shapes host outcome via the NLRP3 inflammasome[J]. Nature Microbiology, 2019, 4(2): 362-374. DOI:10.1038/s41564-018-0318-0 |
| [31] |
Cai Y, Huang TX, Xu YK, Zhou GP, Zou P, Zeng GF, Liu XJ. Genetic and genomic diversity of NheABC locus from Bacillus strains[J]. Archives of Microbiology, 2017, 199(5): 775-785. DOI:10.1007/s00203-017-1350-9 |
| [32] |
Jeßberger N, Krey VM, Rademacher C, Böhm ME, Mohr AK, Ehling-Schulz M, Scherer S, Märtlbauer E. From genome to toxicity: a combinatory approach highlights the complexity of enterotoxin production in Bacillus cereus[J]. Frontiers in Microbiology, 2015, 6: 560. |
| [33] |
Patiño-Navarrete R, Sanchis V. Evolutionary processes and environmental factors underlying the genetic diversity and lifestyles of Bacillus cereus group bacteria[J]. Research in Microbiology, 2017, 168(4): 309-318. DOI:10.1016/j.resmic.2016.07.002 |
| [34] |
Oltuszak-Walczak E, Walczak P. PCR detection of cytK gene in Bacillus cereus group strains isolated from food samples[J]. Journal of Microbiological Methods, 2013, 95(2): 295-301. DOI:10.1016/j.mimet.2013.09.012 |
| [35] |
Adame-Gómez R, Muñoz-Barrios S, Castro-Alarcón N, Leyva-Vázquez MA, Toribio-Jiménez J, Ramírez-Peralta A. Prevalence of the strains of Bacillus cereus group in artisanal Mexican cheese[J]. Foodborne Pathogens and Disease, 2020, 17(1): 8-14. DOI:10.1089/fpd.2019.2673 |
| [36] |
Kumar TDK, Murali HS, Batra HV. Multiplex PCR assay for the detection of enterotoxic Bacillus cereus group strains and its application in food matrices[J]. Indian Journal of Microbiology, 2010, 50(2): 165-171. DOI:10.1007/s12088-010-0002-4 |
| [37] |
Razei A, Sorouri R, Mousavi SL, Nazarian S, Amani J, Aghamollaei H. Presenting a rapid method for detection of Bacillus cereus, Listeria monocytogenes and Campylobacter jejuni in food samples[J]. Iranian Journal of Basic Medical Sciences, 2017, 20(9): 1050-1055. |
| [38] |
Yu S, Yan LN, Wu X, Li F, Wang D, Xu HY. Multiplex PCR coupled with propidium monoazide for the detection of viable Cronobacter sakazakii, Bacillus cereus, and Salmonella spp. in milk and milk products[J]. Journal of Dairy Science, 2017, 100(10): 7874-7882. DOI:10.3168/jds.2017-13110 |
| [39] |
Zhan ZX, Yu B, Li H, Yan LN, Aguilar ZP, Xu HY. Catalytic hairpin assembly combined with graphene oxide for the detection of emetic Bacillus cereus in milk[J]. Journal of Dairy Science, 2019, 102(6): 4945-4953. DOI:10.3168/jds.2018-15812 |
| [40] |
Yang D, Zhao YM, Qin PJ, Feng X, Ma YY, Bian LK. Study on real-time fluorescent PCR detection method of cerea spore bacilli in meat products[J]. Meat Industry, 2017(6): 49-52. (in Chinese) 杨滴, 赵宇明, 秦鹏钧, 封雪, 马颖颖, 卞立坤. 肉制品中蜡样芽胞杆菌实时荧光PCR检测方法的研究[J]. 肉类工业, 2017(6): 49-52. DOI:10.3969/j.issn.1008-5467.2017.06.012 |
| [41] |
Forghani F, Wei S, Oh DH. A rapid multiplex real-time PCR high-resolution melt curve assay for the simultaneous detection of Bacillus cereus, Listeria monocytogenes, and Staphylococcus aureus in food[J]. Journal of Food Protection, 2016, 79(5): 810-815. DOI:10.4315/0362-028X.JFP-15-428 |
| [42] |
Zhou P, Xie GY, Liang TB, Yu B, Aguilar Z, Xu HY. Rapid and quantitative detection of viable emetic Bacillus cereus by PMA-qPCR assay in milk[J]. Molecular and Cellular Probes, 2019, 47: 101437. DOI:10.1016/j.mcp.2019.101437 |
| [43] |
Li Z, Schottroff F, Simpson DJ, Gänzle MG. The copy number of the spoVA2mob operon determines pressure resistance of Bacillus endospores[J]. Applied and Environmental Microbiology, 2019, 85(19): e01596-19. |
| [44] |
De Porcellato D, Narvhus J, Skeie SB. Detection and quantification of Bacillus cereus group in milk by droplet digital PCR[J]. Journal of Microbiological Methods, 2016, 127: 1-6. DOI:10.1016/j.mimet.2016.05.012 |
| [45] |
Pu W, Wang Y, Yang N, Guo G, Li H, Li Q, Ur Rehman N, Zheng L, Wang P, Han S, et al. Investigation of Streptococcus agalactiae using pcsB-based LAMP in milk, tilapia and vaginal swabs in Haikou, China[J]. Journal of Applied Microbiology, 2020, 128(3): 784-793. DOI:10.1111/jam.14501 |
| [46] |
Zhou W, Zhang W, Liu L, Liu D, Wang Z, Qin L, Zhao Y, Zhang Y. Detection of Bacillus cereus in yogurt by loop-mediated isothermal amplification assay[J]. Journal of Dairy Science and Technology, 2013, 36(5): 29-31. (in Chinese) 周巍, 张薇, 刘亮, 刘东, 王赞, 秦丽, 赵勇, 张岩. 环介导等温扩增技术检测酸乳中蜡样芽孢杆菌[J]. 乳业科学与技术, 2013, 36(5): 29-31. |
| [47] |
Jia YJ, Fu BY, Wang Y, Ma XY, Zhang XZ, Yuan N, Zhang W. Detection of Bacillus cereus in milk by real-time fluorescence loop-mediated isothermal amplification method[J]. Food Science, 2016, 37(6): 184-189. (in Chinese) 贾雅菁, 付博宇, 王羽, 马晓燕, 张先舟, 苑宁, 张伟. 实时荧光环介导等温扩增技术检测牛乳中的蜡样芽孢杆菌[J]. 食品科学, 2016, 37(6): 184-189. |
| [48] |
Bentahir M, Ambroise J, Delcorps C, Pilo P, Gala JL. Sensitive and specific recombinase polymerase amplification assays for fast screening, detection, and identification of Bacillus anthracis in a field setting[J]. Applied and Environmental Microbiology, 2018, 84(11): e00506-18. |
| [49] |
Ma L, Zeng FW, Cong F, Yuan W, Zhu YJ, Wu ML, Xu FJ, Huang BH, Lian YX, Huang R, et al. Establishment of a reverse transcriptase recombinase polymerase amplification assay for detection of murine norovirus[J]. Chinese Journal of Veterinary Science, 2019, 39(5): 853-859. (in Chinese) 马磊, 曾繁文, 丛锋, 袁文, 朱余军, 伍妙梨, 徐凤娇, 黄碧洪, 练月晓, 黄韧, 等. 小鼠诺如病毒逆转录重组酶聚合酶扩增检测方法的初步建立[J]. 中国兽医学报, 2019, 39(5): 853-859. |
| [50] |
Liu LB, Nan HZ, Sun XX, Jiang YF, Wang JF, Wang JC. Development and application of real-time recombinase polymerase amplification assay for detection of Bacillus cereus in food[J]. Journal of Food Science and Technology, 2018, 36(1): 89-94. (in Chinese) 刘立兵, 南汇珠, 孙晓霞, 姜彦芬, 王金凤, 王建昌. 食品中蜡样芽胞杆菌实时荧光RPA检测方法的建立与应用[J]. 食品科学技术学报, 2018, 36(1): 89-94. |
| [51] |
Umesha S, Manukumar HM. Advanced molecular diagnostic techniques for detection of food-borne pathogens: current applications and future challenges[J]. Critical Reviews in Food Science and Nutrition, 2018, 58(1): 84-104. DOI:10.1080/10408398.2015.1126701 |
| [52] |
Zhu K, Hölzel CS, Cui YF, Mayer R, Wang Y, Dietrich R, Didier A, Bassitta R, Märtlbauer E, Ding SY. Probiotic Bacillus cereus strains, a potential risk for public health in China[J]. Frontiers in Microbiology, 2016, 7: 718. |
| [53] |
Zhu LJ, He J, Cao XH, Huang KL, Luo YB, Xu WT. Development of a double-antibody sandwich ELISA for rapid detection of Bacillus cereus in food[J]. Scientific Reports, 2016, 6: 16092. DOI:10.1038/srep16092 |
| [54] |
Nakayama M, Miyashita T, Hosoya K, Hitomi J, Sato M, Sunaga Y, Shigematsu Y, Ogasawara J, Takenaka S, Hamasaki M, et al. Evaluation of the immunochromatography kit for detection of Bacillus cereus producing Emetic Toxin[J]. Food Hygiene and Safety Science: Shokuhin Eiseigaku Zasshi, 2012, 53(6): 273-277. DOI:10.3358/shokueishi.53.273 |
| [55] |
Ceuppens S, Rajkovic A, Hamelink S, Van De Wiele T, Boon N, Uyttendaele M. Enterotoxin production by Bacillus cereus under gastrointestinal conditions and their immunological detection by commercially available kits[J]. Foodborne Pathogens and Disease, 2012, 9(12): 1130-1136. DOI:10.1089/fpd.2012.1230 |
 2021, Vol. 48
2021, Vol. 48




