扩展功能
文章信息
- 阮真, 朱鹏飞, 付晓婷, 廖璞
- RUAN Zhen, ZHU Pengfei, FU Xiaoting, LIAO Pu
- 单细胞拉曼技术在病原微生物检测中的研究进展
- Detection of pathogenic microorganism by single-cell raman spectroscopy: a review
- 微生物学通报, 2021, 48(4): 1348-1359
- Microbiology China, 2021, 48(4): 1348-1359
- DOI: 10.13344/j.microbiol.china.200703
-
文章历史
- 收稿日期: 2020-07-12
- 接受日期: 2020-09-28
- 网络首发日期: 2020-12-09
2. 重庆市人民医院检验科 重庆 400013;
3. 中国科学院青岛生物能源与过程研究所单细胞中心 山东 青岛 266101;
4. 重庆市第九人民医院检验科 重庆 400700
2. Department of Laboratory Medicine, Chongqing General Hospital, Chongqing 400013, China;
3. Single-Cell Center, Qingdao Institute of Bioenergy and Bioprocess Technology, Chinese Academy of Sciences, Qingdao, Shandong 266101, China;
4. Department of Laboratory Medicine, The Ninth People's Hospital of Chongqing, Chongqing 400700, China
常规病原微生物检测多为培养后的细胞群体水平检测,而动辄数天的培养时间极大地增加了感染患者的治疗风险,也间接导致“病急乱投医”,造成了耐药微生物产生和蔓延的严峻局面。此外,过去几十年间,微生物学家逐渐意识到自然界中的大部分微生物(> 99%)不能采用常规的方法进行培养,与此同时单个细胞表型异质性开始被报道,其除了会影响微生物形态发生改变,还与微生物耐药性和毒力等都有直接关系[1-3]。
目前非培养的病原微生物研究多采用分子生物学方法(聚合酶链式反应、全基因组测序、基因芯片等)或基质辅助激光解吸电离飞行时间质谱(Matrix-Assisted Laser Desorption Ionization Time-of-Flight Mass Spectrometry,MALDI-TOF MS)检测方法,它们主要针对微生物群体进行分析且需要破坏微生物结构后进行检测,难以用于原位状态下低丰度病原微生物检测和微生物群体内的细胞表型异质性研究[4-6]。近年来,单细胞拉曼技术在病原微生物检测领域的不断发展为解决上述问题提供了新思路。
1 单细胞拉曼技术简介拉曼光谱技术是基于印度科学家Raman在1928年发现的拉曼散射效应而产生的一种分析方法,主要用于分子结构研究。当入射光(多为激光)照射物质分子时,入射光子与分子发生作用,使分子发生不同程度的振动和转动,散射出不同频率的光。当散射光频率与入射光频率相同时为“瑞利散射”,当散射光频率与入射光频率不同时为“拉曼散射”。散射光频率的变化取决于物质内部分子化学键的组成特征,通过拉曼光谱系统,包括激光激发模块、显微聚焦模块、显微成像模块和信号收集模块等收集散射光谱,可以得到物质相应的生物学信息,包括蛋白质、核酸、脂质等,构成生物体特有的“指纹图谱”,图 1展示了单细胞拉曼技术的检测流程[7]。
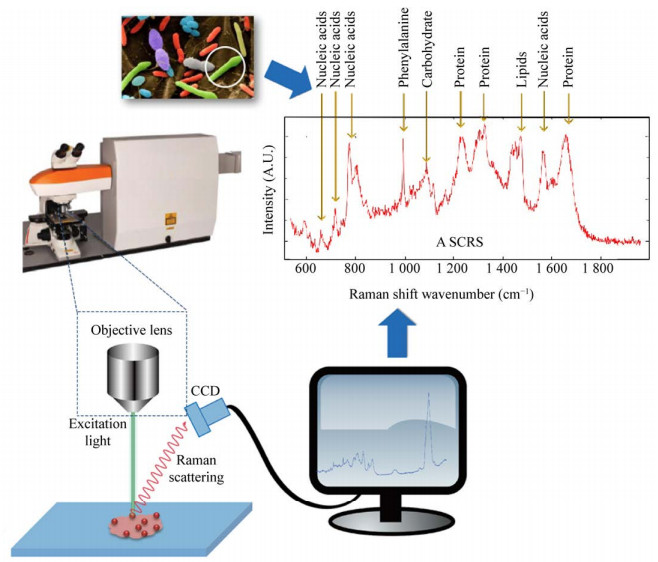
|
| 图 1 单细胞拉曼技术 Figure 1 Single-cell raman spectroscopy (SCRS) |
|
|
虽然早在1928年Raman已经发现了拉曼散射效应,但直到19世纪末拉曼光谱技术才开始应用于医学研究,主要通过指纹图谱与数据信息处理系统(如人工神经网络)相结合探究组织病理学差异和病原微生物快速鉴定[8-10]。由于早期拉曼光谱技术存在光谱灵敏度低、背景信号干扰大、分辨率低等问题,导致拉曼光谱检测在微生物鉴定上无论是光谱收集时间(1-20 min/单个细胞)还是鉴定准确性(约80%)均不太理想[10]。20世纪初研究者们开始对拉曼检测技术和数据处理方法进行改良,大量研究成果的展出揭示拉曼检测技术在微生物鉴定的准确性可媲美“金标准” (16S rRNA基因测序)[11-14]。随着近年来表面增强拉曼(Surface Enhanced Raman Scattering,SERS)、共振拉曼(Resonance Raman Spectroscopy,RRS)、相干反斯托克斯拉曼(Coherent Anti-Stokes Raman Spectroscopy,CARS)、受激拉曼(Stimulated Raman Scattering,SRS)等技术的相继出现,使非侵入性、快速、原位的单个细胞检测成为可能,单细胞拉曼技术应运而生[15-19]。
单细胞拉曼技术在病原微生物检测中的研究集中在病原微生物鉴定、药物敏感性检测和病原微生物自动化检测3个方面,表 1对上述三方面内容进行概述。本综述结合近年来单细胞拉曼技术在病原微生物检测中的研究进展和技术方向,探讨其在临床应用上的可行性。
| 研究内容 Research contents |
原理 Principles |
优势 Advantages |
不足 Disadvantages |
检测内容 Detection contents |
参考文献 References |
| 病原微生物鉴定 Identification of pathogens |
在已建立的微生物拉曼图谱库基础上,结合拉曼分析软件和不同的分析方法对病原微生物进行鉴定 Identify pathogens according to Ramanome Explore (RamEX) and diverse analysis methods on the basis of the established Ramanome Database (RamDB) |
快速、非培养、无破坏性实现原位标本检测和难鉴定菌株准确区分 In-situ detection and accurate discrimination of strains that are difficult to identify in a rapid, non-destructive and culture-independent way |
需要建立数据库和专业的分析软件 Need to establish database and develop professional analysis software for strains identification |
经培养后的细菌、真菌、分枝杆菌鉴定 Identification of bacteria, fungi and Mycobacterium after cultivation |
[11-14, 20-21] |
| 原始样本(透析液、尿液、血液)病原菌直接鉴定 Direct identification of pathogens from the original samples (i.e., dialysate, urine, blood) |
[22-24] | ||||
| 葡萄球菌生物膜检测 Detection of Staphylococcus biofilm |
[25] | ||||
| 高度同源分枝杆菌鉴定 Identification of highly homologous Mycobacterium |
[26] | ||||
| 药物敏感性检测 Antimicrobial susceptibility testing |
对比抗菌药物作用前后“指纹图谱”中生物标志区域“特征峰”差异区分“敏感”与“耐药” Whether a strain is sensitive or resistant to an antibiotic can be determined by the difference of “characteristic peak” in the biomarker bands of Raman fingerprint spectra before and after antibiotic treatment |
快速定性区分“敏感株”和“耐药株”细菌 Rapid discrimination between sensitive strains and resistant strains |
不同抗菌药物和不同微生物相互作用“特征峰”不固定,需要大量前期验证提高灵敏度和特异性 The “characteristic peak” reflecting the interaction between antibiotics and microorganisms is unstable, which requires extensive early validation to improve sensitivity and specificity |
特殊耐药表型(如万古霉素耐药的肠球菌等)快速筛查 Rapid screening of specific resistance phenotypes (e.g., vancomycin-resistant Enterococcus, etc.) |
[27-29] |
| 微生物细胞摄取重水时拉曼峰发生迁移产生重水峰,重水峰所占比例与细胞代谢水平呈正相关 When microbial cells drink heavy water, a “characteristic peak” of heavy water may appear, and the proportion of this “characteristic peak” was positively correlated with the cell metabolism |
代谢能力反映药物敏感性更准确、快速且可定量 It is more accurate, rapid, and quantifiable if the antimicrobial susceptibility is reflected by metabolic activity |
定量检测标准暂不统一,有待进一步标准化 Currently, the quantitative detection standard is not unified, which needs to be standardized in the future |
重水(D2O)标记药敏检测 D2O-labeled antimicrobial susceptibility testing |
[30-31] | |
| 病原微生物自动化检测 Auto-detection of pathogens |
将预处理样本经微流控芯片实现单个细胞流动至SRCS区进行快速拉曼光谱检测,在信息系统的控制下细胞分选系统分选出目标细胞进行进一步分子生物学检测 The pre-processed samples were transferred to the SRCS region at single-cell level via a microfluidic chip for rapid Raman spectroscopy detection, and the target cells were sorted by the cell sorting system under the control of information system for further molecular biological detection |
自动、高效、准确的细胞筛选,且筛选细胞具有活性 The sorting process is automatic, efficient and accurate, and the sorted cells are active |
需要特定仪器和分析系统,价格昂贵 It is expensive due to the requirements of specific instruments and analytical systems |
高通量自动化目标微生物筛选 High-throughput and automatic target microorganism screening |
[32-34] |
| “肠道微生态”检测 “Intestinal microbiome” detection |
[35-36] |
开展常规病原微生物鉴定的临床实验室仍处于分离、培养出微生物菌落后,基于微生物生化反应差异对病原微生物进行鉴定的状态,这导致了病原微生物鉴定周期长、无法区分生化反应特征相似的微生物、无法鉴定超出数据库分类规则的细菌等问题。
自发拉曼光谱,即微生物细胞的“指纹图谱”是最早用于拉曼光谱技术的检测标准,从图 2可以看出,自发拉曼光谱在不同的生物间和同一物种的不同生长时期均存在差异[30]。近年来,随着拉曼光谱数据库的不断完善,以及拉曼数据分析方法如主成分分析(Principal Component Analysis,PCA)、偏最小二乘法判别分析(Partial Least Square-Discriminate Analysis,PLS-DA)、支持向量机分析(Support Vector Machine,SVM)、线性判别分析(Linear Discriminant Analysis,LDA)、人工神经网络(Artificial Neural Networks,ANNs)、层次聚类分析(Hierarchical Cluster Analysis,HCA)等和分析软件的不断升级,越来越多的学者开始研究通过构建微生物“指纹数据库”实现原始标本快速鉴定和难鉴定菌株的准确区分。

|
| 图 2 微生物的自发拉曼光谱 Figure 2 The "fingerprints" of microorganisms 注:A:3种标准菌株变形链球菌(Streptococcus mutans) UA159、血链球菌(Streptococcus sanguis) ATCC10556、发酵乳杆菌(Lactobacillus fermentum) ATCC9338经培养12 h后的拉曼光谱,它们在778-785 cm-1 (胞嘧啶、尿嘧啶)、810-820 cm-1 (核糖核酸、RNA骨架)、1 001-1 003 cm-1 (苯丙氨酸)、1 431-1 481 cm-1 (蛋白质)区域存在差异;B:发酵乳杆菌(Lactobacillus fermentum ATCC9338)在相同条件下培养5 h (a)和12 h (b)后的拉曼光谱,可见在778-785 cm-1 (胞嘧啶、尿嘧啶)、1 431-1 481 cm-1 (蛋白质)区域存在差异 Note: A: After 12 hours of culture, the "fingerprints" of three standards (Streptococcus mutans UA159, Streptococcus sanguis ATCC10556, Lactobacillus fermentum ATCC9338) have difference in 778-785 cm-1 (cytosine, uracil), 810-820 cm-1 (nucleic acids (C-O-P-O-C in RNA backbone)), 1 001-1 003 cm-1 (phenylalanine), 1 431-1 481 cm-1 (protein); B: The "fingerprints" of Lactobacillus fermentum ATCC9338 have difference in 778-785 cm-1 (cytosine, uracil) and 1 431-1 481 cm-1 (protein) after 5 hours (a) and 12 hours (b) culture |
|
|
Montanari等[22]将单细胞拉曼技术与经典微生物检测技术结合,通过与标准菌株拉曼光谱进行对比,鉴定了288份血液透析用水中分离的146株酵母菌,包括临床常见的近平滑假丝酵母和季也蒙假丝酵母,还成功鉴定了临床少见的粘红酵母(Rhodotorula glutinis)、胶红酵母(Rhodotorula mucilaginosa)和墨汁丝孢酵母(Trichosporon inkin)。由此可见,单细胞拉曼技术可用于血液透析用水的病原微生物常规检测,同时也预示了该研究方法在无菌体液标本中检测病原微生物的可行性。
有研究[23]模拟复杂尿路感染的微生物模型,对泌尿系统感染常见病原微生物培养后进行拉曼光谱检测和峰谱分析,再建立2种或3种微生物混合模型,对混合微生物进行拉曼光谱检测,对比PCA、PLS-DA、SVM这3种分析方法,发现采用SVM对混合细菌拉曼光谱进行分析的细菌鉴定准确度达95%,提示SVM分析方法与拉曼检测相结合可用于复杂尿路感染患者的病原微生物直接检测。De Plano等[24]采用噬菌体结合血液中的病原体进行单细胞拉曼检测,极低浓度(1.5 CFU/mL血液)病原菌在6 h内即可成功鉴定,为快速诊断菌血症提供了新思路。
2.2 通过“指纹图谱”差异峰区分高度同源病原微生物常规检测方法对高度同源的病原微生物无法进行区分或鉴定。Samek等[25]通过单细胞拉曼技术成功识别含生物膜和不含生物膜的表皮葡萄球菌,并将生物膜形成细胞用光学镊子直接从菌落中取出。Stöckel等[20]通过单细胞拉曼检测分枝杆菌,经峰谱差异分析,成功将26种不同的分枝杆菌进行分类,准确度可达94%,而且每个样本仅需50-100个完整细胞即可进行检测。Verma等[26]采用单细胞拉曼技术对16S rRNA基因序列有高度相似性的印度分枝杆菌(Mycobacterium indicu spranii)和胞内分枝杆菌(Mycobacterium intracellulare)进行检测,发现2种分枝杆菌在来自脂质和菌酸的光谱信号(1 064、1 127、1 299、1 440、1 460 cm-1)上存在较大差异,采用单变量(强度、比率分析)和多变量(PCA、HCA)分析技术结合,可以快速在各个生长阶段对这2种细菌进行准确区分。可见差异峰谱识别可为鉴定密切相关菌种提供新的鉴定思路。
3 单细胞拉曼技术在药敏检测中的研究进展遏制细菌耐药早已成为全球共同关注的问题,快速、准确的细菌药物敏感性检测是解决问题的关键[37]。单细胞拉曼技术在细菌药敏检测中的研究主要基于对拉曼生物标志光谱区域的检测,特定生物标志区域的拉曼信号强度反映了细菌的代谢活性,通过代谢活性来检测细菌的药物敏感性[14, 38]。表 2列举了部分在微生物研究中常见的拉曼生物标志区域。单细胞拉曼技术和基于细菌生长变化的检测方法相比,其优势在于可以检测处于有代谢活性但不生长的细胞亚群(Non-Growing but Metabolically Active,NGMA),即在药物作用下已经不再生长或增殖但仍然具备代谢活性的存活细胞。这种称为持留菌(Persisters)的细胞在即便是克隆化的微生物群体中也普遍存在,而且在临床上十分常见,被公认为是耐药性产生与传播的根源[43]。学者们主要通过图谱寻找特征峰或重水(D2O)标记2种方式对细菌药物敏感性进行研究。
| 拉曼谱带 Raman bands (cm-1) |
生物标志物 Biomarker |
参考文献 References |
| 405, 542 | β-D-glucose | [39] |
| 484-490 | Glycogen | [40] |
| 504, 894 | Glycine | [39] |
| 573 | Tryptophan/cytosine, guanine | [40] |
| 623 | Adenine | [39] |
| 650 | Guanine | [39] |
| 617, 740, 1 369, 1 671 | Thymine | [39] |
| 792, 1 275 | Cytosine | [39] |
| 786 | DNA: O-P-O, cytosine, uracil, thymine | [40] |
| 790, 1 235 | Uracil | [39] |
| 828, 859 | Tyrosine, collagen | [39-40] |
| 926 | O-P-O stretch (DNA) | [40] |
| 971 | ν(C-C) wagging | [41] |
| 1 004 | Symmetric ring breathing mode of phenylalanine and tryptophan | [42] |
| 1 064 | Skeletal C-C stretching of lipids | [40] |
| 1 117-1 119 | C-C stretching (lipid) | [40] |
| 1 127 | ν(C-N), protein | [41] |
| 1 156, 1 515 | β-Carotene | [39] |
| 1 440 | CH2 and CH3 deformation vibrations (lipid) | [42] |
| 1 450 | CH2 bending | [41] |
| 1 575 | The ring-stretching vibrations of adenine and guanine | [42] |
| 1 607 | Tyrosine, phenylalanine ring vibration | [41] |
| 1 665 | Amide I vibration(protein) | [42] |
| 2 930 | C-H stretching vibration | [42] |
Assmann等[27]发现粪肠球菌在万古霉素作用下,30 min内单细胞拉曼光谱即显现特征性变化,通过拉曼光谱变化反映和解释抗菌药物与微生物细胞作用的过程。Lin等[28]发现采用硫酸粘菌素处理后的大肠埃希菌、鲍曼不动杆菌和铜绿假单胞菌经拉曼光谱检测,在746 cm-1 (腺嘌呤环呼吸模式)、1 125 cm-1 (C-C、C-N拉伸)和1 581 cm-1 (鸟嘌呤环拉伸)谱带均有强度降低,通过HCA分析黏菌素敏感性,全过程仅需1.5 h,总体灵敏度达90.9%,特异性为91.1%。
Kirchhoff等[29]将不同浓度的环丙沙星与15株大肠埃希菌(包含敏感株和耐药株)进行预培养后检测,根据环丙沙星抑制细菌DNA超螺旋从而达到杀菌的作用,筛选与之相关的生物标志区,发现1 458 cm-1 (核酸模式)和1 485 cm-1 (核苷酸嘌呤碱基鸟嘌呤、腺嘌呤)区域强度随环丙沙星浓度增加而增加,当设定两区域强度比值为“1”时即可准确区分敏感株和耐药株,而且对应环丙沙星浓度与参考方法检测的最低抑菌浓度(Minimal Inhibitory Concentration,MIC)高度吻合。
对比抗生素作用前后微生物指纹图谱有差异的特征峰可以快速区分敏感株和耐药株,可在固定的耐药表型筛查(如耐万古霉素的肠球菌、耐甲氧西林的葡萄球菌等)中发挥作用。然而不同类型抗菌药物对微生物细胞的作用机制不同,导致不同抗菌药物作用于不同类型微生物时特征峰并不固定,一定程度上限制了该方法在临床药敏检测上的应用。
3.2 重水(D2O)标记拉曼检测病原微生物药物敏感性研究将稳定同位素(13C、15N、2H)和拉曼光谱检测相结合对微生物进行检测是近年来开始采用的研究方法,可实现更简便、快捷、直观的微生物检测[44]。如图 3所示,培养基中的化合物元素被同位素取代,具有代谢活性的微生物细胞摄入比胞内元素质量更大的同位素时,相应物质所对应的拉曼峰向波数较小的方向迁移。在重水标记(D2O)应用于药物敏感性研究时,代谢活跃的细胞利用氘(2H)标记的水(重水),氘原子会参与细胞脂质合成过程形成C-D键(2 000-2 300 cm-1)并替换原有的C-H键(2 800-3 200 cm-1),这一变化可通过拉曼定量检测出来与细胞代谢活性相关,可用于细菌药物敏感性研究[45]。

|
| 图 3 加入同位素后拉曼峰的迁移 Figure 3 Raman spectra peak is shifted after addition of isotope |
|
|
Ueno等[46]将薄琼脂糖膜和微型注射器泵组合对耻垢分枝杆菌进行灌注培养,在高浓度利福平条件培养后转入含重水(D2O)的培养基进行培养,通过对比C-D峰(重水峰)强度和细菌的生长情况,筛选出克隆群体中的“NGMA”细胞,并证实NGMA细胞并非之前大家认为的持续代谢休眠状态,而是在生长过程中始终保持正常的代谢活性。可见,基于代谢活性的药敏检测较基于生长的药敏检测更准确。
Tao等[30]以导致龋病的变形链球菌(Streptococcus mutans)、重水和3种临床常见抗菌药物为基础构建研究模型,结合多元分析法证明C-D Ratio (重水峰所占比例)能够准确反映细胞利用胞外重水分子的速率,并与细胞的代谢活跃程度呈正相关,全过程仅需20 min,即可在细胞拉曼光谱中反映药物应激特异性的C-D峰,从而区分氟耐受型和氟易感型的变形链球菌。该研究还提出了一种全新的药敏检测标准——基于代谢活性的最低抑菌浓度(MIC-MA)的概念,预示能更准确地反映微生物的药物敏感性。Yang等[31]在C-D Ratio基础上建立抗菌药物的敏感/耐药界限值(S/R Cut Off Value):0.76-0.78,病原微生物在含重水和抗菌药物的培养基中短暂培养后检测其C-D Ratio值,若C-D Ratio≤0.76则说明这种病原微生物对该抗菌药物敏感;C-D Ratio≥0.78则为耐药,这种方法被证实可直接用于尿路感染患者尿液标本中所含病原微生物的药物敏感性检测,从尿液标本到获得敏感性结果全程仅需2.5 h,而且测定结果与传统方法(纸片扩散法)符合率达100%。
由于重水不改变细胞的化学组分,也不影响细胞生长代谢,因此重水标记的微生物药物敏感性研究不受微生物和抗菌药物种类的限制,C-D峰强度与微生物代谢活性的正相关性可为更加准确、快速、便捷的药物敏感性检测方法提供新的思路。
4 单细胞拉曼技术在病原微生物检测中的技术方向近年来,单细胞拉曼技术朝着自动化、智能化、高通量方向不断发展,其中,将单细胞拉曼技术快速、非侵入性检测优势与微流控技术快速细胞筛选及介电电泳(Dielectrophoresis,DEP)、声泳(Acoustophoresis,AP)或光学镊子(Optical Trap,OT)等自动收集目标细胞装置相结合形成的单细胞拉曼分选技术平台(图 4),成功实现高通量、智能化的微生物细胞检测和目标细胞分离,成为目前单细胞拉曼技术在病原微生物检测中最新的技术方向[47-48]。

|
| 图 4 单细胞拉曼分选技术平台 Figure 4 Single-cell raman sorting technology platform |
|
|
Lee等[32]将微流控技术、光学镊子和拉曼光谱技术结合组装形成一种新的“光流控平台”,实现自动筛选经稳定同位素“氘”标记的3种阴性杆菌(大肠埃希菌、鼠伤寒沙门氏菌、海洋细菌Marinobacter adhaerens)和一种阳性杆菌(枯草芽孢杆菌),分选精度达98.3%±1.7%,处理量为200-500个细胞/h,即3.3-8.3个细胞/min。
Wang等[33]首次提出拉曼激活细胞喷射(Raman-Activated Cell Ejection,RACE)的概念,在他们的研究中亚秒级即可获得单个细胞拉曼光谱,经光谱分析后,通过弹出装置成功收集经13C标记的大肠埃希菌,并对收集细胞进行单个细胞基因扩增。在RACE基础上,团队研发了拉曼活化液滴分选(Raman-Activated Droplet Sorting,RADS)自动化平台,提高了分选精度和分选细胞存活率。该技术定制以聚二甲基硅氧烷(Polydimethylsiloxane,PDMS)为结构材料的微流控芯片实现微生物单细胞流动;微流控芯片结合拉曼光谱仪进行单个细胞的拉曼光谱检测,经检测后的单个微生物细胞在芯片上直接进行液滴包埋,包埋后液滴经DEP实现目标细胞分选;RADS分选精度达98.3%,检测效率达260个细胞/min,而且在分选目标细胞中92.7%的细胞存活并能增殖[34]。
4.2 单细胞拉曼分选技术平台在微生物组学中的研究虽然高通量宏基因组测序的出现加速了组学的研究,但拼接式的组学研究技术易忽略微生物表型异质性[49]。在单细胞水平上分析基因组、转录组、蛋白质组、代谢组和甲基化组是当前生命科学中最活跃的前沿研究[50-51]。
Wang等[35]在稀释粪便时加入重水(D2O)和不同种类、浓度的抗生素,经短时间厌氧培养后通过氘同位素探测拉曼显微镜(RamanDIP)观察微生物群C-D区域的代谢状态,使用RACE筛选耐药菌并对筛选菌进行测序,研究人体肠道“微生态”中耐药菌群的分布、产生因素及耐药表型与耐药基因型相关性。与此同时,该课题组将肠道微生物群与重水单独或联合不同底物(葡萄糖、络氨酸等)进行培养,通过RamanDIP研究不同化合物在肠道微生物群作用下的代谢途径,揭示了人体肠道中的葡萄糖在代谢过程中优先合成脂肪酸[36]。该课题组的成果展现也提示将C-D区作为筛选标准结合分选平台高通量检测和筛选优势,可对复杂的肠道“微生态”进行研究。
随着单细胞拉曼分选技术开始用于研究单细胞表型与其基因组、蛋白质组、代谢组的相关性,Teng等[52]首次提出“拉曼组(Ramanome)”的概念,将其定义为在给定时间和条件下从同基因群体中随机选择的若干细胞的单细胞拉曼光谱(SCRS)的集合;由于每个SCRS由超过1 500个拉曼谱带组成,其分别或共同对应检测细胞中代谢物的化学键的共振频率,可以对单个细胞的代谢情况进行实时监测,因此“拉曼组”也是代谢组的一种反映方式,用于揭示和模拟微生物细胞及群体在监测时间内的实时状态。He等[53]首次在“拉曼组”概念的基础上建立“拉曼组”技术平台,将单细胞拉曼分选技术与高通量测序技术结合,使拉曼光谱的“表型大数据”与高通量测序的“基因型大数据”连接,利用分选优势建立目标微生物细胞的单细胞表型-基因型结合研究平台。
5 结语与展望单细胞拉曼技术是基于拉曼光谱分析原理实现非培养、无标签、快速、高效、低成本揭示物质本质的新方法。如文中所述,近年来在病原微生物检测中的研究已实现快速、高效的微生物鉴定、药敏、代谢状态及原位标本检测等,并能高通量、无破坏地获取目标微生物细胞进行后续研究,直观解析各类“组学”关联,虽然单细胞拉曼检测技术的优势在病原微生物检测的各项研究中已得到充分展现,但广泛应用于临床检测仍存在一定的优化空间。
首先,大量生物学信息的处理需要更加智能化的分析系统和数据库。当前MALDI-TOF MS在临床微生物实验室广泛兴起,但单细胞拉曼技术就其非培养、低浓度和非破坏检测方面较MALDI-TOF MS有明显优势。目前大量光谱数据分析和数据模型的建立仍需依赖专业的生物信息团队,这一问题阻碍了单细胞拉曼技术在临床实验室的应用。近年来光谱分析软件和数据库的不断涌现,预示智能化、便捷的分析方法已经成为研究者们关注的问题,这些成果的展出将极大推动该方法的临床应用。
其次,单细胞拉曼技术各项操作的标准化有待进一步建立。拉曼光谱检测方法繁多,不同类型标本背景信号对不同检测方法的干扰存在差异,导致标本的前期处理、参数设置等存在一定差异,这些差异极大地限制了在临床应用中的标准化。同种拉曼检测方法的操作标准化、不同种检测方法的结果差异比对将是单细胞拉曼技术广泛应用于临床亟须解决的问题。
综上所述,虽然单细胞拉曼技术的临床应用仍存在一定问题,但其非培养、无标签、快速、高效、低成本检测和高效分离细胞建立微生物表型与“组学”关联的优势使其在临床微生物检测领域具有极大的发展潜能。
| [1] |
Davey ME, O'toole GA. Microbial biofilms: from ecology to molecular genetics[J]. Microbiology and Molecular Biology Reviews, 2000, 64(4): 847-867. DOI:10.1128/MMBR.64.4.847-867.2000 |
| [2] |
Turner NA, Harris J, Russell AD, Lloyd D. Microbial differentiation and changes in susceptibility to antimicrobial agents[J]. Journal of Applied Microbiology, 2000, 89(5): 751-759. DOI:10.1046/j.1365-2672.2000.01176.x |
| [3] |
Dhar N, McKinney JD. Microbial phenotypic heterogeneity and antibiotic tolerance[J]. Current Opinion in Microbiology, 2007, 10(1): 30-38. DOI:10.1016/j.mib.2006.12.007 |
| [4] |
Peter H, Berggrav K, Thomas P, Pfeifer Y, Witte W, Templeton K, Bachmann TT. Direct detection and genotyping of Klebsiella pneumoniae carbapenemases from urine by use of a new DNA microarray test[J]. Journal of Clinical Microbiology, 2012, 50(12): 3990-3998. DOI:10.1128/JCM.00990-12 |
| [5] |
Moran RA, Anantham S, Holt KE, Hall RM. Prediction of antibiotic resistance from antibiotic resistance genes detected in antibiotic-resistant commensal Escherichia coli using PCR or WGS[J]. Journal of Antimicrobial Chemotherapy, 2017, 72(3): 700-704. |
| [6] |
Jang KS, Kim YH. Rapid and robust MALDI-TOF MS techniques for microbial identification: a brief overview of their diverse applications[J]. Journal of Microbiology, 2018, 56(4): 209-216. DOI:10.1007/s12275-018-7457-0 |
| [7] |
Petry R, Schmitt M, Popp J. Raman spectroscopy: a prospective tool in the life sciences[J]. ChemPhysChem, 2003, 4(1): 14-30. DOI:10.1002/cphc.200390004 |
| [8] |
Liu CH, Alfano RR, Sha WL, Zhu HR, Akins DL, Cleary J, Prudente R, Cellmer E. Human breast tissues studied by IR Fourier-transform Raman spectroscopy[J]. Lasers in the Life Sciences, 1991, 4(1): 23-28. |
| [9] |
Liu CH, Das BB, Glassman WLS, Tang GC, Yoo KM, Zhu HR, Akins DL, Lubicz SS, Cleary J, Prudente R, et al. Raman, fluorescence, and time-resolved light scattering as optical diagnostic techniques to separate diseased and normal biomedical media[J]. Journal of Photochemistry and Photobiology B: Biology, 1992, 16(2): 187-209. DOI:10.1016/1011-1344(92)80008-J |
| [10] |
Goodacre R, Timmins ÉM, Burton R, Kaderbhai N, Woodward AM, Kell DB, Rooney PJ. Rapid identification of urinary tract infection bacteria using hyperspectral whole-organism fingerprinting and artificial neural networks[J]. Microbiology, 1998, 144(5): 1157-1170. DOI:10.1099/00221287-144-5-1157 |
| [11] |
Maquelin K, Choo-Smith LP, Van Vreeswijk T, Endtz HP, Smith B, Bennett R, Bruining HA, Puppels GJ. Raman spectroscopic method for identification of clinically relevant microorganisms growing on solid culture medium[J]. Analytical Chemistry, 2000, 72(1): 12-19. DOI:10.1021/ac991011h |
| [12] |
Kirschner C, Maquelin K, Pina P, Ngo Thi NA, Choo-Smith LP, Sockalingum GD, Sandt C, Ami D, Orsini F, Doglia SM, et al. Classification and identification of enterococci: a comparative phenotypic, genotypic, and vibrational spectroscopic study[J]. Journal of Clinical Microbiology, 2001, 39(5): 1763-1770. DOI:10.1128/JCM.39.5.1763-1770.2001 |
| [13] |
Maquelin K, Kirschner C, Choo-Smith LP, Ngo-Thi NA, Van Vreeswijk T, Stämmler M, Endtz HP, Bruining HA, Naumann D, Puppels GJ. Prospective study of the performance of vibrational spectroscopies for rapid identification of bacterial and fungal pathogens recovered from blood cultures[J]. Journal of Clinical Microbiology, 2003, 41(1): 324-329. DOI:10.1128/JCM.41.1.324-329.2003 |
| [14] |
Huang WE, Griffiths RI, Thompson IP, Bailey MJ, Whiteley AS. Raman microscopic analysis of single microbial cells[J]. Analytical Chemistry, 2004, 76(15): 4452-4458. DOI:10.1021/ac049753k |
| [15] |
Schie IW, Krafft C, Popp J. Applications of coherent Raman scattering microscopies to clinical and biological studies[J]. Analyst, 2015, 140(12): 3897-3990. DOI:10.1039/C5AN00178A |
| [16] |
Evans CL, Xie XS. Coherent anti-stokes Raman scattering microscopy: chemical imaging for biology and medicine[J]. Annual Review of Analytical Chemistry, 2008, 1: 883-909. DOI:10.1146/annurev.anchem.1.031207.112754 |
| [17] |
El-Kurdi R, Patra D. Gold and silver nanoparticles in resonance Rayleigh scattering techniques for chemical sensing and biosensing: a review[J]. Microchimica Acta, 2019, 186(10): 667. DOI:10.1007/s00604-019-3755-4 |
| [18] |
Fu D, Lu FK, Zhang X, Freudiger C, Pernik DR, Holtom G, Xie XS. Quantitative chemical imaging with multiplex stimulated Raman scattering microscopy[J]. Journal of the American Chemical Society, 2012, 134(8): 3623-3626. DOI:10.1021/ja210081h |
| [19] |
Chisanga M, Muhamadali H, Ellis DI, Goodacre R. Surface-enhanced raman scattering (SERS) in microbiology: illumination and enhancement of the microbial world[J]. Applied Spectroscopy, 2018, 72(7): 987-1000. DOI:10.1177/0003702818764672 |
| [20] |
Stöckel S, Meisel S, Lorenz B, Kloss S, Henk S, Dees S, Richter E, Andres S, Merker M, Labugger I, et al. Raman spectroscopic identification of Mycobacterium tuberculosis[J]. Journal of Biophotonics, 2017, 10(5): 727-734. DOI:10.1002/jbio.201600174 |
| [21] |
Ho CS, Jean N, Hogan CA, Blackmon L, Jeffrey SS, Holodniy M, Banaei N, Saleh AAE, Ermon S, Dionne J. Rapid identification of pathogenic bacteria using Raman spectroscopy and deep learning[J]. Nature Communications, 2019, 10(1): 4927. DOI:10.1038/s41467-019-12898-9 |
| [22] |
Montanari LB, Sartori FG, Ribeiro DBM, Leandro LF, Pires RH, De Souza Carvalho Melhem M, De Mello CA, Martins CHG. Yeast isolation and identification in water used in a Brazilian hemodialysis unit by classic microbiological techniques and Raman spectroscopy[J]. Journal of Water and Health, 2018, 16(2): 311-320. DOI:10.2166/wh.2017.334 |
| [23] |
M Y, Chawla K, Bankapur A, Acharya M, D'Souza JS, Chidangil S. A micro-Raman and chemometric study of urinary tract infection-causing bacterial pathogens in mixed cultures[J]. Analytical and Bioanalytical Chemistry, 2019, 411(14): 3165-3177. DOI:10.1007/s00216-019-01784-4 |
| [24] |
De Plano LM, Fazio E, Rizzo MG, Franco D, Carnazza S, Trusso S, Neri F, Guglielmino SPP. Phage-based assay for rapid detection of bacterial pathogens in blood by Raman spectroscopy[J]. Journal of Immunological Methods, 2019, 465: 45-52. DOI:10.1016/j.jim.2018.12.004 |
| [25] |
Samek O, Bernatová S, Ježek J, Šiler M, Šerý M, Krzyžánek V, Hrubanová K, Zemánek P, Holá V, Růžička F. Identification of individual biofilm-forming bacterial cells using Raman tweezers[J]. Journal of Biomedical Optics, 2015, 20(5): 051038. DOI:10.1117/1.JBO.20.5.051038 |
| [26] |
Verma T, Podder S, Mehta M, Singh S, Singh A, Umapathy S, Nandi D. Raman spectroscopy reveals distinct differences between two closely related bacterial strains, Mycobacterium indicus pranii and Mycobacterium intracellulare[J]. Analytical and Bioanalytical Chemistry, 2019, 411(30): 7997-8009. DOI:10.1007/s00216-019-02197-z |
| [27] |
Assmann C, Kirchhoff J, Beleites C, Hey J, Kostudis S, Pfister W, Schlattmann P, Popp J, Neugebauer U. Identification of vancomycin interaction with Enterococcus faecalis within 30 min of interaction time using Raman spectroscopy[J]. Analytical and Bioanalytical Chemistry, 2015, 407(27): 8343-8352. DOI:10.1007/s00216-015-8912-y |
| [28] |
Lin ZQ, Zhao X, Huang J, Liu W, Zheng YT, Yang X, Zhang Y, De La Chapelle ML, Fu WL. Rapid screening of colistin-resistant Escherichia coli, Acinetobacter baumannii and Pseudomonas aeruginosa by the use of Raman spectroscopy and hierarchical cluster analysis[J]. Analyst, 2019, 144(8): 2803-2810. DOI:10.1039/C8AN02220H |
| [29] |
Kirchhoff J, Glaser U, Bohnert JA, Pletz MW, Popp J, Neugebauer U. Simple ciprofloxacin resistance test and determination of minimal inhibitory concentration within 2 h using Raman spectroscopy[J]. Analytical Chemistry, 2018, 90(3): 1811-1818. DOI:10.1021/acs.analchem.7b03800 |
| [30] |
Tao YF, Wang Y, Huang S, Zhu PF, Huang WE, Ling JQ, Xu J. Metabolic-activity-based assessment of antimicrobial effects by D2O-labeled single-cell Raman microspectroscopy[J]. Analytical Chemistry, 2017, 89(7): 4108-4115. DOI:10.1021/acs.analchem.6b05051 |
| [31] |
Yang K, Li HZ, Zhu X, Su JQ, Ren B, Zhu YG, Cui L. Rapid antibiotic susceptibility testing of pathogenic bacteria using heavy-water-labeled single-cell Raman spectroscopy in clinical samples[J]. Analytical Chemistry, 2019, 91(9): 6296-6303. DOI:10.1021/acs.analchem.9b01064 |
| [32] |
Lee KS, Palatinszky M, Pereira FC, Nguyen J, Fernandez VI, Mueller AJ, Menolascina F, Daims H, Berry D, Wagner M, et al. An automated Raman-based platform for the sorting of live cells by functional properties[J]. Nature Microbiology, 2019, 4(6): 1035-1048. DOI:10.1038/s41564-019-0394-9 |
| [33] |
Wang Y, Ji YT, Wharfe ES, Meadows RS, March P, Goodacre R, Xu J, Huang WE. Raman activated cell ejection for isolation of single cells[J]. Analytical Chemistry, 2013, 85(22): 10697-10701. DOI:10.1021/ac403107p |
| [34] |
Wang XX, Ren LH, Su YT, Ji YT, Liu YP, Li CY, Li XR, Zhang Y, Wang W, Hu Q, et al. Raman-activated droplet sorting (RADS) for label-free high-throughput screening of microalgal single-cells[J]. Analytical Chemistry, 2017, 89(22): 12569-12577. DOI:10.1021/acs.analchem.7b03884 |
| [35] |
Wang Y, Xu JB, Kong LC, Li B, Li H, Huang WE, Zheng CM. Raman-activated sorting of antibiotic-resistant bacteria in human gut microbiota[J]. Environmental Microbiology, 2020, 22(7): 2613-2624. DOI:10.1111/1462-2920.14962 |
| [36] |
Wang Y, Xu JB, Kong LC, Liu T, Yi LB, Wang HJ, Huang WE, Zheng CM. Raman-deuterium isotope probing to study metabolic activities of single bacterial cells in human intestinal microbiota[J]. Microbial Biotechnology, 2020, 13(2): 572-583. DOI:10.1111/1751-7915.13519 |
| [37] |
Sugden R, Kelly R, Davies S. Combatting antimicrobial resistance globally[J]. Nature Microbiology, 2016, 1(10): 16187. DOI:10.1038/nmicrobiol.2016.187 |
| [38] |
Jin NF, Zhang DY, Martin FL. Fingerprinting microbiomes towards screening for microbial antibiotic resistance[J]. Integrative Biology, 2017, 9(5): 406-417. DOI:10.1039/c7ib00009j |
| [39] |
De Gelder J, De Gussem K, Vandenabeele P, Moens L. Reference database of Raman spectra of biological molecules[J]. Journal of Raman Spectroscopy, 2007, 38(9): 1133-1147. DOI:10.1002/jrs.1734 |
| [40] |
Stone N, Kendall C, Smith J, Crow P, Barr H. Raman spectroscopy for identification of epithelial cancers[J]. Faraday Discussions, 2004, 126: 141-157. DOI:10.1039/b304992b |
| [41] |
Lakshmi RJ, Kartha VB, Krishna CM, Solomon JGR, Ullas G, Devi P. Tissue raman spectroscopy for the study of radiation damage: brain irradiation of mice[J]. Radiation Research, 2002, 157(2): 157-182. |
| [42] |
Lorenz B, Wichmann C, Stöckel S, Rösch P, Popp J. Cultivation-free Raman spectroscopic investigations of bacteria[J]. Trends in Microbiology, 2017, 25(5): 413-424. DOI:10.1016/j.tim.2017.01.002 |
| [43] |
Brauner A, Fridman O, Gefen O, Balaban NQ. Distinguishing between resistance, tolerance and persistence to antibiotic treatment[J]. Nature Reviews Microbiology, 2016, 14(5): 320-330. DOI:10.1038/nrmicro.2016.34 |
| [44] |
Wang Y, Huang WE, Cui L, Wagner M. Single cell stable isotope probing in microbiology using Raman microspectroscopy[J]. Current Opinion in Biotechnology, 2016, 41: 34-42. DOI:10.1016/j.copbio.2016.04.018 |
| [45] |
Berry D, Mader E, Lee TK, Woebken D, Wang Y, Zhu D, Palatinszky M, Schintlmeister A, Schmid MC, Hanson BT, et al. Tracking heavy water (D2O) incorporation for identifying and sorting active microbial cells[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(2): E194-E203. DOI:10.1073/pnas.1420406112 |
| [46] |
Ueno H, Kato Y, Tabata KV, Noji H. Revealing the metabolic activity of persisters in mycobacteria by single-cell D2O Raman imaging spectroscopy[J]. Analytical Chemistry, 2019, 91(23): 15171-15178. DOI:10.1021/acs.analchem.9b03960 |
| [47] |
Fung CW, Chan SN, Wu AR. Microfluidic single-cell analysis-toward integration and total on-chip analysis[J]. Biomicrofluidics, 2020, 14(2): 021502. DOI:10.1063/1.5131795 |
| [48] |
Song YZ, Yin HB, Huang WE. Raman activated cell sorting[J]. Current Opinion in Chemical Biology, 2016, 33: 1-8. DOI:10.1016/j.cbpa.2016.04.002 |
| [49] |
Goldman SL, MacKay M, Afshinnekoo E, Melnick AM, Wu SX, Mason CE. The impact of heterogeneity on single-cell sequencing[J]. The Frontiers in Genetics, 2019, 10: 8. DOI:10.3389/fgene.2019.00008 |
| [50] |
Turajlic S, Sottoriva A, Graham T, Swanton C. Resolving genetic heterogeneity in cancer[J]. Nature Reviews Genetics, 2019, 20(7): 404-416. DOI:10.1038/s41576-019-0114-6 |
| [51] |
Stuart T, Satija R. Integrative single-cell analysis[J]. Nature Reviews Genetics, 2019, 20(5): 257-272. DOI:10.1038/s41576-019-0093-7 |
| [52] |
Teng L, Wang X, Wang XJ, Gou HL, Ren LH, Wang TT, Wang Y, Ji YT, Huang WE, Xu J. Label-free, rapid and quantitative phenotyping of stress response in E. coli via ramanome[J]. Scientific Reports, 2016, 6(1): 34359. DOI:10.1038/srep34359 |
| [53] |
He YH, Wang XX, Ma B, Xu J. Ramanome technology platform for label-free screening and sorting of microbial cell factories at single-cell resolution[J]. Biotechnology Advances, 2019, 37(6): 107388. DOI:10.1016/j.biotechadv.2019.04.010 |
 2021, Vol. 48
2021, Vol. 48




