扩展功能
文章信息
- 凌茜文, 于洁琼, 彭思远, 褚夫江
- LING Xiwen, YU Jieqiong, PENG Siyuan, CHU Fujiang
- 肠道微生物基因组学与代谢组学分析联合应用的研究进展
- Combined application of intestinal microbial genomics and metabolomics analysis: a review
- 微生物学通报, 2021, 48(4): 1292-1301
- Microbiology China, 2021, 48(4): 1292-1301
- DOI: 10.13344/j.microbiol.china.200585
-
文章历史
- 收稿日期: 2020-06-10
- 接受日期: 2020-07-31
- 网络首发日期: 2020-10-14
2. 广东省生物活性药物研究重点实验室 广东 广州 510006
2. Guangdong Key Laboratory of Pharmaceutical Bioactive Substances, Guangzhou, Guangdong 510006, China
肠道微生物是寄生在肠道内的微生物群体,具有数目庞大、种群繁多、分类复杂的特点,被称为人类的“第二基因组”。近年来,通过临床前和临床研究肠道菌群的代谢功能失调来解释人类疾病和肠道微生物的关系,寻找相关细菌以及具有区分微生物群落相关的细菌基因、年龄相关性黄斑变性(Age-Related Macular Degeneration,AMD)、迟发型败血症(Late-Onset Sepsis,LOS)、炎症性慢性疾病(Inflammatory Bowel Disease,IBD)、类风湿性关节炎(Rheumatoid Arthritis,RA)、肥胖、结肠癌(Colorectal Cancer,CRC)、抗生素诱导的微生物组耗尽(Antibiotic-Induced Microbiome Depletion,AIMD)[1-7]等。
Chu等前期研究发现家蝇幼虫生物活性肽对小鼠腹泻有明显的抑制作用,初步测定了家蝇幼虫生物活性肽对腹泻小鼠肠道微生态的影响,结肠内容物16S rRNA基因测序结果显示家蝇幼虫生物活性肽能增加双歧杆菌和乳酸菌的数量,同时降低肠球菌和肠杆菌的数量[8]。生物活性肽对肠道微生物具有调节作用,肠道微生物调控可能是家蝇幼虫腹泻的主要机制之一。生物活性肽具有易吸收、无毒、多功能等优点,而食物或食物来源的肽会在肠道中被消化和吸收,可以影响肠道菌群组成,进而影响微生物代谢产物。Wang等[9]合成了一种从核桃蛋白水解物中提取的生物活性肽,其可以有效改善小鼠的认知障碍,根据Pearson相关性分析血清代谢产物与粪便16S rRNA基因测序的结果,产生短链脂肪酸(Short-Chain Fatty Acids,SCFAs)的Allobaculum相对丰度变化与血清SCFAs浓度呈负相关,乳杆菌丰度与去甲肾上腺素也呈负相关,乳杆菌丰度的变化可能由于外周循环中去甲肾上腺素生物合成改变。因此,生物活性肽代谢产物的变化可能是特定的肠道微生物群发生改变。我们未来将结合微生物基因组学和代谢组学对生物活性肽影响肠道微生物的相关代谢途径及生物标记物做进一步探索。
微生物基因组学和代谢组学可对肠道细菌进行量化和分类,并且能获取其功能特性与在生物体中引起代谢产物变化的信息,而通过生物信息学和计算建模工具可提供关于复杂微生物群落中细菌基因功能的信息。因此,本文首先对用于描述肠道微生物群特征的代谢组学和微生物基因组学技术研究策略进行综述,并分析其优势和局限性;第2步对代谢组学和微生物基因组学结果之间的统计关联方法进行总结;最后介绍微生物共代谢和宿主-微生物相互作用的新兴的构建预测模型方法并作出展望,有助于确定各种疾病肠道微生物的生物学机制。
1 肠道菌群应用微生物组学与代谢组学技术的研究策略16S rRNA基因测序和宏基因组学的“霰弹枪”测序都可以用于分析肠道微生物组成及其相对丰度。这2种技术都可以直接以操作分类单元(Operational Taxonomic Unit,OTU)对微生物种类进行分类,不需要对菌群进行培养。在没有肠道群落功能特征直接数据的条件下,通过比较不同实验样本的16S rRNA基因序列,可以将肠道群落多样性与疾病以相关性分析进行联系。Liu等[5]对正常组与高脂饮食小鼠及其给药组粪便样本中的16S rRNA基因进行测序后,研究人员能够识别出存在于小鼠体内的核心微生物群,而偏离这一核心(即正常组小鼠微生物多样性的损失或减少)即可能与肥胖相关。但是,该技术在PCR扩增中引入了系统性偏差,这可能会影响所得数据。相较而言,“霰弹枪”测序宏基因组学测序是一种基于16S rRNA单基因靶向DNA的方法,不包含扩增偏倚[10],对样品中所有微生物的基因组进行无限制测序,在进化支水平(菌株、物种等)表征微生物群落,从而推断其生物学作用。基于二代测序技术的扩增子测序方法是微生物多样性研究中的主流,而基于三代测序技术的全长Marker基因测序方法也日益成熟,为微生物多样性的研究带来了新思路。三代测序可以获得基因的全长序列,物种区分较二代测序的基因局部序列分析精细。Xu等[11]采用PacBio SMRT测序和飞行时间质谱(GC-TOF/MS)技术,研究了不同接种条件下玉米青贮中基于物种水平的微生物群落和代谢组,利用Spearman相关性分析主要细菌与主要代谢物之间的关系。目前使用较为成熟的三代测序仪是PacBio Sequel II,用于16S rRNA基因的全长测序。但是目前三代测序技术在微生物多样性研究中受限于价格问题,有效测序量较低,适合于研究丰度较高的物种,测序的准确性仍旧逊于二代测序技术,而且目前适合于全长Marker基因的物种分类数据库严重容量不足,无法跟二代测序数据对应的微生物物种分类数据库相比,导致测序出来的全长基因无法确认物种,从而无法进行物种分类。
微生物组测序仅仅提供肠道细菌构成、基因丰度和功能性信息,而代谢组学是研究生物体中代谢产物变化的科学,可以提供微生物在肠道里的代谢信息。通过检测各种体液(如尿液、血浆和粪便)中的代谢物,可获取肠道微生物和宿主共同代谢物的功能特征图谱,有助于研究受到疾病或药物影响与肠道微生物相关的代谢途径,寻找相关生物标记物以及药物治疗靶点。代谢组学研究策略可以通过靶向或非靶向的方式进行。靶向代谢组学可以准确地定量一组已知的代谢物,而非靶向方法可以覆盖尽可能多的代谢物。Stewart等[2]首次使用UPLC-MS非靶向粪便代谢组学来研究被诊断为LOS的婴儿肠道内微生物的变化,有助于理解其代谢功能与疾病之间的相互作用。随着分析技术的发展,靶向或非靶向策略可以在单个实验中结合使用。代谢组学研究中使用的样品包含大量的代谢物,而不同的代谢组学平台的组合可以比单一方法鉴定更多的代谢物。例如,Li等[12]结合使用核磁共振(Nuclear Magnetic Resonance,NMR)、气相色谱-质谱(Gas Chromatography-Mass Spectrometry,GC-MS)和液相色谱-质谱(Liquid Chromatography-Mass Spectometry,LC-MS)这3种代谢组学方法,最后确定了重度抑郁症(Major Depressive Disorder,MDD)小鼠模型的肝脏代谢图谱变化。
2 整合代谢组和微生物基因组学数据的分析策略为了关联肠道微生物及其功能,对组学数据集进行处理以鉴定肠道微生物中最丰富的微生物种类,肠道微生物基因通过与直系同源基因家族的代表对齐来进行功能注释,然后,这些基因集可以整合到代谢途径中,产生的OTU可以与代谢物水平的显著变化进行统计学关联,从而可以推断出肠道微生物的代谢表型[13] (图 1)。然而整合代谢组和微生物基因组学数据仅在统计相关性上的研究并不能解释潜在的肠道微生物功能,通过开发其他计算方法,利用生物信息学进行全面建模,可能有助于将肠道微生物的研究过渡到机制上。
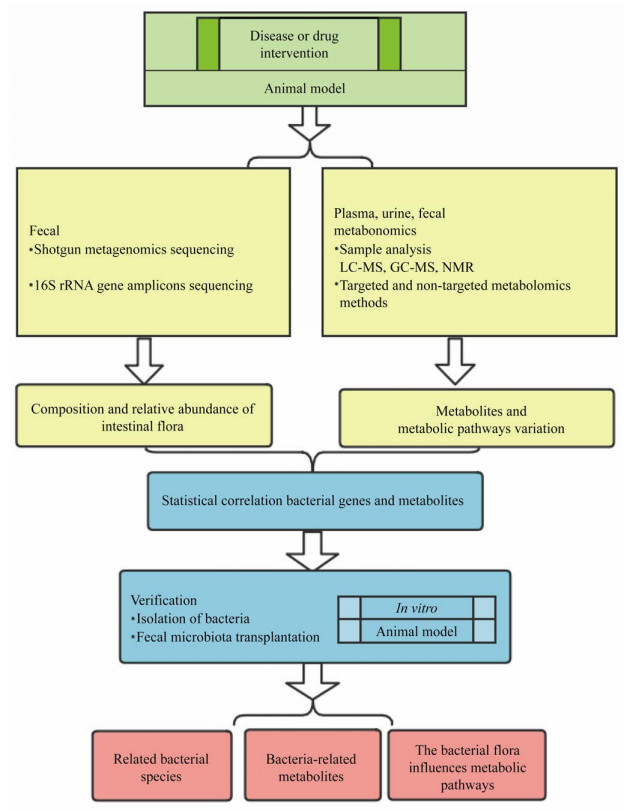
|
| 图 1 整合代谢组和微生物基因组学的实验流程 Figure 1 Experimental flow of integrated metabolome and microbial genomics |
|
|
肠道微生物和代谢图谱改变之间存在明显的相关性。当肠道微生物有关的代谢产物与特定的肠道细菌丰度相关时,可证明肠道微生物和代谢产物之间的功能相关。大多数研究使用单变量Spearman秩相关系数分析对代谢组学数据和微生物基因组分类谱进行分析。例如:Franzosa等[14]为了研究IBD肠道代谢组的变化,采用了基于Spearman的策略来整合代谢组学数据和肠道微生物数据。Lu等[15]使用Pearson的相关系数获取肠道微生物变化与代谢物摄动之间的功能相关性。但是,Pearson的相关性是根据真实值计算来描述线性关系中2个变量,而根据等级计算的Spearman相关性更适用于研究微生物组和代谢组关联的单调关系[16]。此外,单变量相关性分析方法必须通过多次测试校正提高可靠性。
为了解决由于机体复杂性需要同时考虑多个指标之间相关性的问题,有研究采用多元方法来研究高维组学数据矩阵之间的相互作用[17-20]。由于组学数据的高维特点,数据降维已成为其主要分析方法,包括主成分分析(Principal Component Analysis,PCA)和偏最小二乘分析(Partial Least Square,PLS)。例如:Stewart等[2]运用偏最小二乘分析确定了优势细菌属与鉴定出的代谢物之间的相关性。PLS-DA法是一种多元监管技术,使用简便,识别潜在的生物标记物的能力突出,只使用一个模型组和对照组即可分别进行分类[17],研究肠道微生物时更倾向采用此相关性分析方法。近年来,多变量方法不断发展为多组学数据融合提供新的途径,表 1列出了研究肠道微生物的多变量方法的适用性与局限性。
| 多变量分析方法 Multivariate analysis method |
适用性Application | 局限性 Limitation |
相关文献 References |
| 偏最小二乘分析 Partial least squares, PLS |
通过最大化数据集之间的协方差来识别 2个数据矩阵之间的关联 The association between two data matrices is identified by maximizing the covariance between data sets |
与响应向量无关的信息提取为方差大、相关系数小的 成分 Irrelevant information to the response vector is extracted into components with large variance and small correlation coefficients |
[2] |
| 规范相关分析 Canonical correlation analysis, CCA |
基于多元限制性排序的特征提取方法 A feature extraction method based on multivariate restrictive ranking |
数据集中的变量是线性独立的且数据中的协方差未知 Variables in the dataset are linearly independent and the covariance in the data is unknown样本个数不小于变量个数 The number of samples is not less than the number of variables |
[18] |
| 协惯性分析 Coinertia analysis, CIA |
确定具有相同测试对象的两组变量之间的关系 CIA determines the relationship between two sets of variables with the same test subject object |
未对多组学数据关联分析结果进行聚类 CIA cannot cluster the results of multi-omics data association analysis只能获得少数显著差异的特征值, Only a few feature values with significant differences can be obtained无法解释数据整体差异 CIA unable to explain overall difference in data |
[19] |
| 过程分析 Procrustes analysis, PA |
一种强大的最小二乘方法简化了组学比较 A powerful least squares method simplifies omics comparison可能是可视化数据集之间现有关系的补充工具 PA maybe a complementary tool for visualizing existing relationships between data sets |
需与其他方法(包括PLS、CAA和CIA)结合使用 PA need to be combined with other methods (PLS, CCA and CIA) |
[20] |
微生物组不同层面的变化通常存在时间尺度上的差异,运用传统的单、多变量分析方法无法得到解释,多模块方法的出现为此提供了一种分析策略。Garali等[21]利用广义正则CCA (Regularized Generalized CCA,RGCCA)及稀疏广义正则CCA (Sparse Regularized Generalized CCA,SRGCCA)这2个多模块方法,整合已有知识与PCA、PLS、CCA等方法联用;这2个方法都可用于多路形式的纵向数据(无论在时间上还是在采集类型上对给定特征的多次测量数据),而且该计算框架被证明能有效地管理和解释大量的生物数据类型,特别是代谢组学与下一代测序方法(如DNA-Seq、RNA-Seq等)产生的信息,为实现对肠道微生物多组数据的探索性分析提供一种新策略。
另外,肠道微生物多组学技术分析通常需要处理多种数据类型,联合变量和个体变量说明(Joint and Individual Variation Explained,JIVE)[22],独特的COM SCA (DISCO-SCA)[23]也可以为多组学数据融合提供新的分析策略。例如:JIVE是PCA的扩展,可分析多种数据类型的联合变化,降维后能确定特定数据类型独有的特征,为节点和个体结构的可视化研究提供了新的方向;与CCA和PLS方法相比,JIVE有组合组学数据的优势[22]。另外,ANOVA-PCA是PCA的另一个扩展,旨在发现多层数据集上的关系的多块方法,已被广泛用于合并多个分析平台生成的数据并关联2个不同的变量,这种多块方法的主要局限性是目前只能研究2个影响因素[24]。Li等[25]开发了稀疏多块偏最小二乘法(Sparse Mult-Block Partial Least Squares,sMBPLS)来识别在同一组样本上测量的不同类型基因组数据中的多维调控模块。该方法在分析以上2个数据块上优于大多数现有算法,但应用于只有2个数据块时不能优化结果。
传统分析方法具有的假阳性高、分析误差相对较大的局限性,新的联合分析方法基于实际数据自动优化模型mmvec克服了这些问题。mmvec具有结果可靠、应用算法科学、展现形式多样的优势,通过估计某种微生物存在情况下相应代谢物出现的条件概率,从而确定出最可能的微生物与代谢物的相互作用关系[26]。Morton等[26]演示了该方法如何发现肠道微生物产生的代谢物和炎症性肠病之间的关系。但实际研究中如何通过共现概率来获得两者相互作用统计显著性目前尚不清楚,并且对每种微生物-代谢物相互作用强度的置信区间无法计算,未来随着研究人员探索的不断深入可能会有所改善。
2.2 研究肠道微生物中代谢相互作用和途径的建模预测模型构建是利用已有数据库在已知微生物组和代谢组之间联系机制的情况下进行预测。宏基因组学建模依赖于物种的精确描述和注释,不能扩展到代谢物的复杂群落中,因此很多研究将微生物的功能潜能与代谢组学联系起来[27]。同时,全基因组测序的发展也加快了基因组水平代谢网络的构建,使我们可以从注释的基因组中获取代谢网络的组成信息,并使这种整合更具生物学意义[28]。
2.2.1 基于约束或基于网络的建模框架重建社区新陈代谢的预测模型基因组水平的代谢模型(GEMs)是基于生物体的全基因组水平信息,提供基因型与表型的数学关系[29]。将代谢组学数据集成到GEMs可以帮助定义预测,例如一个基因或基因产物如何影响一个特定通路或整个代谢网络的功能[30]。GEM已被用于研究肠道微生物的代谢,但其所提出的建模方法并不能扩大到包括更多肠道微生物的环境代谢网络,包括已知存在于生物体中的所有生化反应(基因、反应和代谢物);此重建过程需要使用一些生化反应数据库(如KEGG[31]、ModelSEED、BiGG[32]或MetaCyc[33]),有时会缺少基因注释,所以GEM的草图也包含代谢网络中的缺失值,即代谢网络中的反应之间没有对应的连接,需要使用新的算法填充间隙,运用化学计量模型预测种间关系[34]。此外,为完善和验证GEM,未来可以把元转录组学、元蛋白质组学和肠道代谢组学的数据逐渐整合到基因组水平的代谢模型(GEMs)上,预测宿主-菌群的相互作用及其在不同条件下(健康与疾病)的影响[35]。在大多数微生物组建模研究中[35],GEM和通量平衡分析(Flux Balance Analysis,FBA)[36]已被用作建模和理解饮食-微生物和微生物-微生物相互作用的首选工具。除了FBA以外,目前也已经开发了几种基于GEM的计算框架,例如COMET[37]、CASINO[35]、MCM[38]和SteadyCom[39]。
2.2.2 基于相对代谢更新率预测的代谢模型构建相对代谢更新率预测(Predicted Relative Metabolic Turnover,PRMT)是通过已知微生物组和代谢组之间联系机制的情况下(KEGG等数据库中获得化学计量酶反应矩阵),预测微生物菌属的代谢活性,进而计算基于社区的代谢物潜力(Community-Wide Metabolic Potential,CMP)分数,这些分数代表给定样本中社区产生或消耗每个代谢物的相对能力且提供软件实现;MIMOSA被评估为目前唯一能够根据人群水平的宏基因组数据预测全群落代谢相对丰度的方法,但其未明确使用数据挖掘和模型构建来验证预测模型,无法对该算法之前未发现的新的元基因组样本进行预测[40]。近期开发的一种算法则没有这个局限,MemonPan从菌群特征预测其代谢组的计算框架,给予模型上配对的代谢组和宏基因组,在新的菌群环境中预测潜在的无法观察的代谢产物[27];而且,Mimosa只能很好地预测少量代谢物化合物,MemonPan则能准确预测绝大多数代谢物。MemonPan与现有的依赖于数量有限、特征明确的分类群、酶和代谢物基于化学计量学的方法不同,功能注释不是必要的,因为该工具的设计目的是通过机器学习,即使是从非特征化的微生物特征来获取信息[40]。但是,MelonnPan不直接预测代谢物通量或峰值(与基于约束的方法相反),其通过合成与结合微生物的序列特征来估计每个代谢物在整个群落范围内的相对丰度,而且由于没有模型能准确跨环境预测,因此其只能作为一种假设生成工具为后续的实验验证研究提供依据。
2.2.3 基于已有数据的贝叶斯网络预测模型基于已有数据的贝叶斯网络(Bayesian Network,BN)模型是因果概率网络模型,用于鉴定对观测数据预测效果最好的概率网络[41]。McGeachie等[42]整合婴儿粪便的16S rRNA基因测序与之前发表的数据,构建了一个能够识别微生物组分类群之间重要关系的肠道微生物生态系统模型,该模型能通过测量或合成的初始条件预测微生物组成突然急剧变化的可能性。
2.2.4 代理人基模型(ABMs)ABMs是基于规则的计算模型,也被称为基于个体的模型(IBMs),结合博弈论、进化规划和蒙特卡罗方法的元素来引入随机性;ABMs也被用于模拟肠道微生物之间的相互作用,预测强调了肠道微生物生态系统中的反馈机制在维持稳定性和发挥关键功能中的必要性[43]。ABM也有助于研究空间组织在处理外部环境条件变化中的重要作用。有研究利用FBA和ABM相结合扩展模型探究了由7个物种组成的合成肠道微生物群落的单个成员之间的相互作用,将生物学规则如运动、趋化性和裂解纳入,建立单个物种代谢网络之间时空相互作用的模型[44]。然而,ABM只能确定整个群体的行为且计算非常费力,通过系统分析很难确定影响ABM输出结果的参数。因此,ABM探索更复杂微生物生态系统的动力学问题仍存在挑战。
3 展望越来越多的研究表明,肠道微生物的组成和功能改变与疾病状态和药物干预之间存在联系。有研究发现家蝇幼虫粉能影响改良三黄鸡肠道中氨基酸与肽类的吸收且使肠道黏膜免疫相关细胞发生变化,而蝇幼虫提取物还能对辐射小鼠具有免疫调节作用[45-46]。但是也有研究报道,家蝇幼虫乙醇提取物可选择性增加盲肠有机酸和调节正常大鼠肠道菌群的双歧杆菌作用[47]。本课题组未来将以家蝇幼虫生物活性肽通过影响肠道菌群来调节腹泻小鼠肠道微生态的角度对其机制进行探索。本文不仅为本课题组后期的研究提供指导,而且为结合肠道微生物基因组学与代谢组学分析联合应用来研究通过药物干预影响肠道菌群调节疾病状态的机制提供新的思路。
家蝇幼虫分泌物中分离得到的抗菌肽与溶菌酶有一定的同源性,但生物学特性有差异,通过破坏细菌细胞膜通透性、阻碍与细胞分裂相关的某些蛋白质合成等途径发挥抗菌作用[48]。本实验室前期研究发现家蝇抗菌肽Cecropin对革兰氏阳性菌和革兰氏阴性菌标准菌株均有较强的抑制活性,而且抗菌作用机制可能与通过结合DNA阻断或减少DNA复制或转录而抑制代谢途径有关[49]。本课题组前期利用水提法所获的罗仙子产物的抑菌活性最优,而且对大肠埃希菌的敏感性高于金黄色葡萄球菌[50]。本实验室研究表明家蝇抗菌肽Cecropin (MDC)是一种新型潜在的抗菌、抗炎和免疫功能抗菌肽,首次阐明了AMP MDC通过调节肠道菌群来改善结肠黏膜屏障的损伤;16S rRNA基因测序结果表明,MDC对肠道菌群具有显著的调节作用,Spearman序列相关分析与Mantel测试证明MDC的肠道菌群调控主要通过调控紧密连接蛋白来实现[51]。紧密连接蛋白功能障碍与多种局部和全体性疾病有关,这可能对鼠伤寒杆菌诱导的肠道菌群紊乱、炎症反应和氧化应激具有决定性影响。
前期研究中,课题组发现中药五谷虫粉对小鼠肠道菌群有一定的良性调节作用,可起到改善肠道内菌群的作用[52]。为了进一步明确五谷虫抑制IBS-D的作用机制及效应物质基础,我们课题组将结合微生物基因组学和代谢组学分析联合应用等技术,阐明其抑制IBS-D的作用机制。然而,不充分的研究设计和缺乏标准化的方法对解释肠道微生物群改变是否是疾病状态改变的结果带来了不确定性。由于机体本身的复杂性和肠道微生物的异构性,整合代谢组和微生物基因组学数据需要利用所有传统和最先进的技术来发现未知的肠道细菌在不同环境条件下建立的代谢变化和疾病之间的联系。基因组水平的代谢模型(GEMs)重建等预测模型应用于肠道微生物失调,将有可能识别紊乱的代谢物,并预估使用药物和菌群替代干预的效果,而肠道微生物生物信息学通过整合和比较不同的实验结果,填补了组学技术的空白并提供了新的计算方法。
| [1] |
Rowan S, Jiang SH, Korem T, Szymanski J, Chang ML, Szelog J, Cassalman C, Dasuri K, McGuire C, Nagai R, et al. Involvement of a gut-retina axis in protection against dietary glycemia-induced age-related macular degeneration[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(22): E4472-E4481. DOI:10.1073/pnas.1702302114 |
| [2] |
Stewart CJ, Embleton ND, Marrs ECL, Smith DP, Fofanova T, Nelson A, Skeath T, Perry JD, Petrosino JF, Berrington JE, et al. Longitudinal development of the gut microbiome and metabolome in preterm neonates with late onset sepsis and healthy controls[J]. Microbiome, 2017, 5(1): 75. DOI:10.1186/s40168-017-0295-1 |
| [3] |
Paramsothy S, Nielsen S, Kamm MA, Deshpande NP, Faith JJ, Clemente JC, Paramsothy R, Walsh AJ, Van Den Bogaerde J, Samuel D, et al. Specific bacteria and metabolites associated with response to fecal microbiota transplantation in patients with ulcerative colitis[J]. Gastroenterology, 2019, 156(5): 1440-1454. DOI:10.1053/j.gastro.2018.12.001 |
| [4] |
Aa LX, Fei F, Qi Q, Sun RB, Gu SH, Di ZZ, Aa JY, Wang GJ, Liu CX. Rebalancing of the gut flora and microbial metabolism is responsible for the anti-arthritis effect of kaempferol[J]. Acta Pharmacologica Sinica, 2020, 41(1): 73-81. DOI:10.1038/s41401-019-0279-8 |
| [5] |
Liu J, Yue SJ, Yang ZR, Feng WW, Meng XT, Wang AT, Peng C, Wang CY, Yan D. Oral hydroxysafflor yellow a reduces obesity in mice by modulating the gut microbiota and serum metabolism[J]. Pharmacological Research, 2018, 134: 40-50. DOI:10.1016/j.phrs.2018.05.012 |
| [6] |
Yang YZ, Misra BB, Liang L, Bi DX, Weng WH, Wu W, Cai SJ, Qin HL, Goel A, Li XX, et al. Integrated microbiome and metabolome analysis reveals a novel interplay between commensal bacteria and metabolites in colorectal cancer[J]. Theranostics, 2019, 9(14): 4101-4114. DOI:10.7150/thno.35186 |
| [7] |
Zarrinpar A, Chaix A, Xu ZZ, Chang MW, Marotz CA, Saghatelian A, Knight R, Panda S. Antibiotic-induced microbiome depletion alters metabolic homeostasis by affecting gut signaling and colonic metabolism[J]. Nature Communications, 2018, 9(1): 2872. DOI:10.1038/s41467-018-05336-9 |
| [8] |
Chu FJ, Jin XB, Ma HY. Anti-diarrhea effects and identification of Musca domestica larvae low molecular weight peptides (LMWP)[J]. Journal of Pharmaceutical and Biomedical Analysis, 2019, 173: 162-168. DOI:10.1016/j.jpba.2019.05.032 |
| [9] |
Wang M, Amakye WK, Guo L, Gong CC, Zhao YH, Yao MJ, Ren JY. Walnut-derived peptide PW5 ameliorates cognitive impairments and alters gut microbiota in APP/PS1 transgenic mice[J]. Molecular Nutrition & Food Research, 2019, 63(18): 1900326. |
| [10] |
Jovel J, Patterson J, Wang WW, Hotte N, O'Keefe S, Mitchel T, Perry T, Kao D, Mason AL, Madsen KL, et al. Characterization of the gut microbiome using 16S or shotgun metagenomics[J]. Frontiers in Microbiology, 2016, 7: 459. |
| [11] |
Xu DM, Ding WR, Ke WC, Li FH, Zhang P, Guo XS. Modulation of metabolome and bacterial community in whole crop corn silage by inoculating homofermentative Lactobacillus plantarum and heterofermentative Lactobacillus buchneri[J]. Frontiers in Microbiology, 2019, 9: 3299. DOI:10.3389/fmicb.2018.03299 |
| [12] |
Li B, Guo KN, Zeng L, Zeng BH, Huo R, Luo YY, Wang HY, Dong MX, Zheng P, Zhou CJ, et al. Metabolite identification in fecal microbiota transplantation mouse livers and combined proteomics with chronic unpredictive mild stress mouse livers[J]. Translational Psychiatry, 2018, 8(1): 34. DOI:10.1038/s41398-017-0078-2 |
| [13] |
McHardy IH, Goudarzi M, Tong MM, Ruegger PM, Schwager E, Weger JR, Graeber TG, Sonnenburg JL, Horvath S, Huttenhower C, et al. Integrative analysis of the microbiome and metabolome of the human intestinal mucosal surface reveals exquisite inter-relationships[J]. Microbiome, 2013, 1(1): 17. DOI:10.1186/2049-2618-1-17 |
| [14] |
Franzosa EA, Sirota-Madi A, Avila-Pacheco J, Fornelos N, Haiser HJ, Reinker S, Vatanen T, Hall AB, Mallick H, McIver LJ, et al. Gut microbiome structure and metabolic activity in inflammatory bowel disease[J]. Nature Microbiology, 2019, 4(2): 293-305. DOI:10.1038/s41564-018-0306-4 |
| [15] |
Lu K, Abo RP, Schlieper KA, Graffam ME, Levine S, Wishnok JS, Swenberg JA, Tannenbaum SR, Fox JG. Arsenic exposure perturbs the gut microbiome and its metabolic profile in mice: an integrated metagenomics and metabolomics analysis[J]. Environmental Health Perspectives, 2014, 122(3): 284-291. DOI:10.1289/ehp.1307429 |
| [16] |
Weiss S, Van Treuren W, Lozupone C, Faust K, Friedman J, Deng Y, Xia LC, Xu ZZ, Ursell L, Alm EJ, et al. Correlation detection strategies in microbial data sets vary widely in sensitivity and precision[J]. The ISME Journal, 2016, 10(7): 1669-1681. DOI:10.1038/ismej.2015.235 |
| [17] |
Triba MN, Le Moyec L, Amathieu R, Goossens C, Bouchemal N, Nahon P, Rutledge DN, Savarin P. PLS/OPLS models in metabolomics: the impact of permutation of dataset rows on the k-fold cross-validation quality parameters[J]. Molecular BioSystems, 2015, 11(1): 13-19. DOI:10.1039/C4MB00414K |
| [18] |
Montoliu I, Cominetti O, Boulange CL, Berger B, Siddharth J, Nicholson J, Martin FPJ. Modeling longitudinal metabonomics and microbiota interactions in c57bl/6 mice fed a high fat diet[J]. Analytical Chemistry, 2016, 88(15): 7617-7626. DOI:10.1021/acs.analchem.6b01343 |
| [19] |
Liu RX, Hong J, Xu XQ, Feng Q, Zhang DY, Gu YY, Shi J, Zhao SQ, Liu W, Wang XK, et al. Gut microbiome and serum metabolome alterations in obesity and after weight-loss intervention[J]. Nature Medicine, 2017, 23(7): 859-868. DOI:10.1038/nm.4358 |
| [20] |
Quinn RA, Navas-Molina JA, Hyde ER, Song SJ, Vázquez-Baeza Y, Humphrey G, Gaffney J, Minich JJ, Melnik AV, Herschend J, et al. From sample to multi-omics conclusions in under 48 hours[J]. mSystems, 2016, 1(2): e00038-16. |
| [21] |
Garali I, Adanyeguh IM, Ichou F, Perlbarg V, Seyer A, Colsch B, Moszer I, Guillemot V, Durr A, Mochel F, et al. A strategy for multimodal data integration: application to biomarkers identification in spinocerebellar ataxia[J]. Briefings in Bioinformatics, 2018, 19(6): 1356-1369. DOI:10.1093/bib/bbx060 |
| [22] |
Chauvel C, Novoloaca A, Veyre P, Reynier F, Becker J. Evaluation of integrative clustering methods for the analysis of multi-omics data[J]. Briefings in Bioinformatics, 2020, 21(2): 541-552. DOI:10.1093/bib/bbz015 |
| [23] |
Schouteden M, Van Deun K, Pattyn S, Van Mechelen I. SCA with rotation to distinguish common and distinctive information in linked data[J]. Behavior Research Methods, 2013, 45(3): 822-833. DOI:10.3758/s13428-012-0295-9 |
| [24] |
Xu Y, Goodacre R. Multiblock principal component analysis: an efficient tool for analyzing metabolomics data which contain two influential factors[J]. Metabolomics, 2012, 8(S1): 37-51. DOI:10.1007/s11306-011-0361-9 |
| [25] |
Li WY, Zhang SH, Liu CC, Zhou XJ. Identifying multi-layer gene regulatory modules from multi- dimensional genomic data[J]. Bioinformatics, 2012, 28(19): 2458-2466. DOI:10.1093/bioinformatics/bts476 |
| [26] |
Morton JT, Aksenov AA, Nothias LF, Foulds JR, Quinn RA, Badri MH, Swenson TL, Van Goethem MW, Northen TR, Vazquez-Baeza Y, et al. Learning representations of microbe-metabolite interactions[J]. Nature Methods, 2019, 16(12): 1306-1314. DOI:10.1038/s41592-019-0616-3 |
| [27] |
Mallick H, Franzosa EA, Mclver LJ, Banerjee S, Sirota-Madi A, Kostic AD, Clish CB, Vlamakis H, Xavier RJ, Huttenhower C. Predictive metabolomic profiling of microbial communities using amplicon or metagenomic sequences[J]. Nature Communications, 2019, 10(1): 3136. DOI:10.1038/s41467-019-10927-1 |
| [28] |
Chan SHJ, Cai JY, Wang L, Simons-Senftle MN, Maranas CD. Standardizing biomass reactions and ensuring complete mass balance in genome-scale metabolic models[J]. Bioinformatics, 2017, 33(22): 3603-3609. DOI:10.1093/bioinformatics/btx453 |
| [29] |
Dufault-Thompson K, Steffensen JL, Zhang Y. Using PSAMM for the curation and analysis of genome-scale metabolic models[J]. New York: Humana Press, 2018, 1716: 131-150. |
| [30] |
Sung J, Hale V, Merkel AC, Kim PJ, Chia N. Metabolic modeling with big data and the gut microbiome[J]. Applied & Translational Genomics, 2016, 10: 10-15. |
| [31] |
Kanehisa M, Furumichi M, Tanabe M, Sato Y, Morishima K. KEGG: new perspectives on genomes, pathways, diseases and drugs[J]. Nucleic Acids Research, 2017, 45(D1): D353-D361. DOI:10.1093/nar/gkw1092 |
| [32] |
Schellenberger J, Park JO, Conrad TM, Palsson BØ. BiGG: a biochemical genetic and genomic knowledgebase of large scale metabolic reconstructions[J]. BMC Bioinformatics, 2010, 11: 213. DOI:10.1186/1471-2105-11-213 |
| [33] |
Caspi R, Billington R, Keseler IM, Kothari A, Krummenacker M, Midford PE, Ong WK, Paley S, Subhraveti P, Karp PD. The MetaCyc database of metabolic pathways and enzymes: a 2019 update[J]. Nucleic Acids Research, 2020, 48(D1): D445-D453. DOI:10.1093/nar/gkz862 |
| [34] |
Benedict MN, Mundy MB, Henry CS, Chia N, Price ND. Likelihood-based gene annotations for gap filling and quality assessment in genome-scale metabolic models[J]. PLoS Computational Biology, 2014, 10(10): e1003882. DOI:10.1371/journal.pcbi.1003882 |
| [35] |
Shoaie S, Ghaffari P, Kovatcheva-Datchary P, Mardinoglu A, Sen P, Pujos-Guillot E, De Wouters T, Juste C, Rizkalla S, Chilloux J, et al. Quantifying diet-induced metabolic changes of the human gut microbiome[J]. Cell Metabolism, 2015, 22(2): 320-331. DOI:10.1016/j.cmet.2015.07.001 |
| [36] |
Lularevic M, Racher AJ, Jaques C, Kiparissides A. Improving the accuracy of flux balance analysis through the implementation of carbon availability constraints for intracellular reactions[J]. Biotechnology and Bioengineering, 2019, 116(9): 2339-2352. DOI:10.1002/bit.27025 |
| [37] |
Harcombe WR, Riehl WJ, Dukovski I, Granger BR, Betts A, Lang AH, Bonilla G, Kar A, Leiby N, Mehta P, et al. Metabolic resource allocation in individual microbes determines ecosystem interactions and spatial dynamics[J]. Cell Reports, 2014, 7(4): 1104-1115. DOI:10.1016/j.celrep.2014.03.070 |
| [38] |
Louca S, Doebeli M. Calibration and analysis of genome-based models for microbial ecology[J]. eLife, 2015, 4: e08208. DOI:10.7554/eLife.08208 |
| [39] |
Chan SHJ, Simons MN, Maranas CD. Steadycom: predicting microbial abundances while ensuring community stability[J]. PLoS Computational Biology, 2017, 13(5): e1005539. DOI:10.1371/journal.pcbi.1005539 |
| [40] |
Larsen PE, Collart FR, Field D, Meyer F, Keegan KP, Henry CS, Mcgrath J, Quinn J, Gilbert JA. Predicted relative metabolomic turnover (PRMT): determining metabolic turnover from a coastal marine metagenomic dataset[J]. Microbial Informatics and Experimentation, 2011, 1(1): 4. DOI:10.1186/2042-5783-1-4 |
| [41] |
Zhu J, Sova P, Xu QW, Dombek KM, Xu EY, Vu H, Tu ZD, Brem RB, Bumgarner RE, Schadt EE. Stitching together multiple data dimensions reveals interacting metabolomic and transcriptomic networks that modulate cell regulation[J]. PLoS Biology, 2012, 10(4): e1001301. DOI:10.1371/journal.pbio.1001301 |
| [42] |
McGeachie MJ, Sordillo JE, Gibson T, Weinstock GM, Liu YY, Gold DR, Weiss ST, Litonjua A. Longitudinal prediction of the infant gut microbiome with dynamic Bayesian networks[J]. Scientific Reports, 2016, 6: 20359. DOI:10.1038/srep20359 |
| [43] |
Shashkova T, Popenko A, Tyakht A, Peskov K, Kosinsky Y, Bogolubsky L, Raigorodskii A, Ischenko D, Alexeev D, Govorun V. Agent based modeling of human gut microbiome interactions and perturbations[J]. PLoS One, 2016, 11(2): e0148386. DOI:10.1371/journal.pone.0148386 |
| [44] |
Bauer E, Zimmermann J, Baldini F, Thiele I, Kaleta C. BacArena: individual-based metabolic modeling of heterogeneous microbes in complex communities[J]. PLoS Computational Biology, 2017, 13(5): e1005544. DOI:10.1371/journal.pcbi.1005544 |
| [45] |
Liu B, Zhang KT, Shen S, Lei CL, Huang W. Immunomodulatory effects of the extract of Musca domestica larvae on irradiated mice[J]. Acta Entomologica Sinica, 2007, 50(9): 889-894. (in Chinese) 刘彬, 张克田, 沈思, 雷朝亮, 黄文. 家蝇幼虫提取物对辐射小鼠的免疫调节作用[J]. 昆虫学报, 2007, 50(9): 889-894. DOI:10.3321/j.issn:0454-6296.2007.09.004 |
| [46] |
Li S. Studies of dynamic absorption of amino acids and peptides from dried-powder of Musca domestica larvae and its effects on changing profile of small intestinal interrelated immunity cell of mucous membrane in improvement San-Huang chickens[D]. Daqing: Master's Thesis of Heilongjiang Bayi Agricultural University, 2009 (in Chinese) 李帅. 家蝇幼虫粉在改良三黄鸡肠道中氨基酸与肽类的动态吸收及对黏膜免疫相关细胞变化的影响[D]. 大庆: 黑龙江八一农垦大学硕士学位论文, 2009 |
| [47] |
Requena T, Burton J, Matsuki T, Munro K, Simon MA, Tanaka R, Watanabe K, Tannock GW. Identification, detection, and enumeration of human Bifidobacterium species by PCR targeting the transaldolase gene[J]. Applied and Environmental Microbiology, 2002, 68(5): 2420-2427. DOI:10.1128/AEM.68.5.2420-2427.2002 |
| [48] |
Zhou YW, Yin YB, Tu ZG, Chen H, Zhang XM, Meng JP, Zhu D. Study on antibacterial activity and mechanism of housefly antimicrobial peptide[J]. Chinese Journal of Antibiotics, 2004, 29(5): 272-274. (in Chinese) 周义文, 尹一兵, 涂植光, 陈辉, 张雪梅, 孟江平, 朱旦. 家蝇抗菌肽抗菌活性及抗菌机制的初步研究[J]. 中国抗生素杂志, 2004, 29(5): 272-274. DOI:10.3969/j.issn.1001-8689.2004.05.005 |
| [49] |
Lu X, Shen J, Jin X, Ma Y, Huang Y, Mei H, Chu F, Zhu J. Bactericidal activity of Musca domestica cecropin (Mdc) on multidrug-resistant clinical isolate of Escherichia coli[J]. Applied Microbiology and Biotechnology, 2012, 95(4): 939-945. DOI:10.1007/s00253-011-3793-2 |
| [50] |
Chu FJ, Jin XB, Liu WB, Zhu JY. Effects on the in vitro antibacterial activity of extraction products of Luo Xianzi (Musca domestica) obtained by different methods[J]. Lishizhen Medicine and Materia Medica Research, 2014, 25(8): 1806-1808. (in Chinese) 褚夫江, 金小宝, 刘文彬, 朱家勇. 不同提取方法对中药罗仙子体外抑菌活性的影响[J]. 时珍国医国药, 2014, 25(8): 1806-1808. |
| [51] |
Zhang L, Gui SQ, Liang ZB, Liu AL, Chen ZX, Tang Y, Xiao MZ, Chu FJ, Liu WB, Jin XB, et al. Musca domestica cecropin (Mdc) alleviates Salmonella typhimurium-induced colonic mucosal barrier impairment: associating with inflammatory and oxidative stress response, tight junction as well as intestinal flora[J]. Frontiers in Microbiology, 2019, 10: 522. DOI:10.3389/fmicb.2019.00522 |
| [52] |
Wang YJ, Chu FJ, Lu XM, Jin XB, Zhu JY. The regulation of the intestinal flora of normal mice by Chinese traditional medicine Cereals[J]. Guangdong Medical Journal, 2015, 36(24): 3761-3762. (in Chinese) 王玉娇, 褚夫江, 卢雪梅, 金小宝, 朱家勇. 中药五谷虫对正常小鼠肠道菌群的调节作用[J]. 广东医学, 2015, 36(24): 3761-3762. |
 2021, Vol. 48
2021, Vol. 48




