扩展功能
文章信息
- 杨兴涛, 董梦瑶, 周紫峣, 刘海峰, 钟志军, 彭广能
- YANG Xingtao, DONG Mengyao, ZHOU Ziyao, LIU Haifeng, ZHONG Zhijun, PENG Guangneng
- 细菌RNA结合蛋白Hfq的DNA压缩和复制作用研究进展
- Research progress on DNA compaction and replication of Hfq, a bacterial RNA binding protein
- 微生物学通报, 2021, 48(4): 1281-1291
- Microbiology China, 2021, 48(4): 1281-1291
- DOI: 10.13344/j.microbiol.china.200579
-
文章历史
- 收稿日期: 2020-06-09
- 接受日期: 2020-07-31
- 网络首发日期: 2020-09-22
RNA结合蛋白(RNA-Binding Protein) Hfq是近半个世纪前在大肠杆菌(Escherichia coli)中作为噬菌体Qβ进行RNA复制所必需的一种寄主因子而被发现的,最初被命名为Host Factor for RNA Phage Qβ Replication (HF-1)[1-2]。Hfq是RNA结合蛋白家族的一员,而这种蛋白质家族在真核生物、原核生物和古细菌中均有发现[3-4]。已完成的细菌全基因组测序结果表明,约50%的细菌中含有Hfq,而且编码该蛋白的基因序列都具有高度的相似性和保守性[3, 5]。
自从发现Hfq可在RNA代谢中发挥调控作用以来,对Hfq的研究大多集中在该蛋白对小分子非编码RNA (Small Non-Coding RNA,sRNA)和mRNA的作用上[6-8]。Hfq最典型的功能是促进sRNA与其靶标mRNA碱基配对,在转录后介导对RNA稳定性和翻译的调控[3, 8-9]。此外,Hfq也能与多种蛋白质直接或间接相互作用,在RNA稳定性、转录终止和核糖体代谢中发挥重要功能[10-12]。然而,Hfq除了在RNA和蛋白质代谢中发挥调控作用外,还被鉴定为细菌的拟核相关蛋白(Nucleoid Associated Proteins,NAP)之一[13]。细胞定位试验表明,细菌中约20%的Hfq与DNA结合,这种结合被认为是非序列特异性的[13-15]。2010年,Updegrove等曾通过碱基突变和凝胶迁移滞后实验(Electrophoretic Mobility Shift Assays,EMSA)证明,Hfq远端面和CTR中的碱基突变会抑制该蛋白与双链(Double Stranded,ds) DNA的结合,而近端面和侧面似乎在Hfq与dsDNA结合方面不起作用[16]。但由于Hfq在RNA代谢和蛋白质代谢中的研究热潮,该蛋白与DNA之间的相互作用在此后的很长一段时间里未得到较好的研究。
随着Hfq在DNA复制和DNA压缩中的作用被逐步揭示,这种功能强大的细菌RNA结合蛋白和拟核相关蛋白在DNA代谢中的潜在作用再次引起了研究者们的关注[17-18]。近年来的一系列研究表明,Hfq可以通过调控DNA桥接(DNA-Bridging)、拟核蛋白自组装(Self-Assembly of Nucleoid Proteins)和DNA超螺旋(DNA Supercoiling)的发生,共同促进DNA压缩(DNA Compaction)[17, 19-20]。此外,Hfq也被证明可直接或间接调控DNA复制效率、错配修复和基因突变等DNA复制(DNA Replication)相关活动,预示着该蛋白对细菌环境适应和基因组进化可能存在更为复杂的调控网络[19, 21-22]。额外的靶标和功能的鉴定将进一步夯实Hfq作为细菌中多种代谢途径核心调控因子的重要地位,也表明该蛋白的功能并不局限于其在RNA和蛋白质代谢中的作用。本文主要介绍了Hfq与其新靶标DNA的相互作用及其相应功能,并讨论了Hfq在DNA代谢调控中的重要价值。
1 Hfq的基本结构和功能通常来说,Hfq的每个亚基均包含1个结构性的氨基(N)末端区域(N-Terminal Region,NTR)和1个非结构性的羧基(C)末端区域(C-Terminal Region,CTR)[23-24]。其中,NTR由1个α螺旋和5个高度扭曲且弯曲的反平行β链组成,α螺旋和β链可共同形成Sm折叠;不同亚基间的Sm折叠可发生相互作用,最终组装成一个含有6个亚基的圆盘状Hfq六聚体;圆盘中心有孔,并存在至少3个RNA结合面(近端面、侧面和远端面)和1个CTR (图 1)[3, 7, 25]。sRNA或mRNA可在转录后过程中与Hfq的3个RNA结合面或CTR进行不同方式的结合[7]。

|
| 图 1 大肠杆菌Hfq的结构模型 Figure 1 Structural model of Escherichia coli Hfq 注:A、B、C、D中不同颜色的箭头分别代表Hfq的近端面(NTR)、远端面、侧面和CTR。其中,黑色箭头代表近端面(NTR),紫色箭头代表远端面,红色箭头代表侧面,绿色箭头代表CTR Note: The different colored arrows in A, B, C and D represent the proximal face (NTR), rim, distal face, and CTR of the Hfq, respectively. Among them, the black arrow represents the proximal face (NTR), the purple arrow represents the distal face, the red arrow represents the rim, and the green arrow represents CTR |
|
|
Hfq最典型的功能是促进sRNA与其靶标mRNA碱基配对,在转录后介导对RNA稳定性和翻译的调控[3, 8-9]。除了sRNA和mRNA,Hfq也能与其他RNA (如rRNA和tRNA)相互作用,正向调控核糖体生物合成、rRNA加工和tRNA成熟等生命活动过程[26-28]。此外,Hfq也能直接或间接调控核糖核酸酶E (RNase E)、PolyA聚合酶I [Poly (A) Polymerase I,PAP I]、Rho和热不稳定延伸因子(Elongation Factor Thermo Unstable,EF-Tu)等蛋白质的活性,在RNA稳定性、转录终止和核糖体代谢中发挥重要作用[10-12]。Hfq缺失后,大多数革兰氏阴性菌在其生长、运动、生物膜形成、毒力和环境胁迫的适应等方面都表现出不同程度的缺陷[29-32]。相比而言,Hfq在革兰氏阳性菌中的研究较少。不过,现在可以明确的是,单核球增多性李斯特菌(Listeria monocytogenes)的生长、生物膜形成和毒力与Hfq的存在密切相关[33-34]。此外,Hfq在枯草芽孢杆菌(Bacillus subtilis)的趋化性、运动性和生长稳定期存活中的作用最近也被证实[35-37]。总而言之,大量研究已经表明,Hfq可作为一种核心调控因子参与细菌中多种代谢途径的直接或间接调控[7, 38]。
2 Hfq在DNA压缩中的研究进展正常情况下,细菌中的DNA双螺旋被压缩成一种紧凑状态并形成拟核[39]。DNA压缩是不同机制协同作用的结果,包括DNA桥接、DNA弯曲(DNA-Bending)、DNA超螺旋、拟核蛋白自组装和相分离(Phase Separation)[19, 40]。截至目前,这种同时作为RNA结合蛋白和拟核相关蛋白的Hfq已被证明可通过调控DNA桥接、拟核蛋白自组装和DNA超螺旋来发挥其在DNA压缩中的作用(图 2)[17, 19-20]。
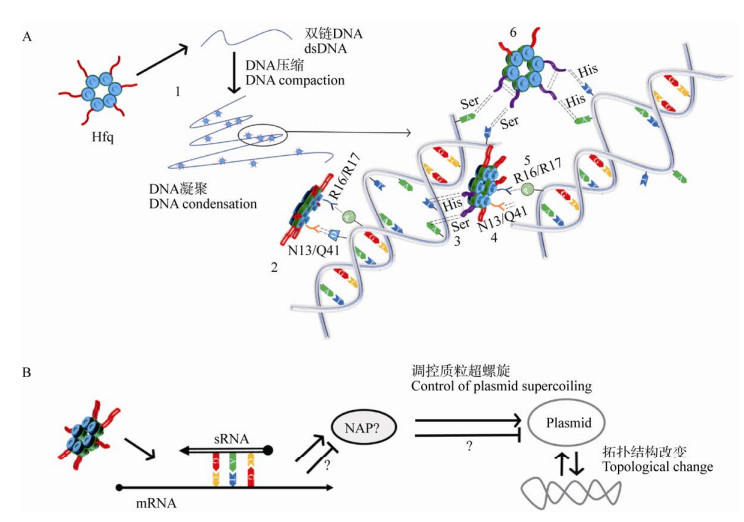
|
| 图 2 Hfq调控DNA压缩的机制 Figure 2 The mechanism of Hfq regulating DNA compaction 注:A:Hfq调控DNA桥接和核蛋白自组装,促进DNA压缩。1:Hfq和dsDNA相互作用后,可将dsDNA压缩成凝聚形式;2:NTR中的残基可分别以静电相互作用(残基R16/R17与dsDNA磷酸基团)和形成氢键(残基N13/Q41与dsDNA磷酸基团中的O原子)的方式与dsDNA的磷酸骨架形成初步不稳定结合;3:CTR中的His/Ser残基和dsDNA (部分富含A/T的双链解开)中的A/T以形成氢键的方式锚定结合,锚定结合后的1个或多个CTR可在dsDNA的诱导下自组装成淀粉样蛋白纤维;4:CTR与dsDNA锚定结合且自组成淀粉样蛋白纤维后,原本与dsDNA形成初步不稳定结合的残基R16/R17或(和) N13/Q41从磷酸骨架上脱落;5:空出结合位点的R16/R17或(和) N13/Q41残基重新与新的dsDNA形成如2中所述的结合;6:已与dsDNA片段锚定结合且自组成淀粉样蛋白纤维的1个或多个CTR可将其剩余的CTR与另一dsDNA片段锚定结合且自组成淀粉样蛋白纤维,实现DNA桥接,从而促进DNA压缩。B:Hfq调控DNA超螺旋,促进DNA压缩。Hfq可能通过促进sRNA (DNA拓扑结构相关)与其靶标mRNA间的多种相互作用,调控某一NAPs的表达,从而实现对DNA拓扑结构的间接调控(维持DNA超螺旋),促进DNA压缩 Note: A: Hfq regulates DNA-bridging and self-assembly of nucleoid proteins to promote DNA compaction. 1: After the interaction between Hfq and dsDNA, dsDNA can be compacted into condensed form; 2: The residues in NTR can form an initial unstable binding with the phosphate skeleton of dsDNA by electrostatic interaction (residue R16/R17 with dsDNA phosphate group) and hydrogen bond (residue N13/Q41 with O atom in dsDNA phosphate group), respectively; 3: The His/Ser residues in CTR and A/T in dsDNA (partial dissociation of double strands rich in uracil) are anchored to form hydrogen bonds. One or more CTR after anchoring binding can be self-assembled into amyloid fibers under the induction of dsDNA; 4: After CTR anchored with dsDNA and self-assembled amyloid fiber, residues R16/R17 or (and) N13/Q41 which initially formed unstable binding with dsDNA fell off from the phosphate skeleton; 5: The R16/R17 or (and) N13/Q41 residues of the vacated binding site are recombined with the new dsDNA, as described in 2; 6: One or more CTR that have anchored dsDNA fragments and self-assembled amyloid fibers can anchor the remaining CTR to another dsDNA fragment and form amyloid fibers themselves to achieve DNA bridging, thereby promoting DNA compaction. B: Hfq regulates DNA supercoiling to promote DNA compaction. Hfq may regulate the expression of a certain NAPs by promoting a variety of interactions between sRNA (related to DNA topology) and its target mRNA, thus indirectly regulating the topology of DNA (maintain DNA supercoiling) and promoting DNA compaction |
|
|
Hfq在DNA桥接中的作用最早是在2015年被发现的[17]。一项基于纳米流体学(Nanofluidics)、中子小角散射(Small Angle Neutron Scattering,SANS)和原子力显微镜(Atomic Force Microscopy,AFM)的试验数据表明,当Hfq的浓度超过阈值时,dsDNA可在纳米通道内被压缩成凝聚形式(Condensed Form),并且这种压缩效应随着缓冲液离子强度、纳米通道横断面积和Hfq浓度的增加而增强[17]。同样,作为NAPs的H-NS和HU也先后被证实可在纳米通道内将dsDNA压缩成凝聚形式,不过相比而言,Hfq在纳米通道内的DNA压缩效率远高于H-NS和HU[17, 41-42]。例如,当缓冲液(含有3 mmol/L NaCl的T-Buffer)和纳米通道横断面积(200×300 nm2)等其他条件均一致时,Hfq、H-NS和HU将dsDNA压缩成凝聚形式的阈值浓度分别为0.005 μmol/L (Hfq: bp=1:1 000)、0.5 μmol/L (Hfq: bp=1:10)和0.5 μmol/L (Hfq: bp= 1:10)[17, 41-42]。Hfq也可以在非限制条件(无纳米通道限制)下将dsDNA压缩成凝聚形式,不过这需要更高的蛋白浓度;在AFM中观察到,高浓度下的Hfq (Hfq: bp=1:15)可与较长的dsDNA片段(10 kb)相互作用,并形成不同片段间的DNA桥接,桥接导致长链DNA片段中的反向折叠(Back Folding)、环化(Looping)和并行聚集(Side-by-Side Aggregation)增多;为了排除DNA分子内的并行聚集等不利因素对持续长度(Persistence Length)和轮廓总长(Contour Length)分析的影响,短链DNA片段(1 000 bp)被用于后续AFM试验,结果表明,低浓度组(Hfq: bp= 1:300)的持续长度和轮廓总长相应为49.1±0.3 nm和350±20 nm,而高浓度组(Hfq: bp=1:6)则分别为60.4±0.3 nm和270±20 nm,简单分析可知,低浓度组的持久长度和轮廓总长与细菌中典型的B型裸露DNA力学性质(持续长度为50 nm,轮廓总长为340 nm)相似,而高浓度组的持续长度和轮廓总长则分别升高和降低约20%,升高的持续长度表明Hfq与dsDNA的结合并不是牢固的对接,相反,该蛋白与dsDNA结合后仍然保持了其“灵活性”[17]。SANS试验结果进一步支持了该观点,分析表明,Hfq和dsDNA间还存在约5 nm的径向距离,Hfq可能仅通过其一小部分位点与dsDNA结合并形成核蛋白复合物,该复合物中的Hfq似乎是“悬挂”在dsDNA上的[17]。这种结合方式与在H-NS和HU中发现的均不同,相比于Hfq,H-NS与dsDNA的结合更加紧密,而HU与dsDNA的结合则会导致dsDNA发生强烈弯曲(105°–140°)[17, 41-42]。此外,研究者认为,高浓度组中轮廓总长的降低可能与Hfq和dsDNA结合过程中富含AT碱基的双链打开有关[17]。总而言之,该研究发现,Hfq和细菌拟核可形成一种核蛋白复合物,后者能改变dsDNA的力学性质,并将dsDNA压缩成凝聚形式[17]。这种强烈的压缩效应与Hfq和dsDNA孵育后在AFM中观察到的DNA桥接密切相关[17]。事实上,已经有人提出,CTR可沿着大沟或小沟与dsDNA磷酸骨架相互作用[7]。结合SANS试验中证明的Hfq在dsDNA上的部分结合,加上该蛋白本身作为六聚体并且含有CTR的结构特性,可以认为Hfq具有较强的桥接DNA的倾向[3, 7, 17]。然而,Hfq是如何介导不同dsDNA片段间的DNA桥接,并最终将dsDNA压缩成凝聚形式的具体作用机制还有待进一步阐明。
最近,CTR在DNA桥接中的作用被进一步强调[20]。研究者们将大肠杆菌的Hfq NTR (残基1–72)和CTR (残基64–102)片段连接表达质粒后在大肠杆菌BL21中进行了蛋白表达和纯化,并对独立的NTR和CTR进行了较为系统的DNA压缩特性分析[20],结果表明,随着Hfq、NTR或CTR浓度的增加,Hfq和CTR在纳米通道和非纳米通道组成的系统中均可将DNA压缩成凝聚形式,但相比于完整的Hfq,CTR要实现DNA凝聚(DNA Condensation)则需要更高的浓度[17, 20]。出现这种浓度梯度的差异可能与CTR中NTR的结构缺失有关,虽然NTR已被证实不能直接将DNA压缩成凝聚形式,但其能促进Hfq与dsDNA的结合[20]。该研究认为,Hfq首先通过其六聚体中带正电荷的侧面或远端面残基与dsDNA中带负电荷的磷酸盐以静电相互作用的方式初步结合,并将六聚体“粘贴”在dsDNA表面;Hfq与dsDNA形成初步不稳定结合后,该蛋白的一个或多个CTR再利用氢键(如CTR中相对丰富的组氨酸和丝氨酸残基)等非静电相互作用与局部解开双链的DNA碱基(如A和/或T)进行锚定结合,从而达到稳定的结合状态;一旦Hfq与dsDNA稳定结合后,该蛋白可将其剩余的一个或多个CTR继续锚定结合在另一dsDNA片段上,实现DNA桥接[20]。
简而言之,Jiang等和Malabirade等的研究结果表明,Hfq主要通过其CTR来发挥桥接DNA的作用,DNA桥接能促进DNA压缩,最终导致dsDNA被压缩成凝聚形式[17, 20]。NTR的单独作用并不能直接导致DNA凝聚的发生,但其可以通过组装形成的六聚体以静电相互作用的方式与dsDNA初步结合;这种初步不稳定结合有利于CTR在dsDNA上的结合锚定,从而使得DNA桥接更容易进行[20]。Orans等对大肠杆菌Hfq NTR (残基2–69)和dsDNA形成的晶体复合物进行了分析,试验结果也支持NTR在DNA桥接中的辅助作用[24],不过该研究与Updegrove等和Malabirade等的研究结果存在差异[16, 20, 24]。比如,Malabirade等认为NTR仅通过静电相互作用与dsDNA结合[20]。然而,最新的晶体复合物研究表明,NTR除了可以借助其R16和R17残基与dsDNA发生静电相互作用外,还可以通过其N13和Q41残基与dsDNA形成氢键[24]。尽管现在还没有文献对CTR和dsDNA形成的晶体复合物进行分析,但现有研究已经表明,CTR在生理pH条件下是电中性的[20]。因此,CTR与dsDNA的结合几乎不可能涉及静电相互作用。考虑到CTR中存在丰富的组氨酸和丝氨酸残基,形成氢键应该是其与dsDNA较为理想的结合方式[20]。不过,Malabirade等和Orans等的研究结果均进一步支持NTR相对于CTR具有更高的亲和力,所以更容易在DNA桥接中产生初步结合,从而间接调控DNA压缩这一事实[20, 24]。此外,晶体复合物结构分析表明,NTR中唯一发现的与dsDNA存在相互作用的4个残基(R16、R17、N13和Q41)均存在于其六聚体近端面的α螺旋中[24]。因此,Updegrove等此前关于Hfq的近端面在该蛋白与dsDNA结合中不发挥作用的论断可被推翻[16, 24]。
2.2 Hfq通过自组装成淀粉样蛋白纤维促进DNA压缩淀粉样蛋白(Amyloids)是一种纳米结构,由多个拷贝的肽或蛋白质单体聚集而成,最终形成纤维[43]。淀粉样蛋白纤维(Amyloid Fibers)通常被认为是神经退行性病变的标志之一,但也存在不少关于细菌表面蛋白淀粉样和纤维化的报道[44-45]。随着细菌DNA压缩概念的阐明,拟核蛋白自组装也被定义为促进DNA压缩的主要机制之一[40]。作为拟核相关蛋白的Hfq,最近被证实可通过其CTR在体外自组装成淀粉样蛋白纤维,但这种体外的自组装似乎是一种相当缓慢的过程,研究还发现,Hfq的完整组装在很大程度上得益于其CTR具有的淀粉样蛋白自组装功能,然而研究者并未将这种强大的自组装能力与DNA压缩联系在一起[46]。
鉴于CTR在DNA压缩中的作用已经显现,Malabirade等利用同步辐射圆二色谱(Synchrotron Radiation Circular Dichroism,SRCD)、傅里叶变换红外光谱(Fourier Transform Infrared Spectroscopy,FTIR Spectroscopy)和小角X射线散射(Small Angle X-Ray Scattering,SAXS)分别研究了DNA与CTR具有的淀粉样蛋白自组装功能之间的潜在联系[19-20, 46]。结果表明,无论CTR是否与dsDNA共同孵育,CTR仍然能在体外自组装成淀粉样蛋白纤维;不过,相比于和dsDNA共同孵育(7 d),单独孵育的CTR要实现淀粉样蛋白纤维化则需要更长的时间(> 14 d),因此该研究发现,DNA能诱导CTR在体外自组装成淀粉样蛋白纤维,在体内可能也是如此;此外,研究者还认为,CTR在其淀粉样蛋白纤维化中发生的自组装促进了DNA桥接和DNA压缩[19]。
总而言之,Hfq现已被证明可通过其CTR在体外自组装成淀粉样蛋白纤维,DNA可诱导CTR实现淀粉样蛋白纤维化,而纤维化过程中发生的自组装促进了DNA桥接和DNA压缩[19-20, 46]。实际上,纤维化后的蛋白质其刚度会变得更高,而刚度的增加正好有利于稳定CTR和dsDNA间形成的DNA桥接,从而进一步促进DNA压缩[47]。
2.3 Hfq通过正向调控DNA超螺旋的发生促进DNA压缩DNA除了作为遗传信息的载体,也是控制基因表达的重要因素,这种调控机制主要源于其拓扑结构的动态变化。超螺旋是DNA拓扑结构的主要形式,其可分为正超螺旋和负超螺旋两类,在一定条件下,这两者可以相互转变[48]。基于Hfq插入失活的早期研究表明,Hfq的失活可减少大肠杆菌中质粒DNA形成的超螺旋,使质粒更加松弛[30]。然而,这种表型差异与Hfq之间的相互作用关系并未阐明。
Malabirade等在体外和体内分别研究了Hfq与质粒超螺旋的相互作用,并讨论了CTR对质粒超螺旋的影响:纯化的Hfq和pHSG298质粒完成共同孵育并经拓扑异构酶以及质粒DNA去蛋白处理后,被用来进行凝胶电泳分析,在此条件下,超螺旋DNA的凝胶迁移速度相比于非超螺旋DNA更快;拓扑异构酶的处理会减少质粒DNA形成的超螺旋,其对应的凝胶迁移速度也就更慢;体外试验观察到,无论Hfq是否与质粒DNA共同孵育,经拓扑异构酶处理后的质粒DNA超螺旋程度均降低至同一水平,这些数据表明,Hfq并不能直接调控质粒DNA超螺旋结构的动态变化[19]。因此,早期研究发现的Hfq在质粒DNA超螺旋结构变化中的调控作用可能是该蛋白在体内间接调控其他基因的结果[19, 30]。体内试验则主要是对分别来源于大肠杆菌野生型、Hfq72 (=∆CTR)和∆Hfq菌株的pHSG298质粒进行同样的凝胶电泳分析,数据显示,来源于大肠杆菌∆Hfq菌株的质粒超螺旋程度明显低于野生型和Hfq72 (=∆CTR)[19]。这与Tsui等的早期研究结果相似,即Hfq的缺失可影响质粒DNA的超螺旋程度[19, 30]。此外,Malabirade等还发现,尽管CTR已被证明可在DNA压缩中发挥重要作用,但CTR与DNA间的直接相互作用并不会显著影响超螺旋形式的拓扑结构,这一点在体内试验中得到了证实,即无论CTR是否存在,从不同菌株中提取的pHSG298在凝胶电泳分析中均表现出相似的质粒DNA超螺旋程度[19]。
总而言之,研究已经表明,Hfq缺失所产生的质粒超螺旋结构改变并不是由该蛋白与质粒DNA直接相互作用的结果[19]。但无论如何,Hfq在DNA超螺旋结构维持中的正向调控作用已经显现,而DNA超螺旋也是促进DNA压缩的主要机制之一[19, 30, 40]。因此,该蛋白也可以通过正向调控DNA超螺旋的形成,进而促进DNA压缩[19, 30, 40]。考虑到Hfq在RNA代谢调控中的多种相互作用,可以假设这种质粒超螺旋的结构变化是通过某些依赖于Hfq的sRNA调控某些DNA拓扑结构相关mRNA的表达而导致的间接效应[3, 7, 19]。至少,现在可以明确的是,Hfq可以在体外结合sRNA-DsrA,该蛋白的体内缺失除了会降低sRNA-DsrA的稳定性以外,还会影响sRNA-DsrA对部分蛋白翻译起始的转录后调控,比如H-NS[49]。H-NS已被证实可通过调控DNA拓扑结构(如超螺旋)的动态变化在DNA压缩等DNA代谢过程中发挥重要作用[50]。
3 Hfq在DNA复制中的研究进展 3.1 Hfq在质粒DNA复制效率中的作用Hfq在DNA复制中的作用最早是在ColE1型质粒中发现的,该研究利用基因敲除探索了Hfq对ColE1型质粒(pET22b、pUC18和pACYC177)和噬菌体λ衍生质粒(pKBamp) DNA复制效率的影响,结果表明,相较于Hfq缺失株,存在于野生型菌株的ColE1型质粒在指数早期的复制效率更高,而在指数晚期和稳定期早期中的复制效率则更低;相反,Hfq对噬菌体λ衍生质粒的DNA复制效率并无显著影响[18]。
Malabirade等同样发现了大肠杆菌Hfq的缺失会导致ColE1型质粒(pHSG298) DNA复制效率相对于野生株更高,这与Cech等的研究结果一致[18-19]。该研究分别在指数晚期和稳定期对来源于大肠杆菌野生型、Hfq72 (=∆CTR)和∆Hfq菌株的pHSG298质粒进行了数量统计,结果发现,野生型和Hfq72中的质粒数量均显著低于∆Hfq菌株[19]。Hfq的缺失已被证明会显著影响质粒DNA超螺旋和DNA压缩,而CTR的缺失只会影响DNA压缩[17, 19-20]。在这种情况下,如果Hfq缺失引起的DNA复制与DNA压缩直接相关,则Hfq72中的CTR缺失也会影响质粒数量;然而,来源于野生型和Hfq72的质粒数量在指数晚期和稳定期均未观察到显著差异,因此该研究认为,Hfq在质粒DNA复制效率中的负调控作用与质粒DNA超螺旋程度的改变直接相关,而与质粒DNA压缩无关[19]。
事实上,虽然大多数质粒DNA的复制都需要负超螺旋形式的拓扑结构来促进环状dsDNA中复制位点的打开,但对于拥有RNA II作为其复制起始位点的ColE1型质粒来说,超螺旋减少导致的质粒松弛将更有利于DNA复制[19, 51]。不过,Hfq对ColE1型质粒DNA复制效率的具体调控机制还有待进一步阐明。
3.2 Hfq在基因突变和错配修复中的作用细菌在DNA复制或转录等过程中可形成G-四链体(G-Quadruplex),这种类型的拓扑结构会阻碍DNA复制,甚至会导致基因突变[21]。研究发现,大肠杆菌中也存在G-四链体,Hfq可在体外特异性结合并稳定该结构,从而促进G-四链体在DNA复制中的负调控(如复制效率降低,基因突变增加)[21]。此外,Hfq对DNA复制的负调控效应比想象中更大,因为这种RNA结合蛋白和拟核相关蛋白还被证明可以在转录后抑制错配修复(MMR)基因mutS和mutH的表达[52]。Chen等证实,Hfq在生长稳定期中对mutS的抑制是通过在其5′非翻译区(UTR)内的直接结合和与sRNA的相互作用而实现[22]。
尽管Hfq在质粒DNA和染色体(拟核) DNA的复制中都表现出了明显的负调控作用,但这种调控的意义尚不清楚[18-19, 21]。研究者们更倾向于将Hfq对染色体(拟核) DNA复制的这种负调控行为(错配修复能力降低,基因突变率增加)归因于该蛋白对环境应激做出的适应,因为这种调控效应无论是对于细菌的应激诱导突变还是基因表达调控的进化而言均是有益的[21-22]。
4 存在的问题和挑战细菌中的DNA大多都以负超螺旋形式的B型DNA存在,在这种情况下,细菌可将通过挫折欠旋而节约的能量(如降低的扭曲压力和碱基对的旋转)重新分配给可用于驱动转录、复制和转移等DNA代谢过程的双链[48]。然而,ColE1型质粒DNA是独立于染色体之外能自主复制的一类双链DNA分子[19, 51]。RNA II是一种作为复制起始引物的RNA,可正调控质粒DNA复制,而RNA I (RNA II的反义RNA)和RNA II形成的复合物则会严重阻碍质粒DNA复制[51]。Rom基因能将RNA I-RNA II复合物进一步稳定,从而负调控DNA复制[19, 51]。考虑到Hfq在RNA代谢调控中介导的多种RNA-RNA相互作用,以及Cech等和Malabirade等在ColE1∆Rom型质粒中观察到的Hfq对DNA复制效率的调控降低,可以假设Hfq对ColE1型质粒的调控作用主要是通过位于其NTR或CTR中的RNA结合位点分别与RNA II和RNA I结合,从而引导RNA I-RNA II复合物的形成[7, 18-19]。这种假设的Hfq功能与Rom类似,即两者均能间接抑制质粒DNA的复制,但Hfq并不参与RNA I-RNA II复合物的进一步稳定。然而,Hfq表现出的对质粒DNA复制效率的负调控到底具有何种意义,仍然是一个悬而未决的问题。一种可能的意义是,Hfq对高拷贝ColE1型质粒DNA复制的抑制是为了将其数量控制在菌体可接受的范围内。此外,体内质粒数量过多也并不利于拟核DNA的表达。
尽管近年来的研究已经揭示了Hfq与RNA、蛋白质和DNA的多种相互作用,但这些研究绝大多数都是在革兰氏阴性菌特别是大肠杆菌中进行的[10, 24, 53]。相比于革兰氏阴性菌,Hfq在革兰氏阳性菌中的研究较少。Hfq在革兰氏阳性菌趋化性和运动性中的正向调控作用已在枯草芽孢杆菌中被证实,此外,研究还发现Hfq可能在枯草芽孢杆菌环境胁迫方面发挥正向调控作用[35]。本实验室先前的工作已经证明了纤维素环境对于枯草芽孢杆菌来说是一种压力胁迫环境,尽管如此,该菌株仍然可以通过降解纤维素来获得维持自身存活的碳源,并通过调控相关基因的表达来逐渐适应这种压力环境[54-56]。值得一提的是,在枯草芽孢杆菌中,内切葡聚糖酶、β-葡萄糖苷酶和类膨胀蛋白BsEXLX1这3种功能蛋白对纤维素的微生物降解至关重要,但该系统的具体作用机制仍未阐明[57-58]。鉴于Hfq与多种蛋白质的直接相互作用已经显现,一种可能的假设是,枯草芽孢杆菌Hfq能直接与上述3种蛋白的一种或几种产生相互作用,从而实现纤维素环境的适应[10, 35, 54]。另一方面,考虑到近年来发现的Hfq在DNA代谢中的强大功能,该蛋白也可能通过间接调控其他NAPs的表达而改变DNA的拓扑结构,从DNA层面实现对上述3种蛋白中的一种或几种的调控,进而有助于枯草芽孢杆菌在纤维素环境中的适应[19, 54, 59]。至少,在大肠杆菌中已经发现了Hfq能与同样作为NAPs的H-NS相互作用[49]。因此,对Hfq与上述3种蛋白相互作用的深入研究将有助于进一步阐明枯草芽孢杆菌适应纤维素环境的具体作用机制。
5 结语综上所述,Hfq的功能已经超出了其在RNA和蛋白质代谢中的作用,除了RNA (sRNA、mRNA、rRNA、tRNA等)和蛋白质这些已知的靶标,Hfq还可以与DNA相互作用。近年来的一系列研究表明:Hfq不仅可以通过调控DNA桥接、拟核蛋白自组装和DNA超螺旋的发生来促进DNA压缩,还被证明可对DNA复制相关活动产生负调控行为(复制效率和错配修复能力降低,而基因突变率增加)。因此,Hfq的调控谱比先前设想的要大。Hfq与DNA的相互作用将有助于进一步理解该蛋白在细菌中的整体功能,也表明Hfq的调控能力并不局限于其在RNA和蛋白质代谢中的作用。
| [1] |
Carmichael GG, Weber K, Niveleau A, Wahba AJ. The host factor required for RNA phage Qβ RNA replication in vitro[J]. The Journal of Biological Chemistry, 1975, 250(10): 3607-3612. DOI:10.1016/S0021-9258(19)41440-3 |
| [2] |
De Fernandez MTF, Eoyang L, August JT. Factor fraction required for the synthesis of bacteriophage Qβ-RNA[J]. Nature, 1968, 219(5154): 588-590. DOI:10.1038/219588a0 |
| [3] |
Vogel J, Luisi BF. Hfq and its constellation of RNA[J]. Nature Reviews Microbiology, 2011, 9(8): 578-589. DOI:10.1038/nrmicro2615 |
| [4] |
Fan B, Chen S, Li YL. Structure, function and mechanisms of bacterial protein Hfq[J]. Journal of Nanjing Forestry University (Natural Sciences Edition), 2016, 40(5): 155-162. (in Chinese) 樊奔, 陈晟, 李昱龙. 细菌Hfq蛋白的结构、功能及作用机制[J]. 南京林业大学学报: 自然科学版, 2016, 40(5): 155-162. |
| [5] |
Sun XG, Zhulin I, Wartell RM. Predicted structure and phyletic distribution of the RNA-binding protein Hfq[J]. Nucleic Acids Research, 2002, 30(17): 3662-3671. DOI:10.1093/nar/gkf508 |
| [6] |
Xu QL, Song YZ, Guo YB. Non-coding RNA and RNA chaperone Hfq in bacteria[J]. Acta microbiologica Sinica, 2018, 58(9): 1511-1520. (in Chinese) 徐巧林, 宋艳钊, 郭岩彬. 细菌非编码RNA及其分子伴侣Hfq[J]. 微生物学报, 2018, 58(9): 1511-1520. |
| [7] |
Updegrove TB, Zhang AX, Storz G. Hfq: the flexible RNA matchmaker[J]. Current Opinion in Microbiology, 2016, 30: 133-138. DOI:10.1016/j.mib.2016.02.003 |
| [8] |
Guo YF, Yuan JY, Wang YF, Cui MQ. Research progress of bacterial RNA binding protein Hfq[J]. Journal of Preventive Medicine of Chinese People's Liberation Army, 2015, 33(5): 584-586. (in Chinese) 郭英飞, 袁玖云, 王玉飞, 崔明全. 细菌RNA结合蛋白Hfq研究进展[J]. 解放军预防医学杂志, 2015, 33(5): 584-586. |
| [9] |
Liu FR, Zhong ZJ, Zhou ZY, Peng GN, Yang P, Wang YP, Liao L. The mechanism of small RNAs response to environmental stresses in bacteria[J]. Microbiology China, 2015, 42(10): 2012-2018. (in Chinese) 刘福芮, 钟志军, 周紫峣, 彭广能, 杨平, 王亚萍, 廖莉. 细菌sRNA对环境胁迫的响应机制[J]. 微生物学通报, 2015, 42(10): 2012-2018. |
| [10] |
Caillet J, Baron B, Boni IV, Caillet-Saguy C, Hajnsdorf E. Identification of protein-protein and ribonucleoprotein complexes containing Hfq[J]. Scientific Reports, 2019, 9(1): 14054. DOI:10.1038/s41598-019-50562-w |
| [11] |
Rabhi M, Espéli O, Schwartz A, Cayrol B, Rahmouni AR, Arluison V, Boudvillain M. The Sm-like RNA chaperone Hfq mediates transcription antitermination at rho-dependent terminators[J]. The EMBO Journal, 2011, 30(14): 2805-2816. DOI:10.1038/emboj.2011.192 |
| [12] |
Mohanty BK, Maples VF, Kushner SR. The Sm-like protein Hfq regulates polyadenylation dependent mRNA decay in Escherichia coli[J]. Molecular Microbiology, 2004, 54(4): 905-920. DOI:10.1111/j.1365-2958.2004.04337.x |
| [13] |
Azam TA, Ishihama A. Twelve species of the nucleoid-associated protein from Escherichia coli sequence recognition specificity and DNA binding affinity[J]. The Journal of Biological Chemistry, 1999, 274(46): 33105-33113. DOI:10.1074/jbc.274.46.33105 |
| [14] |
Diestra E, Cayrol B, Arluison V, Risco C. Cellular electron microscopy imaging reveals the localization of the Hfq protein close to the bacterial membrane[J]. PLoS One, 2009, 4(12): e8301. DOI:10.1371/journal.pone.0008301 |
| [15] |
Azam TA, Hiraga S, Ishihama A. Two types of localization of the DNA-binding proteins within the Escherichia coli nucleoid[J]. Genes to Cells, 2000, 5(8): 613-626. DOI:10.1046/j.1365-2443.2000.00350.x |
| [16] |
Updegrove TB, Correia JJ, Galletto R, Bujalowski W, Wartell RM. E. coli DNA associated with isolated Hfq interacts with Hfq's distal surface and C-terminal domain[J]. Biochimica et Biophysica Acta (BBA)-Gene Regulatory Mechanisms, 2010, 1799(8): 588-596. DOI:10.1016/j.bbagrm.2010.06.007 |
| [17] |
Jiang K, Zhang C, Guttula D, Liu F, Van Kan JA, Lavelle C, Kubiak K, Malabirade A, Lapp A, Arluison V, et al. Effects of Hfq on the conformation and compaction of DNA[J]. Nucleic Acids Research, 2015, 43(8): 4332-4341. DOI:10.1093/nar/gkv268 |
| [18] |
Cech GM, Pakuła B, Kamrowska D, Wegrzyn G, Arluison V, Szalewska-Pałasz A. Hfq protein deficiency in Escherichia coli affects ColE1-like but not λ plasmid DNA replication[J]. Plasmid, 2014, 73: 10-15. |
| [19] |
Malabirade A, Partouche D, Hamoui OE, Turbant F, Geinguenaud F, Recouvreux P, Bizien T, Busi F, Wien F, Arluison V. Revised role for Hfq bacterial regulator on DNA topology[J]. Scientific Reports, 2018, 8(1): 16792. DOI:10.1038/s41598-018-35060-9 |
| [20] |
Malabirade A, Jiang K, Kubiak K, Diaz-Mendoza A, Liu F, Van Kan JA, Berret JF, Arluison V, Van Der Maarel JRC. Compaction and condensation of DNA mediated by the C-terminal domain of Hfq[J]. Nucleic Acids Research, 2017, 45(12): 7299-7308. DOI:10.1093/nar/gkx431 |
| [21] |
Parekh VJ, Niccum BA, Shah R, Rivera MA, Novak MJ, Geinguenaud F, Wien F, Arluison V, Sinden RR. Role of Hfq in genome evolution: instability of G-Quadruplex sequences in E. coli[J]. Microorganisms, 2020, 8(1): 28. |
| [22] |
Chen JD, Gottesman S. Hfq links translation repression to stress-induced mutagenesis in E. coli[J]. Genes & Development, 2017, 31(13): 1382-1395. |
| [23] |
Link TM, Valentin-Hansen P, Brennan RG. Structure of Escherichia coli Hfq bound to polyriboadenylate RNA[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(46): 19292-19297. DOI:10.1073/pnas.0908744106 |
| [24] |
Orans J, Kovach AR, Hoff KE, Horstmann NM, Brennan RG. Crystal structure of an Escherichia coli Hfq core (residues 2–69)-DNA complex reveals multifunctional nucleic acid binding sites[J]. Nucleic Acids Research, 2020, 48(7): 3987-3997. DOI:10.1093/nar/gkaa149 |
| [25] |
Cech GM, Szalewska-Pałasz A, Kubiak K, Malabirade A, Grange W, Arluison V, Węgrzyn G. The Escherichia coli Hfq protein: an unattended DNA-transactions regulator[J]. Frontiers in Molecular Biosciences, 2016, 3: 36. |
| [26] |
Sharma IM, Korman A, Woodson SA. The Hfq chaperone helps the ribosome mature[J]. The EMBO Journal, 2018, 37(11): e99616. |
| [27] |
Andrade JM, Dos Santos RF, Chelysheva I, Ignatova Z, Arraiano CM. The RNA-binding protein Hfq is important for ribosome biogenesis and affects translation fidelity[J]. The EMBO Journal, 2018, 37(11): e97631. |
| [28] |
Lee T, Feig AL. The RNA binding protein Hfq interacts specifically with tRNAs[J]. RNA, 2008, 14(3): 514-523. DOI:10.1261/rna.531408 |
| [29] |
Lei SS. Investigation into B. melitensis hfq deletion mutant on virulence and and immune protective effect[D]. Chengdu: Master's Thesis of Sichuan Agricultural University, 2016 (in Chinese) 雷霜霜. 羊种布鲁氏菌hfq突变株毒力及其免疫保护作用研究[D]. 成都: 四川农业大学硕士学位论文, 2016 |
| [30] |
Tsui HCT, Leung HCE, Winkler ME. Characterization of broadly pleiotropic phenotypes caused by an hfq insertion mutation in Escherichia coli K-12[J]. Molecular Microbiology, 1994, 13(1): 35-49. DOI:10.1111/j.1365-2958.1994.tb00400.x |
| [31] |
Guisbert E, Rhodius VA, Ahuja N, Witkin E, Gross CA. Hfq modulates the σE-mediated envelope stress response and the σ32-mediated cytoplasmic stress response in Escherichia coli[J]. Journal of Bacteriology, 2007, 189(5): 1963-1973. DOI:10.1128/JB.01243-06 |
| [32] |
Wang MC, Chien HF, Tsai YL, Liu MC, Liaw SJ. The RNA chaperone Hfq is involved in stress tolerance and virulence in uropathogenic Proteus mirabilis[J]. PLoS One, 2014, 9(1): e85626. DOI:10.1371/journal.pone.0085626 |
| [33] |
Yao H, Kang MQ, Wang YT, Feng YW, Kong SW, Cai XX, Ling ZT, Chen SS, Jiao XA, Yin YL. An essential role for Hfq involved in biofilm formation and virulence in serotype 4b Listeria monocytogenes[J]. Microbiological Research, 2018, 215: 148-154. DOI:10.1016/j.micres.2018.07.001 |
| [34] |
Lu HT, Qiao J, Meng QL, Peng YL, Chen C, Liu TL, Cai XP, Chen CF. Effect of chaperone hfq gene deletion on Listeria monocytogenes virulence and biofilm formation[J]. Acta Agriculturae Boreali-Sinica, 2017, 32(1): 94-98. (in Chinese) 卢海亭, 乔军, 孟庆玲, 彭叶龙, 陈诚, 刘田莉, 才学鹏, 陈创夫. 分子伴侣hfq基因缺失对单核细胞增生李斯特菌毒力及生物被膜生成的影响[J]. 华北农学报, 2017, 32(1): 94-98. |
| [35] |
Jagtap CB, Kumar P, Rao KK. Bacillus subtilis Hfq: a role in chemotaxis and motility[J]. Journal of Biosciences, 2016, 41(3): 347-358. DOI:10.1007/s12038-016-9618-9 |
| [36] |
Rochat T, Delumeau O, Figueroa-Bossi N, Noirot P, Bossi L, Dervyn E, Bouloc P. Tracking the elusive function of Bacillus subtilis Hfq[J]. PLoS One, 2015, 10(4): e124977. |
| [37] |
Hämmerle H, Amman F, Večerek B, Stülke J, Hofacker I, Bläsi U. Impact of Hfq on the Bacillus subtilis transcriptome[J]. PLoS One, 2014, 9(6): e98661. DOI:10.1371/journal.pone.0098661 |
| [38] |
Kavita K, De Mets F, Gottesman S. New aspects of RNA-based regulation by Hfq and its partner sRNAs[J]. Current Opinion in Microbiology, 2018, 42: 53-61. DOI:10.1016/j.mib.2017.10.014 |
| [39] |
Janissen R, Arens MMA, Vtyurina NN, Rivai Z, Sunday ND, Eslami-Mossallam B, Gritsenko AA, Laan L, De Ridder D, Artsimovitch I, et al. Global DNA compaction in stationary-phase bacteria does not affect transcription[J]. Cell, 2018, 174(5): 1188-1199. DOI:10.1016/j.cell.2018.06.049 |
| [40] |
Joyeux M. Compaction of bacterial genomic DNA: clarifying the concepts[J]. Journal of Physics: Condensed Matter, 2015, 27(38): 383001. DOI:10.1088/0953-8984/27/38/383001 |
| [41] |
Zhang C, Guttula D, Liu F, Malar PP, Ng SY, Dai L, Doyle PS, Van Kan JA, Van Der Maarel JRC. Effect of H-NS on the elongation and compaction of single DNA molecules in a nanospace[J]. Soft Matter, 2013, 9(40): 9593-9601. DOI:10.1039/c3sm51214b |
| [42] |
Guttula D, Liu F, Van Kan JA, Arluison V, Van Der Maarel JRC. Effect of HU protein on the conformation and compaction of DNA in a nanochannel[J]. Soft Matter, 2018, 14(12): 2322-2328. DOI:10.1039/C7SM02118F |
| [43] |
Partouche D, Mathurin J, Malabirade A, Marco S, Sandt C, Arluison V, Deniset-Besseau A, Trépout S. Correlative infrared nanospectroscopy and transmission electron microscopy to investigate nanometric amyloid fibrils: prospects and challenges[J]. Journal of Microscopy, 2019, 274(1): 23-31. DOI:10.1111/jmi.12779 |
| [44] |
Zhou YZ, Blanco LP, Smith DR, Chapman MR. Bacterial amyloids[A]//Sigurdsson E, Calero M, Gasset M. Methods in Molecular Biology (Methods and Protocols)[M]. Switzerland AG: Humana Press, 2012, 849: 303-320
|
| [45] |
Zhou YZ, Smith D, Leong BJ, Brännström K, Almqvist F, Chapman MR. Promiscuous cross-seeding between bacterial amyloids promotes interspecies biofilms[J]. Journal of Biological Chemistry, 2012, 287(42): 35092-35103. DOI:10.1074/jbc.M112.383737 |
| [46] |
Fortas E, Piccirilli F, Malabirade A, Militello V, Trépout S, Marco S, Taghbalout A, Arluison V. New insight into the structure and function of Hfq C-terminus[J]. Bioscience Reports, 2015, 35(2): e00190. DOI:10.1042/BSR20140128 |
| [47] |
Fitzpatrick AWP, Park ST, Zewail AH. Exceptional rigidity and biomechanics of amyloid revealed by 4D electron microscopy[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(27): 10976-10981. DOI:10.1073/pnas.1309690110 |
| [48] |
Dorman CJ, Dorman MJ. DNA supercoiling is a fundamental regulatory principle in the control of bacterial gene expression[J]. Biophysical Reviews, 2016, 8(3): 209-220. DOI:10.1007/s12551-016-0205-y |
| [49] |
Sledjeski DD, Whitman C, Zhang AX. Hfq is necessary for regulation by the untranslated RNA DsrA[J]. Journal of Bacteriology, 2001, 183(6): 1997-2005. DOI:10.1128/JB.183.6.1997-2005.2001 |
| [50] |
Winardhi RS, Yan J, Kenney LJ. H-NS regulates gene expression and compacts the nucleoid: Insights from single-molecule experiments[J]. Biophysical Journal, 2015, 109(7): 1321-1329. DOI:10.1016/j.bpj.2015.08.016 |
| [51] |
Wang ZJ, Yuan ZH, Wen YM. Study on replication and regulation of ColE1 type plasmid DNA vector[J]. Progress in Biochemistry and Biophysics, 2003, 30(6): 863. (in Chinese) 王志军, 袁正宏, 闻玉梅. ColE1型质粒DNA载体的复制与调控研究[J]. 生物化学与生物物理进展, 2003, 30(6): 863. DOI:10.3321/j.issn:1000-3282.2003.06.031 |
| [52] |
Tsui HT, Feng G, Winkler ME. Negative regulation of mutS and mutH repair gene expression by the Hfq and rpoS global regulators of Escherichia coli K-12[J]. Journal of Bacteriology, 1997, 179(23): 7476-7487. DOI:10.1128/JB.179.23.7476-7487.1997 |
| [53] |
Dos Santos RF, Arraiano CM, Andrade JM. New molecular interactions broaden the functions of the RNA chaperone Hfq[J]. Current Genetics, 2019, 65(6): 1313-1319. DOI:10.1007/s00294-019-00990-y |
| [54] |
Zhou ZY, Zhou XX, Li J, Zhong ZJ, Li W, Liu XH, Liu FR, Su HY, Luo YJ, Gu WY, et al. Transcriptional regulation and adaptation to a high-fiber environment in Bacillus subtilis HH2 isolated from feces of the Giant Panda[J]. PLoS One, 2015, 10(2): e0116935. DOI:10.1371/journal.pone.0116935 |
| [55] |
Zhou ZY, Liu FR, Zhang XY, Zhou XX, Zhong ZJ, Su HY, Li J, Li ZH, Feng F, Lan JC, et al. Cellulose-dependent expression and antibacterial characteristics of surfactin from Bacillus subtilis HH2 isolated from the Giant Panda[J]. PLoS One, 2018, 13(1): e0191991. DOI:10.1371/journal.pone.0191991 |
| [56] |
Li J, Zhong ZJ, Su HY, Zhou ZY, Li W, Liu FR, Lan JC, Zhou XX, Zhang WP, Peng GN. Isolation, identification and partial biology research of Bacillus in Giant Panda intestines[J]. Microbiology China, 2016, 43(2): 351-359. (in Chinese) 李进, 钟志军, 苏怀益, 周紫峣, 李魏, 刘福芮, 兰景超, 周潇潇, 张文平, 彭广能. 大熊猫肠道芽孢杆菌的分离鉴定及部分生物学特性[J]. 微生物学通报, 2016, 43(2): 351-359. |
| [57] |
Duan YH, Ma YY, Zhao XD, Huang RL, Su RX, Qi W, He ZM. Real-time adsorption and action of expansin on cellulose[J]. Biotechnology for Biofuels, 2018, 11(1): 317. DOI:10.1186/s13068-018-1318-2 |
| [58] |
Zhang PQ, Ma YY, Cui M, Wang JY, Huang RL, Su RX, Qi W, He ZM, Thielemans W. Effect of sugars on the real-time adsorption of expansin on cellulose[J]. Biomacromolecules, 2020, 21(5): 1776-1784. DOI:10.1021/acs.biomac.9b01694 |
| [59] |
Wien F, Martinez D, Le Brun E, Jones NC, Hoffmann SV, Waeytens J, Berbon M, Habenstein B, Arluison V. The bacterial amyloid-like Hfq promotes in vitro DNA alignment[J]. Microorganisms, 2019, 7(12): 639. DOI:10.3390/microorganisms7120639 |
 2021, Vol. 48
2021, Vol. 48




