扩展功能
文章信息
- 邓祥宜, 李婵娟, 李璐, 李池茜, 张涵池, 李佳鑫
- DENG Xiangyi, LI Chanjuan, LI Lu, LI Chixi, ZHANG Hanchi, LI Jiaxin
- 小龙虾肠道细菌群落多样性及产蛋白酶细菌的筛选
- Diversity of bacterial community and screening of protease producing bacteria in red swamp crayfish (Procambarus clarkii) intestine
- 微生物学通报, 2021, 48(4): 1151-1159
- Microbiology China, 2021, 48(4): 1151-1159
- DOI: 10.13344/j.microbiol.china.200518
-
文章历史
- 收稿日期: 2020-05-27
- 接受日期: 2020-07-07
- 网络首发日期: 2020-10-14
近年来,我国小龙虾(学名:Procambarus clarkii,克氏原螯虾)养殖发展迅猛。2016年,我国已成为世界最大的小龙虾生产国,其中湖北省小龙虾产量占全国的一半。根据中国小龙虾产业发展报告(2018),2017年中国小龙虾全社会经济总产值约2 685亿元,比2016年增长83.15%,我国小龙虾呈现供销两旺的良好局面[1]。
小龙虾养殖规模持续上升,市售小龙虾主要来自人工饲养,野外捕捞量占比逐年下降。小龙虾养殖中,饲料的蛋白质含量在26%-40%,远高于20%,属于蛋白质饲料[2],其价格往往高于粗饲料、青饲料、能量饲料等。不同蛋白源饲料的养殖效果存在明显差异[3],饲料中蛋白质的利用率也有待提高,而养殖动物对饲料的消化利用往往与肠道菌群密切相关。
由于绝大部分(99%以上)微生物无法在人工条件下进行纯培养,因此传统分离培养法不能全面了解肠道微生物群落信息[4],而针对细菌群落宏基因组中16S rRNA基因序列分析的方法(免培养法)能更全面地了解细菌群落的组成,但16S rRNA基因克隆文库测序、变性梯度凝胶电泳(Denatured Gradient Gel Electrophoresis,DGGE)等常规技术只能检测少数优势种类,如李玉宏等研究工厂化养殖凡纳滨对虾肠道微生物群落组成,采用RPLF谱型只鉴定出13个类别,利用DGGE鉴定出21个类别[5]。近年来,高通量测序技术飞速发展,突破了这种局限,能获得更全面更可信的结果[6-7]。目前,通过高通量测序技术对肠道微生物群落开展研究的虾类主要有凡纳滨对虾(Litopenaeus vannamei)[8-11]、罗氏沼虾(Macrobrachium rosenbergii)[12]和小龙虾(P. clarkii)[13-15]等。对于小龙虾肠道菌群的研究,材料主要来自实验室养殖条件且多为哈尔滨市场购买的成虾;湖北养殖产量占全国的半壁江山,湖北小龙虾肠道微生物群落组成有待揭示。
本文以养殖塘的小龙虾为材料,利用高通量测序技术研究小龙虾肠道细菌群落组成,通过与相关研究比较,分析小龙虾肠道细菌群落的核心类群;同时从虾肠道内容物中筛选和鉴定产蛋白酶细菌,以期为开发小龙虾养殖微生态制剂或饲用蛋白酶制剂作铺垫。
1 材料与方法 1.1 样品鲜活小龙虾(20-30 g) 20多只,用地笼从本校小龙虾养殖实验塘捕获(用扶龙牌龙虾饲料投喂,养殖塘2个月内未使用任何微生物制剂或其他药物)。
1.2 主要试剂和仪器及培养基土壤DNA快速提取试剂盒,MP Bio medicals公司;TransStart FastPfu DNA聚合酶,北京全式金生物公司;DNA凝胶回收试剂盒,Axygen公司;MiSeq文库构建试剂盒,Illumina公司;革兰氏染色液试剂盒,海博生物技术有限公司。PCR仪,ABI公司;蓝色荧光定量系统,Promega公司;普通生物显微镜,Leica公司;Illumina MiSeq测序平台,Illumina公司。
含1%酪蛋白的1/5 LB固体培养基(g/L):酪蛋白10.00,胰蛋白胨2.00,酵母抽提物1.00,NaCl 2.00,琼脂粉15.00,pH 7.0。
含1%酪蛋白的SY固体培养基(g/L):酪蛋白10.00,可溶性淀粉2.00,酵母抽提物1.00,KH2PO4 0.20,MgSO4 0.05,琼脂粉15.00,pH 7.0。
1.3 方法 1.3.1 肠道内容物取样方法将捕获的小龙虾(12只)去附肢,75%酒精浸泡15 min,2.5% NaClO浸泡15-20 min,移入超净工作台,无菌水漂洗4次(每次5 min);无菌操作下解剖小龙虾,取出整个肠道,无菌生理盐水中漂洗3次,用镊子将肠道内容物挤出;将所有肠道内容物混合后转移到灭菌的5 mL离心管中,在-80 ℃冰箱冻存,用于后续细菌群落多样性(高通量测序)的研究。
1.3.2 微生物基因组DNA的提取及高通量测序用土壤DNA快速提取试剂盒提取总DNA,经电泳检测后(加干冰)送往上海美吉生物医药科技有限公司,进行细菌16S rRNA基因V3-V4区的PCR扩增,引物为338F (5′-ACTCCTACGGGAG GCAGCAG-3′)和806R (5′-GGACTACHVGGGTW TCTAAT-3′)。PCR反应体系(20 μL):5×Fast Pfu Buffer 4 μL,dNTPs (2.5 mmol/L) 2 μL,引物338F (5 μmol/L) 0.8 μL,引物806R (5 μmol/L) 0.8 μL,Fast Pfu Polymerase (2.5 U/μL) 0.4 μL,模板DNA 10 ng,超纯水补足20 μL。PCR反应条件:95 ℃ 3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,27个循环;72 ℃ 10 min;10 ℃保存。PCR产物经电泳、回收、定量后进行Illumina MiSeq测序[16]。
1.3.3 高通量测序结果分析MiSeq高通量测序结果借助美吉生物云平台进行分析,主要分析流程为:将测序原始序列拼接和质控后进行操作分类单元(Operational Taxonomic Units,OTU)聚类分析,OTU分析使用USEARCH平台(V7.0,http://drive5.com/uparse/),相似性在97%以上的序列聚为一类;选择OTU代表序列比对Silva数据库(Release132,http://www.arb-silva.de)进行物种分类学分析;在OTU水平进行α多样性分析;基于分类学信息,在不同分类学水平上进行群落结构统计分析;使用Excel软件作图。
1.3.4 虾肠道产蛋白酶微生物的筛选按1.3.1中方法取虾肠道内容物(10只)并混合,用无菌生理盐水分散均匀并进行梯度稀释,取10-4、10-5、10-6、10-7、10-8涂布在含1%酪蛋白的1/5 LB平板和SY平板,每个梯度5皿,其中3皿有氧培养(菌株编号中用“+”表示)、2皿厌氧培养(燃烛法,菌株编号中用“-”表示),培养温度30 ℃。另做空白平行2皿(生理盐水代替菌悬液),验证无菌操作。
从涂布的平板挑选单菌落进行划线分离,30 ℃培养;将划线纯化后菌株接种于酪蛋白平板(1/5 LB+1%酪蛋白),30 ℃培养2 d,观察酪蛋白平板上的水解圈,选取水解圈较大的菌株进行鉴定。
1.3.5 产蛋白酶菌株的鉴定用革兰氏染色试剂盒对菌株进行染色观察,同时提取细菌总DNA用于菌株的分子生物学鉴定[17]。以P1 (5′-GAGAGTTTGATCCTGGCTCAG-3′)和P2 (5′-GCCCCCGTCAATTCCTTTGAG-3′)为引物PCR扩增16S rRNA基因,片段长度约900 bp。反应体系(50 μL):10× Fast Pfu Buffer 5.0 μL,dNTPs (10 mmol/L) 1.0 μL,引物P1 (5 μmol/L) 1.0 μL,引物P2 (5 μmol/L) 1.0 μL,Fast Pfu Polymerase (2.5 U/μL) 1.0 μL,模板DNA 1.0 μL,补超纯水至50 μL。PCR反应条件:94 ℃ 4 min;94 ℃ 30 s,52 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 7 min;4 ℃ 5 min。PCR产物经电泳检测后送往昆泰锐(武汉)生物技术有限责任公司测序。利用NCBI数据库中的BLASTn比对16S rRNA基因序列数据库,得到相似程度最高的序列,用MEGA 7.0软件构建系统发育树[18]。
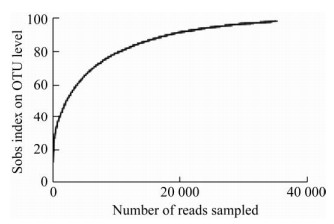
2 结果与分析 2.1 样本序列统计及α多样性分析用高通量测序技术分析小龙虾肠道细菌群落多样性,经质控、拼接,获得36 185条有效序列(细菌16S rRNA基因V3-V4区),长度最长434 bp、最短283 bp,平均长度427.9 bp。细菌群落α多样性指数中,Coverage指数为0.999 7,同时稀释曲线上升逐渐趋于平缓(图 1),说明测序结果覆盖度高,能较好地反映小龙虾肠道样品中的细菌群落组成。
|
| 图 1 样品稀释曲线 Figure 1 Rarefaction curves of samples |
|
|
在97%的相似水平上对小龙虾肠道细菌16S rRNA基因V3-V4区测序结果进行OTU聚类分析,共获得98个OTU。对OTU进行分类学分析,并在不同分类学水平上进行了统计。由于肠道细菌的很多种类亲缘关系较近,而本次Illumina MiSeq测序只覆盖了16S rRNA基因V3-V4区(序列平均长度427.9 bp),包含信息有限,所以很多OTU不能准确鉴定到种,只能鉴定到属水平。因此,在门(图 2A)、科(图 2B)、属(图 2C) 3个分类学水平上对小龙虾肠道细菌群落组成进行分析。
|
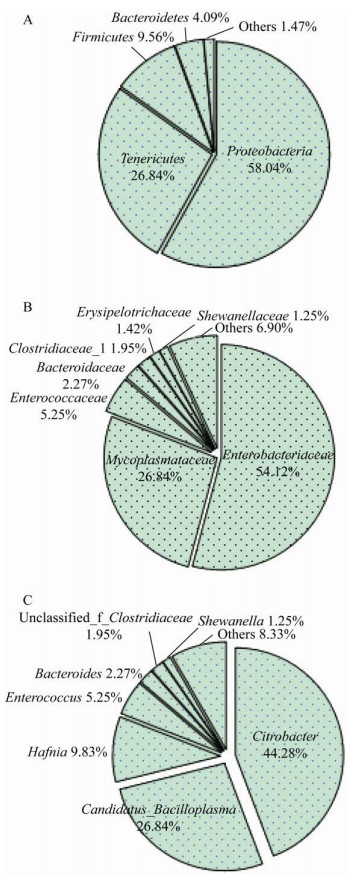
| 图 2 小龙虾肠道的微生物区系 Figure 2 The intestinal microbiota of Procambarus clarkii 注:A:细菌门水平相对丰度;B:细菌科水平相对丰度;C:细菌属水平相对丰度 Note: A: Relative abundance of bacteria on phylum level; B: Relative abundance of bacteria on family level; C: Relative abundance of bacteria on genus level |
|
|
小龙虾肠道细菌分布在变形菌门(Proteobacteria)、软壁菌门(Tenericutes)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)等9个门,包括57个科和83个属。其中,变形菌门占58.04%,主要包括肠杆菌科(Enterobacteriaceae,54.12%)的柠檬酸杆菌属(Citrobacter,44.28%)、哈夫尼菌属(Hafnia,9.83%),以及希瓦氏菌科(Shewanellaceae,1.25%)的希瓦氏菌属(Shewanella,1.25%);软壁菌门占26.84%,主要包括支原体科(Mycoplasmataceae,26.84%)的Candidatus_Bacilloplasma暂定属(26.84%);厚壁菌门占9.56%,主要包括肠球菌科(Enterococcaceae,5.25%)的肠球菌属(Enterococcus,5.25%)、梭菌科未分类属(Unclassified_f_Clostridiaceae,1.95%)和丹毒丝菌科(Erysipelotrichaceae,1.42%);拟杆菌门占4.09%,主要包括拟杆菌科(Bacteroidaceae,2.27%)的拟杆菌属(Bacteroides,2.27%) (图 2)。
按相对丰度 > 1%的细菌属定义为优势属,小龙虾肠道细菌优势属主要包括柠檬酸杆菌属(Citrobacter,44.28%)、支原体科Candidatus_ Bacilloplasma暂定属(26.84%)、哈夫尼菌属(Hafnia,9.83%)、肠球菌属(Enterococcus,5.25%)、拟杆菌属(Bacteroides,2.27%)、梭菌科未分类属(Unclassified_ f_Clostridiaceae,1.95%)、希瓦氏菌属(Shewanella,1.25%)这7个属,累计占比达91.67% (图 2C)。
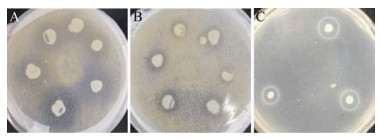
2.3 小龙虾肠道产蛋白酶微生物筛选及鉴定从2种培养基上共计挑取128株菌进行划线纯化,将划线纯化后的菌株点在酪蛋白平板上,一些菌株产生了较明显的水解圈(图 3),表现出较强的蛋白酶活性,挑选20株产酶活性较高的菌株进行鉴定。
|
| 图 3 部分菌株在酪蛋白平板上形成的水解圈 Figure 3 Hydrolysis circles formed by some strains on the casein plate |
|
|
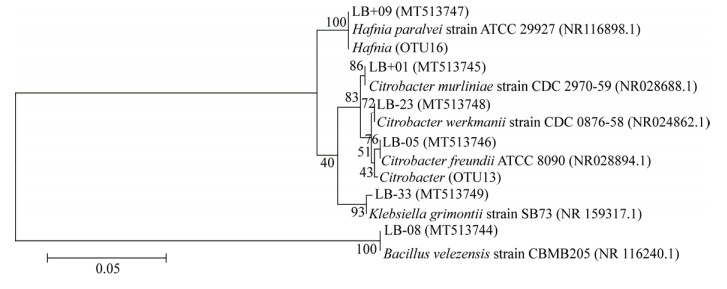
根据16S rRNA基因序列比对和构建系统发育树对菌株进行鉴定,结果见表 1和图 4。LB+09等10株菌初步鉴定为副蜂房哈夫尼菌(Hafnia paralvei,序列相似度100%);LB+01等4株菌初步鉴定为莫林柠檬酸杆菌(Citrobacter murliniae,序列相似度100%);LB-23初步鉴定为乌克曼柠檬酸杆菌(Citrobacter werkmanii,序列相似度99.35%);LB-05等3株菌初步鉴定为弗劳地氏柠檬酸杆菌(Citrobacter freundii,序列相似度100%);LB-33初步鉴定为格里蒙特克雷伯氏菌(Klebsiella grimontii,序列相似度99.48%);LB-08初步鉴定为贝莱斯芽孢杆菌(Bacillus velezensis,序列相似度99.86%)。将高通量测序中相关属的代表序列纳入系统发育树中,可以发现LB+09与Hafnia属代表序列(OTU16)聚在一起,LB+01、LB-23和LB-05与Citrobacter属代表序列(OTU13)聚在一起(图 4),说明分离菌株测序结果与高通量测序结果高度吻合。高通量测序中,没有检出Klebsiella和Bacillus,说明高通量测序虽然灵敏度高于其他研究微生物多样性的方法,但也不能保证检出所有菌株,这一方面受测序反应量的影响,也和样本DNA的提取效率相关。在科水平上,LB+09等19株菌来自肠杆菌科,LB-08来自芽孢杆菌科(表 1)。经染色观察,LB+09等19株菌均为革兰氏阴性杆菌(图 5A),单菌落均呈规则圆形、表面光滑、略微凸起、白色不透明;副蜂房哈夫尼菌菌落表面湿润、易挑起,柠檬酸杆菌属菌落稍湿润、易挑起,格里蒙特克雷伯氏菌菌落表面湿润、较粘稠;LB-08菌株为革兰氏阳性菌(图 5B),而且在培养24 h时开始产生芽孢(图 5C,芽孢呈绿色),菌落呈规则圆形、中心凸起、白色不透明、易挑起,表面光滑湿润但培养48 h以上菌落表面变得干燥皱缩(形成芽孢所致)。
| 菌株 Strains |
最相似菌株 The closest relatives |
GenBank Accession No. | 相似性 Identity (%) |
科 Family |
| LB+09, LB+21, LB-11, LB-12, SY+22, SY-06, SY-07, SY-08, SY-10, SY-19 | 副蜂房哈夫尼菌Hafnia paralvei | NR116898.1 | 100 | Enterobacteriaceae |
| LB+01, LB+11, LB-01, LB-04 | 莫林柠檬酸杆菌Citrobacter murliniae | NR028688.1 | 100 | Enterobacteriaceae |
| LB-23 | 乌克曼柠檬酸杆菌Citrobacter werkmanii | NR024862.1 | 99.35 | Enterobacteriaceae |
| LB-05, LB-06, LB+14 | 弗劳地氏柠檬酸杆菌Citrobacter freundii | NR028894.1 | 100 | Enterobacteriaceae |
| LB-33 | 格里蒙特克雷伯氏菌Klebsiella grimontii | NR159317.1 | 99.48 | Enterobacteriaceae |
| LB-08 | 贝莱斯芽孢杆菌Bacillus velezensis | NR116240.1 | 99.86 | Bacillaceae |
|
| 图 4 基于16S rRNA基因的菌株系统发育树 Figure 4 Phylogenetic tree of strains based on 16S rRNA gene 用最大似然法建树,分支节点处显示1 000次重复检验的置信值;Hafnia (OTU16)、Citrobacter (OTU13)为高通量测序中的代表序列;其他序列在NCBI的登录号,显示在括号中;标尺0.05表示100个核苷酸中有5个被替换 Note: The phylogenetic tree was constructed using the Maximum likelyhood method. Bootstrap values for 1 000 replicates were given in the branch nodes. Hafnia (OTU16) and Citrobacter (OTU13) were representative sequences in high throughput sequencing; The GenBank accession numbers for other sequences were present in the parentheses. The scale bar, 0.05, represented five out of 100 nucleotides were replaced |
|
|
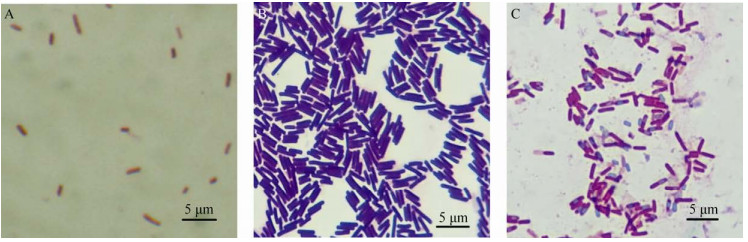
|
| 图 5 革兰氏染色及芽孢染色结果 Figure 5 Results of Gram staining and spore staining 注:A:菌株LB+09,革兰氏阴性;B:菌株LB-08,革兰氏阳性;C:菌株LB-08,芽孢为绿色,菌体为红色 Note: A: Strain LB+09, G-; B: Strain LB-08, G+; C: Spore staining of strain LB-08 showing bacteria (red) and spores (green) |
|
|
本文通过Illumina MiSeq高通量测序对我校
养殖塘中小龙虾肠道细菌群落组成进行了研究,养殖塘2个月以上未使用任何微生物制剂或其他药物,能较好地代表正常生境(室外养殖塘)下的小龙虾肠道菌群组成。16S rRNA基因是微生物生态学研究中广泛使用的分子标记,其有9个高度变异区(V1-V9)。据报道,原核生物单个基因组中平均有3.61个16S rRNA基因的拷贝,这些基因常具有基因组内多态性,在一定程度上会造成物种丰富度的高估,在OTU水平(97%)偏高,达3%-12%,在属水平(95%)及以上影响相对较小[19];V3区和V4-V5区是基因组内多态性较低的区域,选择该区域有利于降低这种偏差[19]。本文选择V3-V4区进行研究,细菌群落的物种丰富度可能也存在一定程度的高估。
一些较新的研究揭示了实验室条件下的小龙虾肠道菌群组成。Guo等从湖北荆州一个养殖场采购虾苗(4.5±0.2 g),在实验室水箱中开展养殖试验,发现变形菌门、软壁菌门、拟杆菌门、厚壁菌门是小龙虾肠道细菌优势门,严重的氮或硫胁迫会导致变形菌门的柠檬酸杆菌属显著下降[13];Zhang等从哈尔滨市场购买成年雄性小龙虾(20.28±2.98 g),养殖于实验室玻璃水箱中,在研究微囊藻毒素(Microcystin-LR)对小龙虾肠道微生物组成时发现,肠道细菌优势门包括变形菌门(70.70%-71.59%)、拟杆菌门(18.21%-20.26%)和厚壁菌门(3.01%-6.21%),而且厚壁菌门在微囊藻毒素处理后显著增加[15];另一研究同样在实验室玻璃水箱中进行,在研究Cd2+对小龙虾(哈尔滨市场购买的雄性成虾,20.96±3.05g)肠道细菌群落影响时发现,肠道细菌优势门包括厚壁菌门(29.04%-58.45%)、变形菌门(18.35%-54.56%)和拟杆菌门(4.30%-47.81%),优势属为拟杆菌属、梭菌ⅩlVb属、哈夫尼菌属、布特菌属(Buttiauxella)、希瓦氏菌属等[14],上述研究与本实验测定的主要优势门和优势属一致。冯光志等也发现,市售小龙虾(成虾,经实验室暂养)肠道细菌群落包括支原体科Candidatus_Bacilloplasma暂定属、拟杆菌属、希瓦氏菌属等本实验检测出的优势属,还包括弧菌属、不动杆菌属、气单胞菌属以及大量未知属细菌(30.48%)[20]。弧菌属很多种类是水产养殖中的条件致病菌,弧菌丰度增加易引起细菌性传染疾病暴发,导致大量虾死亡,降低了养殖效益[11, 21],但也有研究表明弧菌属与凡纳滨对虾的健康状况没有直接关系,应该更关注整个肠道微生物群落状况,而不是只以弧菌为指标[22]。
结合上述前人研究可以发现:变形菌门、拟杆菌门、厚壁菌门是小龙虾肠道的核心菌群,在正常生境条件下,变形菌门往往占据绝对优势,其中以柠檬酸杆菌属、哈夫尼菌属为代表;软壁菌门不是固有核心类群,其受环境影响较大,或占比较高或未检出。这与董学兴等的研究结论相似,他们认为变形菌门、厚壁菌门是罗氏沼虾肠道的核心菌群,软壁菌门受环境影响大[12]。本实验没有从小龙虾肠道内容物中检出弧菌属,说明变形菌门中的弧菌属不是小龙虾肠道菌群的固有类群,其在小龙虾肠道中的丰度受环境影响也较大。
本研究从正常小龙虾肠道分离和鉴定了产蛋白酶细菌,主要来自哈夫尼菌属、柠檬酸杆菌属等虾肠道细菌核心类群,可见小龙虾肠道核心菌群对蛋白质消化有重要作用。有研究从患病小龙虾肝胰腺分离出弗劳地氏柠檬酸杆菌(Citrobacter freundii),认为其具有较强致病性[23],但其在病虾中被检出不代表就是病因,致病原因可能是病毒等其他因素,比如小龙虾感染对虾白斑综合征病毒(White Spot Syndrome Virus,WSSV)后,血淋巴中嗜水气单胞菌(Aeromonas hydrophila)、弗劳地氏柠檬酸杆菌(C. freundii)等条件致病菌明显增加,但致病根源却是对虾白斑综合征病毒的感染[24]。
实验中还发现副蜂房哈夫尼菌等菌株在刚分离鉴定时产蛋白酶活力较高,但经过甘油管-80 ℃冻藏再复苏培养时蛋白酶活力明显下降,经酪蛋白培养基传代几次后活性有所恢复,其蛋白酶表达调控机制值得进一步研究。
肠道微生物对宿主的营养和健康非常重要,参与了宿主很多重要生理功能的调节[25]。相比小龙虾肠道微生物的研究,人们对同属于节肢动物门昆虫的研究更为深入。肠道微生物不仅为昆虫的生长发育提供营养,还合成多种活性物质,调节宿主免疫,抵御病原微生物的感染[26]。昆虫肠道菌群组成具有物种特异性,同时与昆虫性别、发育阶段、摄食以及环境因子(温度、CO2等)密切相关[27]。因此,后续有必要参考昆虫肠道微生物研究方法,对小龙虾肠道微生物进行更深入的研究,以揭示小龙虾在不同发育阶段、环境条件和饲喂方式下的肠道菌群组成,进一步探讨肠道菌群对小龙虾生长及免疫的影响,为小龙虾产业的健康发展提供理论支撑。
| [1] |
Fisheries and Fisheries Administration of Ministry of Agriculture and Rural Affairs of the People's Republic of China, National Fisheries Technology Extension Center, China Society of Fisheries. Report on the development of crayfish industry in China (2018)[J]. China Fisheries, 2018(7): 20-27. (in Chinese) 农业农村部渔业渔政管理局, 全国水产技术推广总站, 中国水产学会. 中国小龙虾产业发展报告(2018)[J]. 中国水产, 2018(7): 20-27. |
| [2] |
Zhao L. The study on China's feed industry[D]. Wuhan: Doctoral Dissertation of Huazhong Agricultural University, 2006 (in Chinese) 赵亮. 我国饲料产业研究[D]. 武汉: 华中农业大学博士学位论文, 2006 |
| [3] |
Yang CG, Su FQ, Wen H, Liu W, Wei KJ. Effects of different protein sources feed on growth and IGF1R mRNA expression of Procambarus clarkia[J]. Freshwater Fisheries, 2019, 49(5): 86-92. (in Chinese) 杨长庚, 苏富强, 文华, 刘伟, 魏开金. 不同蛋白源饲料对克氏原螯虾生长及胰岛素生长因子受体基因表达的影响[J]. 淡水渔业, 2019, 49(5): 86-92. DOI:10.3969/j.issn.1000-6907.2019.05.013 |
| [4] |
Wang HQ, Qu YM, Liu B. Molecular analysis of gut bacterial communities of Oratosquilla oratoria[J]. Chinese Journal of Microecology, 2010, 22(4): 317-320, 324. (in Chinese) 王海青, 曲艳梅, 刘斌. 口虾蛄肠道细菌种群多样性的分子分析[J]. 中国微生态学杂志, 2010, 22(4): 317-320, 324. |
| [5] |
Li YH, Chai PC, Hu XG, Sun Y, Huang J, Song XL. Analysis of intestinal microecology of Litopenaeus vannamei in industrial aquaculture by RFLP and DGGE techniques[J]. Progress in Fishery Sciences, 2014, 35(2): 83-89. (in Chinese) 李玉宏, 柴鹏程, 胡修贵, 孙艳, 黄倢, 宋晓玲. 应用RFLP和DGGE技术分析工厂化养殖凡纳滨对虾肠道微生物群落特征[J]. 渔业科学进展, 2014, 35(2): 83-89. DOI:10.3969/j.issn.1000-7075.2014.02.012 |
| [6] |
Xu G, Wang MJ, Deng BW, Guo MM. Bacterial diversity and community structure of rhizosphere soil of tea plants in different years of planting[J]. Biotechnology Bulletin, 2020, 36(3): 124-132. (in Chinese) 许广, 王梦姣, 邓百万, 郭苗苗. 不同植茶年限茶树根际土壤细菌多样性及群落结构研究[J]. 生物技术通报, 2020, 36(3): 124-132. |
| [7] |
Li DP, Guo MZ, Xu WT. Advances and applications on methodology of 16S rRNA sequencing in gut microbiota analysis[J]. Biotechnology Bulletin, 2015, 31(2): 71-77. (in Chinese) 李东萍, 郭明璋, 许文涛. 16S rRNA测序技术在肠道微生物中的应用研究进展[J]. 生物技术通报, 2015, 31(2): 71-77. |
| [8] |
Hong B, Niu B, Chen P, Li W, Liu HQ, Pan YJ, Zhao Y. Diversity of gut microbiota and antibiotic resistance genes in Litopenaeus vannamei and Macrobrachium rosenbergii[J]. Journal of Fisheries of China, 2019, 43(5): 1347-1358. (in Chinese) 洪斌, 牛犇, 陈萍, 李薇, 刘海泉, 潘迎捷, 赵勇. 凡纳滨对虾和罗氏沼虾肠道微生物及抗生素抗性基因多样性分析[J]. 水产学报, 2019, 43(5): 1347-1358. |
| [9] |
Pei PB, Wu JQ, Liang HH, Du H. Effects of biological water purification grid on intestinal flora composition of pacific white leg shrimp Litopenaeus vannamei[J]. Fisheries Science, 2018, 37(3): 301-308. (in Chinese) 裴鹏兵, 吴洁琼, 梁宏豪, 杜虹. 生物净水栅对凡纳滨对虾肠道菌群组成的影响[J]. 水产科学, 2018, 37(3): 301-308. |
| [10] |
Yu WN, Dai WF, Tao Z, Xiong JB. Characterizing the compositional and functional structures of intestinal microflora between healthy and diseased Litopenaeus vannamei[J]. Journal of Fisheries of China, 2018, 42(3): 399-409. (in Chinese) 郁维娜, 戴文芳, 陶震, 熊金波. 健康与患病凡纳滨对虾肠道菌群结构及功能差异研究[J]. 水产学报, 2018, 42(3): 399-409. |
| [11] |
Ye HB, Fan Y, Wang XL, Li L, Gai CL, Liu HJ, Li TB. Effects of three biological agents on intestinal microbiota of white shrimp (Litopenaeus vannamei) in a water-recirculating cultivation system[J]. Acta Microbiologica Sinica, 2018, 58(9): 1647-1657. (in Chinese) 叶海斌, 樊英, 王晓璐, 李乐, 盖春蕾, 刘洪军, 李天保. 循环水养殖系统中凡纳滨对虾肠道微生物对三种复合益生菌制剂的响应[J]. 微生物学报, 2018, 58(9): 1647-1657. |
| [12] |
Dong XX, Lv LL, Zhao WH, Yu YB, Liu QG. Effects of different cultural patterns on microbial communities in the intestine of Macrobrachium rosenbergii and interactions with environment factors[J]. Journal of Shanghai Ocean University, 2019, 28(4): 501-510. (in Chinese) 董学兴, 吕林兰, 赵卫红, 於叶兵, 刘其根. 不同养殖模式下罗氏沼虾肠道菌群结构特征及其与环境因子的关系[J]. 上海海洋大学学报, 2019, 28(4): 501-510. |
| [13] |
Guo K, Ruan GL, Fan WH, Fang L, Wang Q, Luo MZ, Yi TL. The effect of nitrite and sulfide on the antioxidant capacity and microbial composition of the intestines of red swamp crayfish, Procambarus clarkii[J]. Fish & Shellfish Immunology, 2020, 96: 290-296. |
| [14] |
Zhang Y, Li ZY, Kholodkevich S, Sharov A, Chen C, Feng YJ, Ren NQ, Sun K. Effects of cadmium on intestinal histology and microbiota in freshwater crayfish (Procambarus clarkii)[J]. Chemosphere, 2020, 242: 125105. DOI:10.1016/j.chemosphere.2019.125105 |
| [15] |
Zhang Y, Li ZY, Kholodkevich S, Sharov A, Feng YJ, Ren NQ, Sun K. Microcystin-LR-induced changes of hepatopancreatic transcriptome, intestinal microbiota, and histopathology of freshwater crayfish (Procambarus clarkii)[J]. Science of the Total Environment, 2020, 711: 134549. DOI:10.1016/j.scitotenv.2019.134549 |
| [16] |
Lin GR, Wang CZ, Sun FL, Zheng ZP. Microbial community diversity in seawater polyculture pond of shrimp and clam[J]. Microbiology China, 2018, 45(9): 1989-1999. (in Chinese) 林国荣, 王春忠, 孙富林, 郑志鹏. 海水混养池塘虾蛤肠道与养殖环境的微生物多样性[J]. 微生物学通报, 2018, 45(9): 1989-1999. |
| [17] |
Weisburg WG, Barns SM, Pelletier DA, Lane DJ. 16S ribosomal DNA amplification for phylogenetic study[J]. Journal of Bacteriology, 1991, 173(2): 697-703. DOI:10.1128/JB.173.2.697-703.1991 |
| [18] |
Chen BS, Teh BS, Sun C, Hu SR, Lu XM, Boland W, Shao YQ. Biodiversity and activity of the gut microbiota across the life history of the insect herbivore Spodoptera littoralis[J]. Scientific Reports, 2016, 6: 29505. DOI:10.1038/srep29505 |
| [19] |
Sun DL, Jiang X, Wu QL, Zhou NY. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19): 5962-5969. DOI:10.1128/AEM.01282-13 |
| [20] |
Feng GZ, Zou YN, Wang YL, Wu YT, Shi Y. Screening and identification of xylanase-producing strains isolated from crayfish intestine[J]. Microbiology China, 2019, 46(6): 1364-1370. (in Chinese) 冯光志, 邹颜霓, 王月琳, 吴玉婷, 石玉. 小龙虾肠道产木聚糖酶细菌的分离与鉴定[J]. 微生物学通报, 2019, 46(6): 1364-1370. |
| [21] |
Huang XL, Du SC, Zhang HJ, Chen C, Xiao GQ, Huang L, Zhang DM. Temporal patterns of bacterial communities in shrimp (Litopenaeus vannamei) culture systems loaded with oyster shells[J]. Aquaculture, 2020, 526: 735424. DOI:10.1016/j.aquaculture.2020.735424 |
| [22] |
Zhang DM, Wang X, Xiong JB, Zhu JL, Wang YN, Zhao QF, Chen HP, Guo AN, Wu JF, Dai HP. Bacterioplankton assemblages as biological indicators of shrimp health status[J]. Ecological Indicators, 2014, 38: 218-224. DOI:10.1016/j.ecolind.2013.11.002 |
| [23] |
Chen HL, Song GT, He JX, Hou GJ, Wang YJ. Isolation, identification and antibiotics susceptibility test of Citrobacter freundii from Procambarus clarkii[J]. Freshwater Fisheries, 2014, 44(1): 73-77. (in Chinese) 陈红莲, 宋光同, 何吉祥, 侯冠军, 王永杰. 克氏原螯虾弗氏柠檬酸杆菌的分离鉴定与药敏试验[J]. 淡水渔业, 2014, 44(1): 73-77. |
| [24] |
Qin CT, Yang F, Pan DR. Study on the role of microorganism in the death of crayfish infected with WSSV[J]. Chinese Agricultural Science Bulletin, 2011, 27(32): 72-77. (in Chinese) 秦崇涛, 杨丰, 潘大仁. 微生物在感染WSSV的克氏原鳌虾死亡过程中的作用研究[J]. 中国农学通报, 2011, 27(32): 72-77. |
| [25] |
Tran NT, Zhang J, Xiong F, Wang GT, Li WX, Wu SG. Altered gut microbiota associated with intestinal disease in grass carp (Ctenopharyngodon idellus)[J]. World Journal of Microbiology and Biotechnology, 2018, 34(6): 71. DOI:10.1007/s11274-018-2447-2 |
| [26] |
Wei G, Bai L, Qu S, Wang SB. Insect microbiome and their potential application in the insect pest and vector-borne disease control[J]. Acta Microbiologica Sinica, 2018, 58(6): 1090-1102. (in Chinese) 魏舸, 白亮, 曲爽, 王四宝. 昆虫共生微生物在病虫害和疾病控制上的应用前景[J]. 微生物学报, 2018, 58(6): 1090-1102. |
| [27] |
Wei XY, Guo CL, Chu D. Research progress on the factors influencing microbiota diversity in insect[J]. Journal of Biosafety, 2019, 28(3): 170-176. (in Chinese) 魏晓莹, 郭晨亮, 褚栋. 昆虫体内微生物多样性的影响因素研究进展[J]. 生物安全学报, 2019, 28(3): 170-176. |
 2021, Vol. 48
2021, Vol. 48




