扩展功能
文章信息
- 夏阳, 武斌, 张毅, 李吉达
- XIA Yang, WU Bin, ZHANG Yi, LI Jida
- 核定位信号及其在病毒感染机制中的研究进展
- Research progress of nuclear localization signal in virus infection mechanism
- 微生物学通报, 2021, 48(3): 896-905
- Microbiology China, 2021, 48(3): 896-905
- DOI: 10.13344/j.microbiol.china.200411
-
文章历史
- 收稿日期: 2020-04-24
- 接受日期: 2020-05-30
- 网络首发日期: 2020-07-30
2. 石家庄四药有限公司 河北 石家庄 050000
2. Shijiazhuang No. 4 Pharmaceutical Company Limited, Shijiazhuang, Hebei 050000, China
真核细胞核膜将基因组DNA与细胞中其他成分分隔,核膜分布着核孔复合体(Nuclear Pore Complex,NPC),控制物质进出[1-3]。大部分核酸以及蛋白质通过核孔蛋白(Nucleoporin,NUP)进出细胞核,其中小分子物质可通过核孔复合体以被动扩散的方式进入核内,而大分子物质则需与核质转运受体结合后以主动运输的方式通过核孔复合体方可入核[4]。已知核孔复合体由30多种NUP组成,其中1/3的NUP中存在苯丙氨酸-甘氨酸重复序列(Phenylalanine-Glycine,F-G)结构域,F-G重复序列能够增强NUP的稳定性,并与核质转运受体结合来完成相关蛋白入核[5]。大分子物质的入核转运过程能否顺利是建立在货物蛋白能够与核质转运受体结合的基础之上,而决定货物蛋白能否与核质转运受体结合的关键在于货物蛋白上是否存在一段氨基酸序列,称之为核定位信号(Nuclear Localization Signal,NLS);NLS是一段存在于许多蛋白质上的短肽序列,可与核质转运受体特异性结合来介导货物蛋白以主动运输的方式通过NPC进入细胞核,从而使得货物蛋白能够在细胞核内完成复制[6]。受限于自身基因组编码能力,病毒并不能编码自身所有蛋白,需借助宿主细胞遗传信息编码系统完成。绝大多数DNA病毒自身蛋白的合成需要进入细胞核内完成,虽然大部分RNA病毒能在细胞质中完成自身蛋白的复制过程,但其部分重要蛋白的复制仍需要进入细胞核才能进行。大量研究证实需要进入细胞核内完成复制的蛋白常常为该病毒的关键蛋白,这些蛋白质在影响病毒的毒力、复制能力以及致病力方面往往发挥关键作用[7-10]。基于以上理论,对于病毒蛋白入核机制的研究将有助于病毒感染机制的进一步阐明,也将为抗病毒药物新作用靶点的发现提供思路。
1 NLS及其分类目前已发现的NLS可分为以下几类:(1) 单分型NLS为富含赖氨酸和精氨酸的碱性氨基酸序列,由4-8个氨基酸残基组成,其一致序列为K-K/R-X-K/R,其中X为任意氨基酸;典型的单分型NLS代表为猴空泡病毒40 (Simian Vacuolating Virus 40,SV40) T抗原NLS,其序列为PKKKRKV,该序列于20世纪80年代在一株猴源病毒中发现,也是最早发现的核定位信号肽[11]。(2) 双分型NLS一致序列为(K/R)(K/R)X10-12(K/R)3/5,其中X表示任意氨基酸残基,(K/R)代表赖氨酸或精氨酸残基[11]。经典的双分型NLS为非洲爪蟾核质蛋白NLS,其序列为KRPAATKKAGQAKKKK[5]。不论是单分型NLS还是双分型NLS,统称为经典NLS,这两类NLS均为富含赖氨酸与精氨酸的碱性氨基酸序列,区别在于双分型NLS精氨酸或者赖氨酸分列于两端。(3) 脯氨酸-酪氨酸核定位信号(Proline-Tyrosine Nuclear Localization Signal,PY-NLS)这类NLS的C端富含脯氨酸-酪氨酸序列,一致序列为R/K/H-X(2–5)-P-Y,其中X是任意氨基酸残基;PY-NLS又可分为2种:一类因4个连续的主要疏水残基位于PY残基N-末端而称之为疏水PY-NLS,如异质核核糖核蛋白(Heterogeneous Nuclear Ribonucleoproteins,hnRNP) D、hnRNP F、mRNA核输出因子Tip Associating Protein (TAP)、RNA结合蛋白Human Antigen R (HuR)和hnRNP A1即M9NLS;而另一类则NLS中心区域几乎没有疏水残基,但却在碱性残基中富集,称之为基本PY-NLS,如hnRNP M和多聚谷氨酰胺结合蛋白-1 (Polyglutamine Tract Binding Protein-1,PQBP-1)的NLS[12]。PY-NLS的典型代表为hnRNP A1的M9NLS,其序列为FGYNNQSSNFGPMKGG NFGGRSSGPY[13]。PY-NLS有3个结构独立的表位,可直接与核质转运受体Importin β2结合:1) N端疏水基/碱基序,是R-X的精氨酸表位;2) 2-5P-Y基序表位;3) 脯氨酸-酪氨酸(P-Y)或同源P-Φ (Φ为疏水残基)基序表位[13]。(4) 富含赖氨酸残基的异亮氨酸-赖氨酸核定位信号(Isoleucine- Lysine Nuclear Localization Signal,IK-NLS),其一致序列为K-V/I-X-K-X1-2-K/H/R,其中X为任意氨基酸残基,存在于细胞周期调节因子、核转录因子以及核糖体蛋白之中,IK-NLS的典型代表为转录因子Phosphate Signal Transduction 4 (Pho4)的NLS,其序列为ANKVTKNKSN[14]。核定位信号分类见表 1。
| NLS类型 NLS type |
一致序列 Consensus sequence |
所属蛋白 Protein |
典型序列代表 Typical sequence representative |
| 单分型NLS Monopartite type NLS |
K-K/R-X-K/R | PRRSVN蛋白[15]、丁型肝炎病毒抗原[16]、小鼠p53蛋白[17]等 PRRSVN protein, Hepatitis D virus antigen, murine p53, etc |
SV40 T抗原NLS (PKKKRKV) SV40 T antigen NLS (PKKKRKV) |
| 双分型NLS Bipartite type NLS |
(K/R)(K/R)X10-12(K/R)3/5 | 大鼠糖皮质激素受体[18]、染色体浓缩调节因子1[19]等 Rat glucocorticoid receptor, chromosomal concentration regulator 1, etc |
非洲爪蟾核质蛋白NLS (KRPAATKKAGQAKKKK) Xenopus laevis nucleoplasmin NLS (KRPAATKKAGQAKKKK) |
| PY-NLS | R/K/H-X(2–5)-P-Y | hnRNP D、M9NLS等 hnRNP D, M9NLS etc |
M9NLS (FGYNNQSSNFGPMKGGNFGGRSS GPY) |
| IK-NLS | K-V/I-X-K-X1-2-K/H/R | 周期调节因子、核转录因子、核糖体蛋白等 Cycle regulators, nuclear transcription factors, ribosomal proteins, etc |
Pho4 NLS (ANKVTKNKSN) |
上述4类NLS序列在氨基酸排列顺序上均具有一定的规律性,随着研究者对NLS的深入研究,发现一些目前无规律可循却能介导蛋白入核的序列,如:在人类腺病毒(Human Adenovirus,hadv-5) E1A蛋白的保守区域3 (CR3)中发现的一个NLS,其共识序列为FV(X)7–26MXSLXYM(X)4MF,并不属于目前已总结出规律的NLS,但却能介导E1A蛋白入核[20]。此外,有研究者通过软件对鸡基质蛋白3预测出的几个NLS中,只有其中一个具备核定位功能,其余序列无核定位功能被称为假定NLS[21]。由此可见,通过识别已知一致序列并不能作为寻找入核蛋白NLS的唯一依据,对于NLS的研究如果过分依赖目前已有的NLS一致序列,可能会导致漏掉一些真正具有核定位功能的特殊结构,也有可能误导研究者将精力投入一些假定NLS序列的研究。
2 NLS介导蛋白入核机制目前报道的NLS所介导的入核机制研究中,主要围绕核质转运受体及其介导的核转运通路开展。依据NLS与受体结合方式的不同主要存在2类入核转运通路:(1) 由核质转运受体Importin β家族直接识别NLS介导的入核机制,如Importin β2识别PY-NLS[22]、Transport 3识别RS重复核定位信号[23]、Kap121识别富含赖氨酸残基核定位信号[24];(2) 由进口适配器识别并结合NLS形成二聚体后与Importin β形成三聚体再入核,如Importin α识别经典NLS[3]、Snoportin1识别富含鸟苷酸核糖核蛋白NLS[25]。
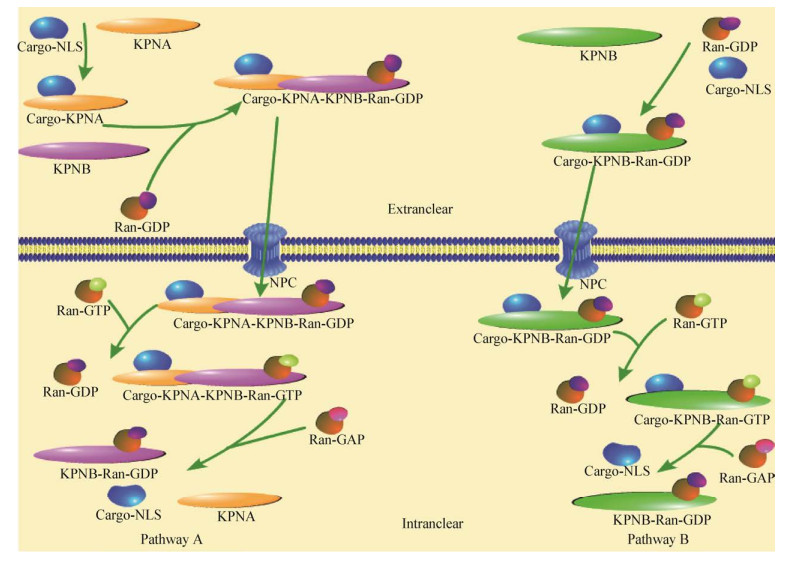
2.1 Importin α/Importin β介导的蛋白入核通路Importin α/Importin β途径介导含有经典NLS的蛋白入核(图 1 Pathway A),该机制中首先由核质转运受体Importin α的Armadillo (ARM)结构域识别货物蛋白(Cargo) NLS并与之结合形成二聚体(Cargo-Importin α),之后二聚体中Importin α的IBB (Important Beta Binding Domain)结构域与核质转运受体Importin β识别并形成三聚体(Cargo-Importin α-Importin β),然后三聚体中IBN-N结构域(Important Beta N-Terminal Binding Domain)与Ran-GDP结合形成四元复合物(Cargo-Importin α-importin β-Ran-GDP),其中Ran-GDP提供能量,四元复合物中Importin β的HEAT (Huntingtin、Elongation Factor 3、Protein Phosphatase 2A和TOR1)结构域与含有F-G重复序列的核孔蛋白结合使得核孔复合体构象发生变化从而进入核内。入核以后,细胞核内高浓度的Ran-GTP取代Ran-GDP形成新复合物(Cargo-Importin α-importin β-Ran-GTP),在Ran-GAP (Ran GTPase Activating Protein)的作用下,Ran-GTP复合物水解为Ran-GDP的形式,水解过程释放的能量使复合物解散为Importin α、Cargo和Importin β-Ran GDP;至此,具有NLS序列的货物蛋白由细胞质进入细胞核[3, 26]。上述蛋白入核途径需要Importin α与Importin β的联合作用才能完成,因此又称之为Importin α/Importin β途径。进一步研究表明Importin α具有3个区域,首先是其N端有一个IBB结构域,该区域能够与Importin β结合;IBB结构域具有自我抑制的序列,使Importin α具有自抑性[27](与NLS结合后,便使得该区域丧失与Importin β结合的能力);中部的多个ARM结构域由10个相对疏水的重复序列组成,每个重复由3个α螺旋构成,约含42-43个氨基酸,该区域与含有NLS的货物蛋白结合;ARM结构域有2个结合位点,分为主要结合位点和次要结合位点[28],其中单分型NLS结合“主要”结合位点,而双分型NLS同时结合“主要”与“次要”结合位点[5];第3个区域为C端结构域,该区域与细胞核内的输出蛋白细胞凋亡敏感性蛋白结合并将α亚基从核内运送到核外,使得Importin α能够再次参与核质转运[28]。
|
| 图 1 入核转运通路示意图 Figure 1 Schematic diagram of the nuclear transfer pathway 注:KPNA:Importin α;KPNB:Importin β;Pathway A:Importin α/Importin β入核转运通路;Pathway B:不需要进口适配器的Importin β入核转运通路 Note: KPNA: Importin α; KPNB: Importin β; Pathway A: The importin α/importin β nuclear transfer pathway; Pathway B: The importin β nuclear transfer pathway that does not require an import adapter |
|
|
人体中Importin α蛋白有7种,根据氨基酸的相似性分为3个亚型,分别为α1亚型:Importin α5、α6和α7;α2亚型:Importin α1和α8;α3亚型:Importin α3和α4[29]。除Importin α6仅存在于睾丸中外,Importin α的所有亚型在机体成体细胞中均有表达[29]。然而值得注意的是,同一亚型Importin蛋白在机体不同的发育阶段分布具有差异性,我们猜测这可能会影响到同一机体不同年龄段对于同一病毒的易感性。为适应流感病毒核蛋白NLS区域的突变最终导致适配蛋白Importin α3变为Importin α7,而这一改变被认为是流感病毒突破种间屏障(禽-人)传播的关键因素[28]。由此可见,病毒入核蛋白NLS区域的突变对于Importin α蛋白具有重新选择性,病毒毒力、传染力等重要特性也可能会随之改变。这说明病毒入核蛋白与核质转运受体的同步研究是必要的,这将有助于对病毒感染机制的重新认识,对于阻断病毒感染及靶向抗病毒药物的筛选具有重要意义。如表 2所示,本文对目前研究报道中已经明确的病毒入核蛋白NLS序列及其核质转运受体Importin α亚型进行了总结,以期为同步研究病毒入核蛋白与核质转运受体提供依据。
| 入核蛋白 Entry nuclear protein |
NLS序列 NLS sequence |
Importin α亚型 Importin α subtype |
病毒核酸类型 Viral nucleic acid type |
| D型流感病毒蛋白NP Influenza D virus NP protein |
(514KRR-X14(VGGADDVTLGT SOP)-KKRGR535) | α7 | RNA[30] |
| 人副流感病毒V/P的公共区域 The P/V common domain of human para influenza virus |
PVKPRRKK PVKPRRKK | α5和/或α7 α5 and/or α7 | RNA[31] |
| 牛腺病毒-3的22K蛋白 BAdV-3 22K protein |
RRRK | α5/α7 | DNA[32] |
| EB病毒的BFLF-2 Epstein-Barr virus BFLF2 |
RRLMHPHHRNYTASKASAH | α7 | DNA[33] |
| 甲型流感病毒的NP蛋白NLS2 Influenza A virus NP protein NLS2 |
NP-NLS-K GRKTR NP-NLS-R GRRTR (weakly) | α7 | RNA[28] |
| 原型泡沫病毒的BELL蛋白 Prototype foamy virus BELL protein |
PRQKRPR PRQKRPR | α1、α6、α7 | RNA[34] |
| 流感病毒的基质蛋白M1 Influenza A virus M1 Protein |
RKLKR | α1、α7 | RNA[35] |
| 单纯疱疹病毒(HSV-1) VP19C Herpes simplex virus (HSV-1) VP19C |
PRGSGPRRAAS | α5除外Except α5 | DNA[36] |
| PRV ICP 22伪狂犬病毒 PRV ICP 22 Pseudorabies virus |
PASTPTPPKRGRYVVEHPEY | α5 | DNA[37] |
Importin β作为Importin家族第二亚型,迄今发现共有20个成员。Importin β可识别并结合不同类型入核蛋白并通过与核孔蛋白的相互作用介导蛋白入核,Importin β的N端均含一个IBN-N结构域,可与Ras相关核蛋白结合;中部为HEAT结构域,与核孔复合体的FG-核孔蛋白结合;因Imprion β亚基HEAT的结构和构象不同导致其对货物有不同选择性,与底物的结合也不同[13, 38-39]。研究发现,Importin β某些亚型(如Importin β2)能够独立完成货物蛋白入核转运过程(图 1 Pathway B),该过程不需要Importin α的参与,其转运效率高于Importin α/Importin β联合作用下介导的蛋白入核;Importin β2也称Transport 2或Karyopherin β2,现阶段发现的可直接识别Importin β2介导蛋白入核的NLS大多为PY-NLS序列[22]。Importin β2上的20个HEAT重复序列构成超螺旋结构,2个重叠拱将该蛋白折叠成S型的超螺旋结构;其中,1-13 HEAT重复序列组成N端拱与RAN-GTP结合;8-20 HEAT重复序列则组成C端拱。Importin β2有2个结合位点与PY-NLS结合,其一位于C端与N端拱重叠的8-13 HEAT重复序列,该位点与PY-NLS亲和力较高,称之为A位点;第2个位点位于C端拱的14-18 HEAT重复序列,该位点与PY-NLS亲和力较低,称为B位点;当PY-NLS与B位点结合后,Importin β2构象会发生改变[12]。Importin β2直接识别的NLS中除PY-NLS外,少数为经典NLS[21]。
2.3 Snurportin 1/Importin β介导的入核通路入核受体蛋白Snurportin 1能够识别富含尿苷核糖核蛋白的NLS并与之结合形成二聚体,然后与Importin β结合形成三聚体之后入核。Snurportin 1结构有两个区域组成,其N端包含一个IBB结构域,可与Importin β进行识别连接,该区域与Importin β有较高的亲和力,解离速度较慢,IBB结构域C末端的螺旋长度增加将导致与Importin β亲和力增加,研究发现Importin α N端的IBB结构域与Snurportin 1 N端的IBB结构域在氨基酸序列上有很高的相似性,但结构上却并无相似性,两者相比较之下后者结构较为复杂;剩余蛋白质折叠成一个三甲基化鸟苷帽结合域,能够对富含尿苷的小核糖核酸蛋白(Uridine-Rich Small Nuclear Ribonucleoproteins,U snRNPs)进行核导入[25]。Snurportin 1 N端的IBB结构域“非比寻常”的亲核特性导致人们误以为货物蛋白可在不含RAN或GTP的情况下进行核导入。研究者通过将内源性RAN消耗掉的方式来研究该信号通路的能量需求发现:将内源性RAN消耗掉之后,未加外源性RAN则货物蛋白不能进入细胞核内,加入外源性RAN之后货物蛋白入核效率大大提高,而未经上述处理也未加外源性RAN该货物蛋白仍然得以进入核内,与经典IBB货物相比,其对外源能量需求仅为30%[24, 40]。
2.4 Kap121p介导的蛋白入核通路Karyopherins 121p (Kap121p)属于核转运蛋白-β超家族的成员,其结构上的24个HEAT重复结构域形成超螺旋结构,作为特征性结构,Kap121p在第15个HEAT重复序列中存在一突起球状区域[14]。Kap121p可识别IK-NLS,研究证实Kap121p与IK-NLS的结合位点为24个HEAT重复结构域中的第7-12个的内表面[24]。此外,研究发现Kap121p也可介导精氨酸和甘氨酸丰富的核定位信号(Arginine-Glycine Nuclear Localization Signal,RG-NLS)入核[14]。
2.5 Transport 3介导的蛋白入核通路Transport 3也称为Tnpo3或Transportin-SR2,同样属于核转运蛋白-β超家族成员,可识别富含精氨酸-丝氨酸-酪氨酸(Arginine-Serine-Tyrosine,RSY)结构域,介导蛋白入核;如冷诱导RNA结合蛋白中含有的该结构可与上述入核转运受体结合后进入细胞核[23]。从结构上来看,Transport 3同其他核转运蛋白-β超家族成员相似,由20个HEAT重复序列环绕成环形,C端结合SR蛋白,N端存在一个Ran GTP结合域,通过结合Ran GTP提供能量将SR蛋白导入核内[41]。
2.6 Xrip α/Importin β通路特异性导入复制蛋白A入核受体蛋白Xrip α包含3个结构域:N端为与Importin α相似的IBB结构域,称XIBB结构域,可特异性地与Importin β识别结合;中部为一个酸性区域;C端为一个锌指基序;复制蛋白A (Replication Protein A,RPA)在DNA复制、损伤修复过程中发挥着重要作用,其入核过程正是通过与Xrip α、Importin β结合形成三聚体,利用能量完成入核[25]。
目前能够介导蛋白入核的受体蛋白有Importin α、Importin β、Snoportin 1、Kap121p、Transport 3、Xrip α这6种,其中Importin β、Kap121p、Transport 3能够识别NLS序列并独立完成入核蛋白的转运,Importin α、Snoportin 1及Xrip α识别NLS序列的同时需要与Importin β结合形成三聚体才能完成入核蛋白的转运。携带NLS序列的蛋白入核过程均需要消耗能量。由表 3可以看出,不同入核蛋白受体识别的NLS序列各有特点。为了更加快速地锁定病毒入核蛋白及其相对应的NLS序列以及验证假定NLS序列,我们认为对入核转运通路的了解是非常有必要的,而入核转运蛋白与病毒入核蛋白亲和力的差异性有助于对病毒感染部位以及感染物种的差异性研究。
| Nuclear transfer pathways | Importin α/Importin β1 | Importin β2 | Snoportin 1/Importin β | Kap121p | Transport 3 | XriP α/Importin β |
| 识别的NLS Recognizd NLS |
经典NLS Classic NLS |
部分经典NLS,PY-NLS A part of classic NLS, PY-NLS |
富含尿苷核糖核蛋白的NLS U snRNPs NLS |
IK-NLS RG-NLS |
RS-NLS | 参与DNA代谢的蛋白RPA Protein RPA involved in DNA metabolism |
| 目标蛋白结合区域 Target protein binding domain |
Importin α中的ARM结构域 ARM domain in importin α |
8–13重复序列、14–18重复序列 8–13 HEAT, 14–18 HEAT |
Snoportin 1中的三甲基化鸟苷帽结合域 Trimethylated guanosine cap binding domain in snoportin 1 |
HEAT重复序列的7–12个内表面 7–12 inner surfaces of HEAT repeat sequence |
Transport3 C端结合域 Transport3 C-terminal binding domain |
Xrip α N末端的76个残基 76 residues at the N-terminus of Xrip α |
| 能量需求情况 Energy requirements |
RAN-GDP | RAN-GDP | RAN-GDP | RAN-GDP | RAN-GDP | RAN-GDP |
| 一致序列 Consensus sequence |
K-K/R-X-K/R, (K/R)(K/R)X10-12(K/R)3/5 | R/K/H-X(2–5)-P-Y | 未见一致序列 No consensus sequence |
K-V/I-X-K-X1-2-K/H/R、RG重复序列 K-V/I-X-K-X1-2-K/H/R,RG repeat sequence |
未见一致序列 No consensus sequence |
未见一致序列 No consensus sequence |
NLS序列所介导的病毒蛋白入核过程对病毒的复制以及宿主的免疫逃逸有着显著的影响。已有研究发现新城疫病毒M蛋白的核定位导致许多转录抑制活性相关的基因表达上调和转录激活物活性相关的基因表达下调,从而抑制宿主细胞的转录,并影响细胞的结合、催化活性、转录调节活性等[42];有研究证实新城疫病毒M蛋白NLS突变可导致病毒的复制能力以及致病性减弱[38];委内瑞拉马脑炎病毒衣壳蛋白入核后主要通过抑制宿主细胞β干扰素的合成提高自身的感染,当入核蛋白NLS序列发生突变后,病毒毒力大大减弱[43];抑制委内瑞拉马脑炎病毒核衣壳蛋白与入核转运受体的相互作用,发现可有效降低病毒滴度,延长细胞存活时间[7];H9N2病毒PA蛋白NLS突变导致该病毒在小鼠体内复制能力及致病性增强,突破从禽类到哺乳动物的种间突破[8];日本脑炎病毒核心蛋白NLS突变使病毒在体内的致病力减弱[9];狂犬病病毒P蛋白的核输入有利于抑制宿主基因的转录,调节病毒基因组的复制和转录,阻断宿主细胞抗病毒信号通路的激活,NLS突变后核进口受到抑制[44];登革热病毒非结构蛋白5 (NS5)中NLS关键残基突变可导致核定位NS5减少80%[45],寨卡病毒非结构蛋白(NS5)的NLS突变可导致宿主免疫应答能力改变[46];通过分离11株PRRSV以及HP-PRRSV参考株JXA1,发现N蛋白的NLS区序列发生了相同的突变,猜测NLS充分影响着病毒的毒力及复制能力[10]。由此可见,病毒入核蛋白及其携带的NLS在病毒的感染机制过程中发挥重要作用。
研究证实,绝大多数病毒蛋白进入细胞核均需要核转运受体蛋白Importin α或Importin β识别和结合靶蛋白携带的NLS序列;寨卡病毒NS5蛋白其受体蛋白Importin α识别NS5自身携带的NLS结构完成入核,该入核过程同样需要Importin β的共同作用,即寨卡病毒NS5与其受体结合是NS5-Importin α-Importin β三聚体的形式[46];A型流感病毒的PB2蛋白同样需要Importin α识别NLS后与Importin β结合以三聚体的形式才能完成入核[47]。研究同时发现,宿主蛋白磷脂爬行酶1 (Phospholipid Scramblase 1,PLSCR 1)可与流感病毒NP蛋白及Importin α形成三聚体,从而抑制NP蛋白入核,最终致使流感病毒复制周期延缓、病毒增殖受到抑制[48],进一步的研究证实竞争性与受体蛋白Importin α7结合的分子药物可有效降低流感病毒毒力及致病力[49]。研究证明,水痘带状疱疹病毒开放阅读区9基因编码的蛋白[50]、新城疫病毒M蛋白[51]等携带有NLS序列的病毒入核蛋白可以直接与Importin β结合,而不需要Importin α参与。人博卡病毒NP1蛋白因自身携带NLS序列的不同能够选择两种入核形式,即同时存在NP1-Importin α-Importin β与NP1-Importin β两种入核结合形态[52]。与此同时,文献资料中大量数据证实了Importin β直接介导的货物蛋白的转运速度高于Importin α/β介导的货物蛋白转运[53-54]。
作为蛋白间相互作用过程,入核蛋白与受体蛋白的结合及解离属于蛋白质配体识别结合过程中的动力学行为,该过程受范德华力、疏水性、氢键、离子键等诸多因素的影响[55]。依据结构生物学的中心法则:蛋白质的功能由其结构决定,而氨基酸的序列决定着蛋白质结构。因此,病毒入核蛋白在入核过程中与受体蛋白的结合方式及动力学行为因自身携带的NLS序列类型以及结构不同而变化,并最终导致其入核效率的差异。不论是入核转运蛋白还是NLS序列都将作为入核转运机制研究的重中之重,例如细小病毒用于基因治疗以及癌症治疗[56],但目前细小病毒进入细胞核效率低下,那么对于其入核转运机制的研究便极为重要。
4 结语及展望NLS及其受体介导的病毒蛋白入核转运机制不仅可以影响该病毒的复制能力、毒力以及致病性,还可影响宿主细胞的细胞周期、宿主细胞的结合、催化活性等,甚至于促使病毒突破种间屏障,而通过探究病毒蛋白入核机制无疑将为抗病毒新药的发现提供新的思路。
| [1] |
Lin DH, Hoelz A. The structure of the nuclear pore complex (an update)[J]. Annual Review of Biochemistry, 2019, 88(1): 725-783. DOI:10.1146/annurev-biochem-062917-011901 |
| [2] |
Kim YH, Han ME, Oh SO. The molecular mechanism for nuclear transport and its application[J]. Anatomy & Cell Biology, 2017, 50(2): 77-85. |
| [3] |
Kapinos LE, Huang B, Rencurel C, Lim RYH. Karyopherins regulate nuclear pore complex barrier and transport function[J]. The Journal of Cell Biology, 2017, 216(11): 3609-3624. DOI:10.1083/jcb.201702092 |
| [4] |
Li C, Goryaynov A, Yang WD. The selective permeability barrier in the nuclear pore complex[J]. Nucleus, 2016, 7(5): 430-446. DOI:10.1080/19491034.2016.1238997 |
| [5] |
Oka M, Yoneda Y. Importin α: functions as a nuclear transport factor and beyond[J]. Proceedings of the Japan Academy, Series B: Physical and Biological Sciences, 2018, 94(7): 259-274. DOI:10.2183/pjab.94.018 |
| [6] |
Fu XK, Liang C, Li FF, Wang LY, Wu XQ, Lu AP, Xiao GZ, Zhang G. The rules and functions of nucleocytoplasmic shuttling proteins[J]. International Journal of Molecular Sciences, 2018, 19(5): 1445. DOI:10.3390/ijms19051445 |
| [7] |
Atasheva S, Fish A, Fornerod M, Frolova EI. Venezuelan equine encephalitis virus capsid protein forms a tetrameric complex with CRM1 and importin α/β that obstructs nuclear pore complex function[J]. Journal of Virology, 2010, 84(9): 4158-4171. DOI:10.1128/JVI.02554-09 |
| [8] |
Xu GL, Zhang XX, Gao WH, Wang CX, Wang JL, Sun HL, Sun YP, Guo L, Zhang R, Chang KC, et al. Prevailing PA mutation K356R in Avian Influenza H9N2 virus increases mammalian replication and pathogenicity[J]. Journal of Virology, 2016, 90(18): 8105-8114. DOI:10.1128/JVI.00883-16 |
| [9] |
Mori Y, Okabayashi T, Yamashita T, Zhao ZJ, Wakita T, Yasui K, Hasebe F, Tadano M, Konishi E, Moriishi K, et al. Nuclear localization of Japanese encephalitis virus core protein enhances viral replication[J]. Journal of Virology, 2005, 79(6): 3448-3458. DOI:10.1128/JVI.79.6.3448-3458.2005 |
| [10] |
Li JD, Yin YB, Guo BQ, Zhou S, Zhang Y, Liu XC, Sun TT. Sequence analysis of the NSP2, ORF5, and ORF7 genes of 11 PRRS virus isolates from China[J]. Virus Genes, 2012, 45(2): 256-264. DOI:10.1007/s11262-012-0763-4 |
| [11] |
Soniat M, Chook YM. Nuclear localization signals for four distinct karyopherin-β nuclear import systems[J]. The Biochemical Journal, 2015, 468(3): 353-362. DOI:10.1042/BJ20150368 |
| [12] |
Lee BJ, Cansizoglu AE, Süel KE, Louis TH, Zhang ZC, Chook YM. Rules for nuclear localization sequence recognition by karyopherin β2[J]. Cell, 2006, 126(3): 543-558. DOI:10.1016/j.cell.2006.05.049 |
| [13] |
Soniat M, Chook YM. Karyopherin-β2 recognition of a PY-NLS variant that lacks the proline-tyrosine motif[J]. Structure, 2016, 24(10): 1802-1809. DOI:10.1016/j.str.2016.07.018 |
| [14] |
Kobayashi J, Matsuura Y. Structural basis for cell-cycle-dependent nuclear import mediated by the karyopherin Kap121p[J]. Journal of Molecular Biology, 2013, 425(11): 1852-1868. DOI:10.1016/j.jmb.2013.02.035 |
| [15] |
Pei YH, Hodgins DC, Lee C, Calvert JG, Welch SKW, Jolie R, Keith M, Yoo D. Functional mapping of the porcine reproductive and respiratory syndrome virus capsid protein nuclear localization signal and its pathogenic association[J]. Virus Research, 2008, 135(1): 107-114. DOI:10.1016/j.virusres.2008.02.012 |
| [16] |
Alves C, Freitas N, Cunha C. Characterization of the nuclear localization signal of the hepatitis delta virus antigen[J]. Virology, 2008, 370(1): 12-21. DOI:10.1016/j.virol.2007.07.034 |
| [17] |
Li HP, Zhang YP. Nuclear import of p53 in relation to MDM2-mediated degradation and ubiquitination[J]. Chinese Journal of Oncology, 2005, 27(2): 86-89. (in Chinese) 李惠平, 张延平. p53核输入功能在鼠双微体2调节的p53蛋白降解和泛素化中的作用[J]. 中华肿瘤杂志, 2005, 27(2): 86-89. |
| [18] |
Carrigan A, Walther RF, Salem HA, Wu DM, Atlas E, Lefebvre YA, Haché RJG. An active nuclear retention signal in the glucocorticoid receptor functions as a strong inducer of transcriptional activation[J]. The Journal of Biological Chemistry, 2007, 282(15): 10963-10971. DOI:10.1074/jbc.M602931200 |
| [19] |
Sankhala RS, Lokareddy RK, Begum S, Pumroy RA, Gillilan RE, Cingolani G. Three-dimensional context rather than NLS amino acid sequence determines importin α subtype specificity for RCC1[J]. Nature Communications, 2017, 8(1): 979. DOI:10.1038/s41467-017-01057-7 |
| [20] |
Marshall KS, Cohen MJ, Fonseca GJ, Todorovic B, King CR, Yousef AF, Zhang ZY, Mymryk JS. Identification and characterization of multiple conserved nuclear localization signals within adenovirus E1A[J]. Virology, 2014, 454-455: 206-214. DOI:10.1016/j.virol.2014.02.020 |
| [21] |
Deng SS. Study on the role of chicken matrix 3 protein in the replication of Newcastle disease virus[D]. Guiyang: Master's Thesis of Guizhou University, 2019 (in Chinese) 邓珊珊. 鸡基质蛋白3在新城疫病毒复制中的作用研究[D]. 贵阳: 贵州大学硕士学位论文, 2019 |
| [22] |
Hu Y, Xiong JM, Zhao JF, Ji XQ, Duan ZQ. Advances in importin β1-mediated nuclear transport of viral proteins in the replication of viruses[J]. Chinese Journal of Cell Biology, 2017, 39(8): 1091-1098. (in Chinese) 胡焱, 熊建民, 赵佳福, 嵇辛勤, 段志强. Importin β1介导的病毒蛋白入核转运在病毒复制中作用的研究进展[J]. 中国细胞生物学学报, 2017, 39(8): 1091-1098. |
| [23] |
Bourgeois B, Hutten S, Gottschalk B, Hofweber M, Richter G, Sternat J, Abou-Ajram C, Göbl C, Leitinger G, Graier WF, et al. Nonclassical nuclear localization signals mediate nuclear import of CIRBP[J]. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(15): 8503-8514. DOI:10.1073/pnas.1918944117 |
| [24] |
Hirano H, Kobayashi J, Matsuura Y. Structures of the karyopherins Kap121p and Kap60p bound to the nuclear pore-targeting domain of the SUMO protease Ulp1p[J]. Journal of Molecular Biology, 2017, 429(2): 249-260. DOI:10.1016/j.jmb.2016.11.029 |
| [25] |
Lott K, Cingolani G. The importin β binding domain as a master regulator of nucleocytoplasmic transport[J]. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research, 2011, 1813(9): 1578-1592. DOI:10.1016/j.bbamcr.2010.10.012 |
| [26] |
Fogelson B, Keener JP. Enhanced nucleocytoplasmic transport due to competition for elastic binding sites[J]. Biophysical Journal, 2018, 115(1): 108-116. DOI:10.1016/j.bpj.2018.05.034 |
| [27] |
Pang XD, Zhou HX. Design rules for selective binding of nuclear localization signals to minor site of importin α[J]. PLoS One, 2014, 9(3): e91025. DOI:10.1371/journal.pone.0091025 |
| [28] |
Wu W, Sankhala RS, Florio TJ, Zhou LX, Nguyun NLT, Lokareddy RK, Cingolani G, Panté N. Synergy of two low-affinity NLSs determines the high avidity of influenza A virus nucleoprotein NP for human importin α isoforms[J]. Scientific Reports, 2017, 7(1): 11381. DOI:10.1038/s41598-017-11018-1 |
| [29] |
Oostdyk LT, McConnell MJ, Paschal BM. Characterization of the importin-β binding domain in nuclear import receptor KPNA7[J]. The Biochemical Journal, 2019, 476(21): 3413-3434. DOI:10.1042/BCJ20190717 |
| [30] |
Donchet A, Oliva J, Labaronne A, Tengo L, Miloudi M, Gerard FCA, Mas C, Schoehn G, Ruigrok RWH, Ducatez M, et al. The structure of the nucleoprotein of influenza D shows that all Orthomyxoviridae nucleoproteins have a similar NPCORE, with or without a NPTAIL for nuclear transport[J]. Scientific Reports, 2019, 9(1): 600. DOI:10.1038/s41598-018-37306-y |
| [31] |
Ohtsuka J, Matsumoto Y, Ohta K, Fukumura M, Tsurudome M, Nosaka T, Nishio M. Nucleocytoplasmic shuttling of the human parainfluenza virus type 2 phosphoprotein[J]. Virology, 2019, 528: 54-63. DOI:10.1016/j.virol.2018.12.005 |
| [32] |
Said A, Wang WX, Woldermariam T, Tikoo SK. Domains of bovine adenovirus-3 protein 22K involved in interacting with viral protein 52K and cellular importins α-5/α-7[J]. Virology, 2018, 522: 209-219. DOI:10.1016/j.virol.2018.07.015 |
| [33] |
Li ML, Chen T, Zou XM, Xu Z, Wang YF, Wang Y, Ou XW, Li YW, Chen DX, Peng T, et al. Characterization of the nucleocytoplasmic transport mechanisms of Epstein-Barr virus BFLF2[J]. Cellular Physiology and Biochemistry, 2018, 51(4): 1500-1517. DOI:10.1159/000495641 |
| [34] |
Duan JH, Tang ZQ, Mu H, Zhang GJ. [Retracted] Nuclear import of prototype foamy virus transactivator Bel1 is mediated by KPNA1, KPNA6 and KPNA7[J]. International Journal of Molecular Medicine, 2017, 39(3): 771. DOI:10.3892/ijmm.2017.2860 |
| [35] |
Liu QF, Bawa B, Ma JJ, Li F, Ma WJ, Richt JA. A crucial role of N-terminal domain of influenza A virus M1 protein in interaction with swine importin α1 protein[J]. Virus Genes, 2014, 49(1): 157-162. DOI:10.1007/s11262-014-1068-6 |
| [36] |
Li Y, Zhao L, Wang S, Xing JJ, Zheng CF. Identification of a novel NLS of herpes simplex virus type 1 (HSV-1) VP19C and its nuclear localization is required for efficient production of HSV-1[J]. The Journal of General Virology, 2012, 93(9): 1869-1875. DOI:10.1099/vir.0.042697-0 |
| [37] |
Li ML, Wang S, Cai MS, Guo H, Zheng CF. Characterization of molecular determinants for nucleocytoplasmic shuttling of PRV UL54[J]. Virology, 2011, 417(2): 385-393. DOI:10.1016/j.virol.2011.06.004 |
| [38] |
Duan ZQ, Xu HX, Ji XQ, Zhao JF, Xu HQ, Hu Y, Deng SS, Hu SL, Liu XF. Importin α5 negatively regulates importin β1-mediated nuclear import of Newcastle disease virus matrix protein and viral replication and pathogenicity in chicken fibroblasts[J]. Virulence, 2018, 9(1): 783-803. DOI:10.1080/21505594.2018.1449507 |
| [39] |
Kumeta M, Konishi HA, Zhang W, Sakagami S, Yoshimura SH. Prolines in the α-helix confer the structural flexibility and functional integrity of importin-β[J]. Journal of Cell Science, 2018, 131(1): jcs206326. DOI:10.1242/jcs.206326 |
| [40] |
Lott K, Bhardwaj A, Mitrousis G, Pante N, Cingolani G. The importin β binding domain modulates the avidity of importin β for the nuclear pore complex[J]. The Journal of Biological Chemistry, 2010, 285(18): 13769-13780. DOI:10.1074/jbc.M109.095760 |
| [41] |
Maertens GN, Cook NJ, Wang W, Hare S, Gupta SS, Öztop I, Lee K, Pye VE, Cosnefroy O, Snijders AP, et al. Structural basis for nuclear import of splicing factors by human transportin 3[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(7): 2728-2733. DOI:10.1073/pnas.1320755111 |
| [42] |
Duan ZQ, Deng SS, Ji XQ, Zhao JF, Yuan C, Gao HB. Nuclear localization of Newcastle disease virus matrix protein promotes virus replication by affecting viral RNA synthesis and transcription and inhibiting host cell transcription[J]. Veterinary Research, 2019, 50(1): 22. DOI:10.1186/s13567-019-0640-4 |
| [43] |
Lundberg L, Fontenot J, Lin SC, Pinkham C, Carey BD, Campbell CE, Kehn-Hall K. Venezuelan equine encephalitis virus capsid implicated in infection-induced cell cycle delay in vitro[J]. Frontiers in Microbiology, 2018, 9: 3126. DOI:10.3389/fmicb.2018.03126 |
| [44] |
Oksayan S, Nikolic J, David CT, Blondel D, Jans DA, Moseley GW. Identification of a role for nucleolin in rabies virus infection[J]. Journal of Virology, 2015, 89(3): 1939-1943. DOI:10.1128/JVI.03320-14 |
| [45] |
Fraser JE, Rawlinson SM, Wang CX, Jans DA, Wagstaff KM. Investigating dengue virus nonstructural protein 5 (NS5) nuclear import[A]//Methods in Molecular Biology[M]. New York: Humana Press, 2014, 1138: 301-328
|
| [46] |
Tan MJA, Chan KWK, Ng IHW, Kong SYZ, Gwee CP, Watanabe S, Vasudevan SG. The potential role of the ZIKV NS5 nuclear spherical-shell structures in cell type-specific host immune modulation during ZIKV infection[J]. Cells, 2019, 8(12): 1519. DOI:10.3390/cells8121519 |
| [47] |
Pumroy RA, Ke S, Hart DJ, Zachariae U, Cingolani G. Molecular determinants for nuclear import of influenza A PB2 by importin α isoforms 3 and 7[J]. Structure, 2015, 23(2): 374-384. DOI:10.1016/j.str.2014.11.015 |
| [48] |
Luo WY, Zhang J, Liang LB, Wang GW, Li QB, Zhu PY, Zhou Y, Li JP, Zhao YH, Sun N, et al. Phospholipid scramblase 1 interacts with influenza A virus NP, impairing its nuclear import and thereby suppressing virus replication[J]. PLoS Pathogens, 2018, 14(1): e1006851. DOI:10.1371/journal.ppat.1006851 |
| [49] |
Resa-Infante P, Paterson D, Bonet J, Otte A, Oliva B, Fodor E, Gabriel G. Targeting importin-α7 as a therapeutic approach against pandemic influenza viruses[J]. Journal of Virology, 2015, 89(17): 9010-9020. DOI:10.1128/JVI.00583-15 |
| [50] |
Cai M, Wang S, Xing J, Zheng C. Characterization of the nuclear import and export signals, and subcellular transport mechanism of varicella-zoster virus ORF9[J]. The Journal of General Virology, 2011, 92(3): 621-626. DOI:10.1099/vir.0.027029-0 |
| [51] |
Duan ZQ, Ji XQ, Xu HQ, Zhao JF, Xu HX, Hu SL, Liu XF. The nuclear import of Newcastle disease virus matrix protein depends on KPNB1 and Ran protein[J]. Acta Microbiologica Sinica, 2017, 57(1): 109-120. (in Chinese) 段志强, 嵇辛勤, 许厚强, 赵佳福, 许海旭, 胡顺林, 刘秀梵. KPNB1和Ran蛋白共同介导新城疫病毒基质蛋白的入核转运[J]. 微生物学报, 2017, 57(1): 109-120. |
| [52] |
Li Q, Zhang ZF, Zheng ZH, Ke XL, Luo HL, Hu QX, Wang HZ. Identification and characterization of complex dual nuclear localization signals in human bocavirus NP1[J]. The Journal of General Virology, 2013, 94(6): 1335-1342. DOI:10.1099/vir.0.047530-0 |
| [53] |
Christie M, Chang CW, Róna G, Smith KM, Stewart AG, Takeda AAS, Fontes MRM, Stewart M, Vértessy BG, Forwood JK, et al. Structural biology and regulation of protein import into the nucleus[J]. Journal of Molecular Biology, 2016, 428(10): 2060-2090. DOI:10.1016/j.jmb.2015.10.023 |
| [54] |
Yoshimura SH, Kumeta M, Takeyasu K. Structural mechanism of nuclear transport mediated by importin β and flexible amphiphilic proteins[J]. Structure, 2014, 22(12): 1699-1710. DOI:10.1016/j.str.2014.10.009 |
| [55] |
Lehne B, Schlitt T. Protein-protein interaction databases: keeping up with growing interactomes[J]. Human Genomics, 2009, 3(3): 291. DOI:10.1186/1479-7364-3-3-291 |
| [56] |
Mäntylä E, Aho V, Kann M, Vihinen-Ranta M. Cytoplasmic parvovirus capsids recruit importin beta for nuclear delivery[J]. Journal of Virology, 2020, 94(4): e01532-19. |
 2021, Vol. 48
2021, Vol. 48