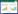扩展功能
文章信息
- 耿忆敏, 任思雨, 于涛, 姜晓冰
- GENG Yimin, REN Siyu, YU Tao, JIANG Xiaobing
- VirAB在单核细胞增生李斯特菌耐药性及生物被膜形成中的作用
- Role of VirAB in antimicrobial resistance and biofilm formation of Listeria monocytogene
- 微生物学通报, 2021, 48(2): 471-479
- Microbiology China, 2021, 48(2): 471-479
- DOI: 10.13344/j.microbiol.china.200122
-
文章历史
- 收稿日期: 2020-02-20
- 接受日期: 2020-03-29
- 网络首发日期: 2020-07-30
2. 新乡学院生命科学技术学院 河南 新乡 453000
2. School of Life Sciences and Technology, Xinxiang University, Xinxiang, Henan 453000, China
单核细胞增生李斯特菌(Listeria monocytogenes,Lm)是一种革兰氏阳性短杆菌,易对肉、蛋、奶制品等食品造成污染,被认为是一种重要的食源性致病菌[1]。摄入被Lm污染的食品可能会引起李斯特菌病(Listeriosis),该病具有较高的致死率[2]。Lm对不利环境的适应能力很强,能在低温、酸、高盐以及长时间干燥等条件下生存和繁殖,增加食品在加工和保藏环节中被其污染的风险[3]。
细菌转运蛋白(Transport Proteins)属于膜蛋白,主要参与营养物质的摄入、代谢产物的释放以及信号转导等生理活动[4]。其中,ABC转运蛋白家族是一大类膜转运蛋白,在细菌中广泛存在,通常由通透酶和ATP结合蛋白组成,能够利用ATP水解产生的能量将与其结合的底物转运出质膜。ABC转运蛋白家族中的一些成员可以把抗生素、消毒剂等抗菌药物排出胞外,从而介导细菌多重耐药[5]。
经转运蛋白数据库(http://www.membranetransport.org/)检索可知,Lm标准菌株EGD-e中存在172个可能的ABC转运蛋白,其中仅有少数几个的生理功能已知[6-8]。早期的研究显示,在Lm菌株LM-49中一个可能的ABC转运蛋白与生物膜的形成有关[9]。经序列比对可知,该蛋白的氨基酸序列与EGD-e的Lmo1746和Lmo1747同源。如图 1所示,lmo1746编码的ABC转运蛋白通透酶VirB由659个氨基酸组成,包含10个跨膜结构域;lmo1747编码ATP结合蛋白VirA。除了可能参与Lm生物膜的形成,VirAB的其他功能仍然未知。因此,本实验在前人研究的基础上探究ABC转运蛋白VirAB在Lm标准菌株EGD-e对抗菌药物耐药、生物膜的形成及运动性中的作用。
|
| 图 1 virA/virB基因结构图 Figure 1 virA/virB gene structure diagram |
|
|
Lm标准菌株EGD-e由华中师范大学罗勤教授惠赠;大肠杆菌DH5α感受态细胞购自新乡市普金生物技术有限公司;大肠杆菌HB101和质粒pPL2由芬兰赫尔辛基大学Hannu Kokeala教授惠赠;穿梭质粒pMAD由本实验室保存。
1.2 主要试剂和仪器苯扎氯铵(Benzalkonium Chloride,BC),国药集团化学试剂有限公司;溴化乙锭(Ethidium Bromide,EB)、DL2000 DNA Marker,北京索莱宝科技有限公司;细菌基因组DNA提取试剂盒,RealMaster Mix (SYBR Green)试剂盒,天根生化科技(北京)有限公司;高保真PCR酶和dNTPs,宝生物工程(大连)有限公司;细菌RNA提取试剂盒,上海碧云天生物技术有限公司;质粒小提试剂盒、PCR产物纯化试剂盒、限制性内切酶、胶回收试剂盒、T4 DNA连接酶,赛默飞世尔科技公司;引物由河南尚亚生物技术有限公司合成。
基因扩增仪,北京东胜创新生物科技有限公司;电热恒温培养箱,北京科伟永兴仪器有限公司;离心机,Sigma公司;凝胶成像分析系统、电转仪、酶标仪,Bio-Rad公司;实时荧光定量PCR仪,ABI公司;微生物全自动生长曲线分析仪,Oy Growth Curves Ab有限公司;倒置显微镜,Leica公司。
1.3 培养基脑心浸液培养基(Brain Heart Infusion,BHI),广东环凯微生物科技有限公司;LB培养基(g/L):酵母提取物5.0,胰蛋白胨10.0,NaCl 10.0;胰蛋白胨大豆肉汤培养基(Tryptic Soy Broth,TSB),北京索莱宝科技有限公司;ALOA培养基,北京缔一生物科技有限公司;BHI+BC培养基:BHI液体培养基中添加BC至终浓度为2 μg/mL。
1.4 方法 1.4.1 基因缺失突变株的构建利用细菌基因组DNA提取试剂盒提取Lm野生株EGD-e的基因组,以EGD-e基因组DNA为模板分别扩增virA、virB和virAB的上游和下游同源臂,引物序列见表 1。利用重叠延伸PCR (Splicing by Overlap Extension PCR,SOE-PCR)[10]技术分别融合3个基因的上、下游同源臂,融合得到的长片段依次经切胶回收、双酶切及纯化后,与穿梭质粒pMAD[11]进行连接反应。利用热激法将构建完成的重组质粒转化至大肠杆菌DH5α感受态细胞中,筛选阳性转化株并送至南京金斯瑞生物科技有限公司进行测序。
| 引物 Primers |
序列 Sequences (5′→3′) |
退火温度 Annealing temperature (℃) |
产物长度 Sizes (bp) |
备注 Remark |
| 1746-1 | CGGGATCCTATGCTCCATTACATTTGCTT (BamH I) | 49 | 705 | virB上游同源臂 virB upstream homology arm |
| 1746-2 | GGGCTTTATTCATTACTTGGTGGGCTTTATTCATTACTTGGT | |||
| 1746-3 | GATTTCTTTTTCAGTAACCCCACCAAGTAATGAATAAAGCCC | 49.7 | 549 | virB下游同源臂 virB downstream homology arm |
| 1746-4 | CGGAATTCATTTTTGCGGATGAACCC (EcoR I) | |||
| 1746-5 | NNNNNNTCTAGATTACTTTTTATTGGCCATCACAATT (BamH I) | 54 | 2 184 | virB回复序列 virB palindrome sequence |
| 1746-6 | NNNNNNGGATCCAAGGACTCTCTCAAGCAAAAG (Xba I) | |||
| 1747-1 | NNNNNNGGATCCACCAAGTAATGAATAAAGCCC (BamH I) | 50 | 547 | virA上游同源臂 virA upstream homology arm |
| 1747-2 | AACGTAACGGGAGGAATACAATTTTTGCGGATGAACCC | |||
| 1747-3 | GGGTTCATCCGCAAAAATTGTATTCCTCCCGTTACGTT | 51 | 559 | virA下游同源臂 virA downstream homology arm |
| 1747-4 | NNNNNNACGCGTAGTTGTTTACTTTGGATGGC (Mlu I) | |||
| 1747-5 | NNNNNNGGTACCTTAAATAACATCGTTTTCGCCACC (Kpn I) | 55 | 927 | virA回复序列 virA palindrome sequence |
| 1747-6 | NNNNNNGGATCCTTGGAATGGAAATCTTTGTGGAAAA (BamH I) | |||
| 1746/1747-1 | NNNNNNGGATCCTCACGACTTACAATGCTACCT (Bam H I) | 50 | 589 | virAB上游同源臂 virAB upstream homology arm |
| 1746/1747-2 | AACGTAACGGGAGGAATACATTGGCTTTTGTGGTTATTTC | |||
| 1746/1747-3 | GAAATAACCACAAAAGCCAATGTATTCCTCCCGTTACGTT | 50 | 559 | virAB下游同源臂 virAB downstream homology arm |
| 1746/1747-4 | NNNNNNACGCGTAGTTGTTTACTTTGGATGGC (Mlu I) | |||
| 1746/1747-5 | NNNNNNGGTACCTTACTTTTTATTGGCCATCACAATT (Kpn I) | 54 | 2 896 | virAB回复序列 virAB palindrome |
| 1746/1747-6 | NNNNNNGGATCCTTGGAATGGAAATCTTTGTGGAAAA (BamH I) | sequence | ||
| 注:斜体部分序列表示保护碱基;下划线部分表示酶切位点 Note: Italic partial sequence indicates protective bases; Underlined part indicates enzyme digestion site | ||||
使用青霉素G法制备EGD-e感受态细胞[12]。将测序完全正确的重组质粒电转至EGD-e感受态细胞,利用红霉素抗性筛选缺失突变株。
1.4.2 回复突变株的构建利用回复引物1746-5/1746-6、1747-5/1747-6和1746/1747-5、1746/1747-6进行PCR[13]扩增基因全长及启动子区域,扩增产物经双酶切后与pPL2质粒连接,重组质粒转化至大肠杆菌DH101中。以阳性转化子为供体菌,缺失突变株为受体菌进行接合转移,在含有氯霉素(7.5 μg/mL)的ALOA平板上筛选接合子,经PCR验证后最终获得回复突变株。同时将质粒pPL2按照上述办法转化至缺失突变株作为对照菌株。
1.4.3 药物敏感性试验按照美国临床实验室标准协会(Clinical and Laboratory Standards Institute,CLSI)推荐的琼脂稀释法检测实验菌株对抗菌药物的最小抑菌浓度(Minimum Inhibitory Concentration,MIC)值[14]。
1.4.4 实时荧光定量PCR检测基因转录水平将EGD-e菌液平均分为2组,一组不加任何药物作为对照,另一组加入头孢噻肟(2 μg/mL),37 ℃、120 r/min继续培养30 min。按照细菌RNA提取试剂盒的使用说明提取细菌总RNA,利用超微量分光光度计测定RNA的浓度和纯度后-80 ℃保存备用。将提取的细菌总RNA反转录为cDNA,以cDNA作为模板,使用RealMaster Mix (SYBR Green)试剂盒进行实时荧光定量PCR[15]。采用2–ΔΔCT法进行基因转录水平的分析。
1.4.5 生长曲线分析从冻存管中挑取实验菌株在BHI固体平板上划线,待长出明显的菌落后,分别挑取3个不同的单克隆接种至BHI液体培养基中37 ℃、120 r/min培养过夜。吸取菌液按1׃100的比例分别转接至BHI和BHI+BC (2 μg/mL)液体培养基中,然后再转移至100孔蜂窝板中。利用微生物全自动生长曲线仪测定菌液的OD600值。利用DMFit软件(英国食品研究所研制)[16]拟合出生长曲线,并计算菌株生长的迟滞期(Lag Phase Duration,LPD)、平均最大生长率(Mean Maximum Growth Rate,MMGR)以及平均最大光密度值(Mean Maximum Optical Density,MMOD)。
1.4.6 点板法测定菌株对卡那霉素和四环素的药物敏感性具体方法参照徐雅梦等[4]的报道进行:将野生株EGD-e和突变株的培养物分别做10倍比梯度稀释,选取若干个连续稀释度的菌液分别点接至不含抗生素的BHI平板和含有不同浓度抗生素的BHI平板上,37 ℃培养24 h。
1.4.7 微孔板法检测菌体的生长和生物被膜的形成具体方法参照Djordjevic等[17]的报道进行:将菌株培养至对数早期(OD600值约为0.4),100倍稀释后取200 μL加入到96孔微孔板中,37 ℃分别培养24、48、72 h后,首先用酶标仪测定菌液OD600值,然后小心弃去孔中的培养基,经草酸铵结晶紫溶液染色、95%的乙醇溶液脱色后,用酶标仪检测其OD570值,同时置于倒置显微镜下观察拍照。
1.4.8 菌株泳动性检测将野生株EGD-e和突变株ΔvirAB接种至3 mL BHI液体培养基中,37 ℃、150 r/min培养过夜。将配制好的半固体胰酪大豆胨液体培养基(TSB)平板倒置,待其水分晾干。将菌液稀释100倍后,吸取1 μL稀释液点在TSB平板上,25 ℃静置培养48 h,观察菌落的形成情况。
1.4.9 数据分析利用Origin 8.5软件对所得的实验数据进行双尾t检验分析。
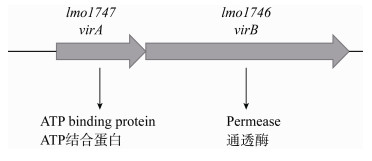
2 结果与分析 2.1 基因缺失株的构建与分子鉴定由图 2可见,利用lmo1746/1747-1/4引物对,以疑似ΔvirAB突变株的基因组为模板扩增得到1 148 bp的短片段;利用lmo1746-1/4引物对,以疑似ΔvirB突变株的基因组为模板扩增得到1 253 bp的短片段;利用lmo1747-1/4引物对,以疑似ΔvirA突变株的基因组为模板扩增得到1 106 bp的短片段。测序结果表明这些疑似突变株均为敲除成功的基因缺失株。
|
| 图 2 PCR检测基因缺失突变株 Figure 2 Determination of the deletion mutant strains by PCR 注:M:DL2000 DNA Marker;1:ΔvirB;2、4:阴性对照;3:ΔvirA;5:ΔvirAB Note: M: DL2000 DNA Marker; 1: ΔvirB; 2, 4: Negative control; 3: ΔvirA; 5: ΔvirAB |
|
|
与野生株EGD-e相比,3株缺失株对头孢噻肟、头孢噻吩、头孢吡肟、乳酸链球菌素(Nisin)和EB的MIC值均下降1/2,对头孢他啶的MIC值下降1/4 (表 2),表明VirAB参与Lm对头孢类抗生素、Nisin和EB的耐药。
| 抗菌药物 Antimicrobial agent |
菌株 Strain | |||
| EGD-e | ΔvirAB | ΔvirB | ΔvirA | |
| 抗生素 Antibiotic | ||||
| 氨苄西林 Ampicillin | 1 | 1 | 1 | 1 |
| 头孢噻肟 Cefotaxime | 8 | 4 | 4 | 4 |
| 头孢噻吩 Cephalothin | 8 | 4 | 4 | 4 |
| 头孢他啶 Ceftazidime | 16 | 4 | 4 | 4 |
| 头孢吡肟 Cefepime | 4 | 2 | 2 | 2 |
| 环丙沙星 Ciprofloxacin | 0.5 | 0.5 | 0.5 | 0.5 |
| 卡那霉素 Kanamycin | 2 | 2 | 2 | 2 |
| 四环素Tetracycline | 0.5 | 0.5 | 0.5 | 0.5 |
| 氯霉素 Chloramphenicol | 4 | 4 | 4 | 4 |
| 抗菌肽 Antimicrobial peptide | ||||
| 乳酸链球菌素 Nisin | 12.5 | 6.25 | 6.25 | 6.25 |
| 染料 Dye | ||||
| 溴化已锭 Ethidium bromide | 20 | 10 | 10 | 10 |
| 消毒液 Disinfectant | ||||
| 苯扎氯铵 Benzalkonium chloride | 6 | 6 | 6 | 6 |
构建的ΔvirAB、ΔvirB、ΔvirA回复突变株分别命名为CΔvirAB、CΔvirB、CΔvirA。这3株回复突变株对头孢噻肟、头孢噻吩、头孢他啶、头孢吡肟、Nisin和EB的MIC值恢复至野生株的水平(表 3),表明本研究中构建的基因缺失株为非极性突变,排除极性效应的影响。
| 菌株 Strain |
头孢噻肟 Cefotaxime |
头孢噻吩 Cephalothin |
头孢他啶 Ceftazidime |
头孢吡肟 Cefepime |
乳酸链球菌素 Nisin |
溴化已锭 EB |
| EGD-e | 8 | 8 | 16 | 4 | 12.5 | 20 |
| CΔvirAB | 8 | 8 | 16 | 4 | 12.5 | 20 |
| CΔvirB | 8 | 8 | 16 | 4 | 12.5 | 20 |
| CΔvirA | 8 | 8 | 16 | 4 | 12.5 | 20 |
| ΔvirAB pERL3 | 4 | 4 | 4 | 2 | 6.25 | 10 |
| ΔvirB pERL3 | 4 | 4 | 4 | 2 | 6.25 | 10 |
| ΔvirA pERL3 | 4 | 4 | 4 | 2 | 6.25 | 10 |
在头孢噻肟的作用下,virAB在野生株EGD-e中的相对转录水平显著提高8.58倍(P < 0.05),表明virAB的转录受到头孢噻肟的诱导。
2.5 生长曲线分析为了进一步调查virAB在Lm对BC耐药中的作用,对EGD-e和ΔvirAB在BC胁迫下的生长曲线进行分析。结果显示,无BC时2种菌株的生长情况一致(图 3A);BC胁迫不仅使ΔvirAB的LPD明显变长,为EGD-e的1.71倍,还使ΔvirAB的MMGR和MMOD则分别降低为EGD-e的44%和41% (图 3B,表 4)。
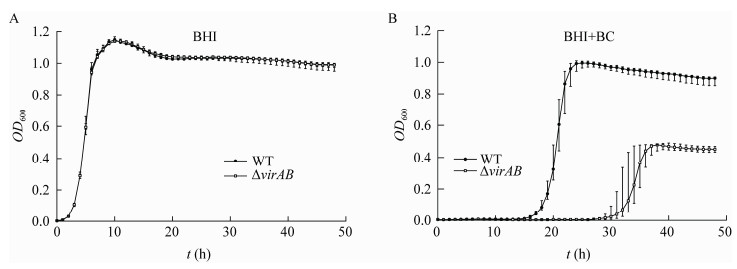
|
| 图 3 野生株EGD-e和突变株ΔvirAB的生长曲线 Figure 3 Growth curves for the wild-type EGD-e and the mutant ΔvirAB 注:A:野生株EGD-e和突变株ΔvirAB在BHI中的生长曲线;B:野生株EGD-e和突变株ΔvirAB在BHI+BC中的生长曲线 Note: A: Growth curves for the wild-type EGD-e and the mutant ΔvirAB in BHI; B: Growth curves for the wild-type EGD-e and the mutant ΔvirAB in BHI+BC |
|
|
| 菌株 Strain |
迟滞期 LPD (h) |
平均最大生长率 MMGR (units/h) |
平均最大光密度值 MMOD (units) |
|||||
| BHI | BC | BHI | BC | BHI | BC | |||
| EGD-e | 3.27±0.192 | 18.63±0.063 | 0.349±0.034 | 0.255±0.016 | 1.15±0.013 | 1.14±0.011 | ||
| ΔvirAB | 3.22±0.197 | 31.87±0.093* | 0.336±0.032 | 0.112±0.004* | 1.10±0.093 | 0.47±0.021* | ||
| 注:*:与野生株EGD-e相比的差异显著性(P < 0.05) Note: Significantly varied values compared to the corresponding value of the wild-type strain EGD-e are indicated by asterisks (P < 0.05) | ||||||||
经平板计数可知野生株EGD-e和突变株ΔvirAB初始菌液的菌落总数分别为2.25×1011 CFU/mL和1.79×1011 CFU/mL,两者数值相近。由图 4可知,在BHI固体平板上,EGD-e和ΔvirAB均能正常生长;在含有0.5 μg/mL卡那霉素的平板上,EGD-e和ΔvirAB的生长显现出差异(稀释倍数大于104时肉眼无法观察到ΔvirAB的生长);在含有0.25 μg/mL四环素的平板上,ΔvirAB的生长明显弱于EGD-e (在所有稀释倍数下均无法观察到ΔvirAB的生长)。
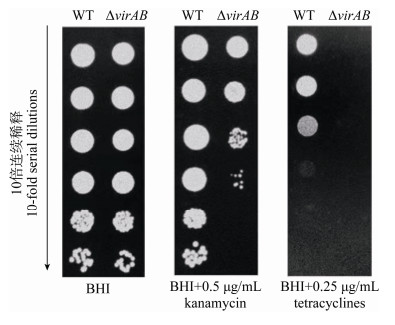
|
| 图 4 平板法检测野生株EGD-e和突变株ΔvirAB对卡那霉素和四环素的敏感性 Figure 4 Determination of kanamycin and tetracyclines susceptibility of the wild-type strain EGD-e and the mutant strain ΔvirAB using the plate-based assay |
|
|
如图 5A所示,在1、2、3 d的培养过程中,野生株EGD-e和突变株ΔvirAB在各时间段的生长量无明显差异;与EGD-e相比,ΔvirAB形成的生物被膜量明显降低,在1、2、3 d分别下降了5%、10%和60% (图 5B)。显微镜下2株菌形成的生物被膜结构如图 5C、D所示:EGD-e形成的链网状生物被膜,结构致密,孔隙很小;而ΔvirAB虽然能形成完整的链网状生物被膜,但其结构疏散、孔隙较大。该结果与微孔板定量检测结果一致,表明virAB缺失使Lm形成生物被膜的能力降低。
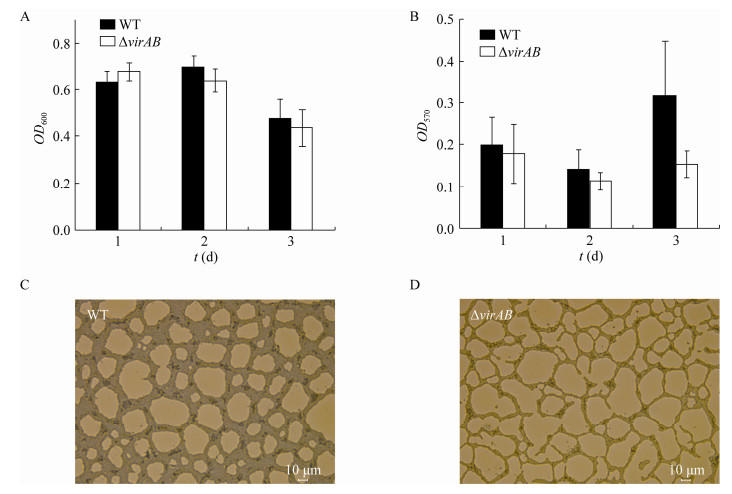
|
| 图 5 野生株EGD-e和突变株ΔvirAB生物被膜的形成 Figure 5 Biofilm formation of the wild-type strain EGD-e and the mutant ΔvirAB 注:A:野生株EGD-e与突变株ΔvirAB的OD600值;B:野生株EGD-e与突变株ΔvirAB的OD570值;C:野生株EGD-e生物被膜图;D:突变株ΔvirAB生物被膜图 Note : A : Wild-type strain EGD-e and the mutant ΔvirAB OD600 values; B: Wild-type strain EGD-e and the mutant ΔvirAB OD570 values; C: Biofilm formation of the wild-type strain EGD-e; D: Biofilm formation of the mutant ΔvirAB |
|
|
在37 ℃培养条件下,由于鞭毛蛋白的合成受到抑制,野生株EGD-e和突变株ΔvirAB均未表现出泳动性,形成较小的紧凑型菌落;在25 ℃条件下,EGD-e和ΔvirAB均能形成边缘整齐的同心环状菌落,表现出良好的泳动性,两者的泳动扩散直径分别为0.6 cm和0.64 cm,无明显差异(图 6)。这表明virAB的缺失并不影响菌株的泳动性。

|
| 图 6 野生株EGD-e和突变株ΔvirAB的泳动性 Figure 6 Motility of the wild-type strain EGD-e and the mutant ΔvirAB 注:A:37 ℃情况下野生株EGD-e的泳动性;B:37 ℃情况下突变株ΔvirAB的泳动性;C:25 ℃情况下野生株EGD-e的泳动性;D:25 ℃情况下突变株ΔvirAB的泳动性 Note: A: Motility of the wild-type strain EGD-e at 37 ℃; B: Motility of the mutant ΔvirAB at 37 ℃; C: Motility of the wild-type strain EGD-e at 25 ℃; D: Motility of the mutant ΔvirAB at 25 ℃ |
|
|
ABC转运蛋白家族在细菌中广泛存在,主要参与营养物质的摄入、代谢产物的排出等生理活动[5]。Lm已测序菌株EGD-e的基因组中存在上百个编码ABC转运蛋白的基因,只有少数基因的功能得到部分解析。Collins等[18]报道,由lmo2114/ lmo2115编码的ABC转运蛋白AnrAB介导EGD-e对包括头孢噻肟、头孢呋辛、氨苄西林和盘尼西林在内的β-内酰胺类抗生素、Nisin和杆菌肽的耐药。AnrAB属于肽7外排转运子(Peptide 7 Exporter,Pep7E)家族,其通透酶AnrB由10个跨膜结构域组成,其中跨膜域7和8之间存在一个大的胞外结构域,该结构域可能直接参与底物的结合[18]。本研究通过软件对VirB的跨膜结构进行了预测,结果发现VirB与AnrB的结构十分相似,也含有10个跨膜域,而且跨膜域7和8之间存在一个大的胞外结构域,据此我们推测VirAB可能也与EGD-e对抗菌药物的耐药有关。为了证实这一推测,本研究构建了菌株EGD-e的基因缺失突变株ΔvirAB,与野生株相比,该突变株对4种头孢类抗生素、Nisin和EB的MIC值下降,表明VirAB介导EGD-e对这几种抗菌药物的耐药性。尽管virAB的缺失没有影响菌株对BC、卡那霉素和四环素的MIC值,但是当培养基中分别添加亚致死浓度的BC、卡那霉素和四环素时,突变株均表现出不同程度的生长缺陷,这表明VirAB与EGD-e对BC、卡那霉素和四环素的耐药也有关。本研究还分别构建了virB和virA2个单基因缺失突变体,经检测发现这2株突变体对受试的4种头孢类抗生素、Nisin和EB的MIC值与ΔvirAB相同,表明virB和virA在介导EGD-e对这些抗菌药物的耐药中均起作用,两者缺一不可。
Zhu等[9]将转座子Tn917插入至Lm菌株4b G的lm.G_1771,得到插入失活突变株LM-49,进一步检测发现LM-49形成生物被膜的能力高于野生株;序列比对结果显示lm.G_1771编码的蛋白与EGD-e的VirB同源,而lm.G_1772编码的蛋白与VirA同源,因此Lm.G_1771/Lm.G_1772这个ABC转运蛋白负调控菌株4b G生物被膜的形成。本研究的结果显示,缺失virAB后菌株形成生物被膜的能力下降,表明VirAB可能参与正调控EGD-e生物被膜的形成,这与Zhu等[9]的研究结果不同。导致本研究与前人研究结果不同的原因可能有两点:(1)研究中所用的Lm菌株不同,Zhu等[9]以4b G为研究对象,该菌株为4b血清型且尚未进行全基因组测序,本研究使用已测序的Lm菌株EGD-e;(2)
构建突变体的方法不同,Zhu等[9]利用转座子Tn917将lm.G_1771基因插入失活,而本研究则通过缺失virAB构建敲除突变体。
Lm形成生物被膜的能力受多种因素的影响,其中泳动性有助于菌体在介质表面的运动,进而影响生物被膜的形成。因此,我们又对野生株EGD-e和突变株ΔvirAB的泳动能力进行检测,结果显示两者泳动能力无明显区别,表明VirAB与EGD-e的泳动能力无关。
本研究证实ABC转运蛋白VirAB介导EGD-e对头孢类抗生素、Nisin和EB耐药,同时该转运蛋白还部分参与EGD-e对BC、卡那霉素和四环素的耐药。此外,VirAB可能参与正调控EGD-e生物被膜的形成。
| [1] |
Jiang XB, Yu T, Liang Y, Ji SD, Guo XW, Ma JM, Zhou LJ. Efflux pump-mediated benzalkonium chloride resistance in Listeria monocytogenes isolated from retail food[J]. International Journal of Food Microbiology, 2016, 217: 141-145. DOI:10.1016/j.ijfoodmicro.2015.10.022 |
| [2] |
Filipello V, Gallina S, Amato E, Losio MN, Pontello M, Decastelli L, Lomonaco S. Diversity and persistence of Listeria monocytogenes within the Gorgonzola PDO production chain and comparison with clinical isolates from the same area[J]. International Journal of Food Microbiology, 2017, 245: 73-78. DOI:10.1016/j.ijfoodmicro.2017.01.012 |
| [3] |
Arslan F, Meynet E, Sunbul M, Sipahi OR, Kurtaran B, Kaya S, Inkaya AC, Pagliano P, Sengoz G, Batirel A, et al. The clinical features, diagnosis, treatment, and prognosis of neuroinvasive listeriosis: a multinational study[J]. European Journal of Clinical Microbiology & Infectious Diseases, 2015, 34(6): 1213-1221. |
| [4] |
Xu YM, Jiang XB, Yu T, Wang HL, Shi L. Role of efflux pump MdrL in tolerance to benzalkonium chloride in Listeria monocytogenes[J]. Microbiology China, 2018, 45(6): 1273-1282. (in Chinese) 徐雅梦, 姜晓冰, 于涛, 王海磊, 石磊. MdrL外排泵在单核细胞增生李斯特菌对苯扎氯铵耐受中的作用[J]. 微生物学通报, 2018, 45(6): 1273-1282. |
| [5] |
Zhu XN, Liu WB, Lametsch R, Aarestrup F, Shi CL, She QX, Shi XM, Knøchel S. Phenotypic, proteomic, and genomic characterization of a putative ABC-transporter permease involved in Listeria monocytogenes biofilm formation[J]. Foodborne Pathogens and Disease, 2011, 8(4): 495-501. DOI:10.1089/fpd.2010.0697 |
| [6] |
Liu YH, Ceruso M, Gunther IV NW, Pepe T, Cortesi LM, Fratamico P. Construction of Listeria monocytogenes mutants with in-frame deletions in putative ATP-Binding Cassette (ABC) transporters and analysis of their growth under stress conditions[J]. Journal of Microbial & Biochemical Technology, 2012, 4(7): 141-146. DOI:10.4172/1948-5948.1000085 |
| [7] |
Ceruso M, Fratamico P, Chirollo C, Taglialatela R, Cortesi ML, Pepe T. The capacity of Listeria monocytogenes mutants with in-frame deletions in putative ATP-binding cassette transporters to form biofilms and comparison with the wild type[J]. Italian Journal of Food Safety, 2014, 3(1): 1657. DOI:10.4081/ijfs.2014.1657 |
| [8] |
Grubaugh D, Regeimbal JM, Ghosh P, Zhou Y, Lauer P, Dubensky Jr TW, Higgins DE. The VirAB ABC transporter is required for VirR regulation of Listeria monocytogenes virulence and resistance to nisin[J]. Infection and Immunity, 2017, 86(3): e00901-17. |
| [9] |
Zhu XN, Long F, Chen YH, Knøchel S, She QX, Shi XM. A putative ABC transporter is involved in negative regulation of biofilm formation by Listeria monocytogenes[J]. Applied and Environmental Microbiology, 2008, 74(24): 7675-7683. DOI:10.1128/AEM.01229-08 |
| [10] |
Zhao C, Zhao JQ, Wang WY, Fan YN, Ma CP, Zhang DH, Lv Y. Expression of MLAA34-HSP70 fusion gene constructed by SOE-PCR[J]. Pakistan Journal of Pharmaceutical Sciences, 2017, 30(3): 1125-1127. |
| [11] |
Arnaud M, Chastanet A, Débarbouillé M. New vector for efficient allelic replacement in naturally nontransformable, low-GC-content, gram-positive bacteria[J]. Applied and Environmental Microbiology, 2004, 70(11): 6887-6891. DOI:10.1128/AEM.70.11.6887-6891.2004 |
| [12] |
Guo L, Chen GW, Xie MM, Liu WK, Ding CC, Wang SJ, Luo Q, Liu Q. Construction and biological activity of a strain of Listeria monocytogenes deleted for positive regulatory factor a gene (prfA)[J]. Food Science, 2017, 38(10): 12-17. (in Chinese) 郭亮, 陈国薇, 谢曼曼, 刘武康, 丁承超, 王淑娟, 罗勤, 刘箐. 单增李斯特菌prfA基因缺失菌株的构建及其生物学特性鉴定[J]. 食品科学, 2017, 38(10): 12-17. DOI:10.7506/spkx1002-6630-201710003 |
| [13] |
Xu YM. MdrL efflux pump mediates the resistance mechanism of Listeria monocytogenes to benzalkonium chloride[D]. Xinxiang: Master's Thesis of Henan Normal University, 2018 徐雅梦. MdrL外排泵介导Listeria monocytogenes对苯扎氯铵的耐受机制[D]. 新乡: 河南师范大学硕士学位论文, 2018 |
| [14] |
CLSI. Performance standards for antimicrobial susceptibility testing; twenty-first informational supplement[R]. Wayne, PA: Clinical and Laboratory Standards Institute, 2011
|
| [15] |
Yu T, Jiang XB, Zhang YG, Ji SD, Gao WJ, Shi L. Effect of benzalkonium chloride adaptation on sensitivity to antimicrobial agents and tolerance to environmental stresses in Listeria monocytogenes[J]. Frontiers in Microbiology, 2018, 9: 2906. DOI:10.3389/fmicb.2018.02906 |
| [16] |
Zhang PP, Liu Y, Fang C, Yu Y, Chen JS, Fang WH. Comparison of growth models of Listeria monocytogenes in chilled pork[J]. Acta Microbiologica Sinica, 2011, 51(12): 1625-1631. (in Chinese) 张培培, 刘媛, 方春, 俞盈, 陈健舜, 方维焕. 单增李斯特菌在冷鲜猪肉中的生长预测模型比较[J]. 微生物学报, 2011, 51(12): 1625-1631. |
| [17] |
Djordjevic D, Wiedmann M, McLandsborough LA. Microtiter plate assay for assessment of Listeria monocytogenes biofilm formation[J]. Applied and Environmental Microbiology, 2002, 68(6): 2950-2958. DOI:10.1128/AEM.68.6.2950-2958.2002 |
| [18] |
Collins B, Curtis N, Cotter PD, Hill C, Ross RP. The ABC transporter AnrAB contributes to the innate resistance of Listeria monocytogenes to nisin, bacitracin, and various β-lactam antibiotics[J]. Antimicrobial Agents and Chemotherapy, 2010, 54(10): 4416-4423. DOI:10.1128/AAC.00503-10 |
 2021, Vol. 48
2021, Vol. 48