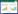扩展功能
文章信息
- 李雪燕, 贾天军
- LI Xueyan, JIA Tianjun
- 沙眼衣原体包涵体膜蛋白CT225与宿主波形蛋白存在相互作用
- Chlamydia trachomatis CT225 interacting with vimentin from HeLa cell
- 微生物学通报, 2021, 48(2): 462-470
- Microbiology China, 2021, 48(2): 462-470
- DOI: 10.13344/j.microbiol.china.200163
-
文章历史
- 收稿日期: 2020-02-28
- 接受日期: 2020-05-21
- 网络首发日期: 2020-07-30
沙眼衣原体(Chlamydia trachomatis,CT)是一种专性胞内寄生的病原微生物,主要引起眼部和生殖器感染,具有特殊的双相发育周期[1]。其包涵体作为衣原体复制和生物合成的场所,不仅保护病原体免遭宿主免疫系统清除,而且衣原体必须通过包涵体从宿主细胞内摄取营养和排泄代谢产物[2]。包涵体膜蛋白(Inclusion Membrane Proteins,Incs)是一类定位于衣原体包涵体膜上的含独特双叶片状疏水性基序结构的衣原体蛋白,大约7%–10%的基因组编码包涵体膜蛋白[3]。可见包涵体膜蛋白在衣原体的生命活动中发挥重要作用,比如参与包涵体膜的运输、影响宿主细胞骨架构建、参与宿主泛素化及增强衣原体抗凋亡活性等[4]。然而许多Incs的生物学功能仍需进一步研究。CT225是已经证实的包涵体膜蛋白[1],未见其生物学功能的研究报道。本实验采用谷胱甘肽巯基转移酶(Glutathione S-Transferase,GST)融合蛋白沉降实验及质谱技术对与其相互作用的蛋白质进行筛选,为进一步研究包涵体膜蛋白的生物学特性及沙眼衣原体的致病机制奠定基础。
1 材料与方法 1.1 主要试剂和仪器pGEX-6p2-CT225重组质粒,钟光明教授惠赠;大肠埃希菌、XL1-Blue感受态细菌和HeLa细胞,本实验室保存;Pierce GST Protein Interaction Pull-Down Kit,Thermo公司;质粒提取试剂盒,Omega公司;小鼠抗Flag抗体,Sigma-Aldrich公司;兔抗Myc抗体、Cy3标记的山羊抗鼠IgG抗体、FITC标记的山羊抗兔IgG抗体、HRP标记羊抗兔IgG和羊抗鼠IgG (HRP标记),Bioss公司;小鼠抗CT225抗体,本实验室保存;兔抗Vimentin抗体、DAPI染色液(即用型),上海碧云天生物技术有限公司。
超声波细胞破碎机,宁波新芝生物科技股份有限公司;Omega Lum G化学发光凝胶成像系统,Aplegen公司;毛细管高效液相色谱仪和电喷雾-组合型离子阱Orbitrap质谱仪,Thermo公司。
1.2 CT225基因编码蛋白的特性分析运用EXPSY Proteomics分析系统(https://web.expasy.org/protparam/)分析蛋白质的分子式、氨基酸组成等基本理化特性。采用PRABIGERLAND (https://npsa-prabi.ibcp.fr/cgibin/secpredsopma.pl)在线软件分析二级结构。使用软件TMNMM Server V2.0 (http://www.cbs.dtu.dk/services/TMHMM-2.0)分析跨膜区域。运用SignalP (http://www.cbs.dtu.dk/services/SignalP/)预测假设蛋白信号肽。运用EXPASY Proteomics软件模拟构建同源结构(三级结构模拟)。利用ProtScale (http://web.expasy.org/protscale)分析该蛋白的亲疏水性。使用PHP蛋白功能预测数据库进行功能预测(http://kiharalab.org/web/index.php)。
1.3 CT225互作分子的初筛 1.3.1 诱饵GST-CT225蛋白的制备将pGEX-6p2-CT225重组质粒的甘油菌接入LB液体培养基中,37 ℃、200 r/min振荡培养16 h,加入IPTG (100 mg/mL) 10 μL诱导表达。收集菌液于4 000 r/min离心30 min保留菌体沉淀,加1 mL裂解液重悬。使用超声波细胞破碎机破裂菌体(功率100 W,超声3 s,间隔4 s,共9 min),4 000 r/min离心15 min后留取上清液。部分上清在4 ℃条件下与谷胱甘肽共轭琼脂糖微珠(GST微珠)吸附2.5 h,使用PBS缓冲液冲洗3次,12 000 r/min离心2 min,弃上清,加等体积上样缓冲液,95 ℃金属浴5 min,进行SDS-PAGE电泳。
1.3.2 固定诱饵蛋白制备洗涤溶液,按1:1比例将TBS缓冲液与Pull-Down裂解缓冲液混合。吸取50 μL的GST微珠浆液加入到Pierce旋柱中,盖上柱的底端向离心柱中加入400 μL洗涤液,盖上柱的顶盖倒转几次,以平衡混匀GST微珠,取下底端盖子,将离心柱置于收集管中,1 250×g离心1 min,重复洗涤5次。将准备的GST-CT225融合蛋白700 μL加入到旋柱中,更换柱子的顶盖,4 ℃孵育2 h并且在旋转平台上轻轻摇动。移除旋柱底盖并置于收集管中,1 250×g离心1 min,弃上清,加入400 μL的洗涤液,吹吸重悬后离心,1 250×g离心1 min,共重复洗涤5次。取部分GST微珠加等体积上样缓冲液,95 ℃金属浴5 min,进行SDS-PAGE电泳。
1.3.3 HeLa细胞总蛋白的制备将培养的细胞用D-Hanks液冲洗,胰酶消化后移至离心管,500×g离心5 min后弃去上清。加入1 mL TBS缓冲液,将细胞重悬后再次离心弃上清。向细胞中加入1 mL Pull-Down裂解液与蛋白酶抑制剂,立即倒转至完全混合,冰上放置30 min,10 min翻转一次管。12 000×g离心5 min取上清至EP管中。
1.3.4 GST-CT225对HeLa细胞互作蛋白的捕获取下装有GST融合蛋白的旋柱顶盖,加入HeLa细胞裂解液上清,4 ℃孵育2 h,并在旋转平台上轻轻摇摆。取下旋柱底盖,置于收集管中,1 250×g离心1 min,弃上清,加入400 μL洗涤液反复吹吸后1 250×g离心1 min,重复5次。取部分GST微珠至EP管中,加等体积上样缓冲液,95 ℃金属浴5 min,进行SDS-PAGE电泳。
1.3.5 质谱分析(1) 前处理
胰蛋白酶(Trypsin)酶解,于GST下拉/沉降实验(Pull-Down Test)得到的GST微珠中加入50 mmol/L NH4HCO3溶液至200 μL,于4 ℃、10 000 r/min离心5 min,弃去上清,并重复操作1次。加入50 mmol/L NH4HCO3溶液100 μL,加入DTT溶液使其终浓度为10 mmol/L,于37 ℃水浴中还原4 h。加入IAA溶液使其终浓度为50 mmol/L,避光反应40 min后,于4 ℃、10 000 r/min离心5 min,弃去上清。加入50 mmol/L NH4HCO3溶液200 μL,于4 ℃、10 000 r/min离心5 min,弃去上清。加入100 μL 50 mmol/L NH4HCO3溶液,按照胰蛋白酶与底物质量比为1:100加入胰蛋白酶,37 ℃酶切4 h,继续按质量比1:100加入胰酶,37 ℃酶切反应过夜(16 h)。酶切后肽段使用自填脱盐柱脱盐,于45 ℃真空离心浓缩仪中挥干溶剂。
(2) 毛细管液相色谱条件
预柱:300 μm i.d.×5 mm,Packed with Acclaim PepMap RPLC C18,5 μm,10 nm。分析柱:75 μm i.d.×150 mm,Packed with Acclaim PepMap RPLC C18,3 μm,10 nm。流动相A:0.1%甲酸,2% ACN;流动相B:0.1%甲酸,80% ACN;流速:300 nL/min;液相色谱梯度:56 min。
(3) 质谱条件
一级质谱参数:Resolution:70 000;AGCtarget:3e6;MaximumIT:40 ms;Scanrange:350-1 800 m/z。二级质谱参数:Resolution:75 000;AGCtarget:1e5;MaximumIT:60 ms;TopN:20;NCE/ steppedNCE:27。
1.3.6 数据库检索质谱采集数据生成Raw格式文件,使用Xcalibur软件打开,可以看到总离子流色谱图。质谱原始文件使用Maxquant (1.5.6.5)分别检索Uniprot-Homo Sapiens (Human)数据库。在得分最高的几个分子中,结合分子功能的分析,选择对得分较高的VIM蛋白进行初步验证。
1.4 CT225互作分子的鉴定 1.4.1 构建真核表达载体以HeLa细胞RNA逆转录的cDNA为模板,与pcDNA3.1(+)His/Myc和pCMV-N-Flag载体进行重组,依据PCR引物设计原则设计特异性引物(表 1),通过PCR扩增VIM、CT225目的片段。PCR反应体系(100 μL):模板/CT基因组1-20 ng,5×Prime STAR Buffer 20 μL,dNTP Mixture (2.5 mmol/L) 8 μL,特异性上、下游引物(10 μmol/L)各4 μL,高保真DNA聚合酶(5 U/μL) 1 μL,ddH2O补齐100 μL。PCR反应条件:98 ℃ 5 min;98 ℃ 10 s,55 ℃ 30 s,72 ℃ 10 min,35个循环;72 ℃ 10 min。使用限制性内切酶酶切、T4 DNA连接酶16 ℃连接过夜。连接好的质粒采用热休克法转化入大肠埃希菌XL1-Blue感受态细胞,37 ℃温箱内LB固体培养基长出菌落后筛选阳性菌落。用大肠埃希菌对质粒进行扩增和纯化,大肠埃希菌在LB液体培养基中37 ℃、200 r/min培养16 h,并配以适当的氨苄青霉素。用质粒提取试剂盒进行质粒提取,送华大基因公司进行测序。
| Primers | Sequence (5′→3′) |
| VIM上游引物VIM upstream primer | CCG-GAATTCGG-ATGTCCACCAGGTCCGTGT (Eco R I) |
| VIM下游引物VIM downstream primer | TTTTCCTTTT-GCGGCCGC-TTATTCAAGGTCATCGTGATGC (Not I) |
| CT225上游引物CT225 upstream primer | CGC-GGATCC-ATGGCTAACAACTCCTTTATTCACC (Bam H I) |
| CT225下游引物CT225 downstream primer | TTTTCCTTTT-GCGGCCGC-TTAATCCCACCCATGAAATTTAGC (Not I) |
HeLa细胞培养在六孔细胞培养板中,待细胞密度达到80%时进行转染。转染36 h后收集六孔板中的HeLa细胞。裂解细胞后13 000 r/min离心10 min得到蛋白上清液,将c-Myc探针抗体共轭琼脂糖微珠(c-Myc微珠)加入到蛋白上清液中翻转吸附2.5 h。1×PBST缓冲液洗涤3次,去除杂蛋白。加入等体积的上样缓冲液,100 ℃下金属浴中孵育10 min。通过Western Blot实验进行验证。
设置实验组:pcDNA3.1(+)His/Myc-CT225与pCMV-N-Flag-VIM。设置对照组:pcDNA3.1(+)His/ Myc与pCMV-N-Flag-VIM;pcDNA3.1(+)His/Myc- CT225;pCMV-N-Flag-VIM。
1.4.3 GST下拉/沉降实验转染pCMV-N-Flag-VIM质粒至HeLa细胞。转染后36 h表达蛋白,将HeLa细胞裂解作为猎物蛋白。使用GST Pull-Down Kit进行实验,将1.3.1部分表达的蛋白GST-CT225固定在GST-微珠上作为诱饵蛋白,孵育诱饵蛋白2 h,用1×PBST洗涤3次后孵育猎物蛋白2 h,Western Blot方法检测。
设置实验组:GST-CT225与pCMV-N-Flag- VIM。同时设置对照组:GST-CT225蛋白组;GST-微珠;VIM猎物蛋白。
1.4.4 亚细胞共定位技术无菌盖玻片放至24孔板,培养HeLa细胞至24孔板,细胞密度达到80%。使用转染试剂将质粒共转染至24孔板中的HeLa细胞,转染36 h后,在室温下用4%丙酮固定15 min。在PBS中洗涤细胞并用0.1% Triton X-100渗透10 min,室温下用10%山羊血清封闭2 h。在PBS中洗涤细胞后加入一抗抗体避光孵育过夜。在PBS中再次洗涤,并用适当浓度的荧光二抗孵育2 h。在PBS中再次洗涤后,使用DAPI染液对细胞核进行染色20 min。玻片滴加抗荧光淬灭剂后被安装在玻璃载玻片上,激光共聚焦显微镜下进行多层面观察拍照,图像由共焦显微镜获取,并使用Zeiss LSM图像浏览器进行处理。
设置实验组:pcDNA3.1(+)1His/Myc-CT225与pCMV-N-Flag-VIM,一抗为兔抗Myc抗体、鼠抗Flag抗体,二抗为FITC标记的羊抗兔IgG抗体、Cy3标记的羊抗鼠IgG抗体。
设置对照组:pcDNA3.1(+)His/Myc与pCMV- N-Flag-VIM,抗体同实验组;pcDNA3.1(+)His/ Myc-CT225与pCMV-N-Flag,抗体同实验组;pcDNA3.1(+)His/Myc-CT225,一抗为鼠抗CT225抗体、兔抗VIM抗体,二抗为FITC标记的羊抗兔IgG抗体、Cy3标记的羊抗鼠IgG抗体。
2 结果与分析 2.1 生物信息学特征通过多种软件对CT225基因编码蛋白进行生物信息学分析,结果显示该蛋白由369个氨基酸组成,分子量为13.31 kD,理论pI为5.33,成分为C1148H1929N369O500S52,不稳定指数为34.24,说明该蛋白质较稳定;TMH数量为2,说明属于跨膜蛋白;D值小于0.450 (阈值),说明无信号肽序列;氨基酸残基数绝大多为亲水性残基,属可溶性蛋白可能性比较大;二级结构为α-螺旋58个(47.54%)、无规则卷曲36个(29.51%)、延伸链28个(22.95%);三级结构建模模型与蛋白数据库比对,发现与5xau.2.B相似性最高,为29.27%;预测蛋白参与的生物学过程为氢转运,分子功能为运输、转运,细胞定位于细胞膜上。
2.2 GST-CT225蛋白的制备通过蛋白表达与亲和层析纯化后,经SDS-PAGE电泳和考马斯亮蓝染色,得到大小为39 kD的诱饵蛋白,与预期一致,如图 1所示。
|
| 图 1 纯化蛋白的SDS-PAGE电泳分析 Figure 1 SDS-PAGE electrophoresis analysis of purified proteins 注:1:CT225纯化蛋白5 µL;2:CT225纯化蛋白10 µL;M:蛋白分子量Marker Note: 1. CT225 purified protein 5 µL; 2: CT225 purified protein 10 µL; M: Protein Marker |
|
|
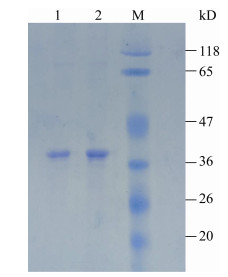
采集质谱结果原始数据可生成总离子流色谱图(图 2)。质谱结果显示得分最高的CT225的互相作用配体为:Alpha-actinin-4、Keratin、Vimentin、Tubulin等(表 2)。
|
| 图 2 总离子流色谱图 Figure 2 Total ions chromatogram |
|
|
| 排名 Ranking |
名称 Name |
得分 Score |
峰度 Kurtosis |
| 1 | Alpha-actinin-4 | 323.31 | 69 364 000 |
| 2 | Keratin, type Ⅱ cytoskeletal 1 | 323.31 | 486 870 000 |
| 3 | Keratin, type Ⅰ cytoskeletal 9 | 323.31 | 249 980 000 |
| 4 | Keratin, type Ⅰ cytoskeletal 10 | 323.31 | 117 700 000 |
| 5 | Vimentin | 302.04 | 141 130 000 |
| 6 | Plectin | 253.32 | 10 942 000 |
| 7 | Keratin, type Ⅰ cuticular Ha3-Ⅱ | 215.62 | 20 774 000 |
| 8 | Actin, cytoplasmic 2; Actin, cytoplasmic 2, N-terminally processed | 197.36 | 371 660 000 |
| 9 | Carbonyl reductase [NADPH] 1 | 195.66 | 174 880 000 |
| 10 | Tubulin beta chain; Tubulin beta-2B chain; Tubulin beta-2A chain | 174.04 | 26 250 000 |
质谱检索结果中Alpha-Actinin-4分值虽高,但峰度值不高(表 2)。Keratin在质谱结果中一般认为是非特异性结合的分子,选择对得分较高的VIM蛋白进行初步验证。
2.4 重构真核质粒构建的真核质粒pcDNA3.1(+)His/Myc-CT225与pCMV-N-Flag-VIM测序结果同数据库序列进行对比序列一致,证明重组质粒成功。
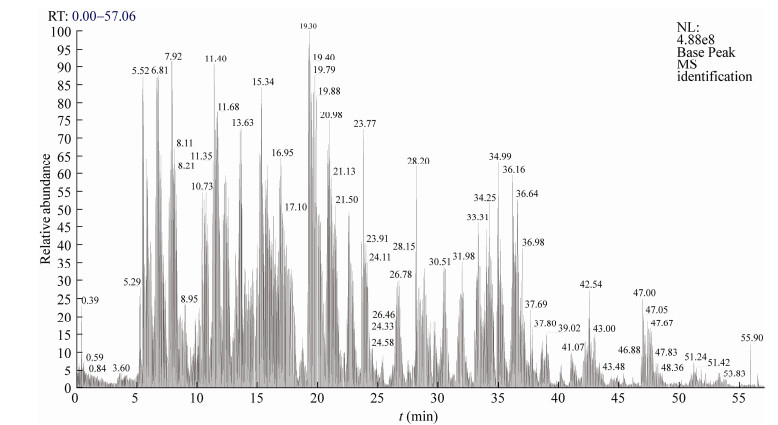
2.5 免疫共沉淀技术证实CT225与VIM相互作用为了验证CT225与VIM在细胞内的相互作用,在免疫共沉淀(Co-Immunoprecipitation,CO-IP)试验中,用2个质粒组合转染HeLa细胞,将转染细胞裂解,经免疫吸附、Western Blot技术进行蛋白检测后分别在13 kD和53 kD处出现与预期相符的目的条带。
为证明VIM与Myc-CT225特异性结合而不是单独与Myc标签结合,设置对照组pcDNA3.1(+)His/Myc与pCMV-N-Flag-VIM,共转染结果无条带。pcDNA3.1(+)His/Myc-CT225质粒转染组在13 kD处有条带。只转染pCMV- N-Flag-VIM质粒且不经免疫沉淀处理的VIM靶蛋白对照条带大小为53 kD,与预期一致,在这些条件下可以检测到CT225和VIM可以在细胞内相互作用(图 3)。
|
| 图 3 免疫共沉淀技术验证CT225与VIM相互作用 Figure 3 Co-immunoprecipitation assay to detect the interaction between CT225 and VIM 注:1:pcDNA3.1(+)His/Myc-CT225质粒转染组(13 kD);2:pcDNA3.1(+)His/Myc-CT225与pCMV-N-Flag-VIM质粒共转组(13 kD和53 kD);3:pcDNA3.1(+)His/Myc与pCMV-N-Flag-VIM质粒共转染组(无条带);4:VIM对照组(53 kD) Note: 1: pcDNA3.1(+)His/Myc-CT225 plasmid transfection group (13 kD); 2: pcDNA3.1(+)His/Myc-CT225 and pCMV-N-Flag-VIM plasmids co-transfection group (13 kD and 53 kD); 3: pcDNA3.1(+)His/Myc and pCMV-N-Flag-VIM plasmids co-transfection group (no stripe); 4: VIM control (53 kD) |
|
|
为检测CT225与VIM在细胞外的直接作用,将Flag-VIM质粒单独转染至HeLa细胞过表达蛋白,与2.2.1部分大肠杆菌表达的GST- CT225融合蛋白共同孵育,经Western Blot检测,在39 kD和53 kD位置有2条带,与预期一致。仅孵育GST-CT225蛋白在39 kD位置获得目的条带,仅孵育VIM蛋白在53 kD位置获得目的条带。阴性对照组上样GST微珠结果无条带,与预期相符(图 4)。
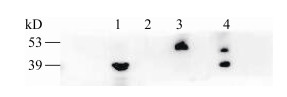
|
| 图 4 GST pull-down实验验证CT225与VIM相互作用 Figure 4 GST pull-down test to confirm the interaction between CT225 and VIM 注:1:诱饵蛋白GST-CT225 (39 kD);2:GST微珠对照组(无条带);3:猎物蛋白pCMV-N-Flag-VIM转染组(53 kD);4:GST-CT225与pCMV-N-Flag-VIM实验组(39 kD和53 kD) Note: 1: Purified bait protein GST-CT225 group (39 kD); 2: Glutathione sepharose agarose control group (no stripe); 3: Prey protein VIM group (53 kD); 4: Bait protein CT225 co-incubated with prey protein VIM group (39 kD and 53 kD) |
|
|
为进一步对转染入细胞的外源性蛋白CT225与VIM间作用进行鉴定,检测转入蛋白CT225与VIM间的共定位,将HeLa细胞转染24 h后,用1 mL丙酮固定细胞,用抗Flag和抗Myc抗体免疫标记,采用三色荧光标记进行染色,使用FITC (黄绿色)对CT225蛋白进行染色,Cy3 (橙红色)对VIM蛋白进行染色,DAPI (蓝色)对DNA染色。
实验组即pcDNA3.1(+)His/Myc-CT225与pCMV-N-Flag-VIM两种质粒共转染组,Myc- CT225蛋白显示绿色荧光,Flag-VIM蛋白显示红色荧光,两者在3个层面均重叠呈现黄色荧光(图 5A)。设置对照组pcDNA3.1(+)His/Myc与pCMV-N-Flag-VIM质粒共转,仅有Flag-VIM蛋白是红色荧光(图 5B);pcDNA3.1(+)His/Myc-CT225与pCMV-N-Flag质粒共转,仅有Myc-CT225蛋白显示绿色荧光(图 5C)。
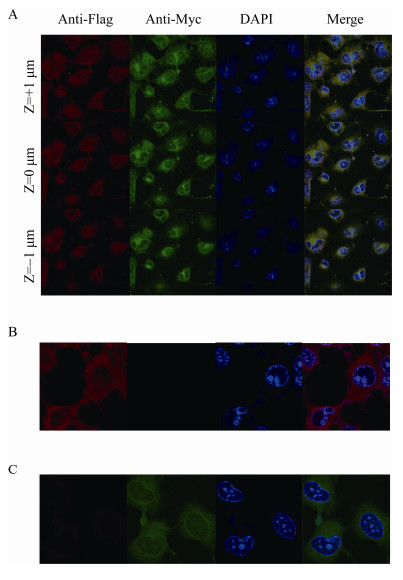
|
| 图 5 间接免疫荧光实验验证CT225和VIM的胞内定位 Figure 5 The indirect immunofluorescence assay to confirm the cellular localization between CT225 and VIM in HeLa cells 注:A:HeLa细胞与编码Myc-CT225和Flag-VIM的重组质粒共转染,分别使用抗Myc和抗Flag抗体检测了蛋白质CT225 (绿色)和VIM (红色);B:用编码c-Myc和Flag-VIM的质粒共转染HeLa细胞,分别使用抗Myc和抗Flag抗体检测了c-Myc蛋白(绿色)和VIM (红色);C:用编码Myc-CT225和Flag的质粒共转染HeLa细胞,分别使用抗Myc和抗Flag抗体检测了蛋白CT225 (绿色)和Flag (红色) Note: A: HeLa cells were co-transfected with recombinant plasmids encoding Myc-CT225 and Flag-VIM. The protein CT225 (green) and VIM (red) were detected with anti-Myc and anti-Flag antibodies, respectively; B: HeLa cells were co-transfected with plasmids encoding c-Myc and Flag-VIM. The protein c-Myc (green) and VIM (red) were detected with anti-Myc and anti-Flag antibodies, respectively; C: HeLa cells were co-transfected with plasmids encoding Myc-CT225 and Flag. The protein CT225 (green) and Flag (red) were detected with anti-Myc and anti-Flag antibodies, respectively |
|
|
内源性蛋白相互作用实验组仅转染pcDNA3.1(+)His/Myc-CT225质粒。结果显示Myc-CT225蛋白经鼠抗CT225标记后出现红色荧光,VIM蛋白经兔抗VIM抗体标记后出现绿色荧光,二者重叠呈现黄色荧光,证明CT225可与内源性VIM蛋白相互作用(图 6)。总的来说,这些实验支持沙眼衣原体包涵体膜蛋白CT225可以与波形蛋白VIM相互作用。
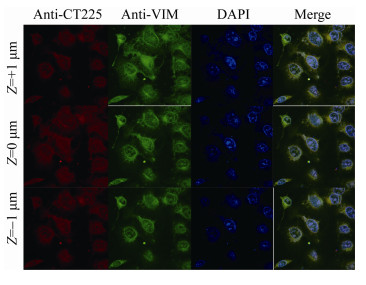
|
| 图 6 间接免疫荧光法确定HeLa细胞中CT225和VIM之间的相互作用 Figure 6 The indirect immunofluorescence assay to confirm the interaction between CT225 and VIM in HeLa cells 注:用编码Myc-CT225的重组质粒转染HeLa,分别使用鼠抗CT225和兔抗VIM抗体检测到CT225 (红色)和VIM (绿色) Note: HeLa were transfected with recombinant plasmids encoding Myc-CT225; The CT225 (red) and VIM (green) were detected with anti-CT225 and anti-VIM antibodies respectively |
|
|
沙眼衣原体是一种专性细胞内致病菌,主要引起眼盲和生殖系统感染。沙眼衣原体感染时宿主与病原体相互作用的组织位于包涵体膜上。在这个界面上,沙眼衣原体建立和维持着它们的细胞内生态位[5]。沙眼衣原体在细胞外和包涵体内都使用Ⅲ型分泌系统将几种效应蛋白传递给宿主细胞的细胞质。这些效应蛋白的很大一部分即包涵体膜蛋白,包涵体膜蛋白暴露在宿主细胞胞浆中,其特殊的双重疏水结构域介导插入包涵体膜,调节包涵体与宿主之间的交互作用[6-7]。
CT223-CT229基因连锁图谱表明其属于同一个基因簇[8]。CT223/IPAM结合宿主中心体蛋白破坏微管功能。CT223与中心体蛋白绑定,在包涵体表面积极地重组组织微管,这导致形成微管上部结构,该上部结构对于保持膜完整性是必需的[9-10]。CT224参与逆向囊泡转运,CT226蛋白质涉及级联途径的信号转导互作于胞质DNA/RNA传感器,调控宿主免疫系统[11];CT228通过与肌球蛋白磷酸酶靶亚基1 (MYPT 1)的相互作用介导衣原体挤压,调节原生小体挤出包涵体[12-14];CT229与GTP酶的Rab分子互作,调控内质体转运,调节CT胞内运输,抑制宿主细胞过早死亡[15-16]。其他涉及多核的效应因子包括CT223、CT224、CT225、CT166,这些蛋白的异位表达阻碍了细胞分裂,导致多核细胞产生,多核提高了高尔基体的含量,使衣原体容易获取高尔基体的脂质[17]。
然而,大多数Incs的功能还有待研究,有些似乎起到结构性作用。某些Incs (IncC、CT101、CT222、CT223、CT224、CT228和CT850)在衣原体包涵体膜的特殊微区中富集;这些微区富含活性Src家族激酶和胆固醇,被认为是与细胞骨架相互作用的平台[18-19]。
CT225蛋白的生物信息学分析显示其定位于膜上,预测其功能主要为运输、转运。VIM附在细胞核、内质网及线粒体的旁边或末端,作为Ⅲ型中间丝蛋白,参与形成细胞骨架的结构框架,对于维持细胞的正常形态和功能、支撑及稳固胞内的细胞器具有重要意义;VIM除维持细胞完整性、稳定细胞骨架外,还参与细胞的附着和迁移、细胞信号转导、传送低密度脂蛋白衍生的胆固醇至酯化位点等[20-23]。
可以推测膜蛋白CT225结合宿主VIM蛋白主要完成运输功能,可运输能量、脂质,参与调节细胞凋亡通路等,这可能因为沙眼衣原体在宿主细胞获取营养、逃避免疫、对细胞凋亡的调控等生物学功能与运输有密切联系。
本实验通过免疫共沉淀、间接免疫荧光和质谱分析等实验证实了CT225与VIM存在相互作用,但相关机制还需要进一步研究。
| [1] |
Li ZY, Chen CQ, Chen D, Wu YM, Zhong YM, Zhong GM. Characterization of fifty putative inclusion membrane proteins encoded in the Chlamydia trachomatis genome[J]. Infection and Immunity, 2008, 76(6): 2746-2757. DOI:10.1128/IAI.00010-08 |
| [2] |
Weber MM, Bauler LD, Lam J, Hackstadt T. Expression and localization of predicted inclusion membrane proteins in Chlamydia trachomatis[J]. Infection and Immunity, 2015, 83(12): 4710-4718. DOI:10.1128/IAI.01075-15 |
| [3] |
Dehoux P, Flores R, Dauga C, Zhong GM, Subtil A. Multi-genome identification and characterization of chlamydiae-specific type Ⅲ secretion substrates: the Inc proteins[J]. BMC Genomics, 2011, 12(1): 109. DOI:10.1186/1471-2164-12-109 |
| [4] |
Omsland A, Sixt BS, Horn M, Hackstadt T. Chlamydial metabolism revisited: interspecies metabolic variability and developmental stage-specific physiologic activities[J]. FEMS Microbiology Reviews, 2014, 38(4): 779-801. DOI:10.1111/1574-6976.12059 |
| [5] |
Bachmann NL, Polkinghorne A, Timms P. Chlamydia genomics: providing novel insights into chlamydial biology[J]. Trends in Microbiology, 2014, 22(8): 464-472. DOI:10.1016/j.tim.2014.04.013 |
| [6] |
Fields KA, Mead DJ, Dooley CA, Hackstadt T. Chlamydia trachomatis type Ⅲ secretion: evidence for a functional apparatus during early-cycle development[J]. Molecular Microbiology, 2003, 48(3): 671-683. DOI:10.1046/j.1365-2958.2003.03462.x |
| [7] |
Muschiol S, Boncompain G, Vromman F, Dehoux P, Normark S, Henriques-Normark B, Subtil A. Identification of a family of effectors secreted by the type Ⅲ secretion system that are conserved in pathogenic Chlamydiae[J]. Infection and Immunity, 2011, 79(2): 571-580. DOI:10.1128/IAI.00825-10 |
| [8] |
Guo Y, Chen CQ. Progress on Chlamydia trachomatis inclusion membrane proteins[J]. Progress in Microbiology and Immunology, 2018, 46(5): 104-109. (in Chinese) 郭芫, 陈超群. 沙眼衣原体包涵体膜蛋白的研究进展[J]. 微生物学免疫学进展, 2018, 46(5): 104-109. |
| [9] |
Dumoux M, Menny A, Delacour D, Hayward RD. A Chlamydia effector recruits CEP170 to reprogram host microtubule organization[J]. Journal of Cell Science, 2015, 128(18): 3420-3434. DOI:10.1242/jcs.169318 |
| [10] |
Wesolowski J, Weber MM, Nawrotek A, Dooley CA, Calderon M, St Croix CM, Hackstadt T, Cherfils J, Paumet F. Chlamydia hijacks ARF GTPases to coordinate microtubule posttranslational modifications and golgi complex positioning[J]. mBio, 2017, 8(3): e02280-16. |
| [11] |
Mirrashidi KM, Elwell CA, Verschueren E, Johnson JR, Frando A, Von Dollen J, Rosenberg O, Gulbahce N, Jang G, Johnson T, et al. Global mapping of the Inc-human interactome reveals that retromer restricts Chlamydia infection[J]. Cell Host and Microbe, 2015, 18(1): 109-121. DOI:10.1016/j.chom.2015.06.004 |
| [12] |
Lutter EI, Barger AC, Nair V, Hackstadt T. Chlamydia trachomatis inclusion membrane protein CT228 recruits elements of the myosin phosphatase pathway to regulate release mechanisms[J]. Cell Reports, 2013, 3(6): 1921-1931. DOI:10.1016/j.celrep.2013.04.027 |
| [13] |
Shaw JH, Key CE, Snider TA, Sah P, Shaw EI, Fisher DJ, Lutter EI. Genetic inactivation of Chlamydia trachomatis inclusion membrane protein CT228 alters MYPT1 recruitment, extrusion production, and longevity of infection[J]. Frontiers in Cellular and Infection Microbiology, 2018, 8: 415. DOI:10.3389/fcimb.2018.00415 |
| [14] |
Belland RJ, Zhong GM, Crane DD, Hogan D, Sturdevant D, Sharma J, Beatty WL, Caldwell HD. Genomic transcriptional profiling of the developmental cycle of Chlamydia trachomatis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(14): 8478-8483. DOI:10.1073/pnas.1331135100 |
| [15] |
Rzomp KA, Moorhead AR, Scidmore MA. The GTPase Rab4 interacts with Chlamydia trachomatis inclusion membrane protein CT229[J]. Infection and Immunity, 2006, 74(9): 5362-5373. DOI:10.1128/IAI.00539-06 |
| [16] |
Slxt BS, Bastidas RJ, Finethy R, Baxter RM, Carpenter VK, Kroemer G, Coers J, Valdivia RH. The Chlamydia trachomatis inclusion membrane protein CpoS counteracts STING-mediated cellular surveillance and suicide programs[J]. Cell Host and Microbe, 2017, 21(1): 113-121. DOI:10.1016/j.chom.2016.12.002 |
| [17] |
Bothe M, Dutow P, Pich A, Genth H, Klos A. DXD motif-dependent and-independent effects of the Chlamydia trachomatis cytotoxin CT166[J]. Toxins, 2015, 7(2): 621-637. DOI:10.3390/toxins7020621 |
| [18] |
Mital J, Miller NJ, Fischer ER, Hackstadt T. Specific chlamydial inclusion membrane proteins associate with active Src family kinases in microdomains that interact with the host microtubule network[J]. Cellular Microbiology, 2010, 12(9): 1235-1249. DOI:10.1111/j.1462-5822.2010.01465.x |
| [19] |
Mital J, Miller NJ, Dorward DW, Dooley CA, Hackstadt T, Kelly KA. Role for chlamydial inclusion membrane proteins in inclusion membrane structure and biogenesis[J]. PLoS One, 2013, 8(5): e63426. DOI:10.1371/journal.pone.0063426 |
| [20] |
Fletcher DA, Mullins RD. Cell mechanics and the cytoskeleton[J]. Nature, 2010, 463(7280): 485-492. DOI:10.1038/nature08908 |
| [21] |
Ivaska J, Pallari HM, Nevo J, Eriksson JE. Novel functions of vimentin in cell adhesion, migration, and signaling[J]. Experimental Cell Research, 2007, 313(10): 2050-2062. DOI:10.1016/j.yexcr.2007.03.040 |
| [22] |
Chernyatina AA, Nicolet S, Aebi U, Herrmann H, Strelkov SV. Atomic structure of the vimentin central α-helical domain and its implications for intermediate filament assembly[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(34): 13620-13625. DOI:10.1073/pnas.1206836109 |
| [23] |
Mendez MG, Kojima S, Goldman RD. Vimentin induces changes in cell shape, motility, and adhesion during the epithelial to mesenchymal transition[J]. The FASEB Journal, 2010, 24(6): 1838-1851. DOI:10.1096/fj.09-151639 |
 2021, Vol. 48
2021, Vol. 48