扩展功能
文章信息
- 王海龙, 王嘉良, 邓子新, 汪志军, 梁晶丹
- WANG Hailong, WANG Jialiang, DENG Zixin, WANG Zhijun, LIANG Jingdan
- 人源脂肪酸合酶在酿酒酵母中的表达纯化和结构的初步分析
- Expression, purification and preliminary structural analysis of human fatty acid synthase in Saccharomyces cerevisiae
- 微生物学通报, 2021, 48(11): 3931-3938
- Microbiology China, 2021, 48(11): 3931-3938
- DOI: 10.13344/j.microbiol.china.210214
-
文章历史
- 收稿日期: 2021-03-03
- 接受日期: 2021-04-25
- 网络首发日期: 2021-05-08
聚酮类化合物(Polyketides,PK)是细菌、真菌等微生物分泌的一种次级代谢产物,在医学领域有重要应用。用作抗生素的普卡霉素(Mithramycin)、抗霉素(Resistomycin),用于降低胆固醇的洛伐他丁(Lovastatin),起抗氧化作用的白藜芦醇(Resveratrol)等都是聚酮类化合物广泛应用的代表[1]。聚酮类化合物在生物体内主要是通过聚酮合酶(Polyketide Synthase,PKS)这一巨型酶催化合成。聚酮合酶目前主要分为三类,即Ⅰ型、Ⅱ型和Ⅲ型聚酮合酶。Ⅰ型聚酮合酶主要合成大环内酯类化合物,Ⅱ型聚酮合酶主要合成芳香类化合物,Ⅲ型聚酮合酶主要合成查尔酮类等其他化合物[2-3],真菌体内主要为Ⅰ型聚酮合酶。聚酮类化合物的研究关键在于聚酮合酶的结构研究和模型构建,目前关于Ⅰ型聚酮合酶的各个结构域的组成和功能已经有明确的研究[4],但聚酮合酶在催化聚酮类化合物合成过程中各个结构域的构象变化及聚酮合酶整体的结构一直没有解决,极大限制了聚酮类化合物的生产应用以及聚酮合酶的研究进展。
人源脂肪酸合酶(Human Fatty Acid Synthase)是催化合成人体内的饱和脂肪酸并且自身分子量较大的合成酶,表达浓度与人体过度肥胖、癌症和糖尿病等相关疾病联系密切[5-6]。目前哺乳动物的脂肪酸合酶各个结构域的结构和功能已经有较为清晰的阐述,但整体三维结构仍未得到。脂肪酸合酶催化脂肪酸合成的过程与Ⅰ型聚酮合酶催化合成聚酮类化合物的过程非常类似,主要区别在于脂肪酸合酶催化底物较为单一且催化过程中没有侧链的变化;而Ⅰ型聚酮合酶的催化底物复杂多样,在合成过程中除了碳链主链的延长外,还涉及侧链的众多甲基化修饰、还原和脱水等过程[7]。两者催化结构域的组成和功能极其类似,但脂肪酸合酶的催化合成过程更为简单。因此,研究人的脂肪酸合酶结构及催化过程中脂肪酸合酶各个结构域的局部重排可以有效地为聚酮合酶的研究提供理论基础,有助于推进聚酮合酶整体的研究进展。
在脂肪酸合酶蛋白的结构研究中,酵母的脂肪酸合酶蛋白已有解析得到的冷冻电镜结构,整体呈现一个类似桶状的结构[8],而且也已通过冷冻电镜直接观察得到脂肪酸合酶底物穿梭过程的构象变化,其底物穿梭机制结构基础已经有详细研究[9-10]。然而在哺乳动物的脂肪酸合酶结构研究中,尚无脂肪酸合酶整体结构的研究。有研究人员对猪的脂肪酸合酶蛋白结构进行模型构建是通过对脂肪酸合酶各个结构域的同源蛋白进行分析比对,同时结合各个同源蛋白的电镜结构分析结果和脂肪酸合酶部分结构域的电镜结果,确定发挥催化作用和连接作用的氨基酸位点,利用其中部分氨基酸连接各个结构域,构建整体模型[10-11],但缺乏脂肪酸合酶蛋白电镜结果的验证。哺乳动物脂肪酸合酶的研究中,现阶段研究思路更多的是对单个结构域分离后进行电镜观察[12-14],但已有研究证明脂肪酸合酶在催化过程中存在结构域的局部重排现象,并且在催化过程中,结构域之间的交界界面也会发生构象的变化[15],因此脂肪酸合酶功能的结构基础研究需要从整体结构层面进行分析,人源脂肪酸合酶蛋白催化过程中呈现二聚体状态[16],蛋白分子量大、纯化难度较大,成为限制人源脂肪酸合酶电镜结构整体研究的重要因素。本实验成功构建起人源脂肪酸合酶蛋白体外纯化体系,通过凝胶电泳实验检测所纯化得到蛋白的分子量,通过液相层析实验确认所纯化蛋白为二聚体状态,与人源脂肪酸合酶的分子量、折叠状态进行比对后验证所纯化蛋白为人源脂肪酸合酶蛋白,为人源脂肪酸合酶的功能研究和催化过程中的局部重排研究提供了新的研究思路。
1 材料与方法 1.1 材料 1.1.1 菌种和质粒酵母表达载体BJ5464、Escherichia coli DH10B及酵母表达质粒pxw55,均由本实验室保存提供。
1.1.2 主要试剂和仪器及培养基质粒提取试剂盒,天根生化科技(北京)有限公司;实验过程中使用的PCR试剂、DNA连接酶、DNA Marker及蛋白Marker (RM014),生工生物工程(上海)股份有限公司;酵母转化试剂盒,睿安公司;其他常用试剂均为国产分析纯。
快速蛋白质液相层析(Fast Protein Liquid Chromatography,FPLC) (AKTA Prime Plus),GE公司;PCR仪,ABI公司;高压破碎仪,永联生物公司;垂直蛋白电泳仪,Bio-Rad公司;−30冰箱,上海盈翔制冷设备有限公司;StrepTrapTMHP预装柱,通用电气公司;简易流量泵,常州普瑞流体技术有限公司。
UDM液体培养基(g/L):酵母提取物水解酪蛋白氨基酸5.0,酵母氮源基础0.067,腺嘌呤硫酸盐0.02,色氨酸0.02,D(+)-葡萄糖2.0,用纯水定容。UDM固体培养基(g/L):酵母提取物水解酪蛋白氨基酸5.0,酵母氮源基础0.067,腺嘌呤硫酸盐0.02,色氨酸0.02,D(+)-葡萄糖2,Bacto agar 2.0。YPD培养基(g/L):酵母粉10.0,蛋白胨20.0,D(+)-葡萄糖20.0。
1.2 方法 1.2.1 引物设计和合成以酿酒酵母的pxw55质粒为模板,根据人源脂肪酸合酶基因设计VhFas-16c-S和VhFas-16c-A (表 1)这对合成引物作为质粒载体的扩增引物,f-hFas78S和f-hFas78A (表 1)含有Strep标签和His标签的片段用于后续与载体拼接。PCR反应体系:上、下游引物(10 μmol/L)各1 μL,质粒模板(5 ng/μL) 1 μL,ddH2O 8.5 μL,2×PCR Buffer 12.5 μL,高保真DNA聚合酶(1 U/μL) 0.5 μL,dNTPs (2.5 mmol/L) 0.5 μL。质粒总长度为13 770 bp。PCR反应条件:95 3 min;95 20 s,58 30 s,72 4 min,35次循环;72 10 min[17]。得到的PCR产物进行0.8%的琼脂糖凝胶电泳分析,根据片段长度确定产物是否正确。
| 引物 Primers |
核苷酸序列 Nucleotide sequence (5′→3′) |
引物长度 Primer length (bp) |
| VhFas-16c-S | GAAAAGTTGGAGGGCCATCACCATCAC | 27 |
| VhFas-16c-A | TCAGCCAAGGAGCTGTGGATGATGCTG | 27 |
| f-hFas78S | ACAGCTCCTTGGCTGAACCAAGAGTTAGTGTTAGAGAAG GTCCATGGTCTCATCCACAATTTGAAAAGTTGGAGGGCC |
78 |
| f-hFas78A | GGCCCTCCAACTTTTCAAATTGTGGATGAGACCATGGAC CTTCTCTAACACTAACTCTTGGTTCAGCCAAGGAGCTGT |
78 |
将得到的正确PCR质粒载体扩增产物用消化酶(Dpn I)进行模板消化,消化后的载体与f-hFas78S和f-hFas78A片段(表 1)用DNA连接酶进行连接,构建人的脂肪酸合酶在酵母中的过表达质粒[18]。在DH10B菌株中繁殖并提取质粒后进行凝胶电泳分析,根据质粒分子量比对,初步验证转入正确质粒,之后,对质粒进行序列检测,并与基因库中人源脂肪酸合酶基因进行比对,验证正确的质粒命名为pxw55-hfas-cSHII。
选用酵母转化试剂盒将验证好的构建的过表达质粒转入到酵母感受态细胞BJ5464。用筛选平板选择阳性克隆,得到表达菌株,命名为BJ5464 hfas cHSII。
1.2.3 人的脂肪酸合酶的诱导表达用含有氨苄青霉素抗性的筛选平板挑选得到阳性单克隆菌落,接种到100 mL UDM液体培养基中,在250 mL规格的锥形瓶中于28条件下培养72 h后能看到明显浑浊的菌液,按照1%的接种量,将在UDM液体培养基中生长达到饱和的种子菌转接到YPD表达培养基中,同时在YPD中加入终浓度为20 g/L的葡萄糖作为碳源,28 220 r培养72 h。培养结束后将菌液于4 16 000 r离心1 h,收集菌体保存在−30冰箱中。
1.2.4 人的脂肪酸合酶的纯化首先进行Ni-NTA纯化,用30 mL缓冲液对5 mL的镍树脂进行平衡。将−30冻存的菌体放置在常温水中进行解冻,菌体和缓冲液按照1:10 (质量体积比)的比例重悬,重悬后的菌体于高压破碎仪中进行破菌,压力控制在1 100 Pa左右,循环4次,期间不断补加缓冲液保证破碎后菌液的pH值在8.0左右。破碎后菌液在4、15 000 r/min离心1 h,取离心后上清与镍树脂按照1:25的体积比在离心管中进行混合,放置于4的旋转摇床上孵育2 h,使目标蛋白充分结合到镍树脂上。孵育结束后将混合液体转移到亲和层析柱空柱中,待菌液充分流净后,用30 mL缓冲液洗涤镍树脂,洗去非特异性结合的杂蛋白。用10 mL高咪唑浓度缓冲液洗涤镍树脂,得到镍树脂纯化后的含有目标蛋白的洗脱液(6 mL)。
再接第二部亲和层析纯化,选用StrepTrapTMHP预装柱,用30 mL缓冲液进行平衡。用简易流量泵以2 mL/min的速率将经过镍树脂纯化后的洗脱液缓慢通过平衡后的预装柱,进行二次亲和层析,洗脱液全部通入树脂后用30 mL缓冲液洗涤树脂进一步去除非特异性结合的杂蛋白,用含有终浓度2.5 mmol/L脱硫生物素的缓冲液缓慢洗脱StrepTrapTMHP预装柱,得到经过镍树脂和StrepTrapTMHP预装柱双重亲和层析后的蛋白。
2 结果与分析 2.1 克隆载体的构建和表达将扩增后的PCR产物用0.8%的琼脂糖凝胶电泳检测,在10 000 bp上方和300 bp下方出现条带(图 1),与设计的13 770 bp和78 bp大小一致,符合预期。同时将构建的质粒送样北京擎科新业生物技术有限公司测序,测序结果与基因库中人源脂肪酸合酶基因和His标签、Strep标签比对,比对结果确认质粒构建正确。

|
| 图 1 载体PCR结果和合成的带有亲和层析标签片段 Figure 1 PCR results of plasmid vector and synthesized fragments with affinity tags 注:M:DNA Marker DL10000;1:人源脂肪酸合酶载体的扩增片段;2:退火后具有His标签和Strep标签的合成片段 Note: M: DNA Marker DL10000; 1: Amplified fragments of human fatty acid synthase vectors; 2: Synthetic fragment with His tag and Strep tag after annealing |
|
|
在酿酒酵母中表达的人源脂肪酸合酶,经过镍树脂和StrepTrapTMHP预装柱亲和层析纯化后,样品进行SDS-PAGE检测。结果显示在245 kD上方的一条特异性目标蛋白条带,大小符合人源脂肪酸合酶的270 kD (图 2A)。对样品进行Native-PAGE电泳检测,出现一条分子量大小在440−660 kD左右的条带(图 2B),符合人源脂肪酸合酶在活性条件下呈现二聚体结构的事实。

|
| 图 2 人源脂肪酸合酶纯化结果 Figure 2 Results of purification of human fatty acid synthase 注:A:人源脂肪酸合酶的SDS-PAGE检测。B:人源脂肪酸合酶的Native-PAGE检测。C:人源脂肪酸合酶的FPLC检测。M:蛋白Marker;NiE:人源脂肪酸合酶蛋白经镍树脂纯化后的结果;SE1、SE2:人源脂肪酸合酶蛋白先后经过镍树脂和StrepTrapTMHP预装柱纯化后收集的第5、6管;8−12:人源脂肪酸合酶蛋白在FPLC中主要紫外吸收峰的检测结果 Note: A: SDS-PAGE assay of human fatty acid synthase. B: Native-PAGE assay of human fatty acid synthase. C: FPLC assay of human fatty acid synthase. M: Protein marker; NiE: Results of human fatty acid synthase protein purified by Ni-NTA; SE1, SE2: The human fatty acid synthase protein was purified by Ni-NTA and StrepTrapTMHP, and the 5th and 6th tubes were collected; 8−12: Detection results of the main UV absorption peaks of human fatty acid synthase protein in FPLC |
|
|
将经过镍树脂和StrepTrapTMHP预装柱纯化的蛋白样品进行FPLC检测,结果能够观察到在8−12 mL处出现明显的蛋白吸收峰(图 2C),证明得到人源脂肪酸合酶蛋白的二聚体结构,对出现蛋白吸收峰的样品进行SDS-PAGE检测,有单一的目标蛋白条带,并且在FPLC中的紫外吸收峰形状左右对称,无其他紫外吸收峰,也证明人源脂肪酸合酶在经过镍树脂和StrepTrapTMHP预装柱纯化后能去除绝大部分杂蛋白,两步亲和层析的效果明显。但蛋白的产量不高,在破碎菌体时需要用到大量的菌体,导致破碎过程中的pH值要求严格,同时大量的杂蛋白会导致纯化效率的降低。因此,在综合考虑镍树脂纯化结果、两步亲和层析结果及FPLC的检测结果之后,对整个蛋白纯化流程中的缓冲液进行优化,在保证蛋白纯度的前提下进一步提高蛋白产量,提高纯化效率。同时,优化缓冲体系后,蛋白样品在电镜观察下的均一性增加,证明优化缓冲体系能有效帮助人源脂肪酸合酶蛋白在体外环境中进行正确的折叠和组装,更有利于在电子显微镜下观察到正确折叠和组装的人源脂肪酸合酶蛋白,提高蛋白构象的均一性,减少错误折叠构象的干扰。因此对缓冲条件的优化可进一步帮助人源脂肪酸合酶蛋白在体外进行正确折叠和组装。
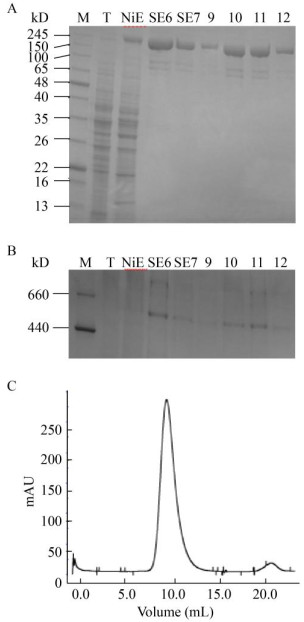
2.4 蛋白纯化条件优化蛋白纯化流程不变,更换缓冲液的种类、pH值、浓度,经过两步亲和层析得到目标蛋白后,根据SDS-PAGE和蛋白产量计算,最终确定合适的蛋白纯化缓冲液条件:镍柱洗脱非特异性结合蛋白的合适缓冲液为25 mmol/L pH 8.0的Tris-HCl,50 mmol/L NaCl,5 mmol/L pH 8.0的咪唑;洗脱特异性目标蛋白的合适缓冲液为25 mmol/L pH 8.0的Tris-HCl,50 mmol/L NaCl,250 mmol/L pH 8.0的咪唑;StrepTrapTMHP预装柱洗脱非特异性结合蛋白的合适缓冲液为25 mmol/L pH 8.0的Tris-HCl,50 mmol/L NaCl;洗脱特异性目标蛋白的合适缓冲液为25 mmol/L pH 8.0的Tris-HCl,50 mmol/L NaCl,2.5 mmol/L脱硫生物素。选用此缓冲体系得到的目的蛋白比例高,产量高且维持蛋白正确折叠构象数目较多,在最适条件下纯化得到的蛋白用SDS-PAGE、Native-PAGE和FPLC检测的结果见图 3。
|
| 图 3 人源脂肪酸合酶在最适缓冲条件下的纯化结果 Figure 3 Results of purification of human fatty acid synthase under optimal buffer conditions 注:A:人源脂肪酸合酶的SDS-PAGE检测。B:人源脂肪酸合酶的Native-PAGE检测。C:人源脂肪酸合酶的FPLC检测。M:蛋白Marker;T:人源脂肪酸合酶表达菌株酿酒酵母细胞破碎后含有的总蛋白;NiE:人源脂肪酸合酶蛋白经镍树脂纯化后的结果;SE6、SE7:人源脂肪酸合酶蛋白先后经过镍树脂和StrepTrapTMHP预装柱纯化后收集的第6、7管;9−12:人源脂肪酸合酶蛋白在FPLC中主要紫外吸收峰的检测结果 Note: A: SDS-PAGE assay of human fatty acid synthase. B: Native-PAGE assay of human fatty acid synthase. C: FPLC assay of human fatty acid synthase. M: Protein marker; T: Total protein contained in human fatty acid synthase-expressing strain Saccharomyces cerevisiae cells after fragmentation. NiE: Results of human fatty acid synthase protein purified by Ni-NTA; SE6, SE7: The human fatty acid synthase protein was purified by Ni-NTA and StrepTrapTMHP, and the 6th and 7th tubes were collected; 9−12: Detection results of the main UV absorption peaks of human fatty acid synthase protein in FPLC |
|
|
对在最适缓冲条件下纯化得到的人源脂肪酸合酶在经过FPLC检测后,蛋白样品进行负染电镜观察,能得到较好的负染照片结果图(图 4),证明蛋白纯化效果良好,能满足后续蛋白电镜结构分析的要求。

|
| 图 4 蛋白负染电镜结果(42 000×) Figure 4 The results of the protein obtained by negative-staining electron microscopy (42 000×) 注:该结果为最适缓冲条件下纯化的人源脂肪酸合酶蛋白在负染电镜观察得到的结果 Note: The negative-stained micrograph results obtained by electron microscopy of human fatty acid synthase protein purified under optimal buffer conditions |
|
|
对处于不同缓冲条件下的人源脂肪酸合酶蛋白进行负染电镜观察后可以发现,首先蛋白的纯度存在一定差异,但差异比较小,说明两步亲和层析对于目标蛋白的纯化非常有效,能得到纯度很高的蛋白。其次两者比较中能明显地看出在非最适环境中得到的蛋白构象不够均一,较难判断颗粒是否属于人源脂肪酸合酶蛋白,电镜观察结果包括从各个角度得到的目标蛋白呈现形式,在构象不均一的情况下很难确定是否是由于蛋白本身的三维结构观察角度造成的差异。因此蛋白的缓冲体系在维持蛋白构象及保证蛋白正确折叠中有重要意义。
考虑到人源脂肪酸合酶在催化过程中存在局部重排现象,在催化的不同阶段也会有不同的构象变化,因此在得到的脂肪酸合酶蛋白中添加底物乙酰辅酶A,让脂肪酸合酶处于催化过程中,对处于该过程中的人源脂肪酸合酶蛋白进行负染电镜观察,从负染电镜中挑选整体构象均一,目标蛋白纯度高的结果进行冷冻电镜观察和数据收集,并对冷冻电镜的数据进行分析,进行二维平均操作,初步分析蛋白结构,但电镜观察到的分辨率较低,后续蛋白二维平均得到的结构分辨率不高,这与脂肪酸合酶蛋白本身的折叠和局部重排有关,考虑可能在得到的体外纯化体系基础上添加辅助因子,进一步提高人源脂肪酸合酶的冷冻电镜观察结果分辨率。虽然蛋白二维平均结果的分辨率不高,但该结果验证了本实验所建立的人源脂肪酸合酶蛋白的体外表达和纯化体系有效,能够在冷冻电镜下观察到人源脂肪酸合酶的整体结构,蛋白的整体结构呈现二聚体形态。人源脂肪酸合酶蛋白体外表达和纯化体系的建立,为后续人源脂肪酸合酶蛋白高分辨率冷冻电镜结构和催化过程中的局部重排研究奠定了良好基础。
3 讨论与结论目前研究蛋白质结构可以通过电子显微镜得到系统准确的蛋白三维结构,研究分析电子显微镜得到的数据,可以有效地进行三维模型的构建[19]。但电子显微镜的研究离不开蛋白质表达、纯化等前期准备,如何能保证蛋白在体外最大限度地保持原有折叠状态、保持原有活性,这是电子显微镜研究蛋白结构的一大难点[20]。
蛋白质的结构决定了其相应的功能,人源脂肪酸合酶整体结构与聚酮合酶类似,催化机制比聚酮合酶简单,因此研究人源脂肪酸合酶的结构对整个聚酮合酶结构的研究及聚酮类化合物的研究都具有十分重要的意义。聚酮合酶独特的催化机制决定了在发挥功能过程中各个结构域会发生构象上的变化,发生局部重排[21-22],因此难以用各个结构域的同源蛋白直接构建三维模型;同时在催化过程中底物的延伸和修饰都需要相应的载体蛋白运送底物及中间产物到各个结构域发生反应,因此研究人源脂肪酸合酶的结构就显得格外重要[23-24];而本实验也成功确定出人源脂肪酸合酶体外纯化的合适条件并进行电子显微镜观察,电镜结果显示蛋白整体构象较为均一,杂蛋白含量少,有利于后续进行目标蛋白的三维模型构建。
本实验通过基因克隆构建,使人源脂肪酸合酶蛋白在酿酒酵母中成功表达;同时结合亲和层析纯化方法,在人源脂肪酸合酶上加上His和Strep标签,使人源脂肪酸合酶能有效地经过两步亲和层析之后与杂蛋白分离开,确定了蛋白体外纯化的合适缓冲环境。在此环境下进行镍树脂和StrepTrapTMHP预装柱亲和层析纯化,以及分子筛的检验之后,都符合后续电子显微镜观察实验中对样品纯度和浓度的要求;同时进行电子显微镜观察得到的负染照片结果显示蛋白样品构象均一,能得到比较好的蛋白结构,为蛋白三维结构的解析奠定良好的基础[25-26]。
| [1] |
Han DD, Wang LY, Luo YP. Isolation, identification, and the growth promoting effects of two antagonistic actinomycete strains from the rhizosphere of Mikania micrantha Kunth[J]. Microbiological Research, 2018, 208: 1-11. DOI:10.1016/j.micres.2018.01.003 |
| [2] |
Chen JY, Chai YR, Yao YH
. Protein expression technologies, structure, activity and evolution of chalcone synthase[J]. Southwest China Journal of Agricultural Sciences, 2012, 25(1): 328-336. (in Chinese) 陈俊毅, 柴友荣, 姚永宏. 查尔酮合酶蛋白表达技术、结构、活性和进化[J]. 西南农业学报, 2012, 25(1): 328-336. DOI:10.3969/j.issn.1001-4829.2012.01.071 |
| [3] |
Shakya G, Rivera H, Lee DJ, Jaremko MJ, La Clair JJ, Fox DT, Haushalter RW, Schaub AJ, Bruegger J, Barajas JF, et al. Modeling linear and cyclic PKS intermediates through atom replacement[J]. Journal of the American Chemical Society, 2014, 136(48): 16792-16799. DOI:10.1021/ja5064857 |
| [4] |
Wu XD, Cheng Q, Du YT, Tian WX
. Structure, function and drug development of ketoacetyl reductase of two kinds of fatty acid synthase[J]. Science & Technology Review, 2005, 23(5): 18-21. (in Chinese) 吴晓东, 程晴, 杜亚涛, 田维熙. 两类脂肪酸合酶中酮酰还原酶的结构、功能和药物开发前景[J]. 科技导报, 2005, 23(5): 18-21. |
| [5] |
Wu HC, Li YS, Liu YC, Lyu SY, Wu CJ, Li TL. Chain elongation and cyclization in type Ⅲ PKS DpgA[J]. Chembiochem, 2012, 13(6): 862-871. DOI:10.1002/cbic.201200051 |
| [6] |
Menendez JA, Lupu R. Fatty acid synthase and the lipogenic phenotype in cancer pathogenesis[J]. Nature Reviews Cancer, 2007, 7(10): 763-777. DOI:10.1038/nrc2222 |
| [7] |
Jones SF, Infante JR. Molecular pathways: fatty acid synthase[J]. Clinical Cancer Research, 2015, 21(24): 5434-5438. DOI:10.1158/1078-0432.CCR-15-0126 |
| [8] |
Singh K, Graf B, Linden A, Sautner V, Urlaub H, Tittmann K, Stark H, Chari A. Discovery of a regulatory subunit of the yeast fatty acid synthase[J]. Cell, 2020, 180(6): 1130-1143.e20. DOI:10.1016/j.cell.2020.02.034 |
| [9] |
Santín O, Yuet K, Khosla C, Moncalián G. Structure and mechanism of the ketosynthase-chain length factor didomain from a prototypical polyunsaturated fatty acid synthase[J]. Biochemistry, 2020, 59(50): 4735-4743. DOI:10.1021/acs.biochem.0c00785 |
| [10] |
Asturias FJ, Chadick JZ, Cheung IK, Stark H, Witkowski A, Joshi AK, Smith S. Structure and molecular organization of mammalian fatty acid synthase[J]. Nature Structural & Molecular Biology, 2005, 12(3): 225-232. |
| [11] |
Leibundgut M, Jenni S, Frick C, Ban N. Structural basis for substrate delivery by acyl carrier protein in the yeast fatty acid synthase[J]. Science, 2007, 316(5822): 288-290. DOI:10.1126/science.1138249 |
| [12] |
Viegas MF, Neves RPP, Ramos MJ, Fernandes PA. Modeling of human fatty acid synthase and in silico docking of acyl carrier protein domain and its partner catalytic domains[J]. The Journal of Physical Chemistry B, 2018, 122(1): 77-85. DOI:10.1021/acs.jpcb.7b09645 |
| [13] |
Mildner AM, Clarke SD. Porcine fatty acid synthase: cloning of a complementary DNA, tissue distribution of its mRNA and suppression of expression by somatotropin and dietary protein[J]. The Journal of Nutrition, 1991, 121(6): 900-907. DOI:10.1093/jn/121.6.900 |
| [14] |
Du DY, Katsuyama Y, Horiuchi M, Fushinobu S, Chen A, Davis TD, Burkart MD, Ohnishi Y. Structural basis for selectivity in a highly reducing type Ⅱ polyketide synthase[J]. Nature Chemical Biology, 2020, 16(7): 776-782. DOI:10.1038/s41589-020-0530-0 |
| [15] |
Joppe M, D'Imprima E, Salustros N, Paithankar KS, Vonck J, Grininger M, Kühlbrandt W. The resolution revolution in cryoEM requires high-quality sample preparation: a rapid pipeline to a high-resolution map of yeast fatty acid synthase[J]. IUCrJ, 2020, 7(2): 220-227. DOI:10.1107/S2052252519017366 |
| [16] |
Fischer M, Joppe M, Mulinacci B, Vollrath R, Konstantinidis K, Kötter P, Ciccarelli L, Vonck J, Oesterhelt D, Grininger M. Analysis of the co-translational assembly of the fungal fatty acid synthase (FAS)[J]. Scientific Reports, 2020, 10(1): 895. DOI:10.1038/s41598-020-57418-8 |
| [17] |
Lynch J. PCR technology: principles and applications for DNA amplification[J]. Journal of Medical Genetics, 1990, 27(8): 536. DOI:10.1136/jmg.27.8.536 |
| [18] |
Backman K, Ptashne M. Maximizing gene expression on a plasmid using recombination in vitro[J]. Cell, 1978, 13(1): 65-71. DOI:10.1016/0092-8674(78)90138-1 |
| [19] |
Tongluan N, Ramphan S, Wintachai P, Jaresitthikunchai J, Khongwichit S, Wikan N, Rajakam S, Yoksan S, Wongsiriroj N, Roytrakul S, et al. Involvement of fatty acid synthase in dengue virus infection[J]. Virology Journal, 2017, 14(1): 28. DOI:10.1186/s12985-017-0685-9 |
| [20] |
Hardwicke MA, Rendina AR, Williams SP, Moore ML, Wang LP, Krueger JA, Plant RN, Totoritis RD, Zhang GF, Briand J, et al. A human fatty acid synthase inhibitor binds β-ketoacyl reductase in the keto-substrate site[J]. Nature Chemical Biology, 2014, 10(9): 774-779. DOI:10.1038/nchembio.1603 |
| [21] |
Seguin F, Carvalho MA, Bastos DC, Agostini M, Zecchin KG, Alvarez-Flores MP, Chudzinski-Tavassi AM, Coletta RD, Graner E. The fatty acid synthase inhibitor orlistat reduces experimental metastases and angiogenesis in B16-F10 melanomas[J]. British Journal of Cancer, 2012, 107(6): 977-987. DOI:10.1038/bjc.2012.355 |
| [22] |
Khoshouei M, Danev R, Plitzko JM, Baumeister W. Revisiting the structure of hemoglobin and myoglobin with cryo-electron microscopy[J]. Journal of Molecular Biology, 2017, 429(17): 2611-2618. DOI:10.1016/j.jmb.2017.07.004 |
| [23] |
Shang J, Zheng Y, Yang Y, Liu C, Geng QB, Tai WB, Du LY, Zhou YS, Zhang W, Li F. Cryo-electron microscopy structure of porcine deltacoronavirus spike protein in the prefusion state[J]. Journal of Virology, 2018, 92(4): e01556-17. |
| [24] |
Harris JR. Transmission electron microscopy in molecular structural biology: a historical survey[J]. Archives of Biochemistry and Biophysics, 2015, 581: 3-18. DOI:10.1016/j.abb.2014.11.011 |
| [25] |
Costa TRD, Ignatiou A, Orlova EV. Structural analysis of protein complexes by cryo electron microscopy[J]. Methods in molecular biology (Clifton, N. J.), 2017, 1615, 377-413. |
| [26] |
Bonomi M, Pellarin R, Vendruscolo M. Simultaneous determination of protein structure and dynamics using cryo-electron microscopy[J]. Biophysical Journal, 2018, 114(7): 1604-1613. DOI:10.1016/j.bpj.2018.02.028 |
 2021, Vol. 48
2021, Vol. 48