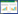扩展功能
文章信息
- 袁维维, 王鸿超, 陈宇涛, 戎一格, 陆文伟, 赵建新, 张灏, 陈卫
- YUAN Weiwei, WANG Hongchao, CHEN Yutao, RONG Yige, LU Wenwei, ZHAO Jianxin, ZHANG Hao, CHEN Wei
- 不同身体质量指数人群肠道菌群结构及共存网络解析
- Feature and co-occurrence network of gut microbiota in different body mass index (BMI) populations
- 微生物学通报, 2021, 48(10): 3776-3790
- Microbiology China, 2021, 48(10): 3776-3790
- DOI: 10.13344/j.microbiol.china.201183
-
文章历史
- 收稿日期: 2020-12-24
- 接受日期: 2021-02-09
- 网络首发日期: 2021-03-09
人体能量稳态与健康状况息息相关,能量稳态的维持是通过能量摄入和能量消耗的协同调节实现的[1]。当能量摄入超过能量消耗则表现为身体质量指数(Body Mass Index,BMI)增加,进而导致肥胖和机体代谢紊乱,反之则表现为由营养不良引起的体重过轻。当今世界,肥胖已经成为西方发达国家巨大的健康负担,许多发展中国家面临着超重和体重过轻的双重负担[2]。肥胖作为一种迅速发展的流行病,不仅给人们的生活带来诸多不便,而且增加了其他疾病的风险,如心脑血管疾病、Ⅱ型糖尿病、冠心病等[3]。体重过轻主要与营养不良有关,与正常体重人群相比,体重过轻人群的传染病率和死亡率均较高[4-5]。
人体中约存在3.9×1013个细菌,其中大部分细菌分布在肠道中,每克湿粪便中约含有1011个细菌[6]。研究表明,人体肠道中已经鉴定出近1 000万条非冗余基因,是人体自身基因组的150倍,因而人体肠道微生物组又被形象的称为“人体第二基因组”[7-8]。因此,人体肠道菌群是一个具有独特微环境的生态系统,肠道菌群的代谢能力远远超过人体细胞的代谢能力,在人类健康中发挥着重要作用。国内外许多研究表明,人体的消化、营养、代谢和免疫等方面与肠道微生态息息相关,肠道菌群的失调与很多疾病之间存在关联[9-10]。
肠道菌群失调与肥胖及体重过轻之间存在因果关系。多项动物实验表明,宿主肠道菌群在能量的获取和储存中发挥作用,因而可能导致肥胖或体重过轻。例如,食用高脂肪饮食无菌小鼠的体重增加率低于食用高脂肪饮食的普通小鼠[11],接受肥胖人群粪便移植的无菌小鼠表现出肥胖症状[12],接受营养不良儿童的菌群的小鼠出现发育不良和体重过轻等症状[13]。众多研究表明,与正常体重的成年人相比,肥胖成年人肠道菌群的组成、多样性均发生了改变[14-15]。然而,相比于正常体重人群,肥胖人群肠道菌群中厚壁菌门/拟杆菌门(Firmicutes/Bacteroidetes)比值变化存在不一致的结论[16]。因此,肥胖人群肠道菌群特征仍需要进一步研究。国内一项研究表明,超重、正常体重及体重过轻3类人群的肠道菌群组成无明显差异,但超重人群及体重过轻人群的肠道菌群α多样性显著低于正常体重人群[17]。近期发表的一项临床研究结果表明,厌食症患者在发病期间(体重过轻)有严重的肠道菌群紊乱[18]。然而,截至目前,关于体重过轻人群肠道菌群特征的研究较少,仍需进一步探究。
本研究从美国肠道计划的公开数据库中选取体重过轻、正常、超重、肥胖人群的肠道菌群样本。在大量样本的基础上,从α多样性、β多样性、物种组成及差异等多方面系统地解析肥胖人群肠道菌群的特征,并构建4类人群肠道菌群共存网络,探究其互作关系。通过对不同BMI人群肠道菌群的结构及共存网络解析,以期为基于肠道菌群干预肥胖及体重过轻等不健康身体状态提供理论依据。
1 材料与方法 1.1 肠道数据来源从美国肠道计划的公开数据集[19]中筛选出最终有效测序序列在1 250条以上的肠道菌群样本,之后再从中筛选具有BMI信息的肠道菌群样本,并根据世界卫生组织规定的BMI划分标准将筛选后的样本分为4类:791个体重过低样本,BMI < 18.5 kg/m2;5 189个正常体重样本,18.5 kg/m2 < BMI < 25 kg/m2;2 055个超重样本,25 kg/m2 < BMI < 30 kg/m2;898个肥胖样本,BMI > 30 kg/m2。α多样性指数(Observed OTUs指数、Faith PD指数和Shannon指数)、β多样性指数(Unweighted UniFrac和Weighted UniFrac距离)和OTU表格均源于美国肠道计划基于QIIME分析平台得来。
1.2 数据分析方法基于AGP数据集中全部样本的Unweighted UniFrac距离矩阵,通过置换多元方差分析(Permutational Multivariate Analysis of Variance Using Distance Matrices,PERMANOVA或Adonis)方法来判定包括经地域、年龄、性别、BMI分类等在内的41种宿主背景信息与其肠道菌群的相关性。通过R语言中Vegan程序包(V2.5-6)中的Adonis函数,置换检验1 000次,计算每个宿主背景信息对于肠道菌群差异的解释度(R2)和判断结果的显著性(P < 0.05则认为具有统计学意义)。基于OTU表,利用Vegan程序包(V2.5-6)的Diversity()函数计算α多样性的Simpson指数。基于Unweighted和Weighted UniFrac距离,采用Kruskal-Wallis检验探究4类BMI人群肠道菌群总体结构的差异。基于门、科、属水平OTU表,利用Amplicon程序包的Tax_stackplot()函数计算门、科、属水平的物种组成并绘制物种组成堆叠图[20]。使用R中的Maaslin2程序包(http://huttenhower.sph.harvard.edu/maaslin),通过多变量相关线性模型来计算不同BMI类别与具体肠道菌群物种之间的相关性。为了降低假阳性率,将未在10%样本中出现的物种删除。根据Adonis分析结果及背景信息覆盖度,选取地域、年龄、性别作为模型校正的混杂因素,并设置Q值的阈值为0.05作为相关性物种的选取标准。
1.3 肠道菌群共存网络分析方法基于物种丰度组成数据进行相关性分析可识别肠道菌群中微生物相互作用。然而,由于测序获得的物种丰度为相对丰度,并未进行绝对定量,可能会产生虚假相关性。SparCC是一种适合测序数据特征的相关性分析方法,可以推断物种之间的相关性,以及构建微生物物种相互作用网络。本研究选取相对丰度在至少一个样本中大于0.1%的属进行相关性分析。基于筛选后的属水平物种丰度表作为原始数据集,使用SparCC分别计算4类BMI人群肠道菌群中菌属相关性。通过Bootstrap方法对原数据集重抽样100次,获得100个随机数据集。对随机数据集中菌属的相关程度进行计算,获得随机数据集的相关性矩阵,从而计算并生成P值矩阵。P值矩阵代表两两菌属之间的相关程度的显著性,P值越低则菌属之间的相关程度越显著(P < 0.05则认为有统计学意义)。筛选出相关系数绝对值大于0.35且P值小于0.05的相关关系构建4类人群肠道菌群的共存网络。
采用R语言中的Igraph程序包(V1.2.5)中的相关函数计算并获得一系列基础的网络参数,以更好了解所构建网络的拓扑结构。网络的基础结构参数包括节点数(Number of Vertices)、边数(Number of Edges)、连接性(Connectance)、平均度(Average Degree)、平均路径长度(Average Path Length)、直径(Diameter)、聚集系数(Clustering Coefficient)、度中心性(Centralization Degree)、介数中心性(Centralization Betweenness)、中心紧密度(Centralization Closeness)、模块性(Modularity)和模块数(Number of Modules)。基于网络节点的平均度及连接性,对网络节点进行重要性排序,获得在4类人群肠道菌群共存网络中的关键菌属。
1.4 统计分析方法统计分析主要基于R 3.6.2 (https://www.r-project.org/)平台,利用Wilcoxon秩和检验进行4类BMI人群肠道菌群之间的α多样性和门水平物种差异性分析,P < 0.05则认为有显著差异;利用Kruskal-Wallis检验进行4类BMI人群肠道菌群之间的β多样性差异分析,P < 0.05则认为有显著差异。
2 结果与分析 2.1 肠道菌群与宿主背景信息的相关性分析基于Unweighted UniFrac距离矩阵,通过基于距离矩阵的Adonis方法计算了宿主肠道菌群的变化与不同宿主背景(如年龄、性别、BMI类别、地域等)之间的关联。结果表明,许多宿主背景因素与肠道菌群显著相关(P < 0.05),按照不同宿主背景信息对肠道菌群差异的解释度(R2值)进行宿主背景信息排序(图 1)。其中,宿主所在地域与肠道菌群的差异之间存在较强的关联。年龄、性别、饮食类型、艰难梭菌感染(Clostridium Difficile Infection,CDI)、炎症性肠病(Inflammatory Bowel Disease,IBD)、自闭症(Autistic Spectrum Disorder,AD)、肠易激综合征(Irritable Bowel Syndrome,IBS)、癌症、阿尔兹海默症(Alzheimer Disease,AD)、糖尿病(Diabetes Mellitus,DM)等宿主背景信息也与肠道菌群显著相关。此外,还发现BMI类别对肠道菌群的差异的解释度也较大,表明体重过轻、正常体重、超重和肥胖人群肠道菌群之间可能存在显著差异,值得进一步探究。
|
| 图 1 基于Unweighted UniFrac距离的Adonis相关性分析 Figure 1 Adonis analysis based on the Unweighted UniFrac distance matrix 注:CDI:艰难梭菌感染;IBD:炎症性肠病;ASD:自闭症;IBS:肠易激综合征;AD:阿尔兹海默症;DM:糖尿病 Note: CDI: Clostridium difficile infection; IBD: Inflammatory bowel disease; ASD: Autistic spectrum disorder; IBS: Irritable bowel syndrome; AD: Alzheimer disease; DM: Diabetes mellitus |
|
|
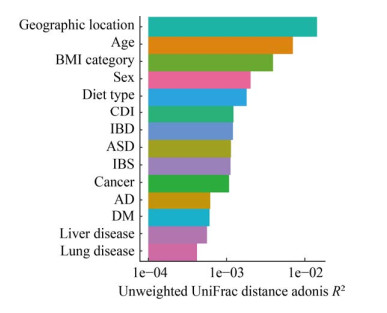
采用Observed OTUs、Faith PD、Shannon和Simpson这4种α多样性指数探究4类BMI人群肠道菌群的丰富度和均匀度(图 2)。Observed OTUs指数主要用来表示某一群落中的物种丰富度,Faith PD指数则主要反应物种进化上的多样性,Shannon指数和Simpson指数则反映群落中物种的丰富度及均匀度。通过Wilcoxon秩和检验对4类BMI人群肠道菌群的多样性进行分析,与正常体重人群相比,4种α多样性指数在体重过轻、超重、肥胖人群中都显著降低,表明体重过轻、超重、肥胖人群中的肠道微生物的丰富度和均匀度都显著低于正常体重人群。此外,肥胖人群肠道菌群的α多样性显著低于超重人群,但与体重过轻人群肠道菌群的α多样性无显著差异,表明肥胖人群肠道菌群丰富度和均匀度显著低于超重人群,但与体重过轻人群肠道菌群的丰富度及均匀度相似。
|
| 图 2 α多样性比对 Figure 2 The comparison of alpha diversity 注:Under:体重过轻;Normal:正常体重;Over:超重。***:P < 0.001;**:P < 0.01;*:P < 0.05 Note: Under: Underweight; Normal: Normalweight; Over: Overweight. ***: P < 0.001; **: P < 0.01; *: P < 0.05 |
|
|
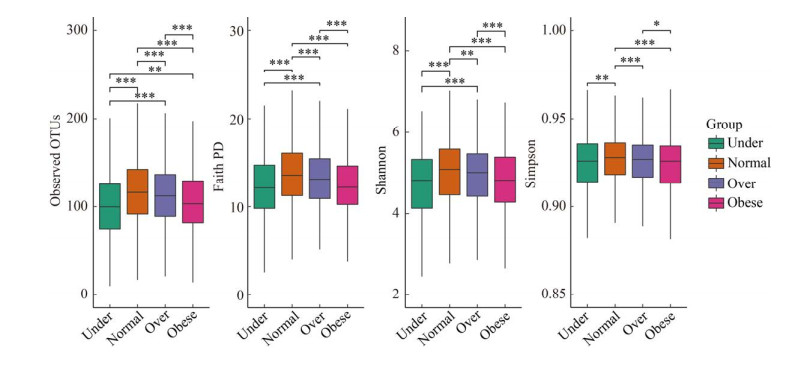
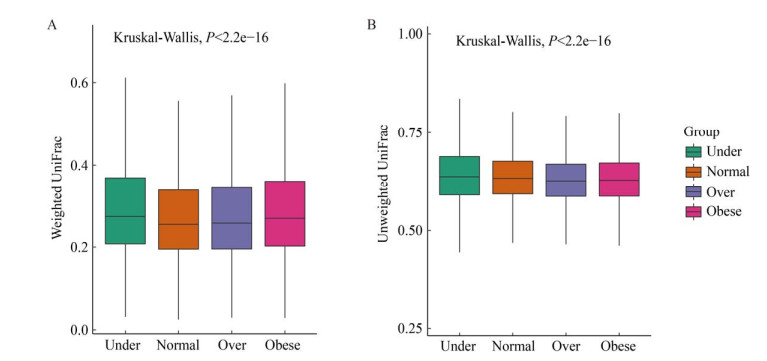
基于2种不同的β多样性距离(Unweighted UniFrac和Weighted UniFrac)对4类BMI人群肠道菌群的整体结构进行分析(图 3A、3B)。UniFrac距离需要通过各个OTU间的进化树,基于系统发育关系来计算样本间的距离,从而反映不同样本基于进化树的肠道菌群差异,以探究2个样本肠道菌群的系统发育关系是否相似。Unweighted UniFrac距离不进行丰度加权,即仅考虑OTU在微生物群落中是否存在,而不考虑其丰度高低。Weighted UniFrac距离进行丰度加权,即不仅仅考虑到OTU序列的存在与否,而且将OTU丰度纳入计算。UniFrac距离的值介于0和1之间,当UniFrac距离的值为0时表明2个样本肠道菌群的种类和数量完全一致,当UniFrac距离的值为1时表明2个样本肠道菌群的种类和数量完全不同。通过Kruskal-Wallis检验,4类BMI人群肠道菌群的Unweighted UniFrac、Weighted UniFrac距离均存在显著差异(P < 2.2e−16),表明4类BMI人群肠道菌群的种类和数量存在显著差异。此外,Adonis分析结果也表明,4类BMI人群肠道菌群的整体结构存在显著差异(Unweighted UniFrac,P=0.001;Weighted UniFrac,P=0.001)。
|
| 图 3 β多样性距离比对 Figure 3 The comparison of beta diversity distance 注:Under:体重过轻;Normal:正常体重;Over:超重 Note: Under: Underweight; Normal: Normalweight; Over: Overweight |
|
|
对4类BMI人群肠道菌群的门、科、属水平进行物种组成分析,并计算厚壁菌门/拟杆菌门比值。在门水平上,4类BMI人群肠道菌群均主要由厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)和疣微菌门(Verrucomicrobia)构成(图 4A)。对4类BMI人群肠道中的厚壁菌门/拟杆菌门(Firmicutes/ Bacteroidetes)比值进行Wilcoxon秩和检验,发现4类BMI人群肠道中的厚壁菌门/拟杆菌门(Firmicutes/Bacteroidetes)比值无显著差异(图 4B)。在科水平上,在4类BMI人群肠道中优势物种主要有:拟杆菌科(Bacteroidaceae)、瘤胃菌科(Ruminococcaceae)、毛螺菌科(Lachnospiraceae)、普雷沃氏菌科(Prevotellaceae)、Porphyromonadacea (图 4C)。在属水平上,在4类BMI人群肠道中拟杆菌属(Bacteroides)、粪杆菌属(Faecalibacterium)、普雷沃氏菌属(Prevotella)、瘤胃球菌属(Ruminococcu)、Parabacteroides、布劳特氏菌属(Blautia)等菌属的相对丰度较高(图 4D)。
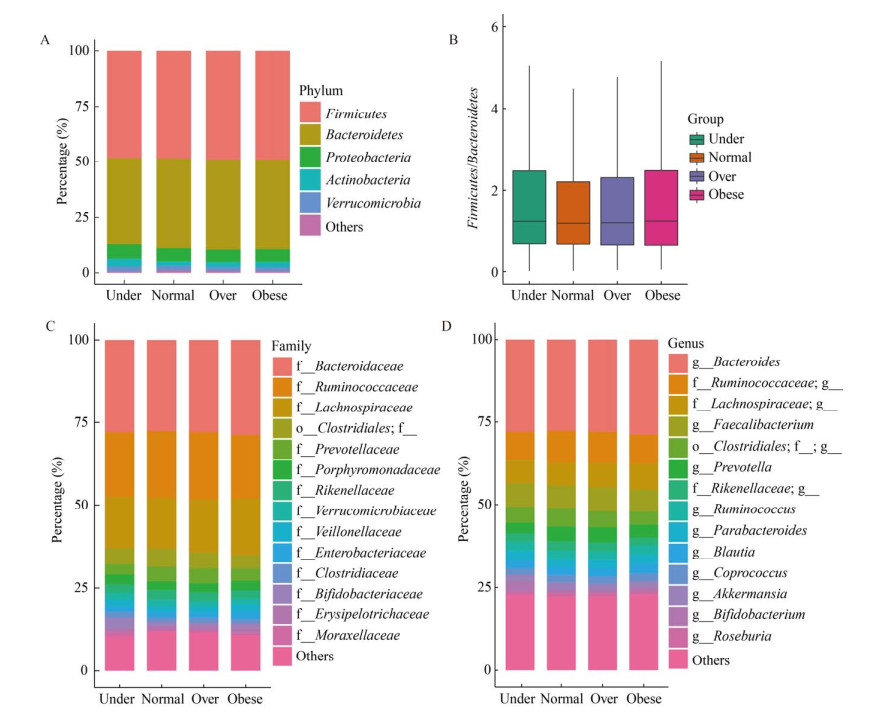
|
| 图 4 门、科、属水平物种组成与厚壁菌门/拟杆菌门比值 Figure 4 The gut microbiome composition at phylum, family and genus level and the Firmicutes/Bacteroidetes ration 注:Under:体重过轻;Normal:正常体重;Over:超重 Note: Under: Underweight; Normal: Normalweight; Over: Overweight |
|
|
通过MaAsLin对不同BMI分类与肠道菌群进行相关性分析,并且将地域、年龄、性别因素作为混杂因素加入到模型中进行校正,共得到49个与BMI类型显著相关的物种(图 5)。在门水平上,放线菌门(Actinobacteria)的相对丰度显著高于正常体重人群;正常体重人群肠道中柔膜菌门(Tenericutes)的相对丰度显著高于其他3类人群。在纲水平上,体重过轻人群的放线菌门(Actinobacteria)显著高于正常体重人群;正常体重人群肠道中柔膜菌纲(Mollicutes)相对丰度显著高于其他3类人群;肥胖人群肠道中产芽胞菌纲(Erysipelotrichi)和红蝽菌纲(Coriobacteriia)相对丰度显著高于正常体重人群。在目水平上,体重过轻人群肠道中双歧杆菌目(Bifidobacteriales)和巴斯德氏菌目(Pasteurellales)相对丰度显著高于正常体重人群;肥胖人群肠道中丹毒丝菌目(Erysipelotrichales)和红蝽菌目(Coriobacteriales)的相对丰度显著高于正常体重人群。在科水平上,体重过轻人群肠道中双歧杆菌科(Bifidobacteriaceae)、梭菌科(Clostridiaceae)、巴斯德氏菌科(Pasteurellaceae)和消化链球菌科(Peptostreptococcaceae)的相对丰度显著高于正常体重人群,帕拉普氏菌科(Paraprevotellaceae)和臭杆菌科(Odoribacteraceae)相对丰度显著低于正常体重人群;肥胖人群肠道中毛螺菌科(Lachnospiraceae)、丹毒丝菌科(Erysipelotrichaceae)、韦荣氏菌科(Veillonellaceae)和红蝽菌科(Coriobacteriaceae)相对丰度显著高于正常体重人群,理研菌科(Rikenellaceae)和Barnesiellaceae的相对丰度显著低于正常体重人群。在属水平上,体重过轻人群肠道中双歧杆菌属(Bifidobacterium)、瘤胃球菌属(Ruminococcus)、韦荣氏球菌属(Veillonella)和Dorea相对丰度显著高于正常体重人群,Coprococcus、帕拉普氏菌属(Paraprevotella)、考拉杆菌属(Phascolarctobacterium)和萨特氏菌属(Sutterella)的相对丰度显著低于正常体重人群;超重人群肠道中毛螺菌属(Lachnospira)和Anaerostipes相对丰度显著低于正常体重人群;肥胖人群肠道菌群中布劳特氏菌属(Blautia)、瘤胃球菌属(Ruminococcus)、考拉杆菌属(Phascolarctobacterium)、巨型球菌属(Megasphaera)、链型杆菌属(Catenibacterium)、Dorea等相对丰度显著高于正常体重人群,毛螺菌属(Lachnospira)和Anaerostipes的相对丰度显著低于正常体重人群。
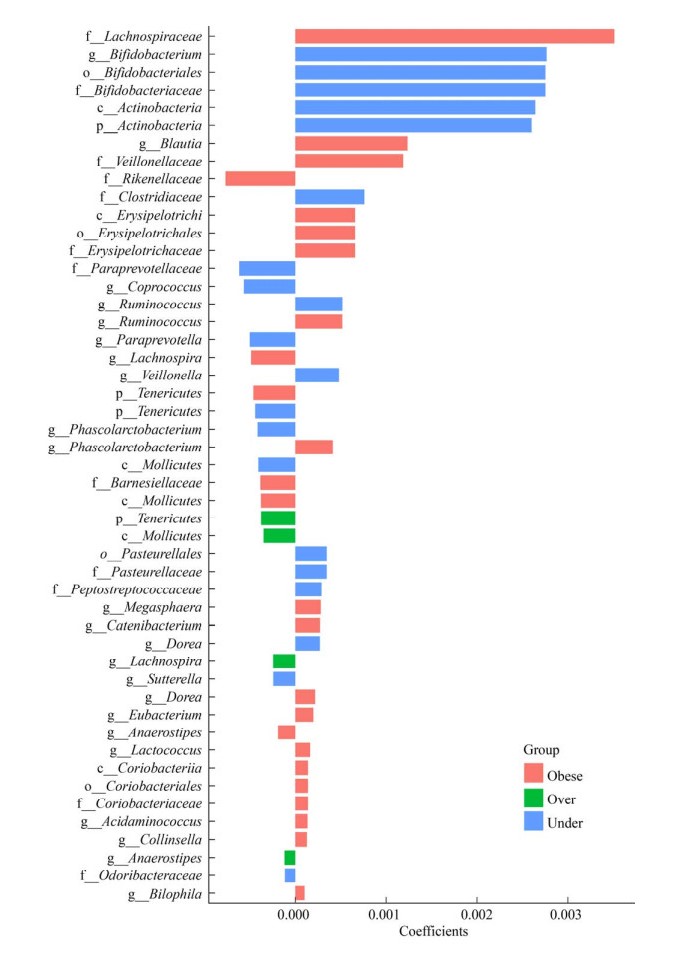
|
| 图 5 不同BMI人群肠道菌群相关性物种分析 Figure 5 Analysis of gut microbiota related species in different BMI populations 注:Under:体重过轻;Normal:正常体重;Over:超重 Note: Under: Underweight; Normal: Normalweight; Over: Overweight |
|
|
对4类BMI人群肠道菌群共存网络的拓扑结构参数进行分析(表 1)。4类BMI人群肠道菌群共存网络的节点数、边数、平均度、连接性、平均路径长度、模块性等参数均存在差异,可初步表明4类BMI人群共存网络拓扑结构复杂性之间的不同。4类BMI人群肠道菌群共存网络共享46.1%的边(70条)与65.2%的节点(43个),表明4类人群肠道菌群网络之间存在一定比例相同的互作关系(图 6)。与其他3类人群相比,体重过轻人群肠道菌群共存网络中特有31条边(占其共存网络的20.4%),6个节点(占其共存网络的9.1%)。正常体重人群肠道菌群共存网络特有4条边(占其共存网络的2.6%),2个节点(占其共存网络的3%)。超重人群肠道菌群共存网络特有3条边(占其共存网络的2%),1个节点(占其共存网络的1.5%)。肥胖人群肠道菌群共存网络特有1条边(占其共存网络的0.7%)。以上统计结果表明,4类人群共存网络的边和节点上具有一定差异性,其中体重过轻人群肠道菌群共存网络结构最复杂。
| Parameters | Underweight | Normal | Overweight | Obese |
| Number of edges | 134.000 | 108.000 | 91.000 | 92.000 |
| Number of positive edges | 133.000 | 107.000 | 90.000 | 91.000 |
| Number of negative edges | 1.000 | 1.000 | 1.000 | 1.000 |
| Number of vertices | 58.000 | 58.000 | 51.000 | 51.000 |
| Connectance | 0.081 | 0.065 | 0.071 | 0.072 |
| Average degree | 4.621 | 3.724 | 3.569 | 3.608 |
| Average path length | 2.329 | 2.470 | 2.549 | 2.306 |
| Diameter | 5.000 | 5.000 | 6.000 | 5.000 |
| Clustering coefficient | 0.574 | 0.480 | 0.601 | 0.528 |
| Centralization degree | 0.200 | 0.198 | 0.169 | 0.168 |
| Centralization betweenness | 0.065 | 0.158 | 0.097 | 0.072 |
| Centralization closeness | 0.018 | 0.017 | 0.015 | 0.016 |
| Modularity | 0.515 | 0.654 | 0.690 | 0.649 |
| Number of modules | 8.000 | 7.000 | 9.000 | 9.000 |
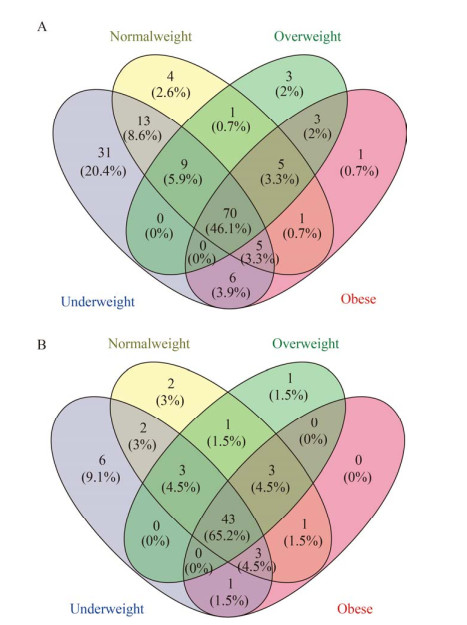
|
| 图 6 肠道菌群网络的边与节点总体差异 Figure 6 Discrepancies of edge and node between the four gut microbiome networks 注:A:4类BMI人群肠道菌群网络边的总体差异;B:4类BMI人群肠道菌群网络节点的总体差异 Note: A: Discrepancies of edge between the four gut microbiome networks; Normal: Discrepancies of node between the four gut microbiome networks |
|
|
4类BMI人群肠道菌群共存网络的模块性均大于0.4,均具有模块化结构。4类BMI人群肠道菌群共存网络中绝大部分节点呈正相关,将这些节点用模块聚类显示,对包含10个及以上节点的较大模块进行分析(图 7)。体重过轻、超重和肥胖人群肠道菌群共存网络拓扑结构中分别具有2个较大模块(模块A和模块B),正常体重人群具有3个较大模块(模块A、模块B、模块C)。其中4类BMI人群肠道菌群的Anaerococcus模块(模块B)内物种组成及复杂程度基本相同,互作物种间的进化距离也相似,均来自厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria)。4类BMI人群肠道菌群的f__Ruminococcaceae_g__模块(模块A)内物种组成及复杂程度有明显差异。从f__Ruminococcaceae_g__模块的复杂程度来看,与正常人群相比,体重过轻人群肠道菌群共存网络中该模块的节点数量及内部复杂度更高,而超重和肥胖人群肠道菌群共存网络中该模块的节点数量及内部复杂度较低。从f__Ruminococcaceae_g__模块的物种组成来看,正常体重人群肠道菌群间的相互作用更趋向于进化距离较远的(来自不同门)菌属之间的相互作用;体重过轻人群肠道菌群共存网络中该模块内物种数目更多,菌群的相互作用更趋向于进化距离较近的菌属之间的相互作用;超重和肥胖人群肠道菌群共存网络中该模块内物种数目均低于正常人群,而且菌群间的相互作用更趋向于进化距离较近的菌属之间的相互作用。
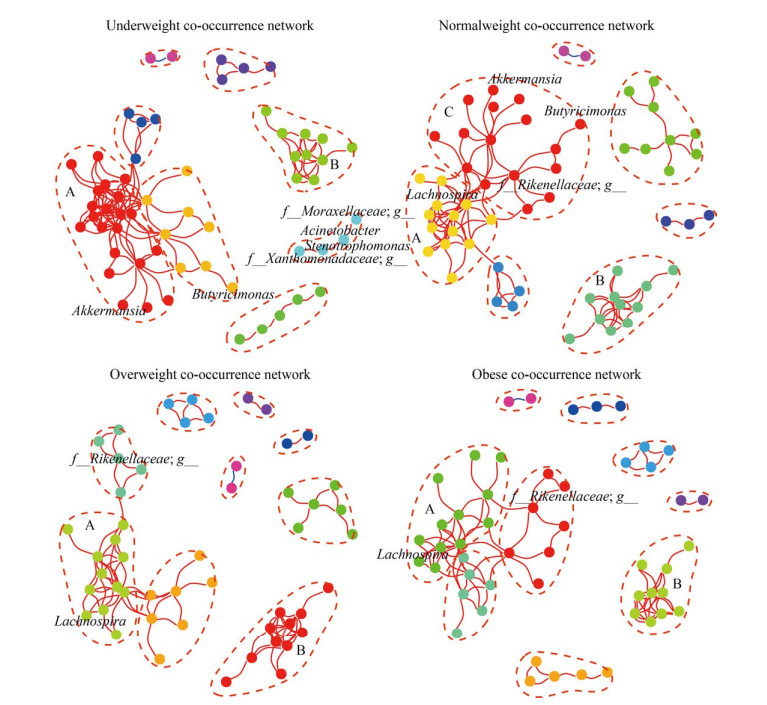
|
| 图 7 肠道菌群共存网络模块差异 Figure 7 Differences of communities of gut microbiome network 注:A:f__Ruminococcaceae_g__模块;B:Anaerococcus模块;C:f__Christensenellaceae_g__模块 Note: A: f__Ruminococcaceae_g__ module; B: Anaerococcus module; C: f__Christensenellaceae_g__ module |
|
|
基于节点的平均度和连接性,分别对4类BMI人群肠道菌群共存网络节点进行重要性排序(图 8)。f__Christensenellaceae_g__、f__Lachnospiraceae_ g__、布劳特氏菌属(Blautia)、Coprococcus、Dorea、罗氏菌属(Roseburia)、f__Ruminococcaceae_ g__、Anaerococcus和Peptoniphilus在4类BMI人群肠道菌群共存网络中均具有较高的度,与其他物种的关联性较强。本研究发现体重过轻与正常体重人群肠道菌群共存网络中的重要节点相似性较高,超重与肥胖人群肠道菌群共存网络中的重要节点相似性较高。瘤胃球菌属(Ruminococcus)、毛螺菌属(Lachnospira)和f__Rikenellaceae_g__在体重过轻和正常体重人群肠道菌群共存网络中的重要性较高,与其他物种的关联较紧密,而在超重和肥胖人群肠道菌群中重要性较低。Finegoldia、f__Tissierellaceae_g__1–68和颤螺菌属(Oscillospira)在超重及肥胖人群肠道菌群共存网络中的重要性较高,在体重过轻与正常人群肠道菌群中与其他物种的关联度较低。此外,Butyricimonas与Akkermansia在体重过轻及正常体重人群肠道菌群网络中特有。在体重过轻人群肠道菌群网络中特有节点为不动杆菌属(Acinetobacter)、Stenotrophomonas、f__Mogibacteriaceae_g__、f__Pasteurellaceae_g__、f__Moraxellaceae_g__、f__Xanthomonadaceae_g__。在超重人群肠道菌群网络中特有节点为Salmonella。
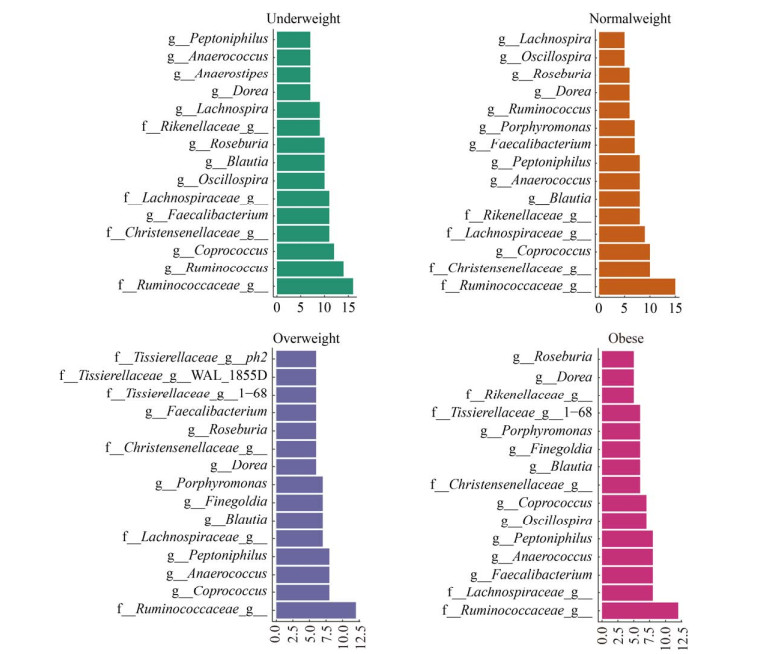
|
| 图 8 肠道菌群网络中重要性节点排序 Figure 8 Ranking of important nodes in the gut microbiome network |
|
|
近年来多个人体肠道微生物组项目表明肠道菌群在能量的储存和消耗中起到重要作用,与人体的BMI状态存在关联[14, 17, 21-22]。研究表明,4类BMI人群肠道菌群的α多样性存在显著差异,体重过轻、肥胖和超重人群肠道菌群的α多样性指数显著低于正常人群,并且肥胖人群肠道菌群的α多样性指数显著低于超重人群[14, 17],本研究的结果与其一致,表明体重过轻、超重和肥胖人群的肠道菌群多样性显著低于正常人群。4类BMI人群肠道菌群的整体结构存在显著差异,与以往研究结果一致[14]。在门水平肠道菌群物种组成上,4类BMI人群肠道菌群均主要由厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)和疣微菌门(Verrucomicrobia)组成。体重过轻、正常体重、超重和肥胖人群Firmicutes和Bacteroidetes的相对含量无显著差异,与多项研究结果相符[14, 17]。然而,有关于肥胖人群的Firmicutes和Bacteroidetes的相对含量与健康人群相比的结论并不一致,早期研究表明Firmicutes/Bacteroidetes在肥胖人群中增加[23-24],后来又有研究表明Firmicutes/ Bacteroidetes在肥胖人群中降低[25-26]。但是,这些研究都集中于单个队列或小型数据库,分析结果可能存在局限性。此外,肠道菌群是一个复杂的生态系统,同一个门水平下不同科、属、种、菌株的功能及其代谢物均存在巨大差异。因此需要在大队列背景下,基于肠道菌群的菌株构成、代谢功能、基因转录以及蛋白表达水平等特征进行深入研究。
MaAsLin分析结果表明,不同BMI分类人群肠道中均存在显著相关性物种。理研菌科(Rikenellaceae)和Barnesiellaceae被证实是正常青少年的肠道菌群标志物[27]。臭杆菌科(Odoribacteraceae)可分解琥珀酸,并且研究表明肥胖患者肠道中其相对丰度显著低于非肥胖人群[28]。在本次研究中我们也有相似的结果,正常体重人群肠道菌群中理研菌科(Rikenellaceae)、Barnesiellaceae和臭杆菌科(Odoribacteraceae)相对丰度显著高于不健康BMI状态人群。此外,本研究发现正常人群肠道中毛螺菌属(Lachnospira)的相对丰度显著高于其他3类人群。毛螺菌属(Lachnospira)被认为可促进维生素D合成并可产短链脂肪酸,对人体健康有益[29-30]。与正常体重人群相比,体重过轻人群肠道菌群的多样性降低,韦荣氏球菌属(Veillonella)和双歧杆菌属(Bifidobacterium)的相对丰度均较高。Chen等研究表明,韦荣氏球菌属(Veillonella)被证实为条件致病菌,Bifidobacterium为人体肠道中益生菌[31]。因此,虽然体重过轻人群肠道菌群多样性显著低于正常体重人群,肠道中的相关条件致病菌较高,但仍然存在一定比例的有益于健康的菌属。在本次研究中,我们发现布劳特氏菌属(Blautia)在肥胖人群肠道菌群中相对丰度较高。Ottosson等的研究表明,布劳特氏菌属(Blautia)与BMI呈正相关[21],与我们的研究相一致。此外,MaAsLin分析结果表明肥胖人群肠道菌群的特征菌还有考拉杆菌属(Phascolarctobacterium)和Eubacterium。肥胖人群肠道内高丰度的考拉杆菌属(Phascolarctobacterium)被证实与减肥成功显著相关[32]。Eubacterium具有较高的系统发育多样性,Eubacterium下的某些种可通过产生短链脂肪酸缓解宿主肥胖症状[33]。这些菌属可能与肥胖干预效果相关,或可作为基于肠道菌群对BMI进行干预的潜在作用靶点。此外,本研究发现,瘤胃球菌属(Ruminococcus)和Dorea在体重过轻及肥胖人群肠道中均为显著相关菌属。Ottosso等研究了674名BMI > 20 kg/m2的瑞典人群的肠道菌群,结果表明瘤胃球菌属(Ruminococcus)和Dorea与BMI呈正相关[21]。在本研究中,瘤胃球菌属(Ruminococcus)和Dorea的相对丰度在正常体重、超重、肥胖人群的肠道中逐渐增加,与上述研究结果一致。我们还发现体重过轻人群(BMI < 18.5 kg/m2)肠道菌群中瘤胃球菌属(Ruminococcus)和Dorea相对丰度显著高于其他3类人群,这在以往的研究中未见报道。瘤胃球菌属(Ruminococcus)和Dorea在非酒精性脂肪肝和非酒精性脂肪肝炎患者肠道中的相对丰度显著高于健康人[34]。因此,我们推测瘤胃球菌属(Ruminococcus)和Dorea可能与能量摄入与消耗失衡及不健康的身体状态相关。
4组BMI人群肠道菌群共存网络的拓扑结构具有一定差异,体重过轻和正常体重人群肠道菌群共存网络的复杂度较高,超重和肥胖人群肠道菌群共存网络的复杂度较低。4组BMI人群肠道菌群共存网络具有46.1%的相同边和65.2%的相同节点,并且均具有物种组成与互作关系基本相同的Anaerococcus模块,这些相同的模块、节点及互作关系可能是人体肠道菌群的基础结构。在我们的研究中,Butyricimonas与Akkermansia是体重过轻及正常体重人群肠道菌群共存网络中的特有节点,在网络中与其他菌属存在直接或间接的正相关(图 7)。Rodriguez等研究表明,Butyricimonas和Akkermansia均与降低肝脏脂质积累有关,并且Akkermansia可驱动菊粉应答者的BMI降低[35]。在超重和肥胖人群肠道菌群共存网络中不存在这2个节点,Butyricimonas和Akkermansia与其他菌属的互作关系的缺失或弱化可能是超重或肥胖的诱因。此外,MaAsLin分析结果表明,毛螺菌属(Lachnospira)和Rikenellaceae为超重及肥胖的显著负相关物种。与正常人群相比,超重及肥胖肠道菌群共存网络Lachnospira和f__Rikenellaceae_g__与其他菌属的互作程度也较低。因此,与健康相关菌属及其互作关系的弱化或缺失,或许是超重及肥胖肠道菌群共存网络复杂度较低的原因。与其他BMI状态人群相比,体重过轻人群的肠道菌群共存网络复杂度最高。存在由不动杆菌属(Acinetobacter)、Stenotrophomonas、f__Moraxellaceae_g__和f__Xanthomonadaceae_g__组成的特有独立模块,模块A的复杂度也最高(图 7)。总之,不同BMI状态肠道菌群共存网络复杂度的差异可能是由于特定节点及互作关系的改变造成的,这需要进一步的实验来验证。
然而,我们的研究存在一些不足。首先,宿主背景因素的复杂性是肠道微生态研究领域的重点和难点,地域、年龄、性别、BMI以及疾病等因素对人体肠道菌群都具有不同程度的影响[36],这也与本研究的分析结果一致。宿主背景因素会对本研究的分析结果产生一定的影响,现有研究并不能完全排除宿主背景混杂因素对疾病与肠道菌群关系的影响[36]。在本研究中,我们校正了地域、年龄、性别因素的影响,在后续的研究中可通过将不同BMI人群的数据集分成考虑地域、年龄因素的子集进行进一步分析。其次,本文数据库所采用的扩增子测序技术存在一定局限性,只能精确到属水平的微生物,无法精确反映样品中精确的菌群结构。因此需要采用宏基因组测序数据进行深入分析,以研究种水平甚至菌株水平的菌群特征。此外,还可扩展数据库进行基因转录和代谢产物层面的研究探索,更加深入解析肠道菌群与不同BMI状态人群的关联。
总之,我们通过大样本量的肠道菌群分析,全面探究了4类BMI人群肠道菌群的特征,同时基于肠道菌群共存网络探究了4类BMI人群肠道菌群共存网络的结构差异及其内部互作关系,为基于肠道菌群来精准干预肥胖及体重过轻等不健康身体状态提供新的理论基础。
| [1] |
Krashes MJ, Lowell BB, Garfield AS. Melanocortin-4 receptor — regulated energy homeostasis[J]. Nature Neuroscience, 2016, 19(2): 206-219. DOI:10.1038/nn.4202 |
| [2] |
Mendez MA, Monteiro CA, Popkin BM. Overweight exceeds underweight among women in most developing countries[J]. The American Journal of Clinical Nutrition, 2005, 81(3): 714-721. DOI:10.1093/ajcn/81.3.714 |
| [3] |
GBD 2015 Obesity Collaborators, Afshin A, Forouzanfar MH, Reitsma MB, Sur P, Estep K, Lee A, Marczak L, Mokdad AH, Moradi-Lakeh M, et al. Health effects of overweight and obesity in 195 countries over 25 years[J]. New England Journal of Medicine, 2017, 377(1): 13-27. DOI:10.1056/NEJMoa1614362 |
| [4] |
Dobner J, Kaser S. Body mass index and the risk of infection — from underweight to obesity[J]. Clinical Microbiology and Infection, 2018, 24(1): 24-28. DOI:10.1016/j.cmi.2017.02.013 |
| [5] |
Flegal KM, Graubard BI, Williamson DF, Gail MH. Cause-specific excess deaths associated with underweight, overweight, and obesity[J]. JAMA, 2007, 298(17): 2028-2037. DOI:10.1001/jama.298.17.2028 |
| [6] |
Sender R, Fuchs S, Milo R. Are we really vastly outnumbered? revisiting the ratio of bacterial to host cells in humans[J]. Cell, 2016, 164(3): 337-340. DOI:10.1016/j.cell.2016.01.013 |
| [7] |
Li JH, Jia HJ, Cai XH, Zhong HZ, Feng Q, Sunagawa S, Arumugam M, Kultima JR, Prifti E, Nielsen T, et al. An integrated catalog of reference genes in the human gut microbiome[J]. Nature Biotechnology, 2014, 32(8): 834-841. DOI:10.1038/nbt.2942 |
| [8] |
Qin JJ, Li RQ, Raes J, Arumugam M, Burgdorf KS, Manichanh C, Nielsen T, Pons N, Levenez F, Yamada T, et al. A human gut microbial gene catalogue established by metagenomic sequencing[J]. Nature, 2010, 464(7285): 59-65. DOI:10.1038/nature08821 |
| [9] |
Shen TCD. Diet and gut microbiota in health and disease[J]. Nestle Nutrition Institute Workshop Series, 2017, 88: 117-126. |
| [10] |
Sánchez-Tapia M, Tovar AR, Torres N. Diet as regulator of gut microbiota and its role in health and disease[J]. Archives of Medical Research, 2019, 50(5): 259-268. DOI:10.1016/j.arcmed.2019.09.004 |
| [11] |
Bäckhed F, Manchester JK, Semenkovich CF, Gordon JI. Mechanisms underlying the resistance to diet-induced obesity in germ-free mice[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(3): 979-984. DOI:10.1073/pnas.0605374104 |
| [12] |
Ridaura VK, Faith JJ, Rey FE, Cheng JY, Duncan AE, Kau AL, Griffin NW, Lombard V, Henrissat B, Bain JR, et al. Gut microbiota from twins discordant for obesity modulate metabolism in mice[J]. Science, 2013, 341(6150): 1241214. DOI:10.1126/science.1241214 |
| [13] |
Blanton LV, Charbonneau MR, Salih T, Barratt MJ, Venkatesh S, Ilkaveya O, Subramanian S, Manary MJ, Trehan I, Jorgensen JM, et al. Gut bacteria that prevent growth impairments transmitted by microbiota from malnourished children[J]. Science, 2016, 351(6275): aad3311. DOI:10.1126/science.aad3311 |
| [14] |
Yun Y, Kim HN, Kim SE, Heo SG, Chang Y, Ryu S, Shin H, Kim HL. Comparative analysis of gut microbiota associated with body mass index in a large Korean cohort[J]. BMC Microbiology, 2017, 17(1): 151. DOI:10.1186/s12866-017-1052-0 |
| [15] |
Sun LJ, Ma LJ, Ma YB, Zhang FM, Zhao CH, Nie YZ. Insights into the role of gut microbiota in obesity: pathogenesis, mechanisms, and therapeutic perspectives[J]. Protein & Cell, 2018, 9(5): 397-403. |
| [16] |
Marchesi JR, Adams DH, Fava F, Hermes GDA, Hirschfield GM, Hold G, Quraishi MN, Kinross J, Smidt H, Tuohy KM, et al. The gut microbiota and host health: a new clinical frontier[J]. Gut, 2016, 65(2): 330-339. DOI:10.1136/gutjnl-2015-309990 |
| [17] |
Wan Y, Yuan JH, Li J, Li H, Yin KH, Wang FL, Li D. Overweight and underweight status are linked to specific gut microbiota and intestinal tricarboxylic acid cycle intermediates[J]. Clinical Nutrition, 2020, 39(10): 3189-3198. DOI:10.1016/j.clnu.2020.02.014 |
| [18] |
Monteleone AM, Troisi J, Fasano A, Dalle Grave R, Marciello F, Serena G, Calugi S, Scala G, Corrivetti G, Cascino G, et al. Multi-omics data integration in anorexia nervosa patients before and after weight regain: a microbiome-metabolomics investigation[J]. Clinical Nutrition, 2021, 40(3): 1137-1146. DOI:10.1016/j.clnu.2020.07.021 |
| [19] |
McDonalda D, Hyde E, Debelius JW, Morton JT, Gonzalez A, Ackermann G, Aksenov AA, Behsaz B, Brennan C, Chen YF, et al. American gut: an open platform for citizen science microbiome research[J]. mSystems, 2018, 3(3): e00031-e00018. |
| [20] |
Zhang JY, Zhang N, Liu YX, Zhang XN, Hu B, Qin Y, Xu HR, Wang H, Guo XX, Qian JM, et al. Root microbiota shift in rice correlates with resident time in the field and developmental stage[J]. Science China Life Sciences, 2018, 61(6): 613-621. DOI:10.1007/s11427-018-9284-4 |
| [21] |
Ottosson F, Brunkwall L, Ericson U, Nilsson PM, Almgren P, Fernandez C, Melander O, Orho-Melander M. Connection between BMI-related plasma metabolite profile and gut microbiota[J]. The Journal of Clinical Endocrinology and Metabolism, 2018, 103(4): 1491-1501. DOI:10.1210/jc.2017-02114 |
| [22] |
Gao XF, Zhang MR, Xue JM, Huang JD, Zhuang RH, Zhou XL, Zhang HY, Fu Q, Hao Y. Body mass index differences in the gut microbiota are gender specific[J]. Frontiers in Microbiology, 2018, 9: 1250. DOI:10.3389/fmicb.2018.01250 |
| [23] |
Turnbaugh PJ, Ley RE, Mahowald MA, Magrini V, Mardis ER, Gordon JI. An obesity-associated gut microbiome with increased capacity for energy harvest[J]. Nature, 2006, 444(7122): 1027-1031. DOI:10.1038/nature05414 |
| [24] |
Ley RE, Turnbaugh PJ, Klein S, Gordon JI. Human gut microbes associated with obesity[J]. Nature, 2006, 444(7122): 1022-1023. DOI:10.1038/4441022a |
| [25] |
Schwiertz A, Taras D, Schäfer K, Beijer S, Bos NA, Donus C, Hardt PD. Microbiota and SCFA in lean and overweight healthy subjects[J]. Obesity: Silver Spring, Md, 2010, 18(1): 190-195. DOI:10.1038/oby.2009.167 |
| [26] |
Collado MC, Isolauri E, Laitinen K, Salminen S. Distinct composition of gut microbiota during pregnancy in overweight and normal-weight women[J]. The American Journal of Clinical Nutrition, 2008, 88(4): 894-899. DOI:10.1093/ajcn/88.4.894 |
| [27] |
Del Chierico F, Abbatini F, Russo A, Quagliariello A, Reddel S, Capoccia D, Caccamo R, Ginanni Corradini S, Nobili V, De Peppo F, et al. Gut microbiota markers in obese adolescent and adult patients: Age-dependent differential patterns[J]. Frontiers in Microbiology, 2018, 9: 1210. DOI:10.3389/fmicb.2018.01210 |
| [28] |
Serena C, Ceperuelo-Mallafré V, Keiran N, Queipo-Ortuño MI, Bernal R, Gomez-Huelgas R, Urpi-Sarda M, Sabater M, Pérez-Brocal V, Andrés-Lacueva C, et al. Elevated circulating levels of succinate in human obesity are linked to specific gut microbiota[J]. The ISME Journal, 2018, 12(7): 1642-1657. DOI:10.1038/s41396-018-0068-2 |
| [29] |
Qian XB, Liu YX, Ye XH, Zheng WJ, Lv S, Mo MJ, Lin JJ, Wang WQ, Wang WH, Zhang XN, et al. Gut microbiota in children with juvenile idiopathic arthritis: characteristics, biomarker identification, and usefulness in clinical prediction[J]. BMC Genomics, 2020, 21(1): 286. DOI:10.1186/s12864-020-6703-0 |
| [30] |
Bosman ES, Albert AY, Lui H, Dutz JP, Vallance BA. Skin exposure to narrow band ultraviolet (UVB) light modulates the human intestinal microbiome[J]. Frontiers in Microbiology, 2019, 10: 2410. DOI:10.3389/fmicb.2019.02410 |
| [31] |
Chen J, Chia N, Kalari KR, Yao JZ, Novotna M, Paz Soldan MM, Luckey DH, Marietta EV, Jeraldo PR, Chen XF, et al. Multiple sclerosis patients have a distinct gut microbiota compared to healthy controls[J]. Scientific Reports, 2016, 6: 28484. DOI:10.1038/srep28484 |
| [32] |
Muñiz Pedrogo DA, Jensen MD, Van Dyke CT, Murray JA, Woods JA, Chen J, Kashyap PC, Nehra V. Gut microbial carbohydrate metabolism hinders weight loss in overweight adults undergoing lifestyle intervention with a volumetric diet[J]. Mayo Clinic Proceedings, 2018, 93(8): 1104-1110. DOI:10.1016/j.mayocp.2018.02.019 |
| [33] |
Mukherjee A, Lordan C, Ross RP, Cotter PD. Gut microbes from the phylogenetically diverse genus Eubacterium and their various contributions to gut health[J]. Gut Microbes, 2020, 12(1): 1802866. DOI:10.1080/19490976.2020.1802866 |
| [34] |
Del Chierico F, Nobili V, Vernocchi P, Russo A, De Stefanis C, Gnani D, Furlanello C, Zandonà A, Paci PL, Capuani G, et al. Gut microbiota profiling of pediatric nonalcoholic fatty liver disease and obese patients unveiled by an integrated meta-omics-based approach[J]. Hepatology, 2017, 65(2): 451-464. DOI:10.1002/hep.28572 |
| [35] |
Rodriguez J, Hiel S, Neyrinck AM, Le Roy T, Pötgens SA, Leyrolle Q, Pachikian BD, Gianfrancesco MA, Cani PD, Paquot N, et al. Discovery of the gut microbial signature driving the efficacy of prebiotic intervention in obese patients[J]. Gut, 2020, 69(11): 1975-1987. DOI:10.1136/gutjnl-2019-319726 |
| [36] |
He Y, Wu W, Zheng HM, Li P, McDonald D, Sheng HF, Chen MX, Chen ZH, Ji GY, Zheng ZDX, et al. Regional variation limits applications of healthy gut microbiome reference ranges and disease models[J]. Nature Medicine, 2018, 24(10): 1532-1535. DOI:10.1038/s41591-018-0164-x |
 2021, Vol. 48
2021, Vol. 48