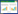扩展功能
文章信息
- 张博雅, 余珂
- ZHANG Bo-Ya, YU Ke
- 微生物基因数据库在氮循环功能基因注释中的应用
- Application of microbial gene databases in the annotation of nitrogen cycle functional genes
- 微生物学通报, 2020, 47(9): 3021-3038
- Microbiology China, 2020, 47(9): 3021-3038
- DOI: 10.13344/j.microbiol.china.200233
-
文章历史
- 收稿日期: 2020-03-14
- 接受日期: 2020-05-27
- 网络首发日期: 2020-06-30
氮循环是重要的生物地球化学循环,过量氮素的滞留会导致水体富营养化,含氮污染物更是污水处理的主要对象,了解和调控氮循环途径对解决环境问题至关重要[1]。微生物体内氮代谢相关作用酶是驱动环境中氮素转化的重要因素[2]。编码这些酶活性亚基的基因称为微生物的功能基因[3]。基于功能基因的检测分析,能够更加深入、全面、细致地揭示环境中微生物的组成、多样性及功能[4]。近年来,随着基因测序技术的发展,基因序列数量迅速增长,各种类型的数据库被开发并应用到功能分析中[5]。然而,目前对不同基因数据库在氮循环功能基因注释中的应用情况提出理论性指导的研究少见报道。
本文详细阐述了由微生物引起的6种无机氮循环途径及其功能基因,并对比了目前最常用的全基因组数据库、结构域数据库和氮循环基因数据库的设计理念、功能特点及氮循环功能基因收录情况,最后以2018年以来引用率较高的52篇文献为样本,从环境介质、表征基因、分析方法和比对方法4个角度分析了研究人员在进行氮循环功能基因注释时,对微生物基因数据库的选择及应用方式,以期为研究人员更好地利用数据库平台解析氮循环基因家族提供一定的参考依据。
1 微生物基因数据库 1.1 全基因组数据库在微生物测序分析中,常常需要对未知的核酸或蛋白质序列进行物种、功能和类别注释,其中最常用的方法是与一些标准数据库进行相似性搜索,即序列相似性比对。因此,数据库的优劣至关重要。常见的包含全基因组序列的核酸或蛋白质数据库如下:
1.1.1 National Center for Biotechnology Information (NCBI)NCBI是目前最权威的生物信息分析平台,其下GenBank数据库每天与European Molecular Biology Laboratory (EMBL)和DNA Data Bank of Japan (DDBJ)交换数据,包含超过7万种生物体的序列,但存在注释错误、术语混乱和序列重复等问题[6];Reference Sequence (RefSeq)数据库经NCBI和其他组织校正,可信度较高,收集了超过5.5万种生物体的核酸序列及其蛋白质产物,多用于物种注释分析[7];Non-Redundant Protein Sequence (NR)数据库是非冗余蛋白质数据库,多用于蛋白质功能注释,其子集Nucleotide Sequence (NT)数据库是非冗余核酸数据库。Yu等[8]提出了一种从NCBI-NR数据库中构建局部子数据库的方法,可用于大型宏基因组数据集的快速相似性搜索和注释,并发现被Metagenome Analyzer (MEGAN)注释为氮循环中氨化作用、反硝化作用、硝化作用和固氮作用的序列共4 318条。Integrated Microbial Genomes (IMG)数据库基于NCBI-RefSeq整合了包含古菌、细菌、真核生物、质粒、病毒和细胞富集物等更为详细的基因组信息,仅细菌基因组数目已超过5万条,而且条目清晰、输出方便[9]。
1.1.2 Universal Protein (UniProt)数据库UniProt数据库是收录信息最全面的非冗余蛋白质数据库,包含3个部分:(1) UniProt Knowledgebase (UniProtKB)可进行交叉引用与物种注释;(2) UniProt Reference Clusters (UniRef)可进行不同相似度序列搜索,并根据序列相似度分为UniRef100、UniRef90和UniRef50;(3) UniProt Archive (UniParc)可进行序列历史资料存储与查询[10-11]。其下Swiss Protein (Swiss-Prot)数据库经手工核对,提供每条序列的详细物种注释、实验结果和计算特征,但更新速度较慢;Translation from EMBL (TrEMBL)数据库是计算机注释的Swiss-Prot补充数据库,能够暂时储存日益增多的蛋白质结构信息[12];Protein Information Resource (PIR)数据库可帮助研究人员鉴别和解释蛋白质序列信息,研究分子进化、功能基因组等[13]。
1.1.3 其他数据库Kyoto Encyclopedia of Genes and Genomes (KEGG)整合了基因组、化学和系统功能信息,具有非常详细的分类模块,能够让研究人员更直观地了解代谢途径;目前已收录超过60种氮循环表征基因,是研究人员绘制氮循环基因表达热图和重构代谢通路图的主要参考依据[14]。Encyclopedia of Metabolic Pathway (MetaCyc)数据库收录了1 400多条代谢途径和相关酶;Encyclopedia of Microbial Genome and Metabolic Pathway (BioCyc)数据库以MetaCyc为参考,提供了500多个生物体的全基因组序列和预测的代谢网络[15]。The SEED Project (SEED)数据库能够提供直系同源基因的准确注释,研究人员常用其下Rapid Annotation using Subsystem Technology (RAST)引擎注释基因组功能和发现新的代谢途径[16]。Message Digest Algorithm 5 Non-Redundant Protein (M5nr)数据库实现了多个数据库的序列共享,包含存储标识符、功能注释和分类信息等,使用户可以在短时间内看到数据的多种解释分析[17]。Similarity Matrix of Proteins (SIMAP)数据库可对蛋白质序列进行同源计算,预测蛋白质序列相似性,并提供专业的序列检索工具[18]。Gene Ontology (GO)数据库基于基因本体论从生物过程、分子功能和细胞组成3个方面对基因和基因产物进行分类注释,是宏转录组分析的常用数据库[19]。
1.2 结构域数据库蛋白质结构域指较大的蛋白质分子中具有特异结构和独立功能的区域。这些结构域共同决定了一个基因转录的蛋白质分子的功能,具有相同蛋白质结构域的基因共同构成一个基因家族。因此,通过蛋白质结构域鉴别微生物功能基因序列更为准确。常见的结构域数据库如下:
(1) Protein Families (Pfam)数据库整合了一系列蛋白质家族,每个蛋白家族均具有隐马尔可夫模型(hidden markov models,HMMs)的表示形式,常用于蛋白质功能结构域的查询和分析[20]。
(2) Functional Gene (FunGene)数据库利用HMMs分类,包含了近30种氮循环基因序列,但其存在冗余序列且一次只允许下载1万条序列,较为不便[21]。
(3) Clusters of Orthologous Groups (COG)/ Clusters of Orthologous Groups for Eukaryotic Complete Genomes (KOG)数据库是由NCBI开发的用于原核/真核生物同源蛋白注释的数据库,其对21种完整微生物基因组的编码蛋白进行了系统发育分类,能够提供直系同源物和旁系同源物的可靠分配,但缺陷是收录的直系同源基因组数量相对较少,仅有4 631个[22]。
(4) Evolutionary Genealogy of Genes: Non- supervised Orthologous Groups (EggNOG)数据库使用无监督聚类算法将COG数据库的直系图扩展到超过19万个直系同源基因组,有效改进了上述问题[23]。
(5) Simple Modular Architecture Research Tool (SMART)数据库是一种模块较为简化的在线搜索和分析平台,常用于蛋白质结构域识别和功能注释,其集成了许多蛋白质结构预测和功能分析的工具,可以预测蛋白质的一些二级结构[24]。
(6) The Institute for Genomic Research defined Protein Families (TIGRfam)数据库能够自动进行蛋白质功能结构域注释和基因组分类,可针对性地用于区分细菌和古菌[25]。
(7) Conserved Domain Database (CDD)是NCBI数据库中蛋白质结构域数据库镜像,收集了来自Pfam、COG、SMART和TIGRfam等数据库中的结构域信息[26]。
1.3 氮循环基因数据库NCycDB数据库专门用于分析环境样品中氮循环相关基因,其手动整合了KEGG、UniProt、COG、EggNOG和SEED数据库,共收录了8个氮循环途径、68种氮循环基因的219 146条参考序列,鉴定了1 958组与氮循环编码蛋白具有相似结构域但不能参与氮循环的蛋白质同系物[27]。Zehr数据库针对性地整理了固氮基因nifH序列,2014年,Heller等[28]开发了一个半自动平台ARBitrator,可识别GenBank数据库中的nifH基因序列;2016年,Frank等[29]利用Classification and Regression Trees (CART)平台快速将nifH序列分类并明确定义其系统发育关系,后用此法定期更新Zehr数据库。此外,康奈尔大学也曾于2014年提供了一个人工检索和管理、用于研究固氮作用的nifH基因蛋白质数据库[30]。
综合上述最新研究结果,可将目前最常用的不同类型微生物基因数据库对比,如表 1所示。
| 数据库 Databases |
冗余 Redundant |
序列类型 Types of sequences |
功能类型 Types of functions |
最新版本 Lastest update |
网址 Websites |
参考文献 References |
|||||
| 全基因组 Whole genome |
蛋白质结构域 Protein domain |
物种注释 Taxonomic annotation |
序列下载 Sequence download |
在线比对 Online alignment |
代谢通路 Metabolic pathway |
||||||
| NCBI-GenBank | √ | √ | √ | √ | √ | √ | 2020 | https://www.ncbinlm.nih.gov/genbank/ | [6] | ||
| NCBI-RefSeq | √ | √ | √ | √ | √ | 2020 | https://www.ncbinlm.nih.gov/refseq/ | [7] | |||
| NCBI-NR | √ | √ | √ | √ | 2020 | https://ftp.ncbi.nlm.nih.gov/blast/db/FASTA | [8] | ||||
| IMG | √ | √ | √ | √ | √ | 2020 | https://ing.jgi.doe.gov | [9] | |||
| UniProt | √ | √ | √ | √ | √ | 2020 | http://www.uniprot.org/ | [10-11] | |||
| KEGG | √ | √ | √ | √ | √ | √ | √ | 2020 | https://www.genomejp/kegg/ | [14] | |
| BioCyc | √ | √ | √ | √ | √ | √ | 2020 | http://biocyc.org/ | [15] | ||
| SEED | √ | √ | √ | √ | √ | √ | 2020 | htp://pubseetheseed.org/ | [16] | ||
| M5nr | √ | √ | √ | √ | 2012 | htp://metagenomics.nmpdr.org | [17] | ||||
| SIMAP | √ | √ | √ | √ | √ | 2013 | htp://mipsgsf.de/simap/ | [18] | |||
| GO | √ | √ | √ | √ | √ | 2020 | http://geneontology.org | [19] | |||
| Pfam | √ | √ | √ | √ | 2018 | http://pfam.xfam.org/ | [20] | ||||
| FunGene | √ | √ | √ | √ | √ | 2019 | http://fungene.cme.msu.edu | [21] | |||
| COG | √ | √ | √ | √ | 2014 | https://www.ncbi.nlm.nih.gov/COG/ | [22] | ||||
| EggNOG | √ | √ | √ | √ | √ | 2016 | http://ggnogdb.embl.de/#/app/home | [23] | |||
| SMART | √ | √ | √ | √ | 2017 | http://smart.embl.de | [24] | ||||
| TIGRfam | √ | √ | √ | 2014 | htpt://igrfarons.jcvi.org/cigi-bin/index.cgi | [25] | |||||
| NCycDB | √ | √ | 2019 | https://github.com/qichao1984NCyc | [27] | ||||||
| Zehr | √ | √ | 2017 | https://www.jzehrlab.com/nifh | [28-29] | ||||||
| 注:√:数据库包含此类信息或功能. Note: √:The database contained such informations or fiunctions. | |||||||||||
同化硝酸盐还原(assimilatory nitrogen reduction,ANRA)、异化硝酸盐还原(dissimilatory nitrogen reduction,DNRA)和反硝化(denitrification)作用均可还原NO3−,是废水处理中生物脱氮的重要途径。其中,ANRA和DNRA均可将NO3−还原为NH4+,但二者的功能基因并不相同,而且ANRA发生于有氧环境中,产生的NH4+被同化为氨基酸[31];DNRA发生于低氧或缺氧环境中,产生的NH4+既可为异化硝酸盐还原菌提供生长所需氮源,又可释放到胞外为其他细菌生长提供氮源[32]。反硝化作用可将NO3−完全还原为N2,发生于低氧或缺氧环境中,其第一步反应(NO3−→NO2−)的功能基因与DNRA相同,因此,反硝化菌与异化硝酸盐还原菌在生物脱氮体系中具有较强的竞争关系[33]。
2.1.2 固氮作用固氮作用(nitrogen fixation)将大气中的N2 还原成NH4+后,可被微生物利用于合成各种含氮化合物,是生态系统中氮素的主要来源。蓝藻(Cyanobacteria)[34]和γ-变形菌(Gammaproteobacteria)[35]是主要的固氮微生物。因为与固氮作用其他功能基因相比,利用nifH基因序列与16S rRNA基因序列构建的系统发育树具有高度一致的进化表征特性,因此,研究人员常将nifH作为固氮作用的表征基因,检测环境中固氮微生物的种群结构及多样性[30]。
2.1.3 硝化作用硝化作用(nitrification)可将NH4+氧化为NO3−,对生态系统生产力、营养物质循环和废水处理均起着至关重要的作用,其作用菌被称为氨氧化古菌(ammonia oxidizing archaea,AOA)、氨氧化细菌(ammonia oxidizing bacteria,AOB)和硝化细菌(nitrite oxidizing bacteria,NOB)。最新研究表明,AOA是大气PM2.5中氨氧化作用的主要原因[36],也是北极泥炭地高N2O排放的主要驱动力[37],因此,近年来有关AOA的研究不断增加。2006年,Costa等[38]预测存在可直接将NH4+氧化为NO3−的微生物,并将其命名为完全氨氧化细菌(complete ammonia oxidizer,Comammox)。2015年,3个科学团队分别发现3种经过富集的细菌(Candidatus Nitrospira nitrosa、Ca. N. nitrificans和Ca. N. inopinata)和1种未经过纯培养的细菌(Nitrospira sp.)均具备单独将氨氧化为硝酸盐的能力,使学术界对硝化作用过程有了新的认识[39-41]。
2.1.4 厌氧氨氧化作用厌氧氨氧化作用(anaerobic ammonium oxidation,Anammox)可将NO2−和NH4+转化为N2,解决废水系统中高氨氮、低碳氮比的问题,是目前最为经济高效的生物脱氮途径,但厌氧氨氧化菌增殖缓慢,如何使之有效富集是工程技术的关键[42]。针对这一问题,研究人员发现相较于常规膨胀颗粒污泥床(expanded granular sludge blanket,EGSB)反应器,生物质载体膨胀颗粒污泥床(carrier expanded granular sludge blanket,CEGSB)反应器对氨氮和亚硝酸盐的去除率均达到90%以上,总脱氮率稳定在70%以上,不仅可以有效提高厌氧氨氧化菌丰度,而且Ca. Brocadia和Asahi BRW2还可在反应体系中共存[43]。此外,Keren等[44]发现DNRA在与成熟氨氧化细菌群落相关的基因组中非常普遍,而且当含有DNRA基因的细菌繁殖速率增加时,可与Brocadia sp.直接竞争氮源。Carreño等[45]发现Comammox虽然会生成厌氧氨氧化菌不想要的硝氮,但也能同时为其提供亚硝氮。
综上所述,环境中由微生物引起的6种无机氮循环途径主要包括:同化硝酸盐还原作用、异化硝酸盐还原作用、反硝化作用、固氮作用、硝化作用(包括完全氨氧化作用)和厌氧氨氧化作用。接下来,反硝化菌与异化硝酸盐还原菌[33]、异化硝酸盐还原菌与厌氧氨氧化菌[44]、Comammox与厌氧氨氧化菌[45]的互作关系,以及根据微生物间的互作关系有效解决环境工程问题、深入解析生物地球化学循环势必成为研究热点。因此,相较于qPCR、DNA指纹图谱和基因芯片等分子检测技术,采用微生物组学技术根据数据库平台解析氮循环功能基因、代谢通路、微生物种群结构及互作关系更为重要。
2.2 氮循环功能基因及其在数据库中的收录情况 2.2.1 统计方法及收录概况本文根据KEGG和NCBI-RefSeq数据库中的氮循环途径,结合最新文献,详细收录了6种无机氮循环途径中50种功能基因的基因名称及注释信息。随后,利用关键词检索并统计了常用数据库KEGG、NCBI-NR、UniProt和NCycDB中所包含的对应功能基因的序列数量(相关基因序列来自https://github.com/EMBL-PKU/RP-N);同时,比对筛选出了每种功能基因的HMMs信息,具体情况如表 2所示。
| 氮循环途径 Nitrogen cycle pathway |
基因 Gene |
注释 Annotation |
Pfam数据库中HMMs编号 The AC number of HMMs in Pfam database |
常用数据库中基因序列数量 The number of gene seuqences in common databases |
||||
| KEGG | NCBI-NR | UniProt | NCycDB | |||||
| 同化硝酸盐还原作用 Assimilatory nitrogen reduction |
NO3−→NO2− | nasA | Assimilatory nitrate reductase catalytic subunit | PF00384.22, PF01568.21, PF04879.16, PF04324.15 | 1 497 | 1 105 | 2 812 | 3 848 |
| nasB | Assimilatory nitrate reductase electron transfer subunit | PF07992.14, PF18267.1, PF04324.15 | 182 | 401 | 213 | 288 | ||
| NR | Nitrate reductase (NAD(P)H) | PF00970.24, PF00175.21, PF03404.16, PF00174.19, PF00173.28, PF08030.12 | 349 | − | − | 1 038 | ||
| narB | Ferredoxin-nitrate reductase | PF00384.22, PF01568.21, PF04879.16 | 206 | 42 | 1 548 | 2 000 | ||
| narC | Cytochrome b-561 | PF13631.6, PF00033.19 | − | 149 | 28 | 68 | ||
| NO2−→NH4+ | nirA | Ferredoxin-nitrite reductase | PF03460.17, PF01077.22 | 713 | 1 264 | 39 | 981 | |
| NIT-6 | Nitrate nonutilizer-6 | PF03460.17, PF01077.22, PF13806.6, PF04324.15, PF07992.14 | 69 | − | 2 | − | ||
| 异化硝酸盐 还原作用 Dissimilatory nitrogen reduction |
NO3−→NO2− | narG | Nitrate reductase 1, alpha subunit | PF00384.22, PF01568.21, PF14710.6, PF04879.16 | 1 525 | 152 | 5 058 | 4 254 |
| narH | Nitrate reductase 1, beta subunit | PF13247.6, PF14711.6 | 1 645 | 116 | 4 230 | 2 295 | ||
| narJ | Nitrate reductase 1, delta subunit | PF02613.15 | − | 220 | 2 852 | 1 716 | ||
| narI | Nitrate reductase 1, gamma subunit | PF02665.14 | 1 718 | 268 | 3 191 | 1 742 | ||
| narZ | Nitrate reductase 2, alpha subunit | PF00384.22, PF01568.21, PF14710.6, PF04879.16 | 357 | 11 | 429 | 2 154 | ||
| narY | Nitrate reductase 2, beta subunit | PF13247.6, PF14711.6 | 185 | 7 | 212 | 891 | ||
| narW | Nitrate reductase 2, delta subunit | PF02613.15 | − | 5 | 195 | 200 | ||
| narV | Nitrate reductase 2, gamma subunit | PF02665.14 | 93 | 7 | 268 | 154 | ||
| napA | Periplasmic nitrate reductase subunit NapA | PF00384.22, PF01568.21, PF04879.16, PF10518.9 | 950 | 3 154 | 5 801 | 2 225 | ||
| napB | Periplasmic nitrate reductase electron transfer subunit | PF03892.14 | 908 | 462 | 1 169 | 622 | ||
| napC | Nitrate reductase cytochrome c-type periplasmic | PF03264.14 | − | 105 | 807 | 1 062 | ||
| NO2−→NH4+ | nirB | Nitrite reductase (NADH) large subunit | PF04324.15, PF07992.14, PF18267.1, PF01077.22, PF03460.17 | 2 961 | 1 240 | 1 067 | 4 058 | |
| nirD | Nitrite reductase (NADH) small subunit | PF13806.6 | 2 358 | 1 352 | 4 872 | 3 061 | ||
| nrfA | Cytochrome c nitrite reductase subunit c552 | PF02335.15 | 758 | 2 517 | 2 504 | 1 483 | ||
| nrfB | Cytochrome c nitrite reductase pentaheme subunit | PF09699.10, PF13435.6 | − | 48 | 487 | 374 | ||
| nrfC | Cytochrome c nitrite reductase Fe-S protein | PF13247.6 | − | 1 305 | 627 | 1 306 | ||
| nrfD | Cytochrome c nitrite reductase subunit NrfD | PF03916.14 | − | 125 | 914 | 363 | ||
| nrfH | Cytochrome c nitrite reductase small subunit | PF03264.14 | 323 | 1 262 | 740 | − | ||
| 反硝化作用 Denitrification |
NO3−→NO2− | 此步与异化硝酸盐还原作用的功能基因相同:narGHJI、narZYWV、napABC Functional genes in this step was the same as dissimilatory nitrogen reduction: narGHJI, narZYWV, napABC |
||||||
| NO2−→NO | nirK | Nitrite reductase (NO-forming) | PF00394.22, PF07731.14, PF07732.15 | 846 | 911 | 16 464 | 11 462 | |
| nirS | Nitrite reductase (NO-forming) | PF02239.16 | 194 | 903 | 23 639 | 14 682 | ||
| NO→N2O | norB | Nitric-oxide reductase subunit B |
PF00115.20 | 1 121 | 5 119 | 2 364 | 1 863 | |
| norC | Nitric-oxide reductase subunit C |
PF00034.21, PF13442.6 | 434 | 246 | 313 | 469 | ||
| N2O→N2 | nosZ | Nitrous-oxide reductase | PF18764.1, PF18793.1, PF13473.6, PF00116.20 | 538 | 4 350 | 15 516 | 10 822 | |
| 固氮作用 Nitrogen fixation |
N2→NH4+ | anfG | Nitrogenase delta subunit | PF03139.15 | 53 | 197 | 79 | 57 |
| nifD | Nitrogenase molybdenumiron protein alpha chain | PF00148.19, PF01968.18 | 794 | 2 766 | 3 873 | 1 755 | ||
| nifK | Nitrogenase molybdenumiron protein beta chain | PF00148.19, PF11844.8 | 815 | 2 703 | 1 620 | 1 006 | ||
| nifH | Nitrogenase iron protein NifH |
PF00142.18 | 900 | 10 001 | 42 083 | 14 757 | ||
| nifW | Nitrogenase-stabilizing/ protective protein |
PF03206.14 | − | 454 | 1143 | 622 | ||
| vnfD | Vanadium-dependent nitrogenase alpha chain | PF00148.19 | 31 | 11 | 108 | − | ||
| vnfK | Vanadium-dependent nitrogenase beta chain | PF00148.19 | 27 | 2 | 37 | − | ||
| vnfG | Vanadium nitrogenase delta subunit | PF03139.15 | 32 | 1 | 51 | − | ||
| vnfH | Vanadium nitrogenase iron protein | PF00142.18 | 27 | 4 | 13 | − | ||
| 硝化作用 Nitrification |
NH4+→NH2OH | amoA | Ammonia monooxygenase subunit A | PF12942.7, PF02461.16 | 80 | 38 546 | 60 652 | 37 797 |
| amoB | Ammonia monooxygenase subunit B | PF04744.12 | 81 | 371 | 140 | 123 | ||
| amoC | Ammonia monooxygenase subunit C | PF04896.12 | 122 | 549 | 75 | 65 | ||
| NH2OH→NO2− | hao | Hydroxylamine dehydrogenase | PF13447.6 | 56 | 22 | 582 | 79 | |
| NO2−→NO3− | nxrA | Nitrite oxidoreductase, alpha subunit |
PF00384.22 | 7 | 276 | 258 | 210 | |
| nxrB | Nitrite oxidoreductase, beta subunit |
PF13247.6 | 7 | 461 | 625 | 284 | ||
| 厌氧氨氧化作用 Anammox |
NO2−→NO | 此步与反硝化作用的功能基因相同:nirKS Functional genes in this step was the same as nitrification: nirKS |
||||||
| NO+NH4+→N2H4 | hzsA | Hydrazine synthase subunit A | PF18582.1 | 2 | 306 | 448 | 250 | |
| hzsB | Hydrazine synthase subunit B | PF00486.28, PF00072.24, PF03150.14 | 2 | 311 | 710 | 514 | ||
| hzsC | Hydrazine synthase subunit C |
PF02239.16, PF10282.9 | 2 | 7 | 235 | 198 | ||
| hzo | Hydrazine oxidoreductase | PF13447.6 | − | 2 078 | 2 374 | 1 140 | ||
| N2H4→N2 | hdh | Hydrazine dehydrogenase | PF00815.20, PF13561.6, PF00106.25, PF13447.6, PF14537.6 | 2 | 59 | 33 | 3 | |
| 注:−:数据库未检索到该基因. Note: −: The gene was not found in the database. | ||||||||
(1) KEGG数据库(版本号93.0,2020年1月1日)线上网页中,对6种氮循环途径进行了单独分类,共收录相关功能基因41种,共计序列22 970条。KEGG基于同源基因具有相似功能的假设,将同源的所有基因归为一类,其中narG、narZ与nxrA为同源基因,narH、narY与nxrB为同源基因,narI与narV为同源基因,amoABC分别与甲烷氧化功能基因pmoABC为同源基因。
(2) NCBI-NR数据库(2019年2月14日)共计121.5 Gb,包含序列1.98亿条,可抽取得到氮循环功能基因48种,共计序列85 971条。显而易见,本地构建NCBI-NR数据库非常占用存储空间,而且氮循环基因序列仅占数据库序列总数的0.04%,用户比对分析时相当耗时。
(3) UniProt数据库(版本号2019_09,2019年10月16日)可利用基因名称检索并批量下载得到氮循环功能基因49种,共计序列213 500条,相较其他数据库,其收录基因数量和序列数量最多。
(4) NCycDB数据库(2019年7月29日)共计106.6 Mb,包含序列219 146条,可抽取得到相关功能基因44种,共计序列134 341条。由此可见,人工构建的氮循环基因数据库NCycDB相较于大型综合数据库更具有针对性,而且大大缩短了用户下载和比对的时间。
本文将从NCBI-NR、UniProt和NCycDB数据库中下载的氮循环功能基因序列合并去冗余后,与Pfam数据库比对得到各功能基因的HMMs信息。基于HMMER[46],发现各功能基因占比50%及以上的比对结果与KEGG和FunGene数据库中已给出的部分氮循环功能基因的HMMs信息相近,因此以50%为截止值筛选得出每种功能基因的HMMs信息。
2.2.2 氮循环各途径功能基因收录情况根据表 2计算可知,从本文统计的4个常用数据库中所收录的无机氮循环各途径功能基因序列数量来看,同化硝酸盐还原作用中nasA、narB和nirA基因序列数量相对较多,占该途径所有基因序列总量的85.21%;异化硝酸盐还原作用中nirB、nirD和nrfA基因序列数量相对较多,占该途径所有基因序列总量的78.19%;反硝化作用中norB和nosZ基因序列数量相对较多,占该途径所有基因序列总量的96.61%;固氮作用中仅nifH基因序列数量就占该途径所有基因序列总量的78.75%;硝化作用中仅amoA基因序列数量就占该途径所有基因序列总量的96.89%;厌氧氨氧化作用中hzsA、hzsB和hzo基因序列数量相对较多,占该途径所有基因序列总量的93.79% (以上计算未包含同时作用于多种途径的功能基因,如narG等)。
narGHJI、narZYWV和napABC基因可同时作用于反硝化与异化硝酸盐还原作用的第一步反应(NO3−→NO2−),nirKS基因可同时作用于反硝化与厌氧氨氧化作用的第一步反应(NO2−→NO),上述多用途基因相较其他基因其存在更为普遍,因此数据库中收录的基因序列数量相对较多,如:在异化硝酸盐还原作用中,多用途基因序列占序列总量的59.67%;在反硝化作用中,多用途基因序列占序列总量的73.95%;在厌氧氨氧化作用中,多用途基因序列占序列总量的88.85%。考虑到多用途基因可同时表征多种作用途径,因此本文对多用途基因进行了单独分析:反硝化与异化硝酸盐还原作用的的第一步反应中,narG、narH和napA基因序列数量相对较多,占该途径所有基因序列总量的58.79%;反硝化与厌氧氨氧化作用的第一步反应中,nirS相较于nirK基因序列数量更多,占比57.04%。
3 数据库在氮循环基因注释中的选择及应用方式氮循环普遍存在于海洋、废水、饮用水、林地、农田甚至动物肠道等多种环境中,本文利用搜索引擎NCBI PubMed、Google Scholar和ScienceDirect,设置关键词‘nitrogen’ and ‘gene (genome)’ and ‘database’ and ‘marine (ocean, sea, etc.)’ or ‘freshwater (river, lake, groundwater, etc.)’ or ‘wastewater’ or ‘reactor’ or ‘pond’ or ‘reservoir (drinking water)’ or ‘soil (land, forest, basin, grass, hillslope, riparian, farm, etc.)’ or ‘microorganism (algae, bacteria, archaea, etc.)’,检索汇总了2018年以来应用微生物基因数据库注释不同环境中氮循环功能基因的引用率较高的相关文献52篇,并根据文献内容从多种角度分析了在进行氮循环功能基因注释时,影响研究人员选择数据库的主要因素及数据库的具体应用方式。具体统计结果见图 1。
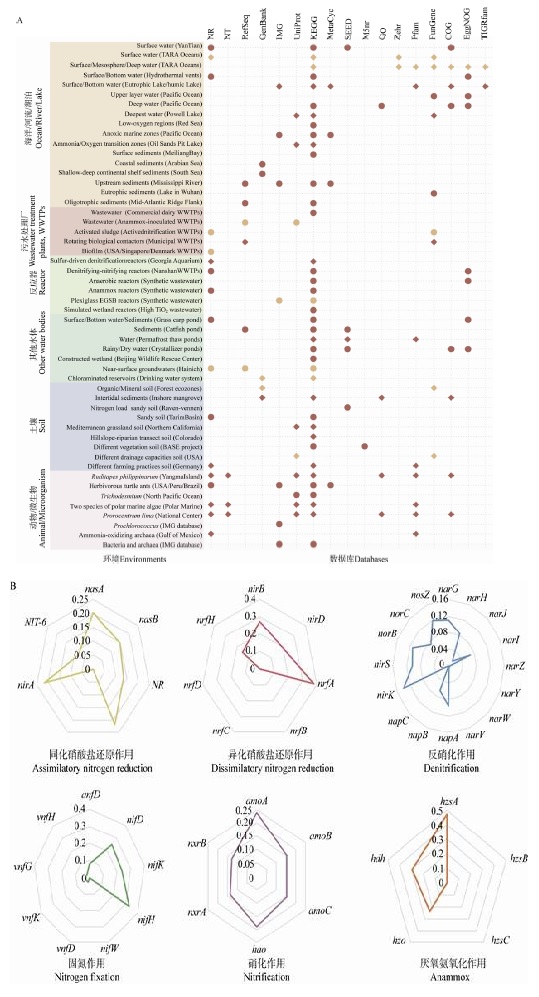
|
| 图 1 文献统计图 Figure 1 Figure of literature statistics 注:A:文献中氮循环功能基因注释所比对数据库,黄色/红色分别表示有/无重构数据库,菱形/圆形分别表示有/无进行蛋白质结构域比对,NR、NT、RefSeq和GenBank均属于NCBI数据库;B:文献中氮循环各途径功能基因研究频率. Note: A: The applications of databases in the annotation of nitrogen cycle functional genes in the literatures; Yellow/red indicates yes/no reconstructed databases; Diamond/circle indicates yes/no aligned protein domains; NR, NT, RefSeq and GenBank belong to NCBI database; B: Frequency of functional genes of different nitrogen cycle pathways in the literatures. |
|
|
本文统计的环境介质主要包括:表层[47-51]、上层[52]、深层[53]和底层[54]等不同地理位置和深度范围,以及低氧[55]、缺氧[56]和氨氧过度区[57]等不同氨氧含量的海洋/河流/湖泊的水体及沉积物[58-63];污水处理厂[64-68]和实验室反应器[69-74]等废水处理系统;养殖场[75-76]、池塘[77-78]、人工湿地[79]、地下水[80]和饮用水[81]等其他水体;林地[82-83]、湿地[84]、盆地[85]、草原[86]、山区[87]和农田[88-90]等土壤环境;菲律宾蛤仔(Ruditapes philippinarum)[91]、分化龟蚁(Cephalotes varians)[92]、藻类[93-96]、细菌和古菌[97-98]等动物及微生物体内。根据图 1A可知,研究人员在注释不同环境介质中氮循环功能基因时所比对的数据库并无规律可循,因此,环境介质对数据库选择无显著影响。
3.2 表征基因对数据库选择的影响根据图 1B可知,narB、nirA和nasA基因多用于表征同化硝酸盐还原作用,nrfA和nirB基因多用于表征异化硝酸盐还原作用,nosZ、norB和多用途基因nirK、narG多用于表征反硝化作用,nifH基因多用于表征固氮作用,amoA和hao基因多用于表征硝化作用,hzsA基因多用于表征厌氧氨氧化作用。值得注意的是,氨氧化菌中的hao基因[99]与厌氧氨氧化菌中的hdh基因都是八面体血红素羟胺氧化还原酶(octaheme hydroxylamine oxidoreductase)的同系物[100],因此hao有时被同时用于表征好氧和厌氧细菌的氨氧化作用[60, 65, 71, 75]。
当研究人员利用nifH基因专注于分析固氮作用时,通常会用到FunGene、Zehr或康奈尔大学的nifH基因数据库[48-49, 52, 82]。当研究人员利用amoA基因表征硝化作用的AOB和Comammox时,考虑到目前数据库未对两种作用菌的基因序列明确分类,通常选择自行补充下载最新报道的Comammox基因组序列,或结合FunGene数据库中的HMMs区分两种作用菌[66, 68, 89]。当研究人员同时分析多种氮循环途径时,数据库的选择不受各途径表征基因的影响,反而表征基因的选择往往与数据库收录基因情况相关[54, 74, 80, 98]。根据表 2和图 1B可知,研究人员在选择氮循环各途径表征基因时主要依据KEGG数据库,因为KEGG数据库中未收录的narC、nrfBCD、napC和nifW基因,在所有文献中均没有被用于表征氮循环作用。
3.3 分析方法对数据库选择的影响本文汇总的52篇文献中,研究人员所采用的分析方法主要包括:16S/18S rRNA基因、宏基因组和宏转录组分析。其中,24篇文献同时提取了微生物16S/18S rRNA基因序列和全基因组DNA序列,先利用16S/18S rRNA基因分析进行序列分类、系统发育和种群结构分析,再通过宏基因组分析进行物种/功能基因注释和代谢途径分析;15篇文献仅利用宏基因组分析进行功能基因注释、代谢途径和物种多样性分析;10篇提取了微生物全基因组RNA序列,利用宏转录组分析或同时结合3种分析方法深入解析了微生物在特定环境或时期的基因表达情况。以上文献均采用了高通量测序(包括16S/18S rRNA基因测序、宏基因组鸟枪测序和宏转录组测序)技术,根据序列长度、碱基质量、错配率、基因组完整度和污染度等条件过滤数据。此外,还有3篇文献提取了微生物功能基因的PCR产物,根据克隆文库测序结果,结合16S rRNA基因或宏基因组分析注释功能基因[59-60, 66]。
值得注意的是,Phylogenetic Investigation of Communities by Reconstruction of Unobserved States (PICRUSt)能够扩展16S rRNA基因分析功能,基于操作分类单元(operational taxonomic units,OTU)表预测细菌宏基因组的功能基因组成[101]。如:Lai等[64]根据16S rRNA基因测序数据,分析了污水处理厂3个渗滤阶段脱氮相关的微生物组成和功能基因丰度变化,基于Greengenes数据库创建参考序列OTU表,利用PICRUSt将OTU表转换为KEGG直系同源基因信息表,获取氮循环功能基因相对丰度。由于Greengenes数据库更新于2013年,而且PICRUSt无法预测Greengenes数据库中没有同源参考基因组序列的物种,因此该方法虽有一定意义,但存在很大局限性和不确定性,无法与真正的宏基因组分析相提并论[101]。
根据52篇文献统计可知,16S/18S rRNA基因分析比对的数据库主要包括SILVA和Greengenes;宏基因组分析比对的数据库主要包括KEGG、NCBI、UniProt、IMG、FunGene、Pfam、COG和EggNOG;宏转录组分析比对的数据库主要包括KEGG、NCBI、Swiss-Prot、COG、GO和Pfam。虽然不同分析方法比对的数据库有所不同,但采用一种分析方法或多种分析方法,对数据库的选择并无显著影响,并且所有文献均利用宏基因组或宏转录组方法进行氮循环功能基因的注释,两种方法所用数据库基本相同。
3.4 比对方法对数据库选择的影响研究人员在进行氮循环功能基因注释时,采用的比对方法主要分为:序列相似性比对和蛋白质结构域比对,不同比对方法使用的微生物基因数据库类型不同。
3.4.1 序列相似性比对根据图 1A可知,KEGG和NCBI数据库在52篇文献中的使用率分别达到75%和59.62%,是研究人员进行序列相似性比对的首选;在NCBI子数据库中NR数据库应用最广,占比为54.84%。考虑到综合数据库中存在未经筛选的基因序列和错误的注释信息,仅使用一种数据库注释氮循环功能基因易产生许多错误信息,76.92%的研究人员同时使用了几种综合数据库对基因组序列进行基因预测和注释,或结合特定功能类型的基因数据库进行比对注释。如:Zhou等[47]同时使用KEGG、COG、SEED和NCBI-NR数据库进行氮循环功能基因注释,探究了海洋链状裸甲藻(Gymnodinium catenatum)赤潮过程中藻际微生物的功能特征。Hu等[92]为证实分化龟蚁肠道共生体细菌能否为宿主固氮,先利用NCBI-NR数据库进行基因分类注释,再根据KEGG和MetaCyc数据库手动构建了分化龟蚁体内氮降解和氨基酸合成途径。Li等[50]为分析浅水生态系统中微生物对碳、硫和氮循环的介导作用,首先将基因组序列与NCBI-NR和KEGG数据库进行相似性比对完成基因功能注释,而未注释的序列,则通过与EggNOG、碳水化合物活性酶(carbohydrate active enzyme)和抗生素抗性基因(antibiotic resistance genes)等特定功能类型数据库比对以获取更多信息。
此外,研究人员有时先筛选出属于氮循环的功能基因,再进行详细的物种和基因注释。如:Black等[61]为得出密西西比河上游沉积物中贻贝(Unionoida)聚集对氮循环基因丰度和组成的影响,首先根据ChocoPhlAn泛基因组数据库比对结果,快速注释泛微生物基因组的功能信息,随后利用MetaCyc和KEGG数据库中氮代谢功能模块注释代谢通路信息;对于确定在氮代谢通路中具有丰度差异的功能基因,利用NCBI-RefSeq数据库比对判定其起源物种,而NCBI-RefSeq中未明确分类的amoA基因,则通过与IMG数据库中参考序列的多序列比对,判定其属于AOB还是Comammox。
3-4-2 蛋白质结构域比对根据图 1A可知,36.54%的研究人员在序列相似性比对的基础上,结合HMMs进行蛋白质结构域的比对分析。其中,FunGene和Pfam数据库在氮循环功能结构域比对上应用最广。蛋白质结构域比对的具体应用情境如下:
(1) 在序列相似性比对注释功能基因前,先使用HMMs筛选基因组序列。如:Diamond等[86]为解析地中海草原土壤生态系统中土壤深浅和降雨量等对未知基因组微生物特征的影响,根据UniProt数据库自定义了HMMs,筛选得到10 158个核糖体蛋白(ribosomal protein,rp) S3序列,随后根据KEGG数据库对rpS3序列进行识别、聚类和多样性分析。
(2) 区分真核、原核和古细菌的基因序列。如:Lavy等[87]使用86个已发表的HMMs和KEGG数据库中KofamKOALA工具,鉴定了真核、原核和古细菌rpS3蛋白序列,并分别注释了山坡-河岸带土壤微生物中参与碳、氮和硫循环的基因。Orellana等[89]在UniProt数据库中抽取得到了古菌和细菌的amoA、hao、nxrA、narG、nirK、nirS、norB、nosZ和nrfA基因序列,并利用FunGene数据库的HMMs进一步检验氮循环序列,分析了不同排水特性土壤对氮肥的响应情况。
(3) 鉴定综合数据库中未收录的功能基因类别。如:Haas等[54]将鲍威尔湖冰期以来海底各层地质微生物的宏基因组数据集,与KEGG数据库比对计算氮循环相关功能基因丰度,而KEGG数据库中未包含的基因,则利用FunGene和UniProt数据库中的HMMs检索鉴定,最终发现了高铵浓度下的固氮作用和化变层潜在的微需氧硝化作用。
3.4.3 基因数据库的重构根据图 1A可知,19.23%的研究人员在单独或结合使用上述两种比对方法的基础上,重新构建数据库以便于更好地分析研究。研究人员重构基因数据库的一般步骤为:首先在综合数据库中下载目标基因的参考序列,随后通过同一性聚类去除冗余序列,最后构建系统发育树筛选所需序列。如:Yang等[65]先从NCBI和UniProtKB/ Swiss-Prot数据库中下载了氮循环相关的所有氨基酸序列,随后使用USEARCH以80%的同一性对所有序列聚类,通过IQ-TREE进行系统发育分析,并与NCBI-RefSeq数据库比对进一步鉴定序列功能,最后用确定属于氮循环功能基因的序列重新构建数据库,以准确分析污水处理厂在直接接种外源Anammox颗粒后的脱氮过程。
在重构基因数据库时,研究人员往往先使用HMMs过滤下载的相关蛋白质序列,以确保数据库的准确性。因此,FunGene数据库是研究人员参考的首选,因为其具有明确的氮循环功能基因分类和蛋白质结构域信息。对于氮循环中唯一具有针对性数据库的固氮作用基因nifH,研究人员在重构数据库时均使用了Zehr等数据库中的参考序列;对于其他无针对性数据库的基因(近两年以Comammox的amoA基因为主),研究人员则根据最新报道补充添加相关的基因组序列。如:Cardenas等[82]为探究北美森林生态圈采伐过程中有机物去除对氮循环基因相对丰度的影响,在康奈尔大学的nifH基因数据库和NCBI-GenBank数据库中检索下载了氮循环相关蛋白质序列,并根据FunGene数据库中的HMMs拟合过滤古菌和细菌的amoA基因序列,自定义了代表固氮(nifH)、硝化(古菌和细菌amoA)、反硝化(nirK、nirS、norB和nosZ)和异化硝酸盐还原作用(nrfA)的氮循环关键酶基因数据库。Salazar等[49]为探究nifH基因的生物地理分布,利用FunGene和Zehr数据库中的nifH基因序列,以及Farnelid数据库中的表层海洋nifH基因扩增子序列重构固氮基因数据库,将海洋微生物参考基因集(Ocean Microbial Reference Gene Catalog,OM-RGC.v2)中检测得到的24个nifH基因序列与之比对,以重新注释编码nifH物种的相对基因和转录丰度。Wang等[66]为比较硝化作用活跃的污水处理厂中AOB和Comammox的基因丰度,利用已报道的4个全长的Comammox amoA基因序列与NCBI-NR数据库比对后,下载相似度较高的基因序列,并以99.5%的同一性聚类,将去冗余后的序列与FunGene数据库中变形杆菌(Proteobacterial)的代表性amoA和pmoA基因序列构建系统发育树,仅保留在Comammox amoA谱系内的序列构建Comammox基因数据库。
4 总结与展望目前,已有大量研究人员通过检测氮循环功能基因的丰度和多样性,分析微生物氮代谢过程中的菌群结构和互作关系。随着基因测序技术的发展,环境中重要功能基因序列数量迅速增长,蛋白质功能结构域的比对受到广泛关注[49, 77, 87]。虽然已存在多种不同功能的综合数据库,但大多都有数据量过大、不便于按功能搜索、不利于本地构建和无法实现比对结果可视化的问题。NCycDB等小型氮循环基因数据库近年来才有所构建,而且大多无法自动更新,不具备线上比对、物种注释等功能。根据本文统计结果可知,UniProt数据库相较其他数据库,收录氮循环功能基因数量和序列数量最多。根据2018−2020年52篇文献中研究人员进行氮循环功能基因注释时对数据库的选择和应用方式可知:
(1) 环境介质、表征基因对研究人员选择数据库无显著影响,KEGG数据库反而是研究人员选择氮循环各途径表征基因的主要依据。
(2) 采用一种或多种分析方法对研究人员选择数据库无显著影响。研究人员多采用宏基因组或宏转录组方法进行氮循环功能基因注释,两种方法所用数据库主要包括:KEGG、NCBI、UniProt、Pfam、FunGene、COG和EggNOG。
(3) 比对方法是影响研究人员选择数据库的主要因素。KEGG和NCBI数据库是研究人员进行序列相似性比对的首选,FunGene和Pfam数据库在蛋白质结构域比对时应用最广。综合数据库中存在未经筛选的基因序列和错误的注释信息,用其注释氮循环功能基因易产生许多错误信息。对此,研究人员应用数据库时的解决方法为:1)在序列相似性比对时,结合使用几种综合数据库或特定功能类型的数据库;2)在序列相似性比对的基础上,结合使用蛋白质结构域比对,预筛选基因组序列、判定数据库中未收录基因的类别和鉴别真核、原核及古细菌基因序列等;3)在单独或结合使用上述两种比对方法的基础上,根据FunGene、Zehr和最新报道的基因组序列重构数据库。
由于微生物基因数据库会不断更新,本文的统计结果和所得结论均限于2020年2月以前的各数据库收录情况。接下来,各大综合数据库在收录基因信息和进行自动注释时,应将序列相似性比对和蛋白质结构域比对更有效地结合,并将目前硝化作用中未明确分类的AOB和Comammox基因序列详细区分。此外,建立一个自动化、可视化的专注于研究氮循环功能基因的数据库平台势在必行,这将对预测环境工程系统中菌间关系,调控和解决氮环境污染起到至关重要的作用。
| [1] |
Kuypers MMM, Marchant HK, Kartal B. The microbial nitrogen-cycling network[J]. Nature Reviews Microbiology, 2018, 16(5): 263-276. DOI:10.1038/nrmicro.2018.9 |
| [2] |
Liu JG, Liu WG. Advances in microbial-mediated nitrogen cycling[J]. Acta Agrestia Sinica, 2018, 26(2): 277-283. (in Chinese) 刘建国, 刘卫国. 微生物介导的氮循环过程研究进展[J]. 草地学报, 2018, 26(2): 277-283. DOI:10.11733/j.issn.1007-0435.2018.02.002 |
| [3] |
Eveillard D, Bouskill NJ, Vintache D, et al. Probabilistic modeling of microbial metabolic networks for integrating partial quantitative knowledge within the nitrogen cycle[J]. Frontiers in Microbiology, 2019, 9: 3298. DOI:10.3389/fmicb.2018.03298 |
| [4] |
Pasin F, Menzel W, Daròs JA. Harnessed viruses in the age of metagenomics and synthetic biology: an update on infectious clone assembly and biotechnologies of plant viruses[J]. Plant Biotechnology Journal, 2019, 17(6): 1010-1026. DOI:10.1111/pbi.13084 |
| [5] |
Hiraoka S, Yang CC, Iwasaki W. Metagenomics and bioinformatics in microbial ecology: current status and beyond[J]. Microbes and Environments, 2016, 31(3): 204-212. DOI:10.1264/jsme2.ME16024 |
| [6] |
Sayers EW, Cavanaugh M, Clark K, et al. GenBank[J]. Nucleic Acids Research, 2019, 47(D1): D94-D99. DOI:10.1093/nar/gky989 |
| [7] |
O'Leary NA, Wright MW, Brister JR, et al. Reference sequence (RefSeq) database at NCBI: current status, taxonomic expansion, and functional annotation[J]. Nucleic Acids Research, 2016, 44(D1): D733-D745. DOI:10.1093/nar/gkv1189 |
| [8] |
Yu K, Zhang T. Construction of customized sub-databases from NCBI-nr database for rapid annotation of huge metagenomic datasets using a combined BLAST and MEGAN approach[J]. PLoS One, 2013, 8(4): e59831. DOI:10.1371/journal.pone.0059831 |
| [9] |
Chen IMA, Chu K, Palaniappan K, et al. IMG/M v.5.0: an integrated data management and comparative analysis system for microbial genomes and microbiomes[J]. Nucleic Acids Research, 2019, 47(D1): D666-D677. DOI:10.1093/nar/gky901 |
| [10] |
Pundir S, Martin MJ, O'Donovan C. UniProt protein knowledgebase[A]//Wu C H, Arighi C N, Ross K E. Protein Bioinformatics[M]. New York: Humana Press, 2017: 41-55
|
| [11] |
The UniProt Consortium. UniProt: a worldwide hub of protein knowledge[J]. Nucleic Acids Research, 2019, 47(D1): D506-D515. DOI:10.1093/nar/gky1049 |
| [12] |
Boutet E, Lieberherr D, Tognolli M, et al. UniProtKB/Swiss-Prot, the manually annotated section of the UniProt knowledgebase: how to use the entry view[A]. Edwards D. Plant Bioinformatics[M]. New York: Humana Press, 2016: 23-54
|
| [13] |
Christensen H, de Vries LE. Databases and protein structures[A]. Christensen H. Introduction to Bioinformatics in Microbiology[M]. Cham: Springer, 2018: 25-50
|
| [14] |
Kanehisa M, Sato Y, Kawashima M, et al. KEGG as a reference resource for gene and protein annotation[J]. Nucleic Acids Research, 2016, 44(D1): D457-D462. DOI:10.1093/nar/gkv1070 |
| [15] |
Caspi R, Billington R, Ferrer L, et al. The MetaCyc database of metabolic pathways and enzymes and the BioCyc collection of pathway/genome databases[J]. Nucleic Acids Research, 2016, 44(D1): D471-480. DOI:10.1093/nar/gkv1164 |
| [16] |
Overbeek R, Olson R, Pusch GD, et al. The SEED and the Rapid Annotation of microbial genomes using Subsystems Technology (RAST)[J]. Nucleic Acids Research, 2014, 42(D1): D206-D214. DOI:10.1093/nar/gkt1226 |
| [17] |
Wilke A, Harrison T, Wilkening J, et al. The M5nr: a novel non-redundant database containing protein sequences and annotations from multiple sources and associated tools[J]. BMC Bioinformatics, 2012, 13: 141. DOI:10.1186/1471-2105-13-141 |
| [18] |
Arnold R, Goldenberg F, Mewes HW, et al. SIMAP-the database of all-against-all protein sequence similarities and annotations with new interfaces and increased coverage[J]. Nucleic Acids Research, 2014, 42(D1): D279-D284. DOI:10.1093/nar/gkt970 |
| [19] |
The Gene Ontology Consortium. The gene ontology resource: 20 years and still GOing strong[J]. Nucleic Acids Research, 2019, 47(D1): D330-D338. DOI:10.1093/nar/gky1055 |
| [20] |
Finn RD, Coggill P, Eberhardt RY, et al. The Pfam protein families database: towards a more sustainable future[J]. Nucleic Acids Research, 2016, 44(D1): D279-D285. DOI:10.1093/nar/gkv1344 |
| [21] |
Fish JA, Chai B, Wang Q, et al. FunGene: the functional gene pipeline and repository[J]. Frontiers in Microbiology, 2013, 4: 291. |
| [22] |
Galperin MY, Kristensen DM, Makarova KS, et al. Microbial genome analysis: the COG approach[J]. Briefings in Bioinformatics, 2019, 20(4): 1063-1070. DOI:10.1093/bib/bbx117 |
| [23] |
Huerta-Cepas J, Szklarczyk D, Forslund K, et al. EggNOG 4.5: a hierarchical orthology framework with improved functional annotations for eukaryotic, prokaryotic and viral sequences[J]. Nucleic Acids Research, 2016, 44(D1): D286-293. DOI:10.1093/nar/gkv1248 |
| [24] |
Letunic I, Bork P. 20 years of the SMART protein domain annotation resource[J]. Nucleic Acids Research, 2018, 46(D1): D493-D496. DOI:10.1093/nar/gkx922 |
| [25] |
Haft DH, Selengut JD, Richter RA, et al. TIGRFAMs and genome properties in 2013[J]. Nucleic Acids Research, 2013, 41(D1): D387-D395. |
| [26] |
Marchler-Bauer A, Bo Y, Han LY, et al. CDD/SPARCLE: functional classification of proteins via subfamily domain architectures[J]. Nucleic Acids Research, 2017, 45(D1): D200-D203. DOI:10.1093/nar/gkw1129 |
| [27] |
Tu QC, Lin L, Cheng L, et al. NCycDB: a curated integrative database for fast and accurate metagenomic profiling of nitrogen cycling genes[J]. Bioinformatics, 2019, 35(6): 1040-1048. DOI:10.1093/bioinformatics/bty741 |
| [28] |
Heller P, Tripp HJ, Turk-Kubo K, et al. ARBitrator: a software pipeline for on-demand retrieval of auto-curated nifH sequences from GenBank[J]. Bioinformatics, 2014, 30(20): 2883-2890. DOI:10.1093/bioinformatics/btu417 |
| [29] |
Frank IE, Turk-Kubo KA, Zehr JP. Rapid annotation of nifH gene sequences using classification and regression trees facilitates environmental functional gene analysis[J]. Environmental Microbiology Reports, 2016, 8(5): 905-916. DOI:10.1111/1758-2229.12455 |
| [30] |
Gaby JC, Buckley DH. A comprehensive aligned nifH gene database: a multipurpose tool for studies of nitrogen-fixing bacteria[J]. Database, 2014, 2014: bau001. DOI:10.1093/database/bau001 |
| [31] |
Zhang XY, Peng DC, Wan Q, et al. Dominant factors of dissimilatory nitrate reduction to ammonia (DNRA) in activated sludge system: a comment[J]. Advances in Environmental Protection, 2018, 8(2): 95-105. (in Chinese) 张新艳, 彭党聪, 万琼, 等. 活性污泥中硝酸盐异化还原成铵(DNRA)过程及其影响因素[J]. 环境保护前沿, 2018, 8(2): 95-105. |
| [32] |
Castro-Barros CM, Jia MS, van Loosdrecht MCM, et al. Evaluating the potential for dissimilatory nitrate reduction by anammox bacteria for municipal wastewater treatment[J]. Bioresource Technology, 2017, 233: 363-372. DOI:10.1016/j.biortech.2017.02.063 |
| [33] |
Nelson MB, Martiny AC, Martiny JB. Global biogeography of microbial nitrogen-cycling traits in soil[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(29): 8033-8040. DOI:10.1073/pnas.1601070113 |
| [34] |
Wang J, Bao JT, Li XR, et al. Molecular ecology of nifH genes and transcripts along a chronosequence in revegetated areas of the tengger desert[J]. Microbial Ecology, 2016, 71(1): 150-163. DOI:10.1007/s00248-015-0657-9 |
| [35] |
Soni R, Suyal DC, Sai S, et al. Exploration of nifH gene through soil metagenomes of the western Indian Himalayas[J]. 3 Biotech, 2016, 6(1): 25. DOI:10.1007/s13205-015-0324-3 |
| [36] |
Gao JF, Fan XY, Pan KL, et al. Diversity, abundance and activity of ammonia-oxidizing microorganisms in fine particulate matter[J]. Scientific Reports, 2016, 6: 38785. DOI:10.1038/srep38785 |
| [37] |
Neal AL, Glendining MJ. Calcium exerts a strong influence upon phosphohydrolase gene abundance and phylogenetic diversity in soil[J]. Soil Biology and Biochemistry, 2019, 139: 107613. DOI:10.1016/j.soilbio.2019.107613 |
| [38] |
Costa E, Pérez J, Kreft JU. Why is metabolic labour divided in nitrification?[J]. Trends in Microbiology, 2006, 14(5): 213-219. DOI:10.1016/j.tim.2006.03.006 |
| [39] |
Daims H, Lebedeva EV, Pjevac P, et al. Complete nitrification by Nitrospira bacteria[J]. Nature, 2015, 528(7583): 504-509. DOI:10.1038/nature16461 |
| [40] |
van Kessel MAHJ, Speth DR, Albertsen M, et al. Complete nitrification by a single microorganism[J]. Nature, 2015, 528(7583): 555-559. DOI:10.1038/nature16459 |
| [41] |
Pinto AJ, Marcus DN, Ijaz UZ, et al. Metagenomic evidence for the presence of comammox Nitrospira-like bacteria in a drinking water system[J]. mSphere, 2016, 1(1): e00054-15. DOI:10.1128/msphere.00054-15 |
| [42] |
Lawson CE, Wu S, Bhattacharjee AS, et al. Metabolic network analysis reveals microbial community interactions in anammox granules[J]. Nature Communications, 2017, 8: 15416. DOI:10.1038/ncomms15416 |
| [43] |
Wu Y, Wang YX, de Costa YG, et al. The co-existence of anammox genera in an expanded granular sludge bed reactor with biomass carriers for nitrogen removal[J]. Applied Microbiology and Biotechnology, 2019, 103(3): 1231-1242. |
| [44] |
Keren R, Lawrence JE, Zhuang WQ, et al. Increased replication of dissimilatory nitrate-reducing bacteria leads to decreased anammox bioreactor performance[J]. Microbiome, 2020, 8: 7. DOI:10.1186/s40168-020-0786-3 |
| [45] |
Carreño NU, Nielsen PH, Willoughby A, et al. Modelling the selective retention of PAOs and Nitrospira (comammox?) in a full-scale implementation of WAS hydrocyclones at the Ejby Mølle WWTP[J]. Proceedings of the Water Environment Federation, 2017(7): 4079-4084. |
| [46] |
Finn RD, Clements J, Eddy SR. HMMER web server: interactive sequence similarity searching[J]. Nucleic Acids Research, 2011, 39(S2): W29-W37. |
| [47] |
Zhou J, Zhang BY, Yu K, et al. Functional profiles of phycospheric microorganisms during a marine dinoflagellate bloom[J]. Water Research, 2020, 173: 115554. DOI:10.1016/j.watres.2020.115554 |
| [48] |
Delmont TO, Quince C, Shaiber A, et al. Nitrogen-fixing populations of Planctomycetes and Proteobacteria are abundant in surface ocean metagenomes[J]. Nature Microbiology, 2018, 3(7): 804-813. DOI:10.1038/s41564-018-0176-9 |
| [49] |
Salazar G, Paoli L, Alberti A, et al. Gene expression changes and community turnover differentially shape the global ocean metatranscriptome[J]. Cell, 2019, 179(5): 1068-1083.E21. DOI:10.1016/j.cell.2019.10.014 |
| [50] |
Li YF, Tang K, Zhang LB, et al. Coupled carbon, sulfur, and nitrogen cycles mediated by microorganisms in the water column of a shallow-water hydrothermal ecosystem[J]. Frontiers in Microbiology, 2018, 9: 2718. DOI:10.3389/fmicb.2018.02718 |
| [51] |
Linz AM, He SM, Stevens SLR, et al. Freshwater carbon and nutrient cycles revealed through reconstructed population genomes[J]. PeerJ, 2018, 6(4): e6075. |
| [52] |
Li YY, Chen XH, Xie ZX, et al. Bacterial diversity and nitrogen utilization strategies in the upper layer of the northwestern pacific ocean[J]. Frontiers in Microbiology, 2018, 9: 797. DOI:10.3389/fmicb.2018.00797 |
| [53] |
Xu L, Sun C, Huang MM, et al. Complete genome sequence of Euzebya sp. DY32-46, a marine Actinobacteria isolated from the Pacific Ocean[J]. Marine Genomics, 2019, 44: 65-69. DOI:10.1016/j.margen.2018.09.008 |
| [54] |
Haas S, Desai DK, LaRoche J, et al. Geomicrobiology of the carbon, nitrogen and sulphur cycles in Powell Lake: a permanently stratified water column containing ancient seawater[J]. Environmental Microbiology, 2019, 21(10): 3927-3952. DOI:10.1111/1462-2920.14743 |
| [55] |
Kharbush JJ, Thompson LR, Haroon MF, et al. Hopanoid-producing bacteria in the Red Sea include the major marine nitrite oxidizers[J]. FEMS Microbiology Ecology, 2018, 94(6): fiy063. DOI:10.1093/femsec/fiy063 |
| [56] |
Plominsky AM, Trefault N, Podell S, et al. Metabolic potential and in situ transcriptomic profiles of previously uncharacterized key microbial groups involved in coupled carbon, nitrogen and sulfur cycling in anoxic marine zones[J]. Environmental Microbiology, 2018, 20(8): 2727-2742. DOI:10.1111/1462-2920.14109 |
| [57] |
Mori JF, Chen LX, Jessen GL, et al. Putative mixotrophic nitrifying-denitrifying gammaproteobacteria implicated in nitrogen cycling within the ammonia/oxygen transition zone of an oil sands pit lake[J]. Frontiers in Microbiology, 2019, 10: 2435. DOI:10.3389/fmicb.2019.02435 |
| [58] |
Li Y, Sun Y, Zhang HJ, et al. The responses of bacterial community and N2O emission to nitrogen input in lake sediment: estrogen as a co-pollutant[J]. Environmental Research, 2019, 179: 108769. DOI:10.1016/j.envres.2019.108769 |
| [59] |
Jabir T, Jesmi Y, Vipindas PV, et al. Diversity of nitrogen fixing bacterial communities in the coastal sediments of southeastern Arabian Sea (SEAS)[J]. Deep Sea Research Part II: Topical Studies in Oceanography, 2018, 156: 51-59. DOI:10.1016/j.dsr2.2018.09.010 |
| [60] |
Zhou ZC, Chen J, Gu WJ, et al. Biogeographic pattern of the nirS gene-targeted anammox bacterial community and composition in the northern South China Sea and a coastal Mai Po mangrove wetland[J]. Applied Microbiology and Biotechnology, 2020, 104(7): 3167-3181. DOI:10.1007/s00253-020-10415-3 |
| [61] |
Black EM, Chimenti MS, Just CL. Metagenomic analysis of nitrogen-cycling genes in upper Mississippi river sediment with mussel assemblages[J]. MicrobiologyOpen, 2019, 8(5): e00739. DOI:10.1002/mbo3.739 |
| [62] |
Yin XJ, Chen LJ, Tang DQ, et al. Seasonal and vertical variations in the characteristics of the nitrogen-related functional genes in sediments from urban eutrophic lakes[J]. Applied Soil Ecology, 2019, 143: 80-88. DOI:10.1016/j.apsoil.2019.05.027 |
| [63] |
Reese BK, Zinke LA, Sobol MS, et al. Nitrogen cycling of active bacteria within oligotrophic sediment of the mid-atlantic ridge flank[J]. Geomicrobiology Journal, 2018, 35(6): 468-483. DOI:10.1080/01490451.2017.1392649 |
| [64] |
Lai E, Hess M, Mitloehner FM. Profiling of the microbiome associated with nitrogen removal during vermifiltration of wastewater from a commercial dairy[J]. Frontiers in Microbiology, 2018, 9: 1964. DOI:10.3389/fmicb.2018.01964 |
| [65] |
Yang YC, Pan J, Zhou ZC, et al. Complex microbial nitrogen-cycling networks in three distinct anammox-inoculated wastewater treatment systems[J]. Water Research, 2020, 168: 115142. DOI:10.1016/j.watres.2019.115142 |
| [66] |
Wang MY, Huang GH, Zhao ZR, et al. Newly designed primer pair revealed dominant and diverse comammox amoA gene in full-scale wastewater treatment plants[J]. Bioresource Technology, 2018, 270: 580-587. DOI:10.1016/j.biortech.2018.09.089 |
| [67] |
Spasov E, Tsuji JM, Hug LA, et al. High functional diversity among Nitrospira populations that dominate rotating biological contactor microbial communities in a municipal wastewater treatment plant[J]. The ISME Journal, 2020, 14(7): 1857-1872. DOI:10.1038/s41396-020-0650-2 |
| [68] |
Annavajhala MK, Kapoor V, Santo-Domingo J, et al. Structural and functional interrogation of selected biological nitrogen removal systems in the United States, Denmark, and Singapore using shotgun metagenomics[J]. Frontiers in Microbiology, 2018, 9: 2544. DOI:10.3389/fmicb.2018.02544 |
| [69] |
Burns AS, Padilla CC, Pratte ZA, et al. Broad phylogenetic diversity associated with nitrogen loss through sulfur oxidation in a large public marine aquarium[J]. Applied and Environmental Microbiology, 2018, 84(20): e01250-18. |
| [70] |
Miao J, Shi YH, Zeng DF, et al. Enhanced shortcut nitrogen removal and metagenomic analysis of functional microbial communities in a double sludge system treating ammonium-rich wastewater[J]. Environmental Technology, 2020, 41(14): 1877-1887. DOI:10.1080/09593330.2018.1551432 |
| [71] |
Zhao YP, Feng Y, Chen LM. Genome-centered omics insight into the competition and niche differentiation of Ca. Jettenia and Ca. Brocadia affiliated to anammox bacteria[J]. Applied Microbiology and Biotechnology, 2019, 103(19): 8191-8202. |
| [72] |
Meng YB, Huang LN, Meng FG. Metagenomics response of anaerobic ammonium oxidation (anammox) bacteria to bio-refractory humic substances in wastewater[J]. Water, 2019, 11(2): 365. |
| [73] |
Li W, Zhuang JL, Zhou YY, et al. Metagenomics reveals microbial community differences lead to differential nitrate production in anammox reactors with differing nitrogen loading rates[J]. Water Research, 2020, 169: 115279. DOI:10.1016/j.watres.2019.115279 |
| [74] |
Yang XY, Chen Y, Guo FC, et al. Metagenomic analysis of the biotoxicity of titanium dioxide nanoparticles to microbial nitrogen transformation in constructed wetlands[J]. Journal of Hazardous Materials, 2020, 384: 121376. DOI:10.1016/j.jhazmat.2019.121376 |
| [75] |
Deng M, Hou J, Song K, et al. Community metagenomic assembly reveals microbes that contribute to the vertical stratification of nitrogen cycling in an aquaculture pond[J]. Aquaculture, 2020, 520: 734911. DOI:10.1016/j.aquaculture.2019.734911 |
| [76] |
Nho SW, Abdelhamed H, Paul D, et al. Taxonomic and functional metagenomic profile of sediment from a commercial catfish pond in Mississippi[J]. Frontiers in Microbiology, 2018, 9: 2855. DOI:10.3389/fmicb.2018.02855 |
| [77] |
Fernandez L, Bertilsson S, Peura S. Non-cyanobacterial diazotrophs dominate nitrogen-fixing communities in permafrost thaw ponds[J]. Limnology and Oceanography, 2020, 65(S1: S180-S193). |
| [78] |
Couto-Rodríguez RL, Montalvo-Rodríguez R. Temporal analysis of the microbial community from the crystallizer ponds in cabo rojo, puerto rico, using metagenomics[J]. Genes, 2019, 10(6): 422. DOI:10.3390/genes10060422 |
| [79] |
Li J, Wang JT, Hu HW, et al. Changes of the denitrifying communities in a multi-stage free water surface constructed wetland[J]. Science of the Total Environment, 2019, 650: 1419-1425. DOI:10.1016/j.scitotenv.2018.09.123 |
| [80] |
Wegner CE, Gaspar M, Geesink P, et al. Biogeochemical regimes in shallow aquifers reflect the metabolic coupling of the elements nitrogen, sulfur, and carbon[J]. Applied and Environmental Microbiology, 2019, 85(5): e02346-18. |
| [81] |
Potgieter SC, Dai ZH, Venter SN, et al. Microbial nitrogen metabolism in chloraminated drinking water reservoirs[J]. mSphere, 2020, 5(2): e00274-20. |
| [82] |
Cardenas E, Orellana LH, Konstantinidis KT, et al. Effects of timber harvesting on the genetic potential for carbon and nitrogen cycling in five North American forest ecozones[J]. Scientific Reports, 2018, 8(1): 3142. DOI:10.1038/s41598-018-21197-0 |
| [83] |
Zhu BT, Zhang XB, Zhao CG, et al. Comparative genome analysis of marine purple sulfur bacterium Marichromatium gracile YL28 reveals the diverse nitrogen cycle mechanisms and habitat-specific traits[J]. Scientific Reports, 2018, 8(1): 17803. DOI:10.1038/s41598-018-36160-2 |
| [84] |
Hester ER, Harpenslager SF, van Diggelen JMH, et al. Linking nitrogen load to the structure and function of wetland soil and rhizosphere microbial communities[J]. mSystems, 2018, 3(1): e00214-17. |
| [85] |
Ren M, Zhang ZF, Wang XL, et al. Diversity and contributions to nitrogen cycling and carbon fixation of soil salinity shaped microbial communities in Tarim Basin[J]. Frontiers in Microbiology, 2018, 9: 431. DOI:10.3389/fmicb.2018.00431 |
| [86] |
Diamond S, Andeer PF, Li Z, et al. Mediterranean grassland soil C-N compound turnover is dependent on rainfall and depth, and is mediated by genomically divergent microorganisms[J]. Nature Microbiology, 2019, 4(8): 1356-1367. DOI:10.1038/s41564-019-0449-y |
| [87] |
Lavy A, McGrath DG, Carnevali PBM, et al. Microbial communities across a hillslope-riparian transect shaped by proximity to the stream, groundwater table, and weathered bedrock[J]. Ecology and Evolution, 2019, 9(12): 6869-6900. DOI:10.1002/ece3.5254 |
| [88] |
Finn D, Yu JL, Penton CR. Soil quality shapes the composition of microbial community stress response and core cell metabolism functional genes[J]. Applied Soil Ecology, 2020, 148: 103483. DOI:10.1016/j.apsoil.2019.103483 |
| [89] |
Orellana LH, Chee-Sanford JC, Sanford RA, et al. Year-round shotgun metagenomes reveal stable microbial communities in agricultural soils and novel ammonia oxidizers responding to fertilization[J]. Applied and Environmental Microbiology, 2017, 84(2): e01646-17. |
| [90] |
Nelkner J, Henke C, Lin TW, et al. Effect of long-term farming practices on agricultural soil microbiome members represented by Metagenomically Assembled Genomes (MAGs) and their predicted plant-beneficial genes[J]. Genes, 2019, 10(6): 424. DOI:10.3390/genes10060424 |
| [91] |
Cong M, Wu HF, Cao TF, et al. Digital gene expression analysis in the gills of Ruditapes philippinarum exposed to short- and long-term exposures of ammonia nitrogen[J]. Aquatic Toxicology, 2018, 194: 121-131. DOI:10.1016/j.aquatox.2017.11.012 |
| [92] |
Hu Y, Sanders JG, Łukasik P, et al. Author correction: herbivorous turtle ants obtain essential nutrients from a conserved nitrogen-recycling gut microbiome[J]. Nature Communications, 2018, 9(1): 2440. DOI:10.1038/s41467-018-04935-w |
| [93] |
Frischkorn KR, Haley ST, Dyhrman ST. Coordinated gene expression between Trichodesmium and its microbiome over day-night cycles in the North Pacific Subtropical Gyre[J]. The ISME Journal, 2018, 12(4): 997-1007. DOI:10.1038/s41396-017-0041-5 |
| [94] |
Mckie-Krisberg ZM, Sanders RW, Gast RJ. Evaluation of mixotrophy-associated gene expression in two species of polar marine algae[J]. Frontiers in Marine Science, 2018, 5: 273. DOI:10.3389/fmars.2018.00273 |
| [95] |
Hou DY, Mao XT, Gu S, et al. Systems-level analysis of metabolic mechanism following nitrogen limitation in benthic dinoflagellate Prorocentrum lima[J]. Algal Research, 2018, 33: 389-398. DOI:10.1016/j.algal.2018.06.004 |
| [96] |
Berube PM, Rasmussen A, Braakman R, et al. Emergence of trait variability through the lens of nitrogen assimilation in Prochlorococcus[J]. eLife, 2019, 8: e41043. DOI:10.7554/eLife.41043 |
| [97] |
Kitzinger K, Padilla CC, Marchant HK, et al. Cyanate and urea are substrates for nitrification by Thaumarchaeota in the marine environment[J]. Nature Microbiology, 2019, 4(2): 234-243. DOI:10.1038/s41564-018-0316-2 |
| [98] |
Albright MBN, Timalsina B, Martiny JBH, et al. Comparative genomics of nitrogen cycling pathways in bacteria and archaea[J]. Microbial Ecology, 2019, 77(3): 597-606. DOI:10.1007/s00248-018-1239-4 |
| [99] |
Kozlowski JA, Stieglmeier M, Schleper C, et al. Pathways and key intermediates required for obligate aerobic ammonia-dependent chemolithotrophy in bacteria and Thaumarchaeota[J]. The ISME Journal, 2016, 10(8): 1836-1845. DOI:10.1038/ismej.2016.2 |
| [100] |
Maalcke WJ, Reimann J, de Vries S, et al. Characterization of Anammox hydrazine dehydrogenase, a key N2-producing enzyme in the global nitrogen cycle[J]. Journal of Biological Chemistry, 2016, 291(33): 17077-17092. DOI:10.1074/jbc.M116.735530 |
| [101] |
Langille MGI, Zaneveld J, Caporaso JG, et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J]. Nature biotechnology, 2013, 31(9): 814-821. DOI:10.1038/nbt.2676 |
 2020, Vol. 47
2020, Vol. 47