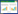扩展功能
文章信息
- 彭玙萍, 曾伟民
- PENG Yu-Ping, ZENG Wei-Min
- 紫金山铜矿酸性矿山废水微生物群落多样性
- Diversity of microbial community in acid mine drainage from Zijinshan copper mine
- 微生物学通报, 2020, 47(9): 2887-2896
- Microbiology China, 2020, 47(9): 2887-2896
- DOI: 10.13344/j.microbiol.china.200279
-
文章历史
- 收稿日期: 2020-03-23
- 接受日期: 2020-08-27
- 网络首发日期: 2020-09-02
2. 生物冶金教育部重点实验室 湖南 长沙 410083
2. Key Laboratory of Biometallurgy, Ministry of Education, Changsha, Hunan 410083, China
金属矿产资源的开发利用过程中,由于硫化矿的氧化会产生大量的酸性废水,包括矿井水、矿坑水、尾矿淋滤水和生物堆浸产生的浸出液等。这些废水的pH低且含有大量的重金属离子[1]。酸性矿山环境广泛分布于全球范围内,如美国的Iron Mountain和西班牙的Rio Tinto River,以及国内的紫金山和大宝山等都是典型的酸性矿山废水区域[2-3]。
微生物在酸性矿山废水的形成过程中起到了重要的作用。一些微生物能够氧化二价铁或还原性无机硫化物来获得能量,从而加速了硫化矿物的溶解。典型的矿水废水中的微生物包括严格自养的酸硫杆菌属(Acidithiobacillus)和钩端螺旋菌属(Leptospirillum),以及可异养生长的硫化杆菌属(Sulfobacillus)、铁质菌属(Ferroplasma)和嗜酸菌属(Acidiphilium)等[4]。目前对酸性矿山废水微生物的研究以原核微生物为主,对于真核微生物仅有少量报道。如Amaral Zettler等发现在Rio Tinto River中,真核藻类的比例较高,占到总生物量的60%以上[5]。研究人员通过构建18S rRNA基因文库发现,Iron Mountain的强酸性废水(pH < 0.9)中存在锁掷酵母(Sporidiobolus pararoseus)以及一些未培养真菌[6]。
酸性矿山废水排放到环境中会对水体和土壤造成污染,是采矿业面临的一个严重的环境问题。为此,人们提出了一些措施来处理酸性矿山废水,包括化学法和生物法等。处理后的矿山废水,其理化性质会发生明显的变化,从而对微生物的生长造成影响,最终将影响整个微生物群落的结构。本研究从紫金山采集了处理前后的矿山废水,分析了处理前后的细菌和真菌群落变化及其与理化参数的关系,以期为矿山废水的处理提供参考指标以及为矿山污染场地的修复提供理论基础。
1 材料与方法 1.1 主要试剂和仪器DNeasy PowerSoil DNA提取试剂盒,Qiagen公司;PCR试剂盒,诺唯赞生物科技有限公司;胶回收纯化试剂盒,天根生化科技(北京)有限公司。pH计,上海仪电科学仪器股份有限公司;PCR仪,北京东胜创新生物科技有限公司;电感耦合等离子体发射光谱仪,Agilent公司。
1.2 样品采集及理化性质测定采集紫金山铜矿3水样,分别为矿坑水、生物堆浸的浸出液及回水。紫金山铜矿设有水处理系统,主要采用中和沉淀法处理含铜酸性溶液,在反应槽中加入石灰乳或液碱进行中和,并加入絮凝剂促进沉淀。部分收集的矿坑水和生物浸出液经萃取和电积处理后产生的废水进入水处理系统。处理后的废水(回水)一部分回用于采矿井下用水(形成矿坑水)、废水处理站用水、生物堆浸用水和碎矿等。每种水样均采集3份,每份10 L,先用定性滤纸过滤除去固体杂质。滤液用0.22 μm的微孔滤膜过滤,用PBS溶液将滤膜上的微生物洗下来,所得菌悬液10 000 r/min离心10 min收集菌体。菌体于-80 ℃保存备用。现场测定水样的温度和pH,以Ag/AgCl电极为参比测定水样的电位。采用电感耦合等离子体发射光谱仪测定过滤后水样的金属离子含量。
1.3 DNA提取及微生物多样性测序采用DNA提取试剂盒提取微生物基因组DNA,该试剂盒采用振荡破碎法,可同时提取细菌和真菌的基因组DNA[7]。采用NanoDrop ND-100测定提取的DNA浓度,琼脂糖凝胶电泳观察提取的DNA完整性。
采用通用引物515F (5′-GTGCCAGCMGCCGCGGTAA-3′)和806R (5′-GGACTACHVGGGTWTCTAAT-3′) PCR扩增细菌和古菌的16S rRNA基因V4区,PCR反应体系和反应条件参照文献[8],采用真菌ITS通用引物ITS3_KYO2 (5′-GATGAAGAACGYAGYRAA-3′)和ITS4 (5′-TCCTCCGCTTATTGATATGC-3′) PCR扩增真菌ITS序列,PCR反应体系和反应条件参照文献[9]。琼脂糖凝胶电泳检测产物,并用胶回收试剂盒回收纯化PCR产物。纯化产物送到罗宁生物科技有限公司采用Illumina HiSeq 2500平台测序。
根据原始序列的质控信息去除低质量的碱基。采用Flash[10]软件将正反向序列进行拼接,用UCHIME[11]检测并去除拼接后序列的嵌合体。使用UPARSE[12]算法在97%的一致性水平上进行OTU聚类,挑选每个OTU中出现频数最高的序列作为OTU的代表序列。使用UCLUST[13]分类法与原核SILVA数据库和真菌UNITE数据库进行物种注释分析。采用R语言中的Vegan[14]软件包计算Shannon指数和Simpson指数。采用Mantel test分析微生物群落结构与环境因子的相关性[15]。
2 结果与分析 2.1 水样理化性质3个样品的理化性质见表 1。矿坑水的pH最低,为1.45,浸出液的pH也低于2.00,回水经过石灰乳和碱液中和后,pH相对于矿坑水和浸出液明显升高,达到4.50以上。回水中的重金属离子含量很低,矿坑水和浸出液的重金属离子浓度很高,其中矿坑水中铜离子含量达到2 608.7 mg/L,总铁离子含量达到7 788.7 mg/L,而浸出液中的总铁含量高达12 143 mg/L。矿坑水和浸出液的电位值明显高于回水。
| 项目Item | S1 | S2 | S3 |
| 酸碱度pH | 4.55±0.01 | 1.45±0.02 | 1.89±0.01 |
| 电位Eh (mV) | 297±3.2 | 576±5.8 | 458±6.9 |
| 铝Al (mg/L) | 1.5±0.6 | 1 486.3±6.7 | 1 876.3±40.2 |
| 砷As (mg/L) | 0.1±0.05 | 35.8±0.6 | 82.7±2.3 |
| 铜Cu (mg/L) | 1.4±0.7 | 2 608.7±11.0 | 1 861.0±47.2 |
| 铁Fe (mg/L) | 1.8±0.5 | 7 788.7±53.5 | 12 143.3±260.1 |
| 锰Mn (mg/L) | 1.8±0.3 | 19.2±0.2 | 23.7±0.5 |
| 锌Zn (mg/L) | 1.9±0.8 | 325.2±3.8 | 325.3±7.4 |
| 注:S1:回水;S2:矿坑水;S3:浸出液.
Note: S1: Back water; S2: Pit water; S3: Leachate from the dump-leaching. | |||
采用HISEQ 2500 PE250分别对原核生物16S rRNA基因V4区和真菌ITS测序。原核微生物V4区测序,S1、S2和S3分别产生33 912、31 748和36 042条有效序列,序列平均长度290 bp,真菌ITS测序,S1、S2和S3分别产生41 779、41 538和42 306条有效序列,序列平均长度为360 bp。原核微生物V4区和真菌ITS测序的覆盖度均达到99.9%以上,表明测序结果已足够代表样品的真实情况。回水、矿坑水和浸出液V4区测序的Shannon指数分别为2.95、2.57和2.76,Simpson指数分别为0.87、0.83和0.87,真菌ITS测序的Shannon指数分别为1.87、0.91和1.27,Simpson指数分别为0.68、0.50和0.53,可见回水的物种多样性要高于矿坑水和浸出液,并且原核微生物的多样性要高于真菌。按照97%的序列相似性划分为一个OTU,原核微生物V4区测序S1、S2和S3分别产生216、216和218个OTU,总OTU数为244个,3组样品共有的OTU有167个,占总OTU数的68.4%,这167个共有OTU的丰度占总生物量的97.3%;真菌ITS测序分别产生133、113和124个OTU,总OTU数为167个,3组样品共有的OTU为72个,占总OTU数的43.1%,这72个共有OTU的丰度占总生物量的90.8%。
2.3 原核微生物群落结构组成3组水样的原核微生物群落结构组成见图 1和图 2。从门水平(图 1)看,样品的原核微生物分布在19个门中,回水中以变形菌门(Proteobacteria)的丰度最高,占比达75.4%,其次为疣微杆菌门(Verrucomicrobia,7.1%)、厚壁菌门(Firmicutes,6.9%)、硝化螺菌门(Nitrospirae,3.5%)和拟杆菌门(Bacteroidetes,2.24%)。矿坑水中的原核微生物丰度最高的是广古菌门(49.81%),丰度较高的有硝化螺菌门(31.85%)、变形菌门(10.3%)和厚壁菌门(4.52%);另外,还有一定比例的奇古菌门(Thaumarchaeota,1.1%)。浸出液中硝化螺菌门(50.9%)丰度最高,比例较高的还有变形菌门(17.9%)、广古菌门(Euryarchaeota,14.3%)和厚壁菌门(14.0%),其他门的微生物丰度均在1%以下。总体来看,回水中原核微生物在门水平的结构组成与矿坑水和浸出液的差别较大,而矿坑水和浸出液的物种类别相似。
|
| 图 1 样品在门水平上的原核微生物群落结构 Figure 1 Prokaryotic microbial community structure of the samples on the phylum level |
|
|
|
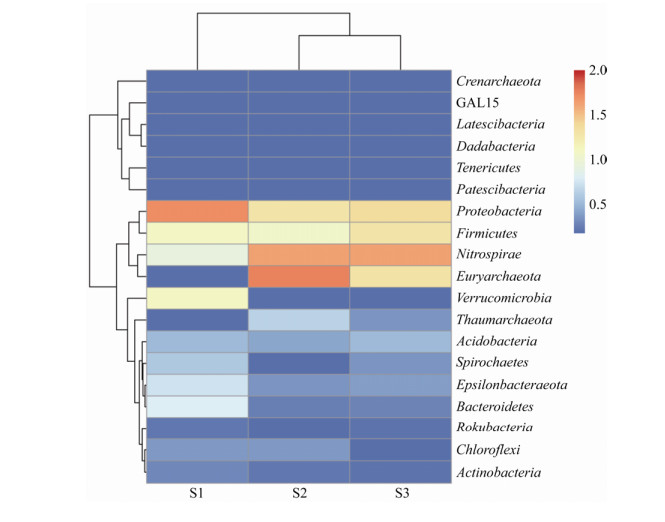
| 图 2 样品在属水平上的原核微生物群落结构 Figure 2 Prokaryotic microbial community structure of the samples on the genus level |
|
|
从属水平(图 2)看,回水中丰度最高的是噬氢菌属(Hydrogenophaga),比例为29.6%,其次为硫幡菌属(Thiovirga,12.4%)和新鞘脂菌属(Novosphingobium,10.8%)。典型极端嗜酸菌的丰度很低,如酸硫杆菌属(Acidithiobacillus,0.23%)、嗜酸菌属(Acidiphilium,0.63%)和硫化杆菌(Sulfobacillus,0.45%),但是钩端螺旋菌(Leptospirillum)有较高的丰度(3.5%)。矿坑水中钩端螺旋菌是优势菌(31.8%),古菌的比例较高,属于古菌的A-plasma、铁质菌属(Ferroplasma)和酸质菌属(Acidiplasma)的丰度分别为16.3%、6.2%和1.3%。值得注意的是,一种Unclassified Ferroplasmaceae的相对丰度达到17.02%,表明该水样中可能存在新的Ferroplasmaceae物种。古菌在矿坑水中的比例较高,可能是因为古菌如铁质菌属等对pH的耐受性很强[16]。此外,酸硫杆菌也有较高的比例,相对丰度为7.5%。与矿坑水相似,浸出液中的优势菌也是钩端螺旋菌属,但是相对丰度更高,为50.9%。钩端螺旋菌属在很多酸性矿山废水中都是优势类群[17]。嗜酸菌属的相对丰度为16.1%,硫化杆菌(Sulfobacillus)为10.4%。A-plasma (6.9%)、铁质菌属(3.1%)和酸质菌属(1.9%)也有一定的比例,但是酸硫杆菌属的比例极低(0.07%)。
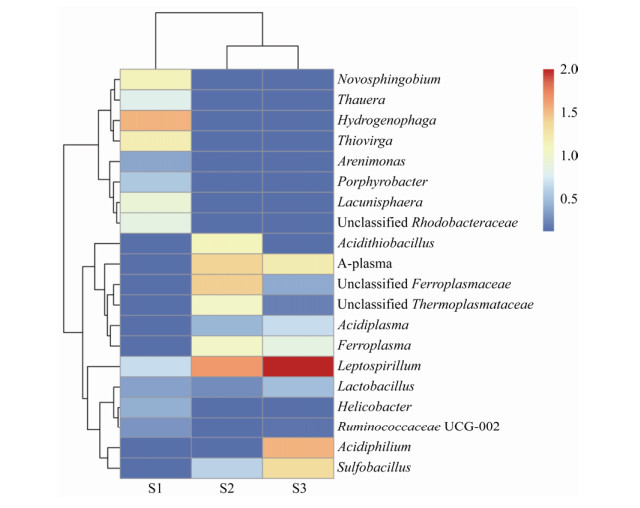
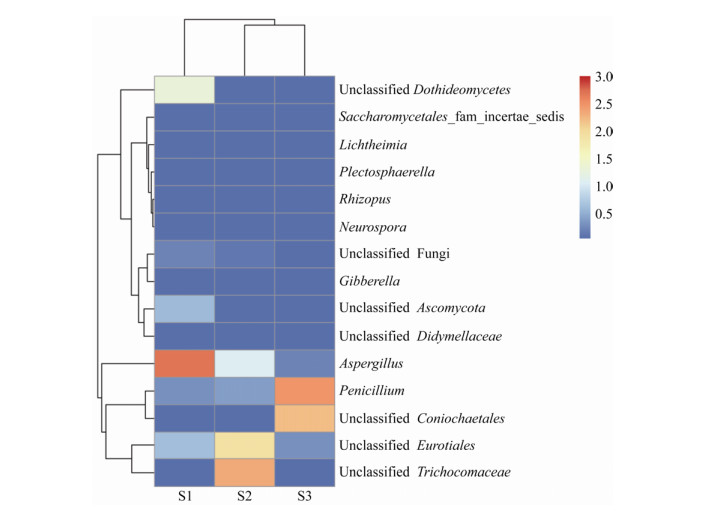
2.4 真菌群落结构组成水样的真菌群落结构组成见图 3和图 4。从门水平(图 3)看,3组样品的差别不明显,子囊菌门(Ascomycota)为绝对优势类群,在3组样品中的丰度分别为98.2%、99.4%和98.9%,可能是因为Ascomycota对酸性环境的耐受较强。从属水平(图 4)看,回水和矿坑水及浸出液的真菌群落结构差别较大。回水中相对丰度最高的为曲霉属(Aspergillus,54.6%),其次为轮枝菌属(Verticillium,23.1%),而顶囊壳属(Gaeumannomyces,2.8%)、外瓶霉属(Exophiala,1.6%)和青霉属(Penicillium,1.1%)也有一定的比例,其他属的物种相对丰度均在1%以下。在矿坑水中,未培养真菌的相对丰度很高,其中Unclassified Trichocomaceae为优势类群,其相对丰度达到64.1%,而来自Eurotiales的未培养真菌相对丰度为26.7%。曲霉和青霉的相对丰度分别为4.3%和1.0%。浸出液中,青霉为优势类群,相对丰度为61.8%;其次为Unclassified Coniochaetales,相对丰度为35.4%,其他类群的相对丰度均很低。

|
| 图 3 样品在门水平上的真菌群落结构 Figure 3 Fungal community structure of the samples on the phylum level |
|
|
|
| 图 4 样品在属水平上的真菌群落结构 Figure 4 Fungal community structure of the samples on the genus level |
|
|
水样环境因子与相对丰度最高的20个原核菌属的相关性见表 2。从表 2可以看出,pH、Al、Mn和Zn与群落组成的相关性最强,而Eh、As、Cu和Fe与群落结构的相关性较弱。pH与Hydrogenophaga、Thiovirga、Novosphingobium、Lacunisphaera、Porphyrobacter、Unclassified Rhodobacteraceae、Thauera、Helicobacter、Arenimonas及Ruminococcaceae UCG-002的相对丰度显著正相关,而Al、Mn、Zn与这些菌属的相对丰度显著负相关。Al、As、Fe、Mn与Leptospirillum的相对丰度显著正相关,Eh与A-plasma和Ferroplasma的相对丰度显著正相关。
| 属Genus | 酸碱度 pH r/(p) |
电位 Eh r/(p) |
铝 Al r/(p) |
砷 As r/(p) |
铜 Cu r/(p) |
铁 Fe r/(p) |
锰 Mn r/(p) |
锌 Zn r/(p) |
| Hydrogenophaga | 0.990 (0.010) |
-0.848 (0.152) |
-0.982 (0.019) |
-0.819 (0.181) |
-0.942 (0.058) |
-0.938 (0.062) |
-0.983 (0.017) |
-1.000 (0.000 1) |
| Thiovirga | 0.991 (0.009) |
-0.852 (0.148) |
-0.980 (0.020) |
-0.815 (0.185) |
-0.945 (0.055) |
-0.936 (0.064) |
-0.982 (0.019) |
-1.000 (0.000 2) |
| Novosphingobium | 0.990 (0.010) |
-0.850 (0.150) |
-0.981 (0.019) |
-0.817 (0.183) |
-0.943 (0.057) |
-0.937 (0.063) |
-0.982 (0.018) |
-1.00 (0.000 1) |
| Lacunisphaera | 0.990 (0.010) |
-0.849 (0.151) |
-0.981 (0.019) |
-0.818 (0.182) |
-0.943 (0.057) |
-0.938 (0.062) |
-0.983 (0.017) |
-1.000 (0.000 1) |
| Unclassified Rhodobacteraceae | 0.990 (0.010) |
-0.850 (0.150) |
-0.981 (0.019) |
-0.817 (0.184) |
-0.944 (0.056) |
-0.937 (0.063) |
-0.982 (0.018) |
-1.000 (0.000 2) |
| Thauera | 0.989 (0.011) |
-0.846 (0.154) |
-0.982 (0.018) |
-0.821 (0.179) |
-0.941 (0.059) |
-0.939 (0.061) |
-0.983 (0.017) |
-1.000 (0.000) |
| Leptospirillum | -0.857 (0.143) |
0.583 (0.417) |
0.979 (0.022) |
0.969 (0.031) |
0.741 (0.259) |
0.999 (0.001) |
0.975 (0.025) |
0.924 (0.076) |
| Porphyrobacter | 0.991 3 (0.009) |
-0.854 (0.146) |
-0.980 (0.021) |
-0.813 (0.187) |
-0.946 (0.054) |
-0.935 (0.065) |
-0.981 (0.019) |
-1.000 (0.000 2) |
| Helicobacter | 0.991 (0.009) |
-0.854 (0.146) |
-0.980 (0.020) |
-0.813 (0.187) |
-0.946 (0.054) |
-0.935 (0.065) |
-0.981 (0.019) |
-1.000 (0.000 3) |
| Arenimonas | 0.989 (0.011) |
-0.845 (0.155) |
-0.983 (0.017) |
-0.822 (0.178) |
-0.940 (0.060) |
-0.940 (0.060) |
-0.984 (0.016) |
-1.000 (0.000 1) |
| Lactobacillus | 0.893 (0.107) |
-0.933 (0.067) |
-0.717 (0.283) |
-0.440 (0.560) |
-0.934 (0.066) |
-0.607 (0.393) |
-0.718 (0.282) |
-0.832 (0.168) |
| Ruminococcaceae UCG-002 |
0.995 (0.006) |
-0.867 (0.133) |
-0.973 (0.027) |
-0.800 (0.202) |
-0.954 (0.046) |
-0.924 (0.076) |
-0.975 (0.025) |
-0.999 (0.001) |
| Acidiphilium | -0.435 (0.565) |
0.040 (0.960) |
0.704 (0.296) |
0.927 (0.073) |
0.251 (0.750) |
0.809 3 (0.191) |
0.694 (0.306) |
0.565 (0.435) |
| Sulfobacillus | -0.488 (0.512) |
0.104 (0.896) |
0.699 (0.301) |
0.921 (0.079) |
0.312 (0.689) |
0.784 (0.216) |
0.681 (0.319) |
0.603 (0.397) |
| Acidithiobacillus | -0.448 (0.552) |
0.766 (0.234) |
0.133 (0.867) |
-0.279 (0.721) |
0.614 (0.386) |
-0.029 (0.972) |
0.147 (0.854) |
0.308 9 (0.691) |
| A-plasma | -0.788 (0.213) |
0.970 (0.030) |
0.552 (0.448) |
0.155 (0.846) |
0.894 (0.106) |
0.411 (0.589) |
0.565 (0.435) |
0.687 (0.313) |
| Unclassified Ferroplasmaceae | -0.504 (0.496) |
0.807 (0.193) |
0.196 (0.804) |
-0.220 (0.781) |
0.664 (0.336) |
0.036 (0.964) |
0.210 (0.790 |
0.369 (0.632) |
| Acidiplasma | -0.873 (0.127) |
0.727 (0.273) |
0.926 (0.074) |
0.775 (0.225) |
0.819 (0.181) |
0.908 (0.092) |
0.935 (0.065) |
0.898 (0.102) |
| Unclassified Thermoplasmataceae | -0.542 (0.458) |
0.832 (0.168) |
0.240 (0.760) |
-0.176 (0.824) |
0.696 (0.304) |
0.080 (0.920) |
0.254 (0.747 |
0.410 (0.590) |
| Ferroplasma | -0.824 (0.176) |
0.982 (0.019) |
0.611 (0.389) |
0.222 (0.778) |
0.919 (0.081) |
0.478 (0.522) |
0.625 (0.375) |
0.733 (0.267) |
水样环境因子与相对丰度最高的10个真菌菌属的相关性见表 3。其中,pH与Unclassified Ascomycota、Unclassified Didymellaceae、Unclassified Dothideomycetes的相对丰度显著正相关,而Al、Mn、Zn则与上述菌属的相对丰度显著负相关。Al、Fe、Mn与Aspergillus、Unclassified Fungi及Gibberella显著负相关。除此之外,Aspergillus的相对丰度还与pH显著正相关,与Zn显著负相关,Gibberella与Zn显著负相关。Eh、As和Cu与真菌群落组成没有显著相关性。
| 属Genus | 酸碱度 pH r/(p) |
电位 Eh r/(p) |
铝 Al r/(p) |
砷 As r/(p) |
铜 Cu r/(p) |
铁 Fe r/(p) |
锰 Mn r/(p) |
锌 Zn r/(p) |
| Penicillium | -0.451 (0.549) |
0.051 (0.949) |
0.709 (0.291) |
0.939 (0.061) |
0.265 (0.735) |
0.810 (0.190) |
0.695 (0.305) |
0.580 (0.420) |
| Unclassified Coniochaetales | -0.430 (0.570) |
0.072 (0.928) |
0.698 (0.302) |
0.873 (0.127) |
0.263 (0.737) |
0.798 (0.202) |
0.695 (0.305) |
0.552 (0.448) |
| Unclassified Eurotiales | -0.414 (0.586) |
0.742 (0.258) |
0.096 (0.904) |
-0.315 (0.685) |
0.584 (0.416) |
-0.066 (0.934) |
0.110 (0.890) |
0.273 (0.727) |
| Aspergillus | 0.979 (0.021) |
-0.814 (0.186) |
-0.992 (0.008) |
-0.852 (0.148) |
-0.920 (0.080) |
-0.958 (0.042) |
-0.992 (0.008) |
-0.998 (0.002) |
| Unclassified Fungi | 0.854 (0.146) |
-0.600 (0.400) |
-0.974 (0.026) |
-0.939 (0.061) |
-0.748 (0.252) |
-0.990 (0.010) |
-0.974 (0.026) |
-0.916 (0.084) |
| Gibberella | 0.938 (0.062) |
-0.725 (0.275) |
-0.100 (0.000) |
-0.911 (0.089) |
-0.854 (0.146) |
-0.980 (0.010) |
-0.999 (0.001) |
-0.978 (0.022) |
| Unclassified Trichocomaceae | -0.464 (0.536) |
0.778 (0.222) |
0.151 (0.850) |
-0.262 (0.738) |
0.629 (0.371) |
-0.010 (0.990) |
0.165 (0.835) |
0.326 (0.674) |
| Unclassified Ascomycota | 0.990 (0.010) |
-0.849 (0.151) |
-0.981 (0.012) |
-0.817 (0.183) |
-0.943 (0.057) |
-0.938 (0.062) |
-0.983 (0.017) |
-1.000 (0.000 1) |
| Unclassified Didymellaceae | 0.977 (0.023) |
-0.829 (0.171) |
-0.987 (0.013) |
-0.826 (0.174) |
-0.926 (0.074) |
-0.950 (0.050) |
-0.990 (0.010) |
-0.992 (0.008) |
| Unclassified Dothideomycetes | 0.989 (0.011) |
-0.846 (0.154) |
-0.982 (0.018) |
-0.82 (0.179) |
-0.941 (0.059) |
-0.940 (0.060) |
-0.983 (0.017) |
-1.000 (0.000 1) |
矿山废水因其pH极低且含有高浓度的重金属离子,如果不经处理而任意排放,将对周边的生态造成危害,如造成鱼类、藻类等水生生物死亡,破坏土壤结构使土壤板结,造成金属管道的腐蚀等[18]。因此,矿山企业在排放废水之前需采用各种方法去除重金属离子及中和废水pH。经处理后的废水除了以各种理化参数来评价处理效果外,还可以考虑用微生物群落结构作为监测水体污染的指标[19]。
本研究采集了紫金山铜矿矿坑水和浸出液,这两组样品的pH很低、重金属离子浓度很高,符合典型的酸性矿山废水的特征。同时采集了中和沉淀处理后的水样(回水),处理后的水样pH显著升高,而重金属离子已经降至很低的浓度。采用基于16S rRNA基因和真菌ITS测序的技术分析了3组水样的原核微生物和真菌群落结构,回水中的微生物群落组成明显不同于矿坑水和浸出液。矿坑水和浸出液中以钩端螺旋菌等典型极端嗜酸微生物为主,而回水中原核微生物以Hydrogenophaga属为优势菌属,Thiovirga和Novosphingobium也有较高的含量,这些菌属常见于弱酸性或近中性且重金属离子浓度低的环境中,如人工湿地等[20-21]。从门水平看,3组水样中,真菌均以Ascomycota的相对丰度最高。张丽娜等[22]分析了安徽某铁矿酸性矿山废水中真核生物的群落结构,发现Ascomycota普遍存在于水样中,并且在其中2个样品中是优势门类。Auld等[23]则研究发现加拿大Copper Cliff酸性水样中,Ascomycota的相对丰度很低,而在其冬季样品中,Basidiomycota下的Microbotryomycetes含量较高。从属水平看,矿坑水中以未分类的真菌丰度较高,浸出液中Penicillium为优势类群,而回水中Aspergillus为优势类群。Amaral-Zettler[24]分析了Rio Tinto某处酸性水样中的真核类群,发现硅藻Pinnularia的相对丰度最高。Baker等[6]发现Richmond mine中的Sporidiobolus占真核微生物的比例最高(37%),其次为Dothideomycetes IM groups II (22%)。这些结果表明相对于原核微生物,真核微生物在酸性矿山废水中的物种分布较为分散,物种的特异性较低。
从水的流向来看,3种水样之间存在关联,因此三者的微生物群落存在一定的相似性,如Leptospirillum和Ferroplasma等极端嗜酸微生物同样存在于pH > 4.00的回水中。但是,环境因子(重金属离子浓度、pH等)对微生物的选择作用,导致了微生物在3种水中的相对丰度产生了差异。通过分析微生物群落结构与理化参数的相关性发现,pH、Al、Mn、Zn与回水中相对丰度较高的菌属如Hydrogenophaga、Thiovirga和Aspergillus有很强的相关性,而在矿坑水与浸出液中有较高丰度的Acidiphilium、Sulfobacillus、Ferroplasma、Penicillium等菌属与环境因子没有显著的相关性。回水和矿坑水及浸出液中微生物群落结构的明显差异性表明,将一些微生物作为矿山废水处理效果的生物性指标具有可行性。随着极端微生物组计划[25]的深入开展,高通量测序技术的发展和大数据的整合将使我们更全面深入地探究酸性矿山废水微生物的全貌,从而揭示矿山环境微生物演变的普遍规律,结合分子生物学技术,未来可能通过分析微生物的变化来监测矿山的生态状况。随着测序成本的进一步降低,采用微生物群落监测水样污染情况将是一种经济有效的方法,是理化参数检测的有益补充。
4 结论(1) 经过中和沉淀处理的矿山废水(回水) pH明显升高,重金属离子浓度显著降低。回水中微生物的群落结构相对于未处理的矿坑水和浸出液有很大差异。
(2) pH、Al、Mn、Zn对回水的微生物群落结构有显著影响,而这些环境因子对矿坑水和浸出液中的微生物群落结构没有显著影响。微生物的群落变化可以作为衡量矿山废水处理效果的指标。
| [1] |
Ding C, Li Q, Guo CL, et al. Responses of microbial communities in paddy soils to the intrusion of acid mine drainage[J]. Acta Scientiae Circumstantiae, 2019, 39(9): 3080-3089. (in Chinese) 丁翠, 李琦, 郭楚玲, 等. 酸性矿山废水对稻田土壤微生物菌群结构的影响[J]. 环境科学学报, 2019, 39(9): 3080-3089. |
| [2] |
Quatrini R, Johnson DB. Microbiomes in extremely acidic environments: functionalities and interactions that allow survival and growth of prokaryotes at low pH[J]. Current Opinion in Microbiology, 2018, 43: 139-147. DOI:10.1016/j.mib.2018.01.011 |
| [3] |
Kuang JL, Huang LN, Chen LX, et al. Contemporary environmental variation determines microbial diversity patterns in acid mine drainage[J]. The ISME Journal, 2013, 7(5): 1038-1050. DOI:10.1038/ismej.2012.139 |
| [4] |
Méndez-García C, Peláez AI, Mesa V, et al. Microbial diversity and metabolic networks in acid mine drainage habitats[J]. Frontiers in Microbiology, 2015, 6: 475. |
| [5] |
Amaral Zettler LA, Gómez F, Zettler E, et al. Microbiology: Eukaryotic diversity in Spain's River of Fire[J]. Nature, 2002, 417(6885): 137. DOI:10.1038/417137a |
| [6] |
Baker BJ, Tyson GW, Goosherst L, et al. Insights into the diversity of eukaryotes in acid mine drainage biofilm communities[J]. Applied and Environmental Microbiology, 2009, 75(7): 2192-2199. DOI:10.1128/AEM.02500-08 |
| [7] |
Ai CB, Yan Z, Zhou H, et al. Metagenomic insights into the effects of seasonal temperature variation on the activities of activated sludge[J]. Microorganisms, 2019, 7(12): 713. DOI:10.3390/microorganisms7120713 |
| [8] |
Ma LY, Wang XJ, Liu XD, et al. Intensified bioleaching of chalcopyrite by communities with enriched ferrous or sulfur oxidizers[J]. Bioresource Technology, 2018, 268: 415-423. DOI:10.1016/j.biortech.2018.08.019 |
| [9] |
Toju H, Tanabe AS, Yamamoto S, et al. High-coverage its primers for the DNA-based identification of ascomycetes and basidiomycetes in environmental samples[J]. PLos One, 2012, 7(7): e40863. DOI:10.1371/journal.pone.0040863 |
| [10] |
Magoč T, Salzberg SL. FLASH: Fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [11] |
Edgar RC, Haas BJ, Clemente JC, et al. Uchime improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-2200. DOI:10.1093/bioinformatics/btr381 |
| [12] |
Edgar RC. UPARSE: Highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [13] |
Edgar RC. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [14] |
Dixon P. VEGAN, a package of R functions for community ecology[J]. Journal of Vegetation Science, 2003, 14(6): 927-930. DOI:10.1111/j.1654-1103.2003.tb02228.x |
| [15] |
Niu JJ, Deng J, Xiao YH, et al. The shift of microbial communities and their roles in sulfur and iron cycling in a copper ore bioleaching system[J]. Scientific Reports, 2016, 6(1): 34744. DOI:10.1038/srep34744 |
| [16] |
Zhou HB, Zhang R, Hu P, et al. Isolation and characterization of Ferroplasma thermophilum sp. nov., a novel extremely acidophilic, moderately thermophilic archaeon and its role in bioleaching of chalcopyrite[J]. Journal of Applied Microbiology, 2008, 105(2): 591-601. DOI:10.1111/j.1365-2672.2008.03807.x |
| [17] |
Rawlings DE, Tributsch H, Hansford GS. Reasons why 'Leptospirillum'-like species rather than Thiobacillus ferrooxidans are the dominant iron-oxidizing bacteria in many commercial processes for the biooxidation of pyrite and related ores[J]. Microbiology, 1999, 145(1): 5-13. |
| [18] |
Cong ZY, Zhao FH. Current situation and prospect of the research on acid mine drainage[J]. China Mining Magazine, 2003, 12(3): 15-18. (in Chinese) 丛志远, 赵峰华. 酸性矿山废水研究的现状及展望[J]. 中国矿业, 2003, 12(3): 15-18. |
| [19] |
He ZL, Zhang P, Wu LW, et al. Microbial functional gene diversity predicts groundwater contamination and ecosystem functioning[J]. American Society for Microbiology, 2018, 9(1): e02435-02417. |
| [20] |
Cheng LF, Fan LY, Zhang X, et al. Diversity of microbial community in winter at constructed wetlands sediment around coal mining area at Zuoyun, Shanxi Province[J]. Microbiology China, 2019, 46(12): 3181-3192. (in Chinese) 程丽芬, 樊兰英, 张欣, 等. 山西左云县采煤区人工湿地冬季沉积物细菌群落多样性[J]. 微生物学通报, 2019, 46(12): 3181-3192. |
| [21] |
Ito T, Sugita K, Yumoto I, et al. Thiovirga sulfuroxydans gen. nov., sp. nov., a chemolithoautotrophic sulfur-oxidizing bacterium isolated from a microaerobic waste-water biofilm[J]. International Journal of Systematic and Evolutionary Microbiology, 2005, 55(3): 1059-1064. DOI:10.1099/ijs.0.63467-0 |
| [22] |
Zhang LN, Hao CB, Wang LH, et al. Characteristics of the eukaryotic community structure in acid mine drainage lake in Anhui Province, China[J]. Acta Microbiologica Sinica, 2012, 52(7): 875-884. (in Chinese) 张丽娜, 郝春博, 王丽华, 等. 安徽某铁矿酸性矿山废水中真核生物的群落结构特征[J]. 微生物学报, 2012, 52(7): 875-884. |
| [23] |
Auld RR, Mykytczuk NCS, Leduc LG, et al. Seasonal variation in an acid mine drainage microbial community[J]. Canadian Journal of Microbiology, 2017, 63(2): 137-152. DOI:10.1139/cjm-2016-0215 |
| [24] |
Amaral-Zettler LA. Eukaryotic diversity at pH extremes[J]. Frontiers in Microbiology, 2013, 3: 441. |
| [25] |
Liu SJ, Shi WY, Zhao GP. China microbiome initiative: Opportunity and challenges[J]. Bulletin of Chinese Academy of Sciences, 2017, 32(3): 241-250. (in Chinese) 刘双江, 施文元, 赵国屏. 中国微生物组计划:机遇与挑战[J]. 中国科学院院刊, 2017, 32(3): 241-250. |
 2020, Vol. 47
2020, Vol. 47