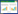扩展功能
文章信息
- 王艳, 郭良栋, 程虎印, 赵乐, 常帆, 万一, 王汉屏
- WANG Yan, GUO Liang-Dong, CHENG Hu-Yin, ZHAO Le, CHANG Fan, WAN Yi, WANG Han-Ping
- 不同生境重楼内生真菌及土壤真菌多样性比较
- Comparison of endophytic and soil fungi of Paris polyphylla diversity from different habitat
- 微生物学通报, 2020, 47(9): 2867-2876
- Microbiology China, 2020, 47(9): 2867-2876
- DOI: 10.13344/j.microbiol.china.200508
-
文章历史
- 收稿日期: 2020-05-25
- 接受日期: 2020-08-19
- 网络首发日期: 2020-08-27
2. 中国科学院微生物研究所 北京 100101;
3. 陕西中医药大学药学院 陕西 西安 712046;
4. 陕西理工大学生物科学与工程学院 陕西 汉中 723001;
5. 西安外事学院医学院 陕西 西安 710077
2. Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
3. College of Pharmacy, Shaanxi University of Chinese Medicine, Xi'an, Shaanxi 712046, China;
4. School of Biological Sciences and Engineering, Shaanxi University of Technology, Hanzhong, Shaanxi 723001, China;
5. College of Medicine, Xi'an International University, Xi'an, Shaanxi 710077, China
植物内生菌(endophyte)是指植物生活史的一定阶段或全部阶段生活于健康植物各种组织和器官内部以及细胞间隙却不引起宿主植物发生病害的一类微生物[1],植物内生菌是影响植物健康和生产力的关键因素之一[2]。微生物组成随土壤来源和植物基因型的不同而变化,同时也可能参与植物的代谢过程[3-5]。药用植物内生菌、根际微生物等植物微生态因素可直接或间接地影响药用植物的生长代谢和化学成分的积累与合成[6],不同地区产的药用植物在品质及效用上往往有巨大的差异[7],这一过程也逐渐被研究者所认识。
像所有高等生物一样,微生物可与植物协同进化并形成特有的处于动态平衡的生态系统,而植物与土壤微生物的相互作用则是植物适应自然环境的关键。植物可以调节其根际营养物质的组成以促进微生物的生长,从而改善特定生态系统中的植物适应性[8],并涉及系统发生相关微生物的选择,不同物种、地理位置、气候和土地管理的植物表现出不同的微生物群落特征[9]。同一种药用植物的次生代谢物含量可能因栽培地生长环境中微生物组成的不同而不同[10-11]。
中草药的品质和效用一直是国内中药材市场关注的热点话题,中草药在种植过程中,微生态则对药材的道地性及优良品质产生着重要影响与贡献[5]。陕西境内的秦巴山区是国内重要的中草药栽培区域,其气候、海拔等自然环境因素均适合中草药的生长。重楼是秦巴山区重要的栽培药用植物之一,本研究通过高通量测序技术对野生及园区环境条件下生长的重楼根际及内生真菌的群落多样性差异进行分析,目的在于了解不同土壤环境对重楼根际及内生真菌的影响,以期为重楼的人工种植提供理论依据,为道地药材仿野生种植中微生态结构的重构提供理论基础。
1 材料与方法 1.1 采样地点样品采集于陕西省汉中市镇巴县大池镇大池社区黎坪小组园区环境及野生环境,样品为药用植物重楼及相关土壤样本,具体样本采集信息见表 1。重楼样本的野生生长环境为林下、阴湿、树木遮光度为70%以上的腐殖土,园区环境为人工管理多年的药园。样地的地理位置坐标为:E107°50′0346″- 107°50′0351″,N32°16′6501″-32°16′6523″。从选定的地点采集生长健康的重楼植株(根、根茎、叶)和相应的根际土及环境土,每个样本生物学重复3个。为保证植株活力,连同根际土及环境土放置于冷链箱中,4 h内带回实验室,待进一步处理。
| 生境 Environment |
样本类型 Type |
样本 Sample |
样本名称 Name |
海拔 Altitude (m) |
| 野生 Wild |
土壤 Soil |
环境土 Environmental soil |
SSSoil | 1 543 |
| 根际土 Rhizosphere soil |
SRSoil | 1 543 | ||
| 植物 Plant |
根 Root |
SRoot | 1 543 | |
| 根茎 Rhizome |
SRhizome | 1 543 | ||
| 叶 Leaf |
SLeaf | 1 543 | ||
| 园区 Garden |
土壤 Soil |
环境土 Environmental soil |
YSSoil | 1 625 |
| 根际土 Rhizosphere soil |
YRSoil | 1 625 | ||
| 植物 Plant |
根 Root |
YRoot | 1 625 | |
| 根茎 Rhizome |
YRhizome | 1 625 | ||
| 叶 Leaf |
YLeaf | 1 625 |
DNA凝胶提取试剂盒,上海百赛生物技术有限公司;Mobio强力土壤DNA提取试剂盒,北京智杰方远科技有限公司;植物基因组DNA提取试剂盒,天根生化科技(北京)有限公司。Illumina HiSeq高通量测序平台,北京诺禾致源生物信息科技有限公司;pH计,Mettler Toledo国际贸易(上海)有限公司;电感耦合原子吸收发光仪,Perkin Elmer (上海)有限公司。
1.3 样品前处理在重楼生长半径10 cm以外、地表以下5 cm处采集环境土。将重楼根部土壤抖落,留存无法抖落的土定义为根际土,根际土壤按照Guo等[12]的描述处理,通过振动平台将根际土振下(20 min,120 r/min),将根际土收集于无菌PE管中。重楼根及根茎部位用流动水清洗,然后分别将根、根茎和叶置于75%的酒精中清洗,时间依次为1.5、2.0和1.0 min,再用5%次氯酸处理3 min,最后用无菌水清洗3次,最终获得表面无菌的重楼根、根茎及叶样本,然后用液氮处理30 s。根际土样本直接用液氮处理,待液氮蒸发完毕后置于-80 ℃超低温冰箱中,待DNA提取。
1.4 土壤理化指标测定方法土壤容重:环刀法[13]。计算公式:容重(g/cm3)=土壤重量/土壤体积。
pH值:取10 g土壤溶于20 mL蒸馏水中,充分搅拌后静置5 min,用pH计测定,每个采样点测定3次,取平均值。
含水量:失重法。取10 g土壤,称重后烘干,待重量不再变化时计算土壤含水量。
土壤矿质元素:电感耦合原子吸收发光仪测定。
1.5 基因组DNA的提取取土壤、根、根茎及叶片每个样本的量约0.5 g用于提取DNA。为了减少DNA提取偏差,提高提取效率,土壤样本和植物组织样本的DNA分别用土壤基因组DNA提取试剂盒和磁珠法植物基因组DNA提取试剂盒提取,其步骤严格按照试剂盒的说明书进行。
1.6 基因组的提取和PCR扩增与测序从土壤样本和植物样本中提取基因组DNA。使用引物ITS1-1F-F (5′-CTTGGTCATTTAGAGGAAGTAA-3′)和ITS1-1F-R (5′-GCTGCGTTCTTCATCGATGC-3′)及ITS5-1737F (5′-GGAAGTAAAAGTCGTAACAAGG-3′)和ITS2-2043R (5′-GCTGCGTTCATCGATGC-3′)对真菌核糖体RNA基因ITS区进行扩增。PCR反应体系(20 μL):5×FastPfu缓冲液4 μL,正、反向引物(5 μmol/L)各0.8 μL,DNA模板10 ng,2.5 mmol/L dNTPs 2 μL,FastPfu聚合酶0.4 μL。PCR反应条件:98 ℃ 1 min;98 ℃ 10 s,50 ℃ 30 s,72 ℃ 30 s,共30个循环;72 ℃ 5 min。PCR扩增产物进行2%琼脂糖凝胶电泳,并从凝胶中提取扩增产物;然后用Axyprep DNA凝胶提取试剂盒进行纯化,用QuantiFluorTM-St进行定量。
根据Illumina基因组DNA文库的制备过程,利用聚合DNA产物构建Illumina对端文库,然后根据标准协议在Illumina HiSeq平台对以上扩增文库进行配对测序。根据标准,原始数据存储在NCBI中(SRA:PRJNA634059)。
1.7 数据处理与统计分析利用Fast QC软件对测序结束的原始数据进行质控,去除低质量序列;利用USEARCH(V11) (http://www.drive5.com/usearch/)流程进行后续生物信息学分析。利用USEARCH流程的Fastq Mergepairs方法进行双端序列合并;利用Cutadapt1.18切除引物;通过USEARCH fastq filter对序列进行质控;Fastx Uniques方法进行去冗余并去除Singleton序列(最小参数为8);通过Unoise3算法进行OTU无参聚类;用Otutab生成OTU表格。聚类后的序列利用Unite数据库进行注释,Sintax方法剪切值为0.8;利用分组中最少序列进行抽平,抽平后得到OTU表格。
运用R3.5.1对所得测序数据进行统计分析[14],并利用USEARCH进行α多样性和β多样性的参数统计分析。在α多样性分析中,利用Anova法进行整体差异分析、利用t检验方法进行组间差异分析,对植物根、根茎和叶等不同生态位的物种多样性指数之间的差异进行研究;在β多样性研究中,使用Binary Jaccard算法进行主坐标分析(principal coordinate analysis,PCoA),对不同生态位之间的距离进行度量;使用真菌数据库Release 6.0对菌群群落结构进行门水平和属水平的分类学统计分析[15-16]。另外,基于Binary Jaccard算法进行UPGMA层次聚类分析;利用维恩(Venn)图展示不同生长环境间共有、特有的真菌OTU数目,直观地表现出样品间OTU的重合情况;用LEfSe [Line Discriminant Analysis (LDA) Effect Size]分析寻找不同生长环境中的差异真菌。
2 结果与分析 2.1 环境土壤理化指标分析土壤容重测定结果显示(表 2),野生环境中的土壤容重值低于园区环境,说明野生环境中的土壤更为疏松、有机质更丰富;疏松土壤的土壤颗粒间有着更大的空隙,即可容纳更多水分,通过土壤含水量测定印证了土壤容重值的可靠性;通过对两种立地环境中土壤pH值的测定发现,野生环境土壤的pH值均低于园区环境,这可能是由于野生环境中存在较多腐殖质、同时产酸微生物大量栖息所致;通过对矿质元素氮、磷、钾的比较发现,野生环境中全氮、全磷、全钾含量也均高于园区环境,但速效磷、速效钾含量低于园区环境,速效磷及速效钾的增高可能是由于人工干预的结果。
| 指标 Index |
野生环境 Wild |
园区环境 Garden |
| 容重 Bulk density (g/cm3) |
1.25±0.08 | 1.47±0.07 |
| 含水量 Moisture content (%) |
39.20±2.01 | 27.21±1.92 |
| pH值 pH value |
6.11±0.10 | 6.57±0.29 |
| 有机质 Organic matter (g/kg) |
94.08±5.10 | 68.40±7.25 |
| 速效氮 Available N (mg/kg) |
538.25±45.4 | 396.58±80.3 |
| 速效磷 Available P (mg/kg) |
3.56±0.90 | 50.28±8.22 |
| 速效钾 Available K (mg/kg) |
269.33±11.33 | 540.05±50.43 |
| 全氮 Total N (g/kg) |
4.83±0.21 | 3.32±0.65 |
| 全磷 Total P (g/kg) |
0.64±0.09 | 0.61±0.06 |
| 全钾 Total K (g/kg) |
12.98±1.11 | 11.17±1.37 |
用USEARCH进行α多样性分析,得到了两种环境的ACE指数、Chao1指数、Shannon指数以及Simpson多样性指数,分别用箱线图显示(图 1)。另外,对不同生境进行t检验,*表示0.05 > P > 0.01。用ACE指数与Chao1指数表明野生环境与园区环境真菌群落丰度的差异性,Shannon指数与Simpson指数表示真菌多样性的差异性。结果如图 1所示,在土壤中,野生环境中环境土壤与根际土壤真菌群落丰度及多样性均高于园区环境,其中,野生环境中根际土壤与园区根际土壤真菌的丰度有显著性差异(P < 0.05);在植物中,园区环境中重楼内生真菌丰度和多样性都要略高于野生环境。
|
| 图 1 不同区系真菌群落的α多样性分析 Figure 1 Alpha diversity of the fungal communities in different samples 注:A:ACE指数;B:Chao1指数;C:Shannon指数;D:Simpson指数.箱线图中每个箱体分别显示每组数据集中的1/4位数(25%)、3/4位数(75%)、中值以及最大和最小计算值;实心箱体表示野生环境样本,空心箱体表示园区环境样本;*表示0.05 > P > 0.01.用箱体颜色区分不同区系. Note: A: ACE index; B: Chao1 index; C: Shannon index; D: Simpson index. The box diagram shows the first (25%) and third (75%) quartiles, median values and maximum and minimum observational values in each data set; the solid box represents the wild environment sample, the hollow box represents the graden environment sample; * represents 0.05 > P > 0.01. Distinguish different floras by box diagram color. |
|
|
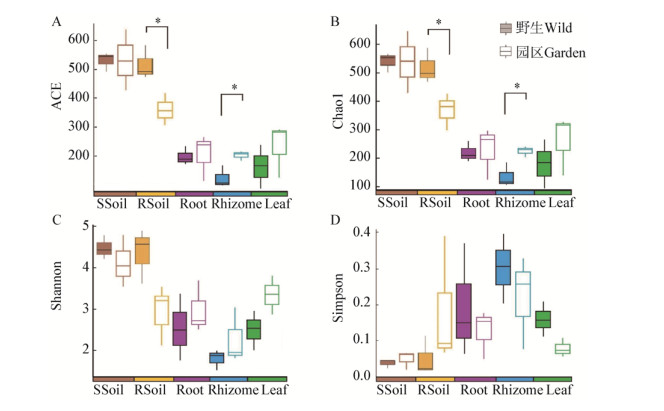
在OTU水平评估两种环境真菌群落的β多样性,比较不同土壤环境中各区系真菌群落结构组成。通过Binary Jaccard相异矩阵算法,进行主坐标分析(PCoA)分析。图 2A显示了样本中真菌群落结构的整体性关系,其中,横坐标解释了关系值的38.66%,纵坐标解释了关系值的12.37%,总解释度为51.03%。PCoA分析结果显示,土壤样本与植物样本有明显的区别,说明土壤中的真菌群落与重楼各部位间有明显差异,但根际土与环境土中的真菌多样性并无较大区别;另外,图 2A中显示园区环境与野生环境真菌群落的β多样性并无明显区分。UPGMA层次聚类分析结果如图 2B所示,植物样本与土壤样本分别形成聚类,不同生长环境间相同区系的进化关系较近,UPGMA层次聚类分析同时印证了Binary Jaccard相异矩阵算法中的PCoA分析结果。另外,从UPGMA层次聚类图中同样可以看出,同一类样本的3个生物重复除野生环境中的一个根样本外,其余都能很好地聚在一起,这同样说明了该分析过程的重复性及可靠性。
|
| 图 2 在OTU水平真菌群落组成PCoA图和UPGMA层次聚类分析图 Figure 2 PCoA map and UPGMA hierarchical cluster analysis of fungal community composition at OTU level 注:PCoA分析与UPGMA层次聚类分析均基于Binary Jaccard算法. A:PCoA分析中,横、纵坐标为样品间差异最大的2个特征值,以百分数的形式体现主要影响程度,PCoA显示样本中真菌群落结构的整体相似性,根际土壤与重楼样本各部分有明显的区分,方形代表采样地为野生环境,圆形代表采样地为园区环境,不同颜色代表真菌不同区系;B:UPGMA层次聚类分析中,样品越靠近,支长越短,说明两个样品的物种组成越相似. Note: Based on the PCoA analysis and Unweighted Pair Group Method with Arithmetic mean (UPGMA) of the binary Jaccard algorithm. A: PCoA analysis, the horizontal and vertical coordinates are the two eigenvalues that lead to the greatest difference between samples, and the main influence degree is reflected as a percentage. PCoA show the similarity of species composition between samples, rhizosphere soil samples were well separated from the plant comparts samples. The square represents the wild environment, the circle represents the garden environment, and the different colors represent the different flora of fungi. B: UPGMA hierarchical cluster analysis, the closer the sample is, the shorter the branch length is, indicating that the species composition of the two samples is more similar. |
|
|
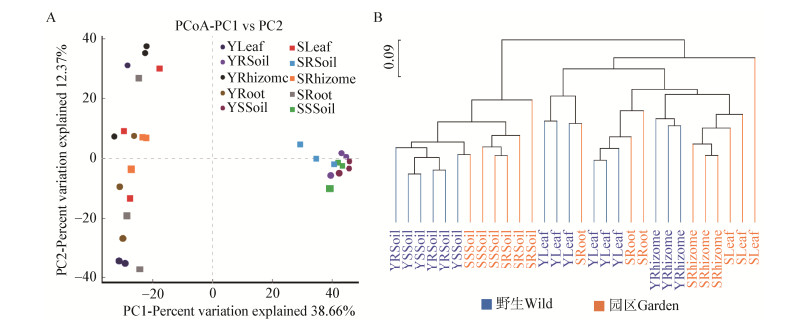
分别在门水平和属水平上统计了不同生长环境的真菌群落组成。如图 3A所示,在门水平上共注释到8个门,分别为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、球囊菌门(Glomeromycota)、被孢霉门(Mortierellomycota)、罗兹菌门(Rozellomycota)、Olpidiomycota、Zoopagomycota和Mucoromycota。该注释结果在野生环境及园区环境中的平均覆盖度分别为75.22%与77.10%,两种环境下主要表现为真菌群落丰度的差异性,多样性并无太大差异。
|
| 图 3 不同生长环境门水平(A)及属水平(B)物种的组成差异 Figure 3 Distribution of OTUs at the phylum (A) and genus (B) levels at different habitat |
|
|
野生环境及园区环境中分别注释到82个和78个属。为清晰显示不同生长环境中真菌群落的相对丰度,在属水平显示前10的属。如图 3B所示,相对丰度前10的属分别为四枝孢属(Tetracladium)、根内球囊霉属(Rhizophagus)、短柄霉属(Aureobasidium)、Inocybe、链格孢属(Alternaria)、Tomentella、油壶菌属(Olpidium)、芽枝霉属(Cladosporium)、外瓶柄霉属(Exophiala)和角担菌属(Ceratobasidium)。由图 3B中可看出各区系真菌群的组成及相对丰度有所差异,丰度最高的是四枝孢属,该属在园区环境较野生环境高37.11%;其次是根内球囊霉属,该属园区中的丰度比野生环境高出89.97%;而短柄霉属在野生环境中的相对丰度是园区环境的3倍。就整体而言,野生环境具有更高的均匀度,而园区环境中菌群具有一定的偏好性,部分菌群表现出更高的优势性。
2.5 不同生长环境间OTU数目Venn分析对不同生长环境的OTU数目进行维恩(Venn)分析[17]。Venn分析结果如图 4所示,交叠部分为共有的OTU数目,野生环境中特有的OTU数目占比为4.04%,园区环境特有的OTU数目占比为4.84%,两种土壤环境共有的OTU数目占比为91.12%。从不同生长环境的分析结果来看,虽然野生环境与园区中有绝大多数的共有OTU,但野生环境与园区环境中都有各自特有的OTU,这些特有的OTU是否对宿主植物的生长代谢有影响需进一步研究。
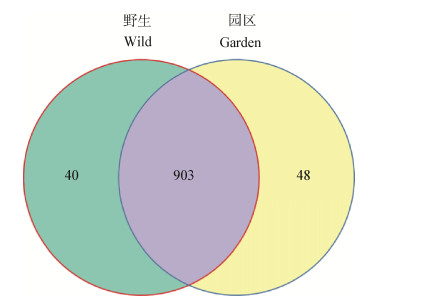
|
| 图 4 野生环境与园区环境真菌OTU数目Venn图 Figure 4 OTU Venn analysis of fungi at wild and garden habitat |
|
|
通过LEfSe[18]分析在两组间寻找具有统计学差异的生物标记物(biomarker),发现不同生长环境中具有差异的生物标记物,结果如图 5所示。图 5显示了两种环境中有显著性差异的生物标记物。其中,野生环境中Ttracladium marohalianum是具有显著性差异的生物标记物,而园区环境中可区分两种土壤环境的生物标记物分别为革菌目(Thelephorales)和革菌科(Thelephoraceae)。此差异可能与园区环境人工干预的作用有关。
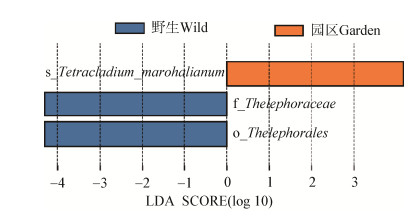
|
| 图 5 野生环境与园区环境环境LEfSe分析结果 Figure 5 Results of LEfSe analysis for wild and garden habitat 注:柱状图的长度代表差异物种的影响大小(即为LDA阈值),不同颜色表示不同生长环境中的物种,蓝色代表野生环境,橙色代表园区环境. Note: Histogram length represents the effect level of different species (LDA threshold), different colors represent species in different growing environment, blue represents wild environment, orange represents park environment. |
|
|
园区环境中,人为因素改变了土壤的理化性质,园区环境中容重的增大会导致土壤透气性的改变,因此好氧菌的生存会受到一定的影响,与此同时含水量也会下降,含水量的测定结果印证了这一点。野生环境比园区环境中土壤的含水量高出44%,而含水量是影响微生物生存和生长发育的重要因素之一。另外,从pH值测定结果中发现,园区土壤环境pH值要高于野生环境,这种变化会导致嗜酸菌群的减少,而某些适合中性环境下生存的病原微生物的数量会随之上升,菌群平衡也会随着这些土壤理化指标的变化而发生变化[19-21],甚至是朝着失衡的方向发展,这可能也是园区更容易暴发病害的原因之一[22]。
由于人工干预的作用,园区环境中速效元素如速效磷、速效钾的含量要明显高于自然环境,这些人为施用的矿质元素往往以无机离子存在于土壤中。众所周知,土壤微生物可转化和帮助植物吸收矿质离子,这些离子的含量同样也影响着微生物的转化和利用,从而改变植物对其的吸收[23]。合理施用氮肥对土壤微生物数量与土壤中营养元素含量具有良好的调节作用,但速效氮在土壤中的存在不够稳定,易受土壤水分条件和生物活动的影响而发生变化[24],因此需要更多的指标来综合评价土壤的营养元素。值得注意的是,野生环境中全氮、全磷、全钾量却高于园区环境,说明野生环境的土壤营养元素含量并不亚于园区环境,而且矿质元素以长效形式存在,从而充分保证了植物生长过程中矿质元素对植物的供给。
土壤环境通常被认为是微生物多样性最丰富的区域[25]。诸多研究发现微生物群落组成及丰度表现出一定的宿主专一性和地域专化性[26],因此,土壤理化指标的差异可能导致土壤中微生态的变化。
3.2 不同生境对重楼内生菌及土壤真菌的影响对野生环境及园区环境的土壤及重楼相关DNA进行了测序分析,确定了不同土壤环境下环境土壤、根际土壤和重楼植株不同部位内生真菌的相对丰度,明确了不同区系之间的差异。结果表明重楼及土壤环境样本中具有高度差异的OTU数,这些差异可归因于真菌对最适生存环境的选择性。
通过对不同土壤环境中真菌的α多样性分析得知,不同生长环境的土壤和重楼真菌群落的丰度及多样性均存在差异。ACE指数和Chao1指数同时反映了野生与园区两种环境中的真菌丰度,野生环境根际土壤中的真菌群落丰度显著高于园区环境根际土。土壤中微生物的多样性及丰度可加快植物对土壤中碳源的利用[27]。同时,根际微生物在促进植物生长发育、防治病害方面起着至关重要的作用[28],根际微生物直接或间接影响着植物的生长以及代谢产物的积累。导致上述两种环境中根际土壤真菌差异的因素很可能是不同土壤环境的自然因素(如降雨、光照、空气扩散条件)以及土壤营养元素(氮、磷、钾等)含量的差异。另外,陆地植物多样性与土壤微生物之间存在着正相关的联系,植物多样性在维持土壤生态系统的运作中起着重要的作用[29]。野生环境与园区环境中植被的多样性截然不同,这同样也可能导致根际土壤真菌的差异。
植物种类、生存环境、植物遗传以及组织状态的不同造成植物内生微生物群落的组成差异[30],由于不同微生物类群对环境的适应性不同,它们更有可能定殖于适合自身生长的环境中[31-32]。我们对不同土壤环境下的环境土、重楼根际及内生真菌的群落差异研究结果表明,不同土壤环境下真菌多样性及群落组成有一定的差异性。但从整体而言,土壤真菌群落与重楼内生真菌群落的差异性要大于野生与园区之间的差异性,这一结果也证明了真菌群落对其定殖环境的选择性。
两种环境中的真菌分别注释到8个门,但在属水平上野生环境注释到82个属,而园区环境注释到78个属,这说明野生环境中真菌的多样性要高于园区环境。众所周知,自然生态系统是一个复杂且稳定的生态系统,与自然生态系统相比,园区生态系统更简单且不稳定,本研究注释结果证明了微生物生态同样存在这一规律。另外,在属水平上可被注释的真菌中,两种环境中丰度最高的均为四枝孢属,但园区中的丰度比野生环境高出37.11%;其次是根内球囊霉属,该属园区中的丰度比野生环境高出89.97%;第三是短柄霉属,该属野生环境中的相对丰度比园区环境高出3倍。由此可见,不同土壤环境对真菌群落丰度有着较为显著的影响。
对不同土壤环境的Venn分析表明,虽然野生环境与园区的样本中有绝大多数是共有OTU,但在野生环境中有4.04%的特有OTU,园区环境中有4.84%的特有OTU。药用植物药用部位的形态建成、有效成分的积累、引种栽培后的质效变迁都与微生物有关[6],这些特有的OTU是否对重楼的生长、代谢产生影响,仍需进一步研究。另外,通过LEfSe分析找到了园区环境与野生环境具有显著性差异的生物标记物,Ttracladium marohalianum、Thelephorales和Thelephoraceae为区别两种环境的生物标记物,这些菌群对重楼植株的形态建成、有效成分的积累、引种栽培后的质效变迁所起到的作用同样是将要研究的内容。
| [1] |
Wang ZW, Ji YL, Chen YG. Studies and biological significances of plant endophytes[J]. Microbiology China, 2015, 42(2): 349-363. (in Chinese) 王志伟, 纪燕玲, 陈永敢. 植物内生菌研究及其科学意义[J]. 微生物学通报, 2015, 42(2): 349-363. |
| [2] |
Compant S, Clément C, Sessitsch A. Plant growth-promoting bacteria in the rhizo- and endosphere of plants: their role, colonization, mechanisms involved and prospects for utilization[J]. Soil Biology and Biochemistry, 2010, 42(5): 669-678. DOI:10.1016/j.soilbio.2009.11.024 |
| [3] |
Zhang JY, Liu YX, Zhang N, et al. NRT1.1B is associated with root microbiota composition and nitrogen use in field-grown rice[J]. Nature Biotechnology, 2019, 37(6): 676-684. DOI:10.1038/s41587-019-0104-4 |
| [4] |
Sessitsch A, Hardoim P, Döring J, et al. Functional characteristics of an endophyte community colonizing rice roots as revealed by metagenomic analysis[J]. Molecular Plant-Microbe Interactions, 2012, 25(1): 28-36. DOI:10.1094/MPMI-08-11-0204 |
| [5] |
Cregger MA, Veach AM, Yang ZK, et al. The Populus holobiont: dissecting the effects of plant niches and genotype on the microbiome[J]. Microbiome, 2018, 6: 31. DOI:10.1186/s40168-018-0413-8 |
| [6] |
Huang WJ, Long CL, Lam E. Roles of plant-associated microbiota in traditional herbal medicine[J]. Trends in Plant Science, 2018, 23(7): 559-562. DOI:10.1016/j.tplants.2018.05.003 |
| [7] |
He DM, Wang H, Chen JL, et al. Microecology and geoherbalism of traditional Chinese medicine[J]. China Journal of Chinese Materia Medica, 2020, 45(2): 290-302. (in Chinese) 何冬梅, 王海, 陈金龙, 等. 中药微生态与中药道地性[J]. 中国中药杂志, 2020, 45(2): 290-302. |
| [8] |
Zhalnina K, Louie KB, Hao Z, et al. Dynamic root exudate chemistry and microbial substrate preferences drive patterns in rhizosphere microbial community assembly[J]. Nature Microbiology, 2018, 3(4): 470-480. |
| [9] |
Chaparro JM, Badri DV, Vivanco JM. Rhizosphere microbiome assemblage is affected by plant development[J]. The ISME Journal, 2013, 8(4): 790-803. |
| [10] |
Brader G, Compant S, Mitter B, et al. Metabolic potential of endophytic bacteria[J]. Current Opinion in Biotechnology, 2014, 27: 30-37. DOI:10.1016/j.copbio.2013.09.012 |
| [11] |
Patten CL, Glick BR. Role of Pseudomonas putida indoleacetic acid in development of the host plant root system[J]. Applied and Environmental Microbiology, 2002, 68(8): 3795-3801. DOI:10.1128/AEM.68.8.3795-3801.2002 |
| [12] |
Guo LD, Hyde KD, Liew ECY. Identification of endophytic fungi from Livistona chinensis based on morphology and rDNA sequences[J]. New Phytologist, 2000, 147(3): 617-630. DOI:10.1046/j.1469-8137.2000.00716.x |
| [13] |
Lin DY. Experimental Guidance of Pedology[M]. Beijing: China Forestry Press, 2004. (in Chinese) 林大仪. 土壤学实验指导[M]. 北京: 中国林业出版社, 2004. |
| [14] |
R Core Team. Core team. R: a language and environment for statistical computing[Z]. Vienna, Austria: R Foundation for Statistical, 2016
|
| [15] |
Kõljalg U, Nilsson RH, Abarenkov K, et al. Towards a unified paradigm for sequence-based identification of fungi[J]. Molecular Ecology, 2013, 22(21): 5271-5277. DOI:10.1111/mec.12481 |
| [16] |
Cheng HY, Wang Y, Zhao L, et al. High-throughput sequencing-based fungal communities of rhizosphere soil and different parts of Paris plant[J]. Chinese Journal of Pharmaceutical analysis, 2020, 40(3): 455-461. (in Chinese) 程虎印, 王艳, 赵乐, 等. 基于高通量测序揭示重楼根际土及不同部位真菌群落差异[J]. 药物分析杂志, 2020, 40(3): 455-461. |
| [17] |
Chen HB, Boutros PC. VennDiagram: a package for the generation of highly-customizable Venn and Euler diagrams in R[J]. BMC Bioinformatics, 2011, 12(1): 35. |
| [18] |
Segata N, Izard J, Waldron L, et al. Metagenomic biomarker discovery and explanation[J]. Genome Biology, 2011, 12(6): R60. DOI:10.1186/gb-2011-12-6-r60 |
| [19] |
Zhou FQ, Ding JJ, Li TT, et al. Plant communities are more sensitive than soil microbial communities to multiple environmental changes in the Eurasian steppe[J]. Global Ecology and Conservation, 2020, 21: e00779. DOI:10.1016/j.gecco.2019.e00779 |
| [20] |
Feng WN, Peng PH. Microbial composition associated with the rhizosphere and seed endosphere of Paeonia szechuanica[J]. Journal of Northeast Forestry University, 2020, 48(1): 88-94. (in Chinese) 冯玮娜, 彭培好. 四川牡丹根际微生物及种子内生菌组成[J]. 东北林业大学学报, 2020, 48(1): 88-94. |
| [21] |
Li ML, Chen YT, Hong XF, et al. Effects of nitrogen management on soil microbial community structure in paddy fields[J]. Acta Agriculturae Zhejiangensis, 2020, 32(2): 308-316. (in Chinese) 李美霖, 陈宇眺, 洪晓富, 等. 不同氮肥管理方式对稻田土壤微生物群落结构的影响[J]. 浙江农业学报, 2020, 32(2): 308-316. |
| [22] |
Li JJ, Luo Y, Zhou LT, et al. Microbial community structure and functional characteristics of rhizosphere soil in different generations of Casuarina equisetifolia plantations[J]. Journal of Forest and Environment, 2020, 40(1): 9-15. (in Chinese) 李建鹃, 罗扬, 周柳婷, 等. 连栽木麻黄根际微生物群落结构和功能特征[J]. 森林与环境学报, 2020, 40(1): 9-15. |
| [23] |
Chen Y, Bonkowski M, Shen Y, et al. Root ethylene mediates rhizosphere microbial community reconstruction when chemically detecting cyanide produced by neighbouring plants[J]. Microbiome, 2020, 8: 4. DOI:10.1186/s40168-019-0775-6 |
| [24] |
Li JT, Huang SX, Wei CZ, et al. Effects of different nitrogen levels on tea rhizosphere microbial community and its role in adjusting soil nutrients[J]. Acta Agriculturae Boreali-Sinica, 2019, 34(S1): 281-288. (in Chinese) 李金婷, 黄少欣, 韦持章, 等. 不同氮素营养水平对茶树根际土壤微生物的影响及其在养分调控中的作用[J]. 华北农学报, 2019, 34(S1): 281-288. |
| [25] |
Roesch LFW, Fulthorpe RR, Riva A, et al. Pyrosequencing enumerates and contrasts soil microbial diversity[J]. The ISME Journal, 2007, 1(4): 283-290. DOI:10.1038/ismej.2007.53 |
| [26] |
Hameed A, Yeh MW, Hsieh YT, et al. Diversity and functional characterization of bacterial endophytes dwelling in various rice (Oryza sativa L.) tissues, and their seed-borne dissemination into rhizosphere under gnotobiotic P-stress[J]. Plant and Soil, 2015, 394(1/2): 177-197. |
| [27] |
Shen FF, Liu Y, Luo CT, et al. Research progress on response and adaptation of plant and soil microbial community diversity to global change in terrestrial ecosystem[J]. Ecology and Environmental Sciences, 2019, 28(10): 2129-2140. (in Chinese) 沈芳芳, 刘影, 罗昌泰, 等. 陆地生态系统植物和土壤微生物群落多样性对全球变化的响应与适应研究进展[J]. 生态环境学报, 2019, 28(10): 2129-2140. |
| [28] |
Fang J, Zhao XQ, Shi GF, et al. Research progress on rhizosphere microorganisms of crops[J]. Journal of Northern Agriculture, 2019, 47(4): 102-107. (in Chinese) 方静, 赵小庆, 史功赋, 等. 农作物根际微生物的研究进展[J]. 北方农业学报, 2019, 47(4): 102-107. |
| [29] |
Chen C, Chen HYH, Chen XL, et al. Meta-analysis shows positive effects of plant diversity on microbial biomass and respiration[J]. Nature Communications, 2019, 10(1): 1332. DOI:10.1038/s41467-019-09258-y |
| [30] |
Edwards J, Johnson C, Santos-Medellín C, et al. Structure, variation, and assembly of the root-associated microbiomes of rice[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(8): E911-E920. DOI:10.1073/pnas.1414592112 |
| [31] |
Whipps JM, Hand P, Pink D, et al. Phyllosphere microbiology with special reference to diversity and plant genotype[J]. Journal of Applied Microbiology, 2008, 105(6): 1744-1755. DOI:10.1111/j.1365-2672.2008.03906.x |
| [32] |
Durán P, Thiergart T, Garrido-Oter R, et al. Microbial interkingdom interactions in roots promote Arabidopsis survival[J]. Cell, 2018, 175(4): 973-983.e14. DOI:10.1016/j.cell.2018.10.020 |
 2020, Vol. 47
2020, Vol. 47