扩展功能
文章信息
- 庄康, 胡晓娟, 曹煜成, 许云娜, 张建设, 文国樑
- ZHUANG Kang, HU Xiao-Juan, CAO Yu-Cheng, XU Yun-Na, ZHANG Jian-She, WEN Guo-Liang
- 不同寡营养培养条件下南海水体细菌群落结构及其对碳源的利用特征
- Bacterial community structure and its utilization characteristics of carbon sources in water of South China Sea under different low-nutrient culture conditions
- 微生物学通报, 2020, 47(9): 2697-2710
- Microbiology China, 2020, 47(9): 2697-2710
- DOI: 10.13344/j.microbiol.china.190875
-
文章历史
- 收稿日期: 2019-10-29
- 接受日期: 2020-07-23
- 网络首发日期: 2020-08-06
2. 中国水产科学研究院南海水产研究所 农业农村部南海渔业资源开发利用重点实验室 广东省渔业生态环境重点实验室 广东 广州 510300;
3. 中国水产科学研究院南海水产研究所深圳试验基地 广东 深圳 518121
2. South China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences; Key Laboratory of South China Sea Fishery Resources Exploitation & Utilization, Ministry of Agriculture and Rural Affairs; Guangdong Provincial Key Laboratory of Fishery Ecology and Environment, Guangzhou, Guangdong 510300, China;
3. Shenzhen Base of South China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Shenzhen, Guangdong 518121, China
海洋微生物是海洋生态系统的重要组成部分,个体虽小,但生物量极大,是海洋生态系统中物质循环和能量流动的主要承担者。海洋微生物因为生活在具有高盐、黑暗、寡营养等环境特点的海洋中[1-2],因此具有温度耐受性、耐盐性、富营养能力强等特点。海洋微生物无论是其物种类群,还是新陈代谢途径、生理生化反应与产物等,都存在着丰富的新颖性和多样性,与陆地微生物相比存在较大的差异,蕴藏的新资源更为丰富和多样化,然而在实验条件下无法完全模拟海洋的环境条件,导致99%以上的海洋微生物不可培养,不能全面地反映海洋微生物多样性,使得大量的具有极大应用价值的海洋微生物资源被埋没[3-4],因此,提高海洋微生物的可培养性,尽量展现其多样性是当前海洋微生物研究的重要任务。
为了研究和挖掘海洋生态系统中可培养的微生物资源,研究者尝试新的培养方法,如改良传统培养方法、微操作技术、极限稀释培养法、模拟原位环境培养法等[5]。其中,寡营养培养法就是改良传统培养方法之一,通过将培养基中的营养元素稀释,提高微生物可培养率[6-7]。戴欣等[8]采用稀释10倍的培养基研究细菌的多样性,获得了更多的微生物。Janssen等[9]比较在正常浓度营养肉汤培养基和稀释100倍的营养肉汤培养基中细菌的生长效果,研究发现稀释后的培养基能获得更好的培养效果。研究者们[10-13]将普通培养基稀释至有机碳水平不高于50 mg/L,比较了稀释培养与未稀释培养的微生物数量,结果发现高营养培养基中的微生物数量并非最多,相反地,高浓度的营养物会抑制一些微生物的生长。此外,Schut等[14]对Resurrection Bay及North Sea海洋微生物样品进行了研究,发现部分细菌仅能在含10 mg/L碳浓度的培养基上形成菌落。由此可见,采用寡营养培养法能有效提高细菌的可培养率。
南中国海(South China Sea)是西太平洋最大的一个边缘海[15],临近西太平洋暖池区,是世界第三大边缘海和我国面积最大的海域。南海北部陆架坡深水区处于陆、洋过渡壳上,不仅蕴藏着丰富的生物资源[16],而且具有重要的战略地位。因南海具有水温高、贫营养和浮游生物占主导地位等特点[17],南海的微生物可能是促进其碳循环和能量流动的主要途径。关于南海微生物多样性的相关研究表明,南海微生物具有较高的多样性[18-22],微生物资源丰富。可见,深入挖掘南海微生物资源,尤其是可培养微生物资源具有重要意义。目前关于南海可培养微生物资源的研究较多采用常规的海洋微生物培养基[23-24],而采用寡营养培养法进行挖掘南海可培养微生物资源的研究较少,因此这一方法值得探讨。
本研究拟以采用原液和稀释10、50倍的2216E培养基培养南海海水细菌,然后基于分子生态的微生物群落结构分析及基于生理生化特征的微生物群落功能分析等多个方面,研究对比不同寡营养培养条件下南海水体细菌的数量、群落结构、群落代谢功能、可培养细菌种类间的差异,以期为南海远洋海域可培养细菌样品的采集及复苏培养方法的优化提供数据支持和理论依据。
1 材料与方法 1.1 实验材料 1.1.1 样品的采集和处理采用有机玻璃采水器从南海不同海域选择3个站点(S1:112°97′E,9°60′N;S2:115°53′E,9°84′N;S3:111°43′E,16°29′N)各采集5 m水层的海水样品3 L,每个站点采集3份水样,每份水样的取水量为1 L。水样经0.2 μm滤膜过滤后,滤膜放入装有海洋细菌保种液的无菌离心管,-20 ℃冷冻保存,带回实验室备用。
1.1.2 主要试剂和仪器及培养基蛋白胨、酵母膏、琼脂粉,广东环凯微生物科技有限公司;磷酸铁,阿拉丁试剂公司;细菌荧光定量PCR检测试剂盒,厦门九龙君合生物科技有限公司;Water DNA Kit,Omega公司;细菌基因组DNA提取试剂盒,天根生化科技(北京)有限公司。0.2 μm滤膜,密理博有限公司(聚碳酸酯材质);Biolog EcoPlateTM微板和微生物鉴定分析仪,Biolog公司;PCR仪,宝生物工程(大连)有限公司;荧光定量PCR仪,Bio-Rad公司。
2216E培养液的配制参照文献[5],稀释10倍和50倍的2216E培养液配置时按比例减量,pH 7.6-7.8。固体培养基加入20.0 g/L琼脂粉。
1.2 实验体系构建实验设置原培养液组(Y)、稀释10倍培养液组(Y-10)和稀释50倍培养液组(Y-50),分别用原液、稀释10倍和50倍的2216E培养液对过滤水样后的滤膜进行复苏培养。每组设置3个平行(即3个站点采集的水样)。将培养瓶置于振荡式微生物培养箱中,30 ℃、100 r/min培养15 d。
1.3 细菌总数的测定从各组中分别取10 mL培养液,使用细菌荧光定量PCR检测试剂盒提取培养液中的细菌DNA,并对其数量进行定量检测。荧光定量的检测体系为(20 μL):PCR混合液10 μL,荧光探针8 μL,待测样品2 μL。反应条件:95 ℃ 2 min;94 ℃ 10 s,60 ℃ 30 s,共40个循环。
1.4 基于16S rRNA基因的细菌群落结构分析从各组中分别取100 mL培养液,采用Water DNA Kit提取水体细菌DNA,利用1%琼脂糖凝胶电泳检测DNA完整性后送明科生物技术(杭州)有限公司,以引物515F (5′-GTGCCAGCMGCCGCGGTAA-3′)和907R (5′-CCGTCAATTCMTTTRAGTTT-3′)特异性扩增16S rRNA基因V4-V5可变区。PCR反应体系(20 μL):5×FastPfu Buffer 4 μL,dNTPs (2.5 mmol/L) 2 μL,FastPfu聚合酶(5 U/μL) 0.4 μL,模板DNA 10 ng,正、反向引物(5 μmol/L)各0.8 μL,补ddH2O至20 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,共27个循环;72 ℃ 10 min。构建扩增文库,通过比对Sliva数据库分析不同样品的细菌群落结构组成。
1.5 异养细菌的数量测定和可培养优势菌的分离及鉴定 1.5.1 异养细菌的数量测定取样进行10倍比系列稀释,取合适稀释度的样品分别涂布于对应稀释度的2216E平板上,置于30 ℃培养箱培养,7 d后平板计数[5]。
1.5.2 可培养优势菌的分离与鉴定在异养细菌的计数平板上标记特征菌株,来源于原培养液组(Y)、稀释10倍培养液组(Y-10)和稀释50倍培养液组(Y-50)的菌株分别标记为:Y-1、Y-2、Y-3等;Y10-1、Y10-2、Y10-3等;Y50-1、Y50-2、Y50-3等。挑取特征菌株至对应稀释度的2216E平板进行多次分离纯化,直至确定为纯菌,制备甘油管将纯化后的菌株冻存备用。
采用细菌基因组DNA提取试剂盒提取纯化单菌的DNA,以细菌16S rRNA基因通用引物进行PCR扩增,引物为8F (5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R (5′-GGTTACCTTGTTACGACTT-3′)。PCR反应体系(50 μL):Premix TaqTM 25 μL,引物8F和1492R (10 μmol/L)各1 μL,模板DNA 2 μL,无菌双蒸水21 μL。PCR反应条件:95 ℃ 4 min;95 ℃ 1 min,48 ℃ 1 min,72 ℃ 2 min,共30个循环;72 ℃ 10 min。PCR扩增产物通过1%的琼脂糖凝胶电泳检测后,送往生工生物工程(上海)股份有限公司测序。测序结果在NCBI数据库BLAST检索中进行核酸序列同源性比较,挑选近缘菌株的16S rRNA基因序列,利用软件MEGA 7.0以邻接法构建系统发育进化树,用Bootstrap (2 000次重复)进行检验。
1.6 可培养细菌群落对碳源的利用特征分析可培养细菌群落对碳源的利用特征采用Biolog EcoPlateTM微板进行测定分析。Biolog EcoPlateTM微板是含有3套31种不同碳源的96微孔板,这31种碳源中包括6类单一碳源底物和四唑染料,另有1个未加碳源的孔加水作对照,每类碳源的微孔各设置3个重复。
取20 mL摇匀的菌液至无菌加样槽,用八道移液枪将菌液加入Biolog EcoPlateTM微板的微孔中,每个微孔加入菌液150 μL,每个样品设3个重复。30 ℃恒温培养360 h,每24 h在590 nm下读取各孔吸光值,后续用于分析细菌群落平均活性、细菌群落功能多样性和细菌群落对各类碳源的利用等[25-28]。
1.7 数据处理 1.7.1 基于16S rRNA基因的细菌群落多样性分析(1) Chao1指数计算公式:

|
Schao1是估计的OTU数,Sobs是实际观测到的OTU数,n1是只含有一条序列的OTU数目,n2是只含有两条序列的OTU数。
(2) Shannon指数计算公式:

|
Sobs是实际观测到的OTU数,ni是第i个OTU所含的序列数,N是所有的序列数。
1.7.2 基于Biolog EcoPlateTM微板的可培养细菌群落对碳源的利用特征分析(1) 细菌群落平均活性的计算
31个孔的平均吸光值(average well color development,AWCD)的计算公式为:AWCD=[∑(Ci-R)]/31
Ci是除对照孔外各孔吸光值,R是对照孔吸光值[26]。
(2) 细菌群落功能多样性的计算
采用Biolog EcoPlateTM微板培养360 h的数据计算Shannon指数、Shannon均度、Simpson指数、McIntosh指数、Mclntosh均度[29]。
Shannon指数计算公式: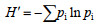
Shannon均度计算公式;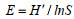
Simpson指数计算公式: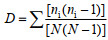
McIntosh指数计算公式:
McIntosh均度计算公式:
(3) 细菌群落对各类碳源的利用
Biolog EcoPlateTM微板包括聚合物类、羧酸类、氨基酸类、糖类、胺类、其他类共6大类碳源底物。根据碳源的分类,以校正后的数据计算各细菌群落对6类碳源的总吸光值来分析不同稀释水平下细菌群落对同一类碳源的利用差异[30]。
1.7.3 数据分析采用软件SPSS 19.0对实验数据进行单因素方差分析(one-way ANOVA),显著水平设为P < 0.05,用以比较不同组间的差异。
2 结果与分析 2.1 细菌总数比较不同稀释水平下细菌数量如图 1所示。经过15 d的培养,Y组细菌总数为(7.70±4.69)×108个/mL,Y-10组细菌总数为(9.28±6.94)×107个/mL,Y-50组细菌总数为(9.00±3.87)×107个/mL,3组间细菌数量无显著差异(P > 0.05)。

|
| 图 1 不同寡营养培养条件下细菌总数比较 Figure 1 The total number of bacteria was compared under different low-nutrient culture conditions 注:图中不同小写字母代表差异性显著(P < 0.05),相同字母代表差异性不显著(P > 0.05). Note: The different letters represent significant difference (P < 0.05), and the same letters represent no significant difference (P > 0.05). |
|
|
虽然不同稀释条件下细菌总数无显著性差异,但其细菌群落结构差异明显(图 2-4)。Y组细菌群落共检测出10门193属,在门水平上,以Proteobacteria和Bacteroides所占比例最高,分别为56.44%和37.27%;优势属为Vibrio、Psychrobacter和Mesonia,分别占14.41%、10.47%、10.39%。Y-10组细菌群落共检测出15门220属,在门水平上,以Proteobacteria、Bacteroides和Firmicutes所占比例最高,分别为40.30%、36.91%和17.30%;优势属为Bacillus,占16.09%。Y-50组细菌群落共检测出14门226属,在门水平上,以Proteobacteria、Bacteroides、Planctomycetes和Firmicutes所占比例最高,分别为45.19%、25.29%、13.58%和11.21%,优势属为Exiguobacterium和Planctomyces,分别占10.39%和10.01%。
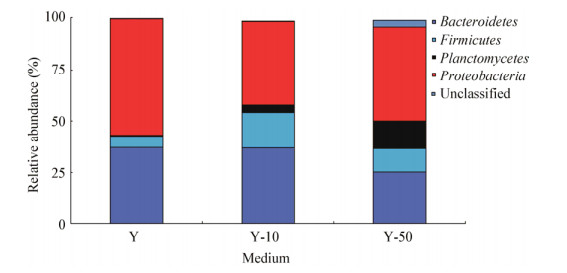
|
| 图 2 不同寡营养培养条件下高数量细菌类群在门分类水平的群落结构组成 Figure 2 Community structure composition of high number of bacterial groups at phylum level under different low-nutrient culture conditions |
|
|

|
| 图 3 不同寡营养培养条件下低数量细菌类群在门分类水平的群落结构组成 Figure 3 Community structure composition of low number bacterial groups at phylum level under different low-nutrient culture conditions |
|
|

|
| 图 4 不同寡营养培养条件下细菌在属分类水平的群落结构组成 Figure 4 Community structure composition of bacteria at genus classification level under different low-nutrient culture conditions |
|
|
不同稀释水平下细菌群落的α多样性指数如表 1所示,Y-10组和Y-50组的OTU数、Chao1值均高于Y组,而且Y-10组和Y-50组细菌群落的Shannon指数显著高于Y组(P < 0.05)。
| Medium | OTU number | Chao1 index | Shannon index |
| Y | 157±22a | 180±25a | 2.65±0.20a |
| Y-10 | 288±136a | 304±123a | 3.26±0.25b |
| Y-50 | 244±53a | 280±42a | 3.26±0.23b |
| 注:数据为平均值±标准差,表中同一列数据后不同小写字母表示不同稀释水平的数据差异性显著(P < 0.05),相同字母表示不同稀释水平的数据差异性不显著(P > 0.05). Note: The data are mean±standard deviation. Different lowercase letters after the same column of data in the table indicate that the data at different dilution levels are significantly different (P < 0.05), and the same letter indicates that the data at different dilution levels are not significant (P > 0.05). | |||
不同稀释水平下异养细菌数量如图 5所示。经过15 d的培养,Y组异养细菌总数为(4.54±0.52)×107 CFU/mL,Y-10组异养细菌总数为(5.47±4.12)×106 CFU/mL,Y-50组异养细菌总数为(5.73±4.62)×106 CFU/mL,Y组异养细菌数量与Y-10组和Y-50组异养细菌数量存在显著差异(P < 0.05),Y-10组与Y-50组无显著性差异(P > 0.05)。
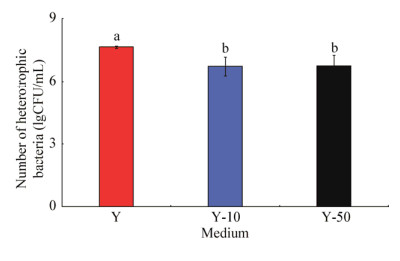
|
| 图 5 不同寡营养培养条件下异养细菌数量比较 Figure 5 The number of heterotrophic bacteria was compared under different low-nutrient culture conditions 注:图中不同小写字母代表差异性显著(P < 0.05),相同字母代表差异性不显著(P > 0.05). Note: The different letters represent significant difference (P < 0.05), and the same letters represent no significant difference (P > 0.05). |
|
|
通过将分离纯化的细菌鉴定,Y组分离到14株细菌,属于3门6属,其中包括Proteobacteria门的Mesorhizobium、Nitratireductor、Microbulbifer和Halomonas 4个属,Bacteroides门的Mesonia属,Firmicutes门的Bacillus属;其优势菌为Bacillus和Microbulbifer,相对丰度分别为57.14%和14.29%。Y-10组分离到14株细菌,属于3门6属,其中包括Proteobacteria门的Microbulbifer、Idiomarina和Vibrio属,Actinobacteria门的Microbacterium属,Firmicutes门的Bacillus和Halobacillus属;其优势菌为Bacillus,占64.29%。Y-50组分离到13株细菌,属于2门7属,其中包括Proteobacteria门的Sulfitobacter、Vibrio、Alcanivorax、Alteromonas和Pseudomonas属,Firmicutes门的Bacillus和Exiguobacterium属;其优势菌为Bacillus、Vibrio和Exiguobacterium,分别占30.77%、23.08%和15.38%。其中Bacillus为共有的优势菌属,稀释10倍培养液筛得的Microbacterium (1株)、Vibro (1株)、Idiomarina (1株)、Halobacillus (1株)共4株优势菌和稀释50倍培养液筛得的Alcanivorax (1株)、Sulfitobacter (1株)、Alteromonas (1株)、Pseudomonas (1株)、Exiguobacterium (2株)、Vibro (3株)共9株优势菌不同于原培养液中所筛菌(图 6)。
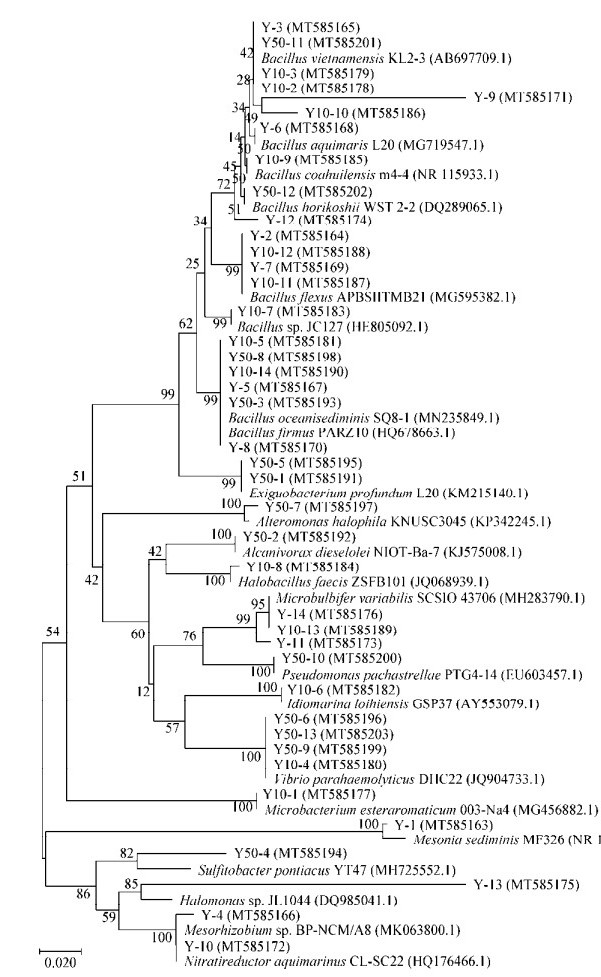
|
| 图 6 可培养优势菌的16S rRNA基因系统发育树 Figure 6 Phylogenetic tree of dominant cultivated bacteria based on 16S rRNA gene sequences 注:括号中的序号表示菌株的GenBank登录号;标尺:核苷酸的替代率为0.02;分支位置中的数字表示Boostrap支持率. Note: The number in brackets denotes the GenBank accession number of the strain; Scale bar: The nucleotide substitution rate was 0.02; The number in each branch points denote the percentages supported by boostrap. |
|
|
AWCD值反映了微生物群落对不同碳源的利用能力以及其代谢活性[31]。Y组细菌群落的AWCD值在0-72 h之间为指数增长期,随后进入稳定期,72 h达到最高,最高值为0.70;Y-10组细菌群落的AWCD值在0-192 h之间为指数增长期,随后进入稳定期,288 h达到最高,最高值为1.41;Y-50组细菌群落的AWCD值在0-168 h之间为指数增长期,随后进入稳定期,336 h达到最高,最高值为1.40。在192 h后,Y-10组和Y-50组细菌群落的AWCD值显著高于Y组(P < 0.05) (图 7)。
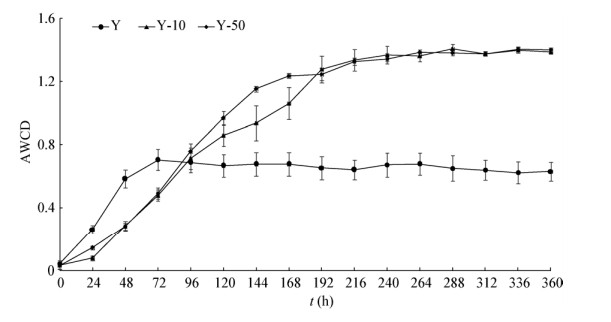
|
| 图 7 不同寡营养培养条件下微生物群落的AWCD值比较 Figure 7 AWCD values of microbial communities under different low-nutrient culture conditions were compared |
|
|
3组细菌群落的Shannon指数、Shannon均度、Simpson指数和McIntosh均度均保持稳定,相较Y组,Y-10组和Y-50组细菌群落的McIntosh指数则有所升高,并且与Y组存在显著性差异(P < 0.05) (表 2)。
| Medium | Shannon index | Shannon uniformity | Simpson index | McIntosh index | McIntosh uniformity |
| Y | 3.35±0.02a | 1.03±0.04a | 28.09±0.70a | 3.86±0.72a | 1.01±0.02a |
| Y-10 | 3.36±0.01a | 1.02±0.02a | 27.76±0.52a | 8.13±0.39b | 1.01±0.01a |
| Y-50 | 3.34±0.01a | 1.07±0.02a | 27.15±0.35a | 8.24±0.23b | 1.02±0.01a |
| 注:数据为平均值±标准差,表中同一列数据后不同小写字母表示不同稀释水平的数据差异性显著(P < 0.05),相同字母表示不同稀释水平的数据差异性不显著(P > 0.05). Note: The data are mean±standard deviation. Different lowercase letters after the same column of data in the table indicate that the data at different dilution levels are significantly different (P < 0.05), and the same letter indicates that the data at different dilution levels are not significant (P > 0.05). | |||||
不同寡营养培养条件下细菌群落对六大类碳源的利用变化情况如图 8所示,从对六大类碳源的利用变化情况来看,Y-10组和Y-50组细菌群落对糖类的利用率最高,其对糖类的总吸光值最高为11.25和12.23,对羧酸、氨基酸、聚合物的利用率次之,对胺类和其他类碳源的利用较少;Y组细菌群落对羧酸的利用率最高,其对羧酸的总吸光值最高为6.17,对糖类、氨基酸的利用率次之,对聚合物、胺类和其他类碳源的利用较少。Y-10组细菌群落对聚合物、羧酸、糖类和氨基酸的利用率分别在168、192、192和312 h后显著高于Y组(P < 0.05);Y-50组细菌群落对聚合物、羧酸、糖类和氨基酸的利用率分别在144、192、168和312 h后显著高于Y组(P < 0.05)。

|
| 图 8 不同寡营养培养条件下微生物群落对同一类型碳源利用的变化 Figure 8 Changes of microbial community utilization of the same type of carbon source under different low-nutrient culture conditions 注:A:聚合物;B:羧酸;C:氨基酸;D:糖类;E:胺类;F:其他 Note: A: Polymer; B: Carboxylic acid; C: Amino acid; D: Carbohydrate; E: Amine; F: Others. |
|
|
海洋微生物研究进展迅速,作为自然资源最大的宝库,蕴藏着巨大的潜力。非培养技术揭示了海洋微生物的多样性,而出于各种各样的原因,其中绝大多数的微生物目前尚未分离出来[32],因此,挖掘更多的可培养海洋微生物资源是海洋微生物研究的方向之一。寡营养培养法是一种根据海洋环境寡营养的特点,将培养基中的营养元素进行稀释,以提高细菌可培养性的研究方法[7],早期由Button等[33]和Schut等[14]开发,用于海水样品的可培养细菌研究。本研究以南海海水样品为研究对象,探讨了不同寡营养培养条件下南海水体细菌数量、群落结构及其对碳源的利用特征。结果发现,培养15 d后,寡营养培养液培养的细菌总数虽低于原培养液培养(P > 0.05),但稀释10倍和稀释50倍培养液培养的细菌群落的Shannon指数显著高于原培养液(P < 0.05),而且稀释10倍培养液和稀释50倍培养液的OTU数和Chao1值均高于原培养液,表明寡营养培养后细菌群落的多样性高于原培养液培养的细菌群落。从细菌群落结构来看,稀释10倍培养液检测出15门220属,稀释50倍培养液检测出14门226属,而原培养液检测出10门193属,寡营养培养后的细菌群落比原培养液更丰富。Sun等[34]采用稀释10倍的2216E培养基和稀释10倍的LB培养基研究海水中细菌的可培养性,测序结果表明寡营养培养基能扩大可以培养的微生物种类范围,本研究与其结果相近。从优势菌组成来看,原培养液培养的细菌群落中优势类群为Proteobacteria和Bacteroides,优势属为Vibrio、Psychrobacter和Mesonia;稀释10倍培养液培养的细菌群落中优势类群为Proteobacteria、Bacteroides和Firmicutes,优势属为Bacillus;稀释50倍培养液培养的细菌群落中优势类群为Proteobacteria、Bacteroides、Planctomycetes和Firmicutes,优势属为Exiguobacterium和Planctomyces,这一结果和白洁等[35]所报道南海南部水体优势类群为Proteobacteria和Bacteroides并且存在Firmicutes和Planctomycetes的结果相近。此外,稀释10倍培养液与稀释50倍培养液的优势类群结果与王蕾等[36]采用原位培养研究南海深海微生物的研究结果相近,两项研究的高通量结果中Proteobacteria、Bacteroides和Firmicutes均为优势类群。
从可培养细菌的状况来看,原培养液培养的异养细菌总数高于寡营养培养液培养的异养细菌总数,且差异显著(P < 0.05)。不同稀释条件下分离的优势菌株种类有所不同,原培养液中分离得到的优势菌为Bacillus和Microbulbifer,稀释10倍培养液中分离的优势菌为Bacillus,稀释50倍培养液中分离得到的优势菌为Bacillus、Vibrio和Exiguobacterium。总体来看,3种培养液所培细菌的分离结果与徐重[37]对南海表层海水中可培养细菌多样性的研究结果一致,其研究表明南海表层海水由Proteobacteria、Bacteroides、Firmicutes和Actinobacteria这4个门组成。此外,本研究分离出的Proteobacteria中γ-变形菌纲所占比例较大,这一结果与李明[38]的研究得以相互印证。由此可见,将南海海水样品经滤膜过滤保存,经复苏培养后的细菌优势类群可基本反映南海水体细菌群落状况。
AWCD值是基于生理生化特性的细菌群落对碳源利用特征的体现,其可反映基于可培养系统的细菌群落对不同碳源的利用能力以及其代谢活性[31]。本研究中,寡营养培养后细菌群落的AWCD值显著高于原培养液(P < 0.05),尤其对聚合物、羧酸、氨基酸和糖类的利用率显著高于原培养液(P < 0.05),这反映出寡营养培养后细菌群落的活性更高,并且提高了代谢能力。值得注意的是,寡营养培养液中异养细菌数量低于原培养液,但其碳源代谢能力仍高于原培养液,说明造成细菌群落代谢功能差异的原因不仅和细菌群落丰富度有关,还和群落结构密切相关[39],寡营养培养后的菌群结构与原培养液相比,分离出更多的Proteobacteria菌株,而Proteobacteria对聚合物类和糖类等碳源具有较高的利用能力[40]。此外,寡营养培养液中的细菌在Biolog EcoPlateTM微板中对部分碳源的利用能力没有显著增加,推测是寡营养培养基的生境导致整个细菌群落的选择压力[41]。
常规的海洋细菌培养基的营养浓度远高于海洋细菌的生长环境,高浓度的营养物质比较适合生长速度快且对高浓度营养物有抵抗能力的细菌,但是对于生长速度慢的细菌可能有抑制作用。在本研究中,将普通的2216E培养基分别进行了10倍和50倍的稀释,在一定程度上降低了培养基的营养水平。通过寡营养培养增加了细菌群落的丰富度和多样性,提高了可培养细菌的代谢活性和对碳源尤其是聚合物、羧酸、氨基酸和糖类的利用率,分离纯化获得原培养基未筛选得到的细菌。因此,在南海远洋海域可培养细菌样品的采集及复苏时,可通过寡营养培养获得更丰富的南海可培养微生物资源。
| [1] |
Xue CB, Wang GL, Jin S, et al. Advances in marine microbial diversity research[J]. Advances in Marine Science, 2004, 22(3): 377-384. (in Chinese) 薛超波, 王国良, 金珊, 等. 海洋微生物多样性研究进展[J]. 海洋科学进展, 2004, 22(3): 377-384. |
| [2] |
Yang JP, Zhang YF, Lv MD. Research and application of marine microorganisms[J]. China Southern Agricultural Machinery, 2018, 49(13): 33. (in Chinese) 杨金鹏, 张翼飞, 吕美东. 海洋微生物的研究与应用[J]. 南方农机, 2018, 49(13): 33. |
| [3] |
Yuan DF, Yu LJ, Liu CG. Progress in high-throughput culturing and sorting of marine microorganisms[J]. Microbiology China, 2014, 41(6): 1180-1187. (in Chinese) 袁东芳, 于乐军, 刘晨光. 海洋微生物高通量培养方法和分选技术的研究进展[J]. 微生物学通报, 2014, 41(6): 1180-1187. |
| [4] |
Ren LC, Li MY, Bao SX. The investigation of the marine archaea diversity[J]. Life Science Research, 2006, 10(2): 67-70. (in Chinese) 任立成, 李美英, 鲍时翔. 海洋古菌多样性研究进展[J]. 生命科学研究, 2006, 10(2): 67-70. |
| [5] |
General Administration of Quality Supervision, Inspection and Quarantine of the People's Republic of China, Standardization Administration of China. GB 17378.7-2007 The specification for marine monitoring-Part 7: Ecological survey for offshore pollution and biological monitoring[S]. Beijing: China Standards Press, 2007 (in Chinese) 中华人民共和国国家质量监督检验检疫总局, 中国国家标准化管理委员会. GB 17378.7-2007海洋监测规范第七部分: 近海污染生态调查和生物监测处理[S].北京: 中国标准出版社, 2007 |
| [6] |
Zhou N, Jiang CY, Liu SJ. Cultivation of microorganisms from environments: nutrient level of the culture medium is of great importance[J]. Microbiology China, 2016, 43(5): 1075-1081. (in Chinese) 周楠, 姜成英, 刘双江. 从环境中分离培养微生物:培养基营养水平至关重要[J]. 微生物学通报, 2016, 43(5): 1075-1081. |
| [7] |
Connon SA, Giovannoni SJ. High-throughput methods for culturing microorganisms in very-low-nutrient media yield diverse new marine isolates[J]. Applied and Environmental Microbiology, 2002, 68(8): 3878-3885. |
| [8] |
Dai X, Wang BJ, Huang Y, et al. Bacterial diversity in the sediments of Taihu Lake by using traditional nutrient medium and dilution nutrient medium[J]. Acta Microbiologica Sinica, 2005, 45(2): 161-165. (in Chinese) 戴欣, 王保军, 黄燕, 等. 普通和稀释培养基研究太湖沉积物可培养细菌的多样性[J]. 微生物学报, 2005, 45(2): 161-165. |
| [9] |
Janssen PH, Yates PS, Grinton BE, et al. Improved culturability of soil bacteria and isolation in pure culture of novel members of the divisions Acidobacteria, Actinobacteria, Proteobacteria, and Verrucomicrobia[J]. Applied and Environmental Microbiology, 2002, 68(5): 2391-2396. |
| [10] |
Maki JS, LaCroix SJ, Hopkins BS, et al. Recovery and diversity of heterotrophic bacteria from chlorinated drinking waters[J]. Applied and Environmental Microbiology, 1986, 51(5): 1047-1055. |
| [11] |
Reasoner DJ, Geldreich EE. A new medium for the enumeration and subculture of bacteria from potable water[J]. Applied and Environmental Microbiology, 1985, 49(1): 1-7. |
| [12] |
Eilers H, Pernthaler J, Glockner FO, et al. Culturability and in situ abundance of pelagic bacteria from the North Sea[J]. Applied and Environmental Microbiology, 2000, 66(7): 3044-3051. |
| [13] |
Cho JC, Giovannoni SJ. Cultivation and growth characteristics of a diverse group of oligotrophic marine Gammaproteobacteria[J]. Applied and Environmental Microbiology, 2004, 70(1): 432-440. |
| [14] |
Schut F, de Vries EJ, Gottschal JC, et al. Isolation of typical marine bacteria by dilution culture: growth, maintenance, and characteristics of isolates under laboratory conditions[J]. Applied and Environmental Microbiology, 1993, 59(7): 2150-2160. |
| [15] |
Zhang WB, Liu HX, Gong XY, et al. Stable nitrogen isotope of size fractionated zooplankton in northern South China Sea[J]. South China Fisheries Science, 2017, 13(6): 48-55. (in Chinese) 张文博, 刘华雪, 巩秀玉, 等. 南海北部不同粒级浮游动物氮稳定同位素研究[J]. 南方水产科学, 2017, 13(6): 48-55. |
| [16] |
Sun DR, Li Y, Wang XH. Seasonal changes of species composition and diversity of fishes in coastal waters of Hainan Island, China[J]. South China Fisheries Science, 2012, 8(1): 1-7. (in Chinese) 孙典荣, 李渊, 王雪辉. 海南岛近岸海域鱼类物种组成和多样性的季节变动[J]. 南方水产科学, 2012, 8(1): 1-7. |
| [17] |
Zhang Z, Gong XY, Hu Y, et al. Abundance of bacterioplankton and virioplankton in the central and northern South China Sea in autumn[J]. South China Fisheries Science, 2016, 12(4): 9-16. (in Chinese) 张喆, 巩秀玉, 胡莹, 等. 南海中北部海域秋季浮游细菌和病毒丰度及其影响因子[J]. 南方水产科学, 2016, 12(4): 9-16. |
| [18] |
Zhang Y, Li J, Cheng XH, et al. Community differentiation of bacterioplankton in the epipelagic layer in the South China Sea[J]. Ecology and Evolution, 2018, 8(10): 4932-4948. DOI:10.1002/ece3.4064 |
| [19] |
Yu TT, Li M, Niu MY, et al. Difference of nitrogen-cycling microbes between shallow bay and deep-sea sediments in the South China Sea[J]. Applied Microbiology and Biotechnology, 2018, 102(1): 447-459. |
| [20] |
Zhang J, Chen M, Huang JF, et al. Diversity of the microbial community and cultivable protease-producing bacteria in the sediments of the Bohai Sea, Yellow Sea and South China Sea[J]. PLoS One, 2019, 14(4): e0215328. DOI:10.1371/journal.pone.0215328 |
| [21] |
Qin M, Wang XT, Jiang L, et al. Bacterial diversity in phosphorus immobilization of the South China Sea[J]. Environmental Technology. DOI:10.1080/09593330.2019.1585479 |
| [22] |
Cui HP, Su X, Chen F, et al. Microbial diversity of two cold seep systems in gas hydrate-bearing sediments in the South China Sea[J]. Marine Environmental Research, 2019, 144: 230-239. DOI:10.1016/j.marenvres.2019.01.009 |
| [23] |
Yu T. Cultivation of deep-sea microorganisms from the South China Sea and taxonomic analysis of four novel bacteria[D]. Qingdao: Master's Thesis of Ocean University of China, 2013 (in Chinese) 于童.南海深海微生物的分离培养及4株海洋新菌的分类鉴定[D].青岛: 中国海洋大学硕士学位论文, 2013 http://d.wanfangdata.com.cn/Thesis_D326783.aspx |
| [24] |
Sun FQ, Wang BJ, Li GY, et al. Diversity of bacteria isolated from the South China Sea Sediments[J]. Acta Microbiologica Sinica, 2008, 48(12): 1578-1587. (in Chinese) 孙风芹, 汪保江, 李光玉, 等. 南海南沙海域沉积物中可培养微生物及其多样性分析[J]. 微生物学报, 2008, 48(12): 1578-1587. |
| [25] |
Cao YC, Li ZJ, Wen GL, et al. Dynamics of microbial community and its metabolisms of different carbon sources in tilapia ponds[J]. Journal of Agro-Environment Science, 2014, 33(1): 172-177. (in Chinese) 曹煜成, 李卓佳, 文国樑, 等. 罗非鱼主养池塘水体微生物群落对碳源代谢的动态变化[J]. 农业环境科学学报, 2014, 33(1): 172-177. |
| [26] |
Hu XJ, Li ZJ, Cao YC, et al. Influence of heavy rain on microbial community in Litopenaeus vannamei culture ponds in western Guangdong Province[J]. Journal of Fishery Sciences of China, 2010, 17(5): 987-995. (in Chinese) 胡晓娟, 李卓佳, 曹煜成, 等. 强降雨对粤西凡纳滨对虾养殖池塘微生物群落的影响[J]. 中国水产科学, 2010, 17(5): 987-995. |
| [27] |
Yang YY, Li ZJ, Liang XH, et al. Influences of Bacillus on metabolic functions of microbial communities in fishponds[J]. Journal of Microbiology, 2009, 29(3): 11-17. (in Chinese) 杨莺莺, 李卓佳, 梁晓华, 等. 芽胞杆菌对鱼池微生物群落代谢功能的影响[J]. 微生物学杂志, 2009, 29(3): 11-17. |
| [28] |
Hu XJ, Li ZJ, Cao YC, et al. Dynamic characteristics of bacterial community in culture ponds for Litopenaeus vannamei in western Guangdong under influence of severe weather[J]. South China Fisheries Science, 2012, 8(5): 52-59. (in Chinese) 胡晓娟, 李卓佳, 曹煜成, 等. 强天气干扰条件下粤西凡纳滨对虾养殖池塘细菌群落动态特征[J]. 南方水产科学, 2012, 8(5): 52-59. |
| [29] |
Yang YH, Yao J, Hua XM. Effect of pesticide pollution against functional microbial diversity in soil[J]. Journal of Microbiology, 2000, 20(2): 23-25, 47. (in Chinese) 杨永华, 姚健, 华晓梅. 农药污染对土壤微生物群落功能多样性的影响[J]. 微生物学杂志, 2000, 20(2): 23-25, 47. |
| [30] |
Li ZJ, Lin L, Yang YY, et al. Influence of Bacillus on the microbial communities in shrimp ponds[J]. Journal of Agro- Environment Science, 2007, 26(3): 1183-1189. (in Chinese) 李卓佳, 林亮, 杨莺莺, 等. 芽孢杆菌制剂对虾池环境微生物群落的影响[J]. 农业环境科学学报, 2007, 26(3): 1183-1189. |
| [31] |
Choi KH, Dobbs FC. Comparison of two kinds of Biolog microplates (GN and ECO) in their ability to distinguish among aquatic microbial communities[J]. Journal of microbiological methods, 1999, 36(3): 203-213. DOI:10.1016/S0167-7012(99)00034-2 |
| [32] |
Ji SQ. The establishment and applications of high-throughput cultivating and sorting method for marine microorganisms[D]. Qingdao: Doctoral Dissertation of Ocean University of China, 2011 (in Chinese) 冀世奇.海洋微生物高通量培养和分选技术的建立及应用[D].青岛: 中国海洋大学博士学位论文, 2011 http://cdmd.cnki.com.cn/Article/CDMD-10423-1011231382.htm |
| [33] |
Button DK, Schut F, Quang P, et al. Viability and isolation of marine bacteria by dilution culture: theory, procedures, and initial results[J]. Applied and Environmental Microbiology, 1993, 59(3): 881-891. |
| [34] |
Sun J, Guo JZ, Yang Q, et al. Diluted conventional media improve the microbial cultivability from aquarium seawater[J]. Journal of Microbiology, 2019, 57(9): 759-768. |
| [35] |
Bai J, Liu XS, Hou R, et al. Community structure and influencing factors of bacterioplankton in the southern South China Sea[J]. China Environmental Science, 2014, 34(11): 2950-2957. (in Chinese) 白洁, 刘小沙, 侯瑞, 等. 南海南部海域浮游细菌群落特征及影响因素研究[J]. 中国环境科学, 2014, 34(11): 2950-2957. |
| [36] |
Wang L, Wang LP, Dong CM, et al. Deep sea in-situ cultivation and diversity analysis of microorganism involved in nitrogen cycling in the South China Sea[J]. Journal of Applied Oceanography, 2019, 38(1): 1-13. (in Chinese) 王蕾, 王丽萍, 董纯明, 等. 南海深海氮循环微生物的原位培养与多样性分析[J]. 应用海洋学学报, 2019, 38(1): 1-13. |
| [37] |
Xu Z. The diversity analysis of culturable bacteria in South China Sea and the polyphasic taxonomy of two deep-sea strains[D]. Liaocheng: Master's Thesis of Liaocheng University, 2014 (in Chinese) 徐重.中国南海样品可培养细菌多样性分析及两株深海细菌的多相分类研究[D].聊城: 聊城大学硕士学位论文, 2014 http://med.wanfangdata.com.cn/Paper/Detail?id=DegreePaper_D502363 |
| [38] |
Li M. Diversity of cultivated bacteria isolated from deep seas in tropical Pacific Ocean and identification of a new species Oceanicola eastpnp[D]. Xiamen: Master's Thesis of Third Institute of Oceanography, State Oceanic Administration, 2007 (in Chinese) 李明.热带太平洋深海可培养细菌多样性与新种Oceanicola eastpnp sp. nov.的鉴定[D].厦门: 国家海洋局第三海洋研究所硕士学位论文, 2007 http://cdmd.cnki.com.cn/Article/CDMD-85303-2007214328.htm |
| [39] |
Lin L. The effect of Bacillus probiotics on microbial communities and shrimp intestinal microflora[D]. Guangzhou: Master's Thesis of Jinan University, 2005 (in Chinese) 林亮.芽孢杆菌制剂对虾池微生物群落以及对虾肠道菌群的影响[D].广州: 暨南大学硕士学位论文, 2005 http://d.wanfangdata.com.cn/Thesis/D464218 |
| [40] |
Shi LN, Ke XL, Liu ZG, et al. Effect of Houttuynia cordata floating-bed on the structure and function of bacterial community in the sediments of tilapia aquacultural system[J]. Chinese Agricultural Science Bulletin, 2015, 31(14): 64-73. (in Chinese) 史丽娜, 可小丽, 刘志刚, 等. 罗非鱼-鱼腥草共生养殖池塘沉积物菌群结构与功能特征[J]. 中国农学通报, 2015, 31(14): 64-73. |
| [41] |
Jiang LL. The effect of biochar on soil microbial diversity and community structure[D]. Shenyang: Doctoral Dissertation of Shenyang Agricultural University, 2016 (in Chinese) 江琳琳.生物炭对土壤微生物多样性和群落结构的影响[D].沈阳: 沈阳农业大学博士学位论文, 2016 http://cdmd.cnki.com.cn/Article/CDMD-10157-1016143344.htm |
 2020, Vol. 47
2020, Vol. 47