扩展功能
文章信息
- 程新宽, 陈曦, 冯进辉, 吴洽庆, 朱敦明
- CHENG Xin-Kuan, CHEN Xi, FENG Jin-Hui, WU Qia-Qing, ZHU Dun-Ming
- 内消旋-二氨基庚二酸脱氢酶关键氨基酸位点的改造提高对烷基取代2-酮酸的催化活力
- Mutation of a key amino acid residue of meso-diaminopimelate dehydrogenase enhances the catalytic activity toward alkyl substituted 2-keto acids
- 微生物学通报, 2020, 47(7): 2119-2127
- Microbiology China, 2020, 47(7): 2119-2127
- DOI: 10.13344/j.microbiol.china.200110
-
文章历史
- 收稿日期: 2020-02-14
- 接受日期: 2020-05-14
- 网络首发日期: 2020-06-04
2. 中国科学院天津工业生物技术研究所 工业酶国家工程实验室 天津市生物催化技术工程中心 天津 300308
2. Tianjin Biocatalysis Technology Engineering Center; National Engineering Laboratory for Industrial Enzymes; Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China
D-氨基酸是许多药物分子[1]和生物活性肽[2]的重要结构单元,这些物质广泛用于治疗微生物感染、高血压和癌症[3-4]。D-丙氨酸可用于合成手性药物和甜味剂[5],而且是维生素B6(vitamin B6)的前体物质[6]。D-2-氨基丁酸主要用于合成各种抑制剂(血管紧缩素转换酶2抑制剂[7]、基质蛋白酶抑制剂[8])和抗增殖剂[9-10]。D-正缬氨酸被广泛用于合成大环内酯类抗生素(16元环的pamamycin-607)[11]以及防御性大环生物碱(r-epilachnene)[12-13]。D-谷氨酸是尼古丁类似物等稠环化合物的关键中间体,可用于抗镇痛药物[14]和心脑血管多肽药物[15],以及新型抗生素的合成[16]。目前,D-氨基酸可通过化学合成法、发酵法以及生物催化法来合成[17-18]。化学法往往需要贵重金属以及强酸强碱等化学试剂参与,以及保护和去保护过程,难以控制选择性,并且需要剧烈的高耗能反应条件。发酵法需要调控复杂的代谢网络,宿主细胞内多种酶的存在会增加底物/产物不必要的消耗,而且干扰目的产物的催化过程,产生的代谢产物也存在对细胞形成毒害的风险。因此,无论是从绿色环保经济节能的角度,还是从操作简便易行的角度出发,生物催化法在合成D-氨基酸方面均具有自身独特的优势。
研究者们利用生物催化法合成D-氨基酸进行了多种不同酶的尝试。例如,D-氨基酸氨基转移酶、D-乙内酰脲酶、丝氨酸消旋酶、N-酰基氨基酸消旋酶、L-氨基酸脱氨酶、苯丙氨酸氨裂解酶等[19-23]。然而这些生物催化法或多或少均存在底物难以获得、底物或产物抑制、需要借助化学试剂、需要昂贵的氨基供体、理论产率低、选择性差等不足。在生物催化法中,内消旋-二氨基庚二酸脱氢酶(meso-diaminopimelate dehydrogenase,DAPDH)可逆催化2-酮酸和游离氨合成D-氨基酸(图 1)。与上述生物催化法相比,DAPDH可以克服上述不足。DAPDH不仅可以高度选择性地催化内消旋-二氨基庚二酸的D-手性中心产生L-2-氨基-6-氧代庚二酸[24],而且与大部分以膜蛋白形式存在的D-氨基酸脱氢酶[25-27]相比,DAPDH高度的可溶性更适合进行D-氨基酸的合成。
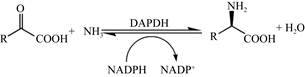
|
| 图 1 DAPDH还原胺化2-酮酸合成D-氨基酸 Figure 1 2-keto acid was reductived amination to produce D-amino acid by DAPDH |
|
|
利用DAPDH催化2-酮酸合成D-氨基酸最早是通过来源于谷氨酸棒状杆菌(Corynebacterium glutamicum)的DAPDH(CgDAPDH)改造实现的[28],2012年Toshihisa Ohshima团队和本实验室分别发表了来源于嗜热球形脲芽胞杆菌(Ureibacillus thermosphaericus strain A1)的DAPDH(UtDAPDH)[29]和来源于嗜热共生杆菌(Symbiobacterium thermophilum)的DAPDH(StDAPDH)[30]合成D-氨基酸。与CgDAPDH和UtDAPDH需要对其进行改造才能合成D-氨基酸不同的是,本实验室报道的StDAPDH不仅具有极好的热稳定性,并且无需任何改造就可以高立体选择性地还原胺化短链2-酮酸合成对应的D-氨基酸[30]。通过对StDAPDH进行三位点突变,其辅酶偏好性由Nicotinamide adenine dinucleotide phosphate(NADPH)转变为Nicotinamide adenine dinucleotide(NADH)[31]。在已经报道的DAPDH家族中,StDAPDH可以催化烷基取代2-酮酸,尤其是丙酮酸(2.87 U/mg)[30],不过其较低的酶活限制了DAPDH的进一步应用。此外,DAPDH家族结构与功能之间的关系,尤其是关键氨基酸残基对不同底物的催化机制缺乏深入研究。DAPDH对烷基取代2-酮酸的低活性也是助推本研究的另一大瓶颈。StDAPDH的晶体结构已经被解析[32],通过改造可以提高其对大位阻芳香族取代2-酮酸的活力[33-34]。本研究将通过分子改造提高StDAPDH对烷基取代2-酮酸底物的催化活力,并进一步解析酶活力提高的催化机制,以期为以后的改造工作提供可靠的依据。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒感受态细胞Escherichia coli BL21(DE3)和表达载体pET-32a(+)及质粒pET-32a-stdapdh均由本实验室保存。
1.1.2 培养基建立突变体库的培养基为Terrific-Broth(TB)培养基:胰蛋白胨12.00 g,酵母提取物24.00 g,甘油4.00 mL,去离子水定容至900 mL,1×105 Pa高压灭菌30 min,冷却至60 ℃以下时,添加100 mL无菌的磷酸盐溶液(2.31 g KH2PO4和12.54 g K2HPO4)。表达纯化的培养基为LB培养基(g/L):胰蛋白胨10.00,酵母提取物5.00,NaCl 10.00,1×105 Pa高压灭菌30 min。
1.1.3 主要试剂和仪器DNA聚合酶KOD Plus,TOYOBO公司;限制性内切酶Dpn I,New England Biolabs公司;烟酰胺辅酶NADP+和NADPH,罗氏公司;GeneRuler 1 kb DNA Ladder,Thermo公司;质粒提取试剂盒和核酸纯化试剂盒,Omega公司;氨苄青霉素(AMP)和异丙基-β-D-硫代半乳糖苷(IPTG),生工生物工程(上海)股份有限公司;2-酮酸底物以及其他分析纯试剂,Sigma公司;引物由华大基因公司合成。高压匀浆机,北京天恩瀚拓科技有限公司;蛋白纯化仪,通用电气公司;电泳仪、凝胶成像仪,Bio-Rad公司;酶标仪,美谷分子仪器(上海)有限公司。
1.2 方法 1.2.1 构建突变体库将来源于Symbiobacterium thermophilum IAM14863的DAPDH编码基因作为目的基因,以实验室之前构建的pET32a-stdapdh质粒作为模板,以NNK作为简并密码子,设计引物(表 1)对H227位进行定点饱和突变,借助QuickChange Mutagenesis协议,利用简并密码子NNK可以编码20种氨基酸的特性,让227位除本身的组氨酸外,可以突变为其他19种氨基酸中的任何一种。PCR反应体系(50 μL):dNTPs(2 mmol/L) 5 μL,10×PCR缓冲液5 μL,MgSO4(25 mmol/L) 3 μL,模板DNA(50−100 ng) 1 μL,上、下游引物(100 μmol/L)各1 μL,KOD PLUS DNA聚合酶(1 U/μL) 1 μL,ddH2O 33 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 30 s,63 ℃ 30 s,68 ℃ 7 min,20个循环;68 ℃ 20 min。得到的PCR产物使用限制性内切酶Dpn I于37 ℃消化3 h。将消化后的PCR产物纯化后转化至E.coli BL21(DE3)感受态中,涂布在含有100 μg/mL AMP的LB平板上,倒置于37 ℃培养箱中培养过夜。
| 引物名称 Primers name |
引物序列 Primers sequence(5′→3′) |
大小 Size(bp) |
| 227F | GCTGATGGACGTTGGTNNKGGT GTTGTTATG |
31 |
| 227R | GTTCCATAACAACACCMNNACC AACGTCCATC |
32 |
| Note: Bold letters represent degenerate bases; N: A/G/C/T; K: G/T; M: A/C. | ||
从LB的AMP抗性平板上挑选单菌落,并将96个单菌落接种到含有400 μL TB培养基(AMP,100 μg/mL)的96孔无菌深孔板中,随后将96孔板在37 ℃、800 r/min孵育培养12−16 h作为种子液。将种子液以2%的接种量接种到含有相同抗性的800 μL TB培养基中,相同的条件下培养至OD600约为0.4−0.6,冷却至25 ℃后,添加终浓度为0.5 mmol/L的IPTG进行诱导,25 ℃、800 r/min培养12 h表达蛋白。诱导表达后的96孔板在4 500 r/min条件下离心20 min收集菌体,收集后的菌体加入到每个孔添加200 μL含有2 mg/mL溶菌酶的Na2CO3-NaHCO3缓冲液(200 mmol/L,pH 9.5)。并置于37 ℃、800 r/min孵育2 h以破碎细胞,破胞后在4 ℃、4 500 r/min条件下离心20 min,得到的上清酶液通过显色法进行突变体库的酶活筛选。
1.2.3 筛选方法因为StDAPDH可催化D-氨基酸氧化脱氨生成对应2-酮酸,同时将辅酶NADP+还原为NADPH[24]。在吩嗪硫酸甲酯(phenazine methosulfate,PMS,0.01 mg/mL)存在的条件下,辅酶NADPH会与氯化硝基蓝四唑(tetranitroblue tetrazolium chloride,NBT,0.15 mg/mL)产生紫色的甲臜[28, 35],利用显色进行筛选。利用DAPDH催化双向反应趋势相同的原则,筛选获得的D-氨基酸脱氢酶突变体具有高效催化2-酮酸合成对应D-氨基酸的潜力。阳性突变体测序工作委托由华大基因有限公司进行。
1.2.4 野生型和有益突变体酶的表达及纯化将野生型和活力提高突变体的单菌落接种至含有100 μg/mL AMP的LB培养基中,37 ℃、200 r/min条件下培养12−16 h。随后以2%的接种量接种至800 mL含有相同抗性的LB培养基中,同样条件下培养至OD600约0.4−0.6时添加终浓度为0.5 mmol/L的IPTG,并于25 ℃、200 r/min条件下诱导表达20 h后,5 000 r/min离心20 min收集菌体。
收集后的菌体采用含10 mmol/L咪唑的Tris-HCl(20 mmol/L,pH 8.0)缓冲液重悬菌体细胞,之后利用高压匀浆机进行破胞。获得的破胞液于4 ℃、10 000 r/min离心20 min收集上清液,收集的上清液经0.45 μm滤膜过滤后利用蛋白纯化仪进行镍柱纯化。
将纯化后的蛋白经SDS-PAGE检测正确后利用10 kD的超滤管置换成Na2CO3-NaHCO3(pH 9.0,100 mmol/L)缓冲液溶解的目的蛋白,并利用BCA法测定纯化后目的蛋白的浓度。
1.2.5 酶活力的测定以30 ℃条件下每分钟消耗1 μmol NADPH所需的酶量定义为1个酶活单位(U)。还原胺化方向活力测定[30]:20 mmol/L 2-酮酸,0.5 mmol/L NADPH,200 mmol/L NH4Cl,1−2 μL(10−30 μg/μL)的纯酶,100 mmol/L Na2CO3-NaHCO3(pH 9.0)缓冲液,反应体积0.2 mL。通过检测340 nm处的NADPH吸光值变化来表征酶活。摩尔吸光系数为6.22 L/(mmol·cm)。每组实验重复3次。
动力学参数的测定与酶活测定条件相同,2-酮酸的底物浓度选择为5−200 mmol/L,每个浓度测3次平行。利用ORIGIN软件进行非线性拟合计算出野生型和有益突变体对2-酮酸底物的Km以及Vmax,并通过测定的蛋白浓度计算出对应的kcat。
1.2.6 野生型和突变体的分子模拟Discover studio是一款基于计算生物学、生命科学和药物化学的综合软件,可以应用于蛋白质结构和功能研究、蛋白结构模拟以及蛋白质与小分子相互作用等领域,是面向生命科学领域的综合分子建模和模拟平台。利用Discover studio 4.1软件的Design protein模块(模拟和设计蛋白质结构的模块,需要已知蛋白质的晶体结构作为模板)构建野生型和H227Q突变体的结构。优化底物和蛋白分子后,借助于Flexible docking模块,并采用默认设置进行蛋白与底物分子D-丙氨酸的对接,从中选出最优构型。之后根据底物在活性中心的位置,利用Structure-Monitor-Distance分析催化距离以及Non-bond interactions分析相互作用力。
2 结果与分析 2.1 突变位点的选择及突变体库的建立和筛选我们早期的研究表明,H227对底物活力具有非常重要的作用[34],对大位阻的2-酮酸的改造工作验证了H227位是提高底物活力的关键位点[33],分别利用D-丙氨酸、D-2-氨基丁酸、D-正缬氨酸、D-谷氨酸作为底物,对H227位建立饱和突变体库并进行筛选(引物见表 1)。
建立的突变体库利用96孔板进行显色筛选,显色越快(1 min内,酶标仪测定OD580)预示突变体的酶活越高。4个突变体库中得到的阳性突变体经华大基因有限公司测序后确定的突变结果见表 2。
| D-丙氨酸 D-alanine |
D-2-氨基丁酸 D-2-aminobutyric acid |
D-正缬氨酸 D-norvaline |
D-谷氨酸 D-glutamic acid |
| H227N | H227N | H227N | H227N |
| H227Q | H227Q | H227Q | H227Q |
| H227T | H227T | H227T | H227T |
| H227S | H227S | H227S | |
| H227M | H227A | H227A | |
| H227I | H227V | H227I | |
| H227C |
将表 2中所有测序验证的突变体,经过对应2-酮酸底物验证活性后,最终根据比较结果确定了H227Q和H227N为两个较优的突变体,对4个筛选底物的比活力均具有明显提高的作用,不同突变体库得到的阳性突变体催化对应2-酮酸酶活的比较结果见图 2。
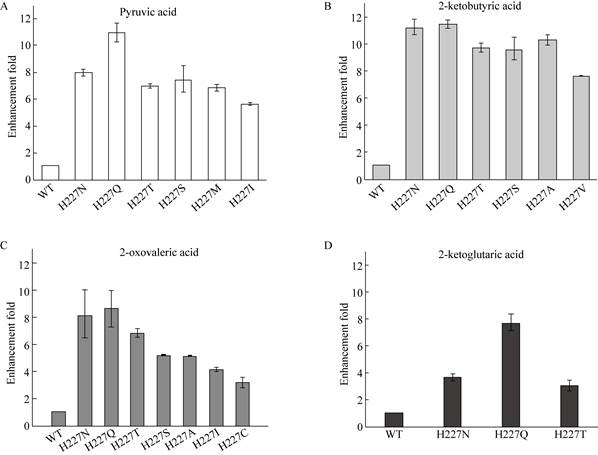
|
| 图 2 突变体酶活提高倍数的比较结果 Figure 2 Comparison results of enhanced fold of activities for mutant enzyme compared to wild-type 注:A:丙酮酸;B:2-氧代丁酸;C:2-氧代戊酸;D:2-酮戊二酸. Note: A: Pyruvic acid; B: 2-ketobutyric acid; C: 2-oxovaleric acid; D: 2-ketoglutaric acid. |
|
|
将从突变体库筛选获得的突变体进行表达纯化,纯化后的蛋白经SDS-PAGE(图 3)检测后进行酶活性质的研究。
|
| 图 3 SDS-PAGE分析野生型和突变体的纯化蛋白 Figure 3 Analysis of wild-type and mutant enzymes by SDS-PAGE 注:M:标准蛋白分子量;1:野生型;2:H227Q;3:H227N. Note: M: Protein marker; 1: Wide-type; 2: H227Q; 3: H227N. |
|
|
利用获得的突变体H227Q、H227N和实验室已有的突变体H227I及对大位阻底物活力明显提升的突变体W121L/H227I对丙酮酸、2-氧代丁酸、2-氧代戊酸、2-酮戊二酸的比活力进行测定(表 3)。
| 底物 Substrate |
菌株名称 Strains |
|||
| Wild-type | H227Q | H227N | W121L/H227I | |
| Pyruvic acid |
2.94±0.08 | 32.03±2.19 | 23.43±0.74 | 31.35±1.24 |
| 2-ketobutyric acid |
1.62±0.09 | 18.57±0.50 | 18.18±0.99 | 32.16±1.84 |
| 2-oxovaleric acid |
1.25±0.13 | 10.80±1.67 | 10.14±0.38 | 30.55±1.24 |
| 2-ketoglutaric acid |
0.81±0.04 | 6.19±0.58 | 2.95±0.22 | 0.44±0.03 |
结果表明,H227Q以及H227N对烷基取代2-酮酸底物表现出比野生型更高的活力,尤其是H227Q,对丙酮酸的比活力为32.03±2.19 U/mg,对2-酮戊二酸的比活力为6.19±0.58 U/mg,均为已报道的最高酶活力,并且是其他来源DAPDH对两个底物最高活力(0.470±0.029 U/mg[36-37],0.025 U/mg[28])的68倍和247倍。当底物的碳原子数逐渐增加到5个碳时,突变体H227Q和H227N的活力则明显下降,突变体H227Q对2-氧代戊酸的比活力仅为丙酮酸的33%;当2-氧代戊酸的5-甲基变为羧基时,酶的比活力下降了37%。突变体W121L/H227I则对丙酮酸、2-氧代丁酸和2-氧代戊酸保持高的催化活力,但是当底物为2-酮戊二酸时酶活下降了98%。
野生型与H227Q突变体的动力学参数结果(表 4)表明,不同于其他2-酮酸底物,当底物为丙酮酸时,H227Q的Km是野生型的3.3倍,同时kcat增加了2.8倍,使其催化效率(kcat/Km)提高了9.4倍;当2-氧代丁酸、2-氧代戊酸和2-酮戊二酸作为底物时,H227Q相比于野生型Km值出现增加,催化活力的提高均是kcat增加的结果,并没有增加对底物的亲和力。
| 底物 Substrate |
Wild-type | H227Q | |||||
| Km(mmol/L) | kcat(1/S) | kcat/Km(L/(mmol·s)) | Km(mmol/L) | kcat(1/S) | kcat/Km(L/(mmol·s)) | ||
| Pyruvic acid | 15.62±2.04 | 7.78±0.28 | 0.50±0.14 | 4.73±0.95 | 22.30±0.72 | 4.71±0.78 | |
| 2-ketobutyric acid | 7.96±1.04 | 2.96±0.07 | 0.37±0.07 | 15.87±3.17 | 16.09±0.86 | 1.01±0.27 | |
| 2-oxovaleric acid | 8.07±1.29 | 0.72±0.02 | 0.09±0.02 | 28.62±2.36 | 7.15±0.21 | 0.25±0.09 | |
| 2-ketoglutaric acid | 12.63±2.40 | 0.62±0.03 | 0.05±0.01 | 27.85±5.93 | 6.54±0.53 | 0.23±0.09 | |
将D-丙氨酸分别与野生型和突变体H227Q对接后,获得D-丙氨酸在野生型以及H227Q的底物结合区域中可能的构型(图 4)。通过对接结果可以看出,由于227位点的谷氨酰胺和丙氨酸的羧酸上的羰基形成氢键,D-丙氨酸的甲基朝向发生了改变,D-丙氨酸的α-H与辅酶烟酰胺环C4原子之间的距离由野生型的3.40 Å(图 4A)缩短为2.97 Å(图 4B)。
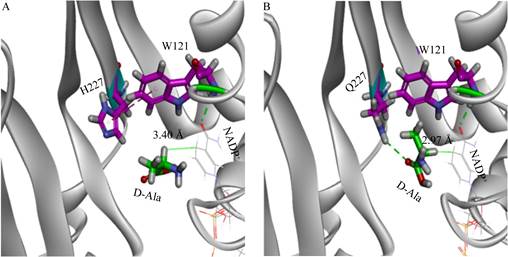
|
| 图 4 D-丙氨酸被对接到野生型和突变体蛋白的底物结合位点 Figure 4 D-alanine were docked into the substrate-binding site 注:A:D-丙氨酸在野生型酶中的位置;B:D-丙氨酸在H227Q突变体酶中的位置.绿色的实线表示D-丙氨酸的α-氢原子到烟酰胺辅酶NADP+的C4原子之间的距离,疏水相互作用和氢键显示为虚线. Note: A: D-alanine posited in wild-type enzyme; B: D-alanine posited in mutant H227Q. Green line is distance between α-hydrogen atom of D-alanine and C4 of nicotinamide mononucleotide(NADP+); Hydrophobic interactions and hydrogen bonds are shown as dotted lines. |
|
|
H227Q突变体提高酶活的可能原因是,谷氨酰胺和底物羧酸上的羰基形成氢键,使得底物的烷基侧链指向W121位点,随着烷基侧链的延长,W121位点的位阻效应越明显。因此,在H227位点上进行W121位点突变才能进一步提高更长烷基侧链取代2-酮酸底物的活力,这与我们进行芳香族取代2-酮酸底物的筛选时发现的结果一致。综上所述,227位的突变通过影响底物和周围氨基酸的相互作用,改变底物催化取向以及底物α-氢原子与烟酰胺核苷酸(NADP+) C4之间的距离,从而提高酶活性。
3 讨论与结论通过定点饱和突变的方法获得了催化烷基取代2-酮酸高活力的StDAPDH突变体。从实验结果可知,H227位突变后对烷基取代2-酮酸的催化活力发生了明显改变,其中H227Q以及H227N对丙酮酸的催化活力提高最为显著,分别提高了10倍和8倍。H227Q是目前所报道的对丙酮酸和2-酮戊二酸酶活(还原胺化方向)最高的突变体酶[28, 33]。通过动力学实验结果可知,227位突变为谷氨酰胺不仅增加了酶的转化数(kcat),并且增加了酶对底物的亲和力;除了丙酮酸外,H227Q对其他烷基取代2-酮酸的催化活力均有不同程度的提高。
对野生型和突变体分别与D-丙氨酸进行分子对接,推测了酶对直链2-酮酸比活力提高可能的催化机制。根据内消旋-二氨基庚二酸脱氢酶催化的本质是发生在底物α-C上的氢和辅酶NADP+的烟酰胺环C4之间,因此,二者之间的距离也被认为是决定酶催化效率高低的关键。实验结果显示,比活力提高的突变体相较于野生型,均不同程度地缩小了两者之间的催化距离。这一结果为StDAPDH催化效率的提高提供了强有力的证据,也进一步证明了底物结合口袋周围的氨基酸,尤其是227位对酶的催化活力起到了非常重要的作用。同时,本研究为接下来进行其他难催化底物合成对应D-氨基酸工作的开展奠定了基础。
| [1] |
Martínez-Rodríguez S, Martinez-Gómez AI, Rodríguez-Vico F, et al. Natural occurrence and industrial applications of D-amino acids: an overview[J]. Chemistry & Biodivers, 2010, 7(6): 1531-1548. DOI:10.1002/cbdv.200900245 |
| [2] |
Strömstedt AA, Pasupuleti M, Schmidtchen A, et al. Evaluation of strategies for improving proteolytic resistance of antimicrobial peptides by using variants of EFK17, an internal segment of LL-37[J]. Antimicrobial Agents and Chemotherapy, 2009, 53(2): 593-602. DOI:10.1128/AAC.00477-08 |
| [3] |
Kovacs M, Schally AV, Csernus B, et al. Luteinizing hormone-releasing hormone (LH-RH) antagonist Cetrorelix down-regulates the mRNA expression of pituitary receptors for LH-RH by counteracting the stimulatory effect of endogenous LH-RH[J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(4): 1829-1834. DOI:10.1073/pnas.98.4.1829 |
| [4] |
Coward RM, Carson CC. Tadalafil in the treatment of erectile dysfunction[J]. Therapeutics and Clinical Risk Management, 2008, 4(6): 1315-1329. |
| [5] |
Xia Q, Huang YH, Lin X, et al. Highly sensitive D-alanine electrochemical biosensor based on functionalized multi-walled carbon nanotubes and D-amino acid oxidase[J]. Biochemical Engineering Journal, 2016, 113: 1-6. DOI:10.1016/j.bej.2016.05.003 |
| [6] |
Snell EE, Guirard BM. Some interrelationships of pyridoxine, alanine and glycine in their effect on certain lactic acid bacteria[J]. Proceedings of the National Academy of Sciences of the United States of America, 1943, 29(2): 66-73. DOI:10.1073/pnas.29.2.66 |
| [7] |
Deaton DN, Graham KP, Gross JW, et al. Thiol-based angiotensin-converting enzyme 2 inhibitors: P1′ modifications for the exploration of the S1′ subsite[J]. Bioorganic & Medicinal Chemistry Letters, 2008, 18(5): 1681-1687. DOI:10.1016/j.bmcl.2008.01.046 |
| [8] |
Behrends M, Wagner S, Kopka K, et al. New matrix metalloproteinase inhibitors based on γ-fluorinated α-aminocarboxylic and α-aminohydroxamic acids[J]. Bioorganic & Medicinal Chemistry, 2015, 23(13): 3809-3818. DOI:10.1016/j.bmc.2015.03.078 |
| [9] |
Ley SV, Priour A. Total synthesis of the cyclic peptide argyrin B[J]. European Journal of Organic Chemistry, 2002, 2002(23): 3995-4004. DOI:10.1002/1099-0690(200212)2002:23<3995::AID-EJOC3995>3.0.CO;2-P |
| [10] |
Shaginian A, Rosen MC, Binkowski BF, et al. Solid-phase synthesis of dihydrovirginiamycin S1, a streptogramin B antibiotic[J]. Chemistry-A European Journal, 2004, 10(17): 4334-4340. DOI:10.1002/chem.200400267 |
| [11] |
Fraser BH, Mulder RJ, Perlmutter P. The total synthesis of pamamycin-607. Part 2: synthesis of the C6-C18 domain[J]. Tetrahedron, 2006, 62(12): 2857-2867. DOI:10.1016/j.tet.2006.01.005 |
| [12] |
Farmer JJ, Attygalle AB, Smedley SR, et al. Absolute configuration of insect-produced epilachnene[J]. Tetrahedron Letters, 1997, 38(16): 2787-2790. DOI:10.1016/S0040-4039(97)00490-5 |
| [13] |
Attygalle AB, Svatoš A, Veith M, et al. Biosynthesis of epilachnene, a macrocyclic defensive alkaloid of the Mexican bean beetle[J]. Tetrahedron, 1999, 55(4): 955-966. DOI:10.1016/S0040-4020(98)01102-8 |
| [14] |
Lennox JR, Turner SC, Rapoport H. Enantiospecific synthesis of annulated nicotine analogues from D-glutamic acid. 7-Azabicyclo[2.2.1]heptano[2.3-c]pyridines[J]. The Journal of Organic Chemistry, 2001, 66(21): 7078-7083. DOI:10.1021/jo010534y |
| [15] |
Ksander GM, Yuan AM, Diefenbacher CG, et al. Angiotensin converting enzyme inhibitors: N-substituted D-glutamic acid gamma-dipeptides[J]. Journal of Medicinal Chemistry, 1985, 28(11): 1606-1611. DOI:10.1021/jm00149a011 |
| [16] |
de Dios A, Prieto L, Martín JA, et al. 4-Substituted D-glutamic acid analogues: the first potent inhibitors of glutamate racemase (MurI) enzyme with antibacterial activity[J]. Journal of Medicinal Chemistry, 2002, 45(20): 4559-4570. DOI:10.1021/jm020901d |
| [17] |
Koeller KM, Wong CH. Enzymes for chemical synthesis[J]. Nature, 2001, 409(6817): 232-240. DOI:10.1038/35051706 |
| [18] |
Hollmann F, Arends IWCE, Holtmann D. Enzymatic reductions for the chemist[J]. Green Chemistry, 2011, 13(9): 2285-2314. DOI:10.1039/c1gc15424a |
| [19] |
Bezsudnova EY, Popov VO, Boyko KM. Structural insight into the substrate specificity of PLP fold type IV transaminases[J]. Applied Microbiology and Biotechnology, 2020, 104(6): 2343-2357. DOI:10.1007/s00253-020-10369-6 |
| [20] |
Aganyants H, Weigel P, Hovhannisyan Y, et al. Rational engineering of the substrate specificity of a thermostable D-hydantoinase (Dihydropyrimidinase)[J]. High-Throughput, 2020, 9(1): 5. DOI:10.3390/ht9010005 |
| [21] |
Wang RY, Li JF, Dang DY, et al. Bacterial production of maize and human serine racemases as partially active inclusion bodies for D-serine synthesis[J]. Enzyme and Microbial Technology, 2020, 137: 109547. DOI:10.1016/j.enzmictec.2020.109547 |
| [22] |
Martínez-Rodríguez S, Soriano-Maldonado P, Gavira JA. N-succinylamino acid racemases: enzymatic properties and biotechnological applications[J]. Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 2020, 1868(4): 140377. DOI:10.1016/j.bbapap.2020.140377 |
| [23] |
Parmeggiani F, Casamajo AR, Colombo D, et al. Biocatalytic retrosynthesis approaches to D-(2, 4, 5-trifluorophenyl) alanine, key precursor of the antidiabetic sitagliptin[J]. Green Chemistry, 2019, 21(16): 4368-4379. DOI:10.1039/C9GC01902B |
| [24] |
Abbott SD, Lane-Bell P, Sidhu KPS. Synthesis and testing of heterocyclic analogs of diaminopimelic acid (DAP) as inhibitors of DAP dehydrogenase and DAP epimerase[J]. Journal of the American Chemical Society, 1994, 116(15): 6513-6520. DOI:10.1021/ja00094a004 |
| [25] |
Olsiewski PJ, Kaczorowski GJ, Walsh C. Purification and properties of D-amino acid dehydrogenase, an inducible membrane-bound iron-sulfur flavoenzyme from Escherichia coli B[J]. The Journal of Biological Chemistry, 1980, 255(10): 4487-4494. |
| [26] |
Jones H, Venables WA. Solubilisation of D-amino acid dehydrogenase of Escherichia coli K12 and ist re-binding to envelope preparations[J]. Biochimie, 1983, 65(3): 177-183. DOI:10.1016/S0300-9084(83)80082-0 |
| [27] |
Tanigawa M, Shinohara T, Saito M, et al. D-amino acid dehydrogenase from Helicobacter pylori NCTC 11637[J]. Amino Acids, 2010, 38(1): 247-255. DOI:10.1007/s00726-009-0240-0 |
| [28] |
Vedha-Peters K, Gunawardana M, Rozzell JD, et al. Creation of a broad-range and highly stereoselective D-amino acid dehydrogenase for the one-step synthesis of D-amino acids[J]. Journal of the American Chemical Society, 2006, 128(33): 10923-10929. DOI:10.1021/ja0603960 |
| [29] |
Akita H, Doi K, Kawarabayasi Y, et al. Creation of a thermostable NADP+-dependent D-amino acid dehydrogenase from Ureibacillus thermosphaericus strain A1 meso-diaminopimelate dehydrogenase by site-directed mutagenesis[J]. Biotechnology Letters, 2012, 34(9): 1693-1699. DOI:10.1007/s10529-012-0952-1 |
| [30] |
Gao XZ, Chen X, Liu WD, et al. A novel meso-diaminopimelate dehydrogenase from Symbiobacterium thermophilum: overexpression, characterization, and potential for D-amino acid synthesis[J]. Applied and Environmental Microbiology, 2012, 78(24): 8595-8600. DOI:10.1128/AEM.02234-12 |
| [31] |
Zhao LM, Liu WD, Chen X, et al. Effect of residue Y76 on co-enzyme specificity of meso-diaminopimelate dehydrogenase from Symbiobacterium thermophilum[J]. Chinese Journal of Biotechnology, 2015, 31(7): 1108-1118. 赵雷明, 刘卫东, 陈曦, 等. 嗜热共生杆菌内消旋-2, 6-二氨基庚二酸脱氢酶中Y76对辅酶偏好性影响[J]. 生物工程学报, 2015, 31(7): 1108-1118. |
| [32] |
Liu WD, Li Z, Huang CX, et al. Structural and mutational studies on the unusual substrate specificity of meso-diaminopimelate dehydrogenase from Symbiobacterium thermophilum[J]. ChemBioChem, 2014, 15(2): 217-222. DOI:10.1002/cbic.201300691 |
| [33] |
Cheng XK, Chen X, Feng JH, et al. Structure-guided engineering of meso-diaminopimelate dehydrogenase for enantioselective reductive amination of sterically bulky 2-keto acids[J]. Catalysis Science & Technology, 2018, 8(19): 4994-5002. |
| [34] |
Gao XZ, Huang F, Feng JH, et al. Engineering the meso-diaminopimelate dehydrogenase from Symbiobacterium thermophilum by site saturation mutagenesis for D-phenylalanine synthesis[J]. Applied and Environmental Microbiology, 2013, 79(16): 5078-5081. DOI:10.1128/AEM.01049-13 |
| [35] |
Mayer KM, Arnold FH. A colorimetric assay to quantify dehydrogenase activity in crude cell lysates[J]. Journal of Biomolecular Screening, 2002, 7(2): 135-140. DOI:10.1177/108705710200700206 |
| [36] |
Hayashi J, Seto T, Akita H, et al. Structure-based engineering of an artificially generated NADP+-dependent D-amino acid dehydrogenase[J]. Applied and Environmental Microbiology, 2017, 83(11): e00491-17. DOI:10.1128/AEM.00491-17 |
| [37] |
Akita H, Hayashi J, Sakuraba H, et al. Artificial thermostable D-amino acid dehydrogenase: creation and application[J]. Frontiers in Microbiology, 2018, 9: 1760. DOI:10.3389/fmicb.2018.01760 |
 2020, Vol. 47
2020, Vol. 47




