扩展功能
文章信息
- 何媛秋, 龙丽娟, 田新朋
- HE Yuan-Qiu, LONG Li-Juan, TIAN Xin-Peng
- 酸微菌纲(Acidimicrobiia)的研究概况
- Recent advances in the class Acidimicrobiia
- 微生物学通报, 2020, 47(6): 1945-1957
- Microbiology China, 2020, 47(6): 1945-1957
- DOI: 10.13344/j.microbiol.china.190774
-
文章历史
- 收稿日期: 2019-09-25
- 接受日期: 2019-12-31
- 网络首发日期: 2020-03-19
2. 中国科学院大学 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
酸微菌是酸微菌纲(Acidimicrobiia)下所有菌的统称,隶属于放线菌门(Actinobacteria),最早由Brierley[1]在1978年从铜浸出矿和温泉中分离获得。起初酸微菌因其具有氧化Fe2+和较强的CO2固定能力,能将酸溶性矿和非酸溶性矿中的重金属沥出[2],而被运用于生物浸矿过程。除了分布于酸性环境中外,酸微菌在中性和弱碱性的河口[3]和海水[4]中也广泛分布,是水体中放线菌的优势类群。随着更多酸微菌的分离与鉴定,人们发现酸微菌具有多样的生理特性和广泛的分布范围,且不同环境中发现的酸微菌在培养条件和理化特征上表现出了较大差异。酸微菌纲目前包含3大科级类群,其中Acidimicrobiaceae类群以中度嗜热、嗜酸、氧化Fe2+和还原Fe3+为特点,主要分离自pH为2.0的酸性矿区环境中[5];Iamiaceae类群普遍分布在中温、中性或偏碱性的海洋、湖泊等水体环境[6];Ilumatobacteraceae类群主要分离自于河口[7]、浅滩[8]和沙漠土壤[9]。此外,位于暂定种状态(Candidatus)的‘Microtrichaceae’类群则被发现于活性污水中[10-11]。目前人们对酸微菌的研究还处在基础的鉴定和分类阶段,免培养生物多样性研究表明很多酸微菌还未获得纯培养菌株。因此,开展酸微菌纲菌种的研究,发掘全新的放线菌资源,有利于对酸微菌独特的生理代谢途径、全基因组功能基因挖掘、极端环境微生物的演化以及全新活性化合物发现及应用等均具有重要意义。
1 酸微菌纲的建立与发展Acidimicrobium ferrooxidans (Am. ferrooxidans)是酸微菌纲中首个成功分离的菌种,早期Am. ferrooxidans通过形态和营养方式被认为是硫化杆菌属(Sulfobacillus)的一个种(S. thermosulfidooxidans)[12]。但随后Clark等[13]于1996年发现该菌与硫化杆菌在细胞长度、基因组(G+C)mol%含量和Fe2+氧化能力存在显著差异,将其重新归类为Acidimicrobium新属下的一个新种。在1996年,Blackall等[10]从澳大利亚工业废水中分离得到酸微菌纲的新属新种‘Candidatus Microthrix parvicella’。Stackebrandt等[14]于1997年运用16S rRNA基因序列对放线菌纲进行了系统发育分析,首次建立了酸微菌亚纲(Acidimicrobidae)并将Acidimicrobium归至Acidimicrobiaceae。在2009年Ferrimicrobium、Ferrithrix[15]、Iamia[16]和Ilumatobacter[7] 4个新属发表,2011年至2018年又相继发表了新属Aciditerrimonas[17]、Aquihabitans[18]、Acidithrix[19]和Desertimona[9]。目前酸微菌纲含有1目、3科、10属、13种。除Ilumatobacter和Microthrix内分别含有3个种和2个种,其余属内只有单一种。表 1展示了目前已分离到的酸微菌菌株的相关信息。
| 时间 Published time |
科名 Family |
属名 Genus |
种名 Species |
分离环境 Isolated environment |
分离培养基/组分 Isolated medium/ingredient |
生长pH Growth pH |
培养温度 Growth temperature (℃) |
参考文献 References |
| 1996 | Acidimicrobiaceae | Acidimicrobium | Ferrooxidans | Geothermal site | Pyrite, Fe2+, yeast extract |
nd | 48 | [13] |
| 2009 | Ferrimicrobium | Acidiphilum | Mine water | Pyrite medium | nd | 30 | [15] | |
| 2009 | Ferrithrix | Thermotolerans | Mine water | Iron/yeast extract medium |
nd | 45 | [15] | |
| 2011 | Aciditerrimonas | Ferrireducens | Solfataric soil | Sulfolobus medium with yeast extract |
2.5 | 55 | [17] | |
| 2015 | Acidithrix | Ferrooxidans | Acid mine drainage | Tryptone soya broth with Fe2+ |
2.5 | 25 | [19] | |
| 1996 | Iamiaceae | Microthrix | Parvicella | Activated sludge plants | R2A agar | nd | 25 | [10] |
| 2006 | Calida | Activated sludge plants | R2A agar | nd | 25 | [11] | ||
| 2009 | Iamia | Majanohamensis | Sea cucumber | SN medium | 7.0-8.0 | 30 | [16] | |
| 2013 | Aquihabitans | Daechungensis | Eutrophic water | 50% R2A agar | 7.0-8.0 | 25 | [18] | |
| 2009 | Ilumatobacteraceae | Ilumatobacter | Fluminis | Estuary sediment | R medium | 7.0-11.0 | 25 | [7] |
| 2011 | Nonamiense | Seashore sand | R medium | 6.0-10.0 | 25 | [8] | ||
| 2011 | Coccineum | Seashore sand | R medium | 6.0-8.0 | 25 | [8] | ||
| 2018 | Desertimonas | Flava | Desert sand | 10% trypticase soya agar |
6.0-7.0 | 28 | [9] | |
| 注:nd:未知. Note: nd: No data available. |
||||||||
1997年Stackebrandt等[14]将酸微菌亚纲(Acidimicrobidae)归入放线菌纲中,2009年Zhi等[20]利用16S rRNA基因分类系统对放线菌纲重新进行了系统发育分析,同时修订了Acidimicrobiaceae的部分16S rRNA基因序列,但并没有改变酸微菌亚纲的分类地位。2012年Ludwig等[21]提议将酸微菌亚纲独立出放线菌纲,作为放线菌门(Actinobacteria)中的酸微菌纲(Acidimicrobiia),以保持放线菌纲中物种特性的一致性。同年,Gao等[22]尝试运用保守特征性插入缺失(conserved signature indels,CSIs)和不同的保守蛋白基因(conserved signature proteins,CSPs),包括细胞色素氧化酶1亚基Cox1、三磷酸胞苷合成酶(cytidine triphosphate synthetase,CTPS)、葡萄糖胺果糖-6-磷酸氨基转移酶(glucosamine-fructose-6-phosphate aminotransferase,GFT)、甘氨酰-tRNA合成酶(glycyl-tRNA synthetase,GlyRS)等9种蛋白基因对放线菌门系统发育框架进行分析,也提议将酸微菌亚纲分离出放线菌纲,升级为独立的酸微菌纲。从图 1 (引自Ludwig等[21])可以看出,酸微菌纲在进化关系上与放线菌门中其他纲相距较远,应作为单独的进化类群。2018年Asem等[9]提议建立新科Ilumatobacteraceae,与Acidimicrobiaceae、Iamiaceae等共同归类于酸微菌目(Acidimicrobiales)。基于16S rRNA基因序列重建的酸微菌纲菌种的系统发育树(图 2)显示出4个主要类群,其中来自海洋、沙漠和活性污泥等中性环境的Iamiaceae、Ilumatobacteraceae和‘Microtrichaceae’类群聚成一支,而来自酸性矿石废水的Acidimicrobiaceae自成一支。Hu等[23]通过分析7个酸微菌纯培养菌株的全基因组序列和从宏基因组数据中拼接获得的13个酸微菌全基因组序列,并与非酸微菌类群进行比较,找到3个酸微菌纲特有的蛋白序列中的CSIs,这些CSIs分别位于RNA聚合酶的β亚基、转录终止因子Rho和CCA tRNA核苷酸转移酶的编码基因。同时Hu等[23]还发现了5个酸微菌纲特有的CSPs。通过对酸微菌基因组数据的比较分析和系统发育树构建,Hu等[23]还提议在Acidimicrobiaceae和Iamiaceae的基础上增加Ilumatobacter类群和‘Microtrix’类群,这和Asem等[9]提议的系统发育地位相符合。

|
| 图 2 基于16S rRNA基因序列的酸微菌纲物种的系统发育树 Figure 2 Reconstructed neighbour-joining tree showing the phylogenetic relationships of the class Acidimicrobiia based on the 16S rRNA gene sequences 注:发育树节点数值表示Bootstrap值;发育树标尺为0.02,表示平均每个核苷酸位置有0.02个差异. Note: Numbers at nodes in the phylogenetic tree represent bootstrap percentages; Bar is 0.02, representing 0.02 substitutions per nucleotide position. |
|
|
由于地理分布环境的多样性,酸微菌不同类群之间表现出明显不同的生理特性。表 2列出了目前Acidimicrobiaceae、Iamiaceae和Ilumatobacteraceae中有效发表的9个属的生理特点。从表 2可以看出,酸微菌一般呈棒状或细杆状,部分种类具有运动能力。酸微菌中Acidimicrobium、Ferrithrix和Aciditerrimonas的菌种表现出中度嗜热的特性,其余菌种为常温菌。Acidimicrobiaceae的菌种最初分离自pH值为2.0左右的酸性矿环境中,具有嗜酸的特点,常用的培养基为含有Fe2+和Fe3+的胰酪大豆胨培养基(tryptic soy broth,TSB)、改良铁硫葡萄糖酵母培养基(glucose yeast extract ferrous sulfate,GYEFe)和改良硫化叶属培养基(modified Sulfolobus,MS)[13, 15, 17];而Iamiaceae的种类分离自中性或弱碱性的湖泊、海洋和沙漠环境,不能在酸性培养基中生长,菌种生长用的培养基为海水琼脂培养基(marine agar 2216,MA,DifcoTM)或R2A琼脂培养基(R2A agar,DifcoTM)[7, 16, 18]。酸微菌细胞壁肽聚糖类型多为meso-DAP (meso-diaminopimelic acid)[5-6],但Ilumatobacteraceae下属种为LL-DAP (LL-diaminopimelic acid)[7-9]。主要的甲基萘醌有MK-9(H8)、MK-9(H4)、MK-9(H6)和MK-8(H10)[5-6]。大部分酸微菌主要脂肪酸类型为i-C16:0,特殊的情况存在于I. majanohamensis为C17:0[16],D. flava为C16:0[9],A. daechungensis为C16:1ω5c[18]。

|
目前发现的酸微菌都能进行异养生长,为好氧或兼性厌氧菌,其中分离自酸性矿环境中的Acidimicrobiaceae下的Am. ferrooxidans和A. ferrireducens还可利用环境中的Fe2+和O2作为电子供体和受体进行化能自养生长,通过Fe2+氧化系统将CO2固定为有机化合物而获得能量[13, 17]。此外,Acidimicrobiaceae下的种在缺氧条件下能以有机物或H2作为电子供体进行生物异化铁还原反应,将电子传递给Fe3+,产生Fe2+和自身生命活动所需能量[24]。Iamiaceae和Ilumatobacteraceae分离自海洋、湖泊和沙漠环境,营异养生长,其氧化Fe2+或还原Fe3+未见报道。‘Microtrichaceae’类群分离自活性污泥,‘M. parvicella’和‘M. calida’营异养生长,由于生长缓慢,极难培养,大部分生理和生化实验无法进行[10]。其中‘M. calida’可利用长链脂肪酸作为碳源,但不能利用单糖和挥发性脂肪酸[25]。然而‘M. calida’可利用的碳源较少,仅发现它能利用挥发性脂肪酸作为碳源[11]。
4 酸微菌的分布及多样性 4.1 酸微菌的地理分布虽然目前成功分离与鉴定的酸微菌来自酸性矿废水、海洋、湖泊和沙漠环境,但宏基因组分析显示酸微菌在海洋和陆地环境的微生物类群中占有一定比例。例如2016年Chen等[26]对西南印度洋脊8个不同地点的样品进行放线菌多样性分析,发现酸微菌纲菌种在该地区所有放线菌中比例最高,其中酸微菌目(Acidimicrobiales)在8个样品中的分布频率范围为2.1%−27.0%,此外未确定分类地位的放线菌16S rRNA基因序列中80%以上可归至酸微菌目。陈柔雯[27]对29个南海沉积物样品(深度42−4 280 m)进行了宏基因组测序与分析,发现酸微菌的平均相对丰度为1.24%−3.34%,约占放线菌类群的75%−85%。陈淇等[28]对三亚鹿回头岸礁区两个优势物种澄黄滨珊瑚和丛生盔型珊瑚中的放线菌种类进行分析,发现酸微菌目的克隆子分别占文库序列总数的56.7%和51.0%。除了印度洋和南海,调查显示在黄海和渤海[29]、红海[30]、地中海东部与挪威海域沉积物[31]、北冰洋沉积物[32]的宏基因组分析中均有酸微菌存在,其中在黄海和渤海海域细菌类群中所占比例分别为3%−12%,在红海占比例约为5%。除海洋环境外,酸微菌在陆地环境也广泛分布,如酸微菌在内陆水库细菌类群中占比1.5%−3.9%[33],在德国东部矿区pH 4.0−6.0的湖泊中酸微菌占细菌类群的13.0%[34],在澳大利亚等地的工厂废水和污泥中酸微菌类群易产生大量泡沫,影响废水中固体污染物的分离效果[35]。中国学者在中国西南部亚热带不同植被森林土壤中发现酸微菌占比1.24%−1.75%[36],在中国滇西北高寒地土壤环境中酸微菌类群在植被退化的草原中含量显著上升[37],在中国西部冰川前沿退缩地土壤中发现酸微菌为优势类群,占所有细菌类群总量的19%[38]。此外,在沙漠土壤环境也发现有酸微菌的存在[9]。这些结果显示出酸微菌类群在全球范围内广泛分布,且大部分为未被发现和培养的种类,资源储量较大,可能具有重要的生态功能。
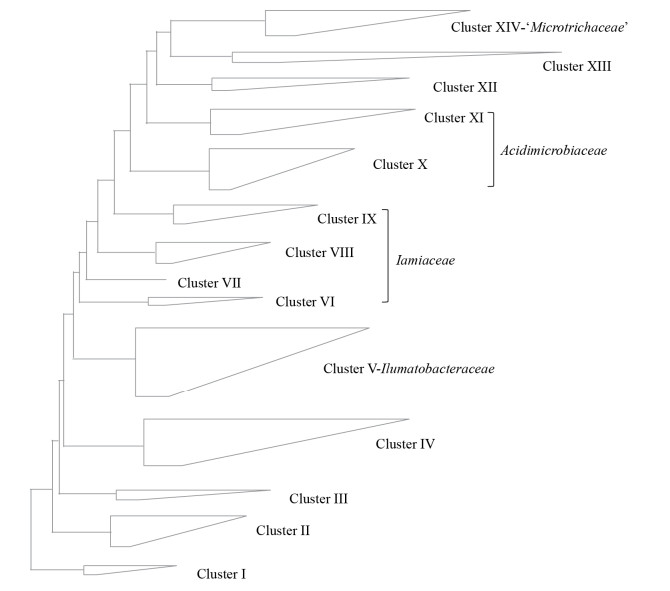
4.2 酸微菌的多样性酸微菌的难培养性限制了人们对酸微菌的深入研究。目前酸微菌纲仅有效发表了10属13个种。核糖体数据库(Ribosomal Database Project,RDP,V11.5)数据显示,至少有75个酸微菌菌株还处在已发现但未被有效描述的阶段,例如‘Acidithiomicrobium’ species[39]、Acidimicrobiaceae新成员RAAP-2和RAAP-3[40]等。从宏基因组样品中得到的酸微菌免培养序列多达17 072条,其中4 286条序列长度大于1 200 bp (表 3)。将核糖体数据库中所有酸微菌的序列(纯培养和免培养中序列长度大于1 200 bp共4 369条)进行建树,结果显示酸微菌群体庞大,至少包括1 168个酸微菌物种,可分为14个类群,其中6个类群:类群十四(Cluster XIV)、类群十一(Cluster XI)、类群十(Cluster X)、类群九(Cluster IX)、类群六(Cluster VI)和类群五(Cluster V)包含有已正式发表的菌种(图 3),其余均为未知类群。值得关注的是,当前Acidimicrobiaceae和Iamiaceae下不同属的菌种分散至不同的类群中,因此随着更多酸微菌纯培养的发现,这两个科级类群有被拆分的可能。现有数据聚类的14个类群中,类群十(Cluster X)至类群十四(Cluster XIV)主要分离自酸性矿区、污水处理厂活性污泥和地热区,类群五(Cluster V)至类群九(Cluster IX)的主要来源为中性或弱碱性的湖泊和海洋水体,而类群一(Cluster I)至类群四(Cluster IV)中至今未获得纯培养菌种,显示出酸微菌类群多样性和巨大的资源潜力。
| 分类地位 Taxon |
已发表种 Published culturable strain |
纯培养序列(未发表) Unpublished culturable strain |
免培养序列 Culture-independent sequences |
| Acidimicrobium | 1 | 7 | 6 |
| Ferrimicrobium | 1 | 12 | 73 |
| Ferrithrix | 1 | 1 | 6 |
| Acidithrix | 1 | 1 | 0 |
| Aciditerrimonas | 1 | 8 | 661 |
| Ilumatobacter | 3 | 4 | 674 |
| Iamia | 1 | 1 | 76 |
| Aquihabitans | 1 | 3 | 453 |
| Desertimonas | 1 | 0 | 0 |
| Unclassified Acidimicrobiaceae | 0 | 3 | 26 |
| Unclassified Iamiaceae | 0 | 17 | 1 306 |
| Unclassified Acidimicrobineae | 0 | 18 | 1 005 |
| 总计Total | 10 | 75 | 4 286 |
| 注:数据来自Ribosomal Database Project (RDP),版本11.5. Note: Data from Ribosomal Database Project (RDP), V11.5. |
|||
|
| 图 3 基于RDP数据库酸微菌纲下4 369条16S rRNA基因序列构建的系统发育树 Figure 3 Reconstructed neighbour-joining tree of the class Acidimicrobiia based on 4 369 16S rRNA gene sequences from RDP database 注:数据来自Ribosomal Database Project (RDP),版本11.5. Note: Data from Ribosomal Database Project (RDP), V11.5. |
|
|
酸微菌中某些化能自养菌能够以CO2为碳源、Fe2+为能量来源,将Fe2+氧化为Fe3+,产物Fe3+可作为酸溶性矿和非酸溶性矿的氧化剂,使矿石中的重金属以溶解硫酸盐的形式沥出或裸露在矿石表面以利于进一步的金属提取。Tanaka等[41]的研究表明,Am. ferrooxidans、Sineobacillus sibiricus和Acidithiobacillus caldus的混合培养物可将矿石中的铁和砷元素从其硫化物的混合物中以溶解态的形式提取出来,并使矿石中金和银的回收率从1.1%和3.2%提升至86%和87%。Davis-Belmar等[39]发现了同时具有S0和Fe2+氧化能力的化能自养菌‘Acidithiomicrobium’物种。同时,Acidimicrobiaceae种类还具有异养或混合营养型的营养方式,可作为浸矿微生物群落中有机物质循环的一部分,分解利用对亚铁氧化菌和硫氧化菌生存不利的有机物质,维持浸矿微生物群落的平衡与稳定。Cleaver等[42]在2007年分离出一种疑似Acidimicrobium的新菌种,研究表明该菌种能持续高效的在镍矿中进行生物浸矿作用,具有很大的工业应用潜力。此外,与高温和常温浸矿菌相比,在实际应用中金属硫化矿的矿堆浸物温度高达50 ℃,恰好满足中等嗜热的酸微菌的生长条件,酸微菌可以在此温度下迅速生长和参与生物浸矿作用,不需冷却设备。刘文彦等[43]在2016年申请了一项中温浸矿复合菌系的专利,其中81%的菌种为Am. ferrooxidans,实践证明该菌系具有较好的铜、铁浸出效率,具有广阔的应用前景。
5.2 化工合成研究人员对酸微菌所产生的某些特殊功能的酶类进行了开发与研究工作,Kim等[44]将来自16个不同种类的细菌ω-转氨酶(ω-transaminases)基因片段进行克隆表达和活性筛选,发现I. coccineus的ω-转氨酶能够促进西他列汀(sitagliptin)的关键前体model β-amino acid (R)-3-amino-4-(2, 4, 5-triflurophenyl) butanoic acid (3-ATfBA)的转化,转化率达81.9%。Koopmeiners等[45]在I. coccineus中发现了卤代醇脱卤素酶(halohydrin dehalogenases,HHDHs),该酶在环己烯氧化物和氧化苎烯的叠氮分解反应过程中表现出较高的催化活性,可进一步应用于化工合成领域。
5.3 污水处理Huang等[46]在美国新泽西州一处河岸湿地中发现一株Acidimicrobiaceae sp. A6菌株,与Am. ferrooxidans、F. acidiphilum 16S rRNA基因序列相似度分别为90%、92%,为潜在酸微菌新属种,该菌可在厌氧条件下进行铁还原氨氮氧化反应(feammox),在还原Fe3+的同时将NH4+氧化为氮气或硝态氮,为工业废水中氨氮的去除提供了新思路。此外,在城市污水处理过程中会产生大量含有重金属的污泥,可利用嗜酸性细菌混培物对其中的重金属进行生物浸出[47],Am. ferrooxidans可作为混培物的一部分,氧化污泥中的Fe2+,提供具有还原力的Fe3+使重金属浸出,从而达到污水的净化作用。
5.4 油气勘探辅助指标Wu等[48]在研究中国苏北北汉庄油田微生物群落对轻烃微渗漏的响应过程中发现,油气藏上方表层沉积物中Aciditerrimonas的菌种具有异常生长现象,这一现象可作为油气勘探的辅助指标,为轻烃微渗漏和相应轻烃氧化微生物异常提供更全面的评估,从而降低油气勘探风险,并提高油气勘探的成功率。
5.5 海洋生态系统物质输送Mori等[49]的研究表明Acidithrix的At. ferrooxidans对湖泊、海洋有机聚集体的形成具有显著的促进作用,比较代谢组学的研究数据显示At. ferrooxidans能分泌聚集诱导信号2-苯乙胺(phenethylamine,PEA),使Acidiphilium cryptum细胞聚集并进一步碰撞和吸引海水中的微型生物、有机碎屑和无机物质,形成海雪(marine snow)。海雪作为有机碳从海洋表层向海底的主要输送形式,对维持深海无光的生态系统生产力和营养物的稳定性具有重要意义。由于目前酸微菌获得纯培养的菌株有限,环境中的16S rRNA基因序列提供的信息也非常有限,因此,对酸微菌在环境中的生态作用、代谢途径和代谢产物的研究均相当有限。但随着更多纯培养菌株的获得和更多基因数据的测序和挖掘,酸微菌的生态作用和和应用价值会得到进一步的阐释与扩展。
6 酸微菌的基因组研究目前酸微菌纲中除A. daechungensis、I. majanohamensis和‘M. calida’暂无基因组数据外,其余10个有效发表的物种(Am. ferrooxidans DSM 10331[50]、I. coccineus YM16-304[51]、At. ferrooxidans Py-F3[52]、F. acidiphilum DSM 19497、F. thermotolerans DSM 19514、A. ferrireducens JCM 15389、I. fluminis DSM 18936、I. nonamiense YM16-303、D. flava SYSU D60003和‘M. parvicella’ RN1)完成了全基因组测序并公布于NCBI网站(National Center for Biotechnology Information Search database,NCBI)。另外,NCBI网站显示还有43个未有效发表的酸微菌基因组已完成测序。大量酸微菌基因组测序完成,对解析酸微菌独特的生理特性、生理机制及其在极端环境中的生态效应等具有很好的支撑作用。酸微菌的基因组大小在2.16−6.22 Mb[50]之间,包含2 122–5 812个基因。其中Am. ferrooxidans基因组内含有卡尔文循环中的关键酶——RubisCO酶的大亚基基因cbbL-1[50]和锌转运蛋白基因MntH、ZntA和CzcD[53],并表现出化能自养和锌耐受的能力。在I. coccineus基因组中初级代谢相关基因有缺失和被替代的现象,如赖氨酸和二氨基庚二酸生物合成途径中dap E基因的缺失,取而代之的是两个替补基因YM304_26990和YM304_19190[51]。At. ferrooxidans基因组中包含编码脲酶的基因类群和I型RubisCO酶基因,可能与维持胞内环境的pH稳定相关[50]。基因组数据显示‘M. parvicella’可通过长链脂肪酸的β氧化获得乙酰辅酶A来进行TCA循环,并具有较强的脂类储存能力和将NO3−还原至NO2−的能力[25]。Pinto等[40]从美国一处酸性矿废水宏基因组数据中拼接出两个Acidimicrobiaceae新成员(菌株RAAP-2和RAAP-3)的基因组,发现这两个新成员相较于Am. ferrooxidans在基因组水平上多了二价铁离子转运系统,同时在RAAP-2基因组中还检测到参与铁混合转运系统和硫酸盐还原途径的蛋白基因。此外,Mizuno等[54]从地中海叶绿素含量最大层(deep chlorophyll maximum,DCM)海水样品中通过宏基因组拼接得到4个酸微菌新类群基因组,并推测它们具有同化C2化合物和光异养的能力。
7 展望 7.1 酸微菌新种资源有待发掘目前,更多的酸微菌正待发现和鉴定,海洋、河口和湖泊等水环境成为近年来酸微菌研究关注的重点区域。酸微菌纲中目前只有效发表了1目、3科、10属、13种。除了Ilumatobacter和Microthrix属内分别含有3个和2个种,其余属内只有单一种,发现的种属过少,很多疑似酸微菌的菌种还处于无法得到纯培养阶段,能够进行系统研究的样品和资料有限。酸微菌作为放线菌中的稀有类群,在实验室条件下难以分离得到纯培养菌株。推测其难培养原因为实验室条件下的营养限制或理化条件不适宜,比如稀有离子、特异的pH、稀有营养组分、特异的温氧光及压力的生长需求或致死等,仍需要现代化的培养组学结合基因组学等深入研究。酸微菌类群目前在矿区发现的菌种多数能够适应pH 2.0左右的环境生活,且具有Fe2+氧化、Fe3+还原等能力,以及需氧的自养和异养生活方式,部分能够耐受高温生长,因此是生物沥滤等领域研究与应用的良好资源。海洋等中性及弱碱性水体中的酸微菌属于优势放线菌类群,但嗜中性及弱碱性的酸微菌仅成功分离并发表了3属、5种,显示出海洋等水体中的酸微菌纯培养新种资源有待深入挖掘。同时,酸微菌对酸性、中性和弱碱性环境的适应性、耐热、耐重金属等生理特性、细胞结构等方面的研究,也是细胞生物学、生理学、代谢机制、极端环境生态学等研究的良好材料和模式生物。
7.2 酸微菌纯培养菌株分离策略目前酸微菌纲菌种生活环境极端、生理学机制特殊导致其大部分仍旧难以获得纯培养,这使得对它们的生理特性、生态功能、代谢机制等全面生命现象研究难以进行。因此,酸微菌的分离和培养仍旧是对其深入研究的一个瓶颈问题。鉴于这样的情况,中国科学院热带海洋生物资源与生态重点实验室通过改良分离培养基、探索了放线菌门难培养种类红色杆菌(Rubrobacteria)的培养条件,成功获得150多株纯培养菌株,其中4株为潜在新属和新种水平分类单元,发表新物种Rubrobacter indicoceani[27]。从红色杆菌等难培养菌株成功分离的经验可知,首先,应通过常规的思路对现有酸微菌分离培养基进行改良,根据存在类群相近的已知物种的生理学特性并结合生态模拟培养的理念,有针对性地优化选择分离方案。实验室同时对海洋来源的酸微菌分离培养基SN进行改良,根据酸微菌类群生理学特点和海洋环境特点,在SN培养基中添加适量的Zn2+、Mn2+等重金属离子,获得4株酸微菌纯培养菌株,其中2株为潜在新属和新种水平分类单元(16S rRNA基因序列与其最相近物种I. majanohamensis相似性分别为93.82%和95.33%)。其次,可以通过高通量测序技术和生物信息学方法探寻环境中酸微菌的
类群,根据存在类群相近的已知物种的生理学特性寻找可能的纯培养对策。第三,分离自海洋等环境的酸微菌可在常用的2216E和R2A培养基上生长,但使用这些常用培养基分离时很少得到酸微菌,一般得到变形菌门和厚壁菌门类群,与刘玉娟等[55]对南海样品的微生物分离结果类似。由此可推测酸微菌在实验室分离条件中可能受到其他微生物的抑制。因此可从微生物相互作用入手,例如在分离培养基中添加抗生素以抑制其他优势菌的生长,加强酸微菌的优势培养。第四,通过宏基因组数据分箱(binning)技术组装获得尚未能获得独立培养的酸微菌基因组,通过对基因组的注释,建立相关初级代谢和次级代谢通路,再根据基因组中存在的基因反推其培养过程中可被利用的营养物质和生长必需的化合物,设计针对性的选择性分离培养基实现对其的纯培养。此外,不断革新的分离技术,如微生物微囊包埋技术[56]、高通量培养法[57]、微流控芯片技术[58]和微生物扩散盒[59]对酸微菌资源的分离提供了新的途径。
7.3 酸微菌基因组数据分析与功能基因发掘酸微菌的基因组大小差异显著,从基因组、比较基因组和转录组层面研究其细胞结构、物种进化、生命特征、适应环境的生理特性及其代谢机制等领域,有望产生更好的成果。虽然目前已发表的酸微菌基因组数量较少,但有文献报道其功能基因具有潜在的开发价值。从酸微菌I. coccineus YM16-304的基因组中检测到两个分别编码ω-转氨酶[44]和卤代醇脱卤素酶[45]的功能基因,其基因产物可运用于制药与化工合成领域。同时,酸微菌嗜酸类群具有耐酸和耐中温的特性,与嗜中性类群的生理特性差异较大,可从基因组水平上进行差异分析,阐明嗜酸类群的耐酸与耐热机制。此外,酸微菌还具有很多未被关注和发掘的功能基因,如亚铁氧化和铁还原基因。到目前为止,酸微菌纲中只有10个菌种进行了全基因组测序和相应的基因组注释及分析,这导致关于酸微菌代谢通路和活性产物的研究较少,难以对酸微菌进行功能和应用方面的深度分析。因此,酸微菌基因组数据的补充和分析十分必要,这不仅有利于揭示酸微菌的相关生理特性,为纯培养提供思路,还能在基因组水平上对具有不同生理代谢能力的酸微菌进行分析和比较,为未来酸微菌工程菌的遗传改造提供必需的遗传基础和背景。
| [1] |
Brierley JA. Thermophilic iron-oxidizing bacteria found in copper leaching dumps[J]. Applied and Environmental Microbiology, 1978, 36(3): 523-525. |
| [2] |
Wakeman K, Auvinen H, Johnson DB. Microbiological and geochemical dynamics in simulated-heap leaching of a polymetallic sulfide ore[J]. Biotechnology and Bioengineering, 2008, 101(4): 739-750. DOI:10.1002/bit.21951 |
| [3] |
Mai YZ, Lai ZN, Li XH, et al. Structural and functional shifts of bacterioplanktonic communities associated with spatiotemporal gradients in river outlets of the subtropical Pearl River Estuary, South China[J]. Marine Pollution Bulletin, 2018, 136: 309-321. DOI:10.1016/j.marpolbul.2018.09.013 |
| [4] |
Choi H, Koh HW, Kim H, et al. Microbial community composition in the marine sediments of Jeju island: next-generation sequencing surveys[J]. Journal of Microbiology and Biotechnology, 2016, 26(5): 883-890. |
| [5] |
Stackebrandt E. The family Acidimicrobiaceae[A]// Rosenberg E, DeLong EF, Lory S, et al. The Prokaryotes[M]. Berlin: Springer, 2014: 5-12
|
| [6] |
Yokota A. The family Iamiaceae[A]//Rosenberg E, DeLong EF, Lory S, et al. The Prokaryotes[M]. Berlin: Springer, 2014: 391-395
|
| [7] |
Matsumoto A, Kasai H, Matsuo Y, et al. Ilumatobacter fluminis gen. nov., sp. nov., a novel actinobacterium isolated from the sediment of an estuary[J]. The Journal of General and Applied Microbiology, 2009, 55(3): 201-205. DOI:10.2323/jgam.55.201 |
| [8] |
Matsumoto A, Kasai H, Matsuo Y, et al. Ilumatobacter nonamiense sp. nov. and Ilumatobacter coccineum sp. nov., isolated from seashore sand[J]. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(9): 3404-3408. |
| [9] |
Asem MD, Shi L, Jiao JY, et al. Desertimonas flava gen. nov., sp. nov. isolated from a desert soil, and proposal of Ilumatobacteraceae fam. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 2018, 68(11): 3593-3599. DOI:10.1099/ijsem.0.003038 |
| [10] |
Blackall LL, Stratton H, Bradford D, et al. "Candidatus Microthrix parvicella, " a filamentous bacterium from activated sludge sewage treatment plants[J]. International Journal of Systematic Bacteriology, 1996, 46(1): 344-346. DOI:10.1099/00207713-46-1-344 |
| [11] |
Levantesi C, Rossetti S, Thelen K, et al. Phylogeny, physiology and distribution of 'Candidatus Microthrix calida', a new Microthrix species isolated from industrial activated sludge wastewater treatment plants[J]. Environmental Microbiology, 2006, 8(9): 1552-1563. DOI:10.1111/j.1462-2920.2006.01046.x |
| [12] |
Norris PR, Clark DA, Owen JP, et al. Characteristics of Sulfobacillus acidophilus sp. nov. and other moderately thermophilic mineral-sulphide-oxidizing bacteria[J]. Microbiology, 1996, 142(4): 775-783. DOI:10.1099/00221287-142-4-775 |
| [13] |
Clark DA, Norris PR. Acidimicrobium ferrooxidans gen. nov., sp. nov.: mixed-culture ferrous iron oxidation with Sulfobacillus species[J]. Microbiology, 1996, 142(4): 785-790. DOI:10.1099/00221287-142-4-785 |
| [14] |
Stackebrandt E, Rainey FA, Ward-Rainey NL. Proposal for a new hierarchic classification system, Actinobacteria classis nov.[J]. International Journal of Systematic Bacteriology, 1997, 47(2): 479-491. DOI:10.1099/00207713-47-2-479 |
| [15] |
Johnson DB, Bacelar-Nicolau P, Okibe N, et al. Ferrimicrobium acidiphilum gen. nov., sp. nov. and Ferrithrix thermotolerans gen. nov., sp. nov.: heterotrophic, iron-oxidizing, extremely acidophilic actinobacteria[J]. International Journal of Systematic and Evolutionary Microbiology, 2009, 59(5): 1082-1089. DOI:10.1099/ijs.0.65409-0 |
| [16] |
Kurahashi M, Fukunaga Y, Sakiyama Y, et al. Iamia majanohamensis gen. nov., sp. nov., an actinobacterium isolated from sea cucumber Holothuria edulis, and proposal of Iamiaceae fam. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 2009, 59(4): 869-873. DOI:10.1099/ijs.0.005611-0 |
| [17] |
Itoh T, Yamanoi K, Kudo T, et al. Aciditerrimonas ferrireducens gen. nov., sp. nov., an iron-reducing thermoacidophilic actinobacterium isolated from a solfataric field[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(6): 1281-1285. DOI:10.1099/ijs.0.023044-0 |
| [18] |
Jin L, Huy H, Kim KK, et al. Aquihabitans daechungensis gen. nov., sp. nov., an actinobacterium isolated from reservoir water[J]. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(8): 2970-2974. |
| [19] |
Jones RM, Johnson DB. Acidithrix ferrooxidans gen. nov., sp. nov.; a filamentous and obligately heterotrophic, acidophilic member of the Actinobacteria that catalyzes dissimilatory oxido-reduction of iron[J]. Research in Microbiology, 2015, 166(2): 111-120. DOI:10.1016/j.resmic.2015.01.003 |
| [20] |
Zhi XY, Li WJ, Stackebrandt E. An update of the structure and 16S rRNA gene sequence-based definition of higher ranks of the class Actinobacteria, with the proposal of two new suborders and four new families and emended descriptions of the existing higher taxa[J]. International Journal of Systematic and Evolutionary Microbiology, 2009, 59(3): 589-608. DOI:10.1099/ijs.0.65780-0 |
| [21] |
Ludwig W, Euzéby J, Schumann P, et al. Road map of the phylum Actinobacteria[A]//Goodfellow M, Kämpfer P, Busse HJ, et al. Bergey's Manual® of Systematic Bacteriology. New York: Springer, 2012: 1-28
|
| [22] |
Gao BL, Gupta RS. Phylogenetic framework and molecular signatures for the main clades of the phylum Actinobacteria[J]. Microbiology and Molecular Biology Reviews, 2012, 76(1): 66-112. |
| [23] |
Hu DY, Cha GH, Gao BL. A Phylogenomic and molecular markers based analysis of the class Acidimicrobiia[J]. Frontiers in Microbiology, 2018, 9: 987. DOI:10.3389/fmicb.2018.00987 |
| [24] |
Hedrich S, Johnson DB. Aerobic and anaerobic oxidation of hydrogen by acidophilic bacteria[J]. FEMS Microbiology Letters, 2013, 349(1): 40-45. |
| [25] |
Mcilroy SJ, Kristiansen R, Albertsen M, et al. Metabolic model for the filamentous 'Candidatus Microthrix parvicella' based on genomic and metagenomic analyses[J]. The ISME Journal, 2013, 7(6): 1161-1172. DOI:10.1038/ismej.2013.6 |
| [26] |
Chen P, Zhang LM, Guo XX, et al. Diversity, biogeography, and biodegradation potential of Actinobacteria in the deep-sea sediments along the Southwest Indian Ridge[J]. Frontiers in Microbiology, 2016, 7: 1340. |
| [27] |
Chen RW. The diversity of the class Rubrobacteria in marine sediment environments[D]. Guangzhou: Doctoral Dissertation of South China Sea Institute of Oceanology, Chinese Academy of Sciences, 2018 (in Chinese) 陈柔雯.海洋沉积环境红色杆菌纲放线菌多样性研究[D].广州: 中国科学院南海海洋研究所硕士学位论文, 2018 |
| [28] |
Chen Q, Long LJ, Zhang S, et al. Diversity of Actinobacteria associated with coral Porites lutea and Galaxea fascicularis[J]. Microbiology China, 2014, 41(4): 691-698. (in Chinese) 陈淇, 龙丽娟, 张偲, 等. 澄黄滨珊瑚和丛生盔型珊瑚非培养放线菌多样性[J]. 微生物学通报, 2014, 41(4): 691-698. |
| [29] |
Wang K. Characteristics of bacterial community in the sediments of the Bohai and Yellow Seas, revealed by 454-Pyrosequencing[D]. Qingdao: Doctoral Dissertation of Ocean University of China, 2015 (in Chinese) 王凯.黄渤海沉积物环境细菌群落特征研究[D].青岛: 中国海洋大学硕士学位论文, 2015 |
| [30] |
Pearman JK, Kürten S, Sarma YVB, et al. Biodiversity patterns of plankton assemblages at the extremes of the Red Sea[J]. FEMS Microbiology Ecology, 2016, 92(3): fiw002. DOI:10.1093/femsec/fiw002 |
| [31] |
Pop Ristova P, Bienhold C, Wenzhöfer F, et al. Temporal and spatial variations of bacterial and faunal communities associated with deep-sea wood falls[J]. PLoS One, 2017, 12(1): e0169906. DOI:10.1371/journal.pone.0169906 |
| [32] |
Zhang GY, Cao TF, Ying JX, et al. Diversity and novelty of Actinobacteria in Arctic marine sediments[J]. Antonie van Leeuwenhoek, 2014, 105(4): 743-754. DOI:10.1007/s10482-014-0130-7 |
| [33] |
Yang H, Zhang GZ, Yang XN, et al. Microbial community structure and diversity in cellar water by 16S rRNA high-throughput sequencing[J]. Environmental Science, 2017, 38(4): 1704-1716. (in Chinese) 杨浩, 张国珍, 杨晓妮, 等. 16S rRNA高通量测序研究集雨窖水中微生物群落结构及多样性[J]. 环境科学, 2017, 38(4): 1704-1716. |
| [34] |
Lu SP, Chourey K, Reiche M, et al. Insights into the structure and metabolic function of microbes that shape pelagic iron-rich aggregates ("Iron Snow")[J]. Applied and Environmental Microbiology, 2013, 79(14): 4272-4281. DOI:10.1128/AEM.00467-13 |
| [35] |
Rossetti S, Tomei MC, Nielsen PH, et al. "Microthrix parvicella", a filamentous bacterium causing bulking and foaming in activated sludge systems: a review of current knowledge[J]. FEMS Microbiology Reviews, 2005, 29(1): 49-64. DOI:10.1016/j.femsre.2004.09.005 |
| [36] |
Meng MJ, Lin J, Guo XP, et al. Impacts of forest conversion on soil bacterial community composition and diversity in subtropical forests[J]. CATENA, 2019, 175: 167-173. DOI:10.1016/j.catena.2018.12.017 |
| [37] |
Jin ZW, Zhong WH, Wu SS, et al. Effect of vegetation degradation on microbial communities in alpine grassland soils in northwest Yunnan[J]. Acta Microbiologica Sinica, 2018, 58(12): 2174-2185. (in Chinese) 金志薇, 钟文辉, 吴少松, 等. 植被退化对滇西北高寒草地土壤微生物群落的影响[J]. 微生物学报, 2018, 58(12): 2174-2185. |
| [38] |
Zhang BL, Wu XK, Zhang GS, et al. The diversity and biogeography of the communities of Actinobacteria in the forelands of glaciers at a continental scale[J]. Environmental Research Letters, 2016, 11(5): 054012. DOI:10.1088/1748-9326/11/5/054012 |
| [39] |
Davis-Belmar CS, Norris PR. Ferrous iron and pyrite oxidation by "Acidithiomicrobium" species[J]. Advanced Materials Research, 2009, 71-73: 271-274. DOI:10.4028/www.scientific.net/AMR.71-73.271 |
| [40] |
Pinto AJ, Sharp JO, Yoder MJ, et al. Draft genome sequences of two novel Acidimicrobiaceae members from an acid mine drainage biofilm Metagenome[J]. Genome Announcements, 2016, 4(1): e01563-15. |
| [41] |
Tanaka M, Yamaji Y, Fukano Y, et al. Biooxidation of gold-, silver, and antimony-bearing highly refractory polymetallic sulfide concentrates, and its comparison with abiotic pretreatment techniques[J]. Geomicrobiology Journal, 2015, 32(6): 538-548. DOI:10.1080/01490451.2014.981645 |
| [42] |
Cleaver AA, Burton NP, Norris PR. A novel Acidimicrobium species in continuous cultures of moderately thermophilic, mineral-sulfide-oxidizing acidophiles[J]. Applied and Environmental Microbiology, 2007, 73(13): 4294-4299. DOI:10.1128/AEM.02658-06 |
| [43] |
Liu WY, Liu XY, Chen BW, et al. Composite bacterial inoculum used for leaching chalcopyrite, comprises Acidithiobacillus caldus 0Y, Acidimicrobium ferrooxidans and Ferroplasma sp: CN, CN105802869-A[P]. 2016-07-27 (in Chinese) 刘文彦, 刘兴宇, 陈勃伟, 等.一种用于黄铜矿浸出的中温浸矿复合菌系及浸矿工艺: 中国, CN105802869A[P]. 2016-07-27 |
| [44] |
Kim GH, Jeon H, Khobragade TP, et al. Enzymatic synthesis of sitagliptin intermediate using a novel ω-transaminase[J]. Enzyme and Microbial Technology, 2019, 120: 52-60. DOI:10.1016/j.enzmictec.2018.10.003 |
| [45] |
Koopmeiners J, Diederich C, Solarczek J, et al. HheG, a halohydrin dehalogenase with activity on cyclic epoxides[J]. ACS Catalysis, 2017, 7(10): 6877-6886. DOI:10.1021/acscatal.7b01854 |
| [46] |
Huang S, Jaffé PR. Characterization of incubation experiments and development of an enrichment culture capable of ammonium oxidation under iron-reducing conditions[J]. Biogeosciences, 2015, 12(3): 769-779. DOI:10.5194/bg-12-769-2015 |
| [47] |
Shi CH. The study of method and mechanism on simultaneous removal of heavy metals and improvement of sewage sludge dewaterability during bioleaching employing acidophilic bacteria[D]. Guangzhou: Master's Thesis of South China University of Technology, 2014 (in Chinese) 石超宏.嗜酸性细菌生物浸出污泥重金属同时改善脱水性能方法及机理研究[D].广州: 华南理工大学硕士学位论文, 2014 http://cdmd.cnki.com.cn/Article/CDMD-10561-1014064483.htm |
| [48] |
Wu XY, Xu XM, Wu CF, et al. Responses of microbial communities to light-hydrocarbon microseepage and novel indicators for microbial prospecting of oil/gas in the Beihanzhuang oilfield, northern Jiangsu, China[J]. Geomicrobiology Journal, 2014, 31(8): 697-707. DOI:10.1080/01490451.2013.843619 |
| [49] |
Mori JF, Ueberschaar N, Lu SP, et al. Sticking together: inter-species aggregation of bacteria isolated from iron snow is controlled by chemical signaling[J]. The ISME Journal, 2017, 11(5): 1075-1086. DOI:10.1038/ismej.2016.186 |
| [50] |
Clum A, Nolan M, Lang E, et al. Complete genome sequence of Acidimicrobium ferrooxidans type strain (ICPT)[J]. Standards in Genomic Sciences, 2009, 1(1): 38-45. DOI:10.4056/sigs.1463 |
| [51] |
Fujinami S, Takarada H, Kasai H, et al. Complete genome sequence of Ilumatobacter coccineum YM16-304T[J]. Standards in Genomic Sciences, 2013, 8(3): 430-440. DOI:10.4056/sigs.4007734 |
| [52] |
Eisen S, Poehlein A, Johnson DB, et al. Genome sequence of the acidophilic ferrous iron-oxidizing isolate Acidithrix ferrooxidans strain Py-F3, the proposed type strain of the novel actinobacterial genus Acidithrix[J]. Genome Announcements, 2015, 3(2): e00382-15. |
| [53] |
Mangold S, Potrykus J, Björn E, et al. Extreme zinc tolerance in acidophilic microorganisms from the bacterial and archaeal domains[J]. Extremophiles, 2013, 17(1): 75-85. DOI:10.1007/s00792-012-0495-3 |
| [54] |
Mizuno CM, Rodriguez-Valera F, Ghai R. Genomes of planktonic Acidimicrobiales: widening horizons for marine actinobacteria by metagenomics[J]. MBio, 2015, 6(1): e02083-14. |
| [55] |
Liu YJ, Tian XP, Huang XF, et al. Diversity of cultivable bacteria isolated from marine sediment environments in South China Sea[J]. Microbiology China, 2014, 41(4): 661-673. (in Chinese) 刘玉娟, 田新朋, 黄小芳, 等. 中国南海沉积环境可培养细菌多样性研究[J]. 微生物学通报, 2014, 41(4): 661-673. |
| [56] |
Zengler K, Walcher M, Clark G, et al. High-throughput cultivation of microorganisms using microcapsules[J]. Methods in Enzymology, 2005, 397: 124-130. DOI:10.1016/S0076-6879(05)97007-9 |
| [57] |
Colin Y, Goñi-Urriza M, Caumette P, et al. Combination of high throughput cultivation and dsrA sequencing for assessment of sulfate-reducing bacteria diversity in sediments[J]. FEMS Microbiology Ecology, 2013, 83(1): 26-37. DOI:10.1111/j.1574-6941.2012.01452.x |
| [58] |
Ma L, Kim J, Hatzenpichler R, et al. Gene-targeted microfluidic cultivation validated by isolation of a gut bacterium listed in Human Microbiome Project's Most Wanted taxa[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(27): 9768-9773. DOI:10.1073/pnas.1404753111 |
| [59] |
Bollmann A, Lewis K, Epstein SS. Incubation of environmental samples in a diffusion chamber increases the diversity of recovered isolates[J]. Applied and Environmental Microbiology, 2007, 73(20): 6386-6390. DOI:10.1128/AEM.01309-07 |
 2020, Vol. 47
2020, Vol. 47