扩展功能
文章信息
- 胡博文, 常冬冬, 王波波, 余志晟
- HU Bo-Wen, CHANG Dong-Dong, WANG Bo-Bo, YU Zhi-Sheng
- 有氧产甲烷研究进展
- Recent advances in aerobic methanogenesis
- 微生物学通报, 2020, 47(6): 1935-1944
- Microbiology China, 2020, 47(6): 1935-1944
- DOI: 10.13344/j.microbiol.china.190773
-
文章历史
- 收稿日期: 2019-09-25
- 接受日期: 2019-12-09
- 网络首发日期: 2020-01-07
甲烷是大气中重要的温室气体之一,其全球增温潜势是二氧化碳的28倍[1]。自工业革命之后,大气中甲烷含量的浓度急剧升高,达到了之前的2.5倍,由0.72 μmol/mol增加至1.82 μmol/mol (2012年)[2]。现在每年排放到大气中的甲烷量约为500−600 Tg,不断加剧着全球温室效应[3]。
甲烷排放的来源分为人为来源和自然来源,人为甲烷排放源包括水稻田、垃圾填埋和一些厌氧反应器,自然来源主要有湿地、淡水、海洋沉积物以及动物肠道等[4]。在这些甲烷排放源中,产甲烷古菌的活动是甲烷产生的重要途径。自然界中结构复杂的有机物在多种微生物的共同作用下被水解,生成如乙酸、甲醇、甲胺等结构简单的有机物作为产甲烷菌可利用的底物,最后产甲烷菌通过代谢此类底物获取能量并释放甲烷作为终产物。
根据产甲烷菌所利用的底物不同将产甲烷途径分为3种:H2/CO2 (CO2还原)途径、乙酸发酵途径和甲基化合物裂解途径,这3种途径所对应的产甲烷菌的种类也有所不同[5]。除这3种途径之外,日本的科学家2016年在地下深层发现了可利用煤炭中的甲氧基芳香族化合物产生甲烷的菌株Methermicoccus shengliensis AmaM及其近亲株Methermicoccus shengliensis ZC-1,这两株菌可以直接利用甲氧基芳香族上的甲基生成甲烷,不仅限于以上几种简单的底物,这一新的产甲烷方式可能成为第4种产甲烷途径[6]。
本课题组在煤炭厌氧转化产甲烷的研究中发现煤层中的细菌同样与甲烷产生有着密切联系。如在我国鄂尔多斯盆地和沁水盆地的煤层气田中检测到了丰富的与甲烷生成相关的产甲烷菌、细菌、真菌菌群,通过实验室培养证实了产出水中微生物的产甲烷能力[7-8];通过对煤矿气溶胶中微生物群落进行定量分析,发现细菌无论在数量还是多样性上都远高于古菌[9];在深入研究煤炭厌氧转化产甲烷的作用机制后发现细菌群落还可以对煤层中大分子物质进行一定程度的分解,增加煤的孔隙度,从而使产甲烷作用更容易进行[10]。
尽管这些细菌在甲烷产生中扮演着重要角色,却也始终不能靠自身代谢直接产生甲烷,还需产甲烷古菌的参与。由于产甲烷菌严格厌氧的特性,长时间以来对于自然界中甲烷产生的研究主要集中在土壤内部以及水底沉积物等厌氧环境中[11]。事实上,人们在不断探索的过程中发现,厌氧条件并非自然界中甲烷产生的唯一条件,甲烷的产生同样也可以在有氧条件下进行,而能够产甲烷的菌种也并非仅限于产甲烷古菌之中[12]。
本综述对自然界中有氧条件下的甲烷产生及产甲烷古菌之外的新型产甲烷方式进行了总结,让人们对微生物产甲烷机理以及碳的地球化学循环有一个更加全面的了解,为甲烷气体的产生和控制提供新的思路。
1 甲烷产生的“悖论”早在1991年的时候,Kiene等就在海洋的上层水中发现了甲烷过饱和的现象,由于海水上层中含有大量的溶解氧,与适合甲烷生成的厌氧条件相悖,所以这一现象被称为“海洋中甲烷产生的悖论(marine methane paradox)”[13]。尽管当时未能引起极大的反响,但这一产甲烷“悖论”在后续的研究中有了新的进展。
2006年Keppler等提到陆生植物可以在有氧条件下产生并释放甲烷[14],同年还有研究发现了海洋上层的束毛藻属(Trichodesmium)可在有氧条件下代谢水中有机膦酸盐产生甲烷的现象[15]。随着2007年再次有研究提到海洋上层甲烷过饱和这一现象[16],人们开始意识到严格厌氧也许并非甲烷产生的唯一方式。2008年Karl等在太平洋中发现,当水中无机磷酸盐匮乏时某些微生物可在有氧条件下代谢海水中的甲基膦酸(methylphosphonate,Mpn)来释放甲烷,并指出这是海洋中有氧甲烷产生的重要方式之一[12]。2011年Grossart等在湖泊中发现水中溶解甲烷的峰值存在于湖泊上层的有氧区域而非深处的厌氧区域中;同时水上层的藻类与有氧甲烷的产生有着密切的关系[17]。
随着一系列相关研究的开展,之前的主流说法“甲烷的产生需在严格厌氧条件下进行”不断遭到质疑。在接下来的几年里,关于“甲烷产生的悖论”这一科学问题的研究在海洋、湿地、湖泊等各个自然环境之中都获得了重要进展,涉及范围更加广泛,相关机理研究更加深入[18-22]。
2 有氧条件下的甲烷产生关于有氧条件下微生物产生甲烷的途径,大体上可分为两种观点:第一种认为甲烷仍然来自于产甲烷菌,通过自身合成的抗氧化途径或由其他特殊因素辅助,使其能够在氧气存在条件下产生甲烷[17, 20, 23-24];第二种观点是甲烷由产甲烷古菌之外的其他微生物产生,如细菌或真菌[19, 25-27]。
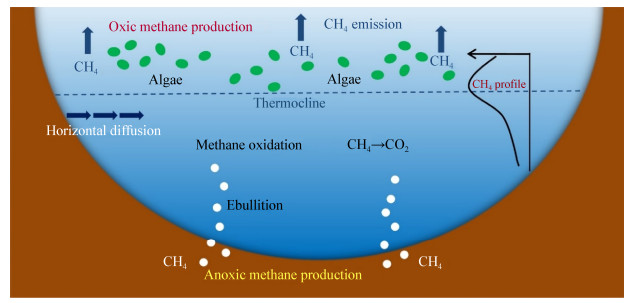
2.1 产甲烷菌的“保护伞”在第一种观点中,水或土壤有氧层中产生的甲烷仍然来源于产甲烷菌的代谢作用,主要发生在湖泊和湿地等淡水环境(图 1)。尽管微生物的厌氧代谢在自然界中十分普遍,但利用中间代谢产物或者无机物作为电子传递链末端电子受体这一途径的产能效率远不如以氧气作为终端电子受体的产能效率高,于是自然界中的许多微生物通过改变其相关基因使自己的代谢途径向好氧代谢转变。产甲烷菌也存在类似的代谢转变过程,目前已经发现沙漠中的Methanosarcina和Methanocella两个产甲烷菌属在一定程度上进化出了自我保护机制来对抗氧化[28]。在对德国施特希林湖中产甲烷作用的研究中发现,湖上层的产甲烷作用与湖中的光合作用生物量有正相关的关系;同时研究人员还通过荧光原位杂交(fluorescence in situ hybridization,FISH)技术拍到产甲烷古菌附着于湖上层水中的藻类之上,这些光合作用生物可以为产甲烷菌提供适合其生长的“厌氧微型生态龛”,并将产甲烷底物直接转移给产甲烷菌来产生甲烷[17]。鉴于此研究的合理性以及诸多证据,这一理论很快就成为主流观点之一。
|
| 图 1 水环境中有氧产甲烷作用 Figure 1 Aerobic methanogenesis in water environment |
|
|
尽管如此,也有其他研究对这种说法提出了质疑。有实验表明,氧气往往可以逐渐渗透到水中有机絮状体、颗粒物和藻类等形成的“厌氧微型生态龛”中,所以水中的这种微厌氧环境并不能持续很长时间,从而极大地限制了这种方式的甲烷生成[19]。
此外,对于水中有氧甲烷生成现象也始终存在着另一种观点:水底和岸边沉积物中会产生大量的甲烷,这些甲烷扩散到了水体上层导致了水中甲烷过饱和现象的发生[29-32]。因此,有研究设计了一个围栏实验[24],将样区的底部和四周与湖水隔开,以排除水底和岸边所产生的甲烷的干扰。实验结果表明,样区水中溶解的甲烷浓度远大于大气中甲烷的平衡浓度,并且甲烷浓度与水中初级生产力(光合作用生物)同样有着正相关的关系。这个实验很好地证明了甲烷的产生来自于有氧水层中,而非其他位置的扩散。同时,甲烷在水中的净积累量不仅仅取决于产甲烷菌的产甲烷作用,与甲烷氧化菌也有着密切的关系。一般来说,水底沉积物中所产生的甲烷会被湖上层中生存的甲烷氧化菌氧化成CO2,从而在很大程度上抑制了水中甲烷的逸出。所以一般情况下,水中甲烷的净积累量是这两类微生物共同作用后的结果。然而,Grossart等在Setchlin湖中发现,甲烷的氧化作用主要出现在水中的变温层而非湖上层[17],Tang等[23]和Murase等[33]的研究则发现甲烷氧化作用随着湖水透光层中光强度的增加而降低。这些原因使得生活在偏下层的甲烷氧化菌仅仅能够氧化湖底逸出的甲烷,却很难对水上层所产生的甲烷进行消耗。因此,这两种作用在空间上的差异导致了湖泊上层甲烷的积累。
有氧产甲烷的相关工作也在湿地中进行了开展。Angle等利用基因组学分析技术发现,湿地中一个新的分布广泛的乙酸型产甲烷菌暂定种Candidatus Paradoxum Methanothrix中存在着相应的抗氧化基因,但转录组学分析结果表明此菌种虽然存在抗氧化的基因,却并未大量表达出相应的蛋白,由此推测这个产甲烷菌种在厌氧微环境中的代谢作用仍然是这个湿地中水和土壤上层甲烷产生的主要原因[20]。
截至目前,产甲烷菌借助“厌氧微型生态龛”产甲烷的说法已经得到了一定程度的认可,相关研究仍在继续开展。尽管如此,目前对于这种产甲烷方式的机理和代谢过程的研究仍然欠缺,使得这一理论始终处于推测阶段,后续仍然需要进一步的研究对其进行证实。
2.2 细菌对特殊底物的代谢作用另一种观点主要围绕一种特殊的产甲烷底物——有机膦酸盐中的甲基膦酸展开。这个观点源于Karl等2008年在太平洋的ALOHA站点中首次发现海洋中的微生物可以好氧代谢甲基膦酸来释放甲烷[12]。这种不依赖于产甲烷古菌的甲烷生成方式极大地更新了人们对自然界中甲烷来源的认识,此后也有许多研究陆续在有机膦酸盐的来源、代谢机理以及生物间的循环中开展[18-19, 34-35]。随着研究的进展,这种产甲烷方式也从海洋延伸到了淡水环境中,预示着这一新的产甲烷机制在自然界中有着更为普遍的存在以及更加重要的意义。
2.2.1 有机膦酸盐有机膦酸盐(phosphonate)是一类在结构上区别于一般无机磷酸盐(phosphate)的自然产物,其特点是与磷酸基团相连的碳由C−P键直接连接,不同于一般磷酸盐中以C−O−P酯键的方式连接[12]。这类有机膦酸盐包括甲基膦酸、2-氨基乙基膦酸(AEP)、2-羟基乙基膦酸(HEP)等众多盐类,这些盐类中都含有C−P键并能彼此之间相互转化。C−P键的结构首次发现于2-氨基乙基膦酸中,这种化学键相比P−O键来说有着更强的化学稳定性及热稳定性,因此更难被生物所利用[36]。
截止目前,几乎全部有机膦酸盐化合物的合成都起始于磷酸烯醇式丙酮酸盐(PEP),在磷酸烯醇式丙酮酸变位酶(PepM)的作用下转变为磷酸丙酮酸盐(PnPy)[37]。此过程中的酶PepM属于异柠檬酸裂合酶的蛋白质家族,首次分离于瘤胃微生物Tetrahymena pyriformis之中[38],而最早发现的有机膦酸盐AEP也来自于这种原生动物[39]。由于PepM是有机膦酸中C−P键产生的关键酶,所以pepM基因可以作为检验有机膦酸盐合成的分子标记。2013年Yu等利用pepM基因作为分子标记对环境中大量微生物进行了检测,结果表明有机膦酸盐的生物合成途径在自然界中有着很强的普遍性与多样性,说明这类物质在自然界中有着广泛的存在[40]。之后有研究利用基因挖掘技术对1 000株放线菌产生有机膦酸盐的潜力进行了测定,所得的基因组序列信息表明其中有278株存在着不同的有机膦酸盐合成基因簇[41]。除此之外,还有许多真核生物也能够产生有机膦酸盐[42],预示着这类物质在自然界中很可能扮演着更为重要的角色。
2.2.2 甲基膦酸甲基膦酸是一种结构最简单的有机膦酸盐,分子式为CH3P(O)(OH2),分子结构为四面体构象,常温下为白色粉末状,易溶于水而难溶于有机溶剂,而海洋上层甲烷过饱和的可能原因就是水中微生物对甲基膦酸的利用[12]。甲基膦酸属于有机膦酸盐中的一种,其合成过程同样起始于磷酸烯醇式丙酮酸盐,在PepM的作用下转变为磷酸丙酮酸盐,接着转变为2-羟基乙基膦酸,最后在以二价铁作为活性中心的加氧酶MpnS作用下转变为甲基膦酸(图 2)[34, 43-44]。此过程中有两种关键酶:PepM和MpnS。其中PepM是有机膦酸盐生成的关键酶,而MpnS是产生甲基膦酸的关键酶。Metcalf等利用纯化后的MpnS蛋白和用13C标记的HEP进行反应,通过核磁共振检测出HEP在MpnS的作用下反应生成了Mpn和HCO3–[34]。

|
| 图 2 甲基膦酸的产生过程 Figure 2 The production process of methylphosphonate |
|
|
尽管可以产生有机膦酸盐的微生物在自然界中十分广泛,但甲基膦酸合成酶的基因却仅在几个物种中被发现(表 1)。有预测称合成MpnS酶的物种在自然界中也有着广泛的分布[34],但到目前为止对于MpnS这种酶来源只在海洋泉古菌门的Nitrosopumilus maritimus SCMI中有过详细的叙述[34]。之后的研究利用qPCR和酶联FISH技术得出Nitrosopumilus maritimus这类氨氧化古菌不仅在海洋中大量存在,同时也存在于淡水环境中,所以这类微生物可能也是淡水环境中Mpn的重要来源[47]。
| 序号 No. |
物种名称 Species |
来源 Resources |
分类 Categories |
文献 References |
| 1 | Nitrosopumilus maritimus SCM1 | Ocean | Thaumarchaeota | [34] |
| 2 | Candidatus Nitrosomarinus catalina | Ocean | Thaumarchaeota | [45] |
| 3 | Methanomethylovorans sp. PtaU1.Bin093 | Paddy field, Soil | Thaumarchaeota | [46] |
| 4 | Candidatus Pelagibacter ubique strain HTCC7217 | Ocean | Proteobacteria | [44] |
海洋中的微生物对甲基膦酸中C−P键的裂解是海洋上层甲烷产生的重要来源[12]。有机膦酸盐中C−P键的裂解机制在很早之前就有过相应研究,微生物可通过这种途径使许多烷基膦酸盐或芳基膦酸盐中的C−P键断裂,从而产生相应的碳氢化合物[48-49]。然而在结构最简单的甲基膦酸中,甲基和磷酸基团直接相连,其C−P键断裂后的直接产物就是甲烷。因此,当水中无机磷酸盐浓度较低时,Mpn可作为水中主要磷源被大肠杆菌、假单胞菌等许多细菌所利用,裂解其中的C−P键来产生甲烷[12, 18]。
C−P键的裂解途径在大肠杆菌这一模式菌株中研究得最为透彻。这一途径由14个基因操纵子编码,分别为phnCDEFGHIJKLMNOP[50-52]。其中phnCDE编码着有机膦酸盐的特异性转移蛋白;phnG到phnM编码着与C−P键裂解相关的蛋白;phnF以及phnN、phnO、phnP编码了这一过程的调节蛋白[53]。
甲基膦酸裂解产甲烷的过程是在PhnG到PhnM这一系列C−P键裂解相关蛋白的共同作用下完成的。在此过程的第一步反应中,PhnI在PhnG、PhnH和PhnL存在的条件下催化甲基膦酸与MgATP反应,形成腺嘌呤(adenine)和甲基膦酸核苷三磷酸(α-D-ribose-1-methylphosphonate-5-triphosphate);接着甲基膦酸核苷三磷酸中的三磷酸盐部分在PhnM作用下得到水解,形成焦磷酸盐(PPi)和甲基膦酸核苷一磷酸(α-D-ribose-1-methylphosphonate- 5-phosphate);最后甲基膦酸核苷一磷酸中的C−P键在腺苷甲硫氨酸(S-adenosyl-L-methionine,SAM)存在的条件下通过PhnJ蛋白催化的自由基反应裂解生成甲烷和环磷酸核苷一磷酸(α-D-ribose-1, 2- cyclic-phosphate-5-phosphate)[18, 35] (图 3)。PhnK蛋白所发挥的功能截至目前还不明确,但有研究通过蛋白表达和显微电镜技术推测出PhnK参与着ABC转运蛋白(ATP-binding cassette transporter)上核苷酸的结合与转移,并且也关系到PhnJ蛋白中心插入位点(central insertion domain)的组成[54-56]。上述途径中的PhnJ蛋白作用于这一系列反应中的最后一步,与甲烷的产生有着直接联系,所以编码这一蛋白的phnJ基因也成为了寻找这一反应最重要的分子标记。

|
| 图 3 甲基膦酸中C−P键裂解产甲烷过程 Figure 3 Methanogenesis by cleavage of the C−P bond in methylphosphonate |
|
|
利用C−P键裂解途径中的有机膦酸盐转移基因(phnC、phnD、phnE)和编码C−P键裂解的基因(phnGHIJKLM)作为分子标记,可以发现携带此类基因的微生物在自然界中十分广泛,如大肠杆菌、假单胞菌这些普遍存在的细菌中都有此类可以代谢有机膦酸盐的基因[12] (表 2)。
| 序号 Number |
物种名称 Species |
基因长度(核苷酸) Gene length (nt) |
蛋白长度(氨基酸) Protein length (aa) |
Gene ID (NCBI) | 文献 References |
| 1 | Burkholderia pseudomallei K96243 | 975 | 324 | 3094145 | [57] |
| 2 | Enterobacter hormaechei subsp.steigerwaltii | 846 | 281 | 34153741 | [58] |
| 3 | Pseudomonas syringae pv. tomato str. DC3000 | 888 | 295 | 1184214 | [59] |
| 4 | Yersinia pestis CO92 | 882 | 293 | 1176277 | [60] |
| 5 | Sinorhizobium fredii NGR234 | 894 | 297 | 7789425 | [61] |
| 6 | Bradyrhizobium diazoefficiens USDA 110 | 897 | 298 | 1049505 | [62] |
| 7 | Sinorhizobium meliloti 1021 | 894 | 297 | 1237786 | [63] |
| 8 | Shigella dysenteriae Sd197 | 846 | 281 | 3798885 | [64] |
| 9 | Escherichia coli IAI39 | 846 | 281 | 7152403 | [65] |
| 10 | Agrobacterium fabrum str. C58 | 903 | 300 | 1132216 | [66] |
| 11 | Rhodobacter capsulatus SB1003 | 864 | 287 | 31490109 | [67] |
| 12 | Acidithiobacillus thiooxidans ATCC19377 | 831 | 276 | 29723131 | [68] |
| 13 | Rhizobium leguminosarum bv.trifolii WSM2304 | 876 | 291 | 34195030 | [69] |
| 14 | Serratia liquefaciens ATCC27592 | 861 | 286 | 29902025 | [70] |
| 15 | Bordetella hinzii | 876 | 291 | 29513971 | [71] |
| 16 | Natronomonas moolapensis 8.8.11 | 1 050 | 349 | 14652945 | [72] |
本研究组在研究中利用phnJ基因设计引物,对湖水中提取到的微生物基因组进行了PCR扩增,也得到了与phnJ基因大小一致的条带。除了异养型微生物之外,海洋中的一些光合自养型微生物也含有此类机制。在以Trichodesmium erythraeum IMS101为代表的Trichodesmium属中,几乎在所有的菌种中都能够发现有机膦酸盐转移和水解基因;在进一步的研究中,对PhnJ蛋白的系统发育分析能够确定出3个含有此类基因的族群,分别为γ-变形菌纲、α-变形菌纲和其他细菌谱系,而Trichodesmium erythraeum IMS101就属于其他细菌谱系;除此之外,化能无机自养型的脱氮硫杆菌Thiobacillus denitrificans (β-变形菌纲)中也存在着类似的phn功能基因簇[15]。建立在功能蛋白的基础上,后续研究利用海洋中其他微生物在实验室中代谢底物Mpn产生了甲烷,证明了这一机制的存在与可行性,其中所涉及的微生物就包括Trichodesmium erythraeum IMS101菌株,以及新发现的聚球藻属Synechococcus OS-A和OS-B′ [12, 73-74]。在此之后,各种各样的相关研究不断在这一领域开展,包括Mpn来源的研究、C−P键裂解途径的研究、PhnJ蛋白结构与功能区域的研究以及寻找海洋或淡水中存在的其他此类产甲烷过程菌种等。
对于相关菌种的研究一直是此领域的热门话题,研究方法也是结合分子生物学和培养实验于一体,不断有新的功能菌被挖掘出来。2013年的研究通过宏转录组学和功能基因组学的分析方法对海洋中代谢Mpn的相关微生物类群做出了鉴定,其中的一些弧菌目(Vibrionales)和红螺菌目(Rhodobacterales)可在无机磷酸盐(Pi)缺乏的时候代谢Mpn来产生甲烷[53]。2014年Carini等通过将Mpn加入到细菌生长的培养基中,在大西洋马尾藻海中筛选到可代谢Mpn产生甲烷的Pelagibacterales sp. strain HTCC7211,同样地,无机磷酸盐的缺乏有着增强基因表达的效果[75];之后有研究在太平洋ALOHA站中发现海水中的假单胞菌株Pseudomonas stutzeri strain (HI00D01)存在着降解海水中的有机膦酸盐来产生甲烷的机制,然而其缺少phnK基因的突变株却无法实现这一过程[19]。这些研究进一步证明了C−P键裂解基因在有机膦酸盐代谢产甲烷过程中所起的的关键作用。不仅限于海洋,在淡水中也发现了相似的结论。有研究表明,从印尼的湖泊中分离出的4种细菌(Agrobacterium tumefaciens,Rhizobium sp.,Agrobacterium tumefaciens,Pantoea ananatis)均可在无机磷酸盐匮乏的情况下,通过phnJ基因编码C−P键裂解酶来降解水中的甲基膦酸释放甲烷[25];后续的研究发现淡水中的假单胞菌属Pseudomonas sp.中也存在着类似机制[26]。
与此同时,在蓝细菌门中,除束毛藻属(Trichodesmium)和聚球藻属(Synechococcus)之外,Nodularia spumigena也可利用phnJ基因编码C−P键裂解酶降解Mpn来产生甲烷[21]。以上这些研究都预示着细菌的代谢作用是水中有氧条件下甲烷产生的重要原因,相信随着相关研究的不断开展,越来越多的菌种将得到挖掘,其中的代谢机理也会得到更加透彻的阐述。
除此之外,还有新的证据表明,微藻物种可以通过去甲基化直接产生甲烷,完全绕过异养微生物的参与[76]。据报道,在一般环境大气条件下,几种有机硫化合物如甲硫氨酸、二甲基亚砜和二甲基巯基丙酸(DMSP)都可以转化生成甲烷,而此类化合物通常也可由藻类产生[77]。因此,如果能够确定藻类中也存在类似的好氧产甲烷过程,那么在有氧水域中的甲烷产生作用将比想象的更加普遍。
3 总结与展望甲烷既是大气中重要的温室气体,也是天然气的主要成分,对于生物甲烷产生的研究既可以为全球温室效应的加剧寻找解决方案,也可以为生物质能源的产生提供新途径。本文从自然界中新型产甲烷方式的角度出发,对有氧甲烷产生途径进行了综述,让人们对于生物甲烷产生有一个新的定义与思考。未来甲烷产生作用的研究方向不应仅局限于传统的厌氧条件,也可以将研究的重心移向有氧条件下的甲烷生成这一较新的领域。
细菌在有氧条件下代谢甲基膦酸产甲烷的作用已在实验室中得到证实,可作为未来新型产甲烷研究的突破口,具体可以从以下几个方面进行探究:(1)仍需进一步解析有氧条件下甲烷产生的生物化学途径;(2)细菌中同样存在着甲烷产生菌,好氧的生存条件为菌种分离培养减轻了难度,后续可对相关菌种进行进一步的分离、培养与鉴定工作;(3)有机膦酸盐在细菌好氧产甲烷中十分关键,后续应对这类产甲烷底物的来源进行更多研究;(4)对湿地、湖泊、土壤和海洋中的好氧甲烷产生量进行测量计算,并将其纳入全球甲烷产生总量,重新评估甲烷在全球气候变化中所占的比重;(5)将新型产甲烷途径与生物质能源方面相联系,充分发挥好氧产甲烷的优势,促进燃气能源的发展。
| [1] |
Knoblauch C, Beer C, Liebner S, et al. Methane production as key to the greenhouse gas budget of thawing permafrost[J]. Nature Climate Change, 2018, 8(4): 309-312. DOI:10.1038/s41558-018-0095-z |
| [2] |
Gu H, Xiao FS, He ZL, et al. Microbial driven methane emission mechanisms in wetland ecosystems[J]. Acta Microbiologica Sinica, 2018, 58(4): 618-632. (in Chinese) 顾航, 肖凡书, 贺志理, 等. 湿地微生物介导的甲烷排放机制[J]. 微生物学报, 2018, 58(4): 618-632. |
| [3] |
Dlugokencky EJ, Nisbet EG, Fisher R, et al. Global atmospheric methane: budget, changes and dangers[J]. Philosophical Transactions of the Royal Society A: Mathematical, Physical and Engineering Sciences, 2011, 369(1943): 2058-2072. DOI:10.1098/rsta.2010.0341 |
| [4] |
Stams AJM, Plugge CM. Electron transfer in syntrophic communities of anaerobic bacteria and archaea[J]. Nature Reviews Microbiology, 2009, 7(8): 568-577. DOI:10.1038/nrmicro2166 |
| [5] |
Cheng L, Zheng ZZ, Wang C, et al. Recent advances in methanogens[J]. Microbiology China, 2016, 43(5): 1143-1164. (in Chinese) 承磊, 郑珍珍, 王聪, 等. 产甲烷古菌研究进展[J]. 微生物学通报, 2016, 43(5): 1143-1164. |
| [6] |
Mayumi D, Mochimaru H, Tamaki H, et al. Methane production from coal by a single methanogen[J]. Science, 2016, 354(6309): 222-225. DOI:10.1126/science.aaf8821 |
| [7] |
Guo HG, Liu RY, Yu ZS, et al. Pyrosequencing reveals the dominance of methylotrophic methanogenesis in a coal bed methane reservoir associated with Eastern Ordos Basin in China[J]. International Journal of Coal Geology, 2012, 93: 56-61. DOI:10.1016/j.coal.2012.01.014 |
| [8] |
Gao HG, Yu ZS, Thompson IP, et al. A contribution of hydrogenotrophic methanogenesis to the biogenic coal bed methane reserves of Southern Qinshui Basin, China[J]. Applied Microbiology and Biotechnology, 2014, 98(21): 9083-9093. DOI:10.1007/s00253-014-5908-z |
| [9] |
Wei M, Yu ZS, Zhang HX. Molecular characterization of microbial communities in bioaerosols of a coal mine by 454 pyrosequencing and real-time PCR[J]. Journal of Environmental Sciences, 2015, 30: 241-251. DOI:10.1016/j.jes.2014.07.035 |
| [10] |
Wang BB, Yu ZS, Zhang YM, et al. Microbial communities from the Huaibei Coalfield alter the physicochemical properties of coal in methanogenic bioconversion[J]. International Journal of Coal Geology, 2019, 202: 85-94. DOI:10.1016/j.coal.2018.12.004 |
| [11] |
Jarrell KF. Extreme oxygen sensitivity in methanogenic archaebacteria[J]. Bioscience, 1985, 35(5): 298-302. DOI:10.2307/1309929 |
| [12] |
Karl DM, Beversdorf L, Björkman KM, et al. Aerobic production of methane in the sea[J]. Nature Geoscience, 2008, 1(7): 473-478. DOI:10.1038/ngeo234 |
| [13] |
Gucinski H. Microbial production and consumption of greenhouse gases: methane, nitrogen oxides, and halomethanes[J]. Journal of Environmental Quality, 1994, 23(1): 211-212. |
| [14] |
Keppler F, Hamilton JTG, Braß M, et al. Methane emissions from terrestrial plants under aerobic conditions[J]. Nature, 2006, 439(7073): 187-191. DOI:10.1038/nature04420 |
| [15] |
Dyhrman ST, Chappell PD, Haley ST, et al. Phosphonate utilization by the globally important marine diazotroph Trichodesmium[J]. Nature, 2006, 439(7072): 68-71. DOI:10.1038/nature04203 |
| [16] |
Reeburgh WS. Oceanic methane biogeochemistry[J]. Chemical Reviews, 2007, 107(2): 486-513. |
| [17] |
Grossart HP, Frindte K, Dziallas C, et al. Microbial methane production in oxygenated water column of an oligotrophic lake[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(49): 19657-19661. DOI:10.1073/pnas.1110716108 |
| [18] |
Kamat SS, Williams HJ, Dangott LJ, et al. The catalytic mechanism for aerobic formation of methane by bacteria[J]. Nature, 2013, 497(7447): 132-136. DOI:10.1038/nature12061 |
| [19] |
Repeta DJ, Ferrón S, Sosa OA, et al. Marine methane paradox explained by bacterial degradation of dissolved organic matter[J]. Nature Geoscience, 2016, 9(12): 884-887. DOI:10.1038/ngeo2837 |
| [20] |
Angle JC, Morin TH, Solden LM, et al. Methanogenesis in oxygenated soils is a substantial fraction of wetland methane emissions[J]. Nature Communications, 2017, 8(1): 1567. DOI:10.1038/s41467-017-01753-4 |
| [21] |
Teikari JE, Fewer DP, Shrestha R, et al. Strains of the toxic and bloom-forming Nodularia spumigena (cyanobacteria) can degrade methylphosphonate and release methane[J]. The ISME Journal, 2018, 12(6): 1619-1630. DOI:10.1038/s41396-018-0056-6 |
| [22] |
Tang KW, McGinnis DF, Ionescu D, et al. Methane production in oxic lake waters potentially increases aquatic methane flux to air[J]. Environmental Science & Technology Letters, 2016, 3(6): 227-233. |
| [23] |
Tang KW, McGinnis DF, Frindte K, et al. Paradox reconsidered: methane oversaturation in well-oxygenated lake waters[J]. Limnology and Oceanography, 2014, 59(1): 275-284. DOI:10.4319/lo.2014.59.1.0275 |
| [24] |
Bogard MJ, Del Giorgio PA, Boutet L, et al. Oxic water column methanogenesis as a major component of aquatic CH4 fluxes[J]. Nature Communications, 2014, 5(1): 5350. DOI:10.1038/ncomms6350 |
| [25] |
Yao MY, Henny C, Maresca JA. Freshwater bacteria release methane as a by-product of phosphorus acquisition[J]. Applied and Environmental Microbiology, 2016, 82(23): 6994-7003. DOI:10.1128/AEM.02399-16 |
| [26] |
Wang Q, Dore JE, McDermott TR. Methylphosphonate metabolism by Pseudomonas sp. populations contributes to the methane oversaturation paradox in an oxic freshwater lake[J]. Environmental Microbiology, 2017, 19(6): 2366-2378. DOI:10.1111/1462-2920.13747 |
| [27] |
Lenhart K, Bunge M, Ratering S, et al. Evidence for methane production by saprotrophic fungi[J]. Nature Communications, 2012, 3(1): 1046. DOI:10.1038/ncomms2049 |
| [28] |
Angel R, Matthies D, Conrad R. Activation of methanogenesis in arid biological soil crusts despite the presence of oxygen[J]. PLoS One, 2011, 6(5): e20453. DOI:10.1371/journal.pone.0020453 |
| [29] |
Murase J, Sakai Y, Kametani A, et al. Dynamics of methane in mesotrophic Lake Biwa, Japan[J]. Ecological Research, 2005, 20(3): 377-385. DOI:10.1007/s11284-005-0053-x |
| [30] |
Hofmann H, Federwisch L, Peeters F. Wave-induced release of methane: littoral zones as source of methane in lakes[J]. Limnology and Oceanography, 2010, 55(5): 1990-2000. DOI:10.4319/lo.2010.55.5.1990 |
| [31] |
Murase J, Sakai Y, Sugimoto A, et al. Sources of dissolved methane in Lake Biwa[J]. Limnology, 2003, 4(2): 91-99. |
| [32] |
Fernández JE, Peeters F, Hofmann H. On the methane paradox: transport from shallow water zones rather than in situ methanogenesis is the major source of CH4 in the open surface water of lakes[J]. Journal of Geophysical Research Biogeosciences, 2016, 121(10): 2717-2726. DOI:10.1002/2016JG003586 |
| [33] |
Murase J, Sugimoto A. Inhibitory effect of light on methane oxidation in the pelagic water column of a mesotrophic lake (Lake Biwa, Japan)[J]. Limnology and Oceanography, 2005, 50(4): 1339-1343. DOI:10.4319/lo.2005.50.4.1339 |
| [34] |
Metcalf WW, Griffin BM, Cicchillo RM, et al. Synthesis of methylphosphonic acid by marine microbes: a source for methane in the aerobic ocean[J]. Science, 2012, 337(6098): 1104-1107. DOI:10.1126/science.1219875 |
| [35] |
Kamat SS, Williams HJ, Raushel FM. Intermediates in the transformation of phosphonates to phosphate by bacteria[J]. Nature, 2011, 480(7378): 570-573. DOI:10.1038/nature10622 |
| [36] |
Kittredge JS, Roberts E. A carbon-phosphorus bond in nature[J]. Science, 1969, 164(3875): 37-42. DOI:10.1126/science.164.3875.37 |
| [37] |
Metcalf WW, van der Donk WA. Biosynthesis of phosphonic and phosphinic acid natural products[J]. Annual Review of Biochemistry, 2009, 78: 65-94. DOI:10.1146/annurev.biochem.78.091707.100215 |
| [38] |
Seidel HM, Freeman S, Seto H, et al. Phosphonate biosynthesis: isolation of the enzyme responsible for the formation of a carbon–phosphorus bond[J]. Nature, 1988, 335(6189): 457-458. DOI:10.1038/335457a0 |
| [39] |
Horiguchi M, Kandatstu M. Isolation of 2-aminoethane phosphonic acid from rumen protozoa[J]. Nature, 1959, 184(4690): 901-902. |
| [40] |
Yu XM, Doroghazi JR, Janga SC, et al. Diversity and abundance of phosphonate biosynthetic genes in nature[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(51): 20759-20764. DOI:10.1073/pnas.1315107110 |
| [41] |
Ju KS, Gao JT, Doroghazi JR, et al. Discovery of phosphonic acid natural products by mining the genomes of 10, 000 actinomycetes[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(39): 12175-12180. DOI:10.1073/pnas.1500873112 |
| [42] |
Horsman GP, Zechel DL. Phosphonate biochemistry[J]. Chemical Reviews, 2017, 117(8): 5704-5783. DOI:10.1021/acs.chemrev.6b00536 |
| [43] |
Peck SC, Chekan JR, Ulrich EC, et al. A common late-stage intermediate in catalysis by 2-hydroxyethyl-phosphonate dioxygenase and methylphosphonate synthase[J]. Journal of the American Chemical Society, 2015, 137(9): 3217-3220. DOI:10.1021/jacs.5b00282 |
| [44] |
Born DA, Ulrich EC, Ju KS, et al. Structural basis for methylphosphonate biosynthesis[J]. Science, 2017, 358(6368): 1336-1339. DOI:10.1126/science.aao3435 |
| [45] |
Ahlgren NA, Chen YY, Needham DM, et al. Genome and epigenome of a novel marine Thaumarchaeota strain suggest viral infection, phosphorothioation DNA modification and multiple restriction systems[J]. Environmental Microbiology, 2017, 19(6): 2434-2452. DOI:10.1111/1462-2920.13768 |
| [46] |
Nobu MK, Narihiro T, Liu MM, et al. Thermodynamically diverse syntrophic aromatic compound catabolism[J]. Environmental Microbiology, 2017, 19(11): 4576-4586. DOI:10.1111/1462-2920.13922 |
| [47] |
Callieri C, Hernández-Avilés S, Salcher MM, et al. Distribution patterns and environmental correlates of Thaumarchaeota abundance in six deep subalpine lakes[J]. Aquatic Sciences, 2016, 78(2): 215-225. DOI:10.1007/s00027-015-0418-3 |
| [48] |
Quinn JP, Kulakova AN, Cooley NA, et al. New ways to break an old bond: the bacterial carbon-phosphorus hydrolases and their role in biogeochemical phosphorus cycling[J]. Environmental Microbiology, 2007, 9(10): 2392-2400. DOI:10.1111/j.1462-2920.2007.01397.x |
| [49] |
White AK, Metcalf WW. Microbial metabolism of reduced phosphorus compounds[J]. Annual Review of Microbiology, 2007, 61: 379-400. DOI:10.1146/annurev.micro.61.080706.093357 |
| [50] |
Metcalf WW, Wanner BL. Involvement of the Escherichia coli phn (psiD) gene cluster in assimilation of phosphorus in the form of phosphonates, phosphite, Pi esters, and Pi[J]. Journal of Bacteriology, 1991, 173(2): 587-600. DOI:10.1128/JB.173.2.587-600.1991 |
| [51] |
Metcalf WW, Wanner BL. Evidence for a fourteen-gene, phnC to phnP locus for phosphonate metabolism in Escherichia coli[J]. Gene, 1993, 129(1): 27-32. |
| [52] |
Metcalf WW, Wanner BL. Mutational analysis of an Escherichia coli fourteen-gene operon for phosphonate degradation, using TnphoA' elements[J]. Journal of Bacteriology, 1993, 175(11): 3430-3442. DOI:10.1128/JB.175.11.3430-3442.1993 |
| [53] |
Martínez A, Ventouras LA, Wilson ST, et al. Metatranscriptomic and functional metagenomic analysis of methylphosphonate utilization by marine bacteria[J]. Frontiers in Microbiology, 2013, 4: 340. |
| [54] |
Jochimsen B, Lolle S, McSorley FR, et al. Five phosphonate operon gene products as components of a multi-subunit complex of the carbon-phosphorus lyase pathway[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(28): 11393-11398. DOI:10.1073/pnas.1104922108 |
| [55] |
Seweryn P, Van LB, Kjeldgaard M, et al. Structural insights into the bacterial carbon-phosphorus lyase machinery[J]. Nature, 2015, 525(7567): 68-72. DOI:10.1038/nature14683 |
| [56] |
Yang KL, Ren ZJ, Raushel FM, et al. Structures of the carbon-phosphorus lyase complex reveal the binding mode of the NBD-like PhnK[J]. Structure, 2016, 24(1): 37-42. |
| [57] |
Holden MTG, Titball RW, Peacock SJ, et al. Genomic plasticity of the causative agent of melioidosis, Burkholderia pseudomallei[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(39): 14240-14245. DOI:10.1073/pnas.0403302101 |
| [58] |
Chavda KD, Chen L, Fouts DE, et al. Comprehensive genome analysis of carbapenemase-producing Enterobacter spp.: new insights into phylogeny, population structure, and resistance mechanisms[J]. mBio, 2016, 7(6): e02093-16. |
| [59] |
Buell CR, Joardar V, Lindeberg M, et al. The complete genome sequence of the Arabidopsis and tomato pathogen Pseudomonas syringae pv. tomato DC3000[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(18): 10181-10186. DOI:10.1073/pnas.1731982100 |
| [60] |
Parkhill J, Wren BW, Thomson NR, et al. Genome sequence of Yersinia pestis, the causative agent of plague[J]. Nature, 2001, 413(6855): 523-527. DOI:10.1038/35097083 |
| [61] |
Schmeisser C, Liesegang H, Krysciak D, et al. Rhizobium sp. strain NGR234 possesses a remarkable number of secretion systems[J]. Applied and Environmental Microbiology, 2009, 75(12): 4035-4045. DOI:10.1128/AEM.00515-09 |
| [62] |
Kaneko T, Nakamura Y, Sato S, et al. Complete genomic sequence of nitrogen-fixing symbiotic bacterium Bradyrhizobium japonicum USDA110 (supplement)[J]. DNA Research, 2002, 9(6): 225-256. DOI:10.1093/dnares/9.6.225 |
| [63] |
Finan TM, Weidner S, Wong K, et al. The complete sequence of the 1, 683-kb pSymB megaplasmid from the N2-fixing endosymbiont Sinorhizobium meliloti[J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(17): 9889-9894. DOI:10.1073/pnas.161294698 |
| [64] |
Yang F, Yang J, Zhang XB, et al. Genome dynamics and diversity of Shigella species, the etiologic agents of bacillary dysentery[J]. Nucleic Acids Research, 2005, 33(19): 6445-6458. DOI:10.1093/nar/gki954 |
| [65] |
Touchon M, Hoede C, Tenaillon O, et al. Organised genome dynamics in the Escherichia coli species results in highly diverse adaptive paths[J]. PLoS Genetics, 2009, 5(1): e1000344. DOI:10.1371/journal.pgen.1000344 |
| [66] |
Goodner B, Hinkle G, Gattung S, et al. Genome sequence of the plant pathogen and biotechnology agent Agrobacterium tumefaciens C58[J]. Science, 2001, 294(5550): 2323-2328. DOI:10.1126/science.1066803 |
| [67] |
Strnad H, Lapidus A, Paces J, et al. Complete genome sequence of the photosynthetic purple nonsulfur bacterium Rhodobacter capsulatus SB 1003[J]. Journal of Bacteriology, 2010, 192(13): 3545-3546. DOI:10.1128/JB.00366-10 |
| [68] |
Valdes J, Ossandon F, Quatrini R, et al. Draft genome sequence of the extremely acidophilic biomining bacterium Acidithiobacillus thiooxidans ATCC 19377 provides insights into the evolution of the Acidithiobacillus genus[J]. Journal of Bacteriology, 2011, 193(24): 7003-7004. DOI:10.1128/JB.06281-11 |
| [69] |
Reeve W, O'Hara G, Chain P, et al. Complete genome sequence of Rhizobium leguminosarum bv trifolii strain WSM2304, an effective microsymbiont of the South American clover Trifolium polymorphum[J]. Standards in Genomic Sciences, 2010, 2(1): 66-76. DOI:10.4056/sigs.44642 |
| [70] |
Nicholson WL, Leonard MT, Fajardo-Cavazos P, et al. Complete genome sequence of Serratia liquefaciens strain ATCC 27592[J]. Genome Announcements, 2013, 1(4): e00548-13. |
| [71] |
Weigand MR, Changayil S, Kulasekarapandian Y, et al. Complete genome sequences of two Bordetella hinzii strains isolated from humans[J]. Genome Announcements, 2016, 4(1): e01639-15. |
| [72] |
Dyall-Smith ML, Pfeiffer F, Oberwinkler T, et al. Genome of the haloarchaeon Natronomonas moolapensis, a neutrophilic member of a previously haloalkaliphilic genus[J]. Genome Announcements, 2013, 1(2): e00095-13. |
| [73] |
White AE, Karl DM, Björkman KM, et al. Phosphonate metabolism by Trichodesmium IMS101 and the production of greenhouse gases[J]. Limnology and Oceanography, 2010, 55(4): 1755-1767. DOI:10.4319/lo.2010.55.4.1755 |
| [74] |
Gomez-Garcia MR, Davison M, Blain-Hartnung M, et al. Alternative pathways for phosphonate metabolism in thermophilic cyanobacteria from microbial mats[J]. The ISME Journal, 2011, 5(1): 141-149. DOI:10.1038/ismej.2010.96 |
| [75] |
Carini P, White AE, Campbell EO, et al. Methane production by phosphate-starved SAR11 chemoheterotrophic marine bacteria[J]. Nature Communications, 2014, 5(1): 4346. DOI:10.1038/ncomms5346 |
| [76] |
Lenhart K, Klintzsch T, Langer G, et al. Evidence for methane production by the marine algae Emiliania huxleyi[J]. Biogeosciences, 2016, 13(10): 3163-3174. DOI:10.5194/bg-13-3163-2016 |
| [77] |
Althoff F, Benzing K, Comba P, et al. Abiotic methanogenesis from organosulphur compounds under ambient conditions[J]. Nature Communications, 2014, 5(1): 4205. DOI:10.1038/ncomms5205 |
 2020, Vol. 47
2020, Vol. 47