扩展功能
文章信息
- 詹学良, 姚严翔, 芦晓红, 郭嘉义, 黄卫东
- ZHAN Xue-Liang, YAO Yan-Xiang, LU Xiao-Hong, GUO Jia-Yi, HUANG Wei-Dong
- 铜绿假单胞菌PAO1中c-di-GMP代谢相关基因PA0575对表型的影响
- Effect of c-di-GMP metabolism related gene PA0575 on phenotype in Pseudomonas aeruginosa PAO1
- 微生物学通报, 2020, 47(6): 1927-1934
- Microbiology China, 2020, 47(6): 1927-1934
- DOI: 10.13344/j.microbiol.china.190810
-
文章历史
- 收稿日期: 2019-10-10
- 接受日期: 2019-12-30
- 网络首发日期: 2020-04-21
2. 宁夏医科大学科技中心 宁夏 银川 750000
2. Science and Technology Center, Ningxia Medical University, Yinchuan, Ningxia 750000, China
铜绿假单胞菌是一种条件致病菌,研究者对野生型铜绿假单胞菌(Pseudomonas aeruginosa) PAO1已完成全基因组测序。作为一种广泛研究的模式菌,野生型铜绿假单胞菌PAO1菌株常被用于细菌表型及生理生化功能的研究[1-2]。近年来发现的一种广泛存在于细菌中的第二信使分子环鸟苷二磷酸(cyclic-di-guanosine monophosphate,c-di-GMP)[3],对细菌的表型(运动能力和生物膜)、毒力等一系列生理生化过程起到重要作用,从而影响了细菌的致病性和耐药性[4-6]。
胞内c-di-GMP分别由二鸟苷酸环化酶(diguanylate cyclase,DGC)合成,并由磷酸二酯酶(phosphodiesterase,PDE)降解。在细菌中具有DGC活性的蛋白通常含GGDEF结构域,具有PDE活性的蛋白通常含有EAL结构域[7]。野生型铜绿假单胞菌PAO1基因组具有17个GGDEF、5个EAL、16个GGDEF/EAL结构域基因,这些结构域可能参与了c-di-GMP代谢,近年来部分蛋白的生物学功能已经得到了深入研究,如FimX、PleD、WspR、BlrP1、RocR、LapD等[8]。研究者们注意到GGDEF/EAL结构域的N端大部分都包含如Per-Arnt-Sim (PAS)等信号感应结构域以响应环境刺激,从而调控细菌的毒力因子、表型等生理生化过程[9]。然而诸多细菌表型中,运动能力尤为重要,运动能力增加侵袭力的同时,对生物膜的定殖也起到了一定的作用;生物膜的形成使得细菌可以抵御外界不利因素的影响,导致患者在短时间内感染该病原菌并且难以治愈[10]。因此,铜绿假单胞菌的表型在病原菌感染时起到了重要作用,研究其表型对未来治疗和预防铜绿假单胞菌感染具有重要意义。
PAO1基因组中,PA0575基因编码的蛋白由1个跨膜结构域、4个PAS结构域、1个GGDEF结构域及1个EAL结构域构成,目前关于PA0575基因GGDEF/EAL串联结构域的研究表明该结构域的PDE活性依赖于GGDEF结构域,与PA0575基因高度同源的PA14_07500基因的研究表明该基因缺失后会导致泳动能力的下降[11],但是PA0575基因的GGDEF结构域及其信号感应结构域与细菌表型的关系至今鲜有报道。
我们对PAO1中与c-di-GMP代谢相关的基因PA0575进行研究,利用刚果红结合实验及电转PcdrA-gfp质粒间接检测胞内c-di-GMP水平;使用转座子插入突变体菌株进行了运动能力的检测,以此来分析PA0575蛋白结构域对细菌表型(运动能力和生物膜)的影响,并探讨4种抗生素对铜绿假单胞菌转座子突变体菌株运动能力的影响;为了更进一步研究该基因的功能,针对PA0575催化结构域和信号感应结构域设计构建了8种融合蛋白的表达载体,并诱导蛋白的表达,以期为后续PA0575的生物学功能及机制研究奠定基础。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒野生型铜绿假单胞菌菌株PAO1及其转座子插入突变体菌株PA0575-1、PA0575-2、PA0575-3和PcdrA-gfp质粒由美国Manoil实验室惠赠。
1.1.2 主要试剂和仪器及培养基Congo Red、氨苄青霉素(ampicillin,AMP)、硫酸卡那霉素(kanamycin sulfate,KAN)、氯霉素(chloramphenicol,CHL)、盐酸四环素(tetracycline hydrochloride,TET)、G-250均购于Solarbio公司。
PCR仪、电泳仪、凝胶成像仪,Bio-Rad公司;恒温培养箱,上海一恒科技有限公司;恒温气浴摇床,上海博迅实业有限公司。
刚果红培养基:LB固体培养基中添加40 μg/mL Congo red和20 μg/mL G-250。在运动能力培养基中添加氨苄青霉素(50 μg/mL)、卡那霉素(25 μg/mL)、四环素(25 μg/mL)、氯霉素(25 μg/mL)[12]。
1.2 方法 1.2.1 c-di-GMP水平检测将PcdrA-gfp质粒电转到PAO1、PA0575-1、PA0575-2、PA0575-3中并培养至OD600为1.0,100 μL菌液加入到1.9 mL 5% LB液体培养基(使用PBS稀释),37 ℃、200 r/min培养6 h,测量OD600及荧光值(激发波长485 nm,发射波长520 nm);刚果红培养基上滴加菌液37 ℃正置培养24 h,拍照记录。
1.2.2 生物膜定量实验及运动能力的表型分析PAO1、PA0575-1、PA0575-2、PA0575-3菌株于37 ℃、200 r/min条件下培养过夜至OD600为0.5,聚苯乙烯试管中加入1 mL LB液体培养基及10 μL菌液,37 ℃、100 r/min孵育,在12、18、24 h分别取出试管,0.2%结晶紫染色,定量时需加入95%酒精5 mL溶解,测量OD595并统计结果。运动能力检测:培养14 h后拍照并测量菌斑的直径,每个菌斑测量3次取平均值,并进行统计分析。
1.2.3 表达载体的构建与蛋白诱导表达根据PA0575基因结构域设计相关引物:
4PASPAC-R:5′-TAGCAAGCTTGAGGGTGG AGATATCGGGCGAAG-3′;
PASPAC1-F:5′-AGAGGATCCCAGCACTAC CGGGGACTGGTGG-3′;
PASPAC1-Rev:5′-TAGCAAGCTTGGCCTCG GTGATGTCGATCATC-3′;
PBPB-Rev:5′-TAGCAAGCTTCAGGCCGCCG ACCCAGGGGGC-3′;
PBPB-Rev:5′-AGAGGATCCAGCATCGCGG TGGGCATGGACACC-3′;
GGDEF-Rev:5′-TAGCAAGCTTGTAGTACTC GATGCGGCTGCGG-3′。
通过PCR扩增目的片段[13],双酶切后连接于载体pET-32a(+),将测序结果无误的质粒分别热激转化到大肠杆菌BL21中,1 mmol/L IPTG诱导蛋白表达,SDS-PAGE检测。
1.2.4 统计学方法采用SPSS 11.5统计软件分析数据,计量资料以均数±标准差(X±SD)表示,多组间比较用方差分析,组间两两比较采用SNK检验方法,P < 0.05为差异有统计学意义。
2 结果与分析 2.1 PCR转座子突变验证以野生型PAO1和PA0575的3个转座子突变体菌株的DNA为模板,以基因特异性引物和转座子引物Hah munus 138分别组合对它们进行转座子插入突变验证(图 1),由第1、2、3、4泳道证明野生型菌株不含转座子;其余泳道条带显示PA0575-1、PA0575-2、PA0575-3基因组存在转座子插入突变且突变位点不同。测序结果显示PA0575-3、PA0575-2的转座子插入位点位于PAS2、PAS3结构域,PA0575-1的突变位点位于GGDEF结构域。

|
| 图 1 PA0575基因转座子插入突变失活验证琼脂糖凝胶电泳图 Figure 1 Verification of PA0575 gene transposon insertion mutation inactivation by agarose gel electrophoresis 注:M: Trans2K Plus DNA Marker;PCR引物组合为:F1+R1、F2+R2、Hah138+R1、Hah138+R2. 1−4:PAO1为DNA模板;5−8:PA0575-1为DNA模板;9−12:PA0575-2为模板;13−16:PA0575-3为DNA模板. Note: M: Trans2K Plus DNA Marker; The combination of PCR primers are: F1+R1, F2+R2, Hah138+R1, Hah138+R2. 1−4: PAO1 as DNA template; 5−8: PA0575-1 as DNA template; 9−12: PA0575-2 as template; 13−16: PA0575-3 as DNA template. |
|
|
为了研究各结构域在c-di-GMP代谢过程中发挥的作用,对野生型PAO1以及PA0575的3个转座子突变体菌株胞内c-di-GMP水平进行检测。利用刚果红结合实验初步分析胞内c-di-GMP水平,结果如图 2A所示,c-di-GMP促进纤维素的合成,纤维素和刚果红结合后会呈现紫色,当c-di-GMP含量偏低时为粉色或者棕色;而PA0575-1菌苔呈紫色,表明其c-di-GMP含量偏高。随后在突变体菌株中电转PcdrA-gfp质粒(将c-di-gmp响应的cdra启动子融合到编码绿色荧光蛋白基因上,用以评估铜绿假单胞菌中c-di-gmp的胞内水平,该结果以荧光值(激发光波长485 nm,发射光波长520 nm)与OD600比值表示,测量结果与刚果红结合实验结果一致,表明PA0575-1菌株中c-di-GMP含量明显高于野生型PAO1菌株(P < 0.05),如图 2B所示。

|
| 图 2 c-di-GMP含量检测 Figure 2 c-di-GMP content detection 注:A:刚果红结合实验;B:将PcdrA-gfp质粒电转移到铜绿假单胞菌中测得的相对荧光值以反映胞内c-di-GMP的水平.与对照相比:*:P < 0.05;**:P < 0.01. Note: A: Congo red binding experiment; B: Relative fluorescence value measured by electrotransfer of PcdrA-gfp plasmid into Pa. The plasmid can reflect the intracellular level of c-di-GMP. Compared with the control group: *: P < 0.05; **: P < 0.01. |
|
|
鉴于不同结构域对c-di-GMP水平的调控存在差异,为了进一步研究各结构域、c-di-GMP及细菌运动能力的关系,对运动能力进行了检测。野生型PAO1以及PA0575基因的3个转座子突变体菌株泳动能力、蜂群运动能力和蹭动运动能力如图 3所示,测量菌苔直径并统计分析后发现:3个转座子突变体菌株的泳动能力明显加强(P < 0.05),PA0575-2和PA0575-3的蜂群运动能力增加(P < 0.05),但是PA0575-1菌株的蜂群泳动能力与野生型无差别(P > 0.05)。

|
| 图 3 PA0575基因转座子突变体菌株运动能力 Figure 3 Movement ability of PA0575 gene transposon mutant strain 注:平板法检测细菌的运动能力(A)以及细菌泳动能力(B)、蜂群运动(C)、蹭行运动(D)柱状统计图.每组测量细菌直径3次,计算平均值. Note: Detection of motility of bacteria by plate method (A) and the bar statistical chart of swimming motility (B), swarming motility (C) and twitching motility (D) of bacteria after adding antibiotics were detected by plate method. Measure the diameter of bacteria three times per group, calculate the average value. |
|
|
为了更进一步了解PA0575在抗生素存在条件下影响突变体菌株运动能力的作用,在运动培养基中分别添加4种抗生素,氨苄青霉素和卡那霉素对野生型菌株以及突变体菌株的运动能力都有抑制作用;氯霉素对野生型PAO1菌株的运动能力无影响(P > 0.05),但是所有的转座子突变体菌株的全部运动表型都被氯霉素抑制(P < 0.05);四环素对野生型PAO1菌株的运动能力起到了抑制作用(P < 0.05),因为转座子中含有四环素抗性,导致转座子突变体菌株的泳动能力和蹭动能力不受影响(P > 0.05),但是蜂群运动能力受到了抑制作用。
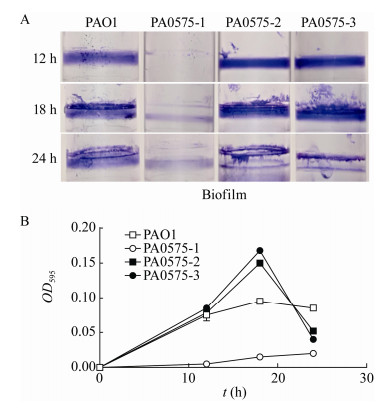
2.4 生物膜检测为了探究PA0575基因对生物膜合成的影响,进行了生物膜定量实验。生物膜经结晶紫染色后会形成紫色环状物,无水乙醇溶解后测量OD595所得实验结果见图 4A,统计分析结果见图 4B。培养18 h后PA0575-2菌株及PA0575-3菌株的生物膜含量比野生型高(P < 0.05),24 h时含量比野生型低(P < 0.05);PA0575-1菌株在12、18、24 h生物膜含量比野生型低(P < 0.05),因此,PA0575蛋白的不同结构域对生物膜的合成也起到了重要作用。
|
| 图 4 生物膜染色结果(A)和定量统计结果(B) Figure 4 Staining results (A) and quantitative results (B) of biofilm |
|
|
为了对PA0575蛋白结构域生化功能进行深入研究,进一步重组表达了PA0575基因的8个结构域蛋白(图 5A)。重组质粒经BamH Ⅰ和Hind Ⅲ双酶切后的琼脂糖凝胶电泳结果如图 5B所示。构建完成的8个表达载体经IPTG诱导后均可得到表达的产物,SDS-PAGE结果如图 5C所示。

|
| 图 5 PA0575基因结构域示意图(A)、双酶切琼脂糖凝胶电泳(B)和SDS-PAGE结果(C) Figure 5 PA0575 gene domain diagram (A), double enzyme digestion result (B) and SDS-PAGE result (C) 注:图A、B、C中编号1−8分别为PBPB、PBPB-PAS1、PBPB-GGDEF、PBPB-EAL、PAS1、PAS1-GGDEF、PAS-EAL、GGDEF结构域基因;图C中9泳道为对照组. M:Marker. Note: 1−8 in figure A, B and C are PBPB, PBPB-PAS1, PBPB-GGDEF, PBPB-EAL, PAS1, PAS1-GGDEF, PAS-EAL and GGDEF domain genes, and lane 9 in figure 5C is the control group. M: Marker. |
|
|
铜绿假单胞菌是引起医院获得性感染的主要病原菌之一[13]。c-di-GMP作为一种广泛存在于细菌中的第二信使,对细菌的运动、生物膜等有重要的影响[14]。细菌的泳动能力依赖于鞭毛的泳动、滑行或旋转梭动[15],蹭行运动依赖于鞭毛的运动和Ⅳ型菌毛的伸展和收缩,蜂群泳动则是一类依赖于Ⅳ型菌毛的群集运动[16]。铜绿假单胞菌在感染时会利用各种形式的运动以便达到感染部位,其中细菌的菌毛可粘附上皮细胞,从而对铜绿假单胞菌慢性感染过程中生物膜的形成起到促进作用,生物膜的形成使细菌可以抵御宿主免疫防御系统及抗生素的影响,同时鞭毛促进了细菌的迁移和扩散,进而促进病原菌与宿主的相互作用[17]。越来越多的患者因耐药铜绿假单胞菌的感染而死亡,因此,针对铜绿假单菌运动表型的相关研究十分急迫。
野生型铜绿假单胞菌PAO1中PA0575蛋白显示PDE催化活性,其具有调控c-di-GMP代谢的GGDEF-EAL结构域[11],以及与胞外信号传递相关的PAS结构域。
本研究利用PCR技术和基因测序对3株PA0575转座子插入突变体菌株的遗传背景进行了验证,突变体PA0575-1菌株的转座子插入位点位于GGDEF结构域,PA0575-2、PA0575-3菌株转座子插入位点分别位于PAS3、PAS2结构域;研究发现PA0575-1菌株的c-di-GMP水平高于PAO1,而PA0575-2、PA0575-3菌株的c-di-GMP水平改变不显著,据此推断PA0575基因可能参与调控胞内c-di-GMP的水平,且GGDEF结构域的插入突变可能与c-di-GMP含量升高有关,此项观察与PA0575蛋白具有PDE活性,具有GGDEF影响PDE活性的结果[11]一致。为了比较转座子插入突变后各菌株表型的变化,我们进行了表型分析:PA0575基因GGDEF结构域的突变引起泳动能力增强,生物膜合成能力降低;然而两个不同的PAS结构域突变后可引起泳动、蜂群运动能力增高的同时引起生物膜含量增高,这表明生物膜的含量不仅与c-di-GMP的水平可能相关,还可能受到PAS结构域的影响;当PAS结构域突变时c-di-GMP水平无变化但运动能力明显增强,表明泳动能力、蜂群运动能力可能受PAS结构域的影响。为了进一步研究各结构域在响应胞外抗生素信号对运动能力抑制时发挥的作用,在运动培养基中添加抗生素后发现:氨苄青霉素和卡那霉素对野生型菌株和突变体菌株的运动能力都有抑制作用;转座子中含有四环素抗性基因,由此转座子突变体菌株的泳动能力和蹭动能力变化不显著,但是蜂群运动能力受到了抑制作用,该现象可能与蜂群运动能力易受外界环境因素的影响有关,在卡那霉素实验组发现细菌的泳动能力和蹭动能力被抑制显著,而蜂群运动能力完全丧失,抑制作用十分显著;同时我们还发现PA0575基因PAS或GGDEF结构域的突变使得氯霉素对细菌3种运动能力都产生抑制作用,表明PAS及GGDEF结构域可能参与了氯霉素对细菌运动能力的抑制途径。
综上所述,我们初步认为:GGDEF结构域参与了c-di-GMP水平的调控;细菌的表型不完全由c-di-GMP直接调控,同时受PAS结构域的影响;PA0575基因的PAS、GGDEF结构域与氯霉素对细菌运动能力抑制过程相关。为了进一步阐明不同结构域、功能、表型的关联,我们构建了PA0575各结构域表达载体,为进一步研究该蛋白的生理生化功能奠定了基础。PA0575基因编码一个多结构域复杂蛋白,对该基因的研究可能对未来抗菌药物的研发、控制细菌感染和扩散起到重要的影响,但是PA0575基因不同结构域的生化功能仍有待进一步探讨。
| [1] |
Chevalier S, Bouffartigues E, Bodilis J, et al. Structure, function and regulation of Pseudomonas aeruginosa porins[J]. FEMS Microbiology Reviews, 2017, 41(5): 698-722. DOI:10.1093/femsre/fux020 |
| [2] |
Doberenz S, Eckweiler D, Reichert O, et al. Identification of a Pseudomonas aeruginosa PAO1 DNA Methyltransferase, Its Targets, and Physiological Roles[J]. mBio, 2017, 8(1): e02312-16. |
| [3] |
Navarro MVAS, de N, Bae N, et al. Structural Analysis of the GGDEF-EAL Domain-Containing c-di-GMP Receptor FimX[J]. Structure, 2009, 17(8): 1104-1116. DOI:10.1016/j.str.2009.06.010 |
| [4] |
Moreira RN, Dressaire C, Barahona S, et al. BolA is required for the accurate regulation of c-di-GMP, a central player in biofilm formation[J]. mBio, 2017, 8(5): e00443-17. |
| [5] |
Yang Y, Li Y, Gao TT, et al. C-di-GMP turnover influences motility and biofilm formation in Bacillus amyloliquefaciens PG12[J]. Research in Microbiology, 2018, 169(4/5): 205-213. |
| [6] |
Ryu MH, Fomicheva A, Moskvin OV, et al. Optogenetic module for dichromatic control of c-di-GMP signaling[J]. Journal of Bacteriology, 2017, 199(18): e00014-17. |
| [7] |
Sarenko O, Klauck G, Wilke FM, et al. More than enzymes that make or break cyclic Di-GMP-local signaling in the interactome of GGDEF/EAL domain proteins of Escherichia coli[J]. mBio, 2017, 8(5): e01639-17. |
| [8] |
Romling U, Galperin MY, Gomelsky M. Cyclic di-GMP: the first 25 years of a universal bacterial second messenger[J]. Microbiology and Molecular Biology Reviews, 2013, 77(1): 1-52. |
| [9] |
Borselli D, Lieutaud A, Thefenne H, et al. Polyamino-isoprenic derivatives block intrinsic resistance of p. aeruginosa to doxycycline and chloramphenicol in vitro[J]. PLoS One, 2016, 11(5): e154490. |
| [10] |
Subedi D, Vijay AK, Willcox M. Overview of mechanisms of antibiotic resistance in Pseudomonas aeruginosa: an ocular perspective[J]. Clin Exp Optom, 2018, 101(2): 162-171. DOI:10.1111/cxo.12621 |
| [11] |
Mantoni F, Paiardini A, Brunotti P, et al. Insights into the GTP-dependent allosteric control of c-di-GMP hydrolysis from the crystal structure of PA0575 protein from Pseudomonas aeruginosa[J]. The FEBS Journal, 2018, 285(20): 3815-3834. DOI:10.1111/febs.14634 |
| [12] |
Opoku-Temeng C, Sintim HO. Targeting c-di-GMP Signaling, Biofilm Formation, and Bacterial Motility with Small Molecules[A]//Sauer K. c-di-GMP Signaling[M]. New York, NY: Humana Press, 2017, 1657: 419-430
|
| [13] |
Chen XH, Zhang ZR, Liu F, et al. Key catalytic sites in the reverse transcription domain of Ll. LtrB intron encoded protein[J]. Acta Microbiologica Sinica, 2019, 59(12): 2357-2366. (in Chinese) 陈相好, 张峥嵘, 刘芳, 等. Ll. LtrB内含子编码蛋白反转录结构域关键催化位点分析及功能验证[J]. 微生物学报, 2019, 59(12): 2357-2366. |
| [14] |
Stover CK, Pham XQ, Erwin A L, et al. Complete genome sequence of Pseudomonas aeruginosa PAO1, an opportunistic pathogen[J]. Nature, 2000, 406(6799): 959-964. DOI:10.1038/35023079 |
| [15] |
Robert-Paganin J, Nonin-Lecomte S, Réty S. Crystal structure of an EAL domain in complex with reaction product 5′-pGpG[J]. PLoS One, 2012, 7(12): e52424. DOI:10.1371/journal.pone.0052424 |
| [16] |
Buensuceso R, Daniel-Ivad M, Kilmury S, et al. Cyclic AMP-Independent Control of Twitching Motility in Pseudomonas aeruginosa[J]. Journal of Bacteriology, 2017, 199(16): e00188-17. |
| [17] |
Ha DG, O'Toole GA. c-di-GMP and its effects on biofilm formation and dispersion: a Pseudomonas aeruginosa review[J]. Microbiol Spectr, 2015, 3(2): MB-0003-2014. |
| [18] |
Zingg W, Hopkins S, Gayet-Ageron A, et al. Health-care-associated infections in neonates, children, and adolescents: an analysis of paediatric data from the European centre for disease prevention and control point-prevalence survey[J]. Lancet Infect Dis, 2017, 17(4): 381-389. |
 2020, Vol. 47
2020, Vol. 47