扩展功能
文章信息
- 佟蕾, 张睿彬, 彭琦, 张杰, Lereclus Didier, 宋福平
- TONG Lei, ZHANG Rui-Bin, PENG Qi, ZHANG Jie, Lereclus Didier, SONG Fu-Ping
- 苏云金芽胞杆菌LM1212菌株和突变株的全基因组序列比较分析
- Comparative analysis of two complete genome sequences from Bacillus thuringiensis strain LM1212 and its mutant
- 微生物学通报, 2020, 47(6): 1888-1900
- Microbiology China, 2020, 47(6): 1888-1900
- DOI: 10.13344/j.microbiol.china.200327
-
文章历史
- 收稿日期: 2020-03-31
- 接受日期: 2020-05-06
- 网络首发日期: 2020-05-19
2. INRA, UMR1319 Micalis, Jouy-en-Josas 78350, France
苏云金芽胞杆菌(Bacillus thuringiensis,Bt)是一种具有多种生态位的革兰氏阳性细菌,其最显著的特征是在产芽胞的过程中会产生由杀虫晶体蛋白(Cry蛋白或Cyt蛋白)组成的伴胞晶体[1]。由于杀虫晶体蛋白具有特异性杀虫活性,目前Bt已成为应用最成功的微生物杀虫剂[2]。苏云金芽胞杆菌染色体为环状DNA,基因组大小为5.0 Mb左右,(G+C)mol%含量一般为32%-35%。Bt菌株包含至少一个质粒DNA,大多数的杀虫晶体蛋白基因定位于质粒DNA上。迄今为止,在NCBI网站上已公布了594株Bt菌株的全基因组序列,这使得高通量测序结合生物信息学分析的方法在Bt基因组数据中深度挖掘各类型基因变得切实可行,如杀虫晶体蛋白基因、营养期杀虫蛋白基因[3]、分泌性杀虫蛋白基因[4]、对癌细胞有特异性活性的基因、苏云金素合成基因簇、双效菌素合成基因簇等。因此,Bt菌株的应用范围也被不断拓宽[2]。
苏云金芽胞杆菌的普遍存在性和高度适应能力可能是由于其具有响应复杂环境变化的生物功能元件。例如,双组分信号系统可通过磷酸基团的级联反应对环境的波动,比如营养缺乏、氧限制及冷/热刺激等,做出快速而有力的反应[5-6],并调控细菌的多种生理行为,包括运动性、趋化性、芽胞形成过程、生物膜形成以及毒素合成等[7-9]。再如,插入序列单元具有高移动性和插入能力,可通过剪切和插入导致基因的突变或移动,从而影响基因的表达。插入序列元件多与信号转导基因或转运蛋白基因相邻,这表明插入序列的转座倾向于影响与环境适应性相关的功能基因的表达[10]。
典型的Bt菌株的芽胞和晶体产生于同一细胞内,其杀虫晶体基因的表达由芽胞依赖型启动子介导[11]。与此形成鲜明对比的是,LM1212菌株的杀虫晶体基因仅在非芽胞细胞中表达且不依赖于芽胞的形成;此外,LM1212菌株具有独特的细胞分化表型,其芽胞和晶体分别产生于芽胞形成细胞和晶体产生细胞中,其芽胞形成细胞和晶体产生细胞的比例约为6:4[12]。前期研究鉴定到LM1212菌株中存在一种新型的转录因子CpcR,目前发现其具有增加晶体细胞比例的功能[13],这表明CpcR是LM1212菌株细胞分化机制的影响因素之一。为了进一步阐明LM1212菌株细胞分化的机制,马丽霞等[14]通过高温处理的方法进行质粒消除实验,得到了一株表型稳定的突变菌株LM1212-DB,该菌株具有更少的芽胞形成细胞和更多的晶体产生细胞。这种能使晶体产生细胞比例显著增加的突变株既可作为研究细胞分化机制的实验材料,又可作为提高Bt菌株杀虫晶体蛋白产量的基础菌株。实验室前期研究表明,与LM1212相比,LM1212-DB突变株的质粒大小差异显著且鉴定到位于质粒上的几个毒素基因已缺失,这意味着二者的基因组结构出现变异,为进一步解析二者之间的差异,对LM1212与LM1212-DB菌株进行了全基因组测序[14]。本研究比较分析了芽胞形成细胞和晶体产生细胞分化比例具有明显差异的LM1212和LM1212-DB菌株的全基因组序列,重点分析了两个菌株的全基因组、双组分信号系统、插入序列等方面的差异,探求导致两个菌株表型产生差异的原因。本研究结果以期为研究LM1212菌株细胞分化机制提供理论依据,进而为Bt制剂的定向遗传改良奠定基础。
1 材料与方法 1.1 供试菌株LM1212菌株和突变株LM1212-DB均为实验室保存。
1.2 主要试剂和仪器蛋白胨、氯化钠、酵母提取物,Sigma公司;RNase A,北京全式金生物技术有限公司。低温离心机,Eppendorf公司;pH计,Thermo Scientific公司。
1.3 培养基LB液体培养基(g/L):NaCl 10.0,Tryptone 10.0,Yeast extract 5.0,pH 7.0。
1.4 基因组制备与测序将LM1212菌株和突变株LM1212-DB以30 ℃、200 r/min的培养条件培养过夜后,按照Stobdan等[15]描述的方法提取并检测基因组DNA,将其送至北京赛墨百合生物科技有限公司进行测序。利用SMRT技术和Pacbio RS Ⅱ测序平台进行测序[16-17]。
1.5 测序数据的组装及注释由于单分子测序技术具有高读长、数据信号捕捉困难且随机性强等特征,因此,在得到原始数据后需要进行严格的数据质量过滤和前处理。利用层次基因组装软件包HGAP对过滤后的高质量Clean reads进行组装。利用Prodigal[18]、Infernal[19]和RNAmmer[20]等软件包对编码序列(coding sequence,CDS)、tRNA和rRNA进行预测。使用Rfam[21]、Swissprot[22]、COG[23]和KEGG[24]数据库对全基因组序列进行基因的功能预测与注释。
1.6 插入序列鉴定及比较基因组分析使用在线网站ISFINDER[25] (https://www-is.biotoul.fr/)对LM1212和LM1212-DB全基因组序列进行插入序列的鉴定与分析。利用MUMmer 3.0[26]软件进行比较基因组分析。
1.7 CpcR同源序列系统发育树构建使用在线网站NCBI-BLASTp (https://blast.ncbi.nlm.nih.gov/Blast)进行CpcR同源序列比对。利用MEGA 7.0软件构建系统发育树。
2 结果与分析 2.1 LM1212菌株和突变株LM1212-DB的全基因组概况LM1212菌株和突变株LM1212-DB的全基因组序列已上传至GenBank,登录号分别为CP024771-CP024779[13]和CP051013-CP051018。根据基因组注释结果显示,LM1212菌株和LM1212-DB菌株的质粒数量、质粒拷贝数以及包含的杀虫基因种类均存在差异。LM1212菌株包含1个环状的染色体、8个环状的质粒和14种杀虫基因,LM1212-DB菌株包含1个环状的染色体、5个环状的质粒和10种杀虫基因(表 1)。
| 菌株 Strains |
染色体/质粒名称 Chromosome/Plasmid name |
碱基数 Sequence size (bp) |
拷贝数 Copy number |
杀虫基因类别 Insecticidal genes types |
| LM1212 | Chromosome | 5 705 934 | 1 | - |
| pLM248 | 248 002 | 3 | cry32Wa1, cry32Va1, cry35-like, cry35-like2, cry41Ca1, cry69Aa-like, cryBP1, cytAa-like, cytCa-like | |
| pLM158 | 157 839 | 3 | cry45ba1, cry74Aa1, nt32-like | |
| pLM113 | 112 709 | 2 | cry32-like, mtx2-like | |
| pLM35 | 35 121 | 3 | - | |
| pLM28 | 28 658 | 2 | - | |
| pLM14 | 14 193 | 10 | - | |
| pLM13 | 13 118 | 6 | - | |
| pLM5 | 4 975 | 8 | - | |
| LM1212-DB | Chromosome | 5 629 931 | 1 | - |
| pLM241 | 241 137 | 4 | cry41Ca1, cry45ba1, cry74Aa1, cry69Aa-like, cryBP1, cytAa-like, cytCa-like, nt32-like | |
| pLM113 | 112 715 | 2 | cry32-like, mtx2-like | |
| pLM35 | 35 175 | 4 | - | |
| pLM14 | 14 375 | 3 | - | |
| pLM14-1 | 14 195 | 2 | - | |
| 注:-:无杀虫基因 Note: -: No insecticidal gene |
||||
对LM1212菌株和突变株LM1212-DB的染色体基因组进行比较分析发现,位于LM1212菌株染色体基因组的2 607 455 bp与2 682 965 bp之间的片段在LM1212-DB菌株中缺失。染色体基因组的缺失片段共含有69个开放阅读框(open reading frame,ORF):4个ORF被注释为转座酶,值得注意的是,位于缺失片段的首末均为同一个转座酶,这可能是导致该片段缺失的原因;38个ORFs被注释为未知功能的假想蛋白基因;31个ORFs被注释为各类别的功能基因,可将这些缺失的功能基因分为2类:一类基因是在基因组未缺失区域仍存在与此基因功能相似或相同的基因;另一类基因是在L-M1212DB菌株中仅存在唯一的拷贝且丢失(表 2)。
| 名称 Name |
蛋白长度 Protein length (aa) |
功能 Function |
COG分类 COG category |
| LM_02887 | 325 | 鸟苷酸环脱氢酶 Ornithine cyclodeaminase |
E |
| LM_02876 | 471 | 藻酸盐乙酰转移酶 Alginate acetyltransferase |
M |
| LM_02926 | 506 | Ⅰ型限制-修饰系统酶M Type Ⅰ restriction-modification system M subunit |
V |
| LM_02927 | 424 | Ⅰ型限制-修饰系统酶S Type Ⅰ restriction-modification system S subunit |
V |
| LM_02928 | 1 031 | Ⅰ型限制-修饰系统酶R Type Ⅰ restriction-modification system R subunit |
V |
| LM_02909 | 117 | XRE家族转录因子 XRE family transcription factors |
K |
| LM_02916 | 225 | 双组分信号系统转录因子 Two-component signaling system transcription factors |
T |
| LM_02906 | 197 | Sigma B | K |
针对第二类在LM1212-DB菌株中完全缺失的基因进行功能分析,未发现与细胞分化过程相关的基因,但发现缺失基因的功能多注释为环境适应性和防御机制相关的基因。按照基因组注释的功能,可将完全缺失的基因分为4类:(1)参与响应外界环境胁迫的转录因子,如SigmaB (LM_02906)、XRE家族转录因子(LM_02909)和未知功能的双组分信号系统的转录因子(LM_02916);(2)与脯氨酸合成途径密切相关的关键酶鸟苷酸环脱氢酶(LM_02887)[27];(3)与细胞壁合成相关的藻酸盐乙酰转移酶(LM_02876)[28];(4)参与细菌防御机制的Ⅰ型限制-修饰系统酶M、S和R (LM_02926、LM_02927和LM_02928)[29]。
综上所述,突变株LM1212-DB的染色体基因组上共缺失了38个未知功能的假想蛋白,其中可能存在着与细胞分化过程相关的基因,但具体的生物学功能有待进一步验证。此外,突变株缺失了一些与响应环境胁迫相关的基因,如可使芽胞杆菌更好地应对环境压力、能量损耗或低温的转录因子Sigma B[30],以及可以通过参与合成脯氨酸来影响细菌渗透调节的鸟苷酸环脱氢酶,由此可以看出,突变株对极端环境的适应能力可能变弱。突变株还缺失了1个可以对藻酸盐进行修饰的藻酸盐乙酰转移酶基因,但在突变株中并未发现藻酸盐合成的基因簇。Ⅰ型限制-修饰系统的缺失则会导致突变株对外源DNA具有更好的兼容性,因此该突变株更适用于进行分子遗传操作。
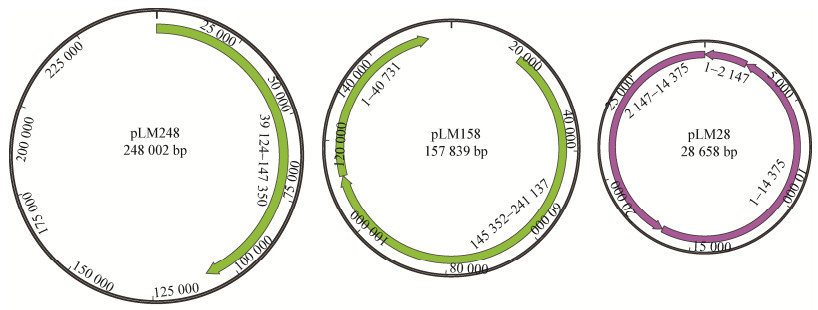
2.3 LM1212菌株和突变株LM1212-DB的质粒差异分析利用MUMmer 3.0软件对LM1212-DB菌株的5个质粒与LM1212菌株的8个质粒进行序列比对发现,LM1212-DB菌株的质粒发生重排以及部分质粒或片段发生缺失:(1)质粒重排及片段缺失:位于LM1212菌株pLM248质粒上的108 222 bp片段与位于pLM158质粒上的两段序列长度分别为40 681 bp和95 786 bp的片段发生重排,在LM1212- DB菌株中形成了一个新的质粒pLM241 (图 1,标注绿色的片段),同时LM1212菌株pLM248和pLM158质粒上未参与重排的片段在LM1212-DB菌株中缺失(图 1,未标注颜色的片段);(2)质粒缺失:LM1212菌株中的pLM13和pLM5在LM1212-DB菌株中缺失,LM1212菌株中的pLM113、pLM35和pLM14在LM1212-DB菌株中未发生重排和缺失,LM1212菌株中的pLM14质粒在LM1212-DB菌株中命名为pLM14-1。
|
| 图 1 质粒重排示意图 Figure 1 Diagram of plasmid rearrangement 注:最内侧的数字表示在LM1212-DB菌株中基因的位置. Note: The innermost number indicates the gene location in LM1212-DB strain. |
|
|
除了发生质粒重排、质粒片段缺失和质粒缺失的情况,还发生了质粒拷贝数的改变:LM1212菌株中的pLM248质粒和pLM158质粒均为3个拷贝,而在LM1212-DB菌株中的pLM241质粒为4个拷贝(表 1),这相当于位于pLM248质粒和pLM158质粒上的未缺失基因在LM1212-DB菌株中均增加了1个拷贝;LM1212菌株中的pLM35质粒为3个拷贝,而在LM1212-DB菌株中pLM35质粒增加了1个拷贝,为4个拷贝(表 1);LM1212-DB菌株中的pLM14质粒源于LM1212菌株中pLM28质粒的一半(图 1,标注粉色的片段),在LM1212-DB菌株中的pLM14质粒为3个拷贝(表 1),比LM1212菌株中pLM28质粒增加了1个拷贝;LM1212菌株pLM14质粒为10个拷贝,而在LM1212-DB菌株中的pLM14-1质粒为2个拷贝,相当于减少了8个拷贝。
2.4 LM1212-DB菌株质粒缺失片段的生物学功能分析在LM1212-DB菌株中缺失的质粒片段共包含184个ORF,包括位于pLM248质粒上的orf118-orf269、位于pLM158质粒上的orf1-orf15和orf172-orf177、位于pLM13质粒上的orf1-9、位于pLM5质粒上的orf1-5。通过COG功能富集分析发现,78个基因编码的蛋白与复制、重组和修复的功能有关,其中大多数基因被注释为各类型的转座酶或质粒重组酶;8个基因编码的蛋白与细胞壁或细胞膜的形成有关,其中包括3个细胞壁相关蛋白、2个Rhs家族蛋白、1个可切割肽聚糖侧链的N-乙酰胞壁酰-L-丙氨酸酰胺酶、1个糖基转移酶和1个与离子转运系统相关的蛋白;6个基因编码的蛋白与信号转导机制有关;4个基因编码的蛋白与防御机制有关;3个基因编码了与芽胞相关的蛋白,其中包括1个芽胞衣相关蛋白和2个芽胞萌发蛋白(图 2);在LM1212-DB菌株的缺失片段中包含了5个杀虫晶体蛋白,分别为Cry35-like (pLM248_00251)、Cry35- like2 (pLM248_00249)、Cry69Aa-like (pLM248_00196)、Cry32Wa1 (pLM248_00197)和Cry32Va1 (pLM248_00227)。

|
| 图 2 缺失基因的COG功能注释 Figure 2 COG functional annotation of the absent genes |
|
|
综上所述,与基因组的缺失片段相比,质粒的缺失片段含有更多的未知功能基因,对于这一部分基因功能的研究势必推动LM1212菌株细胞分化机制的研究。LM1212菌株的质粒上存在着丰富的转座酶和质粒重组酶,这表明LM1212菌株的质粒极易发生重排。这是LM1212菌株的一个重要特性,因此在缺失片段中也包含着大量的转座酶和质粒重组酶。此外,缺失片段中包含2个芽胞衣相关蛋白和1个芽胞萌发蛋白,芽胞衣相关蛋白在芽胞形成过程的晚期发挥作用,芽胞萌发蛋白在芽胞萌发初期发挥作用,因此二者并不会影响芽胞形成过程。值得注意的是,缺失片段中还包含了一个与芽胞形成相关的转录因子AbrB (pLM248_ 00154)。在枯草芽胞杆菌中AbrB可通过抑制sigH表达间接调控Spo0A的磷酸化水平,从而影响芽胞形成过程[31]。因此,转录因子AbrB的缺失可能是导致两个菌株产生表型差异的重要原因。
2.5 LM1212-DB菌株中拷贝数增加的转录因子CpcR的分布情况及系统发育树在LM1212菌株中,cpcR在pLM248质粒上为3个拷贝。在LM1212-DB菌株中,cpcR在pLM241质粒上为4个拷贝。拷贝数增加意味着CpcR的表达量增加,从而导致LM1212-DB菌株增加晶体细胞比例的效果更为明显,这暗示CpcR很可能是导致LM1212-DB菌株和LM1212菌株表型差异的重要影响因素之一。为了进一步探究cpcR是否是导致这种表型差异的特异性基因,以CpcR的氨基酸序列为参考序列,在NCBI数据库中进行序列比对,以氨基酸序列相似性大于50%为标准,共检索到88个同源基因,这些同源基因分属于10个物种,且均被注释为细菌双组分信号系统中OmpR家族转录因子,均含有信号接收结构域和DNA结合结构域。进一步分析发现,这10个物种大多属于厚壁菌门,其中多为蜡样芽胞杆菌族的细菌,如苏云金芽胞杆菌、蜡样芽胞杆菌(Bacillus cereus)、细胞毒素芽胞杆菌(Bacillus cytotoxicus)等;此外,类芽胞杆菌属的Paenibacillus contaminans、葡萄球菌属的Thermoflavimicrobium dichotomicum以及梭菌属的Clostridium cylindrosporum等细菌中也鉴定到cpcR的同源基因。但是,虽然cpcR的同源基因在苏云金芽胞杆菌的近缘物种(一些产胞细菌)中均有分布,但这并不意味着cpcR在产胞细菌中保守,相反,cpcR基因的分布与物种的相关性很差。这一结论可通过包含cpcR同源基因的菌株占该物种中已完成基因组测序的比例即可看出:10个物种中包含cpcR同源基因的菌株仅占88株,而这些菌株所在物种中完成基因组测序的菌株高达2 662株(表 3),这说明cpcR不是芽胞杆菌属菌株的核心基因,其分布很可能与环境或生态位特异性有关。
| 菌株名称 Strain name |
NCBI已测序菌株数目 Number of sequencing strains in NCBI |
包含CpcR同源序列菌株数目 Number of strains containing CpcR homologous genes |
| Bacillus thuringiensis | 594 | 11 |
| Bacillus cereus | 1 113 | 62 |
| Bacillus toyonensis | 216 | 1 |
| Bacillus cytotoxicus | 14 | 1 |
| Bacillus wiedmannii | 158 | 3 |
| Bacillus manliponensis | 1 | 1 |
| Bacillus sp. | 563 | 6 |
| Thermoflavimicrobium dichotomicum | 1 | 1 |
| Paenibacillus contaminans | 1 | 1 |
| Clostridium cylindrosporum | 1 | 1 |
| 总计Total | 2 662 | 88 |
CpcR的保守性与物种相关性差也在其系统发育树中体现出来。88个菌株中CpcR的同源序列存在物种内多个菌株序列100%一致的情况,因此在保证序列的物种信息不损失的前提下,选取可代表全部同源序列特征的21个序列进行系统发育树构建,建树结果显示,根据序列的亲缘关系,CpcR的同源序列被聚类成6个分支,每个分支中的序列不具备物种特异性,例如苏云金芽胞杆菌中的一些亚种在红色、黑色、蓝色分支中均有出现。此外,在苏云金芽胞杆菌yunnanensis亚种Bacillus thuringiensis BGSC4AM1中鉴定到与CpcR氨基酸序列相似性高达85%的序列WP_086410509.1,且有文献记载该菌株具有与LM1212菌株相似的表型[32-33],这也侧面印证了cpcR可能是引起两个菌株表型差异的特异性基因(图 3)。

|
| 图 3 基于cpcR同源序列的系统发育树 Figure 3 Phylogenetic tree of cpcR homologous sequences 注:括号里的序号为菌株GenBank登录号;分支上的数字为Bootstrap值;比例尺长度表示序列的差异度. Note: The numbers in parentheses is the strain accession number in GenBank; The robustness of branching is indicated by bootstrap percentages calculated for 1 000 subsets; Bar indicates 5% estimated sequence divergence. |
|
|
典型的双组分信号系统包含一个具有保守激酶核心区域的组氨酸激酶(histidine kinase,HK)和一个具有保守调控区域的应答调控蛋白(response regulator,RR)。以HK-RR基因对为筛选标准对全基因组数据进行挖掘和筛选,结果发现:LM1212菌株共编码了36个HK-RR基因对,其中包含19个可确定功能的HK-RR基因对,其具体的功能详见表 4;其余的HK-RR基因对仍需要进一步的实验确定其生物学功能。与LM1212菌株不同的是,突变株LM1212-DB中缺失了1个RR基因(表 2),且该RR基因为未知功能的孤儿基因,并未找到与其配对的HK基因。
| 基因位置 Gene locus |
蛋白名称 Protein name |
功能 Function |
| LM_00819/LM_00820 | PhoR/PhoP | 磷酸盐代谢Phosphate metabolism |
| LM_03600/LM_03599 | ResE/ResD | 呼吸作用Respiration |
| LM_01788/LM_01789 | VicK/VicR | 维持细胞壁稳态Maintain cell wall homeostasis |
| LM_00673/LM_00674 LM_01114/LM_01113 LM_04865/LM_04866 LM_06077/LM_06078 |
GraS/GraR | 响应外界压力Response to external stress 细胞壁代谢Cell wall metabolism |
| LM_01380/LM_01379 | VanS/VanR | 万古霉素抗性Vancomycin resistance |
| LM_02513/LM_02514 LM_02534/LM_02535 |
CitS/CitT | 柠檬酸转运Citric acid transport |
| LM_01764/LM_01763 | LytS/LytT | 丙酮酸代谢Pyruvate metabolism |
| LM_04434/LM_04433 | AgrC/AgrA | 毒力基因表达Virulence gene expression |
| LM_01458/LM_01459 | ComP/ComA | 细胞感受态Cellular competence |
| LM_01675/LM_01674 | DesK/DesR | 细胞膜流动性Cell membrane fluidization |
| LM_03561/LM_03562 | LiaS/LiaR | 感应抗生素压力Response to antibiotic stress |
| LM_04277/LM_04276 | SalK/SalR | 分泌系统Secretion system |
| LM_03762/LM_03761 | CheA/ CheY | 细菌趋化性系统Bacterial chemotactic system |
| LM_03883/LM_03884 LM_04326/LM_04327 |
YcbA/YcbB | 谷氨酰胺的利用Utilization of glutamine |
插入序列单元是普遍存在于细菌染色体和质粒中并且具有插入能力的小分子DNA片段。在确保完整性的前提下,LM1212菌株染色体和质粒上分布着属于9个家族的56个完整的插入序列单元,其中24个位于染色体上,17个位于pLM248质粒上,7个位于pLM158质粒上,7个位于pLM113质粒上(表 5)。YBT-1520菌株在染色体上仅含有4个完整的ISBth13拷贝,而LM1212菌株的染色体上含有19个完整的ISBth13拷贝。IS240A发现于苏云金芽胞杆菌以色列亚种[34],LM1212菌株pLM248质粒上含有13个完整的IS240A拷贝。LM1212菌株蕴含丰富的插入序列单元可能是导致原核微生物基因组重排的重要因素。
| 插入序列 Insertion squence |
家族 Family |
插入序列长度 Insertion squence length (bp) |
染色体 Chromosome |
Plasmid pLM248 |
Plasmid pLM158 |
Plasmid pLM113 |
| ISBth166 | IS110 | 1 729 | 3 | 4 | 0 | 1 |
| ISBth13 | IS110 | 1 588 | 19 | 0 | 1 | 1 |
| ISBth15 | IS200/IS605 | 1 490 | 1 | 0 | 0 | 0 |
| ISBth16 | IS200/IS605 | 1 821 | 1 | 0 | 1 | 0 |
| IS231N | IS4 | 1 654 | 0 | 0 | 1 | 1 |
| IS231Y | IS4 | 1 645 | 0 | 0 | 0 | 1 |
| ISBce9 | IS4 | 1 597 | 0 | 0 | 0 | 1 |
| IS240A | IS6 | 861 | 0 | 13 | 0 | 0 |
| IS240B | IS6 | 861 | 0 | 0 | 4 | 2 |
| 总计Total | 24 | 17 | 7 | 7 |
与LM1212菌株相比,在LM1212-DB菌株中共缺失了6个插入序列单元,其中包括2个ISBth13、3个IS240A和1个IS231N。
3 讨论与结论LM1212菌株是一株具有独特表型的苏云金芽胞杆菌,可分化成芽胞形成细胞和晶体产生细胞,比例约为6:4[12]。这样的特性在Bt菌株yunnanensis亚种中也曾被发现,但产生这种现象的分子机制尚未阐明[32, 35]。与野生株LM1212相比,突变株LM1212-DB晶体产生细胞的比例更高,芽胞形成细胞比例更低,为LM1212菌株表型的分子机制解析奠定了重要的基础。此外,与LM1212菌株相比,LM1212-DB菌株中晶体产生细胞比例的增加可大大提高Bt杀虫剂的产量和纯度,从而提高Bt制剂的防治效果。本研究从LM1212和LM1212-DB菌株基因组入手,结合比较基因组学和生物信息学分析的手段,探究二者细胞表型变化的原因。从染色体和质粒变异、双组分信号系统、插入序列等方面对其表型差异原因进行解析。
本研究发现,与野生株LM1212相比,LM1212-DB菌株具有染色体片段缺失、质粒缺失、质粒上部分片段缺失、质粒间重组及质粒拷贝数变化等生物学现象。基于全基因组数据的分析发现,这两个菌株具有丰富的插入序列和重组酶。研究发现,插入序列具有多种转座机制可插入到基因编码区导致基因缺失或倒置,也可插入到基因的启动子区抑制下游基因的表达[3]。重组酶则可以把重组分子重新拼接,导致基因重排及质粒重组等现象。因此,两个菌株中丰富的插入序列和重组酶很可能是导致两个菌株基因组结构变异的原因。伴随着基因组结构的变异,很多影响菌株分化表型的重要基因也会随之缺失或拷贝数增加,从而导致其功能受到影响,进而对菌株的表型产生影响。
基于基因组比较分析的结果,我们推测有5个方面的因素导致了LM1212-DB和LM1212菌株表型的差异:(1) LM1212-DB菌株中缺少了一些参与环境胁迫响应相关的基因,如响应环境胁迫的转录因子Sigma B[30, 36]以及XRE家族转录因子等。这些环境胁迫响应基因的缺失,可能导致启动芽胞形成的信号转导途径被部分阻断,从而大大降低了芽胞形成细胞的比例。事实上,芽胞形成过程是Bt菌应对恶劣环境的一种机制,而环境胁迫响应基因的缺失很可能导致芽胞形成的决策途径被阻断,从而影响芽胞形成起始。例如在枯草芽胞杆菌中已经证实,芽胞形成起始的开关是Spo0A的磷酸化,而Spo0A的磷酸化需要来自Spo0E传递的磷酸信号[37]。Spo0E的启动子中有一个是Sigma B依赖型的,这种由Sigma B激活Spo0E启动子活性的方式是启动Spo0A磷酸化的重要途径[38-39]。这意味着,LM1212-DB菌株中Sigma B的缺失也可能导致芽胞形成分子开关无法正常打开,从而抑制了LM1212-DB菌株的芽胞形成。(2)与芽胞形成相关的重要转录因子AbrB等基因的缺失,可能与表型差异密切相关,但仍需进一步的实验进行验证。(3) LM1212-DB菌株质粒拷贝数的变化,导致某些关键基因的拷贝数也发生了变化,如在LM1212-DB菌株中晶体形成细胞转录因子CpcR增加了1个拷贝,从而导致CpcR的表达量升高,增加晶体细胞比例的作用更为明显,导致菌株表型发生变化。(4)缺失片段含有大量未知功能的基因,其中某些基因可能参与或调控了芽胞形成过程,从而导致表型差异。(5)由于LM1212-DB菌株中缺失了许多重要的调控因子,使LM1212-DB菌株中与表型相关的调控模块网络瘫痪,进而使受其抑制的基因在突变体中表达量上调,从而间接导致表型差异。
本研究还对LM1212和LM1212-DB菌株的特异性基因进行挖掘,也让我们深入了解了这两个菌株的特性,对其应用具有指导意义。基于全基因组数据的分析发现,这两个菌株具有丰富的双组分信号系统以及插入序列,暗示该菌株可响应复杂的环境变化。本研究筛选得到的36对双组分信号系统,如在枯草芽胞杆菌中已经解析的调控细胞分化的转录因子DegU、ComA和Spo0A等[38-39],这表明在LM1212菌株中也存在着复杂的细胞分化调控网络。同时,发现基因组中也存在HK和RR的孤儿基因,因此这两个株菌可能存在一个HK可磷酸化多个RR或一个RR可被多个HK磷酸化的复杂磷酸传递网络,各双组分信号系统间可能也存在着信号交谈(cross talk)的现象。此外,对转录因子CpcR进行进化分析时发现,该基因属于双组分信号系统OmpR家族转录因子,这意味着在LM1212和LM1212-DB菌株中还存在着一个与CpcR互作的HK,而且该基因不是产胞细菌的核心基因,应该是伴随环境进化过程中的水平转移基因;与此同时,与LM1212表型相近的Bt菌株yunnanensis亚种中存在着与CpcR氨基酸序列相似性高达85%的同源基因,这意味着CpcR和其互作的激酶可能是导致LM1212菌株细胞分化表型的重要原因之一,而对CpcR互作激酶的寻找将是有利于LM1212细胞分化机制解析的重要方向。此外,对LM1212菌株的插入序列单元的分布进行分析发现,在LM1212菌株中,与插入序列相邻的基因多为杀虫晶体蛋白基因,这表明插入序列和杀虫晶体蛋白基因可能存在着共同进化的关系。除了杀虫晶体蛋白基因外,部分相邻的基因被注释为假想蛋白,进一步解析这些假想蛋白的功能将有助于阐明插入序列与进化之间的关系。LM1212-DB菌株的染色体上缺失了Ⅰ型限制-修饰系统的操纵子,这使得LM1212-DB菌株与野生菌株相比具有更好的外源DNA兼容性。
通过对苏云金芽胞杆菌LM1212和LM1212-DB菌株进行基因组分析发现,两个菌株表型差异的主要原因可能是由于染色体和质粒变异引发了菌株核心基因及特异性基因之间的系统平衡被打破,进而导致菌株细胞分化的表型趋向于一个极端。上述结果仍需开展进一步实验研究,有利于明确Bt细胞分化的分子机制以及Bt菌株资源和基因挖掘的研究方向。本研究通过对两个菌株的基因组进行比较分析,从基因组层面解析了两个菌株表型差异的原因,为细菌细胞分化的机制研究奠定了基础,也对Bt制剂的遗传改良具有重要的指导意义。
| [1] |
Bravo A, Likitvivatanavong S, Gill SS, et al. Bacillus thuringiensis: a story of a successful bioinsecticide[J]. Insect Biochemistry and Molecular Biology, 2011, 41(7): 423-431. DOI:10.1016/j.ibmb.2011.02.006 |
| [2] |
Jouzani GS, Valijanian E, Sharafi R. Bacillus thuringiensis: a successful insecticide with new environmental features and tidings[J]. Applied Microbiology and Biotechnology, 2017, 101(7): 2691-2711. DOI:10.1007/s00253-017-8175-y |
| [3] |
Estruch JJ, Warren GW, Mullins MA, et al. Vip3A, a novel Bacillus thuringiensis vegetative insecticidal protein with a wide spectrum of activities against lepidopteran insects[J]. Proceedings of the National Academy of Sciences of the United States of America, 1996, 93(11): 5389-5394. DOI:10.1073/pnas.93.11.5389 |
| [4] |
Donovan WP, Engleman JT, Donovan JC, et al. Discovery and characterization of Sip1A: a novel secreted protein from Bacillus thuringiensis with activity against coleopteran larvae[J]. Applied Microbiology and Biotechnology, 2006, 72(4): 713-719. DOI:10.1007/s00253-006-0332-7 |
| [5] |
Aguilar PS, Hernandez-Arriaga AM, Cybulski LE, et al. Molecular basis of thermosensing: a two-component signal transduction thermometer in Bacillus subtilis[J]. The EMBO Journal, 2001, 20(7): 1681-1691. DOI:10.1093/emboj/20.7.1681 |
| [6] |
Jordan S, Hutchings MI, Mascher T. Cell envelope stress response in gram-positive bacteria[J]. FEMS Microbiology Reviews, 2008, 32(1): 107-146. DOI:10.1111/j.1574-6976.2007.00091.x |
| [7] |
Jiang M, Shao WL, Perego M, et al. Multiple histidine kinases regulate entry into stationary phase and sporulation in Bacillus subtilis[J]. Molecular Microbiology, 2000, 38(3): 535-542. DOI:10.1046/j.1365-2958.2000.02148.x |
| [8] |
Lyon GJ, Novick RP. Peptide signaling in Staphylococcus aureus and other gram-positive bacteria[J]. Peptides, 2004, 25(9): 1389-1403. DOI:10.1016/j.peptides.2003.11.026 |
| [9] |
Szurmant H, Ordal GW. Diversity in chemotaxis mechanisms among the bacteria and archaea[J]. Microbiology and Molecular Biology Reviews, 2004, 68(2): 301-319. |
| [10] |
Zaghloul L, Tang C, Chin HY, et al. The distribution of insertion sequences in the genome of Shigella flexneri strain 2457T[J]. FEMS Microbiology Letters, 2007, 277(2): 197-204. DOI:10.1111/j.1574-6968.2007.00957.x |
| [11] |
Deng C, Peng Q, Song FP, et al. Regulation of cry gene expression in Bacillus thuringiensis[J]. Toxins, 2014, 6(7): 2194-2209. DOI:10.3390/toxins6072194 |
| [12] |
Deng C, Slamti L, Raymond B, et al. Division of labour and terminal differentiation in a novel Bacillus thuringiensis strain[J]. The ISME Journal, 2015, 9(2): 286-296. DOI:10.1038/ismej.2014.122 |
| [13] |
Zhang RB, Slamti L, Tong L, et al. The stationary phase regulator CpcR activates cry gene expression in non‐sporulating cells of Bacillus thuringiensis[J]. Molecular Microbiology, 2020, 113(4): 740-754. DOI:10.1111/mmi.14439 |
| [14] |
Ma LX, Peng Q, Didier L, et al. Effect of plasmid curing on cell differentiation of Bacillus thuringiensis strain LM1212[J]. Microbiology China, 2017, 44(3): 574-582. (in Chinese) 马丽霞, 彭琦, Didier L, 等. 苏云金芽胞杆菌LM1212质粒缺失对细胞分化的影响[J]. 微生物学通报, 2017, 44(3): 574-582. |
| [15] |
Stobdan T, Kaur S, Singh A. Cloning and nucleotide sequence of a novel cry gene from Bacillus thuringiensis[J]. Biotechnology Letters, 2004, 26(14): 1153-1156. DOI:10.1023/B:BILE.0000035488.85309.df |
| [16] |
Eid J, Fehr A, Gray J, et al. Real-time DNA sequencing from single polymerase molecules[J]. Science, 2009, 323(5910): 133-138. DOI:10.1126/science.1162986 |
| [17] |
Faino L, Seidl MF, Datema E, et al. Single-molecule real-time sequencing combined with optical mapping yields completely finished fungal genome[J]. Microbiology, 2015, 6(4): e00936-15. |
| [18] |
Hyatt D, Chen GL, LoCascio PF, et al. Prodigal: prokaryotic gene recognition and translation initiation site identification[J]. BMC Bioinformatics, 2010, 11(1): 119. DOI:10.1186/1471-2105-11-119 |
| [19] |
Nawrocki EP, Eddy SR. Infernal 1.1: 100-fold faster RNA homology searches[J]. Bioinformatics, 2013, 29(22): 2933-2935. DOI:10.1093/bioinformatics/btt509 |
| [20] |
Lagesen K, Hallin P, Rødland EA, et al. RNAmmer: consistent and rapid annotation of ribosomal RNA genes[J]. Nucleic Acids Research, 2007, 35(9): 3100-3108. DOI:10.1093/nar/gkm160 |
| [21] |
Nawrocki EP, Burge SW, Bateman A, et al. Rfam 12.0: updates to the RNA families database[J]. Nucleic Acids Research, 2015, 43: D130-D137. DOI:10.1093/nar/gku1063 |
| [22] |
Magrane M, Consortium U. UniProt knowledgebase: a hub of integrated protein data[J]. Database, 2011, 2011: bar009. |
| [23] |
Tatusova T, Ciufo S, Fedorov B, et al. RefSeq microbial genomes database: new representation and annotation strategy[J]. Nucleic Acids Research, 2014, 42(D1): D553-D559. DOI:10.1093/nar/gkt1274 |
| [24] |
Kanehisa M, Araki M, Goto S, et al. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Research, 2008, 36(S1): D480-D484. |
| [25] |
Siguier P, Perochon J, Lestrade L, et al. ISfinder: the reference centre for bacterial insertion sequences[J]. Nucleic Acids Research, 2006, 34(S1): D32-D36. |
| [26] |
Kurtz S, Phillippy A, Delcher AL, et al. Versatile and open software for comparing large genomes[J]. Genome Biology, 2004, 5(2): R12. |
| [27] |
Schindler U, Sans N, Schröder J. Ornithine cyclodeaminase from octopine Ti plasmid Ach5: identification, DNA sequence, enzyme properties, and comparison with gene and enzyme from nopaline Ti plasmid C58[J]. Journal of Bacteriology, 1989, 171(2): 847-854. DOI:10.1128/JB.171.2.847-854.1989 |
| [28] |
Franklin MJ, Ohman DE. Identification of algI and algJ in the Pseudomonas aeruginosa alginate biosynthetic gene cluster which are required for alginate O acetylation[J]. Journal of Bacteriology, 1996, 178(8): 2186-2195. DOI:10.1128/JB.178.8.2186-2195.1996 |
| [29] |
Raleigh EA, Brooks JE. Restriction modification systems: where they are and what they do[A]//de Bruijn FJ, Lupski JR, Weinstock GM. Bacterial Genomes[M]. Boston: Springer, 1998: 78-92
|
| [30] |
Hecker M, Pané-Farré J, Uwe V. SigB-dependent general stress response in Bacillus subtilis and related gram-positive bacteria[J]. Annual Review of Microbiology, 2007, 61(1): 215-236. |
| [31] |
Fujita M, Sadaie Y. Feedback loops involving Spo0A and AbrB in In vitro transcription of the genes involved in the initiation of sporulation in Bacillus subtilis[J]. The Journal of Biochemistry, 1998, 124(1): 98-104. |
| [32] |
Srinivas G, Vennison SJ, Sudha SN, et al. Unique regulation of crystal protein production in Bacillus thuringiensis subsp. yunnanensis is mediated by the cry protein-encoding 103-megadalton plasmid[J]. Applied and Environmental Microbiology, 1997, 63(7): 2792-2797. DOI:10.1128/AEM.63.7.2792-2797.1997 |
| [33] |
Zhang MS, Zhu DK, Wang MS. Transposases in bacterial insertion sequences and their transposition mechanisms[J]. Chinese Journal of Biochemistry and Molecular Biology, 2018, 34(10): 1057-1064. (in Chinese) 张孟思, 朱德康, 汪铭书. 细菌插入序列的转座酶和转座机制[J]. 中国生物化学与分子生物学报, 2018, 34(10): 1057-1064. |
| [34] |
Rosso ML, Delécluse A. Distribution of the insertion element IS240 among Bacillus thuringiensis strains[J]. Current Microbiology, 1997, 34(6): 348-353. DOI:10.1007/s002849900194 |
| [35] |
Ohba M, Aizawa K. Crystals of Bacillus thuringiensis subsp. yunnanensis are produced only in asporogenous cells[J]. Journal of Invertebrate Pathology, 1986, 48(2): 254-256. DOI:10.1016/0022-2011(86)90134-5 |
| [36] |
Kint N, Janoir C, Monot M, et al. The alternative sigma factor σB, plays a crucial role in adaptive strategies of Clostridium difficile during gut infection[J]. Environmental Microbiology, 2017, 19(5): 1933-1958. DOI:10.1111/1462-2920.13696 |
| [37] |
Burbulys D, Trach KA, Hoch JA. Initiation of sporulation in B. subtilis is controlled by a multicomponent phosphorelay[J]. Cell, 1991, 64(3): 545-552. DOI:10.1016/0092-8674(91)90238-T |
| [38] |
López D, Kolter R. Extracellular signals that define distinct and coexisting cell fates in Bacillus subtilis[J]. FEMS Microbiology Reviews, 2010, 34(2): 134-149. DOI:10.1111/j.1574-6976.2009.00199.x |
| [39] |
Reder A, Gerth U, Hecker M. Integration of σB activity into the decision-making process of sporulation initiation in Bacillus subtilis[J]. Journal of Bacteriology, 2012, 194(5): 1065-1074. DOI:10.1128/JB.06490-11 |
 2020, Vol. 47
2020, Vol. 47