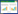扩展功能
文章信息
- 张欢畅, 段云峰, 徐黎明, 蔡峰, 律娜, 朱宝利, 常学东
- ZHANG Huan-Chang, DUAN Yun-Feng, XU Li-Ming, CAI Feng, Lü Na, ZHU Bao-Li, CHANG Xue-Dong
- 两株母乳源植物乳杆菌的全基因组测序分析
- Whole genome sequencing of two Lactobacillus plantarum strains isolated from human milk
- 微生物学通报, 2020, 47(6): 1876-1887
- Microbiology China, 2020, 47(6): 1876-1887
- DOI: 10.13344/j.microbiol.china.190226
-
文章历史
- 收稿日期: 2019-03-21
- 接受日期: 2019-12-09
- 网络首发日期: 2020-04-08
2. 中国科学院病原微生物与免疫学重点实验室 中国科学院微生物研究所 北京 100101;
3. 云南农业大学资源与环境学院 云南 昆明 650201
2. Key Laboratory of Pathogenic Microbiology and Immunology, Institute of Microbiology Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
3. College of Resources and Environment, Yunnan Agricultural University, Kunming, Yunnan 650201, China
人们以往普遍认为母乳是一种无菌液体。但后来的研究发现,母乳中存在一些具有益生功能的微生物[1-2]。2015年研究人员对98名足月生产的瑞典婴儿和他们的母亲进行了肠道微生物基因组分析,发现完全母乳喂养与配方乳喂养的婴儿肠道微生物存在明显差异,纯母乳喂养的婴儿肠道益生菌丰度更高[3],因此母乳是一个重要的益生菌筛选库。
益生菌有着悠久的食用历史[4],被定义为“活的微生物,当给予足够量时,对宿主的健康有益”[5]。其中植物乳杆菌是目前研究比较集中的多功能益生菌,该菌用途广泛、适应性强[6],其产生的抗菌物质可有效替代抗生素抑制单核细胞增生李斯特氏菌54002的生长[7]。2017年,印度进行了一项涉及4 556名新生儿服用植物乳杆菌合生元的大型实验,结果显示植物乳杆菌可明显降低新生儿脓毒症的发病率[6]。此外该菌还可自发性地修饰、删除本身的特定功能基因,调整基因组结构以适应环境生态的需求[8]。比如在植物乳杆菌WCFS1中存在一些必需的基因组,可耐受胆汁消化、在肠道中存活[9]。然而随着益生菌基因组学的发展,我们发现不同菌株具有不同的功能,现有的生理生化方法对其更多潜在特征的研究仍十分有限。采用菌株表型特征与基因型结合的方法来评估益生菌相关特征,将有望为探索其潜在的生物学功能提供更多信息[10],是一条挖掘优质益生菌资源的有效途径[11]。
本研究从健康的母乳中分离筛选植物乳杆菌,并利用二代测序技术对这些分离株的基因组进行测序;同时对菌株的肠道耐受与合成细菌素的关键基因进行挖掘,以助于确定这些菌株是否具有作为工业应用益生菌的潜力,也为这些菌株的进一步研究和应用提供参考。
1 材料与方法 1.1 主要试剂和仪器MRS培养基,Oxoid公司;细菌DNA提取试剂盒,天根生化科技(北京)有限公司;PremixSTAR DNA Polymerase,TaKaRa公司;猪胆盐,Macklin公司;胃蛋白酶,Sigma公司;胰蛋白酶,AMRESCO公司。冷场发射扫描电子显微镜,Hitach公司;生化培养箱,上海博迅实业有限公司;离心机,Beckman公司;PCR仪,Bio-Rad公司。
1.2 菌株的分离与培养收集分娩后1-3 d的10名身体健康的顺产新生儿母亲的母乳样本。在收集前,使用无菌水清洗单侧乳头及周围皮肤后,使用无菌手套丢弃第一滴,收集母乳5 mL置于含有3 mL MRS肉汤培养基的无菌采样管中[12]。将收集好的母乳样品,放置于冰袋上运送至实验室,立即进行梯度稀释、涂布于MRS琼脂平板上,37 ℃厌氧培养48 h后,分离、筛选植物乳杆菌。测试菌株对人工胃、肠液耐受性;两菌株发酵上清液对大肠杆菌ATCC 25922、铜绿假单胞菌ATCC 27553、金黄色葡萄球菌ATCC 27923的抑菌能力[13]。
1.3 菌株DNA提取使用细菌DNA提取试剂盒提取植物乳杆菌菌株的DNA,提取方法见说明书。
1.4 全基因组的测序与组装首先,对DNA样品进行打断、回收目的片段,使用T4 DNA Polymerase、Klenow DNA Polymerase和T4 Polynucleotide Kinase将打断形成的粘性末端修复成平末端,再通过3′端加碱基“A”,使DNA片段与3′端带有“T”碱基的特殊接头连接;使用合格的文库进行二代测序,将下机得到的数据进行生物信息分析。即使用SPAdes[14]短序列组装软件对Clean Data进行组装,多次调整参数、获得最优组装结果;随后将Reads进行de novo拼接,根据Reads的Daired-end和Overlap关系,对组装结果进行局部组装、优化;使用GapCloser[15]软件对组装结果进行内洞修补得到最后的组装结果,提交基因组序列至“gcMeta”平台[16] (PID: 21.86101/gcm.study.b4eaabc849e411e9a992b49 691092464)。
1.5 基因组注释功能与预测采用Prokka (rapid prokaryotic genome annotation)[17]、CAZy (carbohydrate-active enzymes)[18],KEGG (koyto encyclopedia of genes and genomes)[19]和COG (clusters of orthologous genes)[20]对细菌基因组进行快速注释,并得到相应的注释信息;采用Prodigal[21]、RNAmmer[22]等工具对编码序列、核糖体RNA基因进行注释与预测,使用CGView软件绘制两株植物乳杆菌的基因组环形图谱;使用MEGA构建遗传发育树。
2 结果与分析 2.1 菌株信息与生化特征测试菌株的表型和生化特征显示,植物乳杆菌MP37和MP55分离株在形态学上属革兰氏阳性、非运动、不产芽孢的杆状细菌(图 1与表 1)。耐酸性是任何能够在消化道中起作用的菌株所应拥有的特性,但目前很多分离株经过pH值为3.0人工胃液消化3 h后几乎完全失活。由图 2可见,MP37在耐受人工胃液3 h后的菌株活力由处理前的1.1×109 CFU/mL下降到2.0×107 CFU/mL;MP55由处理前的2.8×109 CFU/mL下降到2.1×108 CFU/mL;随即分别接入pH值为8.0的人工肠液,其活菌数量不仅不受影响,而且数量略有增加,考虑是消化液之间缓冲作用对菌株的保护作用所致,最终两菌株在耐受人工胃液3 h、人工肠液24 h后菌株活力均高于1×106 CFU/mL。使用琼脂双层扩散法进行的体外拮抗测试显示,两菌株发酵上清液均有不同程度的抑菌能力,排除乳酸影响后抑菌圈稍有缩小但并没有消失(图 3),说明在上清液中存在抑菌物质。

|
| 图 1 菌株扫描电镜分析 Figure 1 Scanning electron microscopic (SEM) analysis of bacterial isolates 注:A:植物乳杆菌MP37;B:植物乳杆菌MP55. Note: A: Lactobacillus plantarum MP37; B: Lactobacillus plantarum MP55. |
|
|
| 特征 Property |
植物乳杆菌MP37 Lactobacillus plantarum MP37 |
植物乳杆菌MP55 Lactobacillus plantarum MP55 |
| 域Domain | 细菌Bacteria | 细菌Bacteria |
| 门Phylum | 厚壁菌门Firmicutes | 厚壁菌门Firmicutes |
| 纲Class | 杆菌纲Bacilli | 杆菌纲Bacilli |
| 目Order | 乳杆菌目Lactobacillales | 乳杆菌目Lactobacillales |
| 科Family | 乳杆菌科Lactobacillaceae | 乳杆菌科Lactobacillaceae |
| 属Genus | 乳酸杆菌属Lactobacillus | 乳酸杆菌属Lactobacillus |
| 种Species | 植物乳杆菌Lactobacillus plantarum | 植物乳杆菌Lactobacillus plantarum |
| 株Strain | MP37 | MP55 |
| 革兰分型Gram stain | 革兰氏阳性菌Positive | 革兰氏阳性菌Positive |
| 细胞形态Cell shape | 杆状Rod | 杆状Rod |
| 最适培养温度Optimum temperature | 37 ℃ | 37 ℃ |
| 地理位置Geographic location | 中国河北Hebei/China | 中国河北Hebei/China |
|
| 图 2 不同环境下的活菌数量 Figure 2 Survival counts under different conditions |
|
|

|
| 图 3 琼脂扩散法测定植物乳杆菌MP37、MP55上清液对指示菌的抑菌性 Figure 3 Inhibition zones of Lactobacillus plantarum MP37, MP55 on agar plates, obtained from the agar well diffusion method 注:A、C为实验组,分别加入100 μL MP37、MP55发酵上清液;B、D为对照组,加入100 μL使用乳酸调整到相同pH的MRS培养基;指示菌为:大肠杆菌ATCC25922、铜绿假单胞菌ATCC27553、金黄色葡萄球菌ATCC27923. Note: A, C for the experimental group, respectively, add 100 μL of MP37, MP55 fermentation supernatant; B, D for the control group, add 100 μL of MRS medium adjusted to the same pH using lactic acid; The indicator bacteria are: Escherichia coli ATCC25922, Pseudomonas aeruginosa ATCC27553, Staphylococcus aureus ATCC27923. |
|
|
对测序样品进行数据质量评估与短序列组装,组装结果见表 2;整理组装完整序列后,拼接两株植物乳杆菌的基因组环状图(图 4)。
| 特征 Features |
植物乳杆菌MP37 Lactobacillus plantarum MP37 |
植物乳杆菌MP55 Lactobacillus plantarum MP55 |
| Number of scaffolds | 31.00 | 19.00 |
| N50 (bp) | 363 203.00 | 366 013.00 |
| Mean_length (bp) | 106 425.00 | 168 653.00 |
| Max_length (bp) | 632 608.00 | 667 180.00 |
| Full_length (bp) | 3 299 180.00 | 3 204 421.00 |
| Q20% | 98.47 | 98.44 |
| Q30% | 95.95 | 95.87 |
| (G+C)mol% | 44.46 | 44.36 |

|
| 图 4 植物乳杆菌MP37 (A)、MP55 (B)的基因组圆形图谱 Figure 4 Circular map of Lactobacillus plantarum MP37 (A), MP55 (B) genome 注:基因组特征,从外环到中心:1:蛋白编码序列;2:tRNA基因;3:rRNA基因;4:其他;5:GC含量;6:GC正向偏移;7:GC反向偏移. Note: Genomic features, moving from the periphery to the center of the map: 1: CDSs; 2: tRNA genes; 3: rRNA genes; 4: Other; 5: GC content; 6: GC skew+; 7: GC skew-. |
|
|
植物乳杆菌MP37、MP55基因组显示,两菌株全长分别为3 299 180 bp和3 204 421 bp;(G+C)mol%含量分别为44.46%、44.36%;其中分别包含了3 101、3 012个蛋白质编码序列(protein-coding sequence,CDS),统计信息见表 3。
| 特征 Features |
植物乳杆菌MP37 Lactobacillus plantarum MP37 |
植物乳杆菌MP55 Lactobacillus plantarum MP55 |
| Bases | 3 394 812 | 3 286 523 |
| CDS | 3 101 | 3 012 |
| Number_of_Contigs | 340 | 288 |
| tmRNA | 8 | 7 |
| rRNA | 1 | 1 |
| tRNA | 67 | 69 |
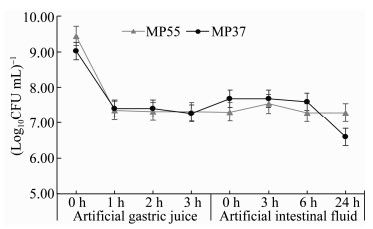
为了探究2株菌株与其他乳酸杆菌之间的进化关系,构建包含MP37、MP55以及具有完整基因组的8株植物乳杆菌与同属不同种的5株乳酸杆菌的系统发育树(图 5)。系统发育树显示MP37、MP55聚集在一起并与植物乳杆菌处于同一分支,与其他乳杆菌属细菌明显分开,其中植物Lactobacillus plantarum DR7与MP37、MP55有最接近的亲缘关系。
|
| 图 5 15株乳酸杆菌构建的遗传发育树 Figure 5 15 construction of a phylogenetic tree from 15 Lactobacillus strains 注:AU:渐近无偏值;BP:结合自助值. Note: AU: Approximately unbiased; BP: Bootstrap probability. |
|
|
CAZy是研究碳水化合物合成与分解酶的重要数据库[23]。根据预测结果,MP37、MP55的蛋白质结构域内氨基酸的序列可划分为不同的种类,包括含糖基转移酶、糖苷水解酶、碳水化合物酯酶、碳水化合物结合模块和附属活力酶。该库成功注释的酶类分别为135个和125个,MP37明显多于MP55 (图 6)。其中糖苷水解酶被注释最多,分别为58个和47个,占总注释的43%、38%;其次为糖基转移酶、碳水化合物结合模块、碳水化合物酯酶,而附属活力酶注释最少,分别为7个和9个。

|
| 图 6 植物乳杆菌MP37、MP55的CAZy功能注释图 Figure 6 CAZy functional classification map of L. plantarum MP37, MP55 注:GH:糖苷水解酶;CE:碳水化合物酯酶;GT:糖基转移酶;CBM:碳水化合物结合模块;AA:附属活力酶. Note: GH: Glycoside hydrolases; CE: Carbohydrate esterase; GT: Glycosyltransferase; CBM: Carbohydrate binding module; AA: Auxiliary activities. |
|
|
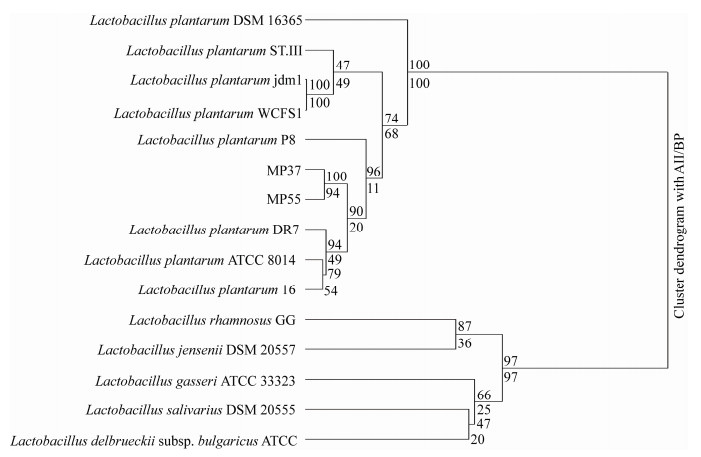
KEGG是在分子水平上对基因、基因组及代谢通路进行标注的数据库[24]。在该数据库中,两菌株中有2 240和2 135个编码基因得到注释,MP37的注释基因多于MP55,并相差105个之多(图 7)。从图 7可以看出,两菌株参与新陈代谢途径最多,其余则分布在环境信息处理、基因信息处理、人类疾病、细胞过程、生物体系统中。
|
| 图 7 植物乳杆菌MP55、MP37的KEGG功能分类图 Figure 7 KEGG functional classification map of L. plantarum MP55, MP37 |
|
|
COG数据库是微生物基因组注释和比较基因组学中常用的工具[20]。常用于对菌株进行基因组功能上的注释[25]。
植物乳杆菌WCFS1的基因组被广泛研究,该菌可存活于人体肠道、提高人体免疫力[26]。因此,将分离的两株菌与其进行对比,以帮助我们对植物乳杆菌MP37、MP55进行更全面的认识。
使用CDS对两株菌的基因组进行23个功能分类,分别获得了2 948、2 863个COG功能注释(表 4)。在预测的CDSs中,可以发现两株菌参与碳水化合物运输与代谢、转录和一般功能预测最多。与植物乳杆菌MP37、WCFS1相比,植物乳杆菌M55参与碳水化合物转运和代谢(G)的基因数量最多,占总注释的11%;三菌株之间,MP37参与能量生产和转换(C)相关的基因最多,占总注释的4.3%之多。另外,MP37、MP55在防御机制(V)也进行了一定数量的注释(分别为88、79),且数量要多于对比菌株(60)。
| COG code | 植物乳杆菌MP37 Lactobacillus plantarum MP37 |
植物乳杆菌MP55 Lactobacillus plantarum MP55 |
植物乳杆菌WCFS1 Lactobacillus plantarum WCFS1 |
| B | 1 | 1 | 0 |
| C | 124 | 124 | 114 |
| D | 43 | 41 | 26 |
| E | 232 | 234 | 267 |
| F | 108 | 107 | 87 |
| G | 309 | 341 | 313 |
| H | 124 | 141 | 91 |
| I | 106 | 104 | 75 |
| J | 214 | 215 | 155 |
| K | 287 | 290 | 305 |
| L | 123 | 121 | 157 |
| M | 174 | 183 | 156 |
| N | 21 | 19 | 11 |
| O | 87 | 92 | 62 |
| P | 134 | 138 | 169 |
| Q | 37 | 37 | 42 |
| R | 269 | 268 | 421 |
| S | 190 | 194 | 224 |
| T | 132 | 136 | 100 |
| U | 23 | 24 | 26 |
| V | 88 | 79 | 60 |
| W | 4 | 4 | 0 |
| 注:B:染色质结构与动力学;C:能量生产与转换;D:细胞周期调控、细胞分裂、染色体分配;E:氨基酸转运与代谢;F:核苷酸转运和代谢;G:碳水化合物转运和代谢;H:辅酶转运和代谢;I:脂质转运和代谢;J:翻译、核糖体结构和生物合成;K:转录;L:DNA复制、重组和修复;M:细胞壁、细胞膜、胞外被膜生物合成;N:细胞运动;O:蛋白质翻译后修饰与转运;P:无机离子转运和代谢;Q:次级代谢产物的生物合成、转运和代谢;R:一般功能预测;S:功能未知;T:信号转导机制;U:胞内运输、分泌和囊泡运输;V:防御机制;W:胞外结构. Note: B: Chromatin structure and dynamics; C: Energy production and conversion; D: Cell cycle control, cell division, chromosome partitioning; E: Amino acid transport and metabolism; F: Nucleotide transport and metabolism; G: Carbohydrate transport and metabolism; H: Coenzyme transport and metabolism; I: Lipid transport and metabolism; J: Translation, ribosomal structure and biogenesis; L: Replication, recombination and repair; M: Cell wall/membrane/envelope biogenesis; N: Cell motility; O: Posttranslational modification, protein turnover, chaperones; P: Inorganic ion transport and metabolism; Q: Secondary metabolites biosynthesis, transport and catabolism; R: General function prediction only; S: Function unknown; T: Signal transduction mechanisms; U: Intracellular trafficking, secretion, and vesicular transport; V: Defense mechanisms; W: Extracellular structures. |
|||
肠道耐受性是筛选益生菌菌株最重要的特性之一,其决定了益生菌在小肠内是否具有生存能力[27]。胆汁盐水解酶(bile saline hydrolase,BSH)是一种可分解胆汁盐的酶类[28],因此益生菌代谢生成BSH的能力理论上可决定菌株耐受肠液的能力[29],同时与之对应的BSH编码基因便成为衡量菌株是否具有益生菌潜力的一个重要指标。另外,BSH还是一种去除胆固醇的重要酶类,其能水解氨基酸和胆固醇之间的酰胺键[30],因此,BSH也与胆固醇清除能力有关,此酶对高脂饮食的人群也是有益的[31]。
植物乳杆菌WCFS1菌株在肠道中具有极高的存活率,该菌的基因组中存在4个与BSH相关的基因(bsh1、bsh2、bsh3、bsh4)[32]。通过对比,在植物乳杆菌MP37和MP55中也找到上述4个基因(表 5),其相似度都在98%以上,基于这些基因组特征,可解释了两个菌株表现良好肠道耐受性的原因。
| 菌株Strains | bsh1 | bsh2 | bsh3 | bsh4 | |||||||
| 位置 Position (bp) |
相似度 Identity (%) |
位置 Position (bp) |
相似度 Identity (%) |
位置 Position (bp) |
相似度 Identity (%) |
位置 Position (bp) |
相似度 Identity (%) |
||||
| Lactobacillus plantarum M55 | 196 139-197 113 | 99.49 | 429 893-430 909 | 99.51 | 39 259-40 245 | 99.59 | 29 646-30 599 | 98.43 | |||
| Lactobacillus plantarum MP37 | 103 807-104 781 | 99.59 | 243 127-244 143 | 98.92 | 40 397-41 383 | 99.49 | 211 919-212 872 | 98.43 | |||
与植物乳杆菌细菌素合成相关基因簇大小约为18-19 kb。以报道较多的WCSF1株为例,该菌株细菌素合成簇分布在6个操纵子上、由23个基因组构成。其中,高度保守部分包括一个细菌素操纵子(plnEFI)和一个转运操纵子(plnGHSTUVW),而保守度较低区或不保守的部分包括一个调节操纵子(plnABCD)、一个细菌素操纵子(plnJKLR)以及两个研究较少、编码未知功能操纵子(plnMNOP、plnQ)[33]。
将植物乳杆菌MP37、MP55的测序结果与WCFS1进行对比(图 8),可以看出,植物乳杆菌MP37存在完整的细菌素合成基因簇;而植物乳杆菌MP55除存在完整的细菌素操纵子(plnEFI)转运操纵子(plnGHSTUVW)外,缺失了4个操作子的部分相关基因,即在调节操纵子(plnABCD)中缺失了plnA、plnB基因;在细菌素操纵子(plnJKLR)中缺失了plnK、plnJ基因;在编码未知功能的操纵子(plnMNOP、plnQ)中则存在整段缺失。

|
| 图 8 3株植物乳杆菌细菌素相关基因比较 Figure 8 Comparison of bacteriocin gene between the three Lactobacillus plantarum strains |
|
|
其中,plnJ、plnK两基因对细菌素合成有着很大的影响,这两个基因编码着一些有抑菌功能的活性肽物质,且当与plnE、plnF同时存在时能使菌株的抑菌效果增加1 000倍[34],因此,该基因的缺失在理论上可直接影响菌株的抑菌能力。但在抑菌实验中发现,MP55具备一定的抑菌性,因此,推测在MP55中,可能还存在一些与抑菌能力相关的其他基因,关于此问题还需要进一步探讨。
3 讨论与结论母乳微生物是一个复杂的细菌群落,失去母乳喂养的婴儿可能会出现肠道菌群失调,甚至出现过敏、哮喘和肥胖等疾病[35],但我们对母乳微生物的确切组成仍知之甚少[36]。本研究收集了健康的母乳样本,分离、筛选出两株植物乳杆菌。在前期的筛选阶段发现,这两株菌表现出良好的肠液耐受性,并同时对肠杆菌ATCC 25922、铜绿假单胞菌ATCC 27553、金黄色葡萄球菌ATCC 27923具有一定的抑制能力。在此基础上,我们对其进行了全基因组测序和分析,将基因序列分别使用CAZy、KEGG和COG数据库进行了注释。注释结果表明,MP37的注释结果明显多于MP55,且两株菌均参与糖基转移酶、新陈代谢、碳水化合物的基因,意味着其可能具有较强的碳水化合物利用能力。另外,我们确定了4个与BSH相关的基因,相似性都在98%以上;还发现了一段与细菌素相关的基因簇,其中MP37存在完整的细菌素合成簇,而MP55则缺失了4个操作子的部分相关基因,在基因层面揭示了植物乳杆菌MP55、MP37的潜在益生机理。
综上所述,本研究通过分离、筛选与基因组装共得到两株母乳源植物乳杆菌的全基因组信息,借助数据分析找到了与肠液耐受相关的基因和与抗性相关的细菌素合成基因簇,说明这两株菌确实是具有潜在功能的益生菌候选菌株。本实验的结果为进一步阐明益生菌特性的功能机制提供了遗传学基础,也为今后研究其他菌株的功能特性奠定了理论基础。
| [1] |
McGuire MK, McGuire MA. Human milk: mother nature's prototypical probiotic food?[J]. Advances in Nutrition, 2015, 6(1): 112-123. DOI:10.3945/an.114.007435 |
| [2] |
Damaceno QS, Souza JP, Nicoli JR, et al. Evaluation of potential probiotics isolated from human milk and colostrum[J]. Probiotics and Antimicrobial Proteins, 2017, 9(4): 371-379. DOI:10.1007/s12602-017-9270-1 |
| [3] |
Bäckhed F, Roswall J, Peng YQ, et al. Dynamics and stabilization of the human gut microbiome during the first year of life[J]. Cell Host & Microbe, 2015, 17(5): 690-703. |
| [4] |
Douillard FP, de Vos WM. Functional genomics of lactic acid bacteria: from food to health[J]. Microbial Cell Factories, 2014, 13(S1): S8. |
| [5] |
Hill C, Guarner F, Reid G, et al. Expert consensus document: the International Scientific Association for Probiotics and Prebiotics consensus statement on the scope and appropriate use of the term probiotic[J]. Nature Reviews Gastroenterology & Hepatology, 2014, 11(8): 506-514. |
| [6] |
Panigrahi P, Parida S, Nanda NC, et al. A randomized synbiotic trial to prevent sepsis among infants in rural India[J]. Nature, 2017, 548(7668): 407-412. DOI:10.1038/nature23480 |
| [7] |
Wang Y, Shang N, Qin YX, et al. The complete genome sequence of Lactobacillus plantarum LPL-1, a novel antibacterial probiotic producing class Ⅱa bacteriocin[J]. Journal of Biotechnology, 2018, 266: 84-88. DOI:10.1016/j.jbiotec.2017.12.006 |
| [8] |
Siezen RJ, van Hylckama Vlieg JE. Genomic diversity and versatility of Lactobacillus plantarum, a natural metabolic engineer[J]. Microbial Cell Factories, 2011, 10(S1): S3. |
| [9] |
Suryavanshi MV, Paul D, Doijad SP, et al. Draft genome sequence of Lactobacillus plantarum strains E2C2 and E2C5 isolated from human stool culture[J]. Standards in Genomic Sciences, 2017, 12(1): 15. DOI:10.1186/s40793-017-0222-x |
| [10] |
Ventura M, O'Flaherty S, Claesson MJ, et al. Genome-scale analyses of health-promoting bacteria: probiogenomics[J]. Nature Reviews Microbiology, 2009, 7(1): 61-71. DOI:10.1038/nrmicro2047 |
| [11] |
Seddik HA, Bendali F, Gancel F, et al. Lactobacillus plantarum and its probiotic and food potentialities[J]. Probiotics and Antimicrobial Proteins, 2017, 9(2): 111-122. DOI:10.1007/s12602-017-9264-z |
| [12] |
Jiang ML, Zhang F, Wan CX, et al. Evaluation of probiotic properties of Lactobacillus plantarum WLPL04 isolated from human breast milk[J]. Journal of Dairy Science, 2016, 99(3): 1736-1746. DOI:10.3168/jds.2015-10434 |
| [13] |
Arena MP, Silvain A, Normanno G, et al. Use of Lactobacillus plantarum strains as a bio-control strategy against food-borne pathogenic microorganisms[J]. Frontiers in Microbiology, 2016, 7: 464. DOI:10.3389/fmicb.2016.00464 |
| [14] |
Palmeira JD, Ferreira H, Madec JY, et al. Draft genome of a ST443 mcr-1- and blaCTX-M-2-carrying Escherichia coli from cattle in Brazil[J]. Journal of Global Antimicrobial Resistance, 2018, 13: 269-270. DOI:10.1016/j.jgar.2018.05.010 |
| [15] |
Zhu LY, Liu N, Wang HQ, et al. Draft genome sequence of broad-spectrum antifungal-producing Bacillus velezensis C4341 isolated from a saline-alkali soil sample in China[J]. Journal of Global Antimicrobial Resistance, 2019, 16: 291-293. DOI:10.1016/j.jgar.2018.12.019 |
| [16] |
Shi WY, Qi HY, Sun QL, et al. gcMeta: a Global Catalogue of Metagenomics platform to support the archiving, standardization and analysis of microbiome data[J]. Nucleic Acids Research, 2019, 47(D1): D637-D648. DOI:10.1093/nar/gky1008 |
| [17] |
Bazinet AL. Pan-genome and phylogeny of Bacillus cereus sensu lato[J]. BMC Evolutionary Biology, 2017, 17(1): 176. |
| [18] |
Huang L, Zhang H, Wu PZ, et al. dbCAN-seq: a database of carbohydrate-active enzyme (CAZyme) sequence and annotation[J]. Nucleic Acids Research, 2018, 46(D1): D516-D521. DOI:10.1093/nar/gkx894 |
| [19] |
Kanehisa M, Sato Y, Morishima K. BlastKOALA and GhostKOALA: KEGG tools for functional characterization of genome and metagenome sequences[J]. Journal of Molecular Biology, 2016, 428(4): 726-731. DOI:10.1016/j.jmb.2015.11.006 |
| [20] |
Galperin MY, Kristensen DM, Makarova KS, et al. Microbial genome analysis: the COG approach[J]. Briefings in Bioinformatics, 2019, 20(4): 1063-1070. DOI:10.1093/bib/bbx117 |
| [21] |
Pendarvis K, Padula MP, Tacchi JL, et al. Proteogenomic mapping of Mycoplasma hyopneumoniae virulent strain 232[J]. BMC Genomics, 2014, 15(1): 576. DOI:10.1186/1471-2164-15-576 |
| [22] |
Singh NK, Bezdan D, Sielaff AC, et al. Multi-drug resistant Enterobacter bugandensis species isolated from the International Space Station and comparative genomic analyses with human pathogenic strains[J]. BMC Microbiology, 2018, 18(1): 175. DOI:10.1186/s12866-018-1325-2 |
| [23] |
Nguyen STC, Freund HL, Kasanjian J, et al. Function, distribution, and annotation of characterized cellulases, xylanases, and chitinases from CAZy[J]. Applied Microbiology and Biotechnology, 2018, 102(4): 1629-1637. DOI:10.1007/s00253-018-8778-y |
| [24] |
Kanehisa M, Furumichi M, Tanabe M, et al. KEGG: new perspectives on genomes, pathways, diseases and drugs[J]. Nucleic Acids Research, 2017, 45(D1): D353-D361. DOI:10.1093/nar/gkw1092 |
| [25] |
Tatusov RL, Galperin MY, Natale DA, et al. The COG database: a tool for genome-scale analysis of protein functions and evolution[J]. Nucleic Acids Research, 2000, 28(1): 33-36. |
| [26] |
van den Nieuwboer M, van Hemert S, Claassen E, et al. Lactobacillus plantarum WCFS1 and its host interaction: a dozen years after the genome[J]. Microbial Biotechnology, 2016, 9(4): 452-465. DOI:10.1111/1751-7915.12368 |
| [27] |
Ruiz L, Margolles A, Sánchez B. Bile resistance mechanisms in Lactobacillus and Bifidobacterium[J]. Frontiers in Microbiology, 2013, 4: 396. |
| [28] |
Xiong ZQ, Wang QH, Kong LH, et al. Short communication: improving the activity of bile salt hydrolases in Lactobacillus casei based on in silico molecular docking and heterologous expression[J]. Journal of Dairy Science, 2017, 100(2): 975-980. DOI:10.3168/jds.2016-11720 |
| [29] |
Begley M, Hill C, Gahan CGM. Bile salt hydrolase activity in probiotics[J]. Applied and Environmental Microbiology, 2006, 72(3): 1729-1738. |
| [30] |
LeBlanc JG, Chain F, Martín R, et al. Beneficial effects on host energy metabolism of short-chain fatty acids and vitamins produced by commensal and probiotic bacteria[J]. Microbial Cell Factories, 2017, 16(1): 79. DOI:10.1186/s12934-017-0691-z |
| [31] |
Dong Z, Zhang J, Lee B, et al. A bile salt hydrolase gene of Lactobacillus plantarum BBE7 with high cholesterol-removing activity[J]. European Food Research and Technology, 2012, 235(3): 419-427. DOI:10.1007/s00217-012-1769-9 |
| [32] |
Yin XC, Lee B, Zaragoza J, et al. Dietary perturbations alter the ecological significance of ingested Lactobacillus plantarum in the digestive tract[J]. Scientific Reports, 2017, 7(1): 7267. DOI:10.1038/s41598-017-07428-w |
| [33] |
Diep DB, Straume D, Kjos M, et al. An overview of the mosaic bacteriocin pln loci from Lactobacillus plantarum[J]. Peptides, 2009, 30(8): 1562-1574. DOI:10.1016/j.peptides.2009.05.014 |
| [34] |
Branch AD. Antisense drug discovery: can cell-free screens speed the process?[J]. Antisense and Nucleic Acid Drug Development, 1998, 8(3): 249-254. DOI:10.1089/oli.1.1998.8.249 |
| [35] |
Gomez-Gallego C, Garcia-Mantrana I, Salminen S, et al. The human milk microbiome and factors influencing its composition and activity[J]. Seminars in Fetal and Neonatal Medicine, 2016, 21(6): 400-405. DOI:10.1016/j.siny.2016.05.003 |
| [36] |
Bode L, McGuire M, Rodriguez JM, et al. It's alive: microbes and cells in human milk and their potential benefits to mother and infant[J]. Advances in Nutrition, 2014, 5(5): 571-573. DOI:10.3945/an.114.006643 |
 2020, Vol. 47
2020, Vol. 47