扩展功能
文章信息
- 张志宇, 刘东格, 卓君雨, 景如贤, 李祎
- ZHANG Zhi-Yu, LIU Dong-Ge, ZHUO Jun-Yu, JING Ru-Xian, LI Yi
- 一株降解纤维素放线菌的产纤维素酶基因克隆与表达
- Cloning and expression of cellulase gene from a strain of cellulose degrading actinomycete
- 微生物学通报, 2020, 47(6): 1730-1739
- Microbiology China, 2020, 47(6): 1730-1739
- DOI: 10.13344/j.microbiol.china.190714
-
文章历史
- 收稿日期: 2019-08-29
- 接受日期: 2019-10-12
- 网络首发日期: 2019-11-04
纤维素是自然界分布最广、含量最多的一种由葡萄糖组成的大分子多糖[1],广泛应用于动物饲料、食品、纺织品、清洁剂和纸张制造[2-3],然而天然纤维素难以降解,在生产生活中对其利用有很大的局限性[4]。过往人们对富含纤维素的木材废料、废纸、作物残渣等进行简单堆积或焚烧,不仅没有充分利用纤维素而且对生态环境造成了污染[5]。在传统工业生产中,一般使用酸碱水解纤维素,但是处理后会引入一些不利于纤维素降解的物质且成本较高[6-7]。近年来面对日益严重的环境和资源匮乏问题[8],越来越多的人把目光投向利用微生物来降解纤维素,这种生物处理方法不仅降解效率高[9]、对环境友好,而且有效降低了成本[10-11],能有效加强对纤维素的开发和利用。微生物降解纤维素关键在于其能否产生纤维素酶,目前对于产纤维素酶菌种研究较多的是曲霉(Aspergillus sp.)、木霉(Trichoderma sp.)、腐质霉(Humicola sp.)等真菌[12],但是用真菌分解木质纤维素往往需要较长时间[13]。
本研究通过筛选分离出一株能降解纤维素的放线菌Lb1,并对其进行全基因组测序,对功能基因进行注释后确定其降解纤维素的通路,发现有3个基因与纤维素酶的合成有关,使用基因工程技术将其中尚未被报道的基因5676进行克隆,将重组DNA分子导入大肠杆菌BL21(DE3),成功表达出纤维素酶,对利用生物方法降解纤维素提供理论基础,为放线菌降解纤维素在生产生活中的应用有重大意义。
1 材料与方法 1.1 材料 1.1.1 培养基富集培养基(g/L):CMC-Na 5.0,滤纸5.0,NaNO3 2.0,K2HPO4 1.0,KCl 0.5,MgSO4·7H2O 0.5,FeSO4 0.001,pH自然。
筛选鉴定培养基(g/L):CMC-Na 5.0,酵母粉2.0,磷酸二氢钾0.5,硫酸镁0.5,琼脂20.0。
液体产酶鉴定培养基(g/L):CMC-Na 10.0,蛋白胨3.0,酵母粉0.2,硫酸铵2.0,磷酸二氢钾4.0,硫酸镁0.3。
LB液体培养基(g/L):酵母粉5.0,蛋白胨10.0,氯化钠10.0,调pH至7.0。
抗生素浓度:氨苄青霉素(Amp)浓度为100 mg/mL。
1.1.2 主要试剂和仪器PBS缓冲液:磷酸二氢钾0.24 g,磷酸氢二钠1.44 g,氯化钠8 g,氯化钾0.2 g,加去离子水800 mL溶解,调pH至7.2,定容至1 L。
DNS试剂:酒石酸钾钠18.2 g,溶解在50 mL蒸馏水中,加入0.63 g 3, 5-二硝基水杨酸,溶解,再依次加入2 g氢氧化钠、0.5 g苯酚和0.5 g无水硫酸钠,溶解后定容至100 mL,避光保存,放置一周后使用。
1% CMC缓冲液:1 g CMC-Na,溶解在100 mL 50 mmol/L柠檬酸-100 mmol/L磷酸氢二钠缓冲液(pH 6.0)中。
EZ-10柱式细菌基因组DNA抽提试剂盒,生工生物工程(上海)股份有限公司;PCR引物、PCR Mix,pETDuet-1载体,T4 DNA连接酶,宝生物工程(大连)有限公司;琼脂糖凝胶回收试剂盒,天根生化科技有限公司;SDS-PAGE凝胶制备试剂盒,北京索莱宝科技有限公司。
高速冷冻离心机,Eppendorf中国有限公司;PCR仪,北京东胜创新生物科技有限公司;凝胶成像分析系统,济南东仪实验室设备有限公司;超微量分光光度仪,赛默飞世尔科技公司;电泳仪,Bio-Rad公司。
1.2 方法 1.2.1 菌株的筛选土样取自河南师范大学花园土壤,土样采集时选择带有枯枝落叶的表层土。取5 g样品放入200 mL富集培养基,30 ℃条件下180 r/min恒温培养3 d,观察菌体生长情况。采用梯度稀释法将富集培养后的菌液涂布于筛选培养基平板。24 h后观察平板菌落,挑取不同形态、颜色的菌落进行分离及纯化,从而获得纯培养物。
将分离纯化的单菌落接种于LB液体培养基,37 ℃、180 r/min培养2 d后,取1 mL菌液于离心管中,4 000 r/min离心2 min,用液体产酶培养基洗涤2次,4 000 r/min离心2 min去上清。无菌水重悬菌体,取5 μL点在筛选鉴定培养基平板中心,37 ℃培养1 d后观察菌落形态,测量菌落直径,使用克氏碘染液将菌落染色5 min,观察透明圈大小,测量并记录透明圈直径。计算透明圈直径与菌落直径的比值,筛选出比值比较大的菌株。
1.2.2 菌株的鉴定将筛选出的菌株送生工生物工程(上海)股份有限公司进行16S rRNA基因测序。将测序结果与NCBI的GenBank数据库以及EzTaxon数据库进行比对[14],选取出与所测序列相似性较高的种或属,最后再用Neighbor-Joining法构建系统进化树[15]。
1.2.3 菌株Lb1的全基因组学分析对Lb1进行全基因组测序分析[16],送测菌株做3个生物学重复。送检样品通过DNA抽提、纯化、建库和测序,得到一系列下机数据。运用统计学方法对所有测序Reads的每个Cycle进行碱基分布和质量波动的统计,从宏观上反映出测序样本的文库构建质量和测序质量,并分析每一个样本的碱基质量、碱基错误率以及碱基分布。
对Lb1基因进行综合预测,采用GeneMark S-native方法[17]和Heuristic模型参数[18],与KEGG、COG、NR、SwissProt和GO 5个数据库进行比对,得到相应的注释信息。在进行BLAST时需要设置筛选条件:E-value小于10-5,大于40%的序列相似性,大于50%的序列覆盖度。在相同条件下将基因的注释信息进行集合组成,用Circos绘制Lb1的基因图谱。
1.2.4 纤维素酶分泌表达载体的构建根据Streptomyces sp. Lb1基因组序列中5676基因序列设计一对引物5676F (5′-GGAATTCCATATGATGGACGTCAGCAGGGGG-3′)和5676R (5′-GGGGTACCTCACTCGCCGGCCCGGGC-3′),在引物5′加Nde I和Kpn I双酶切位点,片段大小为1 044 bp。以Streptomyces sp. Lb1总DNA为模板对基因5676进行扩增。PCR反应体系:DNA 0.5 μg,Premix Taq 25 μL,正、反向引物各0.2 μmol/L,ddH2O补充至50 μL。PCR反应条件:94 ℃ 5 min;98 ℃ 10 s,55 ℃ 30 s,72 ℃ 1.5 min,30个循环;72 ℃ 10 min,8 ℃保存。PCR产物纯化回收后,用Nde I和Kpn I双酶切,纯化回收片段。然后将pETDuet-1空质粒用同样的酶双酶切,纯化回收后与前面得到的片段连接。连接产物转化E. coli DH5α,转化子经菌落PCR验证,PCR反应体系:rTaq (5 U/μL) 0.25 μL,正、反向引物(同上)各1 μmol/L,10×PCR Buffer (Mg2+ plus) 5 μL,dNTP Mixture (各2.5 mmol/L) 4 μL,无菌牙签挑取菌落混入反应液,ddH2O补充至50 μL。PCR反应条件:94 ℃ 5 min;98 ℃ 10 s,55 ℃ 30 s,72 ℃ 1.5 min,30个循环;72 ℃ 10 min,8 ℃保存。质粒抽提和双酶切验证,获得的重组表达质粒pETDuet-1-5676,并送生工生物工程(上海)股份有限公司测序鉴定。
1.2.5 纤维素酶重组表达菌株的构建挑取LB固体平板上大肠杆菌BL21(DE3)单菌落于LB液体培养基中,37 ℃、180 r/min培养至OD600为0.5,4 ℃、5 000 r/min离心10 min,弃上清,用预冷过的无菌水洗涤2次后加入1.5 mL预冷的CaCl2 (100 mmol/L)重悬后即为感受态细胞[19]。取100 μL感受态细胞加入5 μL重组质粒pETDuet-1-5676 (100 ng/μL),冰浴30 min,42 ℃水浴90 s后冰浴2 min,加入350 μL预热的LB液体培养基37 ℃水浴孵育1 h,吸取100 μL涂布于含有氨苄青霉素抗性平板,37 ℃正置培养1 h后倒置培养12 h,挑取转化子,经质粒抽提酶切验证获得阳性转化子,通过该方法构建重组表达菌株并同时构建空质粒pETDuet-1的对照组菌株。
1.2.6 葡萄糖标准曲线由于纤维素在纤维素酶的作用下生成葡萄糖[20-21],因此测得反应液中葡萄糖即可确定纤维素的酶活力。本研究采用DNS法测定葡萄糖,首先绘制葡萄糖标准曲线:取9支10 mL的EP管,向每支管中分别加入1 mg/mL的葡萄糖标准液、DNS试剂和蒸馏水,配制成不同浓度的葡萄糖反应液。混匀后在沸水浴中加热10 min,然后立即用流动水冷却,分别取200 μL加到96孔板中,用酶标仪测定其在540 nm下的吸光值。以葡萄糖质量为横坐标,吸光值为纵坐标,绘制葡萄糖标准曲线。
1.2.7 纤维素酶活力的测定吸取150 μL CMC缓冲液于4 mL EP管中,加入50 μL产酶液,对照组加空质粒pETDuet-1发酵液。50 ℃水浴保温30 min,结束后加入200 μL DNS终止反应,将EP管中的混合液沸水浴5min,加入650 μL蒸馏水,混匀取200 μL加至96孔板,用酶标仪测定540 nm下的吸光度值,实验组与对照组做3个平行重复取平均值[22]。酶活力定义为:在50 ℃的反应条件下,1 min内催化CMC-Na生成1 μmoL葡萄糖所需的酶量[23]。
2 结果与分析 2.1 菌株的筛选通过对样品进行直接稀释涂布分离筛选及纯化,共获得6株纤维素降解菌(表 1)。这6株菌在筛选鉴定培养基平板上培养1 d后菌落为白色或乳白色;菌落形态均为圆形有光泽不透明。分别测量这6株菌的降解圈和菌落的直径发现,菌株D3的降解圈直径最大为2.7 cm,菌株Lb1的降解圈直径最小为1.4 cm,菌株D3的菌落直径最大为1.90 cm,菌株Lb1的菌落直径最小为0.45 cm。单个菌株的大小或是透明圈的大小不能代表菌株的降解能力,所以用每个菌株的透明圈直径与菌落直径的比值来表示菌株的降解能力。菌株Lb1的直径比值最大为3.10,从而反映出菌株Lb1的降解能力最强,因此选择菌株Lb1作为目标菌株进行后续实验。
| 菌株名称 Strain name | 降解圈直径 Degradation circle diameter (cm) | 菌落直径 Colony diameter (cm) | 降解圈直径/菌落直径 Degradation circle/ Colony diameter |
| D1 | 2.6 | 1.75 | 1.49 |
| D3 | 2.7 | 1.90 | 1.42 |
| D4 | 2.2 | 1.50 | 1.47 |
| H1 | 2.3 | 1.16 | 1.44 |
| H2 | 1.8 | 1.15 | 1.57 |
| Lb1 | 1.4 | 0.45 | 3.10 |
将菌株Lb1测序后的16S rRNA基因序列提交至NCBI的GenBank数据库以及EzTaxon数据库进行比对,结果表明菌株Lb1与Streptomyces属的菌株Streptomyces thermoviolaceus subsp. apingens (DSM41392)有最高相似性(表 2),为99.93%。使用MEGA 5软件构建系统发育树,从进化树中可以看出,菌株Lb1与菌株Streptomyces thermoviolaceus subsp. apingens (DSM41392)在同一分支中(图 1)。因此,根据16S rRNA基因序列、系统进化分析及形态特征,确定菌株Lb1为Streptomyces属,命名为Streptomyces sp. Lb1。
| 种属名 Hit taxon name | 种加词 Hit strain name | 登录号 Accession No. | 相似度 Similarity (%) | 完整度 Completeness (%) |
| Streptomyces thermoviolaceus subsp. apingens | DSM41392 | Z68095 | 99.93 | 100.0 |
| Streptomyces thermoviolaceus subsp. thermoviolaceus | DSM40443 | Z68096 | 99.41 | 100.0 |
| Streptomyces mexicanus | CH-M-1035 | AF441168 | 99.12 | 100.0 |
| Streptomyces chiangmaiensis | TA4-1 | AB562507 | 98.81 | 100.0 |
| Streptomyces chromofuscus | NBRC12851 | AB184194 | 98.60 | 99.8 |
| Streptomyces bullii | C2 | HE591384 | 98.59 | 99.2 |
| Streptomyces glomeratus | LMG19903 | AJ781754 | 98.38 | 100.0 |
| Streptomyces thermoalcalitolerans | NBRC16322 | AB249909 | 98.38 | 100.0 |
| Streptomyces thermocarboxydovorans | DSM44296 | U94489 | 89.29 | 100.0 |
| Streptomyces thermodiastaticus | DSM40573 | Z68101 | 88.59 | 100.0 |

|
| 图 1 菌株Lb1基于16S rRNA基因构建的系统进化树 Figure 1 Phylogenetic tree of strain Lb1 based on 16S rRNA gene sequence 注:步长值通过1 000次重复运算在各分支点显示出来;只保留步长值大于50%. Note: Bootstrap values (expressed as percentages of 1 000 replications) are shown at branch points; Only bootstrap values > 50% are showed. |
|
|
依据测序结果及分析,Lb1的全基因组由一条2 184 066 bp的完整环状染色体组成,基因大小为6 275 290,拼接数284,(G+C)mol%含量占比72.45%,编码序列数量5 806个,KEGG基因注释2 820个,COG基因注释数量为4 285个。
2.3.2 Lb1的GO功能注释Lb1的GO功能注释结果如图 2所示,有2 574个基因有具体的功能定义,共获得了12 956个GO功能注释。在所有GO注释中,有3 847个为分子功能,占28.5%;有3 068个为细胞组件,占29.2%;6 041个为生物过程,占46.6%。所有GO注释的基因可归为40个亚类。与分子相关的基因最多,为44.3%。催化活性(catalytic activity)和蛋白结合(binding)相关的基因数量最多,其比例为71.6%和56.0%,并且有178个基因与转录活性(transporter activity)有关,有165个基因与核酸结合转录因子(nucleic acid binding transcription factor activity)有关,有30个基因与分子传感活性(molecular transducer activity)有关,而与水营养活性(nutrient reservoir activity)有关的最少,只有1个。

|
| 图 2 菌株Lb1的GO功能注释 Figure 2 GO function annotation of strain Lb1 |
|
|
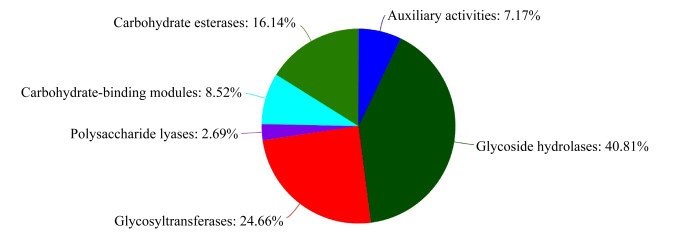
碳水化合物在很多生物学功能中具有重要地位,通过研究碳水化合物相关酶可以得到大量有意义的生物学信息,CAZy数据库专注于分析碳水化合物酶的基因组、结构和生物化学信息。比对CAZy数据库(Carbohydrate Active Enzymes Database)获得碳水化合物活性酶基因注释概况并进行统计。如图 3所示,在菌株Lb1中,比例最高的是水解酶(glycoside hydrolases),占40.81%;其次是糖基转移酶(glycosyltransferases),占24.66%。
|
| 图 3 菌株Lb1的碳水化合物合成酶基因组成 Figure 3 The carbohydrate synthase gene composition of strain Lb1 |
|
|
通过KEGG的计算和推测,获得菌株Lb1的KEGG注释(图 4)。结果显示其基因功能与细胞过程、环境信息处理、遗传信息处理、人类疾病、新陈代谢和生物体系统这6类有关。在代谢通路中发现有39个与菌体生长能力有关的基因,这些基因可映射到5个KEGG pathway通路上,说明这些基因可以在这5个代谢途径上发挥作用。这些基因基本与糖的降解有关,形成一个复杂而又稳定的网络为菌体的生长提供物质和能量,能将来源于细胞外的蔗糖降解为葡萄糖和果糖,将细胞外麦芽糖降解为葡萄糖,这些基因通过产生不同的酶改变糖的构象和种类,最终将多糖和二糖降解成可利用的单糖。降解过程中产生的尿苷二磷酸葡萄糖与氨基糖和核苷酸糖代谢过程互为联系,而产生的腺嘌呤核苷二磷酸葡萄糖与糖异生或糖酵解过程相联系,从而与细胞内的其他过程相联系。

|
| 图 4 菌株Lb1的KEGG通路 Figure 4 KEGG pathway of strain Lb1 |
|
|
从菌株Lb1的KEGG通路中可知,有两条与纤维素降解有关的途径,与纤维素降解有关的基因有3个,如图 4所示,纤维素可以由基因(3.2.1.4)先分解成纤维糊精(cellodextrin),再由β-葡糖苷酶(3.2.1.21)分解成葡萄糖,也可以由基因(3.2.1.91)分解成纤维二糖(cellobiose),最终分解成葡萄糖;纤维素也可以直接由基因(3.2.1.91)或者内切葡聚糖酶(3.2.1.4)分解成纤维二糖,之后再降解成单糖。
如表 3所示,与产纤维素酶有关的基因有3个,其中有两个基因已经被命名为bg1B和cbhA,本研究选取另外一个没有被命名的且标识为5676的基因进行克隆并使其在大肠杆菌中得到表达。
| 基因 Gene | 基因名称 Gene name | 基因长度 Geng long (bp) | 基因标识 Gene ID | 注释 Defination | 与基因5676相似度 Similar to gene 5676 (%) |
| 3.2.1.4 | - | 1 044 | Gene5676 | Engoglucanase | 100.00 |
| 3.2.1.21 | bg1B | 1 359 | Gene0220 | Beta-glucosidase | 48.09 |
| 3.2.1.91 | cbhA | 1 341 | Gene1760 | Cellose 1, 4-beta-cellobiosidase | 46.97 |
| 注:-:未命名的基因名. Note: -: The name of the unnamed gene. | |||||
通过构建pETDuet-1纤维素酶重组表达载体pETDuet-1-5676,转化到大肠杆菌DH5α,挑选克隆子在氨苄青霉素抗性培养基培养过夜,抽提质粒酶切验证结果如图 5所示。重组质粒经过Nde I和Kpn I双酶切后,经1%琼脂糖凝胶电泳结果显示得到两条片段大约为5 366 bp和1 044 bp,通过对重组表达质粒测序,验证连接到载体的基因与目的基因一致,说明重组表达载体构建成功。

|
| 图 5 重组表达质粒pETDuet-1-5676酶切验证 Figure 5 Restriction analysis of expression plasmid pETDuet-1-5676 注:1:重组质粒;2:经Nde I和Kpn I双酶切后的重组质粒;M:1 kb plus DNA ladder. Note: 1: Recombinant plasmid; 2: Recombinant plasmid digested by Nde I and Kpn I; M: 1 kb plus DNA ladder. |
|
|
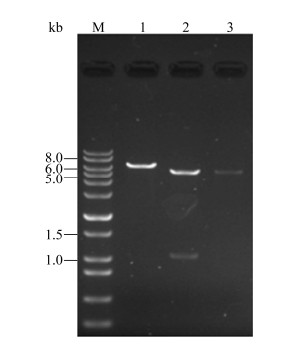
将构建好的纤维素表达载体转入BL21(DE3),得到表达菌株BL21(5676),将空质粒pETDuet-1转入BL21(DE3)得到对照菌株BL21(pETDuet-1),经液体培养抽提质粒酶切验证,结果如图 6所示,抽提重组质粒后测序,目的片段与目的基因一致,表明纤维素酶表达菌种构建成功。
|
| 图 6 表达菌株抽提重组质粒酶切验证 Figure 6 Restriction analysis of expression vector in expression of strain 注:M:1 kb plus DNA ladder;1:经单酶切后的重组质粒;2:经Nde I和Kpn I双酶切后的重组质粒;3:经单酶切后的空质粒. Note:M: 1 kb plus DNA ladder; 1: Recombinant plasmid digested by Nde I and Kpn I; 2: Recombinant plasmid digested by Nde I; 3: Empty plasmid digested by Nde I. |
|
|
配制浓度范围在0-1.6 mg/mL的葡萄糖标准液,加入DNS试剂充分混匀后沸水浴10 min,用酶标仪测定其在540 nm下的吸光值制作标准曲线,根据标准曲线计算线性回归方程为y=2.150x-0.057,相关系数R2=0.972 13。分别取表达菌株发酵液和空载菌株发酵液加入到CMC缓冲液中,使用DNS终止法测定发酵液酶活性,实验组和对照组做3个平行重复,取平均值,结果如图 7所示。

|
| 图 7 重组菌株的纤维素酶活性 Figure 7 Cellulase activity of recombinant strain 注:所有的误差棒代表 3个生物学重复. ***:P < 0.001. Note: All error bars indicate the SE of the three biological repeats. ***: P < 0.001. |
|
|
可以看到对于纤维素降解生成葡萄糖,表达菌株相较于空载菌株有较为明显(P < 0.001)的促进纤维素生成葡萄糖的作用,因此可以判断出表达菌株生成有纤维素酶。
3 讨论目前所发现和研究的降解纤维素的微生物主要为真菌,真菌产纤维素酶较细菌活性高,但发酵过程不易控制且发酵时间过长,所以克隆真菌中产纤维素酶基因使其在原核生物如大肠杆菌中进行表达也是热点研究[24-26]。近年来有学者对真菌-细菌联合使用降解纤维素生成乳酸进行初步研究[27]。本研究分离筛选出一株具有降解纤维素作用的放线菌,并将纤维素酶合成基因在大肠杆菌中表达。对于控制生成纤维素酶的基因的研究主要在于基因bg1B和基因cbhA,本研究首次验证了基因5676产纤维素酶的功能并使其在细菌中表达,为微生物降解纤维素的理论和应用提供新的渠道。
4 结论本研究首先筛选分离出一株具有降解纤维素功能的菌株Lb1,鉴定证实属于链霉菌属,命名为Streptomyces sp. Lb1。通过对Lb1的全基因组测序分析,发现其有3个与纤维素酶表达相关的基因,选取基因5676,构建出纤维素酶表达载体,实现了纤维素酶在大肠杆菌BL21(DE3)中的表达。通过对表达菌株发酵液降解纤维素并对其产物葡萄糖进行测定,其降解纤维素的能力要明显高于空质粒菌株发酵液。未来对其的后续研究可重点放在发酵工艺的优化,使得纤维素酶在菌株中得到更高量的表达,为其在工业中的应用打下良好的基础。
| [1] |
Gilbert HJ. The biochemistry and structural biology of plant cell wall deconstruction[J]. Plant Physiology, 2010, 153(2): 444-455. |
| [2] |
Payne CM, Knott BC, Mayes HB, et al. Fungal cellulases[J]. Chemical Reviews, 2015, 115(3): 1308-1448. |
| [3] |
Ang SK, Shaza EM, Adibah Y, et al. Production of cellulases and xylanase by Aspergillus fumigatus SK1 using untreated oil palm trunk through solid state fermentation[J]. Process Biochemistry, 2013, 48(9): 1293-1302. |
| [4] |
Leonowicz A, Matuszewska A, Luterek J, et al. Biodegradation of lignin by white rot fungi[J]. Fungal Genetics and Biology, 1999, 27(2/3): 175-185. |
| [5] |
Zhang XH, Hong CL, Zhu FX, et al. Current situation and prospect of utilization of agricultural waste resources[J]. Modern Agricultural Technology, 2013(20): 209, 218. (in Chinese) 张俙何, 洪春来, 朱凤香, 等. 农业废弃物资源化利用现状与前景展望[J]. 现代农业科技, 2013(20): 209, 218. |
| [6] |
Ma H, Liu WW, Chen X, et al. Enhanced enzymatic saccharification of rice straw by microwave pretreatment[J]. Bioresource Technology, 2009, 100(3): 1279-1284. |
| [7] |
Sun JS, Su DH, Liu L. Pretreatment technology of corn Stover for ethanol production[J]. Progress in Chemistry, 2007, 19(7): 1122-1128. (in Chinese) 孙君社, 苏东海, 刘莉. 秸秆生产乙醇预处理关键技术[J]. 化学进展, 2007, 19(7): 1122-1128. |
| [8] |
Annamalai N, Rajeswari MV, Elayaraja S, et al. Thermostable, haloalkaline cellulase from Bacillus halodurans CAS 1 by conversion of lignocellulosic wastes[J]. Carbohydrate Polymers, 2013, 94(1): 409-415. |
| [9] |
Zhang YHP, Himmel ME, Mielenz JR. Outlook for cellulase improvement: screening and selection strategies[J]. Biotechnology Advances, 2006, 24(5): 452-481. |
| [10] |
Zheng W, Zheng Q, Xue YY, et al. Influence of rice straw polyphenols on cellulase production by Trichoderma reesei[J]. Journal of Bioscience and Bioengineering, 2017, 123(6): 731-738. |
| [11] |
Libardi N, Soccol CR, Góes-Neto A, et al. Domestic wastewater as substrate for cellulase production by Trichoderma harzianum[J]. Process Biochemistry, 2017, 57: 190-199. |
| [12] |
Gu FY, Chen CY, Shi JJ, et al. Advances in cellulase and its development tendency[J]. Journal of Microbiology, 2008, 28(1): 83-87. (in Chinese) 顾方媛, 陈朝银, 石家骥, 等. 纤维素酶的研究进展与发展趋势[J]. 微生物学杂志, 2008, 28(1): 83-87. |
| [13] |
Dong XQ, Yang JS, Zhu N, et al. Sugarcane bagasse degradation and characterization of three white-rot fungi[J]. Bioresource Technology, 2013, 131: 443-451. |
| [14] |
Chen MH, Sheu SY, Chen CA, et al. Corallomonas stylophorae gen. nov., sp. nov., a halophilic bacterium isolated from the reef-building coral Stylophora pistillata[J]. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(3): 982-988. |
| [15] |
Tamura K, Dudley J, Nei M, et al. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24(8): 1596-1599. |
| [16] |
Ng PC, Kirkness EF. Whole genome sequencing[A]//Barnes MR, Breen G. Genetic Variation: Methods and Protocols[M]. Totowa, NJ: Humana Press, 2010: 215-226
|
| [17] |
Besemer J, Lomsadze A, Borodovsky M. GeneMarkS: a self-training method for prediction of gene starts in microbial genomes. Implications for finding sequence motifs in regulatory regions[J]. Nucleic Acids Research, 2001, 29(12): 2607-2618. |
| [18] |
Claverie JM, Bougueleret L. Heuristic informational analysis of sequences[J]. Nucleic Acids Research, 1986, 14(1): 179-196. |
| [19] |
Yun CH, Bae CS, Ahn T. Transformation of Escherichia coli and protein expression using lipoplex mimicry[J]. Protein Expression and Purification, 2016, 127: 68-72. |
| [20] |
Wood TM, Bhat KM. Methods for measuring cellulase activities[J]. Methods in Enzymology, 1988, 160: 87-112. |
| [21] |
Eveleigh DE, Mandels M, Andreotti R, et al. Measurement of saccharifying cellulase[J]. Biotechnology for Biofuels, 2009, 2: 21. |
| [22] |
Fu L, Ding YF, Zhang C. Studies on methods for determination of cellulase activity[J]. Journal of Xinjiang Agricultural University, 2000, 23(2): 45-48. (in Chinese) 傅力, 丁友昉, 张篪. 纤维素酶测定方法的研究[J]. 新疆农业大学学报, 2000, 23(2): 45-48. |
| [23] |
Lineweaver H, Burk D. The determination of enzyme dissociation constants[J]. Journal of the American Chemical Society, 1934, 56(3): 658-666. |
| [24] |
Zhang CF, Gu WJ, Peng KF, et al. Present situation of research on microbial cellulase[J]. China Journal of Bioinformatics, 2007, 5(1): 34-36. (in Chinese) 张传富, 顾文杰, 彭科峰, 等. 微生物纤维素酶的研究现状[J]. 生物信息学, 2007, 5(1): 34-36. |
| [25] |
Lin MI, Nagata T, Katahira M. High yield production of fungal manganese peroxidases by E. coli through soluble expression, and examination of the activities[J]. Protein Expression and Purification, 2018, 145: 45-52. |
| [26] |
Bai HZ, Zi H, Huang YQ, et al. Catalytic properties of carboxymethyl cellulase produced from newly isolated novel Fungus Penicillium ochrochloron ZH1 in submerged fermentation[J]. Catalysis Letters, 2017, 147(8): 2013-2022. |
| [27] |
Shahab RL, Luterbacher JS, Brethauer S, et al. Consolidated bioprocessing of lignocellulosic biomass to lactic acid by a synthetic fungal-bacterial consortium[J]. Biotechnology and Bioengineering, 2018, 115(5): 1207-1215. |
 2020, Vol. 47
2020, Vol. 47