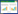扩展功能
文章信息
- 张雨欣, 罗玮, 俞勇, 曾胤新, 赵文玉, 李会荣
- ZHANG Yu-Xin, LUO Wei, YU Yong, ZENG Yin-Xin, ZHAO Wen-Yu, LI Hui-Rong
- 南极恩克斯堡岛土壤中可培养细菌多样性
- Culturable bacterial diversity in soils of Inexpressible Island, Antarctica
- 微生物学通报, 2020, 47(6): 1709-1720
- Microbiology China, 2020, 47(6): 1709-1720
- DOI: 10.13344/j.microbiol.china.191043
-
文章历史
- 收稿日期: 2019-12-13
- 接受日期: 2020-03-24
- 网络首发日期: 2020-04-16
2. 自然资源部中国极地研究中心 上海 200136;
3. 长沙理工大学化学与食品工程学院 湖南 长沙 410114
2. Polar Research Institute of China, Ministry of Natural Resources, Shanghai 200136, China;
3. School of Chemistry and Food Engineering, Changsha University of Science & Technology, Changsha, Hunan 410114, China
长期以来,南极地区一直被认为是地处偏远并保持着原始纯净的环境[1]。该环境的典型特征是极度寒冷、干旱、大幅度温度波动、反复冻融和土壤含盐量高等,这些恶劣的条件对动植物的生存极为不利[2]。一般来说,极端环境和简单的食物网会造成生物量和生物多样性降低[3],但近年来许多研究表明在南极冰层、土壤或湖水等生境发现的生物多样性和丰富度都超出预期[4-5],而且在极地发现的许多微生物由于特殊的环境因素都具有一些独特的性质。
南极恩克斯堡岛(Inexpressible Island)又称难言岛,地处维多利亚地(Victoria Land)北部,罗斯海特拉诺瓦湾南部,面积约50 km2,呈棱形从北向南延伸,基部的侵入岩类型主要是石英二长岩及石英二长闪长岩[6]。维多利亚地的土壤类型多样,整个地区的纬度梯度为8°,处于极地高原和海岸之间,该地土壤受到温度变化和降水的影响。维多利亚地北部低海拔海岸的土壤比南部干谷地更为发达,具有广泛的苔藓层,有海盐输入以及海鸟和企鹅产生的粪便,因此该地区土壤中有机质含量高[7-8]。海岸地区的粪土类型与大陆其他矿质土壤有区别,微生物群落结构和丰度都存在差异[9]。
尽管从环境中通过纯培养可获得的微生物少于1%[10],但对于微生物菌株资源的开发利用来说,分离培养技术是不可或缺的。如研究东南极菲尔德斯半岛和北极苔原等地土壤可培养细菌多样性时,通过直接稀释土壤样品涂布多种不同培养基平板,或对样品进行不同前处理等方法,获得了一定数量的潜在新种,也提供了筛选出具有新活性物质菌株的可能[11-12]。同时,可分离培养的细菌在一定程度上反映了微生物分布具有普遍性。为了获得更多的可培养菌株,研究者们尝试了不同的培养方法,如微生物富集培养、模拟微生物生长的天然环境的原位培养等[13-14]。
不同区域之间和区域内的细菌群落存在差异,随气候和土壤理化条件改变都会发生变化。对于极地还有许多未知的微生物等待发掘,而对维多利亚地区土壤微生物群落多样性的研究主要运用了分子生物学方法。Lee等[15]运用454焦磷酸测序方法研究麦克多干谷地区的微生物群落组成,结果显示细菌在门水平上主要包括Actinobacteria、Proteobacteria、Acidobacteria、Firmicutes、Deinococcus-Thermus、Bacteroidetes和Gemmatimonadetes等;Niederberger等[3]运用限制性内切酶片段长度多态性(restriction fragment length polymorphism,RFLP)分析方法分析维多利亚地北部4个不同地方土壤中微生物群落组成,构建16S rRNA基因克隆文库发现Deinococcus- Thermus和Bacteroidetes主要在干燥、低营养度土壤中存在,而Verrucomicrobia、Gammaproteobacteria中黄单胞菌属仅在高湿度土壤中发现;Aislabie等[9]研究罗斯海地区鸟类粪土细菌群落组成,通过细菌16S rRNA基因的RFLP分析方法发现,企鹅聚集地的土壤中是Firmicutes、Gammaproteobacteria中的Psychrobacter为优势类群,而Actinobacteria和Gammaproteobacteria中的Rhodanobacter、Dokdonella等属在非企鹅聚集地占主导地位。
本文以传统培养方法为主,采用不同培养方法对来自南极恩克斯堡岛的5个土壤样本进行细菌分离培养,以获得更多分离株,并通过16S rRNA基因序列分析获得可分离株多样性,旨在增强对南极地区微生物多样性的认识,同时也为开发和利用新的菌种资源提供依据。
1 材料与方法 1.1 样品采集中国第34次南极科学考察期间,5份土壤样品采集自恩克斯堡岛(74.83°–70.95°S,163.58°–163.76°E),位于维多利亚地北部、特拉诺瓦湾南部。在5个采样点分别用无菌小铲除去表层土壤及小石子,挖取50-100 g样品装入无菌采样袋中,冷冻黑暗运输至国内实验室,实验前存储在-20 ℃冰箱。采集点位置、采样时间及理化性质见表 1。
| 样品 Sample | 采样日期 Date | 地理位置 Geographical coordinates | 土壤类型 Soil type | 湿度 Moisture (%) | 总有机碳 TOC (μg/g) | 总有机氮 TON (μg/g) | 铵盐 NH4+-N (μg/g) | 硅酸盐 SiO42--Si (μg/g) | 硝酸盐 NO3--N (μg/g) | 磷酸盐 PO43--P (μg/g) | 亚硝酸盐 NO2--N (μg/g) | pH |
| InS2 | 20180201 | 74.901 81°S, 163.724 23°E | Penguin activity area | 0.60 | 12.246 | 0.006 6 | 138.212 06 | 14.050 4 | 4.099 06 | 4.850 26 | 3.263 4 | 6.35 |
| InS3 | 20180201 | 74.909 88°S, 163.714 52°E | Vegetation coverage | 4.00 | 0.132 4 | 0.026 | 83.835 08 | 3.941 84 | 0.841 26 | 37.702 51 | 0.344 4 | 7.04 |
| InS4 | 20180201 | 74.896 71°S, 163.724 74°E | Freshwater lake | 2.20 | 0.573 6 | 0.044 5 | 98.371 14 | 4.341 68 | 4.219 6 | 31.445 47 | 0.086 66 | 6.81 |
| InS5 | 20180201 | 74.906 83°S, 163.727 04°E | Typical geological landform | 1.00 | 0.105 7 | 0.019 7 | 49.144 9 | 4.260 76 | 0.782 46 | 29.437 29 | 0.234 78 | 7.35 |
| InS6 | 20180201 | 74.915 78°S, 163.710 59°E | Rare biological background | 0.00 | 0.154 8 | 0.023 5 | 17.958 64 | 5.902 68 | 0.601 72 | 16.788 67 | 0.073 92 | 7.71 |
16S rRNA基因扩增的引物、酶和试剂,生工生物工程(上海)股份有限公司;细菌基因组提取试剂盒、普通琼脂糖凝胶回收试剂盒,天根生化科技(北京)有限公司。PCR仪,赛默飞世尔科技(中国)有限公司;旋涡振荡器,Scientific Industries公司;水平电泳仪,Bio-Rad公司。
参照文献配制NA培养基[16]、R2A培养基[17]和2216E培养基[18],1/10 NA和1/10 2216E培养基是将上述培养基成分减量为1/10配制。富集液配方[19] (g/L):蛋白胨0.2,酵母粉0.2,NH4Cl 1.0,MgSO4·H2O 0.2,CH3COONa 2.0,丙酮酸钠1.25,EDTA 1.0,KH2PO4 0.2,NaHCO3 1.0,去离子水1 L。
1.3 样品分离培养方法 1.3.1 直接涂布培养无菌条件下,称取1 g土壤样品加入分装有9 mL无菌生理盐水的试管中,在旋涡振荡器上充分振荡5 min,使试管中的土壤不沉在底部呈混匀状态,系列稀释至10–1、10–2、10–3三个浓度梯度,每个梯度分别取100 μL涂布到1.2中的5种培养基平板上。置于15 ℃培养箱培养15 d,挑取菌落形态有差异的单菌落进行平板划线纯化。
1.3.2 微好氧富集后涂布培养称取5 g土壤,加入装有2/3灭菌富集液的100 mL蓝盖瓶中,用灭菌富集液将剩余1/3体积补满,形成微厌氧环境。15 ℃静置富集培养,15、30、45和60 d后分别开瓶,取1 mL培养物系列稀释至10–1、10–2、10–3三个梯度后涂布于R2A培养基平板。置于15 ℃培养7–15 d,挑取形态有差异菌落在R2A平板上进行划线纯化。
1.3.3 有氧富集涂布培养称取3 g土壤样品,加入装有100 mL无菌富集液的300 mL锥形瓶中,有氧条件下在15 ℃、150 r/min摇床培养,10、20、30 d后分别开瓶,取1 mL培养物稀释至10–1、10–2、10–3三个浓度梯度后涂布于R2A培养基平板,置于15 ℃培养7–15 d,挑取形态有差异菌落在R2A平板上进行划线纯化。
1.4 菌株保藏、细菌16S rRNA基因测序及其分析 1.4.1 菌株纯化及鉴定对于在15 ℃培养箱中培养后平板上长出的细菌菌落,根据菌落大小、颜色和形态等特征进行描述,挑取形态差异较大的菌落进行划线纯化、编号。将纯化后的单菌落接种至液体培养基,15 ℃、150 r/min培养5-7 d后,取菌液保存在20%甘油管中,保藏于-20 ℃。同时取菌液约20 µL于0.2 mL离心管中,煮沸10 min后,25 ℃下5 000 r/min离心3 min,取上清2 µL作为PCR模板;无法煮沸获得菌体DNA的菌株培养物,采用小量细菌基因组快速提取试剂盒提取细菌DNA,取1-2 μL作为模板,对细菌16S rRNA基因进行PCR扩增。16S rRNA基因通用引物为27F (5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R (5′-GGTTACCTTGTTACGCTT-3′)[20]。PCR反应体系(25 μL):10×Buffer 2.5 μL,dNTPs Mix (2.5 mmol/L) 0.5 μL,引物27F和1492R (10 μmol/L)各0.5 μL,BSA (20 mg/mL) 0.5 μL,rTaq酶(5 U/μL) 0.5 μL,DNA模板2 μL,无菌去离子水18 μL。PCR反应条件:95 ℃ 5 min;94 ℃ 45 s,55 ℃ 45 s,72 ℃ 2 min,30个循环;72 ℃ 10 min。PCR产物经胶回收试剂盒纯化后,连接到pMD18-T载体上,将连接产物转化至大肠杆菌DH5α感受态细胞,经复苏培养后涂布至含100 μg/mL氨苄青霉素的LB培养基上,37 ℃培养过夜,挑取白色克隆子,用T载体通用引物M13+ (5′-GTAAAACGACG GCCAG-3′)和M13- (5′-CAGGAAACAGCTATGA C-3′)检测得到阳性克隆。4 ℃保存,送至生工生物工程(上海)股份有限公司测序。
1.4.2 系统发育分析将所测定的16S rRNA基因序列在EzBioCloud (https://www.ezbiocloud.net/)比对,获得相似性较高的相关模式菌株16S rRNA基因序列,使用ClusterX 1.83和MEGA 6.0分析并构建系统发育树。将分离菌株的16S rRNA基因序列批量提交上传至NCBI数据库,每个样品中分离得到的重复菌株仅选取一个序列上传,获得登录号为MN315387-MN315460。
2 结果与分析 2.1 可培养细菌的多样性及系统发育分析前期实验采用直接涂布法,挑取平板上颜色、形状、透明度等方面有差异的菌落,进行划线纯化及测序,发现在3种平板上分离得到的菌属个数为R2A (14个) > NA (10个) > 2216E (5个),后期富集培养涂布选用R2A平板。
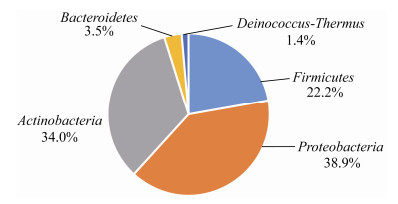
5份土壤样品中共分离得到144株可培养细菌,对分离菌株16S rRNA基因进行PCR扩增、测序和序列比对,发现这些菌株来自5个门,分布在30个属52个种(表 2)。如图 1显示,最多的是变形菌门56株,达到总分离株的38.9%;其次是放线菌门49株,占比34.0%;厚壁菌门32株,占总数的22.2%;异常球菌-栖热菌门5株,占总数的3.5%;拟杆菌门最少,仅有2株,占总数的1.4%。将16S rRNA基因测序结果在EzTaxon数据库中比对获得参考序列,应用MEGA 6.0软件绘制系统发育树,得到进化树如图 2所示。从系统发育分析的结果可以看出,放线菌门中最多的是链霉菌属(Streptomyces) 14株,其次是节杆菌属(Arthrobacter) 7株,最少的类谷氨酸杆菌属(Paeniglutamicibacter)、库克菌属(Kocuria)和韩国生工属(Kribbella)各1株。变形菌门中分别有α、β和γ三个亚门,α变形菌亚门最多的是Aureimonas,有7株;β变形菌类只有贪噬菌属(Variovorax);γ变形菌亚门中最多的是嗜冷杆菌属(Psychrobacter) (22株),其次是假单胞菌属(Pseudomonas) (15株)。对144株菌株测序时发现,有2株菌(1株属于变形菌门,1株属于放线菌门)的16S rRNA基因序列相似度均小于97%,可以认为是潜在新种[21]。这两株潜在新种的菌株鉴定工作正在进行中。
| 样品 Sample | 分离株数目 The number of isolates | 属/种数目 The number of genera/species | 潜在新种数目 The number of potential novel strains |
| InS2 | 20 | 10/14 | 1 |
| InS3 | 46 | 16/18 | 0 |
| InS4 | 36 | 12/16 | 0 |
| InS5 | 20 | 13/15 | 0 |
| InS6 | 22 | 9/10 | 1 |
|
| 图 1 分离菌株在门水平上分布 Figure 1 The distribution of bacterial isolates at phylum level |
|
|
|
| 图 2 分离菌株16S rRNA基因序列与标准株序列采用邻接法构建的系统发育树 Figure 2 Phylogenetic dendrogram of bacterial 16S rRNA gene sequences were inferred using neighbor-joining analysis 注:括号中是16S rRNA基因序登录号;斜线后是在属水平分类上相同的分离株数目;分支上数字表示Bootstrap值;标尺表示每个核苷酸序列位置中碱基替换数. A:放线菌门、厚壁菌门、拟杆菌门和异常球菌-栖热菌门;B:变形菌门. Note: GenBank accession numbers of 16S rRNA gene sequences are given in the parentheses; The number of the same strains at genus level are given after slash; Numbers at the branch points indicated the bootstrap value; Scale bar indicates the estimated number of base changes per nucleotide sequence position. A: Actinobacteria, Firmicutes, Bacteroidetes and Deinococcus-Thermus; B: Proteobacteria. |
|
|
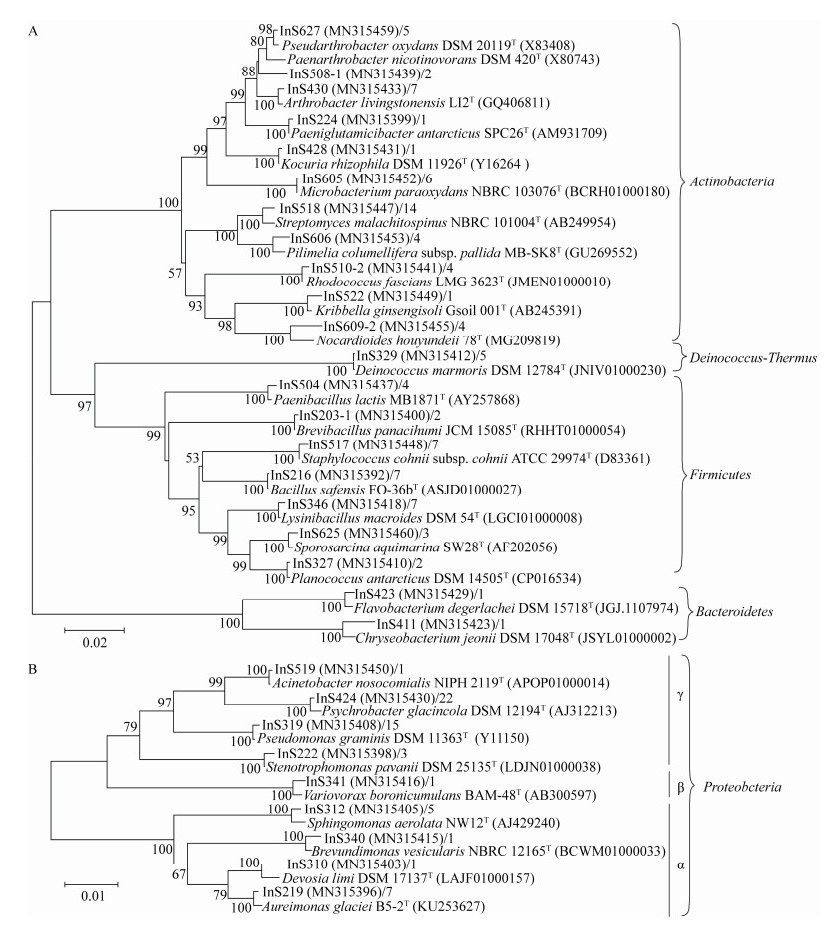
为了使可培养细菌的多样性增加,本实验用了3种方法,分别是直接涂布培养、微好氧富集和有氧富集在不同时间段涂布培养。3种方式在R2A平板上分离到的可培养细菌的种类有区别。直接涂布法分离得到11个属,独有7个属;有氧富集分离得到9个属,独有5个属;微好氧富集独有2个属,分别是Paeniglutamicibacter和Acinetobacter。上述结果说明在R2A平板上,有氧富集和无氧富集均获得特有属,有氧富集涂布与直接涂布法共有的属4个,包括Staphylococcus、Rhodococcus、Pseudarthrobacte和Arthrobacter,微好氧富集获得可培养细菌种类少(图 3)。
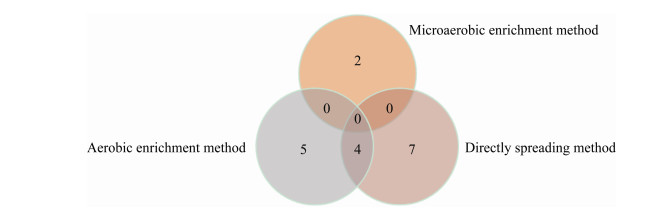
|
| 图 3 3种培养方法在R2A培养基上得到细菌在属水平数目韦恩图 Figure 3 Venn diagram of the number of bacterial genera among three culture methods using R2A medium |
|
|
在门水平上,所有菌株中变形菌门细菌最多(38.9%),其次是放线菌(34.0%)。拟杆菌门细菌仅在InS4样品中出现,InS6样品中没有分离得到异常球菌-栖热菌门(图 4)。

|
| 图 4 可培养细菌在门水平上分布柱状图 Figure 4 The community structure of isolated bacteria in 5 different soils at phylum level |
|
|
在属水平上,5个样品分离培养出的菌株有30个属,其中占比最大的属为γ变形菌亚门的Psychrobacter,在样品InS2、InS3、InS4中均分离得到,而且是InS4中的优势类群。厚壁菌门的Bacillus、Lysinibacillus和γ变形菌门中的Pseudomonas及异常球菌-栖热菌门的Deinococcus在4个样品中都有分布。
从图 5可以看到,不同采样地分离得到的可培养细菌在属水平上有差异。InS2采样点是典型动物企鹅的活动区域,土壤中有企鹅粪便,碳和氮素含量都很高。Bacillus是其优势属,Stenotrophomonas和Sphingomonas也较多,其中Stenotrophomonas pavanii和Paeniglutamicibacter antarcticus是该样品中的特有种类。InS3样品在植被覆盖区域,分离出的菌株中变形菌门的细菌较多,达到48.9%,分离得到的细菌多样性在5个样品中最好,分布在16个属,该样品中的优势属是Streptomyces和Pseudomonas,主要是Pseudomonas graminis和Streptomyces fildesensis。InS4样品来自淡水湖边缘的土壤,土壤较为湿润,Chryseobacterium、Flavobacterium和Kocuria是该样品的特有属,包括Flavobacterium degerlachei、Chryseobacterium jeonii和Kocuria rhizophila菌。InS5样品采集于典型的地质地貌区,分离菌株分布在12个属,特有属有Acinetobacter和Kribbella,分别是Acinetobacter nosocomialis和Kribbella ginsengisoli菌。InS6样品的采样点极其干燥,是生物稀少的本底区,样品中有较多砂砾,分离菌株中的放线菌门细菌丰度很高,约占72.7%。Microbacterium、Pilimelia、Nocardioides在InS6样品中分离株较多,主要有Pilimelia columellifera subsp. pallida、Microbacterium paraoxydans和Nocardioides houyundeii。

|
| 图 5 可培养细菌在属水平上的分布柱状图 Figure 5 The community structure of isolated bacteria at genus level 注:A:厚壁菌门、异常球菌-栖热菌门和放线菌门中的属;B:变形菌门和拟杆菌门中的属. Note: A: The genus in Firmicutes, Deinococcus-Thermus and Actinobacteria; B: The genus in Proteobacteria and Bacteroidetes. |
|
|
本研究对来自南极维多利亚地北部恩克斯堡岛的5个土壤样品主要进行了可培养细菌分离培养,进一步了解维多利亚地在不同地区土壤微生物多样性。我们从所有样品中共分离得到144株细菌,经16S rRNA基因序列测定比对后发现,这些菌株分布在Actinobacteria、Proteobacteria、Firmicutes、Bacteroidetes和Deinococcus-Thermus,这与以往对南极维多利亚地的不同性质土壤微生物多样性研究结果基本一致。Adams等[22]对维多利亚地不同类型土壤中细菌进行了分类,他们发现干冷地带的矿质土在门水平上主要是Actinobacteria、Proteobacteria、Firmicutes和Bacteroidetes,而鸟类粪土中主要是Firmicutes。Aislabie等[23]对来自维多利亚地2个不同区域的4个土壤样品运用分离培养和分子生物学方法研究土壤中的优势细菌,其样品含水量低,总碳和氮量均不高,磷含量较高,他们的分离培养结果显示土壤中主要有Bacteroidetes、Actinobacteria和Proteobacteria门类。我们的实验结果也显示出不同类型土壤在门、属水平上组成有差异,企鹅粪土土壤中主要是Firmicutes和Proteobacteria,在含水量较高的土壤中主要是Proteobacteria,而干燥的土壤中主要是Actinobacteria。土壤中Proteobacteria较多可能是其中有许多属类可以利用不同有机物。
本研究结果显示不同样品之间在属水平上有一定差异。InS2样品采于企鹅活动区域,该样品地土壤呈酸性,总碳量和NH4+-N含量在所有样品中最高。企鹅等海鸟类的粪便不仅是维持该地有机碳和氮含量高的原因之一,还会使土壤的pH值降低[24]。从InS2样品中分离得到的菌株中Firmicutes和Proteobacteria门类占比较大,其中相对优势属是厚壁菌门中的Bacillus,因为芽孢杆菌可以产生抵御环境压力的芽孢,可以存在于动物肠道中,在鸟类粪土中且有机质丰富的地方这类细菌更容易出现[9, 25]。此外,该样品中分离出的Gammaproteobacteria是变形菌门中的主要类群,Stenotrophomonas、Pseudomonas、Psychrobacter均有检出,其中Stenotrophomonas是该样品的特有属。Stenotrophomonas pavanii在以往研究中被发现是内生固氮菌[26]。用有氧富集法分离得到Paeniglutamicibacter antarcticus,该菌属于严格需氧类型,在南极海洋沉积物中被分离出[27]。InS3样品来自于植物覆盖区域土壤,土壤湿度达到4%,氮和磷元素都比较丰富,这两种元素在植物体内参与合成多种化合物。该样品中分离得到细菌的优势属是变形菌门中的Pseudomonas和放线菌门中Streptomyces。Pseudomonas属类中有常见的根际植物促生菌,还有的具有较强的解磷能力,促进植被吸收磷养分,有利于植物生长,对于P元素在植物土壤生态的循环也承担了一定作用[28-29]。在InS3样品分离得到的Pseudomonas属类中,有较多的菌是Pseudomonas graminis,该菌曾在草场中被分离出[30],而Streptomyces属中主要分离出的是Streptomyces fildesensis,该菌也曾在南极土壤中被分离得到[31],而且链霉菌作为放线菌,大多可以产抗生素,对提高植物抗病性等方面都有一定作用[32],可见土壤中优势菌群与土壤类型有关。InS4样品是湖水边缘土壤,该样品中的优势属为Psychrobacter,特有属是Chryseobacterium、Flavobacterium等。Psychrobacter是一种在极冷环境中常见的细菌,在极地土壤、冰层等地均有发现[33-34]。Flavobacterium degerlachei、Chryseobacterium jeonii属于Cytophaga-Flavobacterium-Bacteroides (CFB)菌群(现为Bacteroidetes),在淡水和海洋系统中都非常丰富,有研究认为该类细菌在寒冷水体中对有机物有吸收或降解作用[35-36],土壤中发现的这类细菌可能来自于湖水。InS5样品土壤采于典型地貌区,InS6样品来自于生物稀少的本底区,这两个土壤样品pH均偏碱性,土壤湿度低,有机质含量也较低,InS6样品极其干燥,含水量为零。两个样品分离出的细菌中放线菌门占比大,有研究表明放线菌门在干燥和低营养的土壤中占据优势[37]。两个样品中均分离出Pseudarthrobacter,该属类具有耐干燥特点[38-39]。
在本研究得到的分离可培养细菌中有两株潜在新种,一株为Sphingomonas sp. (菌株名称为InS209-1,登录号MN315391)属于变形菌门,另一株Nocardioides sp. (菌株名称为InS609-2,登录号MN315455)属于放线菌门。Sphingomonas这类耐冷细菌在南极空气中、冻土区均被分离出,在降解有机物污染方面已被研究[40-42]。放线菌门细菌能够在极地的干燥、寒冷土壤中存在,Nocardioides属类细菌在治理环境污染方面也有应用[43-44]。极地的特殊环境赋予了微生物多种特性,还有发现新种的潜力,有许多微生物资源等待挖掘。
5个样品采样地不同,从培养结果可以发现其群落结构有一定差异,尤其是InS2样品与其他样品的区别更大,这与采样地环境有关系。本实验采用分离培养方法分析南极恩克斯堡岛中细菌多样性,可培养细菌在一定程度上反映了该地的优势菌群,而且在分离得到的菌株中放线菌占总分离株的34.0%。放线菌是产生抗生素的重要来源,不同地理位置和土壤条件都可能使放线菌有更多的理化性质。本实验中的一株属于放线菌的潜在新种InS609-2,在新种鉴定过程中发现,该菌株对一定浓度的青霉素、卡那霉素、氨苄西林、四环素和氯霉素等均有抗药性。其他分离菌株待进一步做抗菌活性测定,在南极恩克斯堡岛土壤中放线菌占比大因而具有潜在研究价值。实验中采用的培养方法和条件、人为操作等因素都会影响可培养细菌的分离结果,由于分离培养方法相比非培养方法具有很大的局限性,后续对于极地土壤的细菌分离也可考虑采用改良寡营养培养基等,用更低温条件以获得更多菌种资源。
致谢: 感谢中国第34次南极科学考察队。其中中国科技大学高月嵩博士执行了现场采样,自然资源部第一海洋研究所王能飞副研究员提供了土壤理化参数,在此特别致谢。| [1] |
Wall DH. Biodiversity and ecosystem functioning in terrestrial habitats of Antarctica[J]. Antarctic Science, 2005, 17(4): 523-531. DOI:10.1017/S0954102005002944 |
| [2] |
Cowan DA, Tow LA. Endangered Antarctic environments[J]. Annual Review of Microbiology, 2004, 58: 649-690. DOI:10.1146/annurev.micro.57.030502.090811 |
| [3] |
Niederberger TD, McDonald IR, Hacker AL, et al. Microbial community composition in soils of Northern Victoria Land, Antarctica[J]. Environmental Microbiology, 2008, 10(7): 1713-1724. DOI:10.1111/j.1462-2920.2008.01593.x |
| [4] |
Foght J, Aislabie J, Turner S, et al. Culturable bacteria in subglacial sediments and ice from Two Southern Hemisphere Glaciers[J]. Microbial Ecology, 2004, 47(4): 329-340. |
| [5] |
Brambilla E, Hippe H, Hagelstein A, et al. 16S rDNA diversity of cultured and uncultured prokaryotes of a mat sample from Lake Fryxell, McMurdo Dry Valleys, Antarctica[J]. Extremophiles, 2001, 5(1): 23-33. DOI:10.1007/s007920000169 |
| [6] |
Wang W, Hu JM, Chen H, et al. LA-ICP-MS zircon U-Pb ages and geological constraint of intrusive rocks from the Inexpressible Island, Northern Victoria Land, Antarctica[J]. Geological Bulletin of China, 2014, 33(12): 2023-2031. (in Chinese) 王伟, 胡健民, 陈虹, 等. 南极北维多利亚地难言岛侵入岩LA-ICP-MS锆石U-Pb年龄及其地质意义[J]. 地质通报, 2014, 33(12): 2023-2031. DOI:10.3969/j.issn.1671-2552.2014.12.024 |
| [7] |
Barrett JE, Virginia RA, Hopkins DW, et al. Terrestrial ecosystem processes of Victoria Land, Antarctica[J]. Soil Biology and Biochemistry, 2006, 38(10): 3019-3034. DOI:10.1016/j.soilbio.2006.04.041 |
| [8] |
Hofstee EH, Balks MR, Petchey F, et al. Soils of Seabee Hook, Cape Hallett, northern Victoria Land, Antarctica[J]. Antarctic Science, 2006, 18(4): 473-486. DOI:10.1017/S0954102006000526 |
| [9] |
Aislabie J, Jordan S, Ayton J, et al. Bacterial diversity associated with ornithogenic soil of the Ross Sea region, Antarctica[J]. Canadian Journal of Microbiology, 2009, 55(1): 21-36. DOI:10.1139/W08-126 |
| [10] |
Amann RI, Ludwig W, Schleifer KH. Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J]. Microbiological Reviews, 1995, 59(1): 143-169. DOI:10.1128/MMBR.59.1.143-169.1995 |
| [11] |
Liu CY, Cong BL, Wang NF, et al. Biodiversity, physiological and biochemical identification of culturable microorganisms from the soil of Fildes Peninsula, Antarctica[J]. Haiyang Xuebao, 2016, 38(6): 69-81. (in Chinese) 刘春影, 丛柏林, 王能飞, 等. 南极菲尔德斯半岛可培养土壤微生物多样性及理化性质鉴定[J]. 海洋学报, 2016, 38(6): 69-81. DOI:10.3969/j.issn.0253-4193.2016.06.008 |
| [12] |
He RH, Du ZJ, Yu Y, et al. Isolation of antibacterial and culturable bacteria from Arctic tundra soils[J]. Acta Microbiologica Sinica, 2019, 59(6): 1050-1062. (in Chinese) 贺瑞含, 杜宗军, 俞勇, 等. 北极苔原土壤中可培养细菌的分离及其抗菌活性测定[J]. 微生物学报, 2019, 59(6): 1050-1062. |
| [13] |
González JM, Whitman WB, Hodson RE, et al. Identifying numerically abundant culturable bacteria from complex communities: an example from a lignin enrichment culture[J]. Applied and Environmental Microbiology, 1996, 62(12): 4433-4440. DOI:10.1128/AEM.62.12.4433-4440.1996 |
| [14] |
Kaeberlein T, Lewis K, Epstein SS. Isolating "Uncultivable" microorganisms in pure culture in a simulated natural environment[J]. Science, 2002, 296(5570): 1127-1129. DOI:10.1126/science.1070633 |
| [15] |
Lee CK, Barbier BA, Bottos EM, et al. The Inter-Valley soil comparative survey: the ecology of Dry Valley edaphic microbial communities[J]. The ISME Journal, 2012, 6(5): 1046-1057. DOI:10.1038/ismej.2011.170 |
| [16] |
Chen TS. Manufacture and Application of Microbial Culture Medium[M]. Beijing: China Agriculture Press, 1995: 179-184. (in Chinese) 陈天寿. 微生物培养基的制造与应用[M]. 北京: 中国农业出版社, 1995: 179-184. |
| [17] |
Zhou N, Jiang CY, Liu SJ. Cultivation of microorganisms from environments: nutrient level of the culture medium is of great importance[J]. Microbiology China, 2016, 43(5): 1075-1081. (in Chinese) 周楠, 姜成英, 刘双江. 从环境中分离培养微生物:培养基营养水平至关重要[J]. 微生物学通报, 2016, 43(5): 1075-1081. |
| [18] |
ZoBell CE. Studies on marine bacteria. Ⅰ. the cultural requirements of heterotrophic aerobes[J]. Journal of Marine Research, 1941, 4: 42-75. |
| [19] |
Mu DS, Liang QY, Wang XM, et al. Metatranscriptomic and comparative genomic insights into resuscitation mechanisms during enrichment culturing[J]. Microbiome, 2018, 6: 230. DOI:10.1186/s40168-018-0613-2 |
| [20] |
Bosshard PP, Santini Y, Grüter D, et al. Bacterial diversity and community composition in the chemocline of the meromictic alpine lake Cadagno as revealed by 16S rDNA analysis[J]. FEMS Microbiology Ecology, 2000, 31(2): 173-182. |
| [21] |
Stackebrandt E, Goebel BM. Taxonomic Note: a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J]. International Journal of Systematic Bacteriology, 1994, 44(4): 846-849. |
| [22] |
Adams BJ, Bardgett RD, Ayres E, et al. Diversity and distribution of Victoria Land biota[J]. Soil Biology and Biochemistry, 2006, 38(10): 3003-3018. DOI:10.1016/j.soilbio.2006.04.030 |
| [23] |
Aislabie JM, Chhour KL, Saul DJ, et al. Dominant bacteria in soils of Marble Point and Wright Valley, Victoria Land, Antarctica[J]. Soil Biology and Biochemistry, 2006, 38(10): 3041-3056. DOI:10.1016/j.soilbio.2006.02.018 |
| [24] |
Simas FNB, Schaefer CEGR, Melo VF, et al. Ornithogenic cryosols from Maritime Antarctica: phosphatization as a soil forming process[J]. Geoderma, 2007, 138(3/4): 191-203. |
| [25] |
Sun FQ, Wang BJ, Li GY, et al. Diversity of bacteria isolated from the South China Sea sediments[J]. Acta Microbiologica Sinica, 2008, 48(12): 1578-1587. (in Chinese) 孙风芹, 汪保江, 李光玉, 等. 南海南沙海域沉积物中可培养微生物及其多样性分析[J]. 微生物学报, 2008, 48(12): 1578-1587. DOI:10.3321/j.issn:0001-6209.2008.12.003 |
| [26] |
Ramos PL, van Trappen S, Thompson FL, et al. Screening for endophytic nitrogen-fixing bacteria in Brazilian sugar cane varieties used in organic farming and description of Stenotrophomonas pavanii sp. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(4): 926-931. DOI:10.1099/ijs.0.019372-0 |
| [27] |
Pindi PK, Manorama R, Begum Z, et al. Arthrobacter antarcticus sp. nov., isolated from an Antarctic marine sediment[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(10): 2263-2266. DOI:10.1099/ijs.0.012989-0 |
| [28] |
Rodrı́guez H, Fraga R. Phosphate solubilizing bacteria and their role in plant growth promotion[J]. Biotechnology Advances, 1999, 17(4/5): 319-339. |
| [29] |
Kloepper JW, Leong J, Teintze M, et al. Enhanced plant growth by siderophores produced by plant growth-promoting rhizobacteria[J]. Nature, 1980, 286(5776): 885-886. DOI:10.1038/286885a0 |
| [30] |
Behrendt U, Ulrich A, Schumann P, et al. A taxonomic study of bacteria isolated from grasses: a proposed new species Pseudomonas graminis sp. nov.[J]. International Journal of Systematic Bacteriology, 1999, 49(1): 297-308. |
| [31] |
Li J, Tian XP, Zhu TJ, et al. Streptomyces fildesensis sp. nov., a novel streptomycete isolated from Antarctic soil[J]. Antonie van Leeuwenhoek, 2011, 100(4): 537-543. DOI:10.1007/s10482-011-9609-7 |
| [32] |
Kim YC, Leveau J, McSpadden GBB, et al. The multifactorial basis for plant health promotion by plant-associated bacteria[J]. Applied and Environmental Microbiology, 2011, 77(5): 1548-1555. DOI:10.1128/AEM.01867-10 |
| [33] |
Bowman JP, Cavanagh J, Austin JJ, et al. Novel Psychrobacter species from Antarctic ornithogenic soils[J]. International Journal of Systematic Bacteriology, 1996, 46(4): 841-848. DOI:10.1099/00207713-46-4-841 |
| [34] |
Zeng YX, Yu Y, Liu Y, et al. Psychrobacter glaciei sp. nov., isolated from the ice core of an Arctic glacier[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(4): 1792-1798. DOI:10.1099/ijsem.0.000939 |
| [35] |
Höfle MG. Bacterioplankton community structure and dynamics after large-scale release of nonindigenous bacteria as revealed by low-molecular-weight-RNA analysis[J]. Applied and Environmental Microbiology, 1992, 58(10): 3387-3394. DOI:10.1128/AEM.58.10.3387-3394.1992 |
| [36] |
Bernardet JF, Segers P, Vancanneyt M, et al. Cutting a Gordian knot: emended classification and description of the genus Flavobacterium, emended description of the family Flavobacteriaceae, and proposal of Flavobacterium hydatis nom. nov. (basonym, Cytophaga aquatilis Strohl and Tait 1978)[J]. International Journal of Systematic Bacteriology, 1996, 46(1): 128-148. DOI:10.1099/00207713-46-1-128 |
| [37] |
Niederberger TD, Sohm JA, Gunderson TE, et al. Microbial community composition of transiently wetted Antarctic Dry Valley soils[J]. Frontiers in Microbiology, 2015, 6: 9. |
| [38] |
Busse HJ. Review of the taxonomy of the genus Arthrobacter, emendation of the genus Arthrobacter sensu lato, proposal to reclassify selected species of the genus Arthrobacter in the novel genera Glutamicibacter gen. nov., Paeniglutamicibacter gen. nov., Pseudoglutamicibacter gen. nov., Paenarthrobacter gen. nov. and Pseudarthrobacter gen. nov., and emended description of Arthrobacter roseus[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(1): 9-37. DOI:10.1099/ijsem.0.000702 |
| [39] |
SantaCruz-Calvo L, González-López J, Manzanera M. Arthrobacter siccitolerans sp. nov., a highly desiccation-tolerant, xeroprotectant-producing strain isolated from dry soil[J]. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(11): 4174-4180. |
| [40] |
Piao AL, Feng XM, Nogi Y, et al. Sphingomonas qilianensis sp. nov., isolated from surface soil in the permafrost region of Qilian Mountains, China[J]. Current Microbiology, 2016, 72(4): 363-369. DOI:10.1007/s00284-015-0957-9 |
| [41] |
Busse HJ, Denner EBM, Buczolits S, et al. Sphingomonas aurantiaca sp. nov., Sphingomonas aerolata sp. nov. and Sphingomonas faeni sp. nov., air- and dustborne and Antarctic, orange-pigmented, psychrotolerant bacteria, and emended description of the genus Sphingomonas[J]. International Journal of Systematic and Evolutionary Microbiology, 2003, 53(5): 1253-1260. DOI:10.1099/ijs.0.02461-0 |
| [42] |
Lin SY, Shen FT, Lai WA, et al. Sphingomonas formosensis sp. nov., a polycyclic aromatic hydrocarbon-degrading bacterium isolated from agricultural soil[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(7): 1581-1586. |
| [43] |
Schippers A, Schumann P, Spröer C. Nocardioides oleivorans sp. nov., a novel crude-oil-degrading bacterium[J]. International Journal of Systematic and Evolutionary Microbiology, 2005, 55(4): 1501-1504. DOI:10.1099/ijs.0.63500-0 |
| [44] |
Mattes TE, Coleman NV, Spain JC, et al. Physiological and molecular genetic analyses of vinyl chloride and ethene biodegradation in Nocardioides sp. strain JS614[J]. Archives of Microbiology, 2005, 183(2): 95-106. DOI:10.1007/s00203-004-0749-2 |
 2020, Vol. 47
2020, Vol. 47