扩展功能
文章信息
- 马锐, 郭饶, 王倩, 刘建国, 管晓燕
- MA Rui, GUO Rao, WANG Qian, LIU Jian-Guo, GUAN Xiao-Yan
- 口腔微生物耐氟机制的研究进展
- Research progress in fluoride resistance mechanism of oral microorganism
- 微生物学通报, 2020, 47(5): 1607-1614
- Microbiology China, 2020, 47(5): 1607-1614
- DOI: 10.13344/j.microbiol.china.190795
-
文章历史
- 收稿日期: 2019-09-30
- 接受日期: 2020-02-08
- 网络首发日期: 2020-04-15
2. 遵义医科大学医学与生物学研究中心 贵州省普通高等学校口腔疾病研究特色重点实验室 贵州 遵义 563006;
3. 遵义医科大学附属口腔医院正畸科 贵州 遵义 563099
2. Special Key Laboratory of Oral Disease Research, Higher Education Institution in Guizhou Province, Research Center for Medicine and Biology, Zunyi Medical University, Zunyi, Guizhou 563006, China;
3. Department of Orthodontics, Hospital of Stomatology, Zunyi Medical University, Zunyi, Guizhou 563099, China
氟化物是目前最受欢迎的一种防龋药物,其通过增强牙体硬组织的再矿化和减少牙齿脱矿来降低患龋风险[1]。由于氟化物具有强大的防龋作用,自20世纪中期起就被应用于城市供水系统[2-3],而且还广泛应用于许多口腔护理产品,如含氟牙膏、含氟凝胶、含氟漱口水和氟保护漆等[4-5]。此外,由于氟化物具有很强的抗微生物作用,可通过影响ATP酶(H+/ATPase)与烯醇化酶来抑制微生物的代谢生长[6]。随着氟化物的广泛应用,氟暴露途径不断增加,长期使用氟化物可能会导致耐氟菌株的出现。由于变异链球菌是主要的致龋菌,有研究对其亲本菌株与耐氟菌株进行了比较,以了解其在产酸、脱矿、耐酸、黏附以及糖酵解能力方面的差别,发现在氟环境下,耐氟菌株的产酸和脱矿能力较亲本菌株强;在酸性环境中,耐氟菌株的生长代谢受抑制程度较亲代菌株小;耐氟菌株的葡萄糖摄入能力较亲代菌株强;而耐氟菌株与亲代菌株在黏附率的比较上无明显差异,据此推测耐氟菌株在氟环境下有更强的代谢生长能力,其致龋潜力强于亲代菌株[7]。
长期使用氟化物不仅会造成耐氟菌株的产生,而且可能引起菌群的改变[8-9]。已知氟化物能抑制多种口腔微生物的生长,如唾液链球菌、乳酸杆菌、牙龈卟啉单胞菌、血链球菌、变异链球菌、白色念珠菌等,且不同种类的微生物对氟化物的敏感性不同[10-12]。如变异链球菌对氟化物的敏感性是乳酸杆菌的20-40倍、放线菌的7倍,而有些微生物如韦荣氏菌则根本不受氟的影响[13]。口腔内包含着多种属微生物群落,群落成员间存在着营养竞争与生存的协同作用、拮抗作用和毒力因子的中和作用及生长依靠信号机制干扰等[14]。因此,口腔中一类细菌群落受抑制,可能牵连另一种属菌落抑制或增殖,从而打破口腔微生物稳态。例如菌斑中变异链球菌的数量较高时,往往血链球菌的水平会被抑制;而具核梭杆菌和中间普雷沃菌可以通过产生有机酸和氨,使菌斑环境接近适宜牙龈卟啉单胞菌生存的pH值[15-16]。有研究发现含氟牙膏可对口腔菌斑生物膜产生影响,经含氟牙膏处理后变异链球菌和牙龈卟啉单胞菌的生长受到抑制,而血链球菌数量却增多[17]。Yasuda等[9]模拟人类所处含氟环境建立动物模型,通过16S rRNA基因扩增和基因组测序验证了氟干扰对小鼠口腔微生物群落组成有选择性影响。
以上信息提示,长期暴露于氟环境下的口腔菌群,其组成和结构可能会发生改变,进而影响口腔微生态平衡。人类口腔中的微生物种类繁多,包括细菌、真菌、病毒和古生菌等,形成了一个复杂的微生态环境。这些微生物不仅直接参与口腔及身体其他远隔器官疾病的发生、发展,还可通过与其他微生物相互作用而间接影响人类健康。口腔微生态失衡时,可诱发多种口腔感染性疾病,包括龋病、牙髓病、牙周炎、智齿冠周炎、颌骨骨髓炎等,严重危害口腔局部健康[18]。许多研究表明口腔微生物与一些系统性疾病密切相关,如肿瘤、糖尿病、心血管疾病、类风湿关节炎、早产等,口腔微生态的平衡对人类健康意义重大[19-20]。因此,对口腔微生物抗氟机制进行深入探索,不仅有利于龋病预防的进一步研究,还有利于维持口腔微生态平衡,维护全身健康。
1 氟对多种口腔微生物的抑制作用有研究表明,氟化物能抑制多种口腔微生物的生长,并以多种方式影响口腔中的致龋菌和其他细菌的新陈代谢,如通过直接或间接抑制代谢酶,包括烯醇化酶、H+/ATPase等,导致微生物生长和代谢受抑制[21]。烯醇化酶是糖酵解中催化2-磷酸甘油酯转化为磷酸烯醇丙酮酸酯(phosphoenolpyruvate,PEP)的关键酶,对微生物能量的获得与代谢生长至关重要,当微生物细胞中氟离子聚集时,烯醇化酶受抑制,使三磷酸腺苷(adenosine triphosphate,ATP)生成减少,丙酮酸激酶由于缺少能量供应不能将PEP转化成丙酮酸,进而影响到细胞内的糖酵解过程,并导致磷酸转移酶系统(phosphotransferase system,PTS)对葡萄糖的摄取减少;而H+/ATPase受抑制则会导致细胞膜内外质子浓度梯度的破坏,H+无法排出,细胞质内酸化,从而降低细胞的代谢和耐酸能力[6]。早期时,钱伟等[22]研究了氟化钠对牙龈卟啉单胞菌、伴放线放线杆菌、中间型普氏菌、血链球菌和亲缘链球菌的体外抑制作用,发现氟化钠对这些龈下菌均有抑制作用,其中对伴放线放线杆菌和牙龈卟啉单胞菌的最小抑菌浓度(minimal inhibit concentration,MIC)为1.28×105 μg/L,对亲缘链球菌的MIC为2.56×105 μg/L,对中间型普氏菌的MIC为10.24×105 μg/L,而氟化钠浓度要达到20.48×105- 40.96×105 μg/L才能抑制血链球菌。朱昞等[23]通过模拟接近于人口腔内细菌生存环境选择变异链球菌、唾液链球菌、血链球菌及内氏放线菌4种口腔常驻菌进行连续培养,发现2.28×105 μg/L高氟组氟化钠溶液及1.14×105 μg/L低氟组氟化钠溶液均可抑制这4种细菌产酸,降低培养基中pH值,且细菌受抑制程度随氟化钠浓度增大而加重。有学者从氟化物影响菌斑生物膜形成的角度着手,研究不同氟化钠水平下变异链球菌的生物膜敏感性,发现氟化钠浓度为0.50×105 μg/L时细菌胞外多糖生成减少,浓度为1.00×105 μg/L时细菌生物膜合成大大减少,当浓度大于2.25×105 μg/L时几乎不合成生物膜[24]。
2 口腔微生物的耐氟性目前,氟对口腔微生物的抑制作用已取得了显著的研究成果,但另一方面,口腔微生物对氟的抵抗作用也逐渐引起科研工作者的关注。先有学者Brown等从长期使用氟化物凝胶的干燥症患者中分离得到了变异链球菌耐氟菌株,发现该菌株仅具有短暂的氟抗性[25];后来研究者们在实验室获得了具有稳定抗氟能力的耐氟菌株,抵抗能力达4.00×105-10.00×105 μg/L,且在无氟的情况下这种抗性至少会持续50代[8, 26]。Nassar等[24]研究了氟化钠对7种不同变异链球菌的影响,其中从高危龋病个体分离出的A32-2、NG8菌株有较高的耐氟性,提示在一定氟浓度范围内,生物膜的产生及代谢活性取决于菌株本身,不同菌株对氟毒性的抵抗力不同。有学者研究了耐氟菌株UAFR、UF35的生物膜形成能力及产酸能力,并以它们的亲本菌株UA159作为参考,发现在无氟环境下,耐氟菌株UAFR的生物膜形成能力与产酸能力均强于UF35和UA159,而UF35和UA159无明显差异;当处于有氟环境中时,两种耐氟菌株UAFR和UF35均较UA159表现出更强的生物膜形成能力与产酸能力[27]。还有研究发现,亲本菌株UA159与其耐氟菌株UA159-FR相比,烯醇化酶的活性明显降低,通过基因测序发现UA159-FR的烯醇化酶基因发生突变,这可能是导致烯醇化酶对氟敏感性降低并使细菌能够在高浓度氟化物的存在下代谢的原因[26]。
3 口腔微生物的主要耐氟机制 3.1 氟化物核糖开关2012年,Breaker等[28]通过对具有代表性的2 000多种细菌和古细菌进行分析,发现了细菌用于感测氟化物并降低氟毒性的生物系统——氟化物核糖开关,为研究细菌抗氟机制打开了新的局面。大多数核糖开关是位于mRNA中的代谢物结合结构,它们主要由两部分组成:一是感受外界配体的适体域,二是调控基因表达的结构域;核糖开关的适体域与代谢物或其他配体特异性结合,引起核糖开关的折叠发生改变,从而调控mRNA编码的蛋白质的表达[29]。有学者使用分析晶体结构的方法对氟化物核糖开关进行了研究,发现其在与配体结合时,位于中心的是一个氟离子,周围环绕结合3个镁离子,形成一个类似口袋的结构;由于镁离子和氟离子通过正负电荷而互相吸引,从而导致核糖开关的构象发生变化,引起下游抗氟相关基因的编码改变,并最终使得细菌具有更强的抗氟能力[30]。经过多次验证实验,Breaker[28]确认氟化物是这种核糖开关的天然配体。
有两个基因家族与这种氟化物核糖开关最为相关,其中一个基因家族是eriC,该基因家族属于广泛分布的氯离子通道蛋白(chloride channel,CLC)超家族,参与编码CLC型离子通道蛋白;典型的EriC蛋白与Cl-转运相关,而EriCF同源蛋白离子通道的氨基酸构成与EriC不同,其能对F-起到特异转运的作用,通过从细菌细胞质中输出F-来对抗氟毒性,且EriCF比传统EriC具有更强的质子驱动能力[31]。另一个重要的基因家族是crcB,其位于编码具有不同功能蛋白质的基因上游,并可能调控这些基因编码一些离子转运蛋白(如氯离子、钠离子、质子等),通过增强离子载体对氟的结合能力,转运和调节细胞中的氟离子浓度,从而减少氟对自身的胁迫;还可编码一些基因产物参与各种生理过程(例如普遍的应激反应、DNA修复等)或代谢过程(例如烯醇化酶、甲酸氢裂解酶等),以及编码一些未知功能的蛋白质[28, 32]。
与野生型菌株相比,在大肠杆菌或白色念珠菌中敲除crcB基因可使突变体对氟的敏感性提高200-350倍,其氟抗性又可以通过补充蜡状芽孢杆菌的eriCF来恢复[8]。在同样的基因组位置,有些菌株编码EriC蛋白,另一些则编码CrcB蛋白,表明这两种蛋白具有相同的功能,在抵抗氟毒性过程中扮演了重要角色[33]。为了进一步研究crcB和eriCF基因在口腔链球菌中的分布,Men等[34]对18种口腔链球菌的全基因组序列进行了序列同源性分析,发现口腔链球菌中有两种类型的eriCF基因(eriCF1和eriCF2)和两种类型的crcB基因(crcB1和crcB2),并鉴定了咽峡炎链球菌的耐氟相关基因有eriCF1和eriCF2,其中eriCF1是引起抗氟作用的主要基因;血链球菌的耐氟相关基因有eriCF2、crcB1、crcB2基因,其中crcB1、crcB2基因对血链球菌的耐氟性至关重要。
虽然在真核生物中没有发现氟化物核糖开关,但这些真核生物中有许多具有crcB同源基因,Li等[35]将这些同源基因重新命名为fex,为了证明真核生物中fex基因对氟毒性的抵抗作用,又将白色念珠菌、粗糙脉孢菌及酿酒酵母中的fex基因敲除,发现敲除fex基因的真核生物细胞内氟化物聚集超过10倍,表明在细胞转运出氟化物的过程中Fex蛋白起到重要作用。
3.2 与耐氟性相关的基因突变在对微生物氟抗性的早期研究中,烯醇化酶和H+/ATPase一直是重点。烯醇酶是糖酵解中的关键酶,在丙酮酸的生成中起催化作用,H+/ATPase参与质子浓度梯度的维持及多种溶质如葡萄糖、氨基酸等的转运,从而影响着微生物的生长[6]。烯醇化酶和H+/ATPase对氟化物十分敏感,因此,编码它们的基因曾被认为是氟胁迫下最容易出现突变的位点。Hoelscher等[36]发现变异链球菌耐氟菌株胞膜上的H+/ATPase对氟化物敏感度降低,认为耐氟菌株中H+/ATPase结构发生了改变,并推测这种改变与其酶基因突变有关。Mitsuhata等[37]在耐氟菌株NCH105的烯醇酶编码基因中检测到一个突变位点,该突变以亮氨酸代替脯氨酸作为第173个氨基酸,引起了F-结合位点的构象变化,间接导致了烯醇化酶活性升高,这可能与耐氟菌株的氟抗性有关。
目前的研究多数是探索单核苷酸突变引起的微生物氟抗性改变[38-39]。早在1987年,Brussock等[40]就已经提出细菌表现出的氟抗性可能至少是两种基因突变的累积效应,但一直没有证据证明此观点。近年来,随着测序技术的发展和测序费用的降低,全基因组测序(whole genome sequencing,WGS)和相关的生物信息分析技术得到了广泛的应用,使得研究者可以分析不同个体基因组间的结构差异,同时完成单核苷酸及基因注释,该技术已成功地应用于预测与细菌耐药性和宿主适应性相关的单核苷酸多态性(single nucleotide polymorphism,SNPs)[41-42]。Liao等[39]利用全基因组测序技术对变异链球菌C180-2及其耐氟菌株C180-2FR的基因组序列进行了比较分析,并在耐氟菌株中鉴定出8个单核苷酸突变位点(SNPs),通过对包含(或靠近)这8个SNPs的基因进行表达量检测,发现与C180-2相比较,耐氟菌株C180-2FR中一个包含编码氟化物逆向转运蛋白基因的基因簇被上调了10倍,有两个SNPs位于该基因簇中,一个位于启动子区,另一个位于蛋白编码区;此外,在C180-2FR中有一个基因在早期生长阶段被下调了60%,该基因编码一种摄取甘油的促进蛋白,在其启动子区含有一个SNP。2018年,Liao等[26]在以往研究的基础上,对两株耐氟变异链球菌(UA159-FR和C180-2FR)及其相应野生型菌株(UA159和C180-2)的全基因组序列进行对比分析,研究与变异链球菌的氟化物抗性相关的遗传位点,进一步定位了两种耐氟菌株的共同突变区域,证实了多个基因突变共同参与形成菌株的氟抗性。总之,利用WGS测序能够发现耐氟菌株基因组的遗传变化,这些发现可以为微生物耐氟机制提供新的见解。
3.3 耐氟质粒的转入质粒是细菌、酵母菌和放线菌等生物中染色体以外的DNA分子,其存在于细胞质中,具有自主复制能力,是闭合环状的双链DNA分子。质粒可以使宿主菌获得质粒DNA分子上所含有的某些特殊功能。Brown等[25]从干燥症患者口腔内乳杆菌占优势的菌斑中分离出了变异链球菌的耐氟菌株。盛江筠[43]推测,变异链球菌耐氟菌株的出现很可能是由于变异链球菌体内转入了乳杆菌中的耐氟质粒,因此获得了高水平耐氟能力。研究发现,这种耐氟能力是应激性的,当氟化物的胁迫作用消除时,这类耐氟菌株就会失去相应的质粒,并迅速恢复原来的氟敏感状态[44]。近年来,Fernandez等[45]发现,缺乏编码氟离子转运蛋白基因(fex1和fex2)的酿酒酵母菌对氟化物高度敏感,在氟离子浓度为100 μmol/L时即受到抑制,而在培养基中加入含有FEX1的质粒后,该缺陷菌株能耐受5 mmol/L的高浓度氟化物。
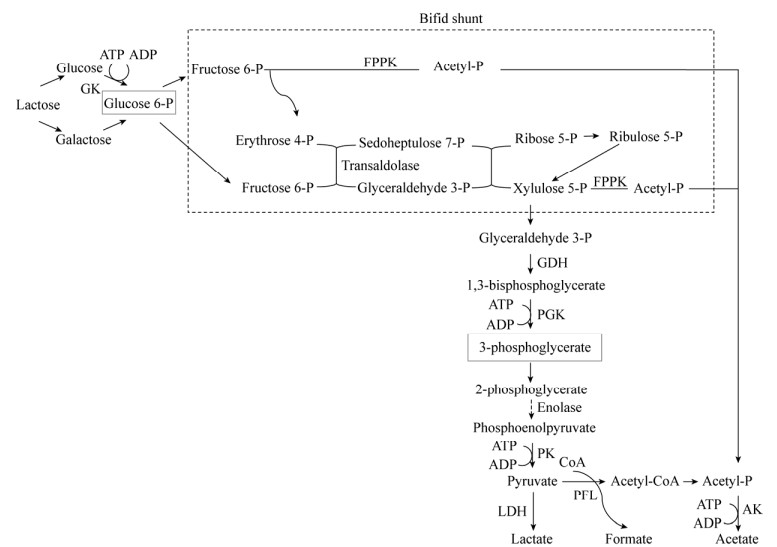
3.4 双歧途径双歧杆菌是一种新型龋相关菌,常在早期儿童龋患儿口腔中检测出。Manome等[46]对双歧杆菌的产酸活性及其对氟化物的敏感性进行了研究,发现双歧杆菌较变异链球菌耐受高氟环境,氟化物对双歧杆菌产酸的半抑制浓度(IC50)比变异链球菌高6.0-14.2倍,双歧杆菌的高氟耐受性被认为是由于其具有独特的代谢途径,即双歧途径(图 1)。已知大部分致龋菌如变异链球菌等通过糖酵解途径代谢碳水化合物,主要产生乳酸[47];而双歧杆菌通过双歧途径代谢碳水化合物,主要生成乳酸和乙酸[48]。当氟化物抑制了双歧杆菌糖酵解过程中的烯醇化酶,引起其上游代谢产物如6-磷酸葡萄糖、3-磷酸甘油酸的积累,以及其下游产物如乳酸盐、ATP生成受阻,此时,双歧杆菌可通过分支途径将6-磷酸果糖分解生成4-磷酸赤藓糖及乙酰磷酸,最后产生乙酸盐和ATP,从而抵抗氟诱导的代谢抑制[46]。
氟化物作为预防龋齿的常用制剂,在临床上已应用较长时间。然而氟过量使用带来的不良影响使氟在临床上的应用受到争议,比如过量的氟接触可导致氟斑牙的形成[49-50]、耐氟菌株的出现降低氟化物防龋效果等。近期还有报道[9]证实氟对口腔微生物群落组成有选择性影响。因此,对口腔微生物耐氟机制进行深入研究,有助于研发新型防龋药物,有利于指导口腔保健和临床用药,并对维持口腔微生态平衡、防治疾病提供新的视角和策略。例如,已知与氟化物核糖开关相关的最常见基因是crcB与eriCF,若能阻断致龋菌编码EriC蛋白与CrcB蛋白,并联合使用氟制剂,理论上可有效抑制致龋菌[31];又如,真菌可以通过解毒、减少吸收或增加排出的机制来抵御环境中有毒的F-离子和化合物,若将阻断氟化物转运与含氟药物联合应用于抗菌药物,将有望对抗真菌感染[51];再如,是否可以利用耐氟机制调控口腔内微生物群落结构与组成,从而维持口腔微生态的平衡,达到防治疾病的效果,这些都需要进一步的探索。
| [1] |
Rošin-Grget K, Peroš K, Šutej I, et al. The cariostatic mechanisms of fluoride[J]. Acta Medica Academica, 2013, 42(2): 179-188. DOI:10.5644/ama2006-124.85 |
| [2] |
Mohd Nor NA, Chadwick BL, Farnell DJJ, et al. The impact of a reduction in fluoride concentration in the Malaysian water supply on the prevalence of fluorosis and dental caries[J]. Community Dentistry and Oral Epidemiology, 2018, 46(5): 492-499. DOI:10.1111/cdoe.12407 |
| [3] |
Allukian M Jr, Carter-Pokras OD, Gooch BF, et al. Science, politics, and communication: the case of community water fluoridation in the US[J]. Annals of Epidemiology, 2018, 28(6): 401-410. DOI:10.1016/j.annepidem.2017.05.014 |
| [4] |
Ahmed F, Prashanth ST, Sindhu K, et al. Antimicrobial efficacy of nanosilver and chitosan against Streptococcus mutans, as an ingredient of toothpaste formulation: An in vitro study[J]. Journal of Indian Society of Pedodontics and Preventive Dentistry, 2019, 37(1): 46-54. DOI:10.4103/JISPPD.JISPPD_239_18 |
| [5] |
Wasiluk A. Fluoride compounds in dental caries prophylaxis in children and adolescents–review of Polish literature[J]. Przeglad Epidemiologiczny, 2017, 71(4): 603-611. |
| [6] |
Hamilton IR. Biochemical effects of fluoride on oral bacteria[J]. Journal of Dental Research, 1990, 69(S2): 660-667. |
| [7] |
Xu H, Liu YQ. Cariogenicity of fluoride-resistant strains of Streptococcus mutans[J]. International Journal of Stomatology, 2013, 40(5): 698-700. (in Chinese) 许华, 刘英群. 变异链球菌耐氟菌株的致龋能力[J]. 国际口腔医学杂志, 2013, 40(5): 698-700. |
| [8] |
Liao Y, Brandt BW, Li JY, et al. Fluoride resistance in Streptococcus mutans: a mini review[J]. Journal of Oral Microbiology, 2017, 9(1): 1344509. DOI:10.1080/20002297.2017.1344509 |
| [9] |
Yasuda K, Hsu T, Gallini CA, et al. Fluoride depletes acidogenic taxa in oral but not gut microbial communities in mice[J]. mSystems, 2017, 2(4): e00047-17. |
| [10] |
Maden EA, Altun C, Ozmen B, et al. Antimicrobial effect of toothpastes containing fluoride, xylitol, or xylitol-probiotic on salivary Streptococcus mutans and Lactobacillus in children[J]. Nigerian Journal of Clinical Practice, 2018, 21(2): 134-138. |
| [11] |
Kulik EM, Waltimo T, Weiger R, et al. Development of resistance of mutans streptococci and Porphyromonas gingivalis to chlorhexidine digluconate and amine fluoride/stannous fluoride-containing mouthrinses, in vitro[J]. Clinical Oral Investigations, 2015, 19(6): 1547-1553. DOI:10.1007/s00784-014-1379-y |
| [12] |
Thomas A, Thakur S, Habib R. Comparison of antimicrobial efficacy of green tea, garlic with lime, and sodium fluoride mouth rinses against Streptococcus mutans, Lactobacilli species, and Candida albicans in children: a randomized double-blind controlled clinical trial[J]. International Journal of Clinical Pediatric Dentistry, 2017, 10(3): 234-239. DOI:10.5005/jp-journals-10005-1442 |
| [13] |
Ma H. Biological effects of fluoride on oral microorganisms[J]. Beijing Stomatology, 1998, 6(3): 132-134. (in Chinese) 马宏. 氟化物对口腔微生物的生物学作用[J]. 北京口腔医学, 1998, 6(3): 132-134. |
| [14] |
Shi WY, Zhou XD. Interaction of human oral microbial community[J]. West China Journal of Stomatology, 2010, 28(1): 1-4. (in Chinese) 施文元, 周学东. 人体口腔微生物群的相互作用[J]. 华西口腔医学杂志, 2010, 28(1): 1-4. |
| [15] |
Yang P, Hao YQ. Research progress on the ecological relationship between Streptococcus mutans and some other oral bacteria[J]. Chinese Journal of Microecology, 2009, 21(11): 1050-1053. (in Chinese) 杨攀, 郝玉庆. 变异链球菌与部分口腔其他细菌生态关系的研究进展[J]. 中国微生态学杂志, 2009, 21(11): 1050-1053. |
| [16] |
Takahashi N. Acid-neutralizing activity during amino acid fermentation by Porphyromonas gingivalis, Prevotella intermedia and Fusobacterium nucleatum[J]. Oral Microbiology and Immunology, 2003, 18(2): 109-113. DOI:10.1034/j.1399-302X.2003.00054.x |
| [17] |
Cheng XQ, Liu JM, Li JY, et al. Comparative effect of a stannous fluoride toothpaste and a sodium fluoride toothpaste on a multispecies biofilm[J]. Archives of Oral Biology, 2017, 74: 5-11. DOI:10.1016/j.archoralbio.2016.10.030 |
| [18] |
He JZ, Xu X, Zhou XD. The oral microbiota and human health[J]. Journal of Microbes and Infections, 2017, 12(3): 139-145. (in Chinese) 何金枝, 徐欣, 周学东. 口腔微生物与全身健康研究进展[J]. 微生物与感染, 2017, 12(3): 139-145. DOI:10.3969/j.issn.1673-6184.2017.03.003 |
| [19] |
Zhang YH, Wang X, Li HX, et al. Human oral microbiota and its modulation for oral health[J]. Biomedicine & Pharmacotherapy, 2018, 99: 883-893. |
| [20] |
Xu X, He JZ, Zhou XD. Oral microbiome in the precision medicine[J]. International Journal of Stomatology, 2017, 44(6): 619-627. (in Chinese) 徐欣, 何金枝, 周学东. 口腔微生物组在口腔精准医疗中的运用[J]. 国际口腔医学杂志, 2017, 44(6): 619-627. |
| [21] |
Marquis RE, Clock SA, Mota-Meira M. Fluoride and organic weak acids as modulators of microbial physiology[J]. FEMS Microbiology Reviews, 2003, 26(5): 493-510. DOI:10.1111/j.1574-6976.2003.tb00627.x |
| [22] |
Qian W, Zhang JC, Xiao XR, et al. Research on inhibition of sodium fluoride on five subgingival bacteria in vitro[J]. West China Journal of Stomatology, 1998, 16(2): 105-107. (in Chinese) 钱伟, 章锦才, 肖晓蓉, 等. 氟化钠对5种常见龈下细菌抑制作用的体外研究[J]. 华西口腔医学杂志, 1998, 16(2): 105-107. |
| [23] |
Zhu B, Li JY, Huang ZW, et al. Comparison of Galla chinesis and sodium fluoride on growth and metabolism of bacteria in biofilm model[J]. West China Journal of Stomatology, 2005, 23(5): 367-369. (in Chinese) 朱昞, 李继遥, 黄正蔚, 等. 五倍子与氟化钠对人工口腔生物膜细菌生长代谢的影响[J]. 华西口腔医学杂志, 2005, 23(5): 367-369. DOI:10.3321/j.issn:1000-1182.2005.05.001 |
| [24] |
Nassar HM, Gregory RL. Biofilm sensitivity of seven Streptococcus mutans strains to different fluoride levels[J]. Journal of Oral Microbiology, 2017, 9(1): 1328265. DOI:10.1080/20002297.2017.1328265 |
| [25] |
Brown LR, White JO, Horton IM, et al. Effect of continuous fluoride gel use on plaque fluoride retention and microbial activity[J]. Journal of Dental Research, 1983, 62(6): 746-751. DOI:10.1177/00220345830620061201 |
| [26] |
Liao Y, Yang JM, Brandt BW, et al. Genetic loci associated with fluoride resistance in Streptococcus mutans[J]. Frontiers in Microbiology, 2018, 9: 3093. DOI:10.3389/fmicb.2018.03093 |
| [27] |
Cai YL, Liao Y, Brandt BW, et al. The fitness cost of fluoride resistance for different Streptococcus mutans strains in biofilms[J]. Frontiers in Microbiology, 2017, 8: 1630. DOI:10.3389/fmicb.2017.01630 |
| [28] |
Breaker RR. New insight on the response of bacteria to fluoride[J]. Caries Research, 2012, 46(1): 78-81. DOI:10.1159/000336397 |
| [29] |
Roth A, Breaker RR. The structural and functional diversity of metabolite-binding riboswitches[J]. Annual Review of Biochemistry, 2009, 78: 305-334. DOI:10.1146/annurev.biochem.78.070507.135656 |
| [30] |
Ren AM, Rajashankar KR, Patel DJ. Fluoride ion encapsulation by Mg2+ ions and phosphates in a fluoride riboswitch[J]. Nature, 2012, 486(7401): 85-89. DOI:10.1038/nature11152 |
| [31] |
Last NB, Stockbridge RB, Wilson AE, et al. A CLC-type F–/H+ antiporter in ion-swapped conformations[J]. Nature Structural & Molecular Biology, 2018, 25(7): 601-606. |
| [32] |
Chouhan S, Tuteja U, Flora SJS. Isolation, identification and characterization of fluoride resistant bacteria: possible role in bioremediation[J]. Applied Biochemistry and Microbiology, 2012, 48(1): 43-50. DOI:10.1134/S0003683812010036 |
| [33] |
Baker JL, Sudarsan N, Weinberg Z, et al. Widespread genetic switches and toxicity resistance proteins for fluoride[J]. Science, 2012, 335(6065): 233-235. DOI:10.1126/science.1215063 |
| [34] |
Men XC, Shibata Y, Takeshita T, et al. Identification of anion channels responsible for fluoride resistance in oral Streptococci[J]. PLoS One, 2016, 11(11): e0165900. DOI:10.1371/journal.pone.0165900 |
| [35] |
Li SS, Smith KD, Davis JH, et al. Eukaryotic resistance to fluoride toxicity mediated by a widespread family of fluoride export proteins[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(47): 19018-19023. DOI:10.1073/pnas.1310439110 |
| [36] |
Hoelscher GL, Hudson MC. Characterization of an unusual fluoride-resistant Streptococcus mutans isolate[J]. Current Microbiology, 1996, 32(3): 156-161. DOI:10.1007/s002849900028 |
| [37] |
Mitsuhata C, Puteri MM, Ohara Y, et al. Possible involvement of enolase in fluoride resistance in Streptococcus mutans[J]. Pediatric Dental Journal, 2014, 24(1): 12-16. DOI:10.1016/j.pdj.2013.10.002 |
| [38] |
Liao Y, Brandt BW, Zhang M, et al. A single nucleotide change in the promoter mutp enhances fluoride resistance of Streptococcus mutans[J]. Antimicrobial Agents and Chemotherapy, 2016, 60(12): 7509-7512. |
| [39] |
Liao Y, Chen JW, Brandt BW, et al. Identification and functional analysis of genome mutations in a fluoride-resistant Streptococcus mutans strain[J]. PLoS One, 2015, 10(4): e0122630. DOI:10.1371/journal.pone.0122630 |
| [40] |
Brussock SM, Kral TA. Effects of pH on expression of sodium fluoride resistance in Streptococcus mutans[J]. Journal of Dental Research, 1987, 66(10): 1594-1596. DOI:10.1177/00220345870660101701 |
| [41] |
Sheppard SK, Didelot X, Meric G, et al. Genome-wide association study identifies vitamin B5 biosynthesis as a host specificity factor in Campylobacter[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(29): 11923-11927. DOI:10.1073/pnas.1305559110 |
| [42] |
Alam MT, Petit Ⅲ RA, Crispell EK, et al. Dissecting vancomycin-intermediate resistance in Staphylococcus aureus using genome-wide association[J]. Genome Biology and Evolution, 2014, 6(5): 1174-1185. DOI:10.1093/gbe/evu092 |
| [43] |
Sheng JY. Effects of fluoride on glucose metabolism of Streptococcus oral[J]. International Journal of Stomatology, 1998, 25(3): 133-135. (in Chinese) 盛江筠. 氟化物对口腔链球菌糖代谢的影响[J]. 国外医学口腔医学分册, 1998, 25(3): 133-135. |
| [44] |
Liu LH, Tian J, Wu NF. Research progress on bacterial fluoride resistance[J]. Journal of Agricultural Science and Technology, 2013, 15(6): 113-118. (in Chinese) 刘丽慧, 田健, 伍宁丰. 细菌氟抗性研究进展[J]. 中国农业科技导报, 2013, 15(6): 113-118. DOI:10.3969/j.issn.1008-0864.2013.06.17 |
| [45] |
Fernandez R, Berro J. Use of a fluoride channel as a new selection marker for fission yeast plasmids and application to fast genome editing with CRISPR/Cas9[J]. Yeast, 2016, 33(10): 549-557. DOI:10.1002/yea.3178 |
| [46] |
Manome A, Abiko Y, Kawashima J, et al. Acidogenic potential of oral Bifidobacterium and its high fluoride tolerance[J]. Frontiers in Microbiology, 2019, 10: 1099. DOI:10.3389/fmicb.2019.01099 |
| [47] |
Takahashi N. Oral microbiome metabolism: from "who are they?" to "what are they doing?"[J]. Journal of Dental Research, 2015, 94(12): 1628-1637. DOI:10.1177/0022034515606045 |
| [48] |
Lee JH, O'Sullivan DJ. Genomic insights into Bifidobacteria[J]. Microbiology and Molecular Biology Reviews, 2010, 74(3): 378-416. |
| [49] |
Wang GP, Gu Y, Wu D, et al. Effect of excessive fluoride on KLK4 protein expression in rat ameloblast cells[J]. Chinese Journal of Control of Endemic Diseases, 2018, 33(3): 260-261. (in Chinese) 王光平, 顾瑜, 吴迪, 等. 过量氟对大鼠成釉细胞KLK4蛋白表达的影响[J]. 中国地方病防治杂志, 2018, 33(3): 260-261. |
| [50] |
Chu KJ, Wang HH, Bai GH, et al. Effects of coal-burning endemic fluorosis on the dental fluorosis and oxidative stress of the rats offspring[J]. Modern Preventive Medicine, 2016, 43(5): 806-809. (in Chinese) 初可嘉, 王海慧, 白国辉, 等. 燃煤型氟中毒对仔鼠氟斑牙和氧化应激的影响[J]. 现代预防医学, 2016, 43(5): 806-809. |
| [51] |
Binder J, Held J, Krappmann S. Impairing fluoride export of Aspergillus fumigatus mitigates its voriconazole resistance[J]. International Journal of Antimicrobial Agents, 2019, 53(5): 689-693. DOI:10.1016/j.ijantimicag.2019.02.003 |
 2020, Vol. 47
2020, Vol. 47