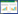扩展功能
文章信息
- 顾燕, 汤伟, 王悦, 戴宝, 卢德鹏, 许向阳, 何增国
- GU Yan, TANG Wei, WANG Yue, DAI Bao, LU De-Peng, XU Xiang-Yang, HE Zeng-Guo
- 耐高温α-淀粉酶分子结构与异源表达研究进展
- Advances in study on structure-function relationship of thermostable α-amylase and its heterologous expression
- 微生物学通报, 2020, 47(5): 1589-1599
- Microbiology China, 2020, 47(5): 1589-1599
- DOI: 10.13344/j.microbiol.china.190435
-
文章历史
- 收稿日期: 2019-05-20
- 接受日期: 2019-11-29
- 网络首发日期: 2020-01-02
2. 青岛海洋生物医药研究院 山东 青岛 266000;
3. 潍坊市信得生物科技有限公司 山东 潍坊 261000;
4. 枣庄市杰诺生物酶有限公司 山东 枣庄 277100
2. Marine Biomedical Research Institute of Qingdao, Qingdao, Shandong 266000, China;
3. Weifang Sinder Biotechnology Company Limited, Weifang, Shandong 261000, China;
4. Zaozhuang Jienuo Enzyme Company Limited, Zaozhuang, Shandong 277100, China
酶是一类非常重要的生物催化剂,因为其来源的天然性及良好的催化功能,数个世纪以来(自古巴比伦时期)就在食品等领域开始广泛应用[1]。淀粉酶是一种重要的水解酶类,是水解淀粉和糖原的一类酶的总称,淀粉酶通常作用于直链淀粉、可溶性淀粉、糖元、糊精等α-1, 4-葡聚糖糖苷键。淀粉酶分布广泛,几乎存在于所有的动植物和微生物中。最近统计表明淀粉酶产量已经占据酶制剂市场的30%左右[2]。
α-淀粉酶是淀粉酶中应用最广泛的酶种,其可以随机地从内部切断淀粉、糖原内的α-1, 4-葡萄糖苷键,将淀粉水解为糊精、低聚糖和单糖[3-4]。该类酶水解产物的末端残基碳原子的构型为A构型,所以称为α-淀粉酶。α-淀粉酶的pH适用范围十分广泛,pH在2.0–12.0之间均适用。根据催化作用温度的不同,α-淀粉酶可分为低温α-淀粉酶、中温α-淀粉酶和耐高温α-淀粉酶。相较于常温α-淀粉酶,耐高温α-淀粉酶有下述优点:(1)热稳定性更好,在工业生产的高温条件下稳定,能够长时间地发挥作用;(2)能够使淀粉的液化更加彻底,缩短糊化和过滤的时间,从而达到节约能源、降低成本的效果;(3)保存条件范围广、易于储存和运输等。因具有热稳定性和较宽的pH适应范围等特质,耐高温α-淀粉酶可直接在温度要求严苛的高温环境中发挥作用,有着较高的开发价值及应用价值。目前,耐高温α-淀粉酶已经逐渐发展为主流淀粉酶品种,广泛应用于淀粉制糖业、味精、啤酒等食品发酵行业以及纺织印染行业等[5-7]。
20世纪60年代末,酶水解理论在国外取得了决定性进展,使得淀粉酶法特别是高温淀粉酶水解工艺取得了重大的突破,逐渐取代了传统酸法,促使淀粉制糖工业获得了快速发展[8]。到了21世纪,丹麦、美国、日本等国家在构建地衣芽孢杆菌(Bacillus licheniformis)、曲霉等基因工程菌的研发技术及蛋白质工程技术上相继取得了突破,生产性状更好的专利酶分子以及性能良好的基因工程菌被相继采用,高温淀粉酶制剂产业逐步发展成为一个为跨国集团(如诺维信等)所垄断的行业。近年来,国内的耐高温α-淀粉酶研发及工业化生产获得了长足发展,形成与跨国公司竞争的态势,尤其表现在玉米深加工行业,耐高温α-淀粉酶和糖化酶的淀粉喷射液化技术和双酶法糖化技术的普遍应用,全面促成了我国味精、淀粉糖、柠檬酸等生产技术的改进及产业的飞跃。
但是,国内耐高温α-淀粉酶的酶分子在性能、产品质量及生产效率诸方面与国外行业巨头(如诺维信等)相比仍有较大差距。如何开发高质量热稳定性更高的耐高温α-淀粉酶、有效地提高产量及生产效率仍是我国淀粉酶业界需要不断探索的问题。由于性能优良的淀粉酶分子已受到国外专利保护,又加之关键的代谢工程及外源表达技术多被行业巨头视为核心技术秘而不宣,密切跟踪α-淀粉酶酶分子相关结构与功能的最新研究成果、充分了解行业内α-淀粉酶的异源过量表达的前沿技术就显得尤为重要。此外,当前α-淀粉酶的热稳定性和嗜酸性已成为淀粉制糖业的关注热点,因此,继续筛选和选择有潜力、兼具耐热及耐酸淀粉酶分子的微生物就有着重要意义[9]。
本文综述了近年来产耐高温α-淀粉酶菌种的筛选研究进展,并对耐高温α-淀粉酶的结构和功能以及耐高温α-淀粉酶活力提升的途径进行了总结,最后对基因工程菌株构建以及耐高温α-淀粉酶异源表达等方面的最新进展进行了回顾。
1 产耐高温α-淀粉酶的微生物最初用于工业生产的α-淀粉酶来自于真菌,曾被用作治疗消化系统疾病的药物辅助剂。随后的研究发现一些细菌来源的α-淀粉酶具有耐高温、耐酸或耐碱等极端环境条件的特性,能在高温工业生产的极端条件下保持催化性能。细菌中Bacillus属的48个种中有32个种可以产生α-淀粉酶,但是只有少数能够产生耐高温α-淀粉酶,常见的有地衣芽孢杆菌、嗜热芽孢杆菌(Bacillus thermophilus)、嗜热脂肪芽孢杆菌(Bacillus stearothermophilus)等。细菌α-淀粉酶目前仍是淀粉液化水解处理的高温工业过程中使用最广泛的工业酶。其中,来自于地衣芽孢杆菌的耐高温α-淀粉酶占据了相当大的市场。表 1列举了几种常见的不同来源耐高温α-淀粉酶产生菌及其最适温度的比较。
| 菌株 Strains |
最适反应温度 Optimum reaction temperature (℃) |
参考文献 References |
| Alicyclobacillus sp. | 75 | [10] |
| Bacillus amyloliquefaciens | 70 | [11] |
| Bacillus flavothermus | 60 | [12] |
| Bacillus lentus | 70 | [13] |
| Bacillus licheniformis | 100 | [14] |
| Bacillus stearothermophilus | 70–80 | [15-16] |
| Bacillus subtilis | 70 | [17] |
| Chloroflexus aurantiacus | 71 | [18] |
| Desulfurococcus mucosus | 100 | [17] |
| Dictyoglomus thermophilum | 90 | [19] |
| Geobacillus bacterium | 80 | [20] |
| Geobacillus stearotermophilus | 70 | [21] |
| Halothermothrix orenii | 60 | [22] |
| Lactobacillus amylovorus | 60–65 | [23] |
| Lactobacillus plantarum | 65 | [24] |
| Lipomyces kononenkoae | 70 | [25] |
| Myceliophthora thermophila | 100 | [26] |
| Pyrococcus furiosus | 100 | [27] |
| Rhizopus oryzae | 60 | [28] |
| Pyrococcus woesei | 100 | [29] |
| Pyrodyctium abyssi | 100 | [30] |
| Rhizomucor pusillus | 70 | [31] |
| Rhodothermus marinus | 61 | [32] |
| Scytalidium thermophilum | 60 | [33] |
| Staphylothermus marinus | 65 | [17] |
| Thermococcus aggregans | 100 | [17] |
| Thermococcus celer | 90 | [17] |
| Thermococcus guaymagensis | 100 | [17] |
| Thermotoga petrophila | 85 | [34] |
通过发现有潜力的产耐高温α-淀粉酶菌株,有可能拓宽高性能耐高温α-淀粉酶的筛选范围。最近,本课题在菌株筛选工作中组分离筛选到近10种性能较好的耐高温α-淀粉酶产生菌株,并且对其中一种高产耐高温α-淀粉酶的较新颖菌株热反硝化地芽孢杆菌(Geobacillus sp.)进行了专利保藏,相关的菌株诱变、发酵条件优化、酶分子功能评估等工作正在进行之中。
2 耐高温α-淀粉酶的结构与功能 2.1 耐高温α-淀粉酶的结构在碳水化合物活性酶(carbohydrate-active enzymes,CAZy)数据库中,α-淀粉酶与GH-13家族中不同种类的糖基水解酶构成α-淀粉酶家族。该家族酶类作用于α-糖苷键并水解,以产生α-异头单糖或寡糖,或者形成α-1, 4或1, 6糖苷键(转糖基化),或者显示两种活性。
不同来源耐高温α-淀粉酶的一级结构一般不同,甚至氨基酸的相异性达到了70%以上(如P. woesei和地衣芽孢杆菌的相异性为71%[35]),但是不同来源耐高温α-淀粉酶的三级结构却高度相似,揭示α-淀粉酶催化活性的高低与三级结构有着更密切的关系。
就三级结构而言,所有耐高温α-淀粉酶均包含3个结构域,即结构域A、结构域B和结构域C。结构域A是由高度对称的α-螺旋和β-折叠交替组成的α/β桶装结构,该折叠结构包括8个平行的β-折叠体排列在一个桶中,边界为8个α-螺旋。结构域A充当催化结构域,酶蛋白质的N末端包含结构域A。此区域表现出较为刚性的特点,用于维持酶的基本构象。结构域B是一个小的突出环,位于TIM桶的第三β-折叠和第三α-螺旋之间。其具有不规则的β-片状结构,形成大的底物结合裂缝,并在不同的α-淀粉酶之间变化。结构域B被认为在α-淀粉酶的底物特异性差异中起作用。耐高温α-淀粉酶的催化活性口袋在结构域A和B之间,位于α/β桶状结构的底部。结构域C则构成α-淀粉酶的C端,其在蛋白质的稳定、折叠和底物结合中起着重要的作用[36]。底物结合位点即蛋白质的活性位点位于结构域A和B的羧基末端之间的长裂缝中。不同结构域间的连接及结构如图 1所示,其中A显示不同区域的分布与连接,B显示A区结构的α/β桶结构,催化位点残基位于其中。这类酶的一级序列具有7个高度保守的催化氨基酸区域,这些氨基酸序列有助于维持保守的TIM桶拓扑结构的稳定。
|
| 图 1 α-淀粉酶的结构 Figure 1 Structure of α-amylase 注:A:α-淀粉酶的结构域组织[37];B:结构域A α/β桶(TIM桶)的示意图[38].结构域A以绿色显示,结构域B以红色显示,结构域C以青色显示. Note: A: The domain organization of α-amylase[37]; B: Schematic representation of the α/β barrel (TIM barrel) of domain A[38]. Domain A is shown in green, domain B is shown in magenta, and domain C is shown in cyan. |
|
|
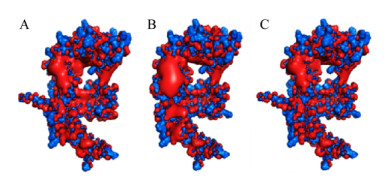
TIM桶包含4个高度保守的区域,分别位于β链3、β链4、β链5和连接β链7与α-螺旋7的回路中(图 2)。第一区域位于具有与底物的葡萄糖残基相互作用的His残基的β链3的C末端;第二区域具有Asp残基的β链4,其在催化过程中充当亲核试剂;第三区域则含有作为质子供体或受体的带有Glu残基的β链5;第四区域带有His残基和可能与底物的葡萄糖残基形成氢键的Asp残基的β链7[36]。
耐高温α-淀粉酶的酶活性与金属离子有着密切的关系。α-淀粉酶是一种金属活化酶,对Ca2+等阳离子具有高亲和力。一分子的α-淀粉酶内部至少含有一个Ca2+,用于维持酶的结构稳定和催化活性。通常认为,Ca2+的存在对耐高温α-淀粉酶的催化活性有着激活的作用。Li等[39]研究表明当耐高温α-淀粉酶AMY121加入5 mmol/L Ca2+时,在75 ℃下酶的半衰期从8.25 min增加到693.14 min,酶的热稳定性得到了大幅度提高。
不同来源的芽孢杆菌淀粉酶在结构上较为相似,但在热稳定性上却表现出很大的差别。Li等[39]对比了地衣芽孢杆菌α-淀粉酶(Bacillus licheniformis α-amylase,BLA)和解淀粉芽孢杆菌α-淀粉酶(Bacillus amyloliquefaciens α-amylase,BAA)的结构,发现BLA与BAA两者氨基酸序列虽具有80%的同一性,但热稳定性却有较显著差异。BAA来源的α-淀粉酶热稳定结构域B较BLA来源的α-淀粉酶的结构域B在209及210位点多出2个氨基酸残基Glu-Gly,但前者的热稳定性显著低于后者。当在BLA的B结构域208、209位点插入Glu-Gly后,突变体BLA的α-淀粉酶热稳定性显著降低,而将该突变体所插入的Glu-Gly去除后,α-淀粉酶热稳定性又恢复如初。可见结构域B中类似位点仅有2个氨基酸的差异即可导致两种芽孢杆菌来源的α-淀粉酶热稳定性的显著差异。这种通过对关键位置氨基酸缺失或定点突变的研究方法,有助于阐明耐高温α-淀粉酶的耐热机理,也为提高芽孢杆菌嗜温α-淀粉酶的热稳定性提供了一种新思路。
通过比较嗜冷、嗜温、嗜热和极端嗜热生物体同源蛋白质稳定性的研究结果发现,天然蛋白质热稳定性的调整似乎是通过积累众多有限个体影响的突变而发生的[40]。在研究巨大芽孢杆菌(B. megaterium) α-淀粉酶耐热机制时,也可通过对钙离子结合位点的点突变实现耐热功能的显著提高。Ghollasi等[41]通过QuikChange定点诱变,用H. orenii耐高温α-淀粉酶(AmyA)的等同位置替代巨大芽孢杆菌WHO α-淀粉酶(Bacillus megaterium WHO α-amylase)假设钙结合环中的残基,如图 3所示,仅通过3个氨基酸取代实现了α-淀粉酶稳定的嗜热突变。该研究结果证实Ca2+的结合对活性和稳定性同时起作用,更紧密的钙结合增加了酶对热变性的抵抗力;通过操纵钙结合位点也可以改善α-淀粉酶的热稳定性。
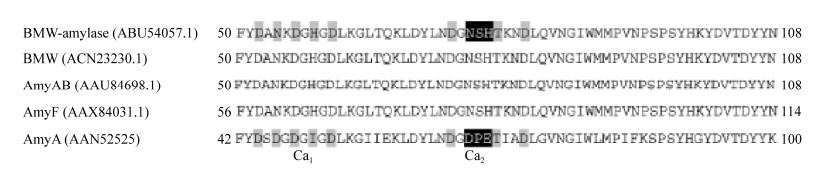
|
| 图 3 GenBank和Swiss-Prot数据库中BMW-淀粉酶和相关蛋白的N末端结构域的多重比对[41] Figure 3 Multiple alignments of N-terminal domain of BMW-amylase and related proteins in GenBank and Swiss-Prot databases[41] 注:氨基酸参与钙结合位点、选择诱变的氨基酸分别用灰色和黑色框标记,BMW-淀粉酶来自巨大芽孢杆菌WHO (ABU54057.1),AmyA来自H. orenii (AAN52525). Note: The amino acids participate in the calciumbinding sites and the ones selected for mutagenesis are marked by gray and black boxes, respectively. BMW-amylase is from B. megaterium WHO (ABU54057.1) and AmyA is from H. orenii (AAN52525). |
|
|
随着分子生物学技术手段在α-淀粉酶研究中的广泛应用以及生物信息学工具的建立,对不同来源α-淀粉酶结构域序列的系统发育分析比较成为可能[42]。Huma等[43]选择古细菌P. furiosus、5个芽孢杆菌(地衣芽孢杆菌、嗜热脂肪地芽孢杆菌、解淀粉芽孢杆菌、枯草芽孢杆菌和B. KSM K38)以及真核细菌米曲霉等7种微生物物种来源的α-淀粉酶进行了分子序列数据的比较分析。尽管发现了淀粉酶分子中3个保守区域,但以NCBI的BioEdit进行的生物信息学分析并没有揭示出分类上接近的芽孢杆菌淀粉酶在耐热序列分布上的相关性。可见,α-淀粉酶耐热功能可能也像前述的催化功能一样,更多取决于酶蛋白的空间结构而不是特定区域氨基酸序列。另外,来自枯草芽孢杆菌的α-淀粉酶在70 ℃具有最佳活性,与嗜热的古细菌激烈热球菌的α-淀粉酶具有更多的序列相似性,它们的主要结构可以在耐热的激烈热球菌α-淀粉酶的基础上进行利用以改变其内在性和稳定性。通过比对大多数热稳定α-淀粉酶的蛋白序列特性,可以在热稳定性较低的α-淀粉酶中引入新的保守热稳定氨基酸序列或优化改进的序列,从而使后者具有更高的耐热性和生产性。随着用于比较分析DNA和蛋白质序列的系统发育方法技术的成熟,分子序列比较及蛋白空间结构预测技术得以相互关联,在此基础上形成用以改善酶催化活性及热稳定性的新的潜在技术手段。
3 耐高温α-淀粉酶的基因工程及外源表达数十年来,耐高温α-淀粉酶工业历经野生菌株发酵、培养基优化及工艺改进等传统技术更新,逐步形成完整的工艺体系。进入21世纪后,引入外源基因、构建重组高效工程菌株并实现耐高温α-淀粉酶的改良表达已成为行业巨头取胜的法宝。用于淀粉酶生产的重组DNA技术之一是选择有效的淀粉酶基因并将基因插入合适的载体系统,且在有效的细菌系统中转化以产生过量表达的重组蛋白[44]。在该技术中,基因的高拷贝数往往能促进淀粉酶的更高产率[45]。
随着基因工程技术的不断发展,构建原核系统外源表达成为了研究的主流。目前研究较多的是将野生菌中的耐高温α-淀粉酶基因导入到外源表达系统中,以此来提高耐高温α-淀粉酶酶活,或根据自己的需要构建筛选出性能良好的菌株。Jiang等[46]将Geobacillus sp. 4j的热稳定α-淀粉酶基因转化到大肠杆菌BL21中进行表达,产量增加了22倍。Ghasemi[47]将来自P. woesei的耐高温α-淀粉酶基因克隆到pTYB2载体并导入大肠杆菌BL21中,成功使酶活提高到185 U/mL。董晨等[48]比较了两种启动子对地衣芽孢杆菌耐高温α-淀粉酶基因在枯草芽孢杆菌A752S中表达水平的差异,研究表明强启动子P43可使耐高温α-淀粉酶表达水平提高8.9倍。刘金岚等[49]将来源于嗜热脂肪地芽孢杆菌NUB3621的耐高温α-淀粉酶基因导入到枯草芽孢杆菌1A751中并进行发酵,酶活可高达1 393.3 U/mL。
相较于原核表达系统,真核表达系统的发展则较为缓慢。但是随着研究者们对于真核生物遗传表达机制研究的不断深入,以及科技水平的不断提高,人们已经开始在昆虫、酵母、哺乳动物细胞中实现了外源基因的表达。一般来说,来源于高等生物的外源基因更适合在真核生物表达系统中表达[8]。毕赤酵母(Pichia pastoris)表达系统因其蛋白产量大、活性高、稳定性和安全性好等优势,近年来得到了充分的开发利用。柯涛等[50]把密码子优化后的超耐热酸性α-淀粉酶基因BD5088转化到毕赤酵母GS115中,使其最适反应温度提高了10 ℃,热稳定性也得到了较大的提高。He等[31]采用RT-PCR从嗜热真菌R. pusillus中克隆耐热α-淀粉酶基因,导入到P. pastoris KM71中进行异源表达,使得酶活最高可达32 100 U/mL。P. furiosus属于极端嗜热厌氧古细菌,需要严格无氧环境,一般原核表达系统都难以实现有效的分泌表达。郭建强等[51]将P. furiosus编码的耐热酸性α-淀粉酶基因在常温好氧的P. pastoris中实现了高效表达,构建了可以在常温有氧的条件下通过优化培养条件、改善生产工艺即可大规模培养获取外源蛋白表达量的基因工程菌。郝帅等[52]将P. furiosus超耐热酸性α-淀粉酶Amy转化到多型汉逊酵母HP-6中获得重组汉逊酵母,使得更容易筛选到高拷贝转化子,更加适用于大规模工业发酵。
要在给定的表达系统中取得淀粉酶的过量表达,规避代谢阻抑有着重要意义。多数淀粉酶的生物合成以糖类物质作为碳源,易受糖分解代谢物阻遏(catabolite repression,CR)。在发酵初期积累的分解代谢产物(如麦芽糖和葡萄糖)往往会导致淀粉酶生物合成受到分解代谢物抑制。因此,设计能规避CR的表达系统是淀粉酶大规模商业化生产中的一种重要策略。Nathan等[53]以地衣芽孢杆菌MSG耐高温α-淀粉酶作为模型,设计了一种来自强异源启动子的分解代谢物无抑制过表达方案,开发了自身可诱导、无分解代谢物抑制的表达系统。构建表达系统时,采用地衣芽孢杆菌CR关键5′cre操纵子缺失的淀粉酶基因与枯草芽孢杆菌磷酸盐饥饿诱导型强pst启动子融合的策略如图 4所示,从而规避葡萄糖抑制并增强淀粉酶的过量表达。在验证试验中,当培养基中加入高葡萄糖并提供有限磷酸盐时,该表达系统在枯草芽孢杆菌中的表达量比野生型菌株提高约300倍,比同基因克隆中天然启动子的产量高18.5倍。
α-淀粉酶外源过量表达成为淀粉酶工业中工程菌构建的重要关注点。必须指出的是,工程菌株的过量表达仅为提高产量可能性提供了保障,要实现这种可能尚需保障所合成的淀粉酶能分泌到胞外,这才是实现产量提高最为关键的一步。为了获得工业上所需的酶产量,至关重要的是确定从转录、翻译到折叠和分泌的蛋白质生产路线中的潜在瓶颈。如果蛋白质在转运后没有快速正确折叠,它们会被存在于膜、细胞壁或细胞外培养基周围的几种蛋白酶降解。Quesada-Ganuza等研究表明[54],为保障α-淀粉酶的有效分泌,外源表达系统中有必要引入PrsA蛋白并与α-淀粉酶共表达。PrsA是一种负责芽孢杆菌胞外蛋白折叠的必不可少的胞外折叠催化剂蛋白。Quesada-Ganuza等[54]着手以枯草芽孢杆菌为宿主,将6个异源淀粉酶分别与6个异源PrsA共表达,构建了包含6个淀粉酶-PrsA对所有组合的菌株矩阵,他们的研究表明,异源PrsA共表达能将5种细胞外α-淀粉酶的最终产量提高2.5倍,并且异源PrsA的过表达对α-淀粉酶分泌的影响对共表达的α-淀粉酶具有特异性。Quesada-Ganuza等[54]还将2个PrsA进行重组,使PrsAB1的6个氨基酸用PrsABN的同位取代,产生新的重组PrsA称为PrsAB1-BN,如图 5所示。重组PrsAB1-BN突变体与α-淀粉酶共表达,与天然PrsAB1和PrsABN折叠酶相比,表面重塑的PrsAB1-BN折叠酶增加了总淀粉酶活性,并且降低了其分泌压力。总的来说,异源PrsA过表达能促进异源淀粉酶分泌,良好的PrsA-淀粉酶配对还可以降低枯草芽孢杆菌的分泌应激反应。
经过半个多世纪的发展,α-淀粉酶产业已从最初的野生菌株分批式发酵生产模式跨入由代谢工程以及蛋白质工程催生的精准调控的工程菌可控式规模化生产阶段[8]。如今,丹麦、美国、日本等国家在芽孢杆菌耐高温α-淀粉酶产业领域仍领先于其他国家。近年来,国内学术界及产业界在诱变育种、工程菌构建以及生产工艺优化等方面进行了长期的研发及开发,使耐高温α-淀粉酶产业得以显著提升和发展。就产业规模而言,也已形成与跨国企业分庭抗礼的态势。但目前纵览国内本土单个生产企业,尚无一家可与国际龙头企业匹敌。如何开发高质量、热稳定性更高的耐高温α-淀粉酶分子、如何构建过量表达的优良工程菌株以及如何开发先进的生产工艺以有效提高生产效率及降低生产成本,仍是我国淀粉酶业界需要不断努力的方向。
在重组耐高温α-淀粉酶酶活性方面如何减弱对Ca2+的依赖以使耐高温α-淀粉酶能够更加广泛、灵活地用于各个领域且适应各种条件,也是亟待解决的主要关键点。超嗜热古细菌一般耐受温度较高,pH稳定范围也比较广泛,特别是其热稳定性并不依赖Ca2+,比如P. woesei[47]、Thermococcus siculi[55]等超嗜热古细菌的耐高温α-淀粉酶基因工程菌的构建已有报道,或许可成为有潜力的耐高温α-淀粉酶基因来源。
耐高温α-淀粉酶的研发如基因工程菌的构建、代谢工程的应用等方面,分子生物学技术主导的精确控制手段将成为研发热点。例如地衣芽孢杆菌和枯草芽孢杆菌的α-淀粉酶启动子在葡萄糖存在下会被抑制10–15倍,使用顺式调节序列的分解代谢物阻遏(CR)抗性突变体或反式调节蛋白如CcpA,淀粉酶表达也仅增加3–10倍[53]。在此情况下,设计无抑制的分解代谢物增强的表达系统、废除CR的策略成为酶过量表达生产中的重要考虑因素。经研究[54],消除这一抑制因素表达量将会比同基因克隆增强数倍,然而目前对这一方向的研究较少,实现无抑制、生长解离且经济高效地生产碳抑制酶和其他重组蛋白是亟待解决的关键环节之一。
必须指出的是,工程菌株的过量表达仅是耐高温α-淀粉酶产量提高的必要条件,要实现这种可能,尚需保障所合成的淀粉酶能分泌到胞外,这才是实现产量提高的最为关键的一步。为了获得工业上所需的酶产量,至关重要的是确定从转录、翻译到折叠和分泌的蛋白质生产路线中的潜在瓶颈,异源蛋白生产遇到的瓶颈在不同阶段均有发现,而克服这一瓶颈也是未来需要致力的另一个关键环节。
综上所述继续筛选性能更好的耐高温α-淀粉酶分子、加强优良工程菌的构建以及基于代谢工程设计的高效外源表达体系的构建及规模化上下游工艺的优化将是一项长期任务[56]。相信随着代谢工程及蛋白质工程技术的进步及新生产工艺的革新,我国的淀粉酶行业,特别是高温α-淀粉酶的研发将迎来光明的前景。
| [1] |
Sun JL. Enzyme Preparation Technology[M]. Beijing: Science Press, 2004: 1. (in Chinese) 孙俊良. 酶制剂生产技术[M]. 北京: 科学出版社, 2004: 1. |
| [2] |
Yin Y, Qu JH, Li HF, et al. Research progress in the acid-resistant thermostable α-amylase and strain breeding[J]. Science and Technology of Cereals, Oils and Foods, 2015, 23(5): 101-105. (in Chinese) 尹伊, 屈建航, 李海峰, 等. 耐酸耐高温α-淀粉酶及其菌种选育研究进展[J]. 粮油食品科技, 2015, 23(5): 101-105. |
| [3] |
Zhang YR, Zhang XY, Tian T, et al. Research progress of acid-stable α-amylase[J]. Journal of Jining Medical University, 2015, 28(5): 310-312, 317. (in Chinese) 张玉然, 张晓云, 田田, 等. 酸性α-淀粉酶研究进展[J]. 济宁医学院学报, 2015, 28(5): 310-312, 317. |
| [4] |
Varalakshmi KN, Tamrakar B, Kumari K, et al. Purification and biochemical characterization of pH tolerant and acid stable α-amylase from Aspergillus oryzae JGI 21 isolated from soil[J]. Croatian Journal of Food Technology, Biotechnology and Nutrition, 2013, 8(1/2): 14-20. |
| [5] |
van der Maarel MJEC, van der Veen B, Uitdehaag JCM, et al. Properties and applications of starch-converting enzymes of the α-amylase family[J]. Journal of Biotechnology, 2002, 94(2): 137-155. |
| [6] |
Reddy NS, Nimmagadda A, Sambasiva KRS. An overview of the microbial α-amylase family[J]. African Journal of Biotechnology, 2003, 2(12): 645-648. |
| [7] |
Rana N, Walia A, Gaur A. α-amylases from microbial sources and its potential applications in various industries[J]. National Academy Science Letters, 2013, 36(1): 9-17. |
| [8] |
Saha BC, Zeikus JG. Biotechnology of maltose syrup production[J]. Process Biochemistry, 1987, 22(3): 78-82. |
| [9] |
Weickert MJ, Chambliss GH. Site-directed mutagenesis of a catabolite repression operator sequence in Bacillus subtilis[J]. Proceedings of the National Academy of Sciences of the United States of America, 1990, 87(16): 6238-6242. |
| [10] |
Wang P, Wang PL, Tian J, et al. A new strategy to express the extracellular α-amylase from Pyrococcus furiosus in Bacillus amyloliquefaciens[J]. Scientific Reports, 2016, 6: 22229. |
| [11] |
Bejuki WM. Developments in industrial microbiology[J]. AIBS Bulletin, 1960, 10(6): 36-37. |
| [12] |
Bolton DJ, Kelly CT, Fogarty WM. Purification and characterization of the α-amylase of Bacillus flavothermus[J]. Enzyme and Microbial Technology, 1997, 20(5): 340-343. |
| [13] |
El-Aassar SA, Omar SH, Gouda MK, et al. Purification of α-amylase from Bacillus lentus cultures[J]. Applied Microbiology and Biotechnology, 1992, 38(3): 312-314. |
| [14] |
Wu XR, Wang YX, Tong BD, et al. Purification and biochemical characterization of a thermostable and acid-stable alpha-amylase from Bacillus licheniformis B4-423[J]. International Journal of Biological Macromolecules, 2018, 109: 329-337. |
| [15] |
Vihinen M, Mäntsälä P. Conserved residues of liquefying α-amylases are concentrated in the vicinity of active site[J]. Biochemical and Biophysical Research Communications, 1990, 166(1): 61-65. |
| [16] |
Vihinen M, Mäntsälä P. Characterization of a thermostable Bacillus stearothermophilus alpha-amylase[J]. Biotechnology and Applied Biochemistry, 1990, 12(4): 427-435. |
| [17] |
Canganella F, Andrade CM, Antranikian G. Characterization of amylolytic and pullulytic enzymes from thermophilic archaea and from a new Fervidobacterium species[J]. Applied Microbiology and Biotechnology, 1994, 42(2/3): 239-245. |
| [18] |
Ratanakhanokchai K, Kaneko J, Kamio Y, et al. Purification and properties of a maltotetraose- and maltotriose-producing amylase from Chloroflexus aurantiacus[J]. Applied and Environmental Microbiology, 1992, 58(8): 2490-2494. |
| [19] |
Ikehata O, Nishiyama M, Horinouchi S, et al. Primary structure of nitrile hydratase deduced from the nucleotide sequence of a Rhodococcus species and its expression in Escherichia coli[J]. European Journal of Biochemistry, 1989, 181(3): 563-570. |
| [20] |
Sudan SK, Narender K, Ishwinder K, et al. Production, purification and characterization of raw starch hydrolyzing thermostable acidic α-amylase from hot springs, India[J]. International Journal of Biological Macromolecules, 2018, 117: 831-839. |
| [21] |
Fincan SA, Enez B. Production, purification, and characterization of thermostable α-amylase from thermophilic Geobacillus stearothermophilus[J]. Starch-Stärke, 2014, 66(1/2): 182-189. |
| [22] |
Tan TC, Yien YY, Patel BKC, et al. Crystallization of a novel α-amylase, AmyB, from the thermophilic halophile Halothermothrix orenii[J]. Acta Crystallographica Section D: Biological Crystallography, 2003, 59(12): 2257-2258. |
| [23] |
Burgess-Cassler A, Imam S. Partial purification and comparative characterization of α-amylase secreted by Lactobacillus amylovorus[J]. Current Microbiology, 1991, 23(4): 207-213. |
| [24] |
Giraud E, Gosselin L, Marin B, et al. Purification and characterization of an extracellular amylase from Lactobacillus plantarum strain A6[J]. Journal of Applied Bacteriology, 1993, 75(3): 276-282. |
| [25] |
Prieto JA, Bort BR, Martínez J, et al. Purification and characterization of a new α-amylase of intermediate thermal stability from the yeast Lipomyces kononenkoae[J]. Biochemistry and Cell Biology, 1995, 73(1/2): 41-49. |
| [26] |
Roy SK, Raha SK, Dey SK, et al. Effect of temperature on the production and location of cellulase components in Myceliophthora thermophila D-14 (ATCC 48104)[J]. Enzyme and Microbial Technology, 1990, 12(9): 710-713. |
| [27] |
Laderman KA, Asada K, Uemori T, et al. α-amylase from the hyperthermophilic archaebacterium Pyrococcus furiosus. Cloning and sequencing of the gene and expression in Escherichia coli[J]. Journal of Biological Chemistry, 1993, 268(32): 24402-24407. |
| [28] |
El-Hadef El-Okki AAK, Gagaoua M, Bennamoun L, et al. Statistical optimization of thermostable α-amylase production by a newly isolated Rhizopus oryzae strain FSIS4 using decommissioned dates[J]. Waste and Biomass Valorization, 2017, 8(6): 2017-2027. |
| [29] |
Koch R, Spreinat A, Lemke K, et al. Purification and properties of a hyperthermoactive α-amylase from the archaeobacterium Pyrococcus woesei[J]. Archives of Microbiology, 1991, 155(6): 572-578. |
| [30] |
Niehaus F, Bertoldo C, Kähler M, et al. Extremophiles as a source of novel enzymes for industrial application[J]. Applied Microbiology and Biotechnology, 1999, 51(6): 711-729. |
| [31] |
He ZG, Yin YC, Mao YZ, et al. Cloning and high-level expression of thermostable α-amylase gene from Rhizomucor pusillus[J]. Journal of Southern Agriculture, 2014, 45(2): 165-172. |
| [32] |
Gomes I, Gomes J, Steiner W. Highly thermostable amylase and pullulanase of the extreme thermophilic eubacterium Rhodothermus marinus: production and partial characterization[J]. Bioresource Technology, 2003, 90(2): 207-214. |
| [33] |
Aquino ACMM, Jorge JA, Terenzi HF, et al. Studies on a thermostable α-amylase from the thermophilic fungus Scytalidium thermophilum[J]. Applied Microbiology and Biotechnology, 2003, 61(4): 323-328. |
| [34] |
Zafar A, Aftab MN, Iqbal I, et al. Pilot-scale production of a highly thermostable α-amylase enzyme from Thermotoga petrophila cloned into E. coli and its application as a desizer in textile industry[J]. RSC Advances, 2019, 9(2): 984-992. |
| [35] |
Bi JF, Dong FK. Studies on enzyme properties of heat-resisting α-amylase[J]. Food Science, 2006, 27(2): 125-128. (in Chinese) 毕金峰, 董福奎. 耐高温α-淀粉酶的酶学性质研究[J]. 食品科学, 2006, 27(2): 125-128. |
| [36] |
Nielsen JE, Borchert TV. Protein engineering of bacterial α-amylases[J]. Biochimica et Biophysica Acta (BBA) - Protein Structure and Molecular Enzymology, 2000, 1543(2): 253-274. |
| [37] |
Prakash O, Jaiswal N. A-amylase: an ideal representative of thermostable enzymes[J]. Applied Biochemistry and Biotechnology, 2010, 160(8): 2401-2414. |
| [38] |
Mehta D, Satyanarayana T. Bacterial and archaeal α-amylases: diversity and amelioration of the desirable characteristics for industrial applications[J]. Frontiers in Microbiology, 2016, 7: 1129. |
| [39] |
Li LZ, Yang J, Li J, et al. Role of two amino acid residues' insertion on thermal stability of thermophilic α-amylase AMY121 from a deep sea bacterium Bacillus sp. SCSIO 15121[J]. Bioprocess and Biosystems Engineering, 2015, 38(5): 871-879. |
| [40] |
Declerck N, Machius M, Joyet P, et al. Hyperthermostabilization of Bacillus licheniformis α-amylase and modulation of its stability over a 50 ℃ temperature range[J]. Protein Engineering, Design and Selection, 2003, 16(4): 287-293. |
| [41] |
Ghollasi M, Ghanbari-Safari M, Khajeh K. Improvement of thermal stability of a mutagenised α-amylase by manipulation of the calcium-binding site[J]. Enzyme and Microbial Technology, 2013, 53(6/7): 406-413. |
| [42] |
Arikan B. Highly thermostable, thermophilic, alkaline, SDS and chelator resistant amylase from a thermophilic Bacillus sp. isolate A3-15[J]. Bioresource Technology, 2008, 99(8): 3071-3076. |
| [43] |
Huma T, Maryam A, Rehman SU, et al. Phylogenetic and comparative sequence analysis of thermostable alpha amylases of kingdom archea, prokaryotes and eukaryotes[J]. Bioinformation, 2014, 10(7): 443-448. |
| [44] |
Gopinath SCB, Anbu P, Arshad MKM, et al. Biotechnological processes in microbial amylase production[J]. BioMed Research International, 2017, 2017: 1272193. |
| [45] |
Son YJ, Ryu AJ, Li L, et al. Development of a high-copy plasmid for enhanced production of recombinant proteins in Leuconostoc citreum[J]. Microbial Cell Factories, 2016, 15: 12. |
| [46] |
Jiang T, Cai MH, Huang MM, et al. Characterization of a thermostable raw-starch hydrolyzing α-amylase from deep-sea thermophile Geobacillus sp.[J]. Protein Expression and Purification, 2015, 114: 15-22. |
| [47] |
Ghasemi A, Ghafourian S, Vafaei S, et al. Cloning, expression, and purification of hyperthermophile α-amylase from Pyrococcus woesei[J]. Osong Public Health and Research Perspectives, 2015, 6(6): 336-340. |
| [48] |
Dong C, Cao J, Zhang J, et al. High-level expression of thermostable α-amylase in Bacillus subtilis[J]. Chinese Journal of Applied & Environmental Biology, 2008, 14(4): 534-538. (in Chinese) 董晨, 曹娟, 张迹, 等. 耐高温α-淀粉酶基因在枯草芽孢杆菌中的高效表达[J]. 应用与环境生物学报, 2008, 14(4): 534-538. |
| [49] |
Liu JL, Fu G, Dong HN, et al. Optimization of thermostable α-amylase secretion by screening of optimal signal peptide in Bacillus subtilis[J]. Industrial Microbiology, 2017, 47(1): 17-23. (in Chinese) 刘金岚, 付刚, 董会娜, 等. 通过信号肽筛选优化耐高温α-淀粉酶在枯草芽孢杆菌中的分泌[J]. 工业微生物, 2017, 47(1): 17-23. |
| [50] |
Ke T, Xiong L, Zhang NQ, et al. Expression of synthetic thermoacidophilic α-amylase gene in Pichia pastoris[J]. Food and Fermentation Industries, 2012, 38(1): 30-35. (in Chinese) 柯涛, 熊蓝, 张乃群, 等. 合成嗜热酸性淀粉酶的毕赤酵母表达[J]. 食品与发酵工业, 2012, 38(1): 30-35. |
| [51] |
Guo JQ, Li YM, Yue LL, et al. Molecular cloning and expression of extremely thermostable and acid-stable amylase gene in Pichia pastoris[J]. Chinese Journal of Biotechnology, 2006, 22(2): 237-242. (in Chinese) 郭建强, 李运敏, 岳丽丽, 等. 超耐热酸性α-淀粉酶基因的克隆及其在酵母细胞中的表达[J]. 生物工程学报, 2006, 22(2): 237-242. |
| [52] |
Hao S, Guo JQ, Li YM, et al. Comparison between the expression of extremely thermostable and acid-stable amylase gene in Hansenula polymorpha and that in Pichia pastoris[J]. Mycosystema, 2006, 25(4): 573-578. (in Chinese) 郝帅, 郭建强, 李运敏, 等. 超耐热酸性α-淀粉酶基因在多型汉逊酵母中表达及与在毕赤酵母中表达的比较研究[J]. 菌物学报, 2006, 25(4): 573-578. |
| [53] |
Nathan S, Nair M. Engineering a repression-free catabolite-enhanced expression system for a thermophilic alpha-amylase from Bacillus licheniformis MSG[J]. Journal of Biotechnology, 2013, 168(4): 394-402. |
| [54] |
Quesada-Ganuza A, Antelo-Varela M, Mouritzen JC, et al. Identification and optimization of PrsA in Bacillus subtilis for improved yield of amylase[J]. Microbial Cell Factories, 2019, 18(1): 158. |
| [55] |
Yao T. Site-directed mutation of gene encoding hypertherthermophiles acid-stable α-amylase from Thermococcus siculi HJ21[D]. Wuxi: Master's Thesis of Jiangnan University, 2011 (in Chinese) 姚婷.超嗜热古菌Thermococcus siculi HJ21高温酸性α-淀粉酶基因的定向改造[D].无锡: 江南大学硕士学位论文, 2011 http://cdmd.cnki.com.cn/Article/CDMD-10295-1011209351.htm |
| [56] |
Ozturk MT, Akbulut N, Oztürk SI, et al. Ligase-independent cloning of amylase gene from a local Bacillus subtilis isolate and biochemical characterization of the purified enzyme[J]. Applied Biochemistry and Biotechnology, 2013, 171(2): 263-278. |
 2020, Vol. 47
2020, Vol. 47