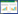扩展功能
文章信息
- 颉兵兵, 刘君, 优丽图孜·乃比, 张春博
- XIE Bing-Bing, LIU Jun, Youlituzi Naibi, ZHANG Chun-Bo
- 西瓜食酸菌抗铜基因cueR的生物信息学分析及功能验证
- Bioinformatics analysis and functional verification of copper resistance gene cueR in Acidovorax citrulli
- 微生物学通报, 2020, 47(5): 1534-1543
- Microbiology China, 2020, 47(5): 1534-1543
- DOI: 10.13344/j.microbiol.china.190679
-
文章历史
- 收稿日期: 2019-08-18
- 接受日期: 2019-11-11
- 网络首发日期: 2019-11-29
2. 新疆农业大学林学与园艺学院 新疆 乌鲁木齐 830052
2. College of Forestry and Horticulture, Xinjiang Agricultural University, Urumqi, Xinjiang 830052, China
革兰氏阴性细菌——西瓜食酸菌(Acidovorax citrulli)[1]可以导致葫芦科植物产生细菌性果斑病(bacterial fruit blotch,BFB)。该病害在高温高湿环境下可以使西瓜、甜瓜以及黄瓜等葫芦科植物发生病斑、萎蔫或果实腐烂[2],是一种可通过种子传播的毁灭性病害。在世界经济一体化的大背景下,该病害已通过全球贸易活动在很多个国家传播开来。在一些商品西瓜地,由BFB导致的经济损失在50%甚至90%以上[3-5]。有研究证实[6-8],BFB的病菌群体中至少存在两个在遗传和生化上有差异的亚群——亚群Ⅰ和亚群Ⅱ。亚群Ⅰ菌株多数对铜不敏感,这表明目前以含铜制剂为主要杀菌剂的防控措施对BFB基本无效。至今为止,尚没有发现抗BFB的种质资源。因此,研究该细菌的抗铜机制,可以为开发有效防控BFB的新方法、新途径提供理论依据。
西瓜食酸菌与大肠杆菌等大多数微生物一样,其代谢过程和呼吸酶需要极少量的铜[9];与此同时,这些生物必须将细胞内的铜含量维持在有毒阈值以下[9]。因此,微生物细胞需要一系列的铜稳态机制来控制铜离子的吸收与外排[9]。Cue系统是大肠杆菌重要的铜离子外排泵之一,在细菌铜稳态中起重要作用。该系统中的转录调控因子CueR与P型ATP酶(CopA)和多铜氧化酶(CueO)共同参与铜离子的调控与转运[10]。2013年赵文龙的研究证实,西瓜食酸菌中的类P型ATP酶(Aave-0034)与该菌的抗铜性相关[11];2014年李强的实验确认注释为多铜氧化酶的基因cueO (Aave-1810)是该菌的抗铜性相关基因[12]。基于以上研究发现可以推测,在西瓜食酸菌中可能存在类似Cue的抗铜系统。本研究以已经鉴定的大肠杆菌等细菌的CueR为参照,利用生物信息学手段分析西瓜食酸菌中调控copA和cueO基因表达的因子CueR,并对该基因进行功能验证,旨在为进一步研究西瓜食酸菌的类Cue系统的机制奠定基础。
1 材料与方法 1.1 实验材料本研究中的CueR蛋白分别来自大肠杆菌(Escherichia coli)、铜绿假单胞菌(Pseudomonas aeruginosa)、沙门氏菌(Salmonella enterica)、霍乱弧菌(Vibrio cholerae)及西瓜食酸菌FC440 (Acidovorax citrulli FC440),其中各参考菌的氨基酸序列来自于NCBI GenBank (www.ncbi.nlm.nih.gov/genbank/) (表 1),西瓜食酸菌FC440由南京农业大学胡白石教授惠赠。
| 蛋白* Protein |
类型 Description |
物种名称 Species |
残基数 Numbers of residue |
蛋白ID Protein ID |
来源 Sources |
| AcCueR | MerR family transcriptional regulator | Acidovorax citrulli FC440 | 143 | QDD67396 | This study |
| EcCueR | DNA-binding transcriptional regulator CueR | Escherichia coli str. K-12 substr. MG1655T | 135 | NP-415020.1 | [13] |
| PaCueR | protein CueR | Pseudomonas aeruginosa PAO1T | 132 | NP-253466.1 | [14] |
| SeCueR | Cu(Ⅰ)-responsive transcriptional regulator | Salmonella enterica subsp. enterica Serovar Typhimurium str. LT2T | 138 | NP-459494.1 | [15] |
| VcCueR | MerR family transcriptional regulator | Vibrio cholera O1 biovar E1 Tor str. N16961T | 139 | NP-230621.1 | [16] |
| 注:*:命名规则为属名与种名的首字母加CueR蛋白.
Note: *: Proteins are named by initials of genus name and species name plus CueR protein. |
|||||
| 菌株、质粒Strains, plasmids | 特性Characteristics | 来源Sources |
| Strains | ||
| Acidovorax citrulli strains | ||
| FC440 | AmpR; Wild type | Our lab |
| FC440(∆cueR) | AmpR; KanR; FC440 mutant defective in cueR | This study |
| FC440(∆cueR-cueR) | AmpR; KanR; GmR; FC440(∆cueR) complemented with cueR gene expressed by vector pBBR1MCS-5 | This study |
| E. coli strains | ||
| Trans110 | RpsL(StrR) thr leu thi-1 lacY galK galT are tonA tsx dan dcm supE44∆(lac-proAB) | TIANGEN |
| Trans110(pBBR1MCS-5-cueR) | GmR; E. coli Trans110 strain containing vector pBBR1MCS-5-cueR | This study |
| S17-1 | E. coli 294, thi RP4-2-Tc::Mu-Kan::Tn7 chromosomally integrated | Our lab |
| S17-1(pK19mob2ΩHMB-TY) | KanR; E. coli S17-1 strain containing vector pK19mob2ΩHMB-TY | This study |
| Plasmids | ||
| pMD19-T | AmpR; Cloning vector | TaKaRa |
| pMD19-T-TY | AmpR; pMD19-T vector containing a 286-bp fragment with the cueR gene | This study |
| pK19mob2ΩHMB | KanR; Suicide vector | Our lab |
| pK19mob2ΩHMB-TY | KanR; pK19mob2ΩHMB vector containing a 286-bp fragment with the cueR gene | This study |
| pBBR1MCS-5 | GmR; Expression vector | Our lab |
| pBBR1MCS-5-cueR | GmR; pBBR1MCS-5 vector containing cueR gene; Used to complement FC440(∆cueR) | This study |
| 注:AmpR、KanR、GmR分别代表氨苄青霉素抗性、卡那霉素抗性和庆大霉素抗性.
Note: AmpR, KanR and GmR represent resistant to ampicillin, kanamycin, and tetracycline, respectively. |
||
| Primers name | Primers sequence (5′→3′) | 来源Sources |
| cueR-F | AACTGCAGGTCGATCCCGTGCCCATC (Pst Ⅰ) | This study |
| cueR-R | CGGAATTCGGCCCAGGTCGTCGATG (EcoR Ⅱ) | This study |
| cueR-QF | CCAAGCTTTCCGGCATGGCACAGGC (Hind Ⅲ) | This study |
| cueR-QR | CGGAATTCCGCACGGCGTTCAGTTCAC (EcoR Ⅰ) | This study |
| AAC-1 | GACCAGCCCACAACTGGGAC | [17] |
| AAC-2 | CTGCCGCACTCCAGCGA | [17] |
| 注:下划线表示酶切位点. Note: The restriction enzyme cutting sites were underlined. |
||
Taq酶、EcoR Ⅰ、Hind Ⅲ,宝生物工程(大连)有限公司;T4 DNA ligase,赛默飞世尔科技(中国)有限公司;细菌基因组提取试剂盒、DNA质粒提取试剂盒及DNA Marker,天根生化科技(北京)有限公司;抗生素,Sigma公司;其余化学试剂均购自国药集团化学试剂有限公司。
Eppendorf台式高速离心机,上海安亭科学仪器厂;PCR仪,宝生物工程(大连)有限公司;电泳仪,北京市六一生物科技有限公司;超净工作台,苏州净化设备有限公司;紫外分光光度计,济南赛富生物科技有限公司;凝胶成像分析系统,上海伯建生物科技有限公司。
1.4 培养基及培养条件KMB固体培养基(g/L):蛋白胨20.0,磷酸氢二钾1.5,硫酸镁1.5,琼脂15.0,调pH至7.0。
NB液体培养基(g/L):蛋白胨5.0,牛肉膏3.0,调pH至7.0。
LB固体培养基(g/L):蛋白胨10.0,酵母粉5.0,氯化钠10.0,琼脂16.0,调pH至7.0。
LB液体培养基(g/L):蛋白胨10.0,酵母粉5.0,氯化钠10.0,调pH至7.0。
SOB培养基(g/L):蛋白胨10.0,酵母粉5.0,氯化钠10.0,氯化钾3.2,调pH至7.0。
西瓜食酸菌在KMB固体培养基或NB液体培养基中于28 ℃、220 r/min下培养;大肠杆菌在LB液体、固体或SOB培养基中于37 ℃、220 r/min下培养。本研究中西瓜食酸菌野生型或其衍生菌使用的抗生素浓度为Amp 30 μg/mL,Kan 50 μg/mL,Gm 50 μg/mL。大肠杆菌的培养中使用的抗生素浓度为Gm 20 μg/mL,Amp 100 μg/mL,Kan 50 μg/mL。
1.5 生物信息学分析采用MEGA 7.0构建进化树[18],以邻近法(neighbor-joining)自检(bootstrap) 1 000次以评估其可靠性。应用SMART (http://smart.embl-heidelberg.de/smart/set_mode.cgi?NORMAL=1)工具进行保守结构域预测及分析;利用ProtParam (https://web.expasy.org/protparam/)工具分析蛋白的理化特性以及亲疏水性质;利用PSORT (http://www.cbs.dtu.dk/services/TatP/)预测蛋白质可能亚细胞定位;利用在线软件SWISS-MODEL (https://swissmodel.expasy.org/)对5种CueR蛋白进行3D同源建模;应用String (https://string-db.org/)数据库预测西瓜食酸菌中AcCueR蛋白与其他蛋白质相互作用关系。利用在线软件SoftBerry (http://linux1.softberry.com/)预测细菌CopA的基因转录启动子序列。
1.6 突变体和基因功能互补菌株的制备以野生型全基因组DNA为模板,使用引物cueR-F/cueR-R (表 3)扩增得到cueR基因约300 bp的同源片段。PCR反应体系:2×Primer STAR GC Buffer (Mg2+) 10 μL,dNTPs (10 mmol/L) 1.6 μL,上、下游引物(10 μmol/L)各1 μL,模板DNA (0.03 mg/L) 1 μL,Primer STARTMHS DNA Polymerase (2.5 U/L) 0.2 μL,ddH2O补至20 μL。PCR反应条件:95 ℃ 5 min;98 ℃ 10 s,68 ℃ 30 s,30个循环;72 ℃ 10 min;4 ℃保存。通过回收、连接转化技术得到自杀质粒pK19mob2ΩHMB-TY。采用双亲杂交法获得基因突变菌株FC440(∆cueR)。使用引物cueR-QF/cueR-QR (表 3)验证目的基因是否被插入突变,即以全基因组DNA为模板进行PCR扩增,PCR反应体系:10×rTaq DNA Buffer 2 μL,MgCl2 (25 mmol/L) 1.6 μL,dNTPs (10 mmol/L) 0.4 μL,cueR-QF和cueR-QR (10 μmol/L)各1 μL,模板DNA (0.03 mg/L) 1 μL,Primer STARTMHS DNA Polymerase (2.5 U/L) 0.2 μL,ddH2O补至20 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 30 s,68 ℃ 30 s,30个循环;72 ℃ 10 min。目的基因被插入突变时,无预期大小片段产生,野生型则有。基因功能互补菌株以野生型基因组DNA为模板,使用引物cueR-QF/cueR-QR扩增cueR基因全长。PCR反应体系:2×Primer STAR GC Buffer (Mg2+) 10 μL,dNTPs (10 mmol/L) 1.6 μL,上、下游引物(10 μmol/L)各1 μL,模板DNA (0.03 mg/L) 1 μL,Primer STARTMHS DNA Polymerase (2.5 U/L) 0.2 μL,ddH2O补至20 μL。PCR反应条件:95 ℃ 5 min;98 ℃ 10 s,68 ℃ 1 min,30个循环;72 ℃ 10 min;4 ℃保存。通过回收、连接转化等步骤得到载体命名为pBBR1MCS-5-cueR;通过双亲杂交法将该载体转入突变菌株FC440(∆cueR)而得到互补菌株FC440(∆cueR-cueR)。
1.7 抗铜性检测用NB培养基将野生型FC440、突变菌株FC440(∆cueR)、互补菌株FC440(∆cueR-cueR) 28 ℃、220 r/min培养12 h,取菌液4 ℃、12 000 r/min离心5 min,去上清,加ddH2O重悬菌体调OD600至0.5,取菌悬液0.2 μL转接至含1.75、2.25、2.75、3.00、3.15、3.30 mmol/L CuSO4的KMB平板上,于28 ℃培养48 h,每隔12 h采集图片。每个浓度至少重复3次。该指标进行了3次生物学重复。
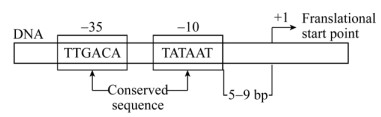
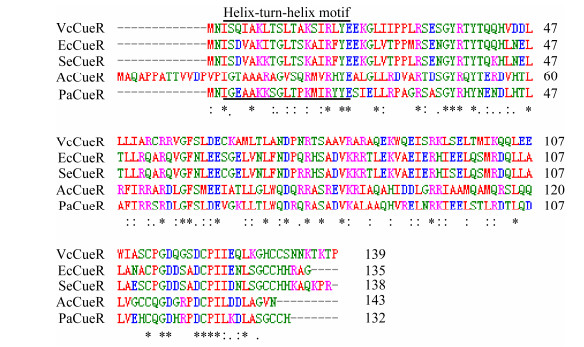
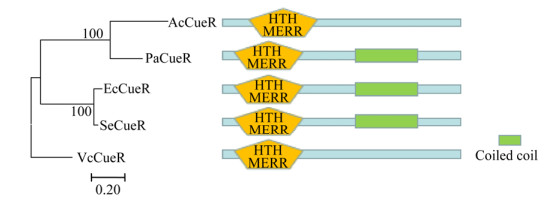
2 结果与分析 2.1 多序列比对及聚类分析从细菌CueR蛋白质序列比对的结果(图 1)可以看出:5种蛋白氨基酸序列具有较高的相似性,均含有保守的Helix-turn-helix (H-T-H) DNA结合基序。细菌CueR蛋白的聚类分析表明:西瓜食酸菌和铜绿假单胞菌的CueR蛋白质序列相似性最高;结构域分析显示出5种蛋白均具有保守的HTH-MerR结构域(图 2)。
|
| 图 1 不同细菌的CueR蛋白多序列比对 Figure 1 Multiple sequence alignment of CueR proteins 注:*表示该列氨基酸完全一致;:表示该列不完全一致,但有高度保守的氨基酸;.表示该列不完全一致,但有一般保守的氨基酸;空白表示该列氨基酸序列差异较大;–表示非同源区域. Note: *: The identical residues; :: The highly conserved residues; .: The conserved residues, while diverse residues are left blank. –: The the non-homologous region. |
|
|
|
| 图 2 细菌CueR蛋白系统进化树及保守结构域 Figure 2 Phylogenetic analysis and domain architecture of CueR protein in bacteria 注:标尺表示遗传距离,0.20表示每100个核苷酸/氨基酸中有20个不同. Note: The scale bar indicates genetic distance, 0.20 indicates 20 differences per 100 nucleotides/amino acids. |
|
|
蛋白特性分析结果显示(表 4):5种蛋白质均为不稳定性蛋白质(< 40稳定;> 40不稳定);由于亲水性平均系数均为负数(< 0亲水;> 0疏水),因此5种CueR蛋白均为亲水性蛋白,且N端均为Met,Met被视为与金属离子结合的氨基酸;除EcCueR为酸性蛋白外,包括AcCueR在内的其他蛋白均为碱性蛋白;从分子量上可以看出,AcCueR蛋白分子量比其他菌的CueR大,其他几个CueR蛋白的分子量相近。蛋白质可能分布的亚细胞位点结果显示,5种CueR蛋白的亚细胞位点均为任何位置。
| 蛋白名称 Proteins name |
分子量 Molecular weight (Da) |
理论等电点 Theoretical pI |
蛋白不稳定指数 Protein instability index (U) |
亲水性平均系数 Average coefficient of hydrophilicity |
N端氨基酸 Amino acids of the N-terminal |
亚细胞位点 Subcellular location |
| AcCueR | 15 898.16 | 9.05 | 57.02 | −0.352 | Met | − |
| EcCueR | 15 235.16 | 5.72 | 64.31 | −0.579 | Met | − |
| PaCueR | 15 015.06 | 7.73 | 54.10 | −0.591 | Met | − |
| SeCueR | 15 647.78 | 7.67 | 58.54 | −0.685 | Met | − |
| VcCueR | 15 693.06 | 8.78 | 47.75 | −0.492 | Met | − |
| 注:−:任何位置.
Note: −: Any other location. |
||||||
为了直观地了解CueR蛋白的结构,通过在线软件SWISS-MODEL对5种CueR蛋白进行3D同源建模(图 3)。预测结果显示:5种CueR蛋白序列与模板的一致性均 > 30%,且5种CueR蛋白氨基酸覆盖率均≥90%,同源建模可信度为100% (表 5)。分析蛋白质构象可知:5种CueR蛋白的主要结构均由α螺旋和无规则卷曲构成,且均含有螺旋-转角-螺旋(helix-turn-helix,HTH)区域。
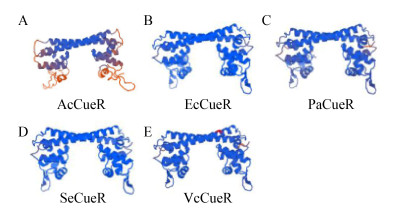
|
| 图 3 不同细菌CueR蛋白的3D结构图 Figure 3 3D structure map of different CueR proteins |
|
|
| 蛋白简称 Proteins name |
一致性 Identity (%) |
覆盖率 Coverage (%) |
模板 Template |
建模质量评价 GMQE |
| AcCueR | 33.59 | 90 | 4ua1.1.B | 0.63 |
| EcCueR | 100.00 | 100 | 4wlw.2.B | 0.98 |
| PaCueR | 48.85 | 99 | 4wlw.2.B | 0.81 |
| SeCueR | 91.79 | 97 | 4wlw.2.B | 0.94 |
| VcCueR | 54.55 | 95 | 4wlw.2.B | 0.81 |
对西瓜食酸菌FC440基因组数据分析显示:AcCueR与CopA (P型ATP酶)和多铜氧化酶CueO之间均具有互作关系,这一结果与大肠杆菌EcCueR蛋白的互作关系结果表现一致(图 4)。
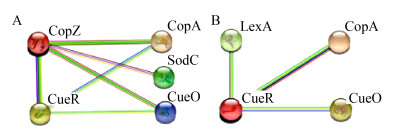
|
| 图 4 CueR的互作蛋白预测 Figure 4 Prediction of CueR interacting proteins 注:A:AcCueR互作蛋白;B:EcCueR互作蛋白. Note: A: AcCueR interacting proteins; B: EcCueR interacting proteins. |
|
|
启动子是通过与RNA聚合酶相互作用,从而影响转录起始频率和位置的DNA序列[19]。细菌启动子具有普遍的一些特征,包括转录起始点、−10区、−35区以及−10和−35区之间较为严格的间隔序列(图 5)[20]和回文基序(palindrome motif)等,RNA聚合酶可以与−10区和−35区结合从而起始转录[19, 21]。在大肠杆菌基因启动子中,−35区(TTGACA)和−10区(TATAAT)的碱基高度保守[19]。对大肠杆菌K-12的copA启动子研究发现(图 6A)[9, 13],其基因上游−10区和−35区之间含19 bp的间隔序列,−35区有5/6的序列与大肠杆菌启动子−35区序列一致(TTGACC和TTGACA比较),−10区的1/2序列与大肠杆菌的−10区序列一致(TAACCT和TATAAT比较);且体外实验证实CueR蛋白可以与该段21 bp的回文结构(两个箭头间包含的区域:5′-ACCTTCCN7GGAAGGT-3′,箭头所示部分为7 bp的二元对称序列(the dyad symmetrical sequence),N7示二元对称序列间的碱基数,下同)结合,从而调控copA基因的转录表达。本研究分析了西瓜食酸菌中copA基因上游序列(图 6B),通过预测发现−10区和−35区之间存在19 bp的间隔序列,且具有长度为19 bp的回文结构(两个箭头包含的区域:5′-TGACCTN7ATGTCA-3′,灰色部分示非对称碱基);与大肠杆菌K12的copA启动子序列相比,其–35区序列完全相同,−10区序列仅存在一个碱基不同,回文结构中的二元对称序列由非连续性的6 bp碱基组成。综合分析表明,西瓜食酸菌copA基因上游存在启动子序列,其特征与大肠杆菌K12的copA启动子高度相似,推测为西瓜食酸菌CueR的结合位点。
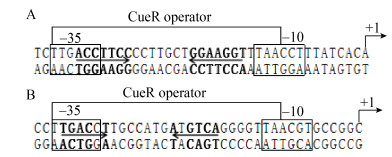
|
| 图 6 西瓜食酸菌和大肠杆菌copA基因上游序列特点 Figure 6 Upstream sequences of copA gene in A. citrulli and E. coli 注:A:大肠杆菌copA启动子核心元件[9];B:西瓜食酸菌copA启动子核心元件. −10和−35区序列用方框标出,回文序列用箭头和粗体标记,转录起始位点用弯曲的箭头表示. Note: A: Core elements of the promoter of copA in E. coli [9]; B: Core elements of the promoter of copA in Acidovorax citrulli. The −10 and −35 sequences are in boxes, and the palindromic sequence is marked with arrows and bold text, the transcriptional start site is indicated by a bent arrow. |
|
|
将cueR基因内部296 bp的片段连接到自杀载体pK19mob2ΩHMB上,获得重组载体pK19mob2ΩHMB-TY;采用双亲杂交法获得基因突变的菌株FC440(∆cueR),经引物cueR-QF/ cueR-QR扩增检测无法得到458 bp左右的目的条带,而野生型基因组DNA则可以扩增获得,证明目的基因已经被插入突变。cueR基因(GenBank登录号MK726380)全长连接到载体pBBR1MCS-5,获得重组载体pBBR1MCS-5-cueR。采用双亲杂交法将互补载体pBBR1MCS-5-cueR转入FC440(∆cueR)中,获得基因互补菌株FC440(∆cueR-cueR)。获得的基因突变体及互补菌株经用西瓜食酸菌特异性引物AAC-1/AAC-2检测均获得246 bp的特异性片段,证明为西瓜食酸菌。
2.7 cueR基因突变体及其互补菌株的抗铜性表型由含铜培养基上的生长情况可以看出(图 7),突变菌株FC440(∆cueR)对铜胁迫有显著的敏感性,随着铜浓度增加生长能力明显减弱,而基因互补菌株FC440(∆cueR-cueR)表现与野生型相似,即对铜的耐受性恢复。研究证实cueR基因与西瓜食酸菌的抗铜性有关。

|
| 图 7 西瓜食酸菌cueR基因突变体及其互补菌株在含铜培养基上的生长表型 Figure 7 The growth phenotype of Acidovorax citrulli cueR gene mutants and complementary strains on copper-containing media 注:图片采集于接菌48 h后. Note: Photos were taken at 48 h after inoculation. |
|
|
研究表明,生活在较高金属浓度环境中的细菌,其胞内金属稳态的维持对于细菌生长至关重要,因此,细胞已经发展出多种调节细胞内金属浓度的方法[10]。大肠杆菌中由转录调控因子CueR、P型ATP酶(CopA)和多铜氧化酶CueO[22]组成的Cue系统对铜离子的调节就是其中之一,其中CueR对细胞内金属离子作出反应,随着胞内铜离子水平的升高,CueR促进copA和cueO的转录表达从而将Cu+转运至周质空间并氧化为Cu2+。研究发现铜离子刺激下Cue系统的转录水平最高可达12倍[13]。在西瓜食酸菌中,类P型ATP酶[11]和多铜氧化酶CueO[12]已经被发现,但这两个基因的转录调控因子尚未被鉴定识别。
研究发现,原核启动子−10区和−35区之间的间距一般在15−20 bp,间距为17 bp时被认为转录效率最高[20],且在−10区和−35区间存在调节因子可以结合的回文基序。前人在革兰氏阴性细菌中的研究发现,MerR家族金属响应转录调节因子(metal responsive transcriptional regulator)作用于一些启动子来抑制或激活金属抗性基因的转录,这些启动子位于基因上游的−10区和−35区,两区之间具有19−20 bp的间隔序列[19],两区间包含一个蛋白质可以结合的回文基序[13]。隶属于MerR家族的大肠杆菌K12的CueR,其结合位点(copA的启动子)即具有上述特征,并且其结合的二元对称序列是连续性的。本研究发现,与大肠杆菌K-12的copA基因启动子比较,西瓜食酸菌copA基因启动子的−10区和−35区序列除符合原核基因启动子的特征外,还表现出其回文基序中二元对称序列为6 bp的非连续性序列。已有研究发现,不同细菌中的回文基序的二元对称序列长度和连续性存在多样性。如,王冬梅等[23]研究发现,淋球菌mtrR启动子区二元对称序列为连续性的6 bp碱基组成;周日成[24]研究发现,玫瑰孢链霉菌depR1启动子由非连续性的11 bp的二元对称序列组成;王慧[25]研究发现,大肠杆菌K12中Fis识别8 bp非连续性的二元对称序列。由此可见,西瓜食酸菌copA启动子中回文基序的不同,即二元对称序列为6 bp且非连续,可能是物种间的差异;而AcCueR与该位点的结合及对copA的调控仍需进一步的实验验证。通过生物信息学分析比较发现,西瓜食酸菌CueR蛋白及其他物种CueR蛋白在分子量、等电点等理化性质上存在差异,出现这些现象的具体原因尚未可知,需要进一步的研究解析。
研究发现,当根据序列或结构信息将某个蛋白质归类到一个超家族,该蛋白可能具有的功能范围会缩小,甚至在某些情况下可以精确地确定其功能[26-27]。在本研究中,AcCueR与另4种CueR蛋白均属于HTH-MerR-SF超家族,它们的一级结构序列、保守结构域等相似但并不完全相同,这可能是导致三级结构不完全相同的原因,但这些差异并不影响它们发挥相似的生物学功能,因为蛋白质的功能主要由其所含有的结构域和三级结构决定。此外,MerR家族调节蛋白均含有螺旋-转角-螺旋(helix-turn-helix,HTH)的DNA结合序列特点,该DNA结合序列接近蛋白质的N-端[13]。本研究中,5种CueR蛋白均含有HTH保守结构域,说明CueR在革兰氏阴性细菌中具有高度的保守性。已有研究发现,不同细菌中的CueR调控的铜稳态相关基因存在相同,但均调控P型ATP酶(CopA)。例如,Martell等[10]研究发现,大肠杆菌中CueR调控耐铜基因的转录,包括内膜铜蛋白ATP酶基因(copA)和胞质多铜氧化酶基因(cueO),这两种基因产物都有助于将铜从细胞中去除;Quintana等[28]研究发现,在铜绿假单胞菌中,CueR调控抗铜基因copA、cusCBA、copZ1及copZ2的转录表达;Espariz等[29]研究证实,沙门氏菌中的铜耐受相关组分P型ATP酶CopA和多铜氧化酶CuiD (沙门氏菌中称为CuiD)基因的表达依赖于CueR的调控;Marrero等[16]研究发现霍乱弧菌的CopA和CueR (CueO尚不确定)确实参与了该菌的铜耐受性过程,且copA基因的表达被CueR激活。这些研究结果显示,在不同细菌中CueR参与调控的铜稳态机制可能不同。本研究中蛋白互作预测显示,大肠杆菌EcCueR与其CopA和CueO具有互作关系,这与前人研究结果完全一致[13];与大肠杆菌相似,西瓜食酸菌AcCueR能与该菌的CopA和CueO发生互作关系;而进一步的基因沉默和基因功能互补实验证实:生物信息学筛选得到的cueR经突变后会导致菌对铜胁迫更加敏感,互补菌株的铜抗性恢复则验证了该基因为西瓜食酸菌抗铜性相关基因。由此推测,AcCueR和大肠杆菌等菌株的CueR具有类似的转录调控功能,在西瓜食酸菌中有关AcCueR对CopA和CueO的作用模式尚需进一步实验探究。
本研究对cueR基因的生物信息学分析及功能验证,为进一步研究西瓜食酸菌CueR蛋白功能和抗铜机制奠定了基础。
| [1] |
Niu QW. Research on occurrence rule and control technology of BFB in the process of grafted watermelon seedling production[D]. Wuhan: Master's Thesis of Huazhong Agricultural University, 2013 (in Chinese) 牛庆伟.西瓜嫁接育苗过程中细菌性果斑病的发生规律及防治技术研究[D].武汉: 华中农业大学硕士学位论文, 2013 http://cdmd.cnki.com.cn/Article/CDMD-10504-1013336788.htm |
| [2] |
Deng WL, Huang TC, Tsai YC. First report of Acidovorax avenae subsp. citrulli as the causal agent of bacterial leaf blight of betelvine in Taiwan[J]. Plant Disease, 2010, 94(8): 1065. |
| [3] |
Latin RX, Rane KK. Bacterial fruit blotch of watermelon in Indiana[J]. Plant Disease, 1990, 74(4): 331. |
| [4] |
Somodi GC, Jones JB, Hopkins DL, et al. Occurrence of a bacterial watermelon fruit blotch in Florida[J]. Plant Disease, 1991, 75(10): 1053-1056. DOI:10.1094/PD-75-1053 |
| [5] |
Liu X. Functional analysis of copper resistance related gene cusB in Acidovorax citrulli[D]. Urumqi: Master's Thesis of Xinjiang Agricultural University, 2015 (in Chinese) 刘星.西瓜食酸菌抗铜性相关基因cusB的功能分析[D].乌鲁木齐: 新疆农业大学硕士学位论文, 2015 http://cdmd.cnki.com.cn/Article/CDMD-10758-1015645934.htm |
| [6] |
Hu YH. Molecular analysis of the copper-responsive CopRSCD of a pathogenic Pseudomonas fluorescens strain[D]. Qingdao: Doctoral Dissertation of the Institute of Oceanology, Chinese Academy of Sciences (Institute of Oceanography), 2009 (in Chinese) 胡永华.荧光假单胞菌抗铜基因簇的表达调控及其致病性分析[D].青岛: 中国科学院研究生院(海洋研究所)博士学位论文, 2009 http://cdmd.cnki.com.cn/Article/CDMD-80068-2009138685.htm |
| [7] |
Bahar O, Burdman S. Bacterial fruit blotch: a threat to the cucurbit industry[J]. Israel Journal of Plant Sciences, 2010, 58(1): 19-31. DOI:10.1560/IJPS.58.1.19 |
| [8] |
King EO, Ward MK, Raney DE. Two simple media for the demonstration of pyocyanin and fluorescin[J]. The Journal of Laboratory and Clinical Medicine, 1954, 44(2): 301-307. |
| [9] |
Outten FW, Outten CE, Hale J, et al. Transcriptional activation of an Escherichia coli copper efflux regulon by the chromosomal MerR homologue, CueR[J]. Journal of Biological Chemistry, 2000, 275(40): 31024-31029. DOI:10.1074/jbc.M006508200 |
| [10] |
Martell DJ, Joshi CP, Gaballa A, et al. Metalloregulator CueR biases RNA polymerase's kinetic sampling of dead-end or open complex to repress or activate transcription[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(44): 13467-13472. DOI:10.1073/pnas.1515231112 |
| [11] |
Zhao WL. Analysis of copper resistance in Acidovorax citrulli[D]. Beijing: Master's Thesis of Chinese Academy of Agricultural Sciences, 2013 (in Chinese) 赵文龙.瓜类细菌性果斑病菌抗铜性研究[D].北京: 中国农业科学院硕士学位论文, 2013 http://cdmd.cnki.com.cn/Article/CDMD-82101-1013357633.htm |
| [12] |
Li Q. Functional analysis of CueO gene in the bacterial fruit blotch of watermelon[D]. Hohhot: Master's Thesis of Inner Mongolia Agricultural University, 2014 (in Chinese) 李强.瓜类果斑病菌多铜氧化酶基因CueO的功能研究[D].呼和浩特: 内蒙古农业大学硕士学位论文, 2014 http://cdmd.cnki.com.cn/Article/CDMD-10129-1014373885.htm |
| [13] |
Stoyanov JV, Hobman JL, Brown NL. CueR (YbbI) of Escherichia coli is a MerR family regulator controlling expression of the copper exporter CopA[J]. Molecular Microbiology, 2001, 39(2): 502-512. DOI:10.1046/j.1365-2958.2001.02264.x |
| [14] |
Bagchi A. Structural characterizations of metal ion binding transcriptional regulator CueR from opportunistic pathogen Pseudomonas aeruginosa to identify its possible involvements in virulence[J]. Applied Biochemistry and Biotechnology, 2015, 175(2): 649-656. DOI:10.1007/s12010-014-1304-5 |
| [15] |
Pérez Audero ME, Podoroska BM, Ibáñez MM, et al. Target transcription binding sites differentiate two groups of MerR-monovalent metal ion sensors[J]. Molecular Microbiology, 2010, 78(4): 853-865. DOI:10.1111/j.1365-2958.2010.07370.x |
| [16] |
Marrero K, Sánchez A, González LJ, et al. Periplasmic proteins encoded by VCA0261-0260 and VC2216 genes together with copA and cueR products are required for copper tolerance but not for virulence in Vibrio cholerae[J]. Microbiology, 2012, 158(8): 2005-2016. DOI:10.1099/mic.0.059345-0 |
| [17] |
Walcott RR, Fessehaie A, Castro AC. Differences in pathogenicity between two genetically distinct groups of Acidovorax avenae subsp. citrulli on cucurbit hosts[J]. Phytopathology, 2004, 152(5): 277-285. DOI:10.1111/j.1439-0434.2004.00841.x |
| [18] |
Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [19] |
Harley CB, Reynolds RP. Analysis of E. coli promoter sequences[J]. Nucleic Acids Research, 1987, 15(5): 2343-2361. DOI:10.1093/nar/15.5.2343 |
| [20] |
Wang XH. The analysis of chemical features in the core promoter of Escherichia coli[D]. Lanzhou: Master's Thesis of Lanzhou University, 2008 (in Chinese) 王晓慧.大肠杆菌核心启动子的化学特性分析[D].兰州: 兰州大学硕士学位论文, 2008 http://www.paper.edu.cn/releasepaper/content/200803-484 |
| [21] |
O'Neill MC. Escherichia coli promoters. Ⅰ. Consensus as it relates to spacing class, specificity, repeat substructure, and three-dimensional organization[J]. Journal of Biological Chemistry, 1989, 264(10): 5522-5530. |
| [22] |
Solioz M. Copper homeostasis in gram-negative bacteria[A]//Solioz M. Copper and Bacteria: Evolution, Homeostasis and Toxicity[M]. Berlin: Springer Press, 2018: 49-80
|
| [23] |
Wang DM, Xia ZD, Qi SW, et al. Multiple resistance of Neisseria gonorrhoeae and mutations in the inverted sequence within the mtrR promoter region[J]. Journal of Central South University (Medical Sciences), 2004, 29(5): 544-547. (in Chinese) 王冬梅, 夏忠弟, 齐素文, 等. 淋球菌mtrR启动子区域中回文序列基因突变与多重耐药的关系[J]. 中南大学学报:医学版, 2004, 29(5): 544-547. |
| [24] |
Zhou RC. Sreening of a transcriptional regulator DepR1 and its regulatory mechanism on daptomycin biosynthesis in Streptomyces Roseosporus SW0702[D]. Hangzhou: Master's Thesis of Zhejiang University, 2015 (in Chinese) 周日成.玫瑰孢链霉菌SW0702中达托霉素生物合成调控因子DepR1的筛选与调控机制研究[D].杭州: 浙江大学硕士学位论文, 2015 |
| [25] |
Wang H. Genome-wide analysis of Fis regulon in Salmonella enterica serovar Typhimurium LT2 and its regulatory mechanism on pathogenicity islands[D]. Tianjin: Doctoral Dissertation of Nankai University, 2013 (in Chinese) 王慧. Fis蛋白在沙门氏菌中的全局调控网络及其对致病岛的调控机制研究[D].天津: 南开大学博士学位论文, 2013 |
| [26] |
Peng B. Analysis of the relationship between protein function and conformation[J]. Farm Products Processing, 2010(12): 65-66, 69. (in Chinese) 彭彬. 蛋白质功能和构象的关系分析[J]. 农产品加工, 2010(12): 65-66, 69. |
| [27] |
Karplus M, McCammon JA. Molecular dynamics simulations of biomolecules[J]. Nature Structural Biology, 2002, 9(9): 646-652. DOI:10.1038/nsb0902-646 |
| [28] |
Quintana J, Novoa-Aponte L, Argüello JM. Copper homeostasis networks in the bacterium Pseudomonas aeruginosa[J]. Journal of Biological Chemistry, 2017, 292(38): 15691-15704. DOI:10.1074/jbc.M117.804492 |
| [29] |
Espariz M, Checa SK, Pérez Audero ME, et al. Dissecting the Salmonella response to copper[J]. Microbiology, 2007, 153(9): 2989-2997. DOI:10.1099/mic.0.2007/006536-0 |
 2020, Vol. 47
2020, Vol. 47