扩展功能
文章信息
- 蒋佩钰, 尚晓冬, 宋春艳, 王瑞娟, 于海龙, 李巧珍, 章炉军, 刘建雨, 谭琦
- JIANG Pei-Yu, SHANG Xiao-Dong, SONG Chun-Yan, WANG Rui-Juan, YU Hai-Long, LI Qiao-Zhen, ZHANG Lu-Jun, LIU Jian-Yu, TAN Qi
- 真菌中赖氨酸生物合成途径研究进展
- Advances in studies on lysine biosynthesis pathway in fungi
- 微生物学通报, 2020, 47(3): 915-922
- Microbiology China, 2020, 47(3): 915-922
- DOI: 10.13344/j.microbiol.china.190514
-
文章历史
- 收稿日期: 2019-06-19
- 接受日期: 2019-09-09
- 网络首发日期: 2019-10-10
2. 上海海洋大学食品学院 上海 201306
2. College of Food Science & Technology, Shanghai Ocean University, Shanghai 201306, China
赖氨酸作为第一限制性必需氨基酸,在蛋白质合成中发挥着重要作用,缺少赖氨酸会使其他氨基酸的利用受阻。在常见的20种蛋白氨基酸中,赖氨酸是唯一的有两个不同生物合成途径的氨基酸,包括存在于植物、细菌及低等真菌中的二氨基庚二酸途径以及存在于眼虫(Euglenoids)、高等真菌和少数细菌中的α-氨基己二酸途径[1-2]。与二氨基庚二酸途径不同的是,α-氨基己二酸途径以α-酮戊二酸为起始物,通过8个酶促反应生成赖氨酸[1, 3]。从生物化学、酶学及基因工程等方向深入探究赖氨酸生物合成途径,对抑制致病性真菌的生长、提高有益真菌的蛋白质水平有重要意义。
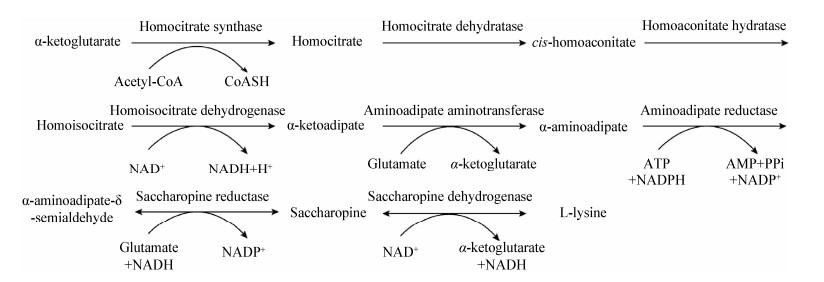
1 真菌中赖氨酸合成途径研究进展 1.1 赖氨酸合成途径的介绍真菌中赖氨酸从头合成途径[4]见图 1,反应分为两部分,发生在不同的场所,最终合成赖氨酸。
α-氨基己二酸途径(α-aminoadipate pathway,AAA途径)的前半部分反应,即以α-酮戊二酸为起始物生成α-氨基己二酸,发生在线粒体中[1, 5],此阶段的反应与亮氨酸生物合成途径以及三羧酸循环有很多相似之处[1, 6-10],而且前3个反应对于高等真菌有很高的特异性[11]。该途径的起始反应是在高柠檬酸合酶(homocitrate synthase,HCS)的催化下,α-酮戊二酸与乙酰辅酶A (acetyl coenzyme A,AcCoA)发生缩合反应生成高柠檬酸,该反应是AAA途径的关键限速步骤[3, 12];文献[13]报道,在产黄青霉(Penicillium chrysogenum)中HCS的活性受pH的调控,降低ATP的浓度会使细胞溶质的pH降低,进而提高HCS的活性,有利于真菌利用赖氨酸合成途径中的中间体产生次级代谢物;除此之外,Na+与游离酶结合也可以调控HCS的活性[14]。HCS反应产物——辅酶A (CoA)会使酿酒酵母(Saccharomyces cerevisiae)中的HCS失活,赖氨酸及α-酮戊二酸可防止该酶失活[15-18]。随后,高柠檬酸在高柠檬酸脱水酶(homocitrate dehydratase,HCD)的催化下生成顺-高乌头酸,后者在高乌头酸水合酶(homoaconitate hydratase,HAH)的作用下生成高异柠檬酸。催化上述两个反应中的两个酶的编码基因在不同的文献报道中有不同的注释[1, 3-4, 19-21],但都是通过脱水作用和水合作用为形成赖氨酸的碳骨架奠定基础。高异柠檬酸在高异柠檬酸脱氢酶(homoisocitrate dehydrogenase,HIDH)的催化下生成α-酮己二酸,至此,赖氨酸碳骨架结构形成[4]。随后,α-酮己二酸在氨基己二酸转氨酶(α-aminoadipate aminotransferase,AAT)的催化下,以谷氨酸为底物生成α-氨基己二酸和α-酮戊二酸。值得注意的是,在酿酒酵母中,两个转氨酶分别位于线粒体和细胞质[21],但在金针菇中,两个转氨酶都位于细胞质[4],推测其差异可能是由于不同物种在进化过程中导致的。
AAA途径的后半部分反应,即由α-氨基己二酸生成L-赖氨酸,发生在细胞质中[1]。首先是以α-氨基己二酸为底物,由氨基己二酸还原酶(aminoadipate reductase,AAR)催化、ATP供能、Mg2+参与、NADPH作为辅酶,生成α-氨基己二酸-δ-半醛;此反应体现了AAA途径的独特性,主要表现在仅存在于真菌中且同时发生腺苷化反应和还原反应[3];经研究发现,虫拟蜡菌(Ceriporiopsis subvermispora)中的腺苷酸化激活结构域是底物腺苷化所必需的,有助于在结构水平上加深对此反应的了解[22];AAR催化的还原反应被认为是真菌赖氨酸合成进化中的关键步骤[23]。然后以谷氨酸作为底物,酵母氨酸还原酶(saccharopine reductase,SDR)催化α-氨基己二酸-δ-半醛生成L-酵母氨酸,后者在酵母氨酸脱氢酶(saccharopine dehydrogenase,SDH)的催化下生成L-赖氨酸和α-酮戊二酸。SDR和SDH是真菌中特有的酶,虽然二者在氨基酸水平上无同源性,但二者催化的反应非常相似[3]。
AAA途径并非真菌所特有,少数细菌如嗜热栖热菌(Thermus thermophilus)[2]、超嗜热古菌(Pyrococcus horikoshii)[24]等也通过此途径合成赖氨酸。细菌和真菌在前半部分的反应是一样的,但是在后半部分通过不同的酶促反应合成赖氨酸,也间接证明了真菌AAA途径中后3个酶的特异性[23]。
1.2 AAA途径的国内外研究进展在前人的研究中,已经确定了赖氨酸的两条合成途径——二氨基庚二酸途径及α-氨基己二酸途径。1960年,Vogel利用14C标记化合物作为示踪剂对低等真菌水霉目中的两性绵霉(Achlya bisexualis)及黑根霉(Rhizopus stolonifer)的赖氨酸途径进行追踪,发现两性绵霉通过二氨基庚二酸途径合成赖氨酸,而黑根霉则是通过AAA途径合成赖氨酸,表明低等真菌中包含两条合成赖氨酸的途径[25]。1965年,Jones等利用14C标记及赖氨酸营养缺陷型菌株,采用纸色谱法和纸电泳法确定了氨基己二酸半醛被胺化后通过新的中间体——酵母氨酸合成赖氨酸的过程[26],这是对AAA途径中的酶促反应更深入的研究。1970年,Sinha等确定了在酿酒酵母中α-氨基己二酸合成氨基己二酸半醛的过程包含3个步骤:首先是氨基己二酸与ATP反应形成腺苷酸衍生物;然后NADPH作为辅酶,该衍生物被还原;最后被还原的腺苷酸衍生物形成α-氨基己二酸-δ-半醛;研究中还发现,Mg2+对α-氨基己二酸参与的第一步反应活性起着关键作用,EDTA抑制半醛的形成[27]。1998年,首次在嗜热栖热菌(Thermus thermophilus)中发现了AAA途径,在此之前认为细菌都是通过二氨基庚二酸途径合成赖氨酸的;Kosuge等从嗜热栖热菌HB27菌株中克隆了3.8 kb的DNA片段,其中2个ORF分别与酿酒酵母的HCS和HAH有55.3%和45.0%的相似度,同时在嗜热栖热菌HB27的赖氨酸营养缺陷突变体TH151补充α-氨基己二酸能够生长,但补充二氨基庚二酸不能生长,由此证明了嗜热栖热菌通过AAA途径合成赖氨酸[2]。
2006年,Xu等对真菌赖氨酸合成途径——α-氨基己二酸途径在生物化学和酶学上进行了总结,α-氨基己二酸途径在真菌中的独特性这一特点可以作为设计抗真菌药物的一个靶标[3]。2011年,Nango等发现(2S, 3S)-硫代异柠檬酸作为抑制剂对嗜热栖热菌高异柠檬酸脱水酶的活性有强烈的抑制,此抑制剂与高异柠檬酸脱水酶结合的二元复合物对设计抗真菌剂和阐明酶反应机制具有指导作用[28]。2014年,Chen等说明了在植物致病菌——稻瘟病菌(Magnaporthe oryzae)中氨基己二酸还原酶Lys2在其生长、分生孢子的萌发及植物致病性发挥着重要作用,因稻瘟病菌侵染植物需要营养物质补给,赖氨酸可作为其中一种营养物,当缺失Lys2时,稻瘟病菌对水稻的致病性急剧下降[29]。嗜热栖热菌中的LysW是由54个氨基酸残基组成的酸性蛋白质,2015年,Yoshida等通过确定TtLysW-γ-AAA及TtLysZ·TtLysW-γ-AAA复合物的晶体结构,以及利用TtLysZ的突变体和模拟研究法,证实了LysW不仅可以保护赖氨酸生物合成中底物的α-氨基基团,而且作为新型载体蛋白通过与生物合成中的酶相互作用更加有效地促进赖氨酸的合成[30]。
自20世纪中期开始,利用示踪法、赖氨酸营养缺陷型菌株及分子生物学等技术逐一确证了一些大型真菌如酵母[31]、粗糙脉孢菌(Neurospora crassa)[32]、白色念珠菌(Candida albicans)[33]等的赖氨酸生物合成途径——AAA途径,而在对真菌赖氨酸合成途径的研究中,研究较多的是模式真菌酿酒酵母和人类致病性真菌念珠菌属。Barnes等利用α-氨基己二酸还原酶的编码基因(lys2)进行基因破坏、基因置换及启动子分析等技术来说明lys2的遗传效应,为其他真菌赖氨酸合成途径中基因的分析提供一个思路[34]。O’Doherty等利用含充足赖氨酸的培养基,暴露于脂质氧化型亚油酸过氧化氢(lipid oxidant-linoleic acid hydroperoxide,LoaOOH)的环境,将酿酒酵母中赖氨酸酵母氨酸脱氢酶基因突变体(lys1Δ)菌株与野生型BY4743相比,发现其生长明显减慢;在含有完整的赖氨酸生物合成基因的培养基中,暴露于LoaOOH的野生型BY4743在缺乏赖氨酸的条件下生长速度也大大降低;通过以上结果认为,赖氨酸生物合成途径在保护细胞免受脂质过氧化物诱导的氧化应激中起着重要作用[35]。2015年,在对光滑念珠菌(Candida glabrata)的研究中发现,两种超氧化物歧化酶(SOD)——Cu, ZnSOD (Sod1)和MnSOD (Sod2)在氧化应激反应中至关重要,缺乏Sod1、Sod2会导致赖氨酸营养缺陷型,而缺失Sod1是导致赖氨酸营养缺陷的主要原因,说明Sod1、Sod2在光滑念珠菌的赖氨酸生物合成过程中起重要作用[36],而光滑念珠菌是感染人体的致病性真菌,此文的研究结果将为研发抗真菌剂提供新的思路。Priyadarshini等探讨了在微生物的进化过程中,如何调节基因的表达以适应环境带来的变化;在对人类真菌病原体白色念珠菌的研究中发现,当出现赖氨酸缺乏的情况时,转录激活因子Gcn4是白色念珠菌的生长所必需的,并且Gcn4是赖氨酸生物合成途径中多个基因的直接激活剂[37]。
因金针菇中赖氨酸含量丰富[38],所以在食用菌中研究较多的是对金针菇的赖氨酸合成途径。2015年,Liu等对金针菇子实体不同发育阶段的HCS基因表达量及赖氨酸含量进行了分析,结果发现FvHCS基因表达量与赖氨酸含量呈正相关,通过全基因组测序确定了金针菇通过α-氨基己二酸途径合成赖氨酸[39],这是第一次在食用菌中对赖氨酸合成途径的报道。2016年,刘建雨等基于全基因组测序结果筛选金针菇中赖氨酸生物合成的关键基因,并分析这些基因编码的蛋白质基本理化性质,最终确定金针菇是通过α-氨基己二酸途径合成赖氨酸,并预测到8个参与α-氨基己二酸途径中的酶[4]。2018年,陶永新等通过对影响金针菇赖氨酸含量的环境因子及其作用机理进行探究发现,蓝光对赖氨酸通路基因的诱导效果最显著,而且在蓝光处理后,HCS基因响应表达上调倍数最高,赖氨酸含量升高倍数最大,体现了HCS作为关键限速酶的重要性[19]。
2 酵母氨酸脱氢酶研究进展目前,对限速酶HCS的研究较为广泛,但是对SDH的研究甚少。从SDH的晶体结构、理化性质及基因序列出发,探究SDH在赖氨酸生物合成途径中的作用,对开发抗真菌药物及作为其他用途具有重要意义。
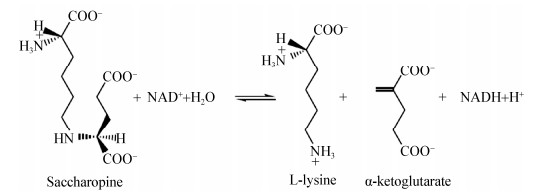
2.1 SDH在整个途径中的位置及反应机理SDH作为AAA途径的最后一个酶,催化依赖吡啶核苷酸的可逆反应,由酵母氨酸氧化脱氨生成赖氨酸和α-酮戊二酸,其中NAD+作为氧化剂[40],如图 2所示。
酶动力学遵循顺序反应机制,大多数与吡啶核苷酸连接的脱氢酶中,辅酶作为底物第一个与酶结合,作为产物最后一个释放。在正反应中,底物与酶结合顺序为NAD+、酵母氨酸[41-42];而在逆反应中,NADH首先与酶结合,赖氨酸与α-酮戊二酸结合顺序随机[42]。
2.2 SDH的理化性质与结构SDH是碱性蛋白,在酿酒酵母中其等电点是10.1[3]。可逆反应中,SDH在正反应的最佳pH为10.0,在逆反应的最佳pH为7.0[43]。
在底物特异性方面,酿酒酵母中的SDH对底物表现出高度特异性,仅有丙酮酸可作为逆反应中α-酮戊二酸的替代酮酸[44]。此外,SDH在逆反应中受高浓度α-酮戊二酸和赖氨酸的抑制,但在正反应中不受酵母氨酸的抑制[45]。SDH对辅酶也表现出严格的特异性,其不能利用NADP+作为辅酶,并且在逆反应中,NADPH与SDH的结合力很弱,没有NADH与SDH的亲和性好[43]。
SDH基因已在白色念珠菌[33]和裂殖酵母[46]中被克隆、表达。SDH含有4个半胱氨酸残基,不含二硫键,且每个酶分子中含一个反应结合位点[47]。
2.3 酵母氨酸脱氢酶的国内外研究进展1972年,Fujioka等研究了SDH与底物类似物的反应,发现在逆反应中α-氨基酸和α-酮酸对SDH有抑制作用,最好的抑制剂是含脂肪族侧链的5–6个碳原子的氨基酸,而二羧酸氨基酸如天冬氨酸、谷氨酸和α-氨基己二酸作为底物类似物没有抑制作用[48]。1984年,Fujioka发现在SDH催化的可逆反应中,在pH较低的情况下,正反应的活性较低而逆反应的活性较高[49]。Burk等从对来自酿酒酵母中的SDH进行克隆表达、晶体结构分析、SDH三元结构建模等方面进行探索,揭示了SDH作为抗真菌药物靶标的适用性[50]。2015年,Wan等发现褐飞虱(Nilaparvata lugens)的SDH (NlysSDH)来源于其体内的共生菌——类酵母共生菌(yeast like symbiotes,YLS),并非自身含有SDH;其研究结果发现NlysSDH在褐飞虱的赖氨酸合成过程中发挥着重要的作用,而赖氨酸在昆虫的发育过程中扮演着重要的角色[51]。LysW作为载体蛋白通过与从AAA转化赖氨酸的过程中的底物结合成复合物,对其α-氨基基团起保护作用,而生物合成中的最后一个酶SDH通过静电相互作用特异性识别LysW的球状结构域使赖氨酸从LysW-γ-Lys复合物中释放出来,完成生物合成反应[52],此发现对SDH催化的最后一步反应在结构水平上有了进一步的认知。
3 展望在20世纪,研究者主要是通过同位素放射性标记法及赖氨酸营养缺陷型菌株对AAA途径进行研究;如今随着计算机技术的成熟,旧的繁琐的方法被替代,通过全基因组测序便可高效快速确定赖氨酸合成途径以及途径中的酶的编码基因。
近些年对α-氨基己二酸途径的生物化学及酶学研究较为深入,但是对于参与该途径中的基因的调控了解甚少。未来的研究方向可以通过选择合适的底物类似物抑制真菌病原体的生长以及利用途径中的基因作为鉴定真菌病原体的分子标记,对于有益真菌可以利用分子手段深入探究其作用机理及调控机理,更加广泛地应用到生产和生活中;而在食用菌中,赖氨酸合成途径的研究是最近几年才开始的,多以金针菇为研究对象。在分子水平上探索赖氨酸生物合成的调控机制将辅助金针菇产业向高质量方向发展,满足消费者更高的营养需求。在生产方面,赖氨酸因其自身的生理功能广泛应用于饲料、医药和食品等行业中,植物、细菌等[53-56]利用基因工程技术选育高赖氨酸菌种已趋近成熟,而真菌中的高赖氨酸菌种培育尚处于空白阶段,因此可以利用基因工程手段结合微生物发酵法提高真菌中的赖氨酸产量,为开发赖氨酸生物合成途径提供新思路。
| [1] |
Zabriskie TM, Jackson MD. Lysine biosynthesis and metabolism in fungi[J]. Natural Product Reports, 2000, 17(1): 85-97. DOI:10.1039/a801345d |
| [2] |
Kosuge T, Hoshino T. Lysine is synthesized through the α-aminoadipate pathway in Thermus thermophilus[J]. FEMS Microbiology Letters, 1998, 169(2): 361-367. |
| [3] |
Xu HY, Andi B, Qian JH, et al. The α-aminoadipate pathway for lysine biosynthesis in fungi[J]. Cell Biochemistry and Biophysics, 2006, 46(1): 43-64. DOI:10.1385/CBB:46:1:43 |
| [4] |
Liu JY, Wang RJ, Zhang D, et al. Analysis of genes related to lysine biosynthesis based on whole genome of Flammulina velutipes[J]. Microbiology China, 2016, 43(10): 2225-2233. (in Chinese) 刘建雨, 王瑞娟, 张丹, 等. 基于金针菇全基因组的赖氨酸合成途径关键酶分析[J]. 微生物学通报, 2016, 43(10): 2225-2233. |
| [5] |
Urrestarazu LA, Borell CW, Bhattacharjee JK. General and specific controls of lysine biosynthesis in Saccharomyces cerevisiae[J]. Current Genetics, 1985, 9(5): 341-344. DOI:10.1007/BF00421603 |
| [6] |
Nishida H, Nishiyama M, Kobashi N, et al. A prokaryotic gene cluster involved in synthesis of lysine through the amino adipate pathway: a key to the evolution of amino acid biosynthesis[J]. Genome Research, 1999, 9(12): 1175-1183. DOI:10.1101/gr.9.12.1175 |
| [7] |
Wulandari AP, Miyazaki J, Kobashi N, et al. Characterization of bacterial homocitrate synthase involved in lysine biosynthesis[J]. FEBS Letters, 2002, 522(1/3): 35-40. |
| [8] |
Miyazaki J, Kobashi N, Nishiyama M, et al. Characterization of homoisocitrate dehydrogenase involved in lysine biosynthesis of an extremely thermophilic bacterium, Thermus thermophilus HB27, and evolutionary implication of β-decarboxylating dehydrogenase[J]. Journal of Biological Chemistry, 2003, 278(3): 1864-1871. DOI:10.1074/jbc.M205133200 |
| [9] |
Jia YH, Tomita T, Yamauchi K, et al. Kinetics and product analysis of the reaction catalysed by recombinant homoaconitase from Thermus thermophilus[J]. Biochemical Journal, 2006, 396(3): 479-485. DOI:10.1042/BJ20051711 |
| [10] |
Miyazaki T, Miyazaki J, Yamane H, et al. α-aminoadipate aminotransferase from an extremely thermophilic bacterium, Thermus thermophilus[J]. Microbiology, 2004, 150(7): 2327-2334. DOI:10.1099/mic.0.27037-0 |
| [11] |
Gabriel I, Rychłowski M. Consequences of lysine auxotrophy for Candida albicans adherence and biofilm formation[J]. Acta Biochimica Polonica, 2017, 64(2): 323-329. |
| [12] |
Schöbel F, Jacobsen ID, Brock M. Evaluation of lysine biosynthesis as an antifungal drug target: biochemical characterization of Aspergillus fumigatus homocitrate synthase and virulence studies[J]. Eukaryotic Cell, 2010, 9(6): 878-893. DOI:10.1128/EC.00020-10 |
| [13] |
Jaklitsch WM, Kubicek CP. Homocitrate synthase from Penicillium chrysogenum. Localization, purification of the cytosolic isoenzyme, and sensitivity to lysine[J]. Biochemical Journal, 1990, 269(1): 247-253. |
| [14] |
Andi B, Cook PF. Regulatory mechanism of histidine-tagged homocitrate synthase from Saccharomyces cerevisiae. Ⅱ. Theory[J]. Journal of Biological Chemistry, 2005, 280(36): 31633-31640. DOI:10.1074/jbc.M502847200 |
| [15] |
Tracy JW, Kohlhaw GB. Reversible, coenzyme-A-mediated inactivation of biosynthetic condensing enzymes in yeast: a possible regulatory mechanism[J]. Proceedings of the National Academy of Sciences of the United States of America, 1975, 72(5): 1802-1806. DOI:10.1073/pnas.72.5.1802 |
| [16] |
Hampsey DM, Kohlhaw GB. Inactivation of yeast alpha-isopropylmalate synthase by CoA. Antagonism between CoA and adenylates and the mechanism of CoA inactivation[J]. Journal of Biological Chemistry, 1981, 256(8): 3791-3796. |
| [17] |
Tracy JW, Kohlhaw GB. Evidence for two distinct CoA binding sites on yeast α-isopropylmalate synthase[J]. Journal of Biological Chemistry, 1977, 252(12): 4085-4091. |
| [18] |
Kohlhaw GB. Leucine biosynthesis in fungi: entering metabolism through the back door[J]. Microbiology and Molecular Biology Reviews, 2003, 67(1): 1-15. DOI:10.1128/MMBR.67.1.1-15.2003 |
| [19] |
Tao YX, Duan JY, Li YN, et al. Identification of genes in Flammulina filiformis L-lysine biosynthesis pathway and their expression in response to light conditions[J]. Acta Edulis Fungi, 2018, 25(4): 1-8. (in Chinese) 陶永新, 段静怡, 李依宁, 等. 金针菇L-赖氨酸合成通路基因鉴定及对不同光质的响应表达[J]. 食用菌学报, 2018, 25(4): 1-8. |
| [20] |
Velasco AM, Leguina JI, Lazcano A. Molecular evolution of the lysine biosynthetic pathways[J]. Journal of Molecular Evolution, 2002, 55(4): 445-449. DOI:10.1007/s00239-002-2340-2 |
| [21] |
Bhattacharjee JK. α-aminoadipate pathway for the biosynthesis of lysine in lower eukaryotes[J]. CRC Critical Reviews in Microbiology, 1985, 12(2): 131-151. DOI:10.3109/10408418509104427 |
| [22] |
Kalb D, Lackner G, Rappe M, et al. Activity of α-aminoadipate reductase depends on the N-terminally extending domain[J]. ChemBioChem, 2015, 16(10): 1426-1430. DOI:10.1002/cbic.201500190 |
| [23] |
Nishida H, Nishiyama M. What is characteristic of fungal lysine synthesis through the α-aminoadipate pathway?[J]. Journal of Molecular Evolution, 2000, 51(3): 299-302. DOI:10.1007/s002390010091 |
| [24] |
Kobashi N, Nishiyama M, Tanokura M. Aspartate kinase-independent lysine synthesis in an extremely thermophilic bacterium, Thermus thermophilus: lysine is synthesized via α-aminoadipic acid not via diaminopimelic acid[J]. Journal of Bacteriology, 1999, 181(6): 1713-1718. DOI:10.1128/JB.181.6.1713-1718.1999 |
| [25] |
Vogel HJ. Two modes of lysine synthesis among lower fungi: evolutionary significance[J]. Biochimica et Biophysica Acta, 1960, 41(1): 172-173. DOI:10.1016/0006-3002(60)90392-9 |
| [26] |
Jones EE, Broquist HP. Saccharopine, an intermediate of the aminoadipic acid pathway of lysine biosynthesis. Ⅱ. Studies in Saccharomyces cereviseae[J]. Journal of Biological Chemistry, 1965, 240: 2531-2536. |
| [27] |
Sinha AK, Bhattacharjee JK. Control of a lysine-biosynthetic step by two unlinked genes of Saccharomyces[J]. Biochemical and Biophysical Research Communications, 1970, 39(6): 1205-1210. DOI:10.1016/0006-291X(70)90689-3 |
| [28] |
Nango E, Yamamoto T, Kumasaka T, et al. Structure of Thermus thermophilus homoisocitrate dehydrogenase in complex with a designed inhibitor[J]. Journal of Biochemistry, 2011, 150(6): 607-614. DOI:10.1093/jb/mvr097 |
| [29] |
Chen Y, Zuo RF, Zhu Q, et al. MoLys2 is necessary for growth, conidiogenesis, lysine biosynthesis, and pathogenicity in Magnaporthe oryzae[J]. Fungal Genetics and Biology, 2014, 67: 51-57. DOI:10.1016/j.fgb.2014.04.001 |
| [30] |
Yoshida A, Tomita T, Fujimura T, et al. Structural insight into amino group-carrier protein-mediated lysine biosynthesis. Crystal structure of the LysZ·LysW complex from Thermus thermophilus[J]. Journal of Biological Chemistry, 2015, 290(1): 435-447. |
| [31] |
Bhattacharjee JK, Strassman M. Accumulation of tricarboxylic acids related to lysine biosynthesis in a yeast mutant[J]. Journal of Biological Chemistry, 1967, 242(10): 2542-2546. |
| [32] |
Hogg RW, Broquist HP. Homocitrate formation in Neurospora crassa. Relation to lysine biosynthesis[J]. Journal of Biological Chemistry, 1968, 243(8): 1839-1845. |
| [33] |
Garrad RC, Bhattacharjee JK. Lysine biosynthesis in selected pathogenic fungi: characterization of lysine auxotrophs and the cloned LYS1 gene of Candida albicans[J]. Journal of Bacteriology, 1992, 174(22): 7379-7384. DOI:10.1128/JB.174.22.7379-7384.1992 |
| [34] |
Barnes DA, Thorner J. Genetic manipulation of Saccharomyces cerevisiae by use of the LYS2 gene[J]. Molecular and Cellular Biology, 1986, 6(8): 2828-2838. DOI:10.1128/MCB.6.8.2828 |
| [35] |
O'Doherty PJ, Lyons V, Tun NM, et al. Transcriptomic and biochemical evidence for the role of lysine biosynthesis against linoleic acid hydroperoxide-induced stress in Saccharomyces cerevisiae[J]. Free Radical Research, 2014, 48(12): 1454-1461. DOI:10.3109/10715762.2014.961448 |
| [36] |
Briones-Martin-Del-Campo M, Orta-Zavalza E, Cañas-Villamar I, et al. The superoxide dismutases of Candida glabrata protect against oxidative damage and are required for lysine biosynthesis, DNA integrity and chronological life survival[J]. Microbiology, 2015, 161(2): 300-310. |
| [37] |
Priyadarshini Y, Natarajan K. Reconfiguration of transcriptional control of lysine biosynthesis in Candida albicans involves a central role for the Gcn4 transcriptional activator[J]. mSphere, 2016, 1(1): e00016-15. |
| [38] |
Wu YY, Bao DP, Wang RJ, et al. Amino acid composition and nutritional evaluation of proteins in six samples of cultivated Flammulina velutipes[J]. Food Science, 2018, 39(10): 263-268. (in Chinese) 吴莹莹, 鲍大鹏, 王瑞娟, 等. 6种市售工厂化栽培金针菇的氨基酸组成及蛋白质营养评价[J]. 食品科学, 2018, 39(10): 263-268. DOI:10.7506/spkx1002-6630-201810040 |
| [39] |
Liu F, Wang W, Chen BZ, et al. Homocitrate synthase expression and lysine content in fruiting body of different developmental stages in Flammulina velutipes[J]. Current Microbiology, 2015, 70(6): 821-828. DOI:10.1007/s00284-015-0791-0 |
| [40] |
Xu HY. Mechanism of saccharopine dehydrogenase: the last enzyme in the lysine biosynthetic pathway in Saccharomyces cerevisiae[D]. Norman: Doctoral Dissertation of the University of Oklahoma, 2007 https://www.researchgate.net/publication/35880056_Mechanism_of_saccharopine_dehydrogenase_the_last_enzyme_in_the_lysine_biosynthetic_pathway_in_Saccharomyces_cerevisiae
|
| [41] |
Fujioka M, Nakatani Y. A kinetic study of saccharopine dehydrogenase reaction[J]. European Journal of Biochemistry, 1970, 16(1): 180-186. DOI:10.1111/j.1432-1033.1970.tb01070.x |
| [42] |
Xu HY, West AH, Cook PF. Overall kinetic mechanism of saccharopine dehydrogenase from Saccharomyces cerevisiae[J]. Biochemistry, 2006, 45(39): 12156-12166. DOI:10.1021/bi0610808 |
| [43] |
Fujioka M, Nakatani Y. Saccharopine dehydrogenase. A kinetic study of coenzyme binding[J]. Journal of Biological Chemistry, 1974, 249(21): 6886-6891. |
| [44] |
Fujioka M, Tanaka M. Enzymic and chemical synthesis of ɛ-N-(L-propionyl-2)-L-lysine[J]. European Journal of Biochemistry, 1978, 90(2): 297-300. DOI:10.1111/j.1432-1033.1978.tb12603.x |
| [45] |
Fujioka M. Saccharopine dehydrogenase. Substrate inhibition studies[J]. Journal of Biological Chemistry, 1975, 250(23): 8986-8989. |
| [46] |
Ford RA, Bhattacharjee JK. Molecular properties of the lys1+ gene and the regulation of α-aminoadipate reductase in Schizosaccharomyces pombe[J]. Current Genetics, 1995, 28(2): 131-137. DOI:10.1007/BF00315779 |
| [47] |
Ogawa H, Okamoto M, Fujioka M. Chemical modification of the active site sulfhydryl group of saccharopine dehydrogenase (L-lysine-forming)[J]. Journal of Biological Chemistry, 1979, 254(15): 7030-7035. |
| [48] |
Fujioka M, Nakatani Y. Saccharopine dehydrogenase. Interaction with substrate analogues[J]. European Journal of Biochemistry, 1972, 25(2): 301-307. DOI:10.1111/j.1432-1033.1972.tb01697.x |
| [49] |
Fujioka M. Chemical mechanism of saccharopine dehydrogenase (NAD+, L-lysine-forming) as deduced from initial rate pH studies[J]. Archives of Biochemistry and Biophysics, 1984, 230(2): 553-559. DOI:10.1016/0003-9861(84)90436-3 |
| [50] |
Burk DL, Hwang J, Kwok E, et al. Structural studies of the final enzyme in the α-aminoadipate pathway-saccharopine dehydrogenase from Saccharomyces cerevisiae[J]. Journal of Molecular Biology, 2007, 373(3): 745-754. DOI:10.1016/j.jmb.2007.08.044 |
| [51] |
Wan PJ, Yang L, Yuan SY, et al. RNA interference-aided knockdown of a putative saccharopine dehydrogenase leads to abnormal ecdysis in the brown planthopper, Nilaparvata lugens (Stål) (Hemiptera: Delphacidae)[J]. Bulletin of Entomological Research, 2015, 105(4): 390-398. DOI:10.1017/S0007485315000231 |
| [52] |
Fujita S, Cho SH, Yoshida A, et al. Crystal structure of LysK, an enzyme catalyzing the last step of lysine biosynthesis in Thermus thermophilus, in complex with lysine: Insight into the mechanism for recognition of the amino-group carrier protein, LysW[J]. Biochemical and Biophysical Research Communications, 2017, 491(2): 409-415. DOI:10.1016/j.bbrc.2017.07.088 |
| [53] |
Zhao DZ. Construction of Escherichia coli L-lysine producing strain based on metabolic engineering[D]. Chongqing: Master's Thesis of Chongqing University, 2017 (in Chinese) 赵德周.基于代谢工程构建大肠杆菌L-赖氨酸高产菌[D].重庆: 重庆大学硕士学位论文, 2017 http://cdmd.cnki.com.cn/Article/CDMD-10611-1017838865.htm |
| [54] |
Xu JZ, Yu HB, Han M, et al. Metabolic engineering of glucose uptake systems in Corynebacterium glutamicum for improving the efficiency of L-lysine production[J]. Journal of Industrial Microbiology & Biotechnology, 2019, 46(7): 937-949. |
| [55] |
Xu JZ, Han M, Zhang JL, et al. Metabolic engineering Corynebacterium glutamicum for the L-lysine production by increasing the flux into L-lysine biosynthetic pathway[J]. Amino Acids, 2014, 46(9): 2165-2175. DOI:10.1007/s00726-014-1768-1 |
| [56] |
Chen JZ. Breeding and evaluation of pyramiding transgenic rice with high lysine[D]. Yangzhou: Master's Thesis of Yangzhou University, 2014 (in Chinese) 陈锦珠.多基因聚合的高赖氨酸转基因水稻的选育及评价[D].扬州: 扬州大学硕士学位论文, 2014 http://cdmd.cnki.com.cn/Article/CDMD-11117-1014359449.htm |
 2020, Vol. 47
2020, Vol. 47