扩展功能
文章信息
- 谷洋, 连佳长, 黄磊, 蔡谨, 徐志南
- GU Yang, LIAN Jia-Zhang, HUANG Lei, CAI Jin, XU Zhi-Nan
- 毕赤酵母基因编辑技术研究进展
- Advances in gene editing of Pichia pastoris
- 微生物学通报, 2020, 47(2): 606-614
- Microbiology China, 2020, 47(2): 606-614
- DOI: 10.13344/j.microbiol.china.190257
-
文章历史
- 收稿日期: 2019-03-29
- 接受日期: 2019-05-28
- 网络首发日期: 2019-06-10
2. 浙江大学化学工程与生物工程学院合成生物学中心 浙江 杭州 310027
2. Center for Synthetic Biology, College of Chemical and Biological Engineering, Zhejiang University, Hangzhou, Zhejiang 310027, China
巴斯德毕赤酵母(Pichia pastoris)是一种甲醇营养型酵母,因其具有调控严谨、表达强度高、易于高密度发酵和能进行蛋白后修饰等优点,许多异源蛋白都选择毕赤酵母作为表达载体,如Liu等利用毕赤酵母和强启动子AOX1表达乙醇氧化酶,表达量能达到562 U/L[1],到目前为止已经有超过5 000种异源蛋白在毕赤酵母中成功表达[2]。此外,由于其具有作为细胞工厂的显著优点,除了重组蛋白外,S-腺苷-L-蛋氨酸[3]和D-阿拉伯糖[4]等初级和次级代谢产物,也开始在毕赤酵母中进行生产。
通过代谢工程改造不仅能够建立新的代谢通路,使菌株能够生产异源代谢产物,还能够调整菌株的代谢通量或删除一些代谢通路,从而提高同源蛋白或同源代谢产物的产量[5-6],例如Prabhu等通过6-磷酸葡萄糖酸酶(SOL3)和D-核酮糖-5-磷酸3-差向异构酶(RPE1)基因的协同合作,提高了PPP途径通量,从而提高了重组人干扰素γ的产量[7]。因此随着毕赤酵母的应用越来越广泛,近年来关于毕赤酵母的代谢工程研究越来越热门,而基因编辑技术作为代谢工程的一个重要工具,也越来越受人关注。除了传统的同源重组外,许多新型基因编辑技术也应运而生,比较热门的技术包括ZFN (zinc finger nuclease)技术、TALEN (transcription activator-like effector nuclease)技术和CRISPR/Cas (clustered regularly interspaced short palindromic repeats/Cas endonuclease)系统,其中CRISPR/Cas9系统是目前比较热门且使用较多的技术。但由于毕赤酵母属于非常规酵母,其遗传背景没有完全清晰,而且因为同源重组效率低等问题的存在,目前毕赤酵母中的基因编辑技术仍存在一些问题,其发展远不及酿酒酵母。基于这一背景,本文对毕赤酵母中基因编辑技术的发展和现状进行了总结,并结合相关邻域对毕赤酵母基因编辑技术的发展前景进行了展望。
1 传统基因编辑技术同源重组和Cre/loxP重组是新型基因编辑工具出现之前使用较多的用于毕赤酵母基因修饰的方法,直到现在仍是毕赤酵母中重要的代谢改造工具。
1.1 同源重组早期对毕赤酵母基因组进行定点基因整合和基因敲除都是通过同源重组(homologous recombination,HR)实现的,一般通过质粒进行基因整合时以单交换形式进行重组,如Liu等利用整合型质粒pPIC9K将乙醇氧化酶通过单交换的方式整合到基因组的HIS4位点[1]。通过片段进行基因整合或基因敲除时一般以双交换形式进行重组,例如Näätsaari等曾通过该方法敲除了毕赤酵母的Ku70基因[8],单交换和双交换的过程如图 1所示。HR是自然界中普遍存在的DNA修复机制,该机制的存在是为了对DNA上因损伤产生的双链断裂(double strand break,DSB)进行修复,因此该机制也是新型基因编辑技术的基础。该方法修复DSB需要同源模板的存在,这是一种保真度较高的修复方法。HR修复的过程如下,首先一个由核酸酶Mre11和Exo1、解螺旋酶Sgs11和核酶Dna2组成的核酶复合体会对DSB的5ʹ末端进行消化,产生带有3ʹ末端的单链DNA。而后ssDNA会在Rad51、Rad52、Dmc1以及解螺旋酶Rad59等相关酶[9]的协助下寻找同源模板进行退火和DNA链的交换。寻找到同源模板后,ssDNA会与同源序列配对形成D-Loop结构,通过D-Loop延伸完成修复过程[10]。在酵母中Rad59可以促进ssDNA的退火过程,Rad52可以促进对同源模板和外源侵入的同源序列的搜索和交换[11-12],因此这两种酶都能促进HR的效率。

|
| 图 1 同源重组和非同源末端连接 Figure 1 Homologous recombination and non-homologous end joining |
|
|
除HR外,机体内还存在另外一种重要的DNA损伤修复方式,那便是非同源末端连接(non- homologous end joining,NHEJ),修复过程如图 1所示。参与NHEJ过程的一种重要的蛋白是Ku蛋白,该蛋白是Ku70和Ku80的异二聚体,用于识别DSB。当Ku蛋白识别到DSB后,便会结合到断裂末端,召集DNA-PKcs (DNA-dependent protein kinase catalytic subunit)通过自磷酸化激活NHEJ修复,而后利用Artemis重组酶加工DNA末端,最后通过DNA连接酶将处理完的末端进行连接,达到修复DNA链断裂的目的[13]。与同源重组不同,非同源末端连接不需要同源模板的参与,利用该过程对DSB进行修复更为简便,但是也更容易导致基因的插入、缺失和点突变等错误修复。
不同物种中HR和NHEJ发生的概率并不相同,在毕赤酵母中NHEJ的效率远高于HR,这就导致在毕赤酵母中同源重组的效率比较低。但是通过提高同源臂长度,例如使用1 000 bp甚至更长的同源臂可以在一定程度上提高同源重组的效率。考虑到蛋白Ku70是识别DSB的Ku蛋白复合体中的一部分,为提高HR效率,Näätsaari等通过敲除表达Ku70蛋白的基因来降低NHEJ效率,敲除该基因后明显提高了同源重组的效率,使用250 bp同源臂对HIS4进行敲除时效率从3%提高到了96.6%[8]。
因为同源重组转化效率低,在使用同源重组进行整合和敲除时会使用一些筛选标签,提高筛选阳性率,而筛选标签的存在往往会对菌体的生长和后续的实验产生影响。为解决这个问题,可以在重组过程中引入一些反选标记,便于后续去除这些筛选标签,比如利用URA3、URA5和来自雄性不育玉米的T-urf13以及来自大肠杆菌的mazf基因来提高筛选效率[14]。
1.2 Cre/loxP系统反选标记虽然可以提高转化筛选的阳性率,但是这些基因的存在容易导致菌体生长缓慢甚至死亡。除使用反选标记之外,还可以采用Cre/loxP系统对常规筛选标记进行剔除,Marx等就曾利用该方法敲除替换启动子时带入的抗性基因[15],该系统操作简便,而且不会对细胞产生毒性,因此应用广泛。
Cre/loxP系统是20世纪80年代末发展起来的一种基因修饰技术,源于P1噬菌体[16]。Cre是一种位点特异性重组酶,是由343个氨基酸组成的一种双结构域蛋白,两个结构域分别是N-端结构域(N-terminal domain,NTD)和C-端结构域(C-terminal domain,CTD)。Cre重组酶能够特异性识别loxP位点,对两个位点间的基因进行重组[17]。loxP位点由两个13 bp的反向重复序列和8 bp的间隔序列组成,间隔序列位于两个反向重复序列中间,决定了loxP位点的方向。两边的两个反向重复序列分别和间隔序列1、8位上的碱基组成两个14 bp的重组酶结合元件(recombinase-binding element,RBE),一个RBE的突变对酶和位点的结合影响不大,但当两个RBE都发生突变时,Cre重组酶与loxP位点的结合效率将会大大下降[14]。当Cre重组酶的NTD识别到loxP位点时,便会结合到loxP位点的一个RBE上,而后两个结构域将DNA双链包围,形成一个如图 2A所示的“C”型的夹子。两个loxP位点一共结合4个Cre重组酶,4个Cre酶会组成一个四聚体,由结构域CTD对DNA序列进行切割,切割位点分别位于两个间隔序列的1/2位和7/8之间[17-18],如图 2B所示。
Cre酶介导两个位点间基因的重组方式取决于两个loxP位点的方向,根据不同的情况,一共分为3种重组方式:(1)当两个位点在同一条DNA链上,且方向相同时,Cre重组酶可以对2个位点间的序列进行切除;(2)当两个位点在同一条链上,且方向相反时,Cre重组酶能够将两个位点间的基因方向调换;(3)当两个位点位于不同的两条链上时,Cre重组酶能够介导两条链之间的基因交换。目前在毕赤酵母中使用较多的是基因敲除功能,倒位和易位的功能使用较少。
虽然通过Cre/loxP系统进行基因敲除操作简便,但是敲除后会留下一个loxP位点,从而影响后续使用此方法进行基因敲除。为解决此问题,根据loxP位点和Cre重组酶的性质,可以使用两个不同loxp位点,每个位点带有一个突变的RBE。Pan等就曾使用各带有一个突变RBE的loxP71和loxP66对抗性筛选标签进行了敲除[14],敲除后剩下了带有两个突变RBE的loxP72,因为两个RBE都发生突变,loxP72和Cre重组酶结合效率极低,因此不会影响后续使用该系统。
2 新型基因组编辑技术近年来基因组编辑技术迅猛发展,许多使用更为方便的基因组编辑技术相继出现。目前比较常用的技术包括ZFN、TALEN和近年来发展迅速的CRISPR/Cas系统。这些技术都是利用特异性核酸酶在相应位点产生切口,而后通过酵母自身的同源重组修复机制来实现基因组的编辑,通过这些技术理论上可以对目的基因进行精确的定点改造。
2.1 ZFN和TALEN技术ZFN属于第一代基因编辑技术,是一种人工合成的融合蛋白,由一个可以识别特异性DNA序列的锌指蛋白(zinc finger protein,ZFP)和一个非特异性核酸内切酶FokⅠ组成。ZFP是由一系列的Cys2-His2锌指蛋白模块(ZF)组成,这种结构是1986年在真核生物转录因子家族的DNA结合区发现的。每个锌指蛋白模块可以识别3−4个连续的碱基,并结合到识别的DNA上。根据这个特性,Kim将核酸内切酶FokⅠ连接到由几个ZF单元组成的ZFP上,利用锌指蛋白对DNA的识别和结合功能,将内切酶引导到目标基因处[19-20]。FokⅠ只在二聚体形式下才表现剪切活性,因此ZFNs都是以二聚体形式来发挥作用的。当两个识别位点距离在6−8 bp时[21],两个ZFNs会分别结合到DNA双链的两侧各剪切一条链,产生DSB,而后通过同源重组修复DNA断裂来实现基因的插入或敲除(图 3)。目前ZFN已经成功在果蝇、斑马鱼和酵母等多种真核细胞中成功应用,但是因为并不是所有的核苷酸三联体都有相应的ZF模块,而且ZFP设计过程复杂,导致使用该方法进行基因编辑耗时耗力,很大程度上限制了该方法的使用。
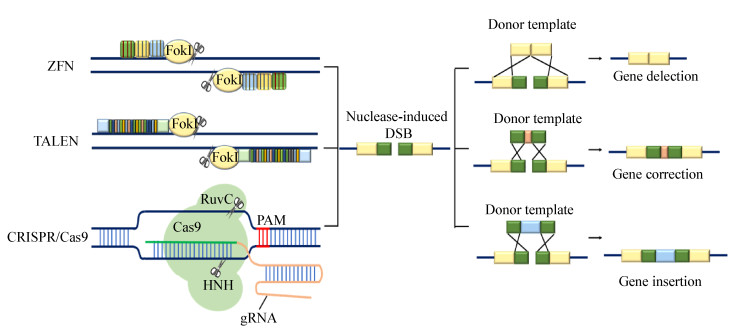
|
| 图 3 三大特异性核酸酶介导的基因编辑技术 Figure 3 Three specific nuclease mediated gene editing techniques |
|
|
TALEN技术与ZFN相似,也是通过位点特异性蛋白实现进行基因编辑(图 3)。与ZFN不同的是,TALEN利用的TALE蛋白来自黄单胞菌,与DNA结合的核心区域由1−33个高度保守的重复序列串联而成,每个重复单位包含33−35个氨基酸。重复单元中的第12和13位上的氨基酸残基决定了该单元识别的特异性,称为重复可变残基(repeat variable residues,RVD)[22]。TALEN切割DNA的方式与ZFN相似,两个TALEN融合蛋白的靶序列间隔一般为16−20 bp[23]。TALEN的设计和构建比ZFN简便,但是因为高度重复的序列的存在,使质粒载体的设计构建具有一定难度,而Golden gate[24]、FLASH (fast ligation-based automatable solid-phase high-throughput)[25]等方法的出现简化了载体构建过程。ZFN和TALEN技术都是新型的基因编辑技术,可以进行定点编辑,但是由于使用起来比较复杂,而且在这两种技术刚出现不久,操作更为简便的CRISPR技术便出现了,因此目前这两种技术并没有在毕赤酵母中得到广泛应用。
2.2 CRISPR/Cas9系统CRISPR/Cas9系统源于细菌和古细菌的免疫防御系统,用于抵御病毒和外源DNA的入侵,是Ⅱ类CRISPR系统中应用最广泛的,该系统只需一种Cas蛋白就能发挥作用,而Ⅰ和Ⅲ类CRISPR系统需要多种蛋白共同发挥作用[26]。与ZFN和TALEN利用蛋白定位不同,CRISPR/Cas9系统中的核酸酶Cas9是通过RNA来进行序列定位的。Cas9是一种能与RNA结合,并由RNA引导的核酸酶,有两个有剪切活性的结构域——HNH和RuvC,能将DNA双链切断[27]。
引导Cas9的RNA由两部分组成,一部分是靶向目标系列的crRNA (CRISPR targeting RNA),该部分是由前体RNA (pre-crRNA)加工而来的一段含有保守重复序列和间隔序列的RNA,可以通过修改间隔序列靶向不同的DNA序列。另一部分是与重复序列互补的反式激活crRNA,tracrRNA (trans-activating crRNA)。这两部RNA相互结合后与Cas9形成一个复合体,将Cas9引导到靶标序列,在crRNA互补序列下游临近PAM位点区对DNA进行剪切,产生DSB,从而激发HR或NHEJ。为方便使用,Jinek等将两部分进行简化,形成了由一个结构RNA和20 bp靶向目标基因的可变RNA组成的single-guide RNA (gRNA或sgRNA)[28],从而使该系统使用更简便。
CRISPR/Cas9系统虽然操作简便,但是Cas9蛋白序列和gRNA序列的选择和表达等多种因素都会影响编辑效率。对于不同的物种,我们需要选择不同的条件来达到较高的效率。Cas的表达主要涉及到启动子的强弱,序列密码子的优化。在酿酒酵母中的研究发现,使用强启动子表达Cas9会对菌株的生长造成负面影响,但选择诱导型启动子又会因表达时间不够,导致编辑效率下降至20%以下,而选用组成型的弱启动子不仅不会影响菌株生长还能保证较高的编辑效率[29]。除启动子外,Cas9蛋白序列的选择也是影响编辑效率的重要因素,目前使用较为广泛的Cas9蛋白是来自于化脓性链球菌(Streptococcus pyogenes)的SpCas9[27, 29],但Weninger等通过研究发现,SpCas9和PpCas9 (由SpCas9经过毕赤酵母表达密码子优化得到)在毕赤酵母中的编辑效率都很低,而经过人源密码子优化的HsCas9在毕赤酵母中能发挥较好的作用[30]。
除Cas9蛋白外,gRNA对编辑效率也有较大影响。首先gRNA序列的选择直接影响到了和靶序列结合的正确率。此外不同的启动子对sgRNA的结构会产生一些影响。RNA聚合酶Ⅱ (RNA pol Ⅱ)和RNA聚合酶Ⅲ (RNA pol Ⅲ)的功能不同,转录的RNA结构也不同。RNA pol Ⅱ用于合成mRNA和snRNA,转录得到的RNA会带有5ʹ帽子和3ʹ polyA尾巴,这些结构对sgRNA与Cas9的复合体形成具有较大影响[29]。RNA pol Ⅲ主要用于合成tRNA、rRNA 5S以及细胞中的一些细小的RNA,因此由RNA pol Ⅲ转录的RNA没有5ʹ帽子和3ʹ polyA尾巴,如图 4所示,转录的sgRNA能在细胞核中停留的时间更长,因此理论上使用RNA pol Ⅲ启动子表达sgRNA更合适[31]。但是由于RNA pol Ⅲ启动子表达sgRNA表达强度低,稳定性差[32-33],导致在多种酵母中利用该启动子的编辑效率都十分低[30, 33]。
为了在提高sgRNA表达量和稳定性的同时保证表达的sgRNA不会因多余的结构导致无法与Cas9蛋白结合,Gao等通过在两端分别加上核酶HH和HDV来切除RNA pol Ⅱ转录出来的sgRNA的5ʹ帽子和3ʹ polyA尾巴[34-35]。Weninger等利用此方法,在毕赤酵母中构建了编辑效率达到80%以上的CRISPR/Cas9系统[30],后续又通过敲除Ku70基因进一步提高了该系统的效率[36]。但是由于HH核酶的前6个碱基需根据gRNA变化,导致质粒构建比较麻烦,为方便使用该系统的使用Prielhofer等利用Golden gate来构建质粒,解决了这一问题[37]。
2.3 碱基编辑器碱基编辑器(base editor)是基于CRISPR/Cas9系统的一种无需通过DSB便能进行基因编辑的技术,通过该技术可对基因组进行单碱基突变或终止密码子突变[38]。该方法将没有剪切活性的Cas9蛋白(dCas9)与胞苷脱氨酶或腺苷脱氨酶进行融合,形成了CBE和ABE两种工具,通过CRISPR/dCas的靶向功能,将胞苷脱氨酶或腺苷脱氨酶引导到目标序列进行基因编辑[39]。其中胞苷脱氨酶可以将胞苷脱氨变成尿嘧啶核苷酸,而后进一步转变为胸腺嘧啶核苷酸,从而实现C·G到A·T的转变,这便是第一代的碱基编辑器——BE1。但BE1在使用时由于自身的修复功能,导致编辑效率低下,为提高编辑效率,出现了二代系统BE2,该系统通过融合尿嘧啶糖基酶抑制蛋白(uracyl glycosylase inhibitor,UGI)来避免U·G被重新修复成C·G,从而提高转化效率。为进一步提高效率,三代系统BE3使用Cas9 nickase替代了dCas9,该系统能在非编辑链上产生切口从而促进C·G转变为A·T[39-40],进一步提高了编辑效率。将腺苷脱氨酶与dCas9融合可以形成ABE系统,该系统的作用与CBE相反,可以将A·T转变为C·G的[41]。理论上利用CBE和ABE可以实现4种碱基之间的转变(C到T,A到G,T到C和G到A)[42],从而随意地对基因组进行修改。但因为该系统不产生DSB,因此难以完成整段基因地整合和敲除,但可以通过终止密码子突变使目标基因失去功能。同时又因为该方法无须在DNA上产生DSB后依靠HR来实现基因编辑,因此对HR效率低的菌株而言这是一种很好的基因编辑手段,目前该技术已经成功在酿酒酵母中使用,但尚未有在毕赤酵母中使用的例子[42]。
3 总结与展望毕赤酵母是一种重要的蛋白表达菌株,建立简洁高效的基因编辑系统对毕赤酵母的代谢改造十分重要,但由于毕赤酵母属于非常规酵母,其遗传背景没有完全清晰,因此基因编辑手段的发展也不如酿酒酵母等常规酵母。目前使用较多的还是传统的同源重组和新发展的CRISPR技术,但是毕赤酵母中同源重组的效率较低,虽然敲除Ku70后同源重组的效率能得到提高,但是因此主要修复方式受损,其生长速率也受到了较大影响[8]。此外CRISPR/Cas9系统也还有待优化。首先因为毕赤酵母中同源重组的效率比较低,因此相较于酿酒酵母,毕赤酵母中的CRISPR/Cas9系统需要较长的同源臂,而且在多重基因同时敲除时的效率也较低[30]。除此之外,毕赤酵母中目前还没有建立起类似于酿酒酵母中集转录激活、转录抑制和基因敲除于一体的多功能CRISPR系统[43]。
要提高同源重组效率,除了抑制或敲除Ku70[8]和Dnl4p[44]等NHEJ相关基因外,还可以过表达RAD52、RAD51[45]等HR相关基因来提高HR效率。此外因为目前毕赤酵母中CRISPR/Cas9系统大多是以质粒形式存在的,因此质粒的稳定性及拷贝数等因素对于编辑效率也会产生一定影响。可以尝试用不同的毕赤酵母复制子构建CRISPR/Cas9质粒系统,例如传统的PARS1复制子和近些年发现的mtDNA[46],panARS[47],以及centromeric DNA[48],寻找稳定性较好,拷贝数较合适的复制子来提高编辑效率,例如Cao等就曾通过使用更稳定的着丝粒DNA构建质粒提高了Scheffersornyces stipitis中CRISPR/Cas9的编辑效率[49]。除目前发现的复制子,后续可以寻找更多的毕赤酵母复制子进行验证,以构建更为高效稳定的编辑系统。
除了优化毕赤酵母中现有的基因编辑系统外,我们还可以借鉴其他基因编辑体系,在毕赤酵母中建立新的基因编辑手段。除上文提到的可以在毕赤酵母中建立CBEs和ABEs系统外,结合目前CRISPR系统的最新进展,还可以在毕赤酵母中建立CRISPR/Cpf1系统来提高编辑效率。CRISPR/Cpf1 (CRISPR/Cas12a)是一种新型的CRIPSR/Cas系统,该系统已经成功在酿酒酵母等物种中使用[50-51],与Cas9相比Cpf1分子量更小且只需一个协助的RNA,因此更方便,剪切后能产生粘性末端,具有更高的编辑效率[52],因此CRISPR/Cpf1的建立或许对毕赤酵母基因编辑技术进步具有重要意义。
| [1] |
Liu YP, Pan JF, Wei PL, et al. Efficient expression and purification of recombinant alcohol oxidase in Pichia pastoris[J]. Biotechnology and Bioprocess Engineering, 2012, 17(4): 693-702. DOI:10.1007/s12257-011-0660-z |
| [2] |
Ahmad M, Hirz M, Pichler H, et al. Protein expression in Pichia pastoris:recent achievements and perspectives for heterologous protein production[J]. Applied Microbiology and Biotechnology, 2014, 98(12): 5301-5317. DOI:10.1007/s00253-014-5732-5 |
| [3] |
Yu P, Shen XQ. Enhancing the production of S-adenosyl-L-methionine in Pichia pastoris GS115 by metabolic engineering[J]. AMB Express, 2012, 2: 57. DOI:10.1186/2191-0855-2-57 |
| [4] |
Cheng HR, Lv JY, Wang HW, et al. Genetically engineered Pichia pastoris yeast for conversion of glucose to xylitol by a single-fermentation process[J]. Applied Microbiology and Biotechnology, 2014, 98(8): 3539-3552. DOI:10.1007/s00253-013-5501-x |
| [5] |
Prabhu AA, Boro B, Bharali B, et al. Gene and process level modulation to overcome the bottlenecks of recombinant proteins expression in Pichia pastoris[J]. Current Pharmaceutical Biotechnology, 2017, 18(15): 1200-1223. |
| [6] |
Baghban R, Farajnia S, Ghasemi Y, et al. New developments in Pichia pastoris expression system, review and update[J]. Current Pharmaceutical Biotechnology, 2018, 19(6): 451-467. DOI:10.2174/1389201019666180718093037 |
| [7] |
Prabhu AA, Veeranki VD. Metabolic engineering of Pichia pastoris GS115 for enhanced pentose phosphate pathway (PPP) flux toward recombinant human interferon gamma (hIFN-γ) production[J]. Molecular Biology Reports, 2018, 45(5): 961-972. DOI:10.1007/s11033-018-4244-2 |
| [8] |
Näätsaari L, Mistlberger B, Ruth C, et al. Deletion of the Pichia pastoris KU70 homologue facilitates platform strain generation for gene expression and synthetic biology[J]. PLoS One, 2012, 7(6): e39720. DOI:10.1371/journal.pone.0039720 |
| [9] |
Bellido A, Hermosa B, Ciudad T, et al. Role of homologous recombination genes RAD51, RAD52, and RAD59 in the repair of lesions caused by γ-radiation to cycling and G2/M-arrested cells of Candida albicans[J]. Cellular Microbiology, 2018, 20(12): e12950. DOI:10.1111/cmi.12950 |
| [10] |
Morrical SW. DNA-pairing and annealing processes in homologous recombination and homology-directed repair[J]. Cold Spring Harbor Perspectives in Biology, 2015, 7(2): a016444. DOI:10.1101/cshperspect.a016444 |
| [11] |
Bi BY, Rybalchenko N, Golub EI, et al. Human and yeast Rad52 proteins promote DNA strand exchange[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(26): 9568-9572. DOI:10.1073/pnas.0403205101 |
| [12] |
Feng Q, Düring L, de Mayolo AA, et al. Rad52 and Rad59 exhibit both overlapping and distinct functions[J]. DNA Repair, 2007, 6(1): 27-37. |
| [13] |
Daley JM, Palmbos PL, Wu DL, et al. Nonhomologous end joining in yeast[J]. Annual Review of Genetics, 2005, 39: 431-451. DOI:10.1146/annurev.genet.39.073003.113340 |
| [14] |
Pan RQ, Zhang J, Shen WL, et al. Sequential deletion of Pichia pastoris genes by a self-excisable cassette[J]. FEMS Yeast Research, 2011, 11(3): 292-298. DOI:10.1111/j.1567-1364.2011.00716.x |
| [15] |
Marx H, Mattanovich D, Sauer M. Overexpression of the riboflavin biosynthetic pathway in Pichia pastoris[J]. Microbial Cell Factories, 2008, 7: 23. DOI:10.1186/1475-2859-7-23 |
| [16] |
van Duyne GD. A structural view of Cre-loxP site-specific recombination[J]. Annual Review of Biophysics and Biomolecular Structure, 2001, 30(30): 87-104. |
| [17] |
Guo F, Gopaul DN, van Duyne GD. Structure of Cre recombinase complexed with DNA in a site-specific recombination synapse[J]. Nature, 1997, 389(6646): 40-46. DOI:10.1038/37925 |
| [18] |
van Duyne GD. Cre recombinase[J]. Microbiology Spectrum, 2015, 3(1): MDNA3-0014-2014. |
| [19] |
Kim YG, Shi Y, Berg JM, et al. Site-specific cleavage of DNA-RNA hybrids by zinc finger/Fok I cleavage domain fusions[J]. Gene (Amsterdam), 1997, 203(1): 43-49. DOI:10.1016/S0378-1119(97)00489-7 |
| [20] |
Kim YG, Cha J, Chandrasegaran S. Hybrid restriction enzymes:zinc finger fusions to Fok I cleavage domain[J]. Proceedings of the National Academy of Sciences of the United States of America, 1996, 93(3): 1156-1160. DOI:10.1073/pnas.93.3.1156 |
| [21] |
Bibikova M, Carroll D, Segal DJ, et al. Stimulation of homologous recombination through targeted cleavage by chimeric nucleases[J]. Molecular and Cellular Biology, 2001, 21(1): 289-297. DOI:10.1128/MCB.21.1.289-297.2001 |
| [22] |
Miller JC, Tan SY, Qiao GJ, et al. A TALE nuclease architecture for efficient genome editing[J]. Nature Biotechnology, 2011, 29(2): 143-148. DOI:10.1038/nbt.1755 |
| [23] |
Geiβler R, Scholze H, Hahn S, et al. Transcriptional activators of human genes with programmable DNA-specificity[J]. PLoS One, 2011, 6(5): e19509. DOI:10.1371/journal.pone.0019509 |
| [24] |
Welker JM, Wierson WA, Wang Y, et al. GoldyTALEN vectors with improved efficiency for golden gate TALEN assembly[J]. Human Gene Therapy, 2016, 27(6): 423-424. DOI:10.1089/hum.2016.012 |
| [25] |
Reyon D, Tsai SQ, Khayter C, et al. FLASH assembly of TALENs for high-throughput genome editing[J]. Nature Biotechnology, 2012, 30(5): 460-465. DOI:10.1038/nbt.2170 |
| [26] |
Barrangou R. The roles of CRISPR-Cas systems in adaptive immunity and beyond[J]. Current Opinion in Immunology, 2015, 32: 36-41. DOI:10.1016/j.coi.2014.12.008 |
| [27] |
Lian JZ, HamediRad M, Zhao HM. Advancing metabolic engineering of Saccharomyces cerevisiae using the CRISPR/Cas system[J]. Biotechnology Journal, 2018, 13(9): 1700601. DOI:10.1002/biot.201700601 |
| [28] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [29] |
Raschmanová H, Weninger A, Glieder A, et al. Implementing CRISPR-Cas technologies in conventional and non-conventional yeasts:Current state and future prospects[J]. Biotechnology Advance, 2018, 36(3): 641-665. DOI:10.1016/j.biotechadv.2018.01.006 |
| [30] |
Weninger A, Hatzl AM, Schmid C, et al. Combinatorial optimization of CRISPR/Cas9 expression enables precision genome engineering in the methylotrophic yeast Pichia pastoris[J]. Journal of Biotechnology, 2016, 235: 139-149. DOI:10.1016/j.jbiotec.2016.03.027 |
| [31] |
DiCarlo JE, Norville JE, Mali P, et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems[J]. Nucleic Acids Research, 2013, 41(7): 4336-4343. DOI:10.1093/nar/gkt135 |
| [32] |
Turowski TW, Tollervey D. Transcription by RNA polymerase Ⅲ:insights into mechanism and regulation[J]. Biochemical Society Transactions, 2016, 44(5): 1367-1375. DOI:10.1042/BST20160062 |
| [33] |
de Vries ARG, de Groot PA, van Den Broek M, et al. CRISPR-Cas9 mediated gene deletions in lager yeast Saccharomyces pastorianus[J]. Microbial Cell Factories, 2017, 16: 222. DOI:10.1186/s12934-017-0835-1 |
| [34] |
Gao YB, Zhao YD. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing[J]. Journal of Integrative Plant Biology, 2014, 56(4): 343-349. DOI:10.1111/jipb.12152 |
| [35] |
Zhang T, Gao Y, Wang R, et al. Production of Guide RNAs in vitro and in vivo for CRISPR using ribozymes and RNA polymerase Ⅱ promoters[J]. Bio-protocol, 2017, 7(4): e2148. DOI:10.21769/BioProtoc.2148 |
| [36] |
Weninger A, Fischer JE, Raschmanová H, et al. Expanding the CRISPR/Cas9 toolkit for Pichia pastoris with efficient donor integration and alternative resistance markers[J]. Journal of Cellular Biochemistry, 2018, 119(4): 3183-3198. DOI:10.1002/jcb.26474 |
| [37] |
Prielhofer R, Barrero JJ, Steuer S, et al. GoldenPiCS:A Golden Gate-derived modular cloning system for applied synthetic biology in the yeast Pichia pastoris[J]. BMC Systems Biology, 2017, 11: 123. DOI:10.1186/s12918-017-0492-3 |
| [38] |
Komor AC, Badran AH, Liu DR. Editing the genome without double-stranded DNA breaks[J]. ACS Chemical Biology, 2018, 13(2): 383-388. DOI:10.1021/acschembio.7b00710 |
| [39] |
Nishida K, Arazoe T, Yachie N, et al. Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems[J]. Science, 2016, 353(6305): aaf8729. DOI:10.1126/science.aaf8729 |
| [40] |
Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature, 2016, 533(7603): 420-424. DOI:10.1038/nature17946 |
| [41] |
Gaudelli NM, Komor AC, Rees HA, et al. Programmable base editing of A·T to G·C in genomic DNA without DNA cleavage[J]. Nature, 2017, 551(7681): 464-471. DOI:10.1038/nature24644 |
| [42] |
Rees HA, Liu DR. Base editing:precision chemistry on the genome and transcriptome of living cells[J]. Nature Reviews Genetics, 2018, 19(12): 770-788. DOI:10.1038/s41576-018-0059-1 |
| [43] |
Lian JZ, Hamedirad M, Hu SM, et al. Combinatorial metabolic engineering using an orthogonal tri-functional CRISPR system[J]. Nature Communications, 2017, 8(1): 1688. DOI:10.1038/s41467-017-01695-x |
| [44] |
Ito Y, Watanabe T, Aikawa S, et al. Deletion of DNA ligase Ⅳ homolog confers higher gene targeting efficiency on homologous recombination in Komagataella phaffii[J]. FEMS Yeast Research, 2018, 18(7): foy074. |
| [45] |
Yasuhara T, Kato R, Hagiwara Y, et al. RAD52 promotes transcription-associated homologous recombination repair[J]. Cancer Discovery, 2018, 8(11): OF7. DOI:10.1158/2159-8290.CD-RW2018-165 |
| [46] |
Schwarzhans JP, Luttermann T, Wibberg D, et al. A mitochondrial autonomously replicating sequence from Pichia pastoris for uniform high level recombinant protein production[J]. Frontiers in Microbiology, 2017, 8: 780. DOI:10.3389/fmicb.2017.00780 |
| [47] |
Camattari A, Goh A, Yip LY, et al. Characterization of a panARS-based episomal vector in the methylotrophic yeast Pichia pastoris for recombinant protein production and synthetic biology applications[J]. Microbial Cell Factories, 2016, 15: 139. DOI:10.1186/s12934-016-0540-5 |
| [48] |
Nakamura Y, Nishi T, Noguchi R, et al. A stable, autonomously replicating plasmid vector containing Pichia pastoris centromeric DNA[J]. Applied and Environmental Microbiology, 2018, 84(15): e02882-17. DOI:10.1128/AEM.02882-17 |
| [49] |
Cao MF, Gao MR, Lopez-Garcia CL, et al. Centromeric DNA facilitates nonconventional yeast genetic engineering[J]. ACS Synthetic Biology, 2017, 6(8): 1545-1553. DOI:10.1021/acssynbio.7b00046 |
| [50] |
Verwaal R, Buiting-Wiessenhaan N, Dalhuijsen S, et al. CRISPR/Cpf1 enables fast and simple genome editing of Saccharomyces cerevisiae[J]. Yeast, 2018, 35(2): 201-211. DOI:10.1002/yea.3278 |
| [51] |
Li ZH, Liu M, Wang FQ, et al. Cpf1-assisted efficient genomic integration of in vivo assembled DNA parts in Saccharomyces cerevisiae[J]. Biotechnology Letters, 2018, 40(8): 1253-1261. DOI:10.1007/s10529-018-2574-8 |
| [52] |
Zhu HX, Liang C. CRISPR-DT:designing gRNAs for the CRISPR-Cpf1 system with improved target efficiency and specificity[J]. Bioinformatics, 2019, 35(16): 2783-2789. DOI:10.1093/bioinformatics/bty1061 |
 2020, Vol. 47
2020, Vol. 47






