扩展功能
文章信息
- 尹珺, 马安周, 宋茂勇, 种国双, 庄绪亮, 庄国强
- YIN Jun, MA An-Zhou, SONG Mao-Yong, CHONG Guo-Shuang, ZHUANG Xu-Liang, ZHUANG Guo-Qiang
- 合成微生物体系研究进展
- Research progress in synthetic microbial systems
- 微生物学通报, 2020, 47(2): 583-593
- Microbiology China, 2020, 47(2): 583-593
- DOI: 10.13344/j.microbiol.china.190464
-
文章历史
- 收稿日期: 2019-05-29
- 接受日期: 2019-10-12
- 网络首发日期: 2019-11-04
2. 中国科学院大学 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
地球上数量及种类最为丰富的便是微生物,其对于地球上的生物地球化学循环具有重要意义。最开始人们将目光集中于微生物单群体,作为微生物群落中的关键功能群体将其分离、改造,以实现目标功能的加强,这是合成生物学的早期研究焦点[1]。然而人们忽视了自然界中的微生物并不是单独存在的,它们处于复杂的、动态的群落中,正是它们的相互作用推动了群落的稳定、功能及进化[2-3]。在面临包括环境修复和传染病在内的许多当代挑战时,功能单一的菌种已经无法满足需求,科学家们对于多种微生物组合成群落的研究兴趣越来越高,由此从合成生物学中拓展了合成生态学这一领域。作为合成生物学的新兴分支,合成生态学能够帮助科学家们构建具有理想特性的微生物群落,形成合成微生物体系,为剖析微生物群落内部的作用机制提供新的途径,为开拓微生物资源提供新的见解[4]。本文将详细介绍合成微生物体系的概念、由来、构建原则及应用理论基础,并且总结概括其近几年在人体健康、工业生产、污染修复方面的应用。
1 合成微生物体系的概念及由来微生物在生物地球化学循环以及人类的健康中扮演着重要的角色,一方面是由于微生物种群数量众多,其功能也是多种多样;另一方面是由于微生物种群并不是独立的个体,而是相互联系、相互作用组成群落。然而也正是因为种群数量巨大以及复杂的相互作用网络,使得直接研究自然环境中微生物种群的难度大大增加,并且难以预测自然环境中的微生物群落行为,这对于了解自然环境体系中的物质迁移与转化、微生物功能作用以及开发利用微生物资源具有很大的局限性。
合成微生物体系的出现很好地解决了这一问题,它是自然环境中微生物体系的简化模型,可以根据研究的需要对其组成成员进行定义,控制影响因素,降低研究对象的复杂度。合成微生物体系指的是在特征良好且受控的环境中,将两个或多个微生物种群组合在一起,通过自下向上的方法创建合理设计的微生物群落[5]。设计合成新的微生物群落既可以选择特定的野生型物种组合,也可以构建转基因物种的生态系统,设计诱导新的物种间相互作用,例如代谢交换、群体感应等[6]。
除了简化性与可定义性,合成微生物体系呈现出的一系列特征还包括模块化、鲁棒性、可预测性、可伸缩性、稳定性[7]。人工合成微生物群落的关键在于搭建微生物物种之间的相互作用关系,在这一过程中需要进行微生物的功能模块划分[8];相互作用的微生物对于外界环境的扰动具有一定的弹性及抗性,构成微生物群落的鲁棒性[9];相较于自然微生物群落,合成微生物群落更透明,因而能够实现精确控制及监测,并预测其种群行为;合成微生物体系所包含的成员可以为1种、2种或3种等,合成群落大小视研究目标而定,微生物在群落中竞争资源的同时交换资源,构成了动态、稳定的群落。
人工构建合成微生物群落的主要目的包括:研究种群之间的自然相互作用,建立种群之间的代谢网络[10]。在早期的研究中,Kato等[11]研究纤维素降解菌群的稳定性机制,即通过构建剔除一个菌群成员的敲除群落,以此来确定各个成员的功能,明确菌群中的相互作用关系。Shou等[12]通过对酵母菌进行基因改造,获得两个代谢能力不同的非交配菌株,混合形成CoSMO系统,并且呈现出一定的稳定性。正是关于CoSMO系统的研究在合成生物学中扩展了合成生态学这一新领域,它提供了一个定量系统,将细胞层面的过程与系统层面的集体行为联系起来。相较于合成生物学倾向于将工程应用于单菌群体,合成生态学更加注重于多菌群体的设计组合。在近几年的研究中,随着基因技术的发展及数学模型的深入,合成微生物体系常常与基因组技术及计算机联合,使得系统的作用机制更为明晰,群落行为也能得到很好的预测[13-14]。
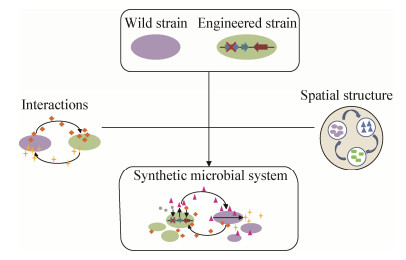
2 合成微生物体系的构建原则在设计与构建合成微生物体系时,主要考虑两点:其一是微生物间的相互作用,其二是合成微生物群落的时空组织结构(图 1)。选择合适的微生物物种来实现微生物之间的相互作用是达到实验目的的关键,而如何从时空结构上来组装这些微生物,产生理想的群落级行为,并且同时实现合成体系的可操纵性与可控性,这对于合成微生物体系的构建具有重要意义。
2.1 微生物之间的相互作用在群落中,微生物通过各种模式相互作用,例如竞争、捕食、共生、寄生等,并且往往参与多个相互作用,在竞争资源的同时也交换资源,这一过程促成了微生物间的依赖关系[16]。因而在合成微生物体系中要构建稳定的种间关系,关键是如何设计微生物间构成相互作用,在这里主要介绍两种方法:其一是通过代谢物质的交换,即代谢作用;其二是通过群感效应(quorum sensing,QS)[17]。
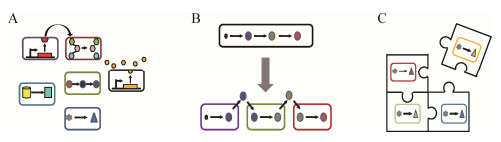
2.1.1 代谢作用以往在合成生物学研究中,研究者通常改变单一种群的代谢通路,以增加细胞代谢物的产量,或是增强单菌种的代谢分解能力,但这一方法往往具有局限性,尤其是对于复杂的代谢网络来说,这将会加重细胞的代谢负担。而在人工合成微生物体系中,将这一代谢工程应用于微生物群体,让不同的微生物种群承担不同的代谢功能,则能够构建微生物种群的相互作用关系,建立代谢网络,实现分工合作[18]。代谢分工合作一方面能够实现在一个微生物体系中综合多个代谢功能,并且减轻单菌种的代谢负担,另一方面代谢功能的模块化更有利于研究人员进行工程改造及操控,以达到理想的目标[19](图 2)。
Mee等[20]通过敲除基因获得14种必需氨基酸的营养缺陷型大肠杆菌,进行两两配对混合培养,发现不同的营养缺陷型之间能够实现代谢互补,并且稀有、代谢成本高的氨基酸能够增强这一作用,在合成相对复杂微生物群落中(代谢互利)交叉喂养作用依旧可行。Lilja等[21]将目光转向了Pseudomonas stutzeri,根据其反硝化途径,通过基因工程获得对硝酸盐和亚硝酸盐还原能力不同的菌株,实现交叉喂养相互作用。这些研究都是基于同基因突变体,即构建亲代菌株的相互作用,这一方法的优点在于同基因突变体之间的遗传差异小,避免出现其他的相互作用,菌株交互更为可控[10]。
另外还能将不同的微生物种或菌株混合形成微生物群落,实现不同基因型之间的相互作用。Zhou等[22]设计组装了Escherichia coli及Saccharomyces cerevisiae联合体,将具有药用价值紫杉烷类的生物合成途径分为两个独立的途径,E. coli产生并分泌紫杉二烯中间体,然后S. cerevisiae完成必要的氧化步骤实现功能化。这一联合体解决了单菌种难以兼容长代谢途径的难题,通过代谢分工使代谢途径各部分得到优化,获得了更高的产量。
2.1.2 群感效应除了通过代谢作用实现理想的群落水平行为外,细菌生态系统中的许多细胞间相互作用都是由多肽、小分子和天然产物等多种分子的分泌、扩散和交换所介导的。这其中最为常见的就是细菌群体间的群感效应。群感效应是一种群体密度依赖性的细胞信号,当群体达到临界密度时会触发行为的变化,其中研究最多的是AHL信号型的群感效应[23-24]。Scott等[25]探索了QS中的不同通信模块,利用AHL信号型QS中的Lux、Las、Rpa系统设计了两个菌株间的同步裂解电路,在达到临界种群密度时每个菌株都被编程进行裂解,他们的这一研究有助于开发微生物群落中更高级的遗传电路。Kim等[26]在E. coli的基因组中整合QS系统的相关调控基因,当E. coli菌群达到浓度阈值时,将会分泌调控蛋白诱导自身生产生物柴油前体物质,实现由QS系统介导生物燃料的全自动生产。群感效应作为一种简单的细菌通信系统,其模块化和工程化的特点使之广泛应用于细菌交流调控[27-28]。
2.2 合成微生物群落的时空组织在自然环境中,群落中的相互作用受到时空的影响,微生物细胞分泌的多肽、抗生素、群体感应信号物等小分子物质扩散差异,以及微生物分布空间位置差异都能够导致群落资源异质性,从而造成局部亚群落之间作用的强弱不同。人工设计合成的微生物体系想要模拟自然微生物群落,同样需要注意共培养体的时空组织,时空组织对微生物间的相互作用将会产生强烈的影响。Jiang等[29]研究硫胺营养缺陷型的籼稻内生菌Serendipita indica与土壤中的Bacillus subtilis之间的相互作用,发现通过让S. indica孢子与B. subtilis在空间上远距离培养,或是在接种S. indica孢子后让其萌发一段时间后再与B. subtilis共培养,二者之间才会发生积极的代谢互补作用,直接混合共培养则会显著抑制S. indica的生长,会出现这种情况,是因为在混合培养过程中B. subtilis会消耗氧气,抑制了S. indica孢子的萌发。
此外,合成微生物群落的空间结构可以通过实验设备实现。目前开发的设备主要有微流控技术,它一方面可以形成细胞间的物理隔离屏障,模拟自然环境中的空间限制,阻止微生物细胞直接接触;另一方面可以使活性物质形成空间梯度,造成物质资源异质性,产生局部作用差异[30]。Jeong等[31]开发了一种微流控静态液滴阵列(static droplet array,SDA),通过化学和物理隔离,分别形成两个细菌种群的密度梯度液滴,使二者在通过QS相互作用时具有不同的种群比例,以此来研究不同种群比例与QS的关系。
另有3D打印技术通过将捕获的细菌定位在交联明胶形成的封闭腔内,构建空间三维结构更为复杂的人工合成微生物群落,并同时能够传递许多具有生物活性的物质[32]。此外,生物膜作为微生物空间分布的一种重要结构,菌群的表面附着作用可以进行有效设计与控制。Nguyen等[14]开发了一种纳米纤维显示平台,他们能够对E. coli的生物膜进行编程,使其粘附在特定的非生物表面和颗粒上。Hong等[28]通过设计生物膜基因电路,在一种新型微流体器件上实现生物膜的转换与移除。
3 合成微生物体系的应用基础在自然生物群落中,数量及种类众多的微生物及其复杂的关系使得研究群落成员的身份、功能、相互作用具有挑战,但通过自下而上人工微生物群落的设计合成,这一局限性能够大大降低。而且,可操作性强的合成微生物体系能够产生一系列的数据集,对数据集进行建模分析及计算机模拟能够提供仅靠实验难以获得的一些独特见解,推动微生物生态的进一步研究,还能够对已知的生态理论进行验证。
3.1 合成微生物体系的生态模型构建自然微生物群落中,其个体的增长、微生物间的相互作用、资源的交换等都能够被参数化,在这些数据的基础上建立适当的数学模型,能够对其进行定量化描述,辅助研究者们对于群落作用机制的理解[33]。但这些数据并不能直接从复杂的自然群落中获得,而是从透明可控的合成微生物体系中采集。图 3介绍了一些基本的计算机建模分析方法。
动态模型主要描述群落随时间的变化,通常采用耦合微分方程对微生物物种丰度、组成随时间的演化进行描述[6]。例如Bush等[38]采用简单的常微分方程模型模拟群落细菌密度、氧气、营养物质随时间的变化,并且得到了与实验观察一致的模型结果。在这个研究中动态模型的建立,主要是根据已知的微生物对营养物质的消耗等生态学理论[38],除此以外,建模还能根据时间序列数据进行。
时间序列数据常用广义的Lotka-Volterra模型来进行描述,该模型主要用于描述两个物种间的交互作用(图 3A),但它本身具有一定的限制性,并不能简单地适用于所有的时间序列分析,有些研究对该模型进行了扩展以克服这些限制[34]。例如Stein等[39]在广义Lotka-Volterra模型中引入微生物对环境扰动的响应项,以描述抗生素扰动下肠道菌群的变化;Momeni等[40]则提出对Lotka-Volterra成对模型进行修正,使其能够更好地描述微生物的交互作用。
3.1.2 代谢模型当微生物通过代谢实现相互作用时,其代谢物质常常满足一定的计量关系式,因而通过构建微生物代谢作用模型,可以描述微生物间的相互作用[35](图 3B)。在代谢作用分析的过程中,通常采用基于约束的重构与分析(constraint-based reconstruction and analysis,COBRA)方法,例如代谢通量平衡分析(flux balance analysis,FBA)重构基因组规模的代谢网络,或是与多种组学技术相结合,从多个角度研究微生物群落中的代谢作用[41-42]。
组学技术并不能展示细胞内的代谢通量,另一个方法13C代谢通量分析(13C metabolic flux analysis,13C-MFA)通过对代谢过程中的葡萄糖进行同位素标定,则能够改善这一局限性,详细描述生物系统内的代谢通量[35]。例如Wolfsberg等[43]采用13C-MFA方法分析研究了生长在琼脂上的E. coli群落代谢,确定了两个不同的E. coli菌群在交叉喂养醋酸盐过程中二者各自的代谢作用。
3.1.3 空间模型空间模型用于描述群落的相互作用随空间的变化,这一变化是由生境的异质性、自然梯度的存在、微生物本身的自组织特性而引起的,主要由偏微分方程(partial deferential equation,PDE)进行模拟[6]。其中所谓的生境异质性即是指生境中存在的物理屏障,这一屏障限制了微生物及其代谢产物的移动与扩散,造成空间位点差异[30]。目前常用的空间模型是反应-扩散方程[36](图 3C),它描述了在微生物相互作用的过程中,由于物质扩散及种群动态导致的每个物种在不同时空下的种群密度[44]。Menon等[36]采用反应-扩散模型描述微生物共享物质浓度的动态变化,发现代谢物的更多共享降低了微生物合作的强度,并通过非平衡相变导致物种灭绝。
现如今有不少研究利用空间模型解析微生物种群中的Pattern formation,以明确这一过程的机制[45]。例如Cao等[46]在研究E. coli工程菌在菌落中形成的核环模式时,利用PDE描述了该过程中的基因通路动力学,其中着重考虑了整个群体中基因表达能力的空间变化,以此揭示了核环模式保持尺度不变性的必要条件。
3.1.4 IBM模型以上3个模型都是基于种群建立的,而IBM模型(individual-based models)是基于个体或主体的模型,它将每一个单独的单元视为交互作用中独立的实体,允许引入个体差异,如生长速率、细胞质量、底物分泌速率等,对种群中每个个体的属性、活动和交互进行建模[37](图 3D)。这一模型通过自下而上的方法,通过对个体行为规则的描述来展现整个群体系统的动态变化,并且允许在系统分析的过程中引入随机性和个别可变性,使得模型描述更为现实具体[47-48]。例如在Estrela等[49]的研究中,采用IBM模型框架对固体表面上的微生物种群增长进行建模,发现两种微生物之间代谢依赖关系的增强推动了二者互惠共生模式的出现,并且促进了种间混合,但在建模的过程中要注意避免构建过于复杂的模型,以免其难以落实于分析中。
在研究合成微生物群落的动态变化时,常常是将多个建模方法结合起来共同描述微生物间的相互作用,并且不同的微生物系统具有不同的特点,需要根据实验的具体情况对生态模型进行优化,以最大程度展现微生物群落的真实动态变化。
3.2 合成微生物体系中的生态学理论反馈微生物生态学的研究需要理论的指导与推动,而已建立的生态理论的普遍性必须通过微生物系统进行检验[50]。具有复杂度低、可操作性强特征的合成微生物体系,成为了科学家们探索一些关键的生态和进化问题的有力工具[51-54]。例如对于自然环境中微生物交叉喂养相互作用维持的机制,Pande等[51]通过设计Acinetobacter baylyi及E. coli营养缺陷型菌株合成群落,研究结构化或非结构化环境中非合作者对于合作者间相互作用的影响,揭示了结构化环境对于相互作用维持的重要性。另一个例子则是关于微生物群落的多样性机制。针对选择过程与中性过程对于理解微生物群落多样性的相对重要性这一问题,Cira等[53]使用可遗传的遗传条码创建了一个合成的微生物群落,并多次稀释且移入一定数量物种后继续培养,在这个过程中持续跟踪群落组成,研究两种机制对于群落组成变化的影响,发现在中性和选择过程之间存在着一种过渡,而这种过渡点取决于外来菌种的比例和适应性的差异。另有Kinnunen等[54]在研究微生物群落的多样性与外来菌种入侵的关系时,通过在亚硝酸盐氧化菌群(nitrite-oxidizing bacteria,NOB)中引入NOB菌种,发现当入侵的微生物与原菌群的系统发育距离较近时,其入侵的成功与否由选择过程控制。
此外,在研究生态作用机制的过程中,常常将生态模型与已知的生态理论相结合,定量化描述微生物群落的生态过程,将实验经验与数值分析整合起来探索微生物间的相互作用。例如在研究微生物群落中的代谢相互作用在代谢物和生物体之间是如何变化时,Zomorrodi等[13]将基因组水平的代谢模型与进化博弈论结合起来,分析E. coli合成群落中相互作用的变化,揭示了生态互作中的细胞内分子机制。Kelsic等[55]则将抗生素的抑制带模型、种群动态模型与微生物的耐药性结合起来,分析在微生物群落中对抗生素具有不同作用的物种共存的机制,展示了抗生素的生成与降解对于自然界微生物多样性的重要性。
然而,复杂的群落系统产生的数据往往是非线性且含有许多噪音,而这会降低数据分析的准确性及计算效率,此时选择合适的模型应用是非常重要的。Wang等[56]对独立单元主体的动力学模型进行了优化,使之近似于独立单元的自动态和耦合动态,以更好地拟合微生物间的非线性作用,减少群落体系内部及外部的噪音污染。Xiao等[57]则开发了一种通过稳态丰度数据推断微生物拓扑网络的方法,研究微生物之间的相互作用网络及交互类型,由此打破了在探索群落生态网络时需要先验动态模型及拟合纵向丰度数据的局限性。他们的研究拓展了微生物群落生态网络研究的方法,有助于加强对微生物群落稳定性和组装规则的理解。
这些通过合成微生物体系建立的理论、方法及模型,同样可以反过来指导微生物生态学的研究工作,在原有的基础上优化人工微生物群落,调控微生物的作用及群落行为,推动人们对于自然微生物群落稳定及进化的深入研究。
4 合成微生物体系的应用关于合成微生物体系的组建原则及理论体系的研究推动了其在实际中的应用,主要包括3个方面:人体健康、工业生产和污染修复(表 1)。
| 应用 Applications |
合成微生物体系组成 Compositions of synthetic microbial systems |
作用 Functions |
参考文献 References |
| 人体健康 Human health |
E. coli及S. cerevisiae联合体 Consortium of E. coli and S. cerevisiae |
合成乙酰化二醇紫杉醇前体 Synthesis of the acetylated diol paclitaxel precursor |
[22] |
| 2株含有不同代谢路径的E. coli细胞 Two E. coli strains with different metabolic pathways |
产姜黄素 Producing curcuminoids |
[58] | |
| 3株含有不同合成模块的E. coli细胞 Three E. coli strains with different synthesis modules |
合成迷迭香酸 Biosynthesis of rosmarinic acid |
[59] | |
| 来自健康个体的粪便微生物群 Fecal microbiota from healthy individuals |
治疗Clostridium difficile感染 Treating Clostridium difficile infection |
[60] | |
| 改造过的Schaedler菌群 Altered schaedler flora |
治疗高氨血症 Treating hyperammonemia |
[61] | |
| 工业生产 Industrial production |
含有不同合成模块的2株E. coli Two E. coli strains with different synthesis modules |
提高cis, cis-muconic acid产量 Increasing the yield of cis, cis-muconic acid |
[62] |
| 四菌复合体系 Consortium of four strains |
直接将纤维素转化为甲烷 Converting cellulose directly to methane |
[63] | |
| Trichoderma reesei及E. coli工程菌 Trichoderma reesei and E. coli engineered strain |
木质纤维素发酵产异丁醇 Lignocellulose fermentation to produce isobutanol |
[64] | |
| E. coli工程菌与S. cerevisiae E. coli engineered strain and S. cerevisiae |
混合糖类发酵产乙醇 Sugar mixture fermentation to produce ethanol |
[65] | |
| 污染修复 Contamination remediation |
工程菌E. coli BL21及野生型菌株LL-1 E. coli BL21 and wild strain LL-1 |
降解农药甲基对硫磷 Degrading methyl parathion |
[66] |
| 含有4种野生型菌的菌群DNC5 DNC5 containing four wild-type strains |
降解农药莠去津 Degrading atrazine |
[67] | |
| Acinetobacter sp. XM-02及Pseudomonas sp. XM-01 Acinetobacter sp. XM-02 and Pseudomonas sp. XM-01 |
降解原油 Degrading crude oil |
[68] | |
| Brevibacillus laterosporus及Galactomyces geotrichum Brevibacillus laterosporus and Galactomyces geotrichum |
降解磺化偶氮染料活性红198 Degrading sulphonated azo dye Reactive Red 198 |
[69] |
合成微生物体系对于提升生物合成药物的生产率具有很大的潜力。例如Zhou等[22]设计组装的E. coli及S. cerevisiae联合体,将具有药用价值的紫杉醇前体的生物合成途径分为两个模块,对这两个独立的代谢功能模块进行了优化,大大缩短了合成时间。姜黄素具有抗氧化、抗癌和抗肿瘤活性,Fang等[58]通过基因改造使得一株E. coli能够利用葡萄糖产对香豆酸,另一株E. coli则能够将对香豆酸转换成最终的产物双二甲基氧基姜黄素,并且这一共培养体系比单培养体系更为有效。对于具有重要营养和药用价值的迷迭香酸,Li等[59]采用模块化生物合成策略,将其合成路径分为咖啡酸、丹参酚酸A及迷迭香酸模块,分别位于3个单独的E. coli细胞中,经过共培养其产量提高了38倍。此外,近年的研究表明生活在人体内的微生物群落对于宿主的生理和健康具有重大的影响,例如肠道微生物群与肥胖、糖尿病和炎症性肠病之间具有密切关系,因而有不少研究者探索重组微生物群以治疗人类疾病。例如van Nood等[60]使用粪便微生物群移植治疗Clostridium difficile感染,即将来自健康个体的粪便细菌移植到C. difficile感染的患者体内,其治疗效果比抗生素更有效。Shen等[61]在小鼠模型上移植重组菌群,发现可以降低肝损伤小鼠的发病率和死亡率,具有治疗高氨血症的潜力。
4.2 工业生产合成生物学在工业产品生产上应用广泛,但存在单一菌种无法兼并完整的合成途径、代谢产品产率低、有毒副产物累积等问题[18]。合成微生物体系则能够通过不同细胞间的代谢分工合作减少单个菌株的代谢负担,并且能够使得各个反应模块都达到最优。Zhang等[62]通过对E. coli的基因电路进行设计及优化,将cis, cis-muconic acid (MA)的合成途径分为两个独立的模块,分布在两个单独的E. coli细胞中,由此解决了代谢中间产物积累及混合糖类底物利用率低的问题,显著提高了cis, cis-MA的产量。除此以外,合成微生物体系也被广泛应用于生物燃料的生产。吴俊妹等[63]获得了可直接将纤维素转化为甲烷的四菌复合体系。而Minty等[64]则设计了TrEc体系,该体系中Trichoderma reesei分泌纤维素酶水解木质纤维素生物质成可溶性糖类,E. coli工程菌则将可溶性糖类代谢成异丁醇,由此实现了木质纤维素向生物燃料的直接转换,并且产量高达理论最大值的62%。Wang等[65]将无法利用葡萄糖的产乙醇E. coli工程菌与S. cerevisiae共培养,提高了这一体系在葡萄糖与木糖混合底物上的发酵产乙醇效率。
4.3 污染修复环境中的污染物常常成分复杂,或是分子量较高的有机污染物,少有单一菌种能够满足完全降解污染物的需求。合成微生物体系在这一方面具有巨大的潜力,它能够将具有不同代谢能力的菌株组装起来,在实现对污染物完全降解的同时,也能提高降解效率[70]。Li等[66]利用改造过的E. coli BL21及分离得到的Ochrobactrum sp. strain LL-1对农药甲基对硫磷(methyl parathion,MP)进行降解,E. coli BL21分泌甲基对硫磷水解酶(methyl parathion hydrolase,MPH)使MP水解产生对硝基苯酚(p-nitrophenol,PNP),菌株LL-1则可利用PNP作为唯一的碳源、氮源和能源。PNP作为一种稳定的有毒中间体,为了避免它的产生,有研究改造单一菌株使其同时具有MPH活性及PNP矿化活性,但这增加了菌株的代谢负担[71],而E. coli BL21及Ochrobaltrum sp. strain LL-1体系能够实现对污染物MP的完全矿化,同时代谢的分工合作避免了对单一菌株造成负担。除了污染降解工程菌的构建,具有不同代谢功能野生菌的组合也能够提高污染物的降解效率及矿化程度。Zhang等[67]分离得到一个稳定的含有4种菌的菌群DNC5,该菌群通过微生物间的协同作用能够有效地降解农药莠去津。另有Chen等[68]将分离得到的Acinetobacter sp. XM-02以及Pseudomonas sp. XM-01混合培养,显著提高了原油中的烷烃降解率,且代谢产物中的重组分相比单一菌种降解产物要少。Kurade等[69]利用Brevibacillus laterosporus及Galactomyces geotrichum联合降解磺化偶氮染料活性红198 (reactive red 198,RR198),发现联合体相比于单一菌种具有更高效的降解能力,能够在18 h内使92%的RR198脱色,而这得益于这一细菌-真菌联合体在共培养过程中能够分泌多种氧化还原酶。
5 展望合成微生物体系作为自下而上建立起来的微生物系统,它在微生物生态学的研究中具有巨大的潜力。近年来在人工合成群落的设计、组装、分析、计算机模拟方面的理论和研究进展,使合成微生物体系广泛应用于微生物生态学的研究中,推动了生态理论框架的形成与已知理论的验证。
但面对复杂庞大的自然微生物群落,想要更进一步、更全面地理解自然群落的生态过程,如何构建规模更大、生态网络更为复杂的合成微生物体系仍然是努力的方向。这其中,如何设计微生物间的相互作用、调控群落空间结构以实现理想的群落系统行为是研究中的关键。譬如对其他的微生物相互作用如竞争、捕食、寄生等的探索,以及微生物通信系统的合理模块划分,还需要更进一步的研究。
其次,目前研究中建立的生态模型是比较粗略的,需要对人工合成微生物群落进行反复的测试与调整,以系统地评估受控环境中的参数,优化生态模型,更好地整合经验方法和模型驱动的分析。另外,合成微生物体系与计算机技术的联合也是研究的热点之一,一方面是实现对合成群落动态变化的更好拟合,另一方面是通过模型预测能够展现实验无法达到的效果及效率。
对新的合成微生物体系的探索和表征将进一步促进研究者对于生态理论的理解,从而指导研究者对于微生物资源应用的探索,推动人工合成微生物群落工程化应用。
| [1] |
Bartley BA, Kim K, Medley JK, et al. Synthetic biology: engineering living systems from biophysical principles[J]. Biophysical Journal, 2017, 112(6): 1050-1058. DOI:10.1016/j.bpj.2017.02.013 |
| [2] |
Song H, Ding MZ, Jia XQ, et al. Synthetic microbial consortia: from systematic analysis to construction and applications[J]. Chemical Society Reviews, 2014, 43(20): 6954-6981. DOI:10.1039/C4CS00114A |
| [3] |
Ren G, Ma AZ, Zhang YF, et al. Electron acceptors for anaerobic oxidation of methane drive microbial community structure and diversity in mud volcanoes[J]. Environmental Microbiology, 2018, 20(7): 2370-2385. DOI:10.1111/1462-2920.14128 |
| [4] |
Fredrickson JK. Ecological communities by design[J]. Science, 2015, 348(6242): 1425-1427. DOI:10.1126/science.aab0946 |
| [5] |
de Roy K, Marzorati M, van Den Abbeele P, et al. Synthetic microbial ecosystems: an exciting tool to understand and apply microbial communities[J]. Environmental Microbiology, 2014, 16(6): 1472-1481. DOI:10.1111/1462-2920.12343 |
| [6] |
Zomorrodi AR, Segrè D. Synthetic ecology of microbes: mathematical models and applications[J]. Journal of Molecular Biology, 2016, 428(5): 837-861. DOI:10.1016/j.jmb.2015.10.019 |
| [7] |
Goers L, Freemont P, Polizzi KM. Co-culture systems and technologies: taking synthetic biology to the next level[J]. Journal of the Royal Society Interface, 2014, 11(96): 20140065. DOI:10.1098/rsif.2014.0065 |
| [8] |
Klitgord N, Segrè D. Ecosystems biology of microbial metabolism[J]. Current Opinion in Biotechnology, 2011, 22(4): 541-546. DOI:10.1016/j.copbio.2011.04.018 |
| [9] |
Stenuit B, Agathos SN. Deciphering microbial community robustness through synthetic ecology and molecular systems synecology[J]. Current Opinion in Biotechnology, 2015, 33: 305-317. DOI:10.1016/j.copbio.2015.03.012 |
| [10] |
Dolinšek J, Goldschmidt F, Johnson DR. Synthetic microbial ecology and the dynamic interplay between microbial genotypes[J]. FEMS Microbiology Reviews, 2016, 40(6): 961-979. DOI:10.1093/femsre/fuw024 |
| [11] |
Kato S, Haruta S, Cui ZJ, et al. Stable coexistence of five bacterial strains as a cellulose-degrading community[J]. Applied and Environmental Microbiology, 2005, 71(11): 7099-7106. DOI:10.1128/AEM.71.11.7099-7106.2005 |
| [12] |
Shou WY, Ram S, Vilar JMG. Synthetic cooperation in engineered yeast populations[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(6): 1877-1882. DOI:10.1073/pnas.0610575104 |
| [13] |
Zomorrodi AR, Segrè D. Genome-driven evolutionary game theory helps understand the rise of metabolic interdependencies in microbial communities[J]. Nature Communications, 2017, 8(1): 1563. DOI:10.1038/s41467-017-01407-5 |
| [14] |
Nguyen PQ, Botyanszki Z, Tay PKR, et al. Programmable biofilm-based materials from engineered curli nanofibres[J]. Nature Communications, 2014, 5: 4945. DOI:10.1038/ncomms5945 |
| [15] |
Shong J, Diaz MRJ, Collins CH. Towards synthetic microbial consortia for bioprocessing[J]. Current Opinion in Biotechnology, 2012, 23(5): 798-802. DOI:10.1016/j.copbio.2012.02.001 |
| [16] |
Faust K, Raes J. Microbial interactions: from networks to models[J]. Nature Reviews Microbiology, 2012, 10(8): 538-550. DOI:10.1038/nrmicro2832 |
| [17] |
Johns NI, Blazejewski T, Gomes ALC, et al. Principles for designing synthetic microbial communities[J]. Current Opinion in Microbiology, 2016, 31: 146-153. DOI:10.1016/j.mib.2016.03.010 |
| [18] |
Tsoi R, Wu FL, Zhang C, et al. Metabolic division of labor in microbial systems[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(10): 2526-2531. DOI:10.1073/pnas.1716888115 |
| [19] |
Tsoi R, Dai ZJ, You LC. Emerging strategies for engineering microbial communities[J]. Biotechnology Advances, 2019, 37(6): 107372. DOI:10.1016/j.biotechadv.2019.03.011 |
| [20] |
Mee MT, Collins JJ, Church GM, et al. Syntrophic exchange in synthetic microbial communities[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(20): E2149-E2156. DOI:10.1073/pnas.1405641111 |
| [21] |
Lilja EE, Johnson DR. Segregating metabolic processes into different microbial cells accelerates the consumption of inhibitory substrates[J]. The ISME Journal, 2016, 10(7): 1568-1578. DOI:10.1038/ismej.2015.243 |
| [22] |
Zhou K, Qiao KJ, Edgar S, et al. Distributing a metabolic pathway among a microbial consortium enhances production of natural products[J]. Nature Biotechnology, 2015, 33(4): 377-383. DOI:10.1038/nbt.3095 |
| [23] |
Abisado RG, Benomar S, Klaus JR, et al. Bacterial quorum sensing and microbial community interactions[J]. mBio, 2018, 9(3): e02331-17. |
| [24] |
Cheng FF, Ma AZ, Zhuang GQ, et al. Exogenous N-acyl-homoserine lactones enhance the expression of flagella of Pseudomonas syringae and activate defence responses in plants[J]. Molecular Plant Pathology, 2018, 19(1): 104-115. DOI:10.1111/mpp.12502 |
| [25] |
Scott SR, Din MO, Bittihn P, et al. A stabilized microbial ecosystem of self-limiting bacteria using synthetic quorum-regulated lysis[J]. Nature Microbiology, 2017, 2(8): 17083. DOI:10.1038/nmicrobiol.2017.83 |
| [26] |
Kim EM, Woo HM, Tian T, et al. Autonomous control of metabolic state by a quorum sensing (QS)-mediated regulator for bisabolene production in engineered E. coli[J]. Metabolic Engineering, 2017, 44: 325-336. DOI:10.1016/j.ymben.2017.11.004 |
| [27] |
Scott SR, Hasty J. Quorum sensing communication modules for microbial consortia[J]. ACS Synthetic Biology, 2016, 5(9): 969-977. DOI:10.1021/acssynbio.5b00286 |
| [28] |
Hong SH, Hegde M, Kim J, et al. Synthetic quorum-sensing circuit to control consortial biofilm formation and dispersal in a microfluidic device[J]. Nature Communications, 2012, 3: 613. DOI:10.1038/ncomms1616 |
| [29] |
Jiang X, Zerfaß C, Feng S, et al. Impact of spatial organization on a novel auxotrophic interaction among soil microbes[J]. The ISME Journal, 2018, 12(6): 1443-1456. DOI:10.1038/s41396-018-0095-z |
| [30] |
Aleklett K, Kiers ET, Ohlsson P, et al. Build your own soil: exploring microfluidics to create microbial habitat structures[J]. The ISME Journal, 2018, 12(2): 312-319. DOI:10.1038/ismej.2017.184 |
| [31] |
Jeong HH, Jin SH, Lee BJ, et al. Microfluidic static droplet array for analyzing microbial communication on a population gradient[J]. Lab on A Chip, 2015, 15(3): 889-899. DOI:10.1039/C4LC01097C |
| [32] |
Connell JL, Ritschdorff ET, Whiteley M, et al. 3D printing of microscopic bacterial communities[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(46): 18380-18385. DOI:10.1073/pnas.1309729110 |
| [33] |
Liu WW, Wu B, Xiang MC, et al. From microbiome to synthetic microbial community[J]. Microbiology China, 2017, 44(4): 881-889. (in Chinese) 刘炜伟, 吴冰, 向梅春, 等. 从微生物组到合成功能菌群[J]. 微生物学通报, 2017, 44(4): 881-889. |
| [34] |
Gonze D, Coyte KZ, Lahti L, et al. Microbial communities as dynamical systems[J]. Current Opinion in Microbiology, 2018, 44: 41-49. DOI:10.1016/j.mib.2018.07.004 |
| [35] |
Gebreselassie NA, Antoniewicz MR. 13C-metabolic flux analysis of co-cultures: a novel approach[J]. Metabolic Engineering, 2015, 31: 132-139. DOI:10.1016/j.ymben.2015.07.005 |
| [36] |
Menon R, Korolev KS. Public good diffusion limits microbial mutualism[J]. Physical Review Letters, 2015, 114(16): 168102. DOI:10.1103/PhysRevLett.114.168102 |
| [37] |
Hellweger FL, Clegg RJ, Clark JR, et al. Advancing microbial sciences by individual-based modelling[J]. Nature Reviews Microbiology, 2016, 14(7): 461-471. DOI:10.1038/nrmicro.2016.62 |
| [38] |
Bush T, Diao M, Allen RJ, et al. Oxic-anoxic regime shifts mediated by feedbacks between biogeochemical processes and microbial community dynamics[J]. Nature Communications, 2017, 8(1): 789. DOI:10.1038/s41467-017-00912-x |
| [39] |
Stein RR, Bucci V, Toussaint NC, et al. Ecological modeling from time-series inference: insight into dynamics and stability of intestinal microbiota[J]. PLoS Computational Biology, 2013, 9(12): e1003388. DOI:10.1371/journal.pcbi.1003388 |
| [40] |
Momeni B, Xie L, Shou WY. Lotka-Volterra pairwise modeling fails to capture diverse pairwise microbial interactions[J]. eLife, 2017, 6: e25051. DOI:10.7554/eLife.25051 |
| [41] |
Magnúsdóttir S, Thiele I. Modeling metabolism of the human gut microbiome[J]. Current Opinion in Biotechnology, 2018, 51: 90-96. DOI:10.1016/j.copbio.2017.12.005 |
| [42] |
Embree M, Liu JK, Al-Bassam MM, et al. Networks of energetic and metabolic interactions define dynamics in microbial communities[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(50): 15450-15455. DOI:10.1073/pnas.1506034112 |
| [43] |
Wolfsberg E, Long CP, Antoniewicz MR. Metabolism in dense microbial colonies: 13C metabolic flux analysis of E. coli grown on agar identifies two distinct cell populations with acetate cross-feeding[J]. Metabolic Engineering, 2018, 49: 242-247. DOI:10.1016/j.ymben.2018.08.013 |
| [44] |
Datta MS, Korolev KS, Cvijovic I, et al. Range expansion promotes cooperation in an experimental microbial metapopulation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(18): 7354-7359. DOI:10.1073/pnas.1217517110 |
| [45] |
Scholes NS, Isalan M. A three-step framework for programming pattern formation[J]. Current Opinion in Chemical Biology, 2017, 40: 1-7. DOI:10.1016/j.cbpa.2017.04.008 |
| [46] |
Cao YXL, Ryser MD, Payne S, et al. Collective space-sensing coordinates pattern scaling in engineered bacteria[J]. Cell, 2016, 165(3): 620-630. DOI:10.1016/j.cell.2016.03.006 |
| [47] |
Hellweger FL, Bucci V. A bunch of tiny individuals — individual-based modeling for microbes[J]. Ecological Modelling, 2009, 220(1): 8-22. DOI:10.1016/j.ecolmodel.2008.09.004 |
| [48] |
Ferrer J, Prats C, López D. Individual-based modelling: an essential tool for microbiology[J]. Journal of Biological Physics, 2008, 34(1/2): 19-37. |
| [49] |
Estrela S, Brown SP. Metabolic and demographic feedbacks shape the emergent spatial structure and function of microbial communities[J]. PLoS Computational Biology, 2013, 9(12): e1003398. DOI:10.1371/journal.pcbi.1003398 |
| [50] |
Prosser JI, Bohannan BJM, Curtis TP, et al. The role of ecological theory in microbial ecology[J]. Nature Reviews Microbiology, 2007, 5(5): 384-392. DOI:10.1038/nrmicro1643 |
| [51] |
Pande S, Kaftan F, Lang S, et al. Privatization of cooperative benefits stabilizes mutualistic cross-feeding interactions in spatially structured environments[J]. The ISME Journal, 2016, 10(6): 1413-1423. DOI:10.1038/ismej.2015.212 |
| [52] |
Burghardt LT, Epstein B, Guhlin J, et al. Select and resequence reveals relative fitness of bacteria in symbiotic and free-living environments[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(10): 2425-2430. DOI:10.1073/pnas.1714246115 |
| [53] |
Cira NJ, Pearce MT, Quake SR. Neutral and selective dynamics in a synthetic microbial community[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(42): E9842-E9848. DOI:10.1073/pnas.1808118115 |
| [54] |
Kinnunen M, Dechesne A, Albrechtsen HJ, et al. Stochastic processes govern invasion success in microbial communities when the invader is phylogenetically close to resident bacteria[J]. The ISME Journal, 2018, 12(11): 2748-2756. DOI:10.1038/s41396-018-0202-1 |
| [55] |
Kelsic ED, Zhao J, Vetsigian K, et al. Counteraction of antibiotic production and degradation stabilizes microbial communities[J]. Nature, 2015, 521(7553): 516-519. DOI:10.1038/nature14485 |
| [56] |
Wang S, Herzog ED, Kiss IZ, et al. Inferring dynamic topology for decoding spatiotemporal structures in complex heterogeneous networks[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(37): 9300-9305. DOI:10.1073/pnas.1721286115 |
| [57] |
Xiao YD, Angulo MT, Friedman J, et al. Mapping the ecological networks of microbial communities[J]. Nature Communications, 2017, 8: 2042. DOI:10.1038/s41467-017-02090-2 |
| [58] |
Fang Z, Jones JA, Zhou JW, et al. Engineering Escherichia coli co-cultures for production of curcuminoids from glucose[J]. Biotechnology Journal, 2018, 13(5): 1700576. DOI:10.1002/biot.201700576 |
| [59] |
Li ZH, Wang XN, Zhang HR. Balancing the non-linear rosmarinic acid biosynthetic pathway by modular co-culture engineering[J]. Metabolic Engineering, 2019, 54: 1-11. DOI:10.1016/j.ymben.2019.03.002 |
| [60] |
van Nood E, Vrieze A, Nieuwdorp M, et al. Duodenal infusion of donor feces for recurrent Clostridium difficile[J]. The New England Journal of Medicine, 2013, 368(5): 407-415. DOI:10.1056/NEJMoa1205037 |
| [61] |
Shen TCD, Albenberg L, Bittinger K, et al. Engineering the gut microbiota to treat hyperammonemia[J]. Journal of Clinical Investigation, 2015, 125(7): 2841-2850. DOI:10.1172/JCI79214 |
| [62] |
Zhang HR, Pereira B, Li ZJ, et al. Engineering Escherichia coli coculture systems for the production of biochemical products[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(27): 8266-8271. DOI:10.1073/pnas.1506781112 |
| [63] |
Wu JM, Ma AZ, Cui MM, et al. Bioconversion of cellulose to methane by a consortium consisting of four microbial strains[J]. Environmental Science, 2014, 35(1): 327-333. (in Chinese) 吴俊妹, 马安周, 崔萌萌, 等. 降解纤维素产甲烷的四菌复合系[J]. 环境科学, 2014, 35(1): 327-333. |
| [64] |
Minty JJ, Singer ME, Scholz SA, et al. Design and characterization of synthetic fungal-bacterial consortia for direct production of isobutanol from cellulosic biomass[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(36): 14592-14597. DOI:10.1073/pnas.1218447110 |
| [65] |
Wang L, York SW, Ingram LO, et al. Simultaneous fermentation of biomass-derived sugars to ethanol by a co-culture of an engineered Escherichia coli and Saccharomyces cerevisiae[J]. Bioresource Technology, 2019, 273: 269-276. DOI:10.1016/j.biortech.2018.11.016 |
| [66] |
Li L, Yang C, Lan WS, et al. Removal of methyl parathion from artificial off-gas using a bioreactor containing a constructed microbial consortium[J]. Environmental Science & Technology, 2008, 42(6): 2136-2141. |
| [67] |
Zhang Y, Cao B, Jiang Z, et al. Metabolic ability and individual characteristics of an atrazine-degrading consortium DNC5[J]. Journal of Hazardous Materials, 2012, 237-238: 376-381. DOI:10.1016/j.jhazmat.2012.08.047 |
| [68] |
Chen Y, Li C, Zhou ZX, et al. Enhanced biodegradation of alkane hydrocarbons and crude oil by mixed strains and bacterial community analysis[J]. Applied Biochemistry and Biotechnology, 2014, 172(7): 3433-3447. DOI:10.1007/s12010-014-0777-6 |
| [69] |
Kurade MB, Waghmode TR, Jadhav MU, et al. Bacterial-yeast consortium as an effective biocatalyst for biodegradation of sulphonated azo dye Reactive Red 198[J]. RSC Advances, 2015, 5(29): 23046-23056. DOI:10.1039/C4RA15834B |
| [70] |
Tang HZ, Wang WW, Zhang LG, et al. Application of synthetic biology in environmental remediation[J]. Chinese Journal of Biotechnology, 2017, 33(3): 506-515. (in Chinese) 唐鸿志, 王伟伟, 张莉鸽, 等. 合成生物学在环境修复中的应用[J]. 生物工程学报, 2017, 33(3): 506-515. |
| [71] |
Yang C, Cai N, Dong M, et al. Surface display of MPH on Pseudomonas putida JS444 using ice nucleation protein and its application in detoxification of organophosphates[J]. Biotechnology and Bioengineering, 2008, 99(1): 30-37. |
 2020, Vol. 47
2020, Vol. 47