扩展功能
文章信息
- 王勇超, 刘志华, 安柳迪, 张丛兴, 王金杰
- WANG Yong-Chao, LIU Zhi-Hua, AN Liu-Di, ZHANG Cong-Xing, WANG Jin-Jie
- 绿木霉Gv29-8丝氨酸蛋白酶S8/S53超家族基因特性及功能
- Characterization of S8/S53 serine protease superfamily gene of Trichoderma virens Gv29-8
- 微生物学通报, 2020, 47(2): 532-541
- Microbiology China, 2020, 47(2): 532-541
- DOI: 10.13344/j.microbiol.china.190376
-
文章历史
- 收稿日期: 2019-04-28
- 接受日期: 2019-11-20
- 网络首发日期: 2019-11-29
木霉菌(Trichoderma spp.)广泛存在于土壤、植物根围、叶围、种子和球茎等生态环境中,是重要的植物病害生防菌[1-2]。木霉菌产生的丝氨酸蛋白酶(serine protease)具有抗真菌和重寄生作用,以及诱导寄主植物防卫反应等作用[3-4]。Geremia等在诸多的研究后证明了哈茨木霉(T. harzianum)的丝氨酸蛋白酶Prb1与重寄生效应关系密切[5]。Pozo等发现丝氨酸蛋白酶基因tvsp1在绿木霉(T. virens)中的过表达显著提高了棉花幼苗抗立枯丝核菌(Rhizoctonia solani)的能力[3]。
丝氨酸蛋白酶是许多真菌侵染线虫的一种关键酶,作为一种毒力因子,在降解线虫体壁的过程中发挥着非常重要的作用[6]。近年来,人们已发现多种木霉对根结线虫具有一定的抑制作用[7-8]。Szabó等研究表明哈茨木霉的几丁质酶、丝氨酸蛋白酶和天冬氨酸蛋白酶等基因协同表达,在对线虫卵寄生过程中起重要作用[9]。来自绿木霉(T. virens) G1-3的培养滤液可以抑制南方根结线虫(Meloidogyne incognita)卵的孵化和二龄幼虫(J2s)的活动[10]。土壤中最普遍的一种捕食性真菌少孢节丛孢菌(Arthrobotrys oligospora)也可以分泌丝氨酸蛋白酶,从而发挥杀线虫效应。Zhao等扩增得到少孢节丛孢菌丝氨酸蛋白酶P186的基因序列,在毕赤酵母中成功表达,纯化后的蛋白酶reP186具有杀线虫作用[11]。这些结果表明,丝氨酸蛋白酶在真菌生物防治过程中起到重要的作用。
鉴于此,本文利用生物信息学方法,对绿木霉Gv29-8菌株丝氨酸蛋白酶S8/S53超家族基因结构、蛋白酶理化性质、进化关系和结构等方面进行预测分析,旨在为研究该蛋白酶的结构功能、外源表达以及为绿木霉更有效地防治植物病害提供理论依据。
1 材料与方法 1.1 丝氨酸蛋白酶基因的鉴定本实验室从中国农业微生物菌种保藏管理中心(ACCC)购买一株绿木霉ACCC 32490 (T. virens),其生防效果较好,从中克隆了一个丝氨酸蛋白酶基因(GenBank登录号为MN399377),进一步对同种已知基因组信息的绿木霉Gv29-8丝氨酸蛋白酶进行研究,将该基因的cDNA序列与JGI数据库中绿木霉Gv29-8基因组信息(https://genome.jgi.doe.gov/TriviGv29_8_2/TriviGv29_8_2.home.html)进行BLAST比对,得到23个丝氨酸蛋白酶基因的DNA和cDNA序列,并在少孢节丛孢菌ATCC 24927基因组中(https:// genome.jgi.doe.gov/Artol1/Artol1.home.html)搜索到4个丝氨酸蛋白酶基因,统计27个基因在染色体上的位置、外显子和内含子数量等。
1.2 丝氨酸蛋白酶保守结构域及家族预测利用NCBI上的BLASTp (https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行保守结构域及蛋白家族的预测,找出丝氨酸蛋白酶家族的保守结构域,并记录其氨基酸序列。利用ExPASy PROSITE (https://prosite.expasy.org/)分析丝氨酸蛋白酶的功能区和活性位点。
1.3 丝氨酸蛋白酶基本性质预测利用在线工具ExPASy Protparm (https://web.expasy.org/protparam/)分析蛋白的分子量、等电点、不稳定系数、总平均亲水性等理化性质。利用SignalP4.1 (http://www.cbs.dtu.dk/services/SignalP/)预测蛋白的信号肽。利用TMHMM在线软件(http://www.cbs.dtu.dk/services/TMHMM/)预测蛋白的跨膜结构域。利用CBS Prediction serves (http://www.cbs.dtu.dk/services/TargetP/)进行亚细胞定位预测。利用NetNGlyc 1.0 Server (http://www.cbs.dtu.dk/services/NetNGlyc/)预测蛋白的N-糖基化位点。
1.4 丝氨酸蛋白酶功能区多序列比对及进化分析利用BioEdit软件对27个丝氨酸蛋白酶的氨基酸序列进行多序列比对,对其功能区进行分析,从而推测它们结构和功能上的联系。利用MEGA 7.0软件的N-J法(neighbor-joining method)构建系统进化树,Bootstrap重复次数为1 000次,对蛋白酶的进化关系进行分析。利用DNAMAN软件进行多序列相似性分析。
1.5 同源建模和三维结构分析通过生物在线软件SOPMA和SWISS-MODEL (http://swissmodel.expasy.org/)对丝氨酸蛋白酶的二级、三级结构进行预测。
2 结果与分析 2.1 丝氨酸蛋白酶保守结构域及家族预测经过BLASTp比对分析,27个丝氨酸蛋白酶氨基酸序列均具有丝氨酸蛋白酶家族的保守结构域,属于丝氨酸蛋白酶家族S8/S53超家族。保守区预测显示,27个丝氨酸蛋白酶分为S53、S8_5、S8_7、S8_Kp43_protease、S8_PCSK9_ProteinaseK_ like、S8_Subtilisin_subset这6个亚家族,不同的亚家族保守区长度差别较大,同一亚家族的保守区长度相近。其中少孢节丛孢菌的4个丝氨酸蛋白酶与绿木霉的6个蛋白酶同属于S8_PCSK9_ProteinaseK_like亚家族,且保守区长度相近。
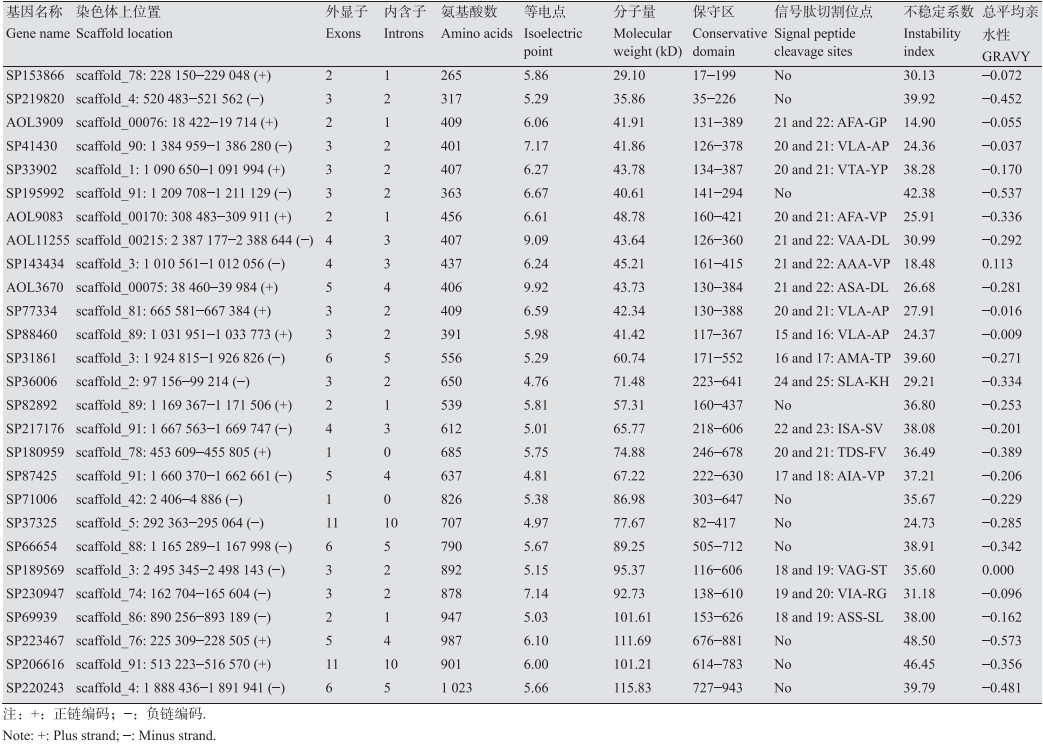
2.2 丝氨酸蛋白酶基因的结构和理化性质预测27个丝氨酸蛋白酶基因的内含子数量差异显著(图 1),表明了生物体内基因的多样性。这些基因编码蛋白的氨基酸数量差别较大,由265-1 023个氨基酸组成,蛋白分子量介于29.10-111.69 kD (表 1)。它们的理论等电点大小不等,除SP41430、SP230947外,其余蛋白均为酸性蛋白。不稳定系数分析表明,除SP195992、SP206616、SP223467为不稳定蛋白,其余蛋白均为稳定蛋白。总平均亲水性预测显示,除SP143434和SP189569外,其余蛋白均为亲水性蛋白,总平均亲水性为-0.573。N-糖基化位点预测发现,27个蛋白均有N-糖基化位点。亚细胞定位显示有17个蛋白为分泌蛋白,它们均有N-端信号肽,主要分布在S8_PCSK9_ProteinaseK_ like、S53和S8_5亚家族。跨膜区预测显示,只有SP41430和SP143434蛋白具有跨膜区,其余蛋白均位于膜外。

|
| 图 1 绿木霉和少孢节丛孢菌丝氨酸蛋白酶基因外显子及内含子分析 Figure 1 Exons and introns analysis of serine proteases genes in T. virens and A. oligospora 注:方框和直线分别代表外显子和内含子,数字表示基因的长度(bp). SP:绿木霉丝氨酸蛋白酶基因;AOL:少孢节丛孢菌丝氨酸蛋白酶基因. Note: The boxes and lines represent exonic and intronic regions respectively. Numbers indicate the length of genes (bp). SP: Serine protease gene of T. virens; AOL: Serine protease gene of A. oligospora. |
|
|
多序列比对结果显示(图 2),这些丝氨酸蛋白酶的保守性低,但是在催化残基(Glu/Asp/His/Ser)及周围的序列都很保守,说明其在丝氨酸蛋白酶功能行使上的重要性,对酶的催化活性是必需的。其中,SP31861、SP217176、SP180959、SP36006和SP87425这5个蛋白含有S53家族的Glu-Asp-Ser催化三联体结构,其余蛋白除SP153866外,均含有S8家族的Asp-His-Ser催化三联体结构[12],这与之前的亚家族预测一致。

|
| 图 2 27个丝氨酸蛋白酶催化基序多序列比对 Figure 2 Multiple sequence alignment of catalytic motif of 27 serine proteases 注:催化残基用灰色底纹显示. SP:绿木霉丝氨酸蛋白酶;AOL:少孢节丛孢菌丝氨酸蛋白酶. Note: The catalytic residues are shown in gray shade. SP: Serine protease of T. virens; AOL: Serine protease of A. oligospora. |
|
|
使用MEGA 7.0软件对27个丝氨酸蛋白酶的氨基酸序列进行聚类分析,并建立系统进化树(图 3)。结果表明,进化树主要分为6支,同一亚家族的蛋白酶聚在一起。来自少孢节丛孢菌的4个蛋白酶与绿木霉的6个蛋白酶较有规律地排列在进化树上并形成第一个分支,表明其亲缘关系非常近。S8_PCSK9_ProteinaseK_like和S53亚家族的蛋白酶位于系统进化树的后端,处于较为进化的地位,其中绿木霉S8_PCSK9_ProteinaseK_like亚家族的蛋白酶可能具有与少孢节丛孢菌的4个蛋白酶功能相似的杀线虫作用,推测杀线虫作用可能是后来随蛋白酶进化产生的,而S53亚家族的蛋白酶组氨酸活性位点缺失,出现谷氨酸活性位点,可能导致其发生进化。相似性比较发现这些蛋白酶的相似性相差较大,但同一亚家族的蛋白酶相似性较高(表 2)。以上结果表明丝氨酸蛋白酶存在种内差异性,可能产生不同的生物学特性,并且在同一亚家族内具有一定的保守性。

|
| 图 3 MEGA 7.0构建27个丝氨酸蛋白酶的系统进化树 Figure 3 Phylogenetic tree of 27 serine proteases generated by MEGA 7.0 |
|
|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | 16 | 17 | 18 | 19 | 20 | 21 | 22 | 23 | 24 | 25 | 26 | 27 | |
| 1 | 100 | ||||||||||||||||||||||||||
| 2 | 78.2 | 100 | |||||||||||||||||||||||||
| 3 | 42.4 | 41.6 | 100 | ||||||||||||||||||||||||
| 4 | 38.7 | 40.4 | 35.5 | 100 | |||||||||||||||||||||||
| 5 | 34.3 | 35.6 | 31.5 | 39.9 | 100 | ||||||||||||||||||||||
| 6 | 39.0 | 38.8 | 36.5 | 41.1 | 32.0 | 100 | |||||||||||||||||||||
| 7 | 37.0 | 38.9 | 34.6 | 42.0 | 39.9 | 42.9 | 100 | ||||||||||||||||||||
| 8 | 36.4 | 36.8 | 33.7 | 41.1 | 35.6 | 40.7 | 55.9 | 100 | |||||||||||||||||||
| 9 | 38.4 | 36.8 | 33.8 | 36.5 | 33.3 | 38.4 | 46.2 | 43.8 | 100 | ||||||||||||||||||
| 10 | 39.0 | 39.6 | 32.8 | 38.6 | 34.9 | 39.8 | 45.6 | 44.4 | 61.8 | 100 | |||||||||||||||||
| 11 | 10.7 | 12.2 | 11.5 | 8.9 | 10.9 | 9.2 | 10.0 | 8.3 | 10.9 | 9.3 | 100 | ||||||||||||||||
| 12 | 10.6 | 10.9 | 9.2 | 9.1 | 8.8 | 10.0 | 11.0 | 9.5 | 11.0 | 9.8 | 21.2 | 100 | |||||||||||||||
| 13 | 9.2 | 9.0 | 9.1 | 10.5 | 8.5 | 9.4 | 10.0 | 8.1 | 9.3 | 11.6 | 9.5 | 9.9 | 100 | ||||||||||||||
| 14 | 7.5 | 8.7 | 9.6 | 10.0 | 9.4 | 9.6 | 11.7 | 11.5 | 11.5 | 12.1 | 9.0 | 8.9 | 25.1 | 100 | |||||||||||||
| 15 | 10.9 | 10.4 | 9.3 | 11.0 | 9.3 | 10.1 | 10.3 | 11.4 | 8.8 | 11.1 | 8.9 | 15.3 | 13.4 | 14.3 | 100 | ||||||||||||
| 16 | 15.2 | 16.6 | 15.0 | 12.3 | 13.0 | 14.9 | 16.1 | 17.5 | 15.3 | 14.7 | 7.7 | 8.0 | 10.1 | 10.0 | 7.5 | 100 | |||||||||||
| 17 | 14.4 | 15.4 | 11.1 | 14.4 | 10.2 | 13.5 | 12.5 | 14.1 | 14.2 | 13.0 | 8.6 | 6.6 | 9.6 | 8.7 | 7.5 | 57.8 | 100 | ||||||||||
| 18 | 14.7 | 14.3 | 11.2 | 13.7 | 12.5 | 12.1 | 12.6 | 11.7 | 11.7 | 11.7 | 8.2 | 7.5 | 8.2 | 6.0 | 8.1 | 19.7 | 15.6 | 100 | |||||||||
| 19 | 14.0 | 13.6 | 10.0 | 12.7 | 12.2 | 10.5 | 12.7 | 13.0 | 11.8 | 12.8 | 7.0 | 7.8 | 7.8 | 7.8 | 8.9 | 21.3 | 15.3 | 11.9 | 100 | ||||||||
| 20 | 12.8 | 12.2 | 13.4 | 14.2 | 10.8 | 11.6 | 15.0 | 13.7 | 13.7 | 12.5 | 8.1 | 7.2 | 8.1 | 6.1 | 6.6 | 20.5 | 13.4 | 9.7 | 11.6 | 100 | |||||||
| 21 | 14.3 | 14.3 | 15.2 | 15.6 | 12.7 | 12.9 | 13.4 | 11.8 | 13.5 | 12.2 | 8.4 | 6.2 | 9.7 | 7.9 | 8.8 | 15.7 | 13.0 | 15.4 | 13.6 | 31.5 | 100 | ||||||
| 22 | 11.2 | 10.6 | 13.7 | 14.8 | 12.0 | 11.1 | 12.8 | 12.5 | 12.7 | 11.7 | 7.2 | 5.8 | 7.1 | 6.9 | 8.4 | 17.6 | 11.4 | 9.3 | 11.5 | 18.0 | 53.3 | 100 | |||||
| 23 | 9.1 | 9.4 | 7.1 | 8.7 | 7.8 | 9.8 | 10.8 | 8.3 | 9.1 | 9.6 | 6.3 | 6.7 | 7.3 | 5.6 | 6.3 | 8.7 | 8.8 | 7.0 | 7.2 | 6.4 | 5.8 | 6.3 | 100 | ||||
| 24 | 9.6 | 9.2 | 8.0 | 10.6 | 10.4 | 10.0 | 7.9 | 8.2 | 8.9 | 8.4 | 6.5 | 5.5 | 5.2 | 7.3 | 6.0 | 7.5 | 6.6 | 7.4 | 6.1 | 6.4 | 5.4 | 5.6 | 8.2 | 100 | |||
| 25 | 6.8 | 7.8 | 9.3 | 9.0 | 6.6 | 6.9 | 9.1 | 7.8 | 8.8 | 7.9 | 5.1 | 8.1 | 7.2 | 7.3 | 9.3 | 4.2 | 5.8 | 5.9 | 5.2 | 4.9 | 7.6 | 6.0 | 6.6 | 6.0 | 100 | ||
| 26 | 7.5 | 7.6 | 7.4 | 10.0 | 8.1 | 7.1 | 6.8 | 8.0 | 7.6 | 8.1 | 6.3 | 8.2 | 7.5 | 4.8 | 8.3 | 6.4 | 8.8 | 7.1 | 5.7 | 5.3 | 4.7 | 7.2 | 7.5 | 6.3 | 8.2 | 100 | |
| 27 | 11.3 | 10.5 | 7.1 | 6.4 | 7.5 | 6.6 | 5.9 | 7.1 | 6.9 | 7.1 | 8.6 | 6.7 | 6.5 | 6.5 | 8.4 | 5.7 | 6.3 | 7.5 | 6.2 | 6.1 | 7.6 | 5.9 | 6.7 | 5.9 | 11.7 | 11.8 | 100 |
| Note: 1: SP41430; 2: SP77334; 3: SP33902; 4: SP88460; 5: SP82892; 6: SP143434; 7: AOL3909; 8: AOL9083; 9: AOL11255; 10: AOL3670; 11: SP31861; 12: SP217176; 13: SP180959; 14: SP36006; 15: SP87425; 16: SP153866; 17: SP195992; 18: SP223467; 19: SP220243; 20: SP206616; 21: SP219820; 22: SP66654; 23: SP71006; 24: SP37325; 25: SP230947; 26: SP189569; 27: SP69939. | |||||||||||||||||||||||||||
利用SOPMA在线程序预测结果表明,27个丝氨酸蛋白酶的二级结构以α-螺旋(alpha helix)、无规则卷曲(random coil)、延伸链(extended strand)为主,超过85%,而β-转角(beta turn)少于15%。从每个亚家族选择一种蛋白酶,利用SWISS-MODEL获得蛋白质的三级结构(图 4),A、B、C、D、E、F分别是对SP69939、SP71006、SP66654、SP217176、SP77334、AOL3909的结构预测。由于S8_7亚家族丝氨酸蛋白酶未具有较为适合的同源建模模板或模板相似性较低,因此未能构建模拟出三级结构的模型。预测结果显示,这些蛋白质三级结构的相似度较低,但它们每个结构域均含有至少一个折叠片,构成一个折叠桶,活性部位位于两个折叠桶之间,为Asp-His-Ser或Glu-Asp-Ser催化三联体。

|
| 图 4 丝氨酸蛋白酶三级结构模型 Figure 4 The model on tertiary structure of serine proteases 注:圆球代表Ca2+结合位点. A:SP69939;B:SP71006;C:SP66654;D:SP217176;E:SP77334;F:AOL3909. Note: The Ca2+ binding sites are shown as spheres. A: SP69939; B: SP71006; C: SP66654; D: SP217176; E: SP77334; F: AOL3909. |
|
|
SP77334和AOL3909的三维结构高度相似,两个Ca2+结合位点和催化三联体的位置基本相同,说明它们很可能具有相似的功能,这与之前的分析结果一致,推测绿木霉S8_PCSK9_ProteinaseK_like亚家族的蛋白酶和少孢节丛孢菌的丝氨酸蛋白酶功能相似,均对线虫有抑制作用。
3 讨论与结论丝氨酸蛋白酶家族成员具有多种多样的生理功能,在生物机体中发挥着重要的生理作用,具有广泛的研究和应用价值[13]。本研究从绿木霉Gv29-8菌株的基因组信息中得到了27个丝氨酸蛋白酶基因的信息,它们的长度相差较大,内含子数量差异显著。这些基因编码的丝氨酸蛋白酶分为6个亚家族,同一亚家族的蛋白酶保守区长度相近,理化性质相似,说明同一亚家族的蛋白酶具有一定的保守性。
丝氨酸蛋白酶家族氨基酸序列差异较大,某些蛋白质序列的相似度甚至低于20%,但该蛋白质的活性结构域却极其保守,都有一个类似于口袋状的核心催化结构域Asp-His-Ser[14]。通过进一步对27个丝氨酸蛋白酶的结构研究发现,虽然它们的相似度较低,但在催化残基(Asp/His/Ser或Glu/Asp/Ser)及周围的序列都很保守,同一亚家族的蛋白酶保守性更高。正是由于丝氨酸蛋白酶在结构上的微小变化,导致其在功能上的进化[13]。在少孢节丛孢菌的丝氨酸蛋白酶中也有相似的高度保守片段,与绿木霉S8_PCSK9_ProteinaseK_like亚家族的蛋白酶结构相似,这说明真菌丝氨酸蛋白酶基因在物种进化上具有很强的保守性,并且暗示着它们可能在某类重要途径中具有相似的功能。少孢节丛孢菌的这4个丝氨酸蛋白酶均具有杀线虫作用,而且已报道的体壁降解酶主要是S8_PCSK9_ ProteinaseK_like和S8_Subtilisin_subset亚家族中的成员,是真菌重要的毒力因子[11, 15-19]。系统进化树将同一亚家族的蛋白酶聚在一起,亲缘关系较近,并且相似性分析显示,同一亚家族的蛋白酶相似性更高,表明丝氨酸蛋白酶可能由于催化基序的氨基酸残基发生变化,导致不同亚家族的蛋白酶具有独特的功能。已有研究表明,寄生真菌能够通过表达其他蛋白酶而补偿由于寄主产生丝氨酸蛋白酶抑制剂而损失的蛋白酶活力[20-21],一些蛋白酶的相似性不高,但都具有丝氨酸蛋白酶结构域和保守的催化基序,表明它们的存在可能是为了补偿侵染性蛋白酶的酶活力。同源建模分析显示,不同亚家族的丝氨酸蛋白酶在三维结构上差别较大,但都具有催化三联体结构和β折叠桶,它们结构的差异可能是这些酶具有独特水解作用的原因。丝氨酸蛋白酶的活性及稳定性主要和Ca2+结合位点有关,SP69939没有预测到Ca2+结合位点,SP77334和AOL3909具有2个Ca2+结合位点,SP71006、SP66654和SP217176具有一个Ca2+结合位点,距酶活性中心有一定距离,有助于稳定构象[22]。
丝氨酸蛋白酶广泛存在于捕食线虫真菌中,其杀线虫作用已被广泛研究,这些蛋白酶大多为生化特性相似、序列相似的底物酶,是对线虫体识别作用中重要的酶类之一,其中类枯草杆菌(subtilisin-like)丝氨酸蛋白酶在捕食线虫真菌对线虫的致病性进化过程中可能是至关重要的[18, 23]。越来越多的研究证明,木霉丝氨酸蛋白酶S8/S53超家族的基因在木霉抗真菌和杀线虫过程中起着重要的作用[3, 10, 24-28],而且它们大部分属于S8_PCSK9_ ProteinaseK_like亚家族。在该研究中,绿木霉有6个丝氨酸蛋白酶和少孢节丛孢菌的4个丝氨酸蛋白酶同属于S8_PCSK9_ProteinaseK_like亚家族,它们的相似性较高,多序列比对显示它们的催化基序十分保守,系统进化树将它们归为一支,并且三级结构非常相似。少孢节丛孢菌的4个丝氨酸蛋白酶均为已报道的体壁降解丝氨酸蛋白酶,它们都参与到侵染、降解线虫体壁的过程,可能发挥类似的作用[11, 15, 18, 21, 29],绿木霉的SP77334蛋白酶为已报道的Tvsp1丝氨酸蛋白酶[3]。综合以上结果,我们推测绿木霉S8_PCSK9_ProteinaseK_like亚家族的蛋白酶可能具有降解细胞壁或体壁,杀菌或杀线虫的作用。
目前对另外几个亚家族的丝氨酸蛋白酶功能报道较少,也无法预测其具体功能,还需要通过基因克隆及表达分析来验证。Szabó等发现哈茨木霉S53亚家族的P5216蛋白酶与其他蛋白酶共同表达,可能在线虫卵寄生过程中起关键作用[9]。虽然这个家族和S8家族属于同一个超家族,但是两个家族之间的催化机制是非常不同的[30]。来自芽孢杆菌(Bacillus sp.) KSM-KP43菌株的丝氨酸蛋白酶KP-43属于S8_Kp43_protease亚家族,由于其具有水解活性,可用作洗涤剂酶[31]。但经过与绿木霉丝氨酸蛋白酶基因比对,这两个蛋白与该亚家族蛋白酶相似性较低,无法判断绿木霉丝氨酸蛋白酶是否具有相似的功能。
来自绿木霉的23个基因编码的蛋白质均具有丝氨酸蛋白酶的催化残基,它们可能作为一种重要的毒力因子在抑制植物病原真菌和降解线虫体壁的过程中发挥着重要的作用,但其结构的不同导致其与底物结合部位的差异决定了各自对底物的专一性。了解绿木霉丝氨酸蛋白酶S8/S53超家族的生物学信息后,将为进一步研究丝氨酸蛋白酶的生物学功能和开发高效、稳定的生防制剂奠定理论基础,对病原物的生物防控也具有重要的理论和实践意义。
| [1] |
Schuster A, Schmoll M. Biology and biotechnology of Trichoderma[J]. Applied Microbiology and Biotechnology, 2010, 87(3): 787-799. DOI:10.1007/s00253-010-2632-1 |
| [2] |
Qualhato TF, Lopes FAC, Steindorff AS, et al. Mycoparasitism studies of Trichoderma species against three phytopathogenic fungi: evaluation of antagonism and hydrolytic enzyme production[J]. Biotechnology Letters, 2013, 35(9): 1461-1468. DOI:10.1007/s10529-013-1225-3 |
| [3] |
Pozo MJ, Baek JM, García JM, et al. Functional analysis of tvsp1, a serine protease-encoding gene in the biocontrol agent Trichoderma virens[J]. Fungal Genetics and Biology, 2004, 41(3): 336-348. DOI:10.1016/j.fgb.2003.11.002 |
| [4] |
Ferre FS, Santamarina MP. Efficacy of Trichoderma harzianum in suppression of Fusarium culmorum[J]. Annals of Microbiology, 2010, 60(2): 335-340. DOI:10.1007/s13213-010-0047-y |
| [5] |
Geremia RA, Goldman GH, Jacobs D, et al. Molecular characterization of the proteinase-encoding gene, prb1, related to mycoparasitism by Trichoderma harzianum[J]. Molecular Microbiology, 1993, 8(3): 603-613. DOI:10.1111/j.1365-2958.1993.tb01604.x |
| [6] |
Tang LY, Yang L, Xing XJ, et al. Advances in pathogenicity-related serine proteases in nematophagous fungi[J]. Microbiology China, 2017, 44(4): 911-919. (in Chinese) 汤丽燕, 杨乐, 邢新婧, 等. 食线虫真菌致病相关丝氨酸蛋白酶的研究进展[J]. 微生物学通报, 2017, 44(4): 911-919. |
| [7] |
Daragó A, Szabó M, Hrács K, et al. In vitro investigations on the biological control of Xiphinema index with Trichoderma species[J]. Helminthologia, 2013, 50(2): 132-137. DOI:10.2478/s11687-013-0121-7 |
| [8] |
Affokpon A, Coyne DL, Htay CC, et al. Biocontrol potential of native Trichoderma isolates against root-knot nematodes in West African vegetable production systems[J]. Soil Biology and Biochemistry, 2011, 43(3): 600-608. DOI:10.1016/j.soilbio.2010.11.029 |
| [9] |
Szabó M, Urbán P, Virányi F, et al. Comparative gene expression profiles of Trichoderma harzianum proteases during in vitro nematode egg-parasitism[J]. Biological Control, 2013, 67(3): 337-343. DOI:10.1016/j.biocontrol.2013.09.002 |
| [10] |
Meyer SLF, Massoud SI, Chitwood DJ, et al. Evaluation of Trichoderma virens and Burkholderia cepacia for antagonistic activity against root-knot nematode, Meloidogyne incognita[J]. Nematology, 2000, 2(8): 871-879. DOI:10.1163/156854100750112815 |
| [11] |
Zhao HL, Qiao J, Meng QL, et al. Expression of serine proteinase P186 of Arthrobotrys oligospora and analysis of its nematode-degrading activity[J]. Antonie Van Leeuwenhoek, 2015, 108(6): 1485-1494. DOI:10.1007/s10482-015-0595-z |
| [12] |
Yan HB. Genome-wide analysis of proteases and inhibitors sequences identified through bioinformatics data mining in four tapeworms[D]. Beijing: Doctoral Dissertation of Chinese Academy of Agricultural Sciences, 2013 (in Chinese) 闫鸿斌.四种绦虫蛋白酶及其抑制剂的系统挖掘与功能分析[D].北京: 中国农业科学院博士学位论文, 2013 http://cdmd.cnki.com.cn/Article/CDMD-82101-1013357011.htm |
| [13] |
Wang SH, Wang WY, Huang YZ, et al. On serine protease[J]. Fujian Journal of Agricultural Sciences, 2007, 22(4): 453-456. (in Chinese) 汪世华, 王文勇, 黄益洲, 等. 丝氨酸蛋白酶研究进展[J]. 福建农业学报, 2007, 22(4): 453-456. DOI:10.3969/j.issn.1008-0384.2007.04.026 |
| [14] |
Ji XL, Sun ZR. Structure based evolution analysis of the serine protease superfamily[J]. Acta Electronica Sinica, 2001, 29(12A): 1756-1758. (in Chinese) 季星来, 孙之荣. 基于结构的丝氨酸蛋白酶超家族进化分析[J]. 电子学报, 2001, 29(12A): 1756-1758. |
| [15] |
Wang JW, Meng QL, Qiao J, et al. The recombinant serine protease XAoz1 of Arthrobotrys oligospora exhibits potent nematicidal activity against Caenorhabditis elegans and Haemonchus contortus[J]. FEMS Microbiology Letters, 2013, 344(1): 53-59. DOI:10.1111/1574-6968.12154 |
| [16] |
Zhao HL, Meng QL, Qiao J, et al. Cloning and bioinformatics analysis of associated gene of serine protease family of Arthrobotrys oligospora[J]. Southwest China Journal of Agricultural Sciences, 2015, 28(1): 414-418. (in Chinese) 赵海龙, 孟庆玲, 乔军, 等. 少孢节丛孢菌丝氨酸蛋白酶家族基因的克隆及生物信息学分析[J]. 西南农业学报, 2015, 28(1): 414-418. |
| [17] |
Wang RB, Yang J, Lin C, et al. Purification and characterization of an extracellular serine protease from the nematode-trapping fungus Dactylella shizishanna[J]. Letters in Applied Microbiology, 2006, 42(6): 589-594. |
| [18] |
Åhman J, Ek B, Rask L, et al. Sequence analysis and regulation of a gene encoding a cuticle-degrading serine protease from the nematophagous fungus Arthrobotrys oligospora[J]. Microbiology, 1996, 142(7): 1605-1616. DOI:10.1099/13500872-142-7-1605 |
| [19] |
Li YS. The molecular mechanism underlying resistance against entomopathogenic fungi by protease inhibitors in Bombyx mori[D]. Chongqing: Doctoral Dissertation of Southwest University, 2013 (in Chinese) 李游山.家蚕蛋白酶抑制剂抵御昆虫致病性真菌入侵的分子机理研究[D].重庆: 西南大学博士学位论文, 2013 http://cdmd.cnki.com.cn/Article/CDMD-10635-1013269990.htm |
| [20] |
Zhao ML. Study on gene cloning and expression of infectious extracellular proteases from nematophagous fungi[D]. Kunming: Doctoral Dissertation of Yunnan University, 2003 (in Chinese) 赵明莲.食线虫真菌侵染性胞外蛋白酶的基因克隆与表达[D].昆明: 云南大学博士学位论文, 2003 http://cdmd.cnki.com.cn/article/cdmd-10673-2006030838.htm |
| [21] |
Yang JK, Wang L, Ji XL, et al. Genomic and proteomic analyses of the fungus Arthrobotrys oligospora provide insights into nematode-trap formation[J]. PLoS Pathogens, 2011, 7(9): e1002179. DOI:10.1371/journal.ppat.1002179 |
| [22] |
Wang N. Heterogeneous expression of protease gene SS42 from Trichoderma atroviride and characterization of recombinant protein in biocontrol progress[D]. Harbin: Master's Thesis of Northeast Forestry University, 2014 (in Chinese) 王娜.深绿木霉蛋白酶基因SS42异源表达及重组蛋白生防特性分析[D].哈尔滨: 东北林业大学硕士学位论文, 2014 http://cdmd.cnki.com.cn/article/cdmd-10225-1014408987.htm |
| [23] |
Han HB. Induction of traps and analysis of serine protease gene sequence of Arthrobotrys oligospora[D]. Hohhot: Master's Thesis of Inner Mongolia Agricultural University, 2008 (in Chinese) 韩海宾.少孢节丛孢菌捕食器诱导及丝氨酸蛋白酶基因序列分析[D].呼和浩特: 内蒙古农业大学硕士学位论文, 2008 http://cdmd.cnki.com.cn/Article/CDMD-10129-2008132042.htm |
| [24] |
Chen LL, Liu LJ, Shi M, et al. Characterization and gene cloning of a novel serine protease with nematicidal activity from Trichoderma pseudokoningii SMF2[J]. FEMS Microbiology Letters, 2009, 299(2): 135-142. DOI:10.1111/j.1574-6968.2009.01746.x |
| [25] |
Yan L, Qian Y. Cloning and heterologous expression of SS10, a subtilisin-like protease displaying antifungal activity from Trichoderma harzianum[J]. FEMS Microbiology Letters, 2009, 290(1): 54-61. DOI:10.1111/j.1574-6968.2008.01403.x |
| [26] |
Liu ZH, Yang XX, Sun DM, et al. Expressed sequence tags-based identification of genes in a biocontrol strain Trichoderma asperellum[J]. Molecular Biology Reports, 2010, 37(8): 3673-3681. DOI:10.1007/s11033-010-0019-0 |
| [27] |
Vieira PM, Coelho AS, Steindorff AS, et al. Identification of differentially expressed genes from Trichoderma harzianum during growth on cell wall of Fusarium solani as a tool for biotechnological application[J]. BMC Genomics, 2013, 14: 177. DOI:10.1186/1471-2164-14-177 |
| [28] |
Liu Y, Yang Q. Cloning and heterologous expression of serine protease SL41 related to biocontrol in Trichoderma harzianum[J]. Journal of Molecular Microbiology and Biotechnology, 2013, 23(6): 431-439. DOI:10.1159/000346830 |
| [29] |
Yang JK, Liang LM, Li J, et al. Nematicidal enzymes from microorganisms and their applications[J]. Applied Microbiology and Biotechnology, 2013, 97(16): 7081-7095. DOI:10.1007/s00253-013-5045-0 |
| [30] |
Elad Y, Kapat A. The role of Trichoderma harzianum protease in the biocontrol of Botrytis cinerea[J]. European Journal of Plant Pathology, 1999, 105(2): 177-189. DOI:10.1023/A:1008753629207 |
| [31] |
Saeki K, Ozaki K, Kobayashi T, et al. Detergent alkaline proteases: enzymatic properties, genes, and crystal structures[J]. Journal of Bioscience and Bioengineering, 2007, 103(6): 501-508. DOI:10.1263/jbb.103.501 |
 2020, Vol. 47
2020, Vol. 47